Method Article
Marcatura terminale fluorescente e incapsulamento di RNA lunghi per microscopia FRET-TIRF a singola molecola
In questo articolo
Riepilogo
Questo articolo descrive la marcatura a due colori di RNA lunghi in posizione terminale e la loro immobilizzazione superficiale tramite incapsulamento in vescicole fosfolipidiche per applicazioni di microscopia FRET TIRF a singola molecola. La combinazione di queste tecniche consente una visualizzazione e un'analisi precise della dinamica dell'RNA a livello di singola molecola.
Abstract
Il trasferimento di energia di risonanza di Förster a singola molecola (smFRET) eccelle nello studio delle biomolecole dinamiche, consentendo un'osservazione precisa dei loro cambiamenti conformazionali nel tempo. Per monitorare la dinamica dell'RNA con smFRET, abbiamo sviluppato un metodo per marcare covalentemente gli RNA ai loro terminali con una coppia di fluorofori FRET. Questa strategia di marcatura diretta delle estremità ha come obiettivo il 5'-fosfato mediante attivazione della carbodiimmide (EDC)/N-idrossisuccinimide (NHS) e il 3'-ribosio mediante ossidazione del periodato, che possono essere adattati ad altri RNA indipendentemente dalle loro dimensioni e sequenza per studiarli indipendentemente dalle modifiche artificiali. Inoltre, l'attivazione del 5'-EDC/NHS è di interesse generale per tutti gli acidi nucleici con un 5'-fosfato. L'uso di sostanze chimiche disponibili in commercio elimina la necessità di sintetizzare sonde specifiche per RNA.
La microscopia a fluorescenza a riflessione interna totale (TIRF) richiede che le molecole di interesse immobilizzate in superficie si trovino all'interno del campo evanescente da illuminare. Un modo sofisticato per mantenere le molecole di RNA all'interno del campo evanescente è quello di incapsularle in vescicole fosfolipidiche. L'incapsulamento beneficia del meglio di entrambi i mondi, legando la molecola alla superficie e consentendo la libera diffusione della molecola. Ci assicuriamo che ogni vescicola contenga solo una singola molecola di RNA, consentendo l'imaging di una singola molecola. Dopo la marcatura dual-end e l'incapsulamento dell'RNA di interesse, le misure smFRET offrono una visione dinamica e dettagliata del comportamento dell'RNA.
Introduzione
Il trasferimento di energia di risonanza di Förster (FRET) è una tecnica potente e sensibile per lo studio delle interazioni inter- e intramolecolari di biomolecole su scala nanometrica. Si basa sul trasferimento di energia non radiativa da una molecola donatrice eccitata a una molecola accettore vicina, che avviene su distanze tipicamente comprese tra 1 nm e 10 nm. La distanza tra il colorante donatore e quello accettore determina l'efficienza di questo trasferimento di energia, rendendo FRET uno strumento prezioso per lo studio delle dinamiche molecolari, dei cambiamenti conformazionali e delle interazioni in un'ampia gamma di sistemi biologici 1,2, compresi gli RNA. La microscopia a fluorescenza a riflessione interna totale (TIRF) ha dimostrato di essere una tecnica potente per le indagini smFRET, in quanto illumina selettivamente le molecole solo vicino alla superficie, consentendo la dinamica FRET delle singole molecole con un'elevata risoluzione spaziale e temporale. Tuttavia, prima di eseguire esperimenti smFRET-TIRF, la molecola di interesse deve essere prima marcata in fluorescenza con un'appropriata coppia FRET e quindi immobilizzata sulla superficie del microscopio. Il protocollo smFRET-TIRF qui descritto è stato convalidato utilizzando l'introne Sc.ai5γ di gruppo II wild-type dai mitocondri di Saccharomyces cerevisiae, affiancato dalle sue due sequenze esoniche (915 nucleotidi)3. Per una visione più dettagliata degli introni del gruppo II di marcatura fluorescente e della loro immobilizzazione per la microscopia smFRET TIRF, fare riferimento alla nostra recensione4.
Una strategia ideale di marcatura dell'RNA sito-specifica consentirebbe l'incorporazione precisa dei coloranti donatori e accettori in posizioni predeterminate senza alterare la struttura o la funzione dell'RNA, garantendo misure FRET accurate ed efficienti. Questo è difficile a causa delle somiglianze chimiche tra le quattro basi nucleotidiche, che complicano l'etichettatura selettiva. La marcatura delle estremità lega i coloranti donatore e accettore alle estremità dell'RNA prendendo di mira il 5'-fosfato e il 3'-ribosio. Questo approccio offre un approccio minimamente invasivo, pur fornendo preziose informazioni sulle dinamiche strutturali e sulle interazioni. La capacità dell'introne del gruppo II di auto-splicare in presenza di Mg2+ limita l'uso di enzimi metallo-ioni-dipendenti. Qui, presentiamo un approccio a RNA a doppia marcatura finale lunghi e cataliticamente attivi (ribozimi) che aggirano la necessità di enzimi o la sintesi di sonde specializzate.
Un approccio comune per legare le molecole di RNA alla superficie per la microscopia TIRF consiste nel legare covalentemente una porzione di biotina direttamente all'RNA o nell'ibridare un oligonucleotide antisenso (ASO) che trasporta biotina5,6,7. Tuttavia, questo metodo di immobilizzazione diretta può introdurre artefatti dovuti alle interazioni RNA-superficie, con conseguente potenziale ripiegamento errato di RNA8. Una soluzione elegante per mitigare questi artefatti di immobilizzazione è quella di incapsulare l'RNA in vescicole fosfolipidiche su scala nanometricaattaccate alla superficie 9,10,11. Queste vescicole, che hanno un diametro di circa 100 nm, sono ancorate alla superficie attraverso un legame biotina-streptavidina12,13,14, consentendo all'RNA di diffondersi liberamente all'interno e consentendo lo scambio di ioni attraverso la membrana lipidica10. Dopo aver marcato covalentemente un grande RNAfunzionale 3, presentiamo un approccio per incapsulare tali RNA in vescicole fosfolipidiche combinando protocolli consolidati per la passivazione superficiale e l'incapsulamento delle vescicole, adattati per preservare la funzionalità dell'RNA 10,11,14. Questo approccio dual-end di marcatura e incapsulamento consente di ottenere un alto tasso di mono-incapsulamento di RNA funzionali per la microscopia smFRET TIRF.
Protocollo
1. Marcatura a doppia estremità dell'RNA
NOTA: Il seguente protocollo descrive la marcatura sito-specifica di RNA con una coppia di fluorofori FRET mediante l'attaccamento covalente di un colorante donatore (sCy3) al 5'-fosfato e di un colorante accettore (Cy5) al 3'-ribosio. Un RNA lungo cataliticamente attivo, l'introne ribozima del gruppo II, viene scelto come RNA di interesse. La Tabella 1 e la Figura 1 riassumono questo protocollo di etichettatura dual-end. Eseguire tutti i passaggi che coinvolgono i fluorofori in condizioni di oscurità.
| Giorno 0 | ▪ Aliquotare 50-75 μg di RNA per un volume totale di 55 μl per provetta da 1,5 mL. | |||||
| Giorno 1 | Attivazione del 5′-fosfato | |||||
| ▪ Aggiungere 45 μL di soluzione EDC-NHS, pH 6.0 appena preparata all'RNA in ddH2O fino a un volume finale di 100 μL, mescolare bene e incubare per 4 ore a 25 °C e 500 giri/min. | ||||||
| ▪ Ciclo di purificazione 1: precipitazione notturna di EtOH. | ||||||
| Giorno 2 | ▪ Far precipitare l'RNA attivato dal 5', lavare e asciugare. | |||||
| 5′-Attacco per colorante | ||||||
| ▪ Risospendere in 95 μl di 100 mM MOPS, pH 7,5. | ||||||
| ▪ Aggiungere 5 μl di soluzione colorante funzionalizzata con ammina 2 mM. | ||||||
| ▪ Mescolare bene e incubare per 16 ore a 25 °C e 500 giri/min. | ||||||
| Giorno 3 | ▪ Ciclo di purificazione 2: precipitazione di EtOH. | |||||
| Giorno 4 | ▪ Far precipitare l'RNA attivato dal 5', lavare e asciugare. | |||||
| Passaggio di blocco | ||||||
| ▪ Risospendere in 100 μl di 100 mM Tris-HCl, pH 7,5 e incubare per 2 ore a 25 °C e 500 giri/min. | ||||||
| ▪ Ciclo di purificazione 3: filtrazione centrifuga. | ||||||
| → Eluire l'RNA marcato con 5'. | ||||||
| Giorno 5 | Ossidazione 3′-periodato | |||||
| ▪ Incubare l'RNA con 20 mM di NaIO4 in 50 mM di tampone NaOAc, pH 5,5 in un volume finale di 100 μL per 2 ore a 25 °C e 500 giri/min. | ||||||
| ▪ Spegnere il periodato in eccesso: aggiungere 30 μl di glicerolo al 50%, mescolare bene e incubare per 30 minuti a 25 °C e 500 giri/min. | ||||||
| ▪ Ciclo di purificazione 4: Precipitazione notturna di EtOH. | ||||||
| Giorno 6 | ▪ Far precipitare l'RNA ossidato 3', lavare e asciugare. | |||||
| Attacco 3′-Dye | ||||||
| ▪ Risospendere in 95 μL di 50 mM di NaOAc, pH 6,0. | ||||||
| ▪ Aggiungere 5 μl di soluzione colorante funzionalizzata con idrazide 2 mM. Mescolare bene e incubare per 16 ore a 25 °C e 500 giri/min. | ||||||
| Giorno 7 | ▪ Ciclo di purificazione 5: precipitazione EtOH. | |||||
| Giorno 8 | ▪ Precipitare l'RNA marcato, lavare e asciugare. | |||||
| ▪ Filtrazione centrifuga. | ||||||
| → Eluire l'RNA marcato a doppia estremità. | ||||||
Tabella 1: Riepilogo del protocollo per la marcatura dual-end dell'RNA. Clicca qui per scaricare questa tabella.
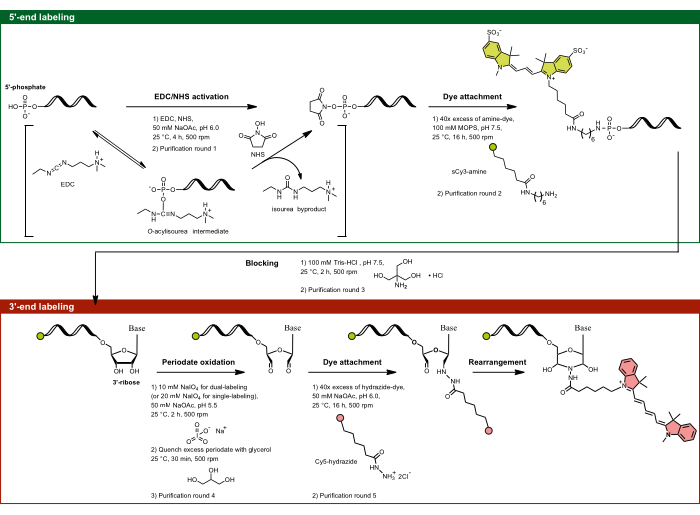
Figura 1: Il flusso sperimentale della marcatura dual-end mirando allo zucchero 5'-fosfato e allo zucchero 3'-ribosio. Il 5'-fosfato viene attivato mediante EDC in presenza di NHS e successivamente viene accoppiato con il colorante funzionalizzato con ammina. La porzione 3'-diolo dell'RNA viene ossidata dall'attività periodica a dialdeide, che reagisce ulteriormente con il colorante funzionalizzato con idrazide. Per l'etichettatura a doppia estremità, è importante iniziare con l'etichettatura a 5' per evitare l'etichettatura incrociata, seguita dall'etichettatura a 3' con un passaggio di blocco intermedio. Abbreviazioni: EDC = carbodiimmide; NHS = N-idrossisuccinimide; MOPS = acido 3-morfolino-propano-1-solfonico; NaOAc = acetato di sodio. Clicca qui per visualizzare una versione più grande di questa figura.
- Marcatura dell'estremità 5': Targeting del 5'-fosfato mediante attivazione EDC/NHS
NOTA: Questo metodo di marcatura 5' si applica non solo all'RNA, ma a qualsiasi acido nucleico a filamento singolo contenente un 5'-fosfato. La specificità della reazione è governata dalla dipendenza dal pH, consentendo solo al 5'-fosfato di essere mirato in modo sito-specifico, nonostante la presenza di più fosfati nella spina dorsale dell'RNA. A pH 6,0, la reattività unica del 5'-fosfato nei confronti delle carbodiimmidi è dovuta al suo specifico stato di protonazione, in cui due ossigeni vengono deprotonati e uno rimane protonato. Ciò rende il 5'-fosfato reattivo, mentre i fosfati della spina dorsale completamente deprotonati rimangono non reattivi, consentendo la marcatura selettiva dell'estremità 5' tramite il targeting EDC/NHS.- Preparare la soluzione EDC-NHS-NaOAc, pH 6.0. Miscelare 1,5 mg di EDC e 2,0 mg di NHS per aliquota in 35 μL di ddH2O e 10 μL di 0,5 M NaOAc, pH 6,0 (pH aggiustato con acido acetico glaciale).
- Aliquotare l'RNA di interesse per avere circa 50-75 μg di RNA per provetta in 55 μL di ddH2O. Aggiungere 45 μL della miscela EDC-NHS-NaOAc, pH 6.0 all'RNA, raggiungendo un volume totale di 100 μL con le concentrazioni finali di 78 mM EDC, 174 mM NHS e 50 mM NaOAc, pH 6.0. Incubare per 4 ore a 25 °C agitando a 500 giri/min.
NOTA: Qui, 70 μg corrispondono a 250 pmol di un RNA 915 nt trascritto in vitro e 2,5 μM nel volume di reazione finale di 100 μL. - Ciclo di purificazione 1: purificare l'RNA attivato dal 5'-fosfato mediante precipitazione di EtOH.
- Preparare una soluzione di cianina 3 ammina solfonata (sCy3-ammina) da 2 mM in acqua.
- Risospendere il pellet di RNA attivato in 95 μL di tampone 100 mM di acido 3-morfolinopropano-1-solfonico (MOPS), pH 7,5 (pH regolato con NaOH). Aggiungere 5 μL di soluzione di 2 mM di sCy3-ammina all'RNA attivato. Accoppiare il fluoroforo al 5'-fosfato attivato mediante incubazione per 16 ore a 25 °C, agitando a 500 giri/min.
NOTA: Conservare il tampone MOPS a 4 °C al buio. Il NaOH viene scelto per la regolazione del pH per mantenere inattivo l'introne ribozima del gruppo II. Il fluoroforo dovrebbe essere almeno 40 volte superiore all'RNA. - Ciclo di purificazione 2: Aumentare il volume aggiungendo 200 μL di ddH2O per migliorare la separazione. Purificare l'RNA marcato con 5'-fosfato mediante precipitazione EtOH e ripetere fino a quando il surnatante è incolore (di solito sono necessari due giri).
- Fase di blocco: risospendere l'RNA in 100 μL di Tris-HCl 100 mM, pH 7,5 e incubare per 2 ore a 25 °C e 500 giri/min. Solo per l'etichettatura singola con estremità 5', saltare la fase di blocco, risospendere l'RNA in ddH2O e procedere con il passaggio 1.1.8. Tuttavia, per semplicità, se si esegue l'etichettatura doppia, separare il controllo con etichetta singola a 5' solo dopo il passaggio 1.1.9.
NOTA: Questo passaggio serve a bloccare i 5'-fosfati attivati che non sono stati accoppiati a un fluoroforo funzionalizzato con ammina reagendo con una fonte di ammina primaria relativamente più piccola (ad esempio, Tris) per ridurre al minimo il rischio di marcatura incrociata con il fluoroforo funzionalizzato con idrazide utilizzato per il protocollo di marcatura dell'estremità 3'. - Ciclo di purificazione 3: Rimuovere i coloranti liberi lavando l'RNA marcato mediante filtrazione centrifuga con un totale di almeno 10 mL di ddH2O, quindi eluire in ddH2O.
NOTA: Il taglio molecolare del filtro deve essere inferiore alla metà delle dimensioni dell'acido nucleico. Il tampone di scelta può essere utilizzato al posto di ddH2O. L'eluizione deve seguire le istruzioni del produttore, assicurandosi che il campione non venga centrifugato fino alla completa secchezza. - Determinare le concentrazioni di RNA e colorante coniugato mediante spettroscopia UV-Vis.
- Marcatura dell'estremità 3': Targeting del 3'-ribosio mediante ossidazione periodato
- Aliquotare l'RNA marcato con l'estremità 5' in ddH2O per avere circa 50-75 μg di RNA per provetta in 90 μL. Se il volume di eluizione della fase precedente era elevato, risultando in una bassa concentrazione, concentrare l'RNA precipitando e risospendendo il pellet in ddH2O. Aggiungere 5 μL di tampone NaOAc 1,0 M, pH 5,5 (corrispondente a 50 mM NaOAc, pH 5,5 per un volume di reazione di 100 μL).
NOTA: Simile all'RNA marcato singolo con estremità 5', prepariamo abitualmente RNA marcato singolo con estremità 3' come controllo. Per fare ciò, aliquotare l'RNA non marcato in modo che abbia circa 50-75 μg di RNA per provetta in 86 μL di ddH2O. - Aggiungere 4 μL di soluzione madre di metaperiodato di sodio (NaIO4) da 500 mM appena preparata (corrispondenti a 10 mM di NaIO4 per un volume di reazione di 100 μl). Per la marcatura singola con estremità 3', aggiungere 8 μL di soluzione madre NaIO4 (corrispondenti a 20 mM NaIO4 per un volume di reazione di 100 μL). Mescolare accuratamente e incubare per 2 ore a 25 °C agitando a 500 giri/min in condizioni di oscurità poiché NaIO4 è sensibile alla luce.
NOTA: Non modificare l'ordine di aggiunta o aumentare ulteriormente il tempo di incubazione, poiché ciò potrebbe causare il fotosbiancamento del colorante donatore attaccato. La concentrazione di NaIO4 utilizzata è diminuita della metà per l'etichettatura dual-end per ridurre al minimo la tempra del colorante già attaccato all'estremità 5'. - Arrestare la reazione aggiungendo 30 μl di glicerolo al 50%. Incubare per 30 minuti a 25 °C agitando a 500 giri/min.
NOTA: Il glicerolo funge da diolo per estinguere l'eccesso di periodato. - Ciclo di purificazione 4: aggiungere 400 μl di miscela di precipitazione EtOH-NaOAc ghiacciata ed eseguire la precipitazione con etanolo.
- Risospendere il pellet di RNA ossidato in 95 μL di 50 mM di NaOAc, pH 6,0.
- Attacco idrazide-colorante: Preparare la soluzione fluoroforica sciogliendo alcuni cristalli di idrazide cianina5 (Cy5-idrazide) in DMSO e quindi diluendo con ddH2O alla concentrazione di 2 mM. Se il fluoroforo scelto è solubile in acqua, preparare la soluzione in ddH2O. Aggiungere 5 μL di soluzione di Cy5-idrazide 2 mM all'RNA ossidato. Mescolare bene e incubare per 16 ore a 25 °C agitando a 500 giri/min.
NOTA: Il fluoroforo deve essere almeno 40 volte superiore all'RNA. - Ciclo di purificazione 5: purificare l'RNA marcato a doppia estremità (o RNA marcato a singola estremità a 3' estremità) mediante precipitazione EtOH e filtrazione centrifuga, simile ai passaggi 1.2.6 e 1.2.8, rispettivamente, ed eluire in ddH2O.
- Calcolare le concentrazioni di RNA e colorante coniugato mediante spettroscopia UV-Vis.
- Caratterizzare l'RNA marcato utilizzando saggi analitici basati su gel, spettroscopia di fluorescenza d'insieme (vedere la sezione Risultati rappresentativi ) e/o HPLC analitica come mostrato altrove3.
- Aliquotare l'RNA marcato con l'estremità 5' in ddH2O per avere circa 50-75 μg di RNA per provetta in 90 μL. Se il volume di eluizione della fase precedente era elevato, risultando in una bassa concentrazione, concentrare l'RNA precipitando e risospendendo il pellet in ddH2O. Aggiungere 5 μL di tampone NaOAc 1,0 M, pH 5,5 (corrispondente a 50 mM NaOAc, pH 5,5 per un volume di reazione di 100 μL).
2. Preparazione della camera microfluidica
NOTA: Si consiglia di maneggiare sei o otto camere alla volta. La sonicazione viene eseguita a temperatura ambiente, salvo diversa indicazione. Questo protocollo limita l'uso di solventi organici, come l'acetone, per prevenire la solubilizzazione delle impurità in tracce. Per le alternative, fare riferimento ai riferimenti15,16.
- Pulitura
- Praticare quattro fori nelle slitte di quarzo con un trapano diamantato secondo lo schema indicato nella Figura 2A per formare due canali.
NOTA: Sebbene possano rompersi più facilmente durante la perforazione, i vetrini sono un'alternativa economica ai vetrini al quarzo per le configurazioni di microscopia TIRF basate su obiettivi in cui il vetrino coprioggetti è la superficie di imaging (Figura 2B). Questa non è un'opzione per le configurazioni TIRF basate su prismi, in cui il vetrino per microscopia è la superficie di imaging a causa della fluorescenza di fondo.
NOTA: In alternativa, riciclare le camere microfluidiche usate16. Per questo, immergere le camere usate per una notte in acetone (avvolgere il barattolo di colorazione Coplin in un foglio di alluminio per ridurre al minimo l'evaporazione) nella cappa aspirante e successivamente sonicare per 5 minuti. Smontare la camera e gettare i vetrini coprioggetti e gli adesivi. Se lo smontaggio non funziona immediatamente, sonicare la camera in acetone per altri 10 minuti. Procedere con il passaggio 2.1.2. - Posizionare i vetrini di quarzo forati e circa il doppio dei vetrini coprioggetti (poiché potrebbero rompersi facilmente) in un barattolo di vetro Coplin con coperchio. Risciacquare 3 volte con ddH2O. Sonicare in ddH2O per 5 minuti, quindi risciacquare 3 volte con ddH2O.
- Sonicare in una soluzione detergente per vetreria da laboratorio al 10% (vedere la tabella dei materiali) per 30 minuti a 50 °C. Risciacquare almeno 3 volte con ddH2O fino a quando le bolle di detersivo non sono sparite. Sonicare in ddH2O per 5 min. Risciacquare 3 volte con ddH2O.
- Sonicare in soluzione 1 M KOH per 30 minuti, quindi lasciare riposare per una notte. Risciacquare 3 volte con ddH2O. Sonicare in ddH2O per 5 min. Risciacquare 3 volte con ddH2O.
NOTA: Sebbene la corrosione dovuta a un'eccessiva incubazione sia una preoccupazione precedentemente sollevata da Chandradoss et al.15, raccomandiamo questa lunga incubazione che consente di evitare l'incisione dei Piranha. - Asciugare i vetrini e i vetrini coprioggetti con N2(g).
- Trattare i vetrini e i vetrini coprioggetti asciutti con un detergente al plasma all'ossigeno per 30 minuti secondo le istruzioni del produttore.
- Praticare quattro fori nelle slitte di quarzo con un trapano diamantato secondo lo schema indicato nella Figura 2A per formare due canali.
- Aminosilianizzazione
- Preparare la soluzione APTES-EtOH al 3% mescolando accuratamente 288,5 mL di EtOH assoluto, 1,5 mL di ddH2O e 9 mL di (3-amminopropil)trietossisilano (APTES) in un pallone di Erlenmeyer da 500 mL.
- Collocare i vetrini e i vetrini coprioggetti puliti in un barattolo di colorazione Coplin, immergerli nella soluzione APTES-EtOH al 3%, sonicare per 1 minuto e incubare per 30 minuti.
- Risciacquare 3 volte con EtOH assoluto, quindi 3 volte con ddH2O.
- Asciugare i vetrini e i vetrini coprioggetti sotto il flusso N2(g).
- Passivazione superficiale e biotinilazione
- Preparare una scatola umida riempiendo una scatola di puntali per pipette vuota fino alla metà con ddH2O. Posizionare i vetrini all'interno della scatola, con il lato da trattare rivolto verso l'alto.
- Preparare la miscela bPEG-mPEG in una provetta da microcentrifuga sterile da 1,5 mL mescolando delicatamente 2 mg di biotina-polietilenglicole-succinimidil valerato 5000 (biotina-PEG-SVA) e 80 mg di metossi polietilenglicole-succinimidil valerato (mPEG-SVA) in 640 μL di tampone 100 mM di bicarbonato di sodio (NaHCO3), pH 8,3. Centrifugare la miscela bPEG-mPEG a 16.000 × g per 1 minuto per eliminare eventuali bolle d'aria.
- Rimuovere con cautela il surnatante e aggiungere una goccia da 30 μl al centro del vetrino per coprire entrambi i canali. Posiziona una gocciolina aggiuntiva su un lato del vetrino per avere un vetrino coprioggetti PEGilato extra, poiché si rompono facilmente e avere un ricambio è utile. Infine, coprire la goccia con un vetrino coprioggetti pulito, chiudere la scatola umida ed eseguire la PEGilazione durante la notte in condizioni di oscurità.
- Sciacquare accuratamente i vetrini e i vetrini coprioggetti passivati con PEG e biotinilati con ddH2O. Notare il cambiamento nell'idrofobicità delle superfici trattate. Asciugare con flusso N2(g).
- Assemblaggio della camera microfluidica
- Taglia un adesivo biadesivo per creare i canali. Attacca l'adesivo alla diapositiva, assicurandoti che copra l'area di interesse. Posizionare con cura il vetrino coprioggetti sulla parte superiore, allineandolo in modo che le superfici PEGilate siano rivolte l'una verso l'altra.
- Posizionare ciascuna camera assemblata in una provetta da centrifuga da 50 mL e riempire le provette con N2(g) e conservare a -20 °C per un massimo di 1 mese.
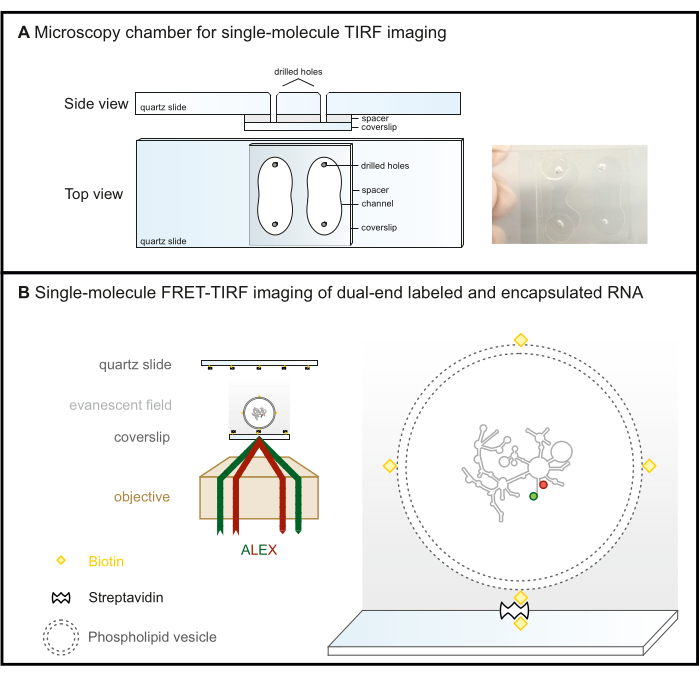
Figura 2: Microscopia FRET-TIRF a singola molecola. (A) Camera microfluidica per imaging TIRF. (B) L'RNA marcato con FRET è incapsulato in una vescicola fosfolipidica biotinilata e immobilizzato su una superficie di vetro rivestita di steptavidina. Ciò mantiene la molecola di interesse all'interno del campo evanescente (gradiente di grigio), che viene creato dalla luce incidente che viene totalmente riflessa all'angolo critico nella microscopia TIRF. Qui, entrambi i fluorofori vengono successivamente eccitati con lo schema ALEX. Abbreviazioni: FRET = Trasferimento di energia di risonanza di Förster; TIRF = fluorescenza totale a riflessione interna; ALEX = eccitazione laser alternativa. Clicca qui per visualizzare una versione più grande di questa figura.
3. Incapsulamento delle vescicole fosfolipidiche
- Preparazione del panello lipidico
NOTA: Maneggiare sempre il cloroformio sotto una cappa aspirante.- Utilizzando un ago sterile, praticare dei fori nel coperchio (dall'interno verso l'esterno) di provette per microcentrifuga sterili da 2,0 ml, come mostrato nella Figura 3A.
- Preparare la soluzione madre di 1,2-dipalmitoil-sn-glicero-3-fosfoetanolammina-N-cap biotinile (bPE) sciogliendo almeno 2 mg di bPE nella quantità appropriata di cloroformio fino a una concentrazione finale di 1 mg/mL.
- Preparare la soluzione madre di 1,2-dimiristoil-sn-glicero-3-fosfocolina (DMPC) sciogliendo almeno 10 mg di DMPC nella quantità appropriata di cloroformio fino a una concentrazione finale di 10 mg/mL.
- Preparare la miscela bPE-DMPC (mantenendo il rapporto 1:99 p/p) mescolando 100 μL di bPE e 990 μL di soluzioni madre DMPC. Distribuire 109 μl della miscela bPE-DMPC per provetta da 2 mL.
- Posizionare un inserto divisorio di cartone di una scatola di stoccaggio per microprovette in un pallone Schlenk, che funge da supporto per provette. Utilizzando una pinzetta lunga, posizionare le provette contenenti la miscela lipidica nel supporto della provetta in un pallone Schlenk da 500 ml (Figura 3B).
NOTA: Assicurarsi che siano in posizione verticale e non inclinati. - Far evaporare il cloroformio a basso flusso di N2(g) per una notte (o per almeno 2 ore fino a quando il solvente non è completamente evaporato).
- Sigillare i tubi con Parafilm per coprire i fori. Conservare i panetti lipidici a -20 °C per un massimo di 1 mese.
- Incapsulamento dell'RNA
- Preparare i seguenti tamponi e soluzioni regolando le concentrazioni di K+ e Mg2+ in base al sistema biologico di interesse.
- Preparare il buffer standard 5x (5x SB): 2,5 M KCl, 400 mM MOPS; regolare il pH a 6,9 con KOH, filtro sterile e conservare a 4 °C al buio (rispettivamente 1x SB: 500 mM KCl, 80 mM MOPS, pH 6,9).
- Preparare il tampone antilampeggio (AB): 100 mM di MgCl2, Trolox, 1x SB, filtro sterile e conservare a 4 °C al buio per un massimo di 1 settimana.
NOTA: Utilizzare la punta di una spatola di Trolox per un volume finale di 10 ml, agitare per mescolare e regolare nuovamente il pH.
- Assemblare l'estrusore utilizzando una membrana in policarbonato (PC) da 100 nm e un disco di drenaggio in poliestere (PE) da 10 mm, equilibrare la siringa e le membrane con AB e riscaldare a 30 °C (o essenzialmente al di sopra della temperatura di transizione vetrosa DMPC di 24 °C).
- Miscelare 5 μL di RNA marcato a doppia estremità da 1 μM e 45 μL di AB (per un volume totale di 50 μL).
- Idratare la torta lipidica con questa soluzione di RNA. A 30 °C, mescolare per 5 minuti agitando a 1.400 giri/min, quindi per 15 minuti a 700 giri/min. Centrifugare per 2 minuti a 13.000 g e trasferire con cura il surnatante in una nuova provetta.
- Diluire il campione con 250 μl di AB.
- Riempire la siringa con la sospensione di RNA-lipidi. Estrudere 35x a 30 °C su un blocco riscaldante per incapsulare l'RNA marcato a doppia estremità in vescicole fosfolipidiche di 100 nm di diametro.
NOTA: La distribuzione dimensionale delle vescicole dopo l'utilizzo dell'estrusore può essere convalidata mediante diffusione dinamica della luce (DLS), come mostrato nel riferimento11.
- Preparare i seguenti tamponi e soluzioni regolando le concentrazioni di K+ e Mg2+ in base al sistema biologico di interesse.

Figura 3: Preparazione del panello lipidico. (A) I tappi delle provette vengono perforati per consentire l'evaporazione del solvente. (B) I tubi contenenti la miscela lipidica vengono posti in un pallone di Schlenk e il cloroformio viene evaporato per ottenere un panello lipidico. Clicca qui per visualizzare una versione più grande di questa figura.
4. microscopia smFRET-TIRF
- Immobilizzazione superficiale
- Preparare i seguenti tamponi e soluzioni.
- Preparare il tampone anti-zucchero (SAB): 1% di D-glucosio (p/v) in AB, filtro sterile, e conservare a 4 °C al buio per un massimo di 1 settimana.
- Preparare il 100x OSS: 100x Sistema di evacuazione dell'ossigeno (OSS): 3 mg di glucosio ossidasi (corrispondente a 1,7 U), 10 μL di catalasi (corrispondente a 22 U) in 90 μL di 1x SB, conservare a 4 °C al buio per un massimo di 1 settimana.
- Preparare nuovamente il tampone di imaging (IB) mescolando 198 μL di SAB e 2 μL di soluzione OSS 100x.
- Lasciare che la camera microfluidica raggiunga la temperatura ambiente. Lavare la camera 2x con 200 μl di 1x SB.
- Riempire la camera con 50 μL di soluzione di streptavidina da 20 μL/mL e incubare per 5 minuti.
- Lavare la camera con 200 μl di 1x SB e poi con 100 μl di AB.
- Immobilizzare l'RNA incapsulato sulla superficie aggiungendo 75 μL di sospensione vescicola e incubare per 10 minuti.
- Lavare la camera con 200 μL di IB appena preparato e incubare per 5 minuti.
- La camera è ora pronta per l'acquisizione dei dati (Figura 2B). Per evitare l'evaporazione durante le misurazioni lunghe, sigillare i fori come mostrato nella Figura 2A. Se le perdite da un canale all'altro destano preoccupazione, sigillare il canale inutilizzato prima di iniziare il protocollo di immobilizzazione della superficie.
NOTA: Si consiglia di eseguire misurazioni in bianco come controllo di qualità per la pulizia delle camere microfluidiche e delle vescicole fosfolipidiche contro le contaminazioni da fluorescenza.
- Preparare i seguenti tamponi e soluzioni.
Risultati
Presentiamo la marcatura sito-specifica a singola e doppia fluorescenza dell'RNA 915-nt di interesse, l'introne mitocondriale di lievito Sc.ai5γ gruppo II, affiancato da sequenze esoniche. La coppia di fluorofori FRET è posizionata alle estremità dell'RNA tramite l'attivazione EDC/NHS del 5'-fosfato e l'ossidazione periodica del 3'-ribosio, seguita dai rispettivi attacchi coloranti. Abbiamo quindi verificato i coniugati RNA-colorante mediante elettroforesi su gel fluorescente, come presentato nella Figura 4A. La co-migrazione dell'RNA e dei fluorofori sul gel di agarosio conferma il successo dell'etichettatura. Successivamente, come mostrato nella Figura 4B, è stata utilizzata la spettroscopia di fluorescenza d'insieme per caratterizzare l'introne del gruppo II a doppia marcatura. Il trasferimento di energia, cioè FRET, è stato osservato dopo l'eccitazione del colorante donatore dimostrando la doppia marcatura dell'RNA. In particolare, coerentemente con la natura dipendente dalla distanza di FRET, il ripiegamento dell'RNA intronico del gruppo II in presenza di ioni metallici ha portato ad un aumento dell'efficienza di FRET, come evidenziato dalla diminuzione dell'emissione del donatore (freccia verde) e da un corrispondente aumento dell'emissione dell'accettore (freccia rossa). Ciò indica che questa marcatura FRET tiene traccia dei cambiamenti conformazionali del ribozima.
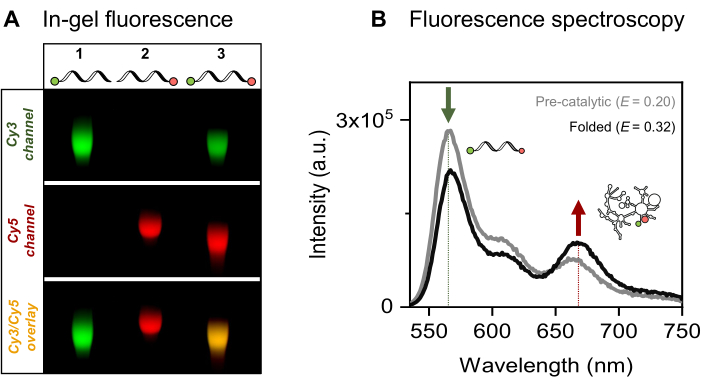
Figura 4: Caratterizzazione dei coniugati RNA-colorante. (A) L'analisi analitica basata su gel di RNA marcato a fluorescenza singola e doppia mostra la co-migrazione dei coloranti con l'RNA su un gel di agarosio al 2%. La co-localizzazione dei fluorofori nel campione a doppia marcatura è indicata dalla banda gialla nell'immagine unita (in basso) dei canali Cy3 (in alto, verde) e Cy5 (in centro, in rosso), visualizzati rispettivamente con un'illuminazione di 532 nm e 635 nm. Corsia 1: 5'-sCy3 solo RNA marcato, corsia 2: 3'-Cy5 solo RNA marcato e corsia 3: RNA marcato a doppia estremità (5'-sCy3 e 3'-Cy5). (B) La spettroscopia di fluorescenza d'insieme conferma la doppia marcatura. Il trasferimento di energia all'eccitazione del donatore (λex = 515 nm, λem = 670 nm) verifica che entrambi i coloranti siano accoppiati con successo all'RNA. La curva grigia rappresenta il profilo di emissione dell'RNA pre-catalitico, mentre la curva nera dimostra un aumento dell'efficienza FRET nell'RNA intronico del gruppo II ripiegato (incubato con 500 mM di KCl a 70 °C per 3 minuti, raffreddato a 42 °C per 5 minuti, seguito dall'aggiunta di 100 mM di MgCl2). Questa figura è stata adattata da Ahunbay et al.3 Fare clic qui per visualizzare una versione più grande di questa figura.
Con in mano l'introne del gruppo II wild-type marcato in modo fluorescente, siamo ora in grado di esplorare le sue dinamiche a livello di singola molecola. Una volta incapsulato in vescicole fosfolipidiche, l'RNA marcato viene immobilizzato su una superficie di microscopia a densità superficiali molto basse per ottenere la risoluzione di una singola molecola per smFRET-TIRF. Come si vede nella Figura 5A, diverse singole molecole possono essere tracciate contemporaneamente. La microscopia TIRF consente il monitoraggio in tempo reale dell'efficienza FRET e delle sue variazioni nel tempo. La Figura 5B esemplifica le tracce FRET statiche e dinamiche a singola molecola dell'RNA marcato e incapsulato. Una tipica traccia dinamica mostra un'anticorrelazione tra i segnali del donatore e dell'accettore che fluttuano l'efficienza del FRET. Quando l'emissione dell'accettore all'aumento dell'eccitazione del donatore, l'emissione del donatore diminuisce di conseguenza, indicando un cambiamento dinamico nella distanza tra i coloranti. Questa anticorrelazione suggerisce cambiamenti conformazionali nella molecola di RNA.
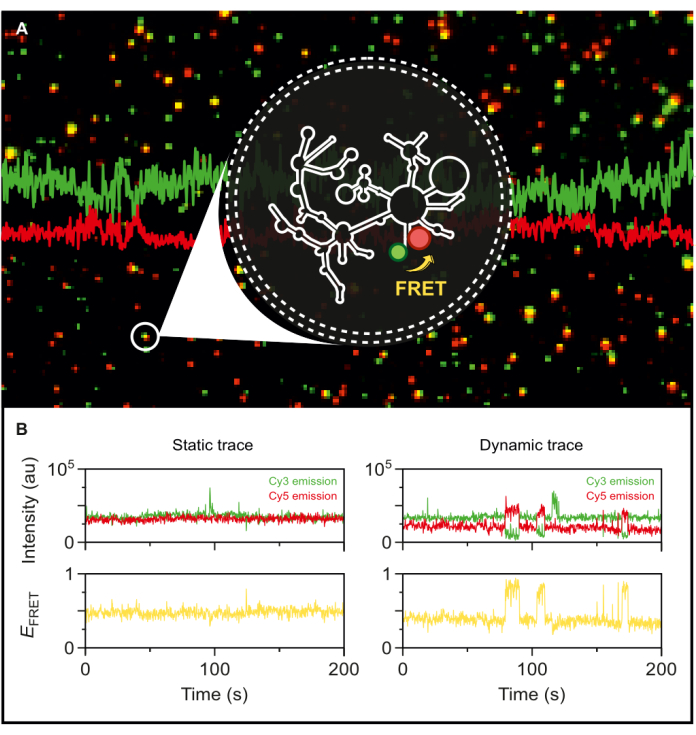
Figura 5: Comportamento altamente dinamico dell'RNA intronico del gruppo II rivelato da smFRET. L'RNA dell'introne del gruppo II marcato a doppia estremità viene incapsulato in una vescicola fosfolipidica e immobilizzato sulla superficie per l'imaging con un microscopio TIRF basato su obiettivo. (A) Immagine unita di singole molecole di RNA marcate che esibiscono emissione donatrice (sCy3, verde) ed emissione accettore (Cy5, rosso) dopo eccitazione a 532 nm. (B) Le traiettorie tipiche di smFRET di (a sinistra) di una molecola di RNA statica in cui le intensità del donatore e dell'accettore non fluttuano nel tempo e (a destra) di una molecola di RNA dinamica, in cui le intensità del donatore e dell'accettore sono anticorrelate, con le efficienze FRET in giallo. Le singole molecole vengono localizzate e analizzate utilizzando MASH-FRET17. Vengono applicati l'eccitazione diretta, il sanguinamento e le correzioni dei fattori γ. Abbreviazioni: smFRET = trasferimento di energia di risonanza di Förster a molecola singola. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
La FRET a livello di singola molecola è unica perché consente l'osservazione e l'analisi di singole molecole, rivelando l'eterogeneità del campione e catturando stati transitori che possono essere oscurati nelle misure d'insieme 1,2. L'osservazione delle singole molecole di RNA utilizzando smFRET fornisce informazioni ad alta risoluzione sui loro percorsi di ripiegamento e sulle loro dinamiche. Questo protocollo descrive la marcatura chimica dual-end dell'RNA e la sua immobilizzazione superficiale tramite incapsulamento di vescicole fosfolipidiche, che insieme consentono di seguire cambiamenti conformazionali dinamici tramite microscopia smFRET-TIRF.
Lo studio della dinamica dell'RNA è un campo in costante crescita con la necessità di nuove strategie di marcatura fluorescente sito-specifiche. Etichettiamo le estremità dell'RNA prendendo di mira il 5'-fosfato con carbodiimmidi e lo zucchero 3'-ribosio con periodato. Questi approcci sono stati descritti in precedenza (5'-end18,19 e 3'-end19,20,21,22) ma non sono stati precedentemente applicati a un RNA di dimensioni simili al ribozima intronico wild-type Sc.ai5γ group II, che richiedeva un'ottimizzazione. L'attivazione della carbodiimmide (ad es. EDC) del 5'-fosfato è reversibile. Pertanto, l'imidazolo è stato utilizzato per reagire irreversibilmente con l'intermedio O-acilisourea per formare un fosforimidazolide altamente reattivo 19,20. Tuttavia, è ora noto che a pH più elevato, le carbodiimmidi possono modificare le basi nucleotidiche, in particolare le guanine e gli uracili, il che ha recentemente portato al loro utilizzo come agenti di sondaggio strutturale23,24.
Per evitare la cross-reattività, è fondamentale purificare l'RNA attivato dall'EDC prima di aumentare il pH a 7,5 per la fase di accoppiamento del colorante. Tuttavia, quando abbiamo introdotto una fase di purificazione tra l'attivazione e l'attacco del colorante25, abbiamo ottenuto rese molto basse. Analogamente, nella marcatura delle proteine, i residui di lisina accessibili in superficie possono essere attivati con le carbodiimmidi. Tuttavia, al posto dell'imidazolo, che impedisce l'inversione dell'attivazione, viene utilizzato di routine il NHS. Abbiamo adottato anche questa strategia, sostituendo così l'intermedio fosforimidazolide con un intermedio NHS-fosfato. In questo modo abbiamo ottenuto il controllo del pH e una maggiore densità di marcatura a temperature più basse e tempi di incubazione più brevi, ovvero 25 °C per 4 ore rispetto a 37 °C per 16 ore. Sviluppata per l'RNA, questa strategia di marcatura del 5' può essere applicata a qualsiasi altro acido nucleico a filamento singolo con un 5'-fosfato.
L'attivazione del 5'-fosfato e l'ossidazione del 3'-ribosio si escludevano a vicenda, perché le sostanze chimiche non sono ortogonali. Per superare questa sfida ed evitare la marcatura incrociata, abbiamo iniziato con l'estremità 5', seguita da una fase di blocco per inibire i siti attivati ma non marcati prima di procedere con la marcatura dell'estremità 3'. Durante l'ossidazione del 3'-diolo, l'eccesso di sodio meta-periodato (NaIO4) potrebbe estinguere il fluoroforo già attaccato all'estremità 5'. Pertanto, abbiamo ridotto la concentrazione di NaIO4 utilizzata per l'etichettatura singola da 20 mM a 10 mM.
Si consiglia di lavorare con più aliquote in parallelo invece di aumentare le reazioni. Questo protocollo richiede più fasi di precipitazione dell'etanolo (EtOH). Quando si lavora con più aliquote in parallelo, preparare una miscela di precipitazione (30 mL di EtOH assoluto al 100% e 1 mL di NaOAc 3 M, pH 5,2). NaCl non viene utilizzato a causa della sua bassa solubilità in EtOH. Precipitare l'RNA con 3,1 vol. di questa miscela mediante incubazione notturna a -20 °C, seguita da centrifugazione. Lavare due volte il pellet di RNA con 500 μl di EtOH ghiacciato al 70%, centrifugare a 4 °C dopo ogni volta e asciugare sotto vuoto. La precipitazione di EtOH sfrutta l'insolubilità dell'RNA e la solubilità dei coloranti liberi in etanolo al 70%. La filtrazione centrifuga rimuove efficacemente i coloranti liberi a causa della loro significativa differenza dimensionale rispetto all'RNA e facilita lo scambio di tamponi, eliminando i sali. Oltre ai metodi di precipitazione e filtrazione centrifuga con EtOH, i coloranti liberi possono essere rimossi anche utilizzando tecniche di estrazione con gel e/o cromatografia (ad esempio, HPLC); Tuttavia, la scala dovrebbe essere adattata di conseguenza. Non agitare gli RNA lunghi per risospenderli, poiché ciò potrebbe causare un taglio meccanico26. Il momento ottimale per mettere in pausa il protocollo è quando l'RNA viene pellettato. Utilizziamo tubi a basso legame del DNA per migliorare il recupero dell'acido nucleico. Sebbene il volume finale di reazione sia di 100 μL, le provette da 1,5 mL (e non di volume inferiore) sono preferite per una migliore purificazione mediante precipitazione di EtOH.
Una volta rimossi i coloranti liberi non reagiti mediante precipitazione e filtrazione centrifuga, abbiamo confermato la marcatura mediante elettroforesi su gel fluorescente (Figura 4A), spettroscopia UV-Vis, HPLCanalitica 3. Tuttavia, è importante notare che questi metodi non sono in grado di distinguere tra una molecola di RNA che trasporta sia fluorofori che una miscela di RNA, ciascuno etichettato con un singolo colore. Allo stesso modo, non possono essere utilizzati per determinare se una molecola di RNA trasporta più fluorofori dello stesso colore. La spettrometria di massa non può essere utilizzata a causa delle limitazioni dimensionali. Lespettroscopie FRET a singola molecola corroborano la marcatura a doppia fluorescenza, come mostrato nelle Figure 4B e 5B. La stechiometria di 0,5 (rapporto sCy3/Cy5 di 1:1) negli esperimenti smFRET conferma l'uguale coniugazione dei due fluorofori. Una preoccupazione era la doppia marcatura all'estremità 3' etichettando entrambe le aldeidi invece della ciclizzazione proposta. La mancanza di specie con una stechiometria di 0,25 (rapporto sCy3 a Cy5 di 1:2) negli esperimenti smFRET suggerisce che l'attaccamento del colorante ostacola stericamente e impedisce l'attaccamento di un secondo colorante.
Con questa marcatura a doppia fluorescenza, le modifiche del segnale FRET possono essere attribuite a riarrangiamenti strutturali durante il ripiegamento e la catalisi dell'RNA. Per mantenere l'RNA marcato in fluorescenza all'interno del campo evanescente per l'imaging TIRF a singola molecola, l'incapsulamento è preferibile al tethering diretto della superficie. Questo approccio prevede l'intrappolamento di singole molecole di RNA all'interno di doppi strati lipidici di vescicole, creando un ambiente controllato favorevole all'osservazione del loro comportamento dinamico. Il protocollo descritto arricchisce la mono-incapsulazione, come dimostra il fotosbiancamento in un unico passaggio11. Per comprendere il ripiegamento e la funzione dell'RNA, è essenziale colmare il divario in vitro e in vivo 27. Gli agenti di affollamento molecolare possono imitare le condizioni all'interno delle cellule per migliorare la catalisi dell'RNA da parte degli introni del gruppo II 7,28. In alternativa, l'incapsulamento crea microambienti vincolati che promuovono il ripiegamento dell'RNA29, avvicinando la nostra comprensione della struttura e della dinamica dell'RNA a un contesto cellulare realistico.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Si ringrazia il sostegno finanziario del Fondo nazionale svizzero per la ricerca scientifica [200020_192153 a RKOS], della UZH Forschungskredit [FK-20-081 a EA], della UZH Stiftung für wissenschaftliche Forschung [a RKOS e SZP], della Graduate Research Campus (GRC) Short Grant [2024__SG_092 a EA], della Graduate School of Chemical and Molecular Sciences Zurich (CMSZH) e dell'Università di Zurigo.
Materiali
| Name | Company | Catalog Number | Comments |
| RNA labeling | |||
| 1.5 mL DNA LoBind tubes | Eppendorf | 30108035 | |
| 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide hydrochloride (EDC-HCl or EDC) | Thermo Scientific | 11844071 | Pierce EDC, No-Weigh Format. Store at -20 °C |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration) |
| Acetic acid (glacial) | Sigma-Aldrich | 1.00063 | 100%, anhydrous for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Centrifugal filtration unit | Sartorius | VS0132 | Vivaspin 500, MWCO 50.000, PES, 500 μL |
| Cyanine5 hydrazide (Cy5-hydrazide) | Lumiprobe | 23070 | 5 mg, CAS number 1427705-31-4 |
| Dimethyl sulfoxide (DMSO) | New England Biolabs | 12611P | Molecular biology grade |
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glycerol | Sigma-Aldrich | G5516 | for molecular biology, ≥99.0% |
| Hydrochloric acid (HCl) | Sigma-Aldrich | 1.00317 | fuming 37%, for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| N-hydroxysuccinimide (NHS) | Sigma-Aldrich | 130672 | 98% |
| Sodium acetate (NaOAc) | Sigma-Aldrich | S8750 | anhydrous, ReagentPlus, ≥99.0% |
| Sodium meta-periodate (NaIO4) | Thermo Fisher Scientific, Life Technologies | 20504 | Pierce product line |
| Sulfo-Cyanine3 amine (sCy3-amine) | Lumiprobe | 213C0 | 5 mg, CAS number 2183440-43-7 |
| Tris(hydroxymethyl)aminomethane (Tris) | Biosolve | 200923 | Molecular biology grade |
| Chamber preparation | |||
| 3-aminopropyl)triethoxysilane (APTES) | Sigma-Aldrich | 440140 | 99% |
| Acetone | Sigma-Aldrich | 1.00014 | for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Biotin-Polyethylene glycol-Succinimidyl valerate (biotin-PEG-SVA, bPEG) and Methoxy polyethylene glycol-Succinimidyl valerate (mPEG-SVA) | Laysan Bio, Inc. | BIO-PEG-SVA, MW 5,000 and MPEG-SVA, MW 5,000 - Combo Kit | |
| Coverslips | Carl Roth | H876.2 | 24 x 24 mm, 0.13-0.16 mm thickness |
| Deconex 11 universal | Borer Chemie AG | 17416492 | Laboratory glassware cleaning solution |
| Diamond coated core drill bit | Crystalite corporation | 1 mm thickness | |
| Diamond driller | |||
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glass Coplin staining jar with cover | |||
| Imaging spacer | Grace Bio-Labs | 654008 | SecureSeal, 8 wells, 9 mm × 0.12 mm |
| Oxygen plasma cleaner | Zepto One | Diener | |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Quartz slides | G. Finkenbeiner, Inc. | 7.5 x 2.5 cm, 1 mm thickness | |
| Sodium bicarbonate (NaHCO3) | Sigma-Aldrich | S6297 | BioXtra, 99.5-100.5% |
| Lipid cake preparation | |||
| 16:0 Biotinyl Cap PE, bPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(cap biotinyl) (sodium salt), C53H98N4O11PNaS) | Avanti Polar Lipids | 870277P | Stable for 1 year at -20 °C |
| 14:0 PC, DMPC (1,2-ditetradecanoyl-sn-glycero-3-phosphocholine or 1,2-dimyristoyl-sn-glycero-3-phosphocholine, C36H72NO8P) | Avanti Polar Lipids | 850345P | |
| 2.0 mL microcentrifuge tubes, Safe-Lock | Eppendorf | 0030120094 | Autoclave to sterilize |
| 500 mL Schlenk flask | |||
| Chloroform | Merck | 1.02445 | for analysis EMSURE ACS, ISO, Reag. Ph Eur |
| Parafilm | |||
| Surface immobilization via encapsulation | |||
| (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid (Trolox®) | Thermo Fischer | 53188-07-1 | 97% |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration). Store at +4 °C in the dark. |
| Adhesive seal tabs | Grace Bio-Labs | 629200 | |
| Catalase | Sigma-Aldrich | 9001-05-2, C30 | from bovine liver, aqueous suspension, 10,000-40,000 units/mg protein |
| D-glucose | Sigma-Aldrich | G7528 | ≥99.5% (GC), BioXtra |
| Extruder polycarbonate (PC) membrane | Avanti Polar Lipids | 610005-1EA | 0.1 μm, 19 mm |
| Extruder set with heating block | Avanti Polar Lipids | 610000 | |
| Glucose oxidase | Sigma-Aldrich | 9001-37-0 | from Aspergillus niger, Type VII, lyophilized powder, ≥100,000 units/g solid (without added oxygen) |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 7786-30-3, M1028 | for molecular biology, 1.00 M ± 0.01 M solution |
| Polyester (PE) drain disc, membrane | Whatman, Cytiva | 230300 | 10 mm |
| Potassium chloride (KCl) | Sigma-Aldrich | 60128 | BioUltra, for molecular biology, ≥99.5% (AT) |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Streptavidin | Thermo Fischer | 434301 |
Riferimenti
- Eitan, L., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416 (2021).
- Taekjip, H., et al. Fluorescence resonance energy transfer at the single-molecule level. Nat Rev Methods Primers. 4, 22 (2024).
- Ahunbay, E., Steffen, F. D., Zelger-Paulus, S., Sigel, R. K. O. Chemical dual end-labeling of large ribozymes. Methods Mol Biol. 2439, 191-204 (2022).
- Ahunbay, E., Zelger-Paulus, S., Sigel, R. K. O. Group II Introns: Highly structured yet dynamic. Chimia (Aarau). 77 (4), 235-241 (2023).
- Steiner, M., Karunatilaka, K. S., Sigel, R. K. O., Rueda, D. Single-molecule studies of group II intron ribozymes. Proc Natl Acad Sci USA. 105 (37), 13853-13858 (2008).
- Karunatilaka, K. S., Solem, A., Pyle, A. M., Rueda, D. Single-molecule analysis of Mss116-mediated group II intron folding. Nature. 467 (7318), 935-939 (2010).
- Paudel, B. P., Fiorini, E., Börner, R., Sigel, R. K. O., Rueda, D. S. Optimal molecular crowding accelerates group II intron folding and maximizes catalysis. Proc Natl Acad Sci USA. 115 (47), 11917-11922 (2018).
- Saha, R., Verbanic, S., Chen, I. A. Lipid vesicles chaperone an encapsulated RNA aptamer. Nat Commun. 9 (1), 2313 (2018).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in surface-tethered lipid vesicles as a new tool for single biomolecule spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Cisse, I., Okumus, B., Joo, C., Ha, T. Fueling protein DNA interactions inside porous nanocontainers. Proc Natl Acad Sci USA. 104 (31), 12646-12650 (2007).
- Zelger-Paulus, S., Hadzic, M. C. A. S., Sigel, R. K. O., Börner, R. Encapsulation of fluorescently labeled RNAs into surface-tethered vesicles for single-molecule FRET studies in TIRF microscopy. Methods Mol Biol. 2331, 1-16 (2020).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Ishitsuka, Y., Okumus, B., Arslan, S., Chen, K. H., Ha, T. Temperature-independent porous nanocontainers for single-molecule fluorescence studies. Anal Chem. 82 (23), 9694-9701 (2010).
- Liu, B., Mazouchi, A., Gradinaru, C. C. Trapping single molecules in liposomes: surface interactions and freeze-thaw effects. J Phys Chem B. 114 (46), 15191-15198 (2010).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. J Vis Exp. (86), e50549 (2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protoc. 3 (1), 101152 (2022).
- Hadzic, M. C. A. S., Kowerko, D., Börner, R., Zelger-Paulus, S., Sigel, R. K. O., Farkas, D. L., Nicolau, D. V., Leif, R. C. Detailed analysis of complex single molecule FRET data with the software MASH. Imaging, Manipulation, and Analysis of Biomolecules, Cells, and Tissues IX. SPIE 971119, (2016).
- Chu, B. C., Wahl, G. M. Le Orgel. Derivatization of unprotected polynucleotides. Nucleic Acids Res. 11 (18), 6513-6529 (1983).
- Qin, P. Z., Pyle, A. M. Site-specific labeling of RNA with fluorophores and other structural probes. Methods. 18 (1), 60-70 (1999).
- Whitfeld, P. R. A method for the determination of nucleotide sequence in polyribonucleotides. Biochem J. 58 (3), 390-396 (1954).
- Zamecnik, P. C., Stephenson, M. L., Scott, J. F. Partial purification of soluble RNA. Proc Natl Acad Sci USA. 46 (6), 811-822 (1960).
- Proudnikov, D., Mirzabekov, A. Chemical methods of DNA and RNA fluorescent labeling. Nucleic Acids Res. 24 (22), 4535-4542 (1996).
- Wang, P. Y., Sexton, A. N., Culligan, W. J., Simon, M. D. Carbodiimide reagents for the chemical probing of RNA structure in cells. RNA. 25 (1), 135-146 (2019).
- Mitchell, D., et al. In vivo RNA structural probing of uracil and guanine base-pairing by 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide (EDC). RNA. 25 (1), 147-157 (2019).
- Rinaldi, A. J., Suddala, K. C., Walter, N. G. Native purification and labeling of RNA for single molecule fluorescence studies. Methods Mol Biol. 1240, 63-95 (2015).
- Liu, T., Patel, S., Pyle, A. M. Making RNA: Using T7 RNA polymerase to produce high yields of RNA from DNA templates. Methods in Enzymology. , (2023).
- Leamy, K. A., Assmann, S. M., Mathews, D. H., Bevilacqua, P. C. Bridging the gap between in vitro and in vivo RNA folding. Q Rev Biophys. 49, e10 (2016).
- Fiorini, E., Börner, R., Sigel, R. K. O. Mimicking the in vivo Environment--The Effect of Crowding on RNA and Biomacromolecular Folding and Activity. Chimia. 69 (4), 207-212 (2015).
- Peng, H., Lelievre, A., Landenfeld, K., Müller, S., Chen, I. A. Vesicle encapsulation stabilizes intermolecular association and structure formation of functional RNA and DNA. Curr Biol. 32 (1), 86-96.e6 (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon