Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Extraktion von DNA mit hohem Molekulargewicht von mikrobiellen Matten
In diesem Artikel
Zusammenfassung
Wir bieten ein verbessertes Protokoll zur Extraktion von DNA mit hohem Molekulargewicht aus hypersaline mikrobielle Matten. Mikrobielle Zellen sind von der Matte Matrix vor der DNA-Extraktion und Reinigung getrennt. Dies erhöht die Konzentration, Qualität und Größe der DNA. Das Protokoll kann für andere feuerfeste Proben verwendet werden.
Zusammenfassung
Erfolgreiche und genaue Analyse und Interpretation von Daten metagenomische ist abhängig von der effizienten Gewinnung von hochwertigen, mit hohem Molekulargewicht (HMW)-Community DNA. Doch Umwelt-mat Proben stellen häufig Schwierigkeiten zu erhalten hohe Konzentrationen von hoher Qualität, HMW-DNA. Hypersalinen mikrobiellen Matten enthalten hohe Mengen an extrazellulären polymeren Substanzen (EPS) 1 und Salze, die Downstream-Anwendungen der extrahierten DNA hemmen können. Direkte und harte Methoden sind oft in der DNA-Extraktion aus feuerfestem Proben verwendet. Diese Methoden sind in der Regel verwendet, da das EPS in Matten, Klebstoff-Matrix, bindet DNA 2,3 während der direkten Lyse. Als Folge der härteren Extraktionsmethoden, DNA wird in kleinen Größen 4,5,6 fragmentiert.
Die DNA wird somit ungeeignet für große Insert-Vektor Klonen. Um diese Einschränkungen zu umgehen, melden wir eine verbesserte Methodik zur HMW-DNA von guter Qualität und Quantität von hypersaline mikrobiellen Matten extrahieren. Wir verwendeten eine indirekte Methode, die die Trennung von mikrobiellen Zellen aus dem Hintergrund Matte Matrix durch Mischen und differentielle Zentrifugation. Eine Kombination aus mechanischen und chemischen Verfahren wurde verwendet, um zu extrahieren und zu reinigen DNA aus den extrahierten mikrobieller Zellen. Unser Protokoll ergibt etwa 2 ug HMW-DNA (35-50 kb) pro Gramm Matte Probe, mit einer A 260/280 Ratio von 1,6. Darüber hinaus schlägt Amplifikation von 16S rRNA-Gene 7, die das Protokoll in der Lage, minimieren oder zu eliminieren hemmenden Wirkungen von Verunreinigungen ist. Unsere Ergebnisse liefern eine geeignete Methode zur Extraktion von HMW-DNA aus mikrobiellen Matten für funktionale metagenomische Studien und kann auf andere Umwelt-Proben, aus denen die DNA-Extraktion ist eine Herausforderung.
Protokoll
1. Microbial Cell Extraction:
- Homogenisieren mikrobiellen Matten mit einer sterilen Schleifen Pistill durch Mischen gründlich. Legen Sie etwa alle 30 g (Feuchtgewicht) homogenisiert Matte Material in sterilen Behälter von Waring, etwa 100 ml 1 M NaCl (oder eine Konzentration spezifischen Probe in use) hinzufügen, und fügen sich dreimal bei mittlerer Geschwindigkeit für 1 min mit intermittierende Kühlung in einem -20 ° C Gefrierschrank für 1 min. Übertragen Sie die Gülle in einen 250 mL Zentrifuge Flasche füllen und die restlichen leeren Volumen mit 1 M NaCl. Bereiten NaCl-Lösung in autoklaviert DI Wasser und Filter sterilisieren.
- Weitere verdrängen die mikrobielle Zellen aus der Matrix durch Schütteln (150 rpm) mit einem Vortexer (Vortex-Genie R 2) bei Raumtemperatur für 30 min.
- Zentrifugation bei niedriger Geschwindigkeit (500x g) für 15 min bei 4 ° C. Vorsichtig den Überstand in einen Kolben mit minimaler Störung des Sediments.
- Mit dem pelletiert Sediment, wiederholen Sie die Schritte Mischen und 1,2 und 1,3 weitere vier Male mit der Zugabe von frischem NaCl jedem Sediment Pellet am Ende von Schritt 1.3. Der Überstand aus jeder NaCl-Extraktion ist in einem frischen Kolben überführt.
- Kombinieren Sie die Überstände aus den 5 Zellen Extraktionen und Zentrifuge in Aliquots von 200 ml mit hoher Geschwindigkeit (25.000 xg) für 15 min bei 4 ° C. Überstand verwerfen, resuspendieren jedem Zellpellet in 10 ml 2% Natriumhexametaphosphat, kombinieren Pellets und machen auf ein Volumen von 200 ml mit 2% Natriumhexametaphosphat. Wash-Zellen durch Schütteln (150 rpm) bei Raumtemperatur für 30 min. Am Ende dieses Schrittes sollten Sie nur eine Röhre von Zellen, um zum nächsten Schritt übergehen.
- Zentrifuge mit hoher Geschwindigkeit (25.000 xg) für 15 min bei 4 ° C. Überstand verwerfen, Zellpellet resuspendieren in 200 ml TE (50 mM EDTA), und waschen Sie die Zellen durch Schütteln (150 rpm) bei Raumtemperatur für 10 min.
- Zentrifuge mit hoher Geschwindigkeit (25.000 xg) für 15 min bei 4 ° C, den Überstand verwerfen, in 15 mL TE (10 mM EDTA) resuspendiert, und speichern 200-ul Aliquots bei -80 ° C bis zum Gebrauch für die DNA-Extraktion.
2. DNA Extraktion und Reinigung:
- Geben Sie 200 ul 5 M NaCl, 200 ul 10% SDS und 100 ul 14,3 Mio. β-Mercaptoethanol auf die 200-ul mikrobielle Zelle Aliquot. Vorsichtig mischen durch Invertieren 4-6 mal.
- Vorbehaltlich der Mischung auf 3 Runden Frost-Tau durch Eintauchen in flüssigen Stickstoff für 2 min durch Auftauen in einem 65 ° C Wasserbad für 5 min. Extend endgültigen Auftauen bis 10 min.
- Geben Sie 200 ul 5 M Kaliumacetat (pH 5,5) und auf Eis für 10 min. Zentrifuge bei 10.000 xg für 10 min bei 4 ° C und den Überstand in ein neues Röhrchen mit einem weit Bohrung Pipettenspitze. Add 3 ul RNase A und Inkubation bei 37 ° C für 1 Stunde.
- Add gleichen Volumen Chloroform, kurz schütteln durch Umdrehen und Zentrifuge bei 15.000 xg für 10 min bei Raumtemperatur.
- Sorgfältig Transfer die oberste Schicht der Überstand in ein neues Röhrchen mit einem weit Bohrung Pipettenspitze. Vermeiden Pipettieren der weiße Grenzschicht zwischen der oberen und unteren Schichten entfernt. Wiederholen Sie die Chloroform-Extraktion.
- Fügen Sie dem gleichen Volumen gekühlt (bei -20 ° C) Isopropanol zum Überstand und inkubieren auf Eis für 30 min zur Fällung DNA.
- Zentrifuge bei Höchstgeschwindigkeit (20.800 xg) für 10 min bei 4 ° C bis Pellet DNA.
- Überstand verwerfen und waschen DNA-Pellet mit 1 ml gekühlt (-20 ° C) 70% Ethanol.
- Zentrifuge bei Höchstgeschwindigkeit (20.800 xg) für 10 min bei 4 ° C, den Überstand verwerfen, wiederholen Sie 70% Ethanol waschen und an der Luft trocknen DNA-Pellet für 10 min. Wiederholen Sie die 70% Ethanol waschen.
- Resuspendieren DNA in 25 ul TE (10 mM EDTA), warm bei 65 ° C für 5 min in einer Röhre zu verbinden, wenn mehrere Rohre verwendet wurden, und Make-up-Volumen auf 500 ul mit TE.
- Add 500 mL eiskaltem (oder bei 4 ° C) 20% Polyethylenglykol (PEG) (hergestellt in 1,2 M NaCl). Vorsichtig mischen durch Umdrehen, auf Eis für 10 min und Zentrifuge inkubieren bei maximaler Geschwindigkeit für 10 min bei 4 ° C bis Pellet DNA. Entfernen und den Überstand verwerfen und waschen Sie die pelletierte DNA mit 1 ml gekühltem 70% Ethanol, an der Luft trocknen für 10 min und Resuspendieren in 30-50 ul TE oder molekulare reines Wasser. Erleichterung Resuspension der DNA durch Erwärmung auf 65 ° C für 5 min und DNA bei -80 ° C.
3. DNA-Reinheit, Konzentration und Größe Bestimmung:
- Bestimmen DNA-Qualität durch die Messung des A 260/280 und A 260/230 Verhältnisse mit einem Nanodrop 1000 Spektralphotometer oder sonstige geeignete Instrumente.
- Bestimmen DNA-Konzentration mit dem Quant-iT dsDNA Assay Kit nach den Anweisungen des Herstellers.
- Bestimmen DNA Größe mit Puls-Feld-Gelelektrophorese. Bereiten Sie 1% Agarosegel mit 0,5 X TBE und verwenden Sie den folgenden Parameter auf der CHEF Mapper XA System; ersten Einschalten der Zeit von 0,35 s, eine endgültige Zeit umschalten von 7,67 s und 120 ° Öffnungswinkel für ein Gefälle von 6,0 V / cm bei eine lineare rAmping-Faktor. Stain Gel mit 1 x SYBR Green I für 30 min. Legen Sie zwischen 700-1000 ng Gesamt-DNA auf einem Gel aufgetrennt. Siehe Abb.. 3 für eine verkürzte Form der komplette Protokoll.
Repräsentative Ergebnisse:
Zell-Extraktion:
Sequential mikrobiellen Zellextrakten (Überstände) haben gezeigt, dass die Trübung der Extrakte als die Zahl der Extraktionen erhöhen abnimmt. Dies deutet auf eine Reduzierung der Anzahl der Zellen nach jeder aufeinander folgenden Zelle Extraktion. Wichtig hierbei ist, dass da jeder zusätzliche Zelle Extraktionsschritt bietet die Möglichkeit für Kontaminanten Einführung, die Anzahl der Zellen Extraktionen minimiert werden sollte, so dass die endgültige Zellpellet repräsentativ für die Gesamtheit der mikrobiellen Gemeinschaft ist. Andere Quellen von Verunreinigungen wurden durch die Verabschiedung angemessener Labor sterile Techniken gesteuert. Zum Beispiel wurden die Lösungen in autoklaviert DI Wasser hergestellt und sterilisiert. Die Behälter wurden mit Alkohol sterilisiert, autoklaviert und behandelt unter UV-Licht und UV-Vernetzer.
DNA-Konzentration und Qualität Bestimmung:
Das Protokoll ergab etwa 2 ug HMW-DNA (35-50 kb) (Abb. 4) pro Gramm Matte Probe mit einem A 260/280 Ratio von 1,6 und ein A 260/230 Ratio von 0,7 (Tabelle 1). Obwohl die A 260/230 Ratio schien gering zu sein, keine Hemmung der nachgeschalteten molekularen-basierten Anwendungen wie PCR-Amplifikation des 16S rRNA-Gene in einer separaten Studie 7 beobachtet wurde. Es ist wichtig, dass DNA aus hypersaline Matten hat zwei Hauptquellen der Kontamination beachten Sie, EPS und Salze. Es ist daher möglich, dass Spuren dieser Verunreinigungen kann Einfluss auf die A 260 / 230 Verhältnisse trotz der enormen Anstrengungen, um ihre Auswirkungen auf die Downstream-Anwendungen zu reduzieren.
DNA Größenbestimmung:
Pulse-Feld-Gel-Elektrophorese verwendet eine pulsierende Stromfluss mit Intervallschaltung direktionale Schalter was zu einem DNA-Abstrich, wie in Abbildung 3 dargestellt. Unser Protokoll ergab einen HMW-DNA von ungefähr 35-50 kb. Während einige DNA Größe kann kleiner sein als 30 kb, ist es wichtig, einen Teil der Abstrich über 35 kb da Fosmid Klonen ~ 40 kb DNA-Inserts erfordert und größere DNA-Fragmente bieten einen besseren Zugang zu erhalten Biosynthesewege haben. In unseren Untersuchungen wurde die DNA-Abstrich über 35 kb ausgeschnitten und gereinigt für große Insert-Vektor Klonierung und andere molekulare Anwendungen.

Abbildung 1. Salzwasserwanne mikrobiellen Matte Probenahmestelle (Big Pond) auf Eleuthera, Bahamas entfernt.

Abbildung 2. Ein Querschnitt der hypersaline mikrobiellen Matte in dieser Studie verwendet. Die mikrobielle Matte wurde aus einer hypersaline Teich auf Eleuthera, Bahamas befindet sich erhalten.
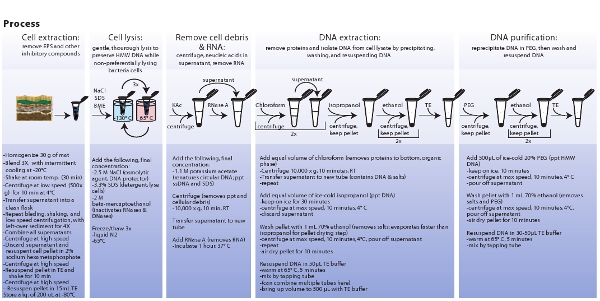
Abbildung 3. Schematische Darstellung der Verfahren in mikrobielle Zelle Extraktion, Zell-Lyse, Extraktion und Reinigung von DNA metagenomische beteiligt. Klicken Sie hier, um ein größeres Bild zu sehen.

Abbildung 4. Molekulargewicht Charakterisierung der extrahierten DNA mittels Puls-Feld-Gelelektrophorese. Lane M ist ein HMW-Marker und die Spuren 1 und 2 sind Wiederholungen von Metagenom DNA aus dem Eleuthera hypersaline Matte mit dem obigen Protokoll extrahiert.
| Extraction Method | Konzentration ng / g | A 260/280 | A 260/230 | |
| PEG | REP 1 | 1806,1 | 1,64 | 0,76 |
| Rep 2 | 2010,8 | 1,61 | 0,75 |
Tabelle 1. Die Messung der Konzentration und Qualität der DNA extrahiert aus mikrobiellen hypersaline Matte.
Access restricted. Please log in or start a trial to view this content.
Diskussion
Da die gesamte Zelle Entnahme aus komplexen und sehr unterschiedlichen mikrobiellen Matte Proben nicht praktikabel ist, ist die wichtigste Angelegenheit, wie gut die extrahierten Zellen die gesamte mikrobielle Matte Gemeinschaft darstellen. In einer früheren Studie zeigte PCR-DGGE Analyse mikrobieller 16S rRNA-Gene, dass die fünf Zelle Ausbauschritte in diesem Protokoll verwendeten Zellen, die repräsentativ für die Gesamtheit mikrobiellen Matte Gemeinschaft 7 sind Extrakte. Die tatsächliche Anzahl der Ze...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Danksagungen
Diese Arbeit wurde von der National Science Foundation Environmental Genomics Program (Grant No EF-0723707) gefördert.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenz | Firma | Katalog-Nummer | Kommentare |
|---|---|---|---|
| β-Mercaptoethanol | Sigma-Aldrich | M3148 | |
| Polyethylenglycol 8000 | Promega | V3011 | 20% in 1,2 M NaCl |
| Kaliumacetat | Fisher Scientific | Fisher Scientific | |
| Quant-iT dsDNA Assay Kit | Invitrogen | Q33130 | |
| RNase | Epicentre | MRNA092 | |
| Kochsalz | BDH Chemicals | BDH8014 | Entsprechende konz. |
| Natriumdodecylsulfat | Fisher Scientific | 03-500-509 | 10% in Wasser |
| Natriumhexametaphosphat | EMD Chemicals | SX0583-3 | 2% in Wasser |
| TBE | Fisher Scientific | BP1333-1 | |
| CHEF Mapper XA-System | Bio-Rad Laboratories | 170-3670 | |
| NanoDrop 1000 Spektralphotometer | Thermo Scientific | ND-1000 | |
| Vortexer | Scientific Industries Inc. | ||
| Ultraviolet Crosslinker | UVP | ||
| Waring | Waring Labor | LB10S |
Referenzen
- Decho, A. W. Microbial biofilms in intertidal systems: an overview. Cont. Shelf Res. 20, 1257-1273 (2000).
- Dupraz, C., Visscher, P. T. Microbial lithification in marine stromatolites and hypersaline mats. Trends Microbiol. 13, 429-438 (2005).
- Steffan, R. J., Goksoyr, J., Boj, A. K., Atlas, R. M. Recovery of DNA from soils and sediments. Appl. Environ. Microbiol. 54, 2908-2915 (1988).
- Lee, Y. K., Kim, H. W., Liu, C. L., Lee, H. K. A simple method for DNA extraction from marine bacteria that produce extracellular materials. J. Microbiol. Methods. 52, 245-250 (2003).
- Roose-Amsaleg, C. L., Garnier-Sillam, E., Harry, M. Extraction and Purification of Microbial DNA from Soil and Sediment Samples. Appl. Soil Ecol. 18, 47-60 (2001).
- de Lipthay, J. R., Enzinger, C., Johnsen, K., Aamand, J., Sørensen, S. J. Impact of DNA extraction method on bacterial community composition measured by denaturing gradient gel electrophoresis. Soil Biol. Biochem. 36, 1607-1614 (2004).
- Bey, B. S., Fichot, E. B., Dayama, G., Decho, A. W., Norman, R. S. Extraction of High Molecular Weight DNA from Microbial Mats. Biotechniques. 49, 631-640 (2010).
- Kakirde, S. K., Parsley, L. C., Liles, M. R. Size does matter: Application-driven approaches for soil metagenomics. Soil Biology & Biochemistry. 42, 1911-1923 (2010).
- Rodon, M. R., August, P. R., Bettermann, A. D., Brady, S. F., Grossman, T. H., Liles, M. R., Loiacono, K. A., Lynch, B. A., MacNeil, I. A., Minor, C., Tiong, C. L., Gilman, M., Osburne, M. S., Clardy, J., Handelsman, J., Goodman, R. M. Cloning the Soil Metagenomic: A Strategy for Accessing the Genetic and Functional Diversity of Uncultured Microorganisms. Appl. Environ. Microbiol. 66, 2541-2547 (2000).
- Beja, O., Aravind, L., Koonin, E. V., Suzuki, M. T., Hadd, A., Nguyen, L. P., Jovanovich, S. B., Gates, C. M., Feldman, R. A., Spudich, J. L., Spudich, E. N., DeLong, E. F. Bacterial Rhodopsin: Evidence for a New Type of Phototrophy in the Sea. Science. 289, 1902-1906 (2000).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten