Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Extraction d'ADN de haut poids moléculaire de tapis microbiens
Dans cet article
Résumé
Nous fournissons un protocole amélioré pour l'extraction ADN de haut poids moléculaire à partir de tapis microbiens hypersalin. Les cellules microbiennes sont séparés de la matrice mat avant l'extraction de l'ADN et de purification. Cela améliore la concentration, la qualité et la taille de l'ADN. Le protocole peut être utilisé pour d'autres échantillons réfractaires.
Résumé
Analyse réussie et exacte et l'interprétation des données métagénomiques dépend de l'extraction efficace de haute qualité, l'ADN de haut poids moléculaire de la communauté (HMW). Cependant, les échantillons environnementaux posent souvent des tapis de difficultés à obtenir de grandes concentrations de haute qualité, l'ADN HMW. Tapis microbiens hypersalin contiennent des quantités élevées de substances polymériques extracellulaires (EPS) 1 et les sels qui peuvent inhiber les applications en aval de l'ADN extrait. Les méthodes directes et dures sont souvent utilisés dans l'extraction d'ADN à partir d'échantillons réfractaires. Ces méthodes sont généralement utilisées parce que le BPA dans des nattes, une matrice adhésive, lie l'ADN 2,3 lors de la lyse directe. En raison de méthodes d'extraction plus sévères, l'ADN se fragmente en petites tailles 4,5,6.
L'ADN devient donc inapproprié pour le vecteur de clonage à grande insérer. Afin de contourner ces limitations, nous présentons une méthodologie améliorée pour en extraire l'ADN HMW de bonne qualité et la quantité de tapis microbiens hypersalin. Nous avons utilisé une méthode indirecte impliquant la séparation des cellules microbiennes de la matrice mat fond par mélange et centrifugation différentielle. Une combinaison de procédés mécaniques et chimiques a été utilisé pour extraire et purifier l'ADN des cellules microbiennes extrait. Notre protocole de rendements d'environ 2 pg d'ADN HMW (35-50 ko) par gramme d'échantillon mat, avec un A 260/280 ratio de 1,6. Par ailleurs, l'amplification des gènes d'ARNr 16S 7 suggère que le protocole est en mesure de minimiser ou éliminer les effets inhibiteurs de contaminants. Nos résultats fournissent une méthodologie appropriée pour l'extraction d'ADN à partir HMW tapis microbiens fonctionnels pour les études métagénomiques et peut être applicable à d'autres échantillons environnementaux à partir de laquelle l'extraction d'ADN est un défi.
Protocole
1. Extraction de cellule microbienne:
- Homogénéiser les tapis microbiens avec un pilon stériles meulage en mélangeant soigneusement. Placer environ tous les 30 g (poids humide) de matériau mat homogénéisé dans un contenant stérile d'un mélangeur Waring, ajouter environ 100 ml de NaCl 1 M (ou une concentration spécifique à l'échantillon en utilisation), et mélanger à trois reprises à vitesse moyenne pendant 1 min avec refroidissement intermittent dans un congélateur à -20 ° C pendant 1 min. Transférer la pâte dans une bouteille de 250 mL centrifugeuse et de remplir le volume restant vide avec 1 M de NaCl. Préparer une solution de NaCl dans l'autoclave DI eau et filtrer le stériliser.
- En outre déloger les cellules microbiennes de la matrice par agitation (150 rpm) en utilisant un Vortexer (Vortex-Genie R 2) à température ambiante pendant 30 min.
- Centrifuger à basse vitesse (500x g) pendant 15 min à 4 ° C. Délicatement le transfert du surnageant dans un flacon avec une perturbation minimale de la sédimentation.
- En utilisant les sédiments granulés, répétez les étapes de mélange et 1,2 et 1,3 quatre fois supplémentaires avec l'ajout de frais de NaCl à chaque pastille sédiments à la fin de l'étape 1.3. Le surnageant de chaque extraction de NaCl est transféré dans un flacon de frais.
- Combinez les surnageants des extractions de cellule et centrifuger 5 aliquots de 200 ml à haute vitesse (25 000 xg) pendant 15 min à 4 ° C. Jeter le surnageant, resuspendre chaque culot cellulaire dans 10 ml d'hexamétaphosphate de sodium à 2%, mélanger boulettes et faire monter le volume à 200 ml avec hexamétaphosphate de sodium à 2%. Laver les cellules par la secousse (150 rpm) à température ambiante pendant 30 min. A la fin de cette étape, vous devriez avoir un seul tube de cellules pour procéder à l'étape suivante.
- Centrifugeuse à vitesse élevée (25 000 xg) pendant 15 min à 4 ° C. Jeter le surnageant, resuspendre le culot cellulaire dans 200 ml de TE (50 mM d'EDTA), et laver les cellules par agitation (150 rpm) à température ambiante pendant 10 min.
- Centrifugeuse à vitesse élevée (25 000 xg) pendant 15 min à 4 ° C, éliminer le surnageant, remettre en suspension dans 15 ml de TE (10 mM d'EDTA), et stocker 200 uL d'aliquotes à -80 ° C jusqu'au moment de l'extraction d'ADN.
2. Extraction de l'ADN et purification:
- Ajouter 200 ul 5 M de NaCl, 200 ul SDS à 10%, et 100 uL 14,3 M β-mercaptoéthanol à l'aliquote de 200 uL microbienne cellule. Mélanger doucement en inversant 4-6 fois.
- Objet le mélange à trois tours de gel-dégel en submergeant dans l'azote liquide pendant 2 min puis décongélation dans un bain d'eau à 65 ° C pendant 5 min. Étendre finale dégel à 10 min.
- Ajouter 200 ul d'acétate de potassium 5 M (pH 5,5) et le placer sur la glace pendant 10 min. Centrifuger à 10000 xg pendant 10 min à 4 ° C et le transfert du surnageant dans un nouveau tube à l'aide d'une pipette de grand diamètre. Ajouter 3 pi de RNase A et incuber à 37 ° C pendant 1h.
- Ajouter un volume égal de chloroforme, agiter brièvement par inversion, et centrifuger à 15 000 xg pendant 10 min à température ambiante.
- Soigneusement transfert de la couche supérieure de surnageant dans un nouveau tube à l'aide d'une pipette de grand diamètre. Évitez de pipetage la couche d'interface blanche située entre les couches supérieure et inférieure. Répétez la procédure chloroforme-extraction.
- Ajouter un volume égal de froid (à -20 ° C) d'isopropanol au surnageant et incuber sur glace pendant 30 min pour précipiter l'ADN.
- Centrifuger à vitesse max (20 800 xg) pendant 10 min à 4 ° C à culot d'ADN.
- Jeter le surnageant et laver l'ADN culot avec 1 mL de froid (à -20 ° C) 70% d'éthanol.
- Centrifuger à vitesse max (20 800 xg) pendant 10 min à 4 ° C, éliminer le surnageant, répétez lavage à l'éthanol 70%, et sécher à l'air culot d'ADN pendant 10 min. Répétez lavage à l'éthanol 70%.
- Resuspendre l'ADN dans 25 ul de TE (10 mM d'EDTA), chaude à 65 ° C pendant 5 min, mélanger dans un tube, si plusieurs tubes ont été utilisés, et le maquillage volume à 500 ul avec TE.
- Ajouter 500 ul glacée (ou à 4 ° C) de polyéthylène glycol de 20% (PEG) (préparé en 1.2 M de NaCl). Mélanger doucement par inversion, incuber sur de la glace pendant 10 min et centrifuger à vitesse maximale pendant 10 min à 4 ° C à culot d'ADN. Retirez et jetez le surnageant et laver l'ADN granulés avec 1 ml d'éthanol à 70% réfrigérés, séchés à l'air pendant 10 min, et remettre en 30-50 uL TE ou moléculaire de l'eau de qualité. Faciliter la remise en suspension de l'ADN par le réchauffement à 65 ° C pendant 5 min et entreposer l'ADN à -80 ° C.
3. Pureté d'ADN, de concentration et de détermination Taille:
- Déterminer la qualité de l'ADN par la mesure de la 260/280 A et A 260/230 ratios en utilisant un spectrophotomètre ou Nanodrop 1000 toute autre instrumentation appropriée.
- Déterminer la concentration d'ADN en utilisant Quant-iT Kit de dosage ADNdb conformément aux instructions du fabricant.
- Déterminer la taille d'ADN par électrophorèse sur gel d'impulsion sur le terrain. Préparer 1% gel d'agarose avec 0.5X TBE et utiliser les paramètres suivants sur le système CHEF Mapper XA; changer l'heure initiale de 0,35 s, un temps final de passer 7,67 s et 120 ° angle inclus pour une pente de 6,0 V / cm à une r linéairesamplification facteur. Tache de gel avec 1 x SYBR j'ai vert pendant 30 min. Charge entre 700-1000 ng d'ADN total sur gel. Voir Fig. 3 pour une forme condensée du protocole complet.
Les résultats représentatifs:
Extraction de cellule:
Séquentielle des extraits de cellules microbiennes (surnageants) ont montré que la turbidité des extraits diminue à mesure que le nombre d'augmentation des extractions. Ceci suggère une réduction du nombre de cellules après chaque extraction de cellule successives. Important à noter ici est que chaque étape d'extraction de cellules supplémentaires prévoit la possibilité pour l'introduction de contaminants, le nombre d'extractions de cellule doit être minimisée pour que le culot cellulaire final est représentatif de la communauté microbienne globale. D'autres sources de contamination ont été contrôlés par l'adoption de techniques de laboratoire adéquats stérile. Par exemple, les solutions ont été préparées dans de l'eau déminéralisée à l'autoclave et stérilisés par filtration. Conteneurs ont été stérilisés avec de l'alcool, autoclave, et traités sous la lumière UV et réticulant ultraviolets.
Concentration de l'ADN et la détermination de la qualité:
Le protocole a donné environ 2 pg d'ADN HMW (35-50 ko) (Fig. 4) par gramme d'échantillon tapis avec un A 260/280 ratio de 1,6, et un A 260/230 ratio de 0,7 (tableau 1). Bien que le rapport A 260/230 semblait être faible, pas d'inhibition de l'aval moléculaires à base d'application tels que la PCR d'amplification des gènes d'ARNr 16S a été observée dans une étude distincte 7. Il est important de noter que l'ADN de hypersalin tapis a deux principales sources de contamination; EPS et des sels. Il est donc possible que des traces de ces contaminants peuvent influencer l'260/230 Un ratio en dépit des efforts énormes pour réduire leurs effets sur les applications en aval.
L'ADN détermination de la taille:
Électrophorèse en champ pulsé utilise un débit pulsé courant avec interrupteurs intermittents directionnelle traduit par un frottis d'ADN comme le montre la figure 3. Notre protocole donné un ADN HMW d'environ 35 à 50 ko. Alors que certains de taille d'ADN peut être inférieur à 30 ko, il est important d'avoir une partie du frottis-dessus de 35 ko, étant donné que le clonage fosmide nécessite environ 40 kb inserts d'ADN et des fragments d'ADN plus de fournir un meilleur accès aux voies de biosynthèse intacte. Dans nos études, le frottis d'ADN de plus de 35 kb a été excisée et purifiée pour le vecteur de clonage à grande insertion et d'autres applications moléculaires.

Figure 1. Microbienne hypersalin tapis de site d'échantillonnage (Big Pond) situé sur Eleuthera, aux Bahamas.

Figure 2. Une coupe transversale du tapis microbien hypersalin utilisées dans cette étude. Les tapis microbiens a été obtenue à partir d'un étang situé sur hypersalin Eleuthera, aux Bahamas.
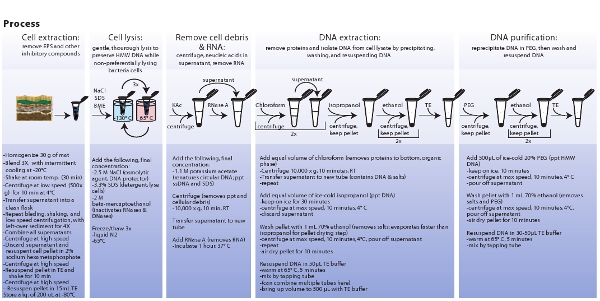
Figure 3. Représentation schématique des procédures impliquées dans l'extraction de cellules microbiennes, la lyse cellulaire, l'extraction et la purification d'ADN métagénomique. Cliquez ici pour voir une image plus grande.

Figure 4. Caractérisation du poids moléculaire de l'ADN extrait par électrophorèse en champ pulsé. Lane M est un marqueur HMW et des voies 1 et 2 sont de l'ADN se réplique métagénomique extraites du tapis de Eleuthera hypersalin utilisant le protocole ci-dessus.
| Méthode d'extraction | Concentration ng / g | A 260/280 | A 260/230 | |
| PEG | Rep 1 | 1806.1 | 1,64 | 0,76 |
| Rep 2 | 2010.8 | 1,61 | 0,75 |
Tableau 1. Mesure de la concentration et la qualité de l'ADN extrait de tapis microbiens hypersalin.
Access restricted. Please log in or start a trial to view this content.
Discussion
Étant donné que l'élimination totale des cellules du complexe et très diversifié échantillons des tapis microbiens n'est pas pratique, la principale préoccupation est de savoir comment bien les cellules extraites de représenter la communauté microbienne globale tapis. Dans une étude précédente, la PCR-DGGE analyse des gènes microbiens ARNr 16S ont montré que les cinq étapes d'élimination des cellules utilisées dans le présent protocole d'extraits des cellules qui sont représentatifs de...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Remerciements
Ce travail a été financé par la National Science Foundation Programme génomique environnementale (Subvention n ° 0723707 EF-).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Nom de réactif | Société | Numéro de catalogue | Commentaires |
|---|---|---|---|
| β-mercaptoéthanol | Sigma-Aldrich | M3148 | |
| Le polyéthylène glycol 8000 | Promega | V3011 | 20% dans 1,2 M de NaCl |
| L'acétate de potassium | Fisher Scientific | Fisher Scientific | |
| Quant-iT Assay Kit ADNdb | Invitrogen | Q33130 | |
| RNase | Epicentre | MRNA092 | |
| Chlorure de sodium | BDH Chemicals | BDH8014 | Conc appropriées. |
| Dodécyl sulfate de sodium | Fisher Scientific | 03-500-509 | 10% dans de l'eau |
| hexamétaphosphate de sodium | EMD Chemicals | SX0583-3 | 2% dans de l'eau |
| TBE | Fisher Scientific | BP1333-1 | |
| CHEF Mapper XA système | Bio-Rad Laboratories | 170-3670 | |
| NanoDrop 1000 spectrophotomètre | Thermo Scientific | ND-1000 | |
| Vortexer | Scientific Industries Inc | ||
| Crosslinker ultraviolet | UVP | ||
| Mélangeur Waring | Waring de laboratoire | LB10S |
Références
- Decho, A. W. Microbial biofilms in intertidal systems: an overview. Cont. Shelf Res. 20, 1257-1273 (2000).
- Dupraz, C., Visscher, P. T. Microbial lithification in marine stromatolites and hypersaline mats. Trends Microbiol. 13, 429-438 (2005).
- Steffan, R. J., Goksoyr, J., Boj, A. K., Atlas, R. M. Recovery of DNA from soils and sediments. Appl. Environ. Microbiol. 54, 2908-2915 (1988).
- Lee, Y. K., Kim, H. W., Liu, C. L., Lee, H. K. A simple method for DNA extraction from marine bacteria that produce extracellular materials. J. Microbiol. Methods. 52, 245-250 (2003).
- Roose-Amsaleg, C. L., Garnier-Sillam, E., Harry, M. Extraction and Purification of Microbial DNA from Soil and Sediment Samples. Appl. Soil Ecol. 18, 47-60 (2001).
- de Lipthay, J. R., Enzinger, C., Johnsen, K., Aamand, J., Sørensen, S. J. Impact of DNA extraction method on bacterial community composition measured by denaturing gradient gel electrophoresis. Soil Biol. Biochem. 36, 1607-1614 (2004).
- Bey, B. S., Fichot, E. B., Dayama, G., Decho, A. W., Norman, R. S. Extraction of High Molecular Weight DNA from Microbial Mats. Biotechniques. 49, 631-640 (2010).
- Kakirde, S. K., Parsley, L. C., Liles, M. R. Size does matter: Application-driven approaches for soil metagenomics. Soil Biology & Biochemistry. 42, 1911-1923 (2010).
- Rodon, M. R., August, P. R., Bettermann, A. D., Brady, S. F., Grossman, T. H., Liles, M. R., Loiacono, K. A., Lynch, B. A., MacNeil, I. A., Minor, C., Tiong, C. L., Gilman, M., Osburne, M. S., Clardy, J., Handelsman, J., Goodman, R. M. Cloning the Soil Metagenomic: A Strategy for Accessing the Genetic and Functional Diversity of Uncultured Microorganisms. Appl. Environ. Microbiol. 66, 2541-2547 (2000).
- Beja, O., Aravind, L., Koonin, E. V., Suzuki, M. T., Hadd, A., Nguyen, L. P., Jovanovich, S. B., Gates, C. M., Feldman, R. A., Spudich, J. L., Spudich, E. N., DeLong, E. F. Bacterial Rhodopsin: Evidence for a New Type of Phototrophy in the Sea. Science. 289, 1902-1906 (2000).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon