È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Estrazione di DNA ad alta peso molecolare da Mats microbica
In questo articolo
Riepilogo
Forniamo un protocollo migliore per l'estrazione del DNA alto peso molecolare da stuoie microbica hypersaline. Cellule microbiche sono separate dalla matrice tappetino prima dell'estrazione del DNA e la purificazione. Questo aumenta la concentrazione, la qualità e le dimensioni del DNA. Il protocollo può essere utilizzato per altri campioni refrattari.
Abstract
Analisi di successo e preciso e l'interpretazione dei dati metagenomica dipende l'estrazione efficiente di alta qualità, ad elevato peso molecolare (HMW) del DNA della comunità. Tuttavia, i campioni di mat ambientali costituiscono spesso difficoltà ad ottenere grandi concentrazioni di alta qualità, il DNA HMW. Stuoie microbica hypersaline contengono elevate quantità di sostanze polimeriche extracellulari (EPS) 1 e sali che possono inibire le applicazioni a valle di DNA estratto. Metodi diretti e duri sono spesso utilizzati per l'estrazione del DNA da campioni di refrattari. Questi metodi sono generalmente utilizzati perché l'EPS in stuoie, una matrice adesiva, lega il DNA 2,3 durante la lisi diretta. Come risultato di metodi di estrazione più severe, il DNA viene frammentato in piccole dimensioni 4,5,6.
Il DNA diventa così inadeguato per la grande inserto clonazione vettore. Per aggirare queste limitazioni, si riporta un metodo migliore per estrarre il DNA HMW di buona qualità e la quantità di stuoie microbica hypersaline. Abbiamo impiegato un metodo indiretto che coinvolge la separazione di cellule microbiche dalla matrice sfondo opaco attraverso la miscelazione e la centrifugazione differenziale. Una combinazione di procedure meccaniche e chimiche è stato utilizzato per estrarre e purificare il DNA da cellule microbiche estratti. Il nostro protocollo produce circa 2 mg di HMW DNA (35-50 kb) per grammo di campione tappeto, con un 260/280 Un rapporto di 1,6. Inoltre, l'amplificazione dei geni rRNA 16S 7 si evince che il protocollo è in grado di minimizzare o eliminare gli effetti inibitori dei contaminanti. I nostri risultati forniscono una metodologia adeguata per l'estrazione del DNA da HMW stuoie microbica per gli studi funzionali metagenomica e può essere applicabile ad altri campioni ambientali dai quali l'estrazione del DNA è una sfida.
Protocollo
1. Cella di estrazione microbica:
- Omogeneizzare stuoie microbica con un pestello sterile macinazione mescolando accuratamente. Mettere circa 30 g tutti (peso a umido) di materiale opaco omogeneizzato in un contenitore sterile di Waring frullatore, aggiungere circa 100 ml di 1 M NaCl (o una specifica concentrazione di campione in uso), e si fondono tre volte a velocità media per 1 minuto con raffreddamento intermittente in un congelatore di -20 ° C per 1 min. Trasferire l'impasto in una bottiglia da 250 ml centrifuga e riempire il volume vuoto rimasto con 1 M di NaCl. Preparare la soluzione di NaCl in acqua autoclavata DI e filtro sterilizzarlo.
- Ulteriori rimuovere le cellule microbiche dalla matrice agitando (150 giri) con un vortex (Vortex-Genie R 2) a temperatura ambiente per 30 min.
- Centrifugare a bassa velocità (500x g) per 15 minuti a 4 ° C. Delicatamente trasferire il surnatante in un pallone con il minimo disturbo per il sedimento.
- Utilizzando il sedimento pellet, ripetere la miscelazione e punti 1.2 e 1.3 quattro volte supplementare con l'aggiunta di NaCl fresca ad ogni sedimento pellet alla fine del passo 1.3. Il supernatante di ogni estrazione NaCl viene trasferita in un pallone fresca.
- Combinare i surnatanti dalla cella di 5 estrazioni e centrifugare in aliquote da 200 ml ad alta velocità (25.000 xg) per 15 minuti a 4 ° C. Eliminare il surnatante, risospendere ogni pellet di cellule in 10 ml di esametafosfato di sodio al 2%, si combinano pellet e portare il volume a 200 ml con esametafosfato di sodio al 2%. Lavare le cellule agitando (150 rpm) a temperatura ambiente per 30 min. Al termine di questa fase si dovrebbe avere un solo tubo di cellule per procedere alla fase successiva.
- Centrifuga ad alta velocità (25.000 xg) per 15 minuti a 4 ° C. Eliminare il surnatante, risospendere il pellet in 200 ml di TE (50 mM EDTA), e lavare le cellule agitando (150 rpm) a temperatura ambiente per 10 min.
- Centrifuga ad alta velocità (25.000 xg) per 15 minuti a 4 ° C, scartare il surnatante, risospendere in 15 ml di TE (10 mM EDTA), e memorizzare 200 microlitri aliquote a -80 ° C fino al momento della estrazione del DNA.
2. Estrazione e purificazione del DNA:
- Aggiungere 200 ul 5 M NaCl, 200 ul SDS 10%, e 100 ul 14,3 M β-mercaptoetanolo al 200 microlitri aliquota di cellule microbiche. Mescolare delicatamente capovolgendo 4-6 volte.
- Soggetto la miscela a 3 turni di gelo-disgelo con immersione in azoto liquido per 2 minuti seguito da scongelamento in un bagno d'acqua a 65 ° C per 5 min. Estendere scongelamento finale per 10 min.
- Aggiungere 200 ul di acetato di potassio 5 M (pH 5,5) e il luogo in ghiaccio per 10 min. Centrifugare a 10000 xg per 10 minuti a 4 ° C e trasferire il surnatante in una nuova provetta utilizzando un ampio tunnel punta della pipetta. Aggiungere 3 ml di RNasi A e incubare a 37 ° C per 1 ora.
- Aggiungere uguale volume di cloroformio, agitare per inversione, e centrifugare a 15000 xg per 10 minuti a temperatura ambiente.
- Con attenzione trasferire lo strato superiore di supernatante in un tubo nuovo usando un ampio calibro punta della pipetta. Evitare di pipettare lo strato bianco di interfaccia si trova tra gli strati superiori e inferiori. Ripetere la procedura di estrazione cloroformio.
- Aggiungere un volume equivalente di acqua refrigerata (a -20 ° C) isopropanolo al surnatante e incubare in ghiaccio per 30 minuti al DNA precipitato.
- Centrifugare alla massima velocità (20.800 xg) per 10 minuti a 4 ° C a pellet di DNA.
- Eliminare il supernatante e lavare il DNA pellet con 1 ml di acqua refrigerata (a -20 ° C) etanolo al 70%.
- Centrifugare alla massima velocità (20.800 xg) per 10 minuti a 4 ° C, scartare il surnatante, ripetere lavare etanolo al 70%, e aria secca DNA pellet per 10 minuti. Ripetere il lavaggio etanolo al 70%.
- Risospendere DNA in 25 microlitri TE (10 mM EDTA), caldo a 65 ° C per 5 minuti, si combinano in un tubo, se i tubi multipli sono stati utilizzati, e make-up del volume di 500 microlitri di TE.
- Aggiungere 500 microlitri ghiacciata (o 4 ° C) polietilene glicole 20% (PEG) (preparato in 1,2 M di NaCl). Mescolare delicatamente per inversione, incubare in ghiaccio per 10 minuti, e centrifugare alla massima velocità per 10 minuti a 4 ° C a pellet di DNA. Rimuovere e scartare il surnatante e lavare il DNA pellet con 1 ml di acqua refrigerata etanolo al 70%, aria secca per 10 minuti, e risospendere nel 30-50 microlitri TE o molecolare dell'acqua grado. Facilitare la risospensione del DNA da riscaldamento a 65 ° C per 5 minuti e il DNA Conservare a -80 ° C.
3. DNA purezza, concentrazione e determinazione Dimensione:
- Determinare la qualità del DNA misurando la A 260/280 e 260/230 Un rapporto con un NanoDrop 1000 spettrofotometro o qualsiasi altra strumentazione appropriata.
- Determinare la concentrazione di DNA utilizzando Quant-iT kit di analisi dsDNA secondo le istruzioni del produttore.
- Determinare le dimensioni del DNA tramite elettroforesi impulso gel. Preparare 1% gel con TBE 0.5X e utilizzare i seguenti parametri sul Mapper XA CHEF System; Interruttore orario iniziale di 0,35 s, un tempo di passaggio finale di 7,67 s, e 120 ° incluso angolo per un dislivello di 6,0 V / cm a lineare ramping fattore. Macchia gel con 1 x SYBR Green I per 30 min. Carico tra 700-1000 ng di DNA totale su gel. Vedi fig. 3 per una forma condensata del protocollo completo.
Rappresentante dei risultati:
Celle di estrazione:
Sequenziale estratti di cellule microbiche (surnatanti) hanno dimostrato che la torbidità di estratti diminuisce il numero di aumento di estrazioni. Ciò suggerisce una riduzione del numero di cellule a seguito di estrazione ogni cella successiva. Importante da notare qui è che, come ogni ulteriore fase di estrazione delle cellule offre l'opportunità per l'introduzione di contaminanti, il numero di estrazioni cella dovrebbe essere ridotto al minimo in modo che il finale pellet cellulare è rappresentativo della comunità microbica totale. Altre fonti di contaminazione sono stati controllati con l'adozione di adeguate tecniche di laboratorio sterile. Per esempio, le soluzioni sono state preparate in autoclave acqua deionizzata e filtro sterilizzato. I contenitori sono stati sterilizzati con alcol, in autoclave, e trattati alla luce UV ed reticolante ultravioletti.
DNA, la concentrazione e la determinazione di qualità:
Il protocollo ha dato circa 2 mg di HMW DNA (35-50 kb) (Fig. 4) per grammo di campione tappeto con un 260/280 Un rapporto di 1,6, e una A 260/230 rapporto di 0,7 (Tabella 1). Anche se la A 260/230 rapporto sembra essere bassa, non l'inibizione della valle molecolare-based come la PCR-amplificazione del gene 16S rRNA è stata osservata in uno studio separato 7. E 'importante notare che il DNA da hypersaline stuoie ha due principali fonti di contaminazione; EPS e sali. E 'quindi possibile che le tracce di questi contaminanti possono influenzare la A 260/230 rapporti nonostante gli enormi sforzi per ridurre i loro effetti sulle applicazioni a valle.
DNA stabilire la misura:
Elettroforesi su gel campo Pulse utilizza un flusso pulsante con intermittente interruttori direzionale con conseguente striscio DNA, come mostrato in figura 3. Il nostro protocollo ha prodotto un DNA HMW di circa 35-50 kb. Mentre alcune dimensioni del DNA può essere inferiore a 30 kb, è importante avere una parte dello striscio sopra i 35 kb, dato che la clonazione fosmid richiede ~ 40 kb inserti di DNA e frammenti di DNA più grandi offrono un maggiore accesso alle vie biosintetiche intatto. Nei nostri studi, lo striscio DNA superiori a 35 kb è stato asportato e purificato per la grande inserto clonazione vettoriale e altre applicazioni molecolare.

Figura 1. Ipersalina microbica campionamento sito mat (Big Pond) che si trova su Eleuthera, Bahamas.

Figura 2. Una sezione del tappeto microbica hypersaline utilizzato in questo studio. Il tappetino microbica è stato ottenuto da uno stagno hypersaline situato su Eleuthera, Bahamas.
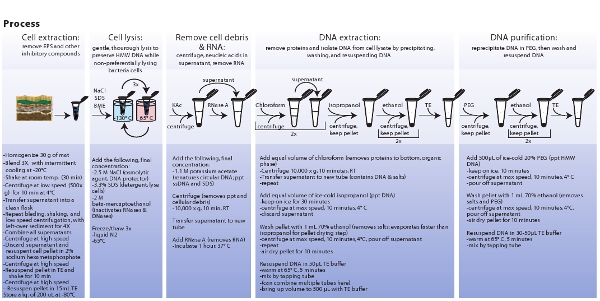
Figura 3. Rappresentazione schematica di procedure necessarie per l'estrazione delle cellule microbiche, la lisi cellulare, estrazione e purificazione del DNA metagenomica. Clicca qui per visualizzare l'immagine ingrandita.

Figura 4. Caratterizzazione peso molecolare del DNA estratto mediante elettroforesi su gel campo di impulsi. Corsia M è un marker HMW, e corsie 1 e 2 sono repliche di metagenomica DNA estratto dal materassino Eleuthera hypersaline utilizzando il protocollo di cui sopra.
| Metodo di estrazione | Concentrazione ng / g | A 260/280 | A 260/230 | |
| PEG | Rep 1 | 1806,1 | 1,64 | 0,76 |
| Rep 2 | 2010,8 | 1,61 | 0,75 |
Tabella 1. Misurazione della concentrazione e della qualità del DNA estratto da microbica tappetino hypersaline.
Access restricted. Please log in or start a trial to view this content.
Discussione
Dato che la rimozione totale di cellule da campioni microbiche complesse e molto diverse tappetino non è pratico, la preoccupazione principale è come le cellule estratte rappresentano la complessiva comunità microbica tappeto. In uno studio precedente, PCR-DGGE analisi dei geni rRNA 16S microbica ha mostrato che le cinque fasi di rimozione delle cellule utilizzate in questo protocollo estratti di cellule che sono rappresentativi della comunità microbica complessiva tappeto 7. Il numero effettivo di estraz...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Riconoscimenti
Questo lavoro è stato finanziato dal Programma National Science Foundation Environmental Genomica (Grant No. EF-0723707).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti |
|---|---|---|---|
| β-mercaptoetanolo | Sigma-Aldrich | M3148 | |
| Polietilene glicole 8000 | Promega | V3011 | 20% in 1,2 M di NaCl |
| Acetato di potassio | Fisher Scientific | Fisher Scientific | |
| Quant-iT Assay kit dsDNA | Invitrogen | Q33130 | |
| RNasi | Epicentre | MRNA092 | |
| Cloruro di sodio | Prodotti chimici BDH | BDH8014 | Conc appropriato. |
| Sodio dodecil solfato | Fisher Scientific | 03-500-509 | 10% in acqua |
| sodio esametafosfato | EMD chimici | SX0583-3 | 2% in acqua |
| TBE | Fisher Scientific | BP1333-1 | |
| CHEF Mapper XA sistema | Bio-Rad Laboratories | 170-3670 | |
| NanoDrop 1000 spettrofotometro | Thermo Scientific | ND-1000 | |
| Vortex | Scientific Industries Inc. | ||
| Crosslinker ultravioletti | UVP | ||
| Waring Blender | Waring laboratorio | LB10S |
Riferimenti
- Decho, A. W. Microbial biofilms in intertidal systems: an overview. Cont. Shelf Res. 20, 1257-1273 (2000).
- Dupraz, C., Visscher, P. T. Microbial lithification in marine stromatolites and hypersaline mats. Trends Microbiol. 13, 429-438 (2005).
- Steffan, R. J., Goksoyr, J., Boj, A. K., Atlas, R. M. Recovery of DNA from soils and sediments. Appl. Environ. Microbiol. 54, 2908-2915 (1988).
- Lee, Y. K., Kim, H. W., Liu, C. L., Lee, H. K. A simple method for DNA extraction from marine bacteria that produce extracellular materials. J. Microbiol. Methods. 52, 245-250 (2003).
- Roose-Amsaleg, C. L., Garnier-Sillam, E., Harry, M. Extraction and Purification of Microbial DNA from Soil and Sediment Samples. Appl. Soil Ecol. 18, 47-60 (2001).
- de Lipthay, J. R., Enzinger, C., Johnsen, K., Aamand, J., Sørensen, S. J. Impact of DNA extraction method on bacterial community composition measured by denaturing gradient gel electrophoresis. Soil Biol. Biochem. 36, 1607-1614 (2004).
- Bey, B. S., Fichot, E. B., Dayama, G., Decho, A. W., Norman, R. S. Extraction of High Molecular Weight DNA from Microbial Mats. Biotechniques. 49, 631-640 (2010).
- Kakirde, S. K., Parsley, L. C., Liles, M. R. Size does matter: Application-driven approaches for soil metagenomics. Soil Biology & Biochemistry. 42, 1911-1923 (2010).
- Rodon, M. R., August, P. R., Bettermann, A. D., Brady, S. F., Grossman, T. H., Liles, M. R., Loiacono, K. A., Lynch, B. A., MacNeil, I. A., Minor, C., Tiong, C. L., Gilman, M., Osburne, M. S., Clardy, J., Handelsman, J., Goodman, R. M. Cloning the Soil Metagenomic: A Strategy for Accessing the Genetic and Functional Diversity of Uncultured Microorganisms. Appl. Environ. Microbiol. 66, 2541-2547 (2000).
- Beja, O., Aravind, L., Koonin, E. V., Suzuki, M. T., Hadd, A., Nguyen, L. P., Jovanovich, S. B., Gates, C. M., Feldman, R. A., Spudich, J. L., Spudich, E. N., DeLong, E. F. Bacterial Rhodopsin: Evidence for a New Type of Phototrophy in the Sea. Science. 289, 1902-1906 (2000).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon