JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
미생물 매트에서 높은 분자량의 DNA의 추출
요약
우리는 hypersaline 미생물 매트에서 높은 분자량의 DNA를 추출하기위한 개선된 프로토콜을 제공합니다. 미생물 세포는 DNA 추출 및 정화하기 전에 매트 매트릭스에서 분리됩니다. 이것은 농도, 품질 및 DNA의 크기를 향상시킵니다. 프로토콜은 다른 내화물 샘플에 사용할 수 있습니다.
초록
metagenomic 데이터의 성공적인 정확한 분석과 해석은 고품질, 높은 분자량 (HMW) 커뮤니티 DNA의 추출 효율에 의존적일 수 밖에 없습니다. 그러나, 환경 매트 샘플은 종종 높은 품질 HMW DNA의 큰 농도를 구하기 위해 어려움을 제기. Hypersaline의 미생물 매트 높은 세포외 고분자 물질의 양의 (EPS) 1 추출한 DNA의 다운 스트림 애플 리케이션을 억제 수 염분이 포함되어 있습니다. 직접적이고 거친 방법은 자주 내화물 샘플에서 DNA 추출에 사용됩니다. 매트, 접착제 매트릭스에 EPS 직접 용해하는 동안 DNA 2,3를 바인딩하기 때문에 이러한 방법은 일반적으로 사용됩니다. 엄격한 추출 방법의 결과로, DNA되고 작은 크기 4,5,6으로 조각.
DNA 따라서 대형 삽입 벡터 복제에 대한 부적 절한됩니다. 이러한 제한을 우회하기 위해, 우리는 hypersaline 미생물 매트에서 좋은 품질과 수량의 HMW DNA를 추출하는 개선된 방법을보고합니다. 우리는 혼합 및 차동 원심 분리를 통해 배경 매트 매트릭스에서 미생물 세포의 분리와 관련된 간접적인 방법을 고용. 기계적 및 화학적 절차 조합이 추출된 미생물 세포에서 DNA를 추출하고 정화하는 데 사용되었다. 우리 프로토콜 1.6 280분의 260의 비율로, 매트 샘플 g 당 HMW DNA (35~50킬로바이트)의 약 2 μg을 산출. 또한, 16 rRNA 유전자 7 증폭이 프로토콜은 오염 물질의 억제 효과를 최소화하거나 제거할 수 있다고 제안합니다. 우리의 결과는 기능 metagenomic 연구 미생물 매트에서 HMW의 DNA의 추출을위한 적절한 방법을 제공하고 DNA 추출이 어려운있는에서 다른 환경 시료에 적용할 수 있습니다.
프로토콜
1. 미생물 세포 추출 :
- 철저하게 혼합하여 멸균 분쇄 유봉과 미생물 매트를 균질. 약 워링 믹서기의 멸균 용기에 무균 매트 재료의 모든 30g (서부 유럽 표준시 무게), 1 분 1 M NaCl (또는 사용 예제 특정 농도) 100 ML에 대한 추가하고, 중간 속도로 세 번 조화를 배치 1 분 -20 ° C의 냉동고에 간헐적으로 냉각. 250 ML의 원심 분리기 병에 슬러리를 전송하고 1 M NaCl과 함께 남아있는 빈 볼륨을 작성하십시오. autoclaved DI 워터의 NaCl 용액을 준비하고 소독 필터.
- 또한 30 분 실온에서 vortexer (소용돌이 - 지니 R 2)를 사용하여 (150 RPM) 흔들림으로 모체의 미생물 세포를 이동시키다.
- 4 15 분 ° C. 낮은 속도 (500x g)에서 원심 분리기 부드럽게 퇴적물에 최소한의 방해와 플라스크에 뜨는를 전송합니다.
- pelleted 퇴적물을 사용하면, 단계 1.3의 끝 부분에 각각의 퇴적물 펠렛 신선한 NaCl의 추가와 함께 1.2 1.3 4 개의 추가 시간을 혼합하여 단계를 반복합니다. 각 NaCl 추출에서 뜨는은 신선한 플라스크로 전송됩니다.
- 4 15 분에 대한 고속 (25,000 XG) 200 ML의 aliquots에 5 세포 extractions 및 원심 분리기에서 supernatants을 결합 ° C. 뜨는을 취소, 2 % 나트륨 hexametaphosphate 10 ML 각 세포 펠렛을 resuspend, 알약을 결합하고 2 % 나트륨 hexametaphosphate 200 ML에 볼륨을합니다. 30 분 상온에서 (150 RPM)에 의해 세포를 흔들어 씻으십시오. 이 단계의 끝에서 당신은 다음 단계로 진행하는 세포 중 하나만 튜브가 있어야합니다.
- 4 15 분 ° C.에 대한 고속 (25,000 XG)에서 원심 분리기 TE (50 MM EDTA (에틸렌 다이아 민 테트라 초산)) 200 ML에 뜨는, resuspend 세포 펠렛을 무시, 10 분 상온에서 (150 RPM)에 의해 세포를 흔들어 씻는다.
- 4 15 분에 대한 고속 (25,000 XG)에서 원심 분리기 ° C가, 15 ML TE (10 MM EDTA (에틸렌 다이아 민 테트라 초산))에 resuspend 뜨는을 폐기하고, -80에서 보관 200 μL aliquots ° C까지 DNA extractions 필요.
2. DNA 추출 및 정제 :
- 200 μL 5 M NaCl, 200 μL 10% SDS와 200 μL 미생물 세포 나누어지는 100 μL 14.3 M의 β - 메르 캅 토 에탄올을 추가합니다. 반전 4-6 배 부드럽게 섞는다.
- 5 분 65 ° C 물 욕조에서 해동 다음 2 분에 대한 액화 질소에 잠수함이 잠수하여 동결 - 해동의 3 라운드에 따라 혼합물. 최종 10 분에 해동 연장.
- 200 μL 5 M의 칼륨 아세테이트 (산도 5.5) 10 분 얼음에 장소를 추가합니다. 4 10 분 10,000 XG에 원심 분리기 ° C와 넓은 구멍 피펫 팁을 사용하여 새로운 튜브에 뜨는을 전송하기만하면됩니다. 1 시간을 위해 RNase 37과 부화는 ° C의 3 μL를 추가합니다.
- 클로로포름의 동일한 음량을 추가, 역전에 의해 잠시 흔들하고, 실온에서 10 분 15,000 XG에 원심 분리기.
- 신중하게 넓은 구멍 피펫 팁을 사용하여 새로운 튜브에 뜨는의 상단에 레이어를 전송합니다. 상단 및 하단 레이어 사이에있는 흰색 인터페이스 레이어를 pipetting하지 마십시오. 클로로포름 - 추출 절차를 반복합니다.
- 냉장의 동일한 음량 (-20에서 ° C) 촉진의 DNA로 30 분 얼음에 뜨는와 부화 이소프로판올. 추가
- 4 10 분 최대 속도 (20,800 XG)에서 원심 분리기 ° C 펠렛 DNA 있습니다.
- 냉장 1 ML 70 % 에탄올 (-20 ° C에서)로 뜨는 및 세척 DNA 펠렛 폐기하십시오.
- 4 10 분 최대 속도 (20,800 XG) ° C에서 원심은, 표면에 뜨는를 버리고 10 분 70 %의 에탄올 세척 및 공기 건조 DNA 펠렛를 반복합니다. 70 % 에탄올 세척을 반복합니다.
- 25 μL TE (10 MM EDTA (에틸렌 다이아 민 테트라 초산))에 Resuspend의 DNA, 65 ° 따뜻한 5 분 C는 여러 개의 튜브가 사용되었습니다면 한 튜브에 결합하고, TE 500 μL에 볼륨을 구성합니다.
- 추가 500 μL 얼음처럼 차가운 (또는 4 ° C) 20 %의 폴리에틸렌 글리콜 (PEG) (1.2 M NaCl에서 준비). 펠렛 DNA 4 10 분 최대 속도로 10 분, 그리고 원심 분리기를위한 얼음에 품어 역전, ° C에 의해 부드럽게 섞는다. 제거하고 폐기 뜨는하고 냉장 70 % ethanol, 10 분 대한 항공 건기 1 mL로 pelleted DNA를 씻고, 그리고 30-50 μL TE or 분자 학년 물에 resuspend. 65 온난화에 의한 DNA의 resuspension을 촉진 ° -80시 5 분 및 저장 DNA에 대한 C ° C.
3. DNA 순도, 농도 및 크기 결정 :
- 280분의 260과 Nanodrop 1000 분광 광도계 또는 다른 적절한 장비를 사용하여 230분의 260 비율을 측정하여 DNA의 품질을 확인합니다.
- 제조 업체의 지시에 따라 퀀트 - IT dsDNA 분석 키트를 사용하여 DNA 농도를 결정합니다.
- 펄스 필드 겔 전기 영동을 사용하여 DNA의 크기를 결정합니다. 0.5X TBE와 1퍼센트 아가로 오스 겔을 준비하고 다음 주방장 매퍼 XA 시스템에 대한 매개 변수를 사용; 0.35 s의 초기 스위치 시간, 7.67 s의 마지막 스위치 시간, 120 °는 다음 위치에서 6.0 V / cm의 그라데이션에 대한 각도를 포함 리니어 R요소를 좋아지고. 30 분 1 X SYBR 그린 I와 젤 얼룩. 겔에 700-1000 NG 총 DNA 사이에로드합니다. 그림을 참조하십시오. 전체 프로토콜의 응축 형태 3.
대표 결과 :
세포 추출 :
순차 미생물 세포 추출물은 (supernatants) 추출물의 탁도가 extractions 증가의 숫자로 감소하는 것으로 나타났습니다. 이것은 각각의 연속된 세포 추출 다음과 같은 세포의 수를 감소를 제안합니다. 여기서주의해야할 중요 각 추가 셀 추출 단계는 오염 물질 도입에 대한 기회를 제공으로, 휴대 extractions의 번호가 최종 세포 펠렛은 전체 미생물 커뮤니티의 대표되도록 최소화해야한다는 것입니다. 오염의 다른 소스는 적절한 실험실 멸균 기술을 채택하여 제어했습니다. 예를 들어, 솔루션은 autoclaved DI 물의 준비와 필터를 소독했다. 컨테이너는 알코올로 소독 autoclaved하고, 자외선과 자외선 crosslinker에 따라 치료를했다.
DNA 농도 및 품질 결정 :
프로토콜은 1.6 280분의 260 비율로 매트 샘플 g 당 HMW DNA (35-50킬로바이트) (그림 4)의 약 2 μg을 굴복하고, 0.7의 230분의 260 비율 (표 1). 230분의 260 비율이 같은 rRNA 유전자는 별도의 연구 7 관찰했던 16의 PCR - 증폭 등 하류 분자 기반 응용 프로그램에 대한 억제 낮은하지 나타나지만. EPS와 소금, hypersaline 매트에서 해당 DNA 오염의 두 가지 주요 출처가에 유의하는 것이 중요합니다. 그것은 이러한 오염 물질의 흔적은 다운 스트림 애플 리케이션에 미치는 영향을 줄이기 위해 엄청난 노력에도 불구하고 230분의 260 비율에 영향을 미치는있을 수 있으므로 가능합니다.
DNA 크기 결정 :
펄스 필드 겔 전기 영동은 그림 3과 같이 DNA의 얼룩의 발생 간헐적인 방향 스위치와 pulsating 전류 흐름을 사용합니다. 우리 프로토콜은 약 35-50킬로바이트의 HMW DNA를 나왔고. 일부 DNA 크기가 30킬로바이트보다 작아야 수 있지만, 그것은 fosmid 복제가 ~ 40킬로바이트 DNA 삽입이 필요하며 큰 DNA 조각은 그대로 biosynthetic 경로에 큰 액세스를 제공하는 주어진 35킬로바이트 위의 얼룩의 일부를 가지고하는 것이 중요합니다. 우리의 연구에, 35킬로바이트 위의 DNA의 얼룩은 excised되었으며 대형 삽입 벡터 클로닝 및 기타 분자 애플 리케이션을위한 정화.

엘류테라, 바하마에있는 그림 1. Hypersaline의 미생물 매트 샘플링 사이트 (빅 폰드).

그림 2. 본 연구에서 사용되는 hypersaline 미생물 매트의 단면. 미생물 매트는 엘류테라, 바하마에있는 hypersaline 연못로부터 얻은 것입니다.
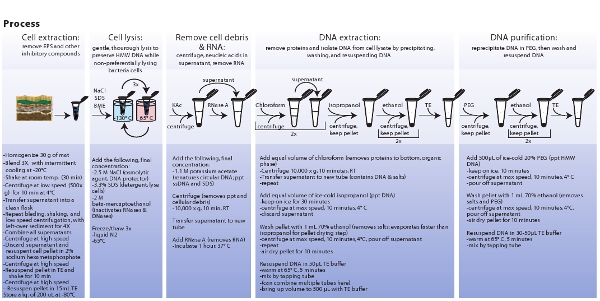
그림 3. 미생물의 세포 추출, 세포 용해, 추출, metagenomic DNA의 정화에 참여 절차 도식 표현은. 큰 이미지를 보려면 여기를 클릭하십시오.

그림 4. 펄스 필드 겔 전기 영동을 사용하여 추출한 DNA의 분자량 특성화. 레인 M은 HMW 마커이며, 골목길 1과 2는 위의 프로토콜을 사용 엘류테라 hypersaline 매트에서 추출한 DNA의 복제 metagenomic 있습니다.
| 추출 방법 | 농도 NG / G | 280분의 260 | 230분의 260 | |
| PEG | 담당자 1 | 1806.1 | 1.64 | 0.76 |
| 담당자 2 | 2010.8 | 1.61 | 0.75 |
표 1. DNA의 농도 및 품질 측정은 미생물의 hypersaline 매트에서 추출한.
Access restricted. Please log in or start a trial to view this content.
토론
복잡하고 매우 다양한 미생물 매트 샘플에서 전체 세포 제거 실용적 아니라는 것을 감안할 때, 기본 우려가 추출된 세포가 전체 미생물 매트 커뮤니티를 대표하는 얼마나 잘됩니다. 이전 연구에서는 미생물의 유전자 16 rRNA의 PCR - DGGE 분석이 프로토콜에 사용되는 다섯 셀 제거 단계를 전체 미생물 매트 커뮤니티 7 대표하는 세포를 추출 것으로 나타났다. 세포 추출의 실제 번호는 전체 미?...
Access restricted. Please log in or start a trial to view this content.
공개
감사의 말
이 작품은 국립 과학 재단 (National Science Foundation) 환경 게놈 프로그램 (그랜트 번호 EF - 0723707)에 의해 재정 지원되었다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 시약의 이름 | 회사 | 카탈로그 번호 | 댓글 |
|---|---|---|---|
| β - 메르 캅 토 에탄올 | 시그마 - 알드리치 | M3148 | |
| 폴리에틸렌 글리콜 8000 | Promega | V3011 | 1.2 M NaCl에 최고 20 % |
| 칼륨 아세테이트 | 피셔 사이 언티픽 | 피셔 사이 언티픽 | |
| 퀀트 - IT dsDNA 분석 키트 | Invitrogen | Q33130 | |
| RNase | Epicentre | MRNA092 | |
| 나트륨 염화물 | BDH 화학 | BDH8014 | 적절한 CONC. |
| 나트륨 Dodecyl의 황산 | 피셔 사이 언티픽 | 03-500-509 | 물에서 10 % |
| 나트륨 hexametaphosphate | EMD 화학 | SX0583 - 3 | 물에 2% |
| TBE | 피셔 사이 언티픽 | BP1333 - 1 | |
| 요리사 매퍼 XA 시스템 | 바이오 래드 연구소 | 170-3670 | |
| NanoDrop 1000 분광 광도계 | 써모 과학 | ND - 1000 | |
| Vortexer | 과학 산업 주식 회사 | ||
| 자외선 Crosslinker | UVP | ||
| 워링 믹서기 | 워링 실험실 | LB10S |
참고문헌
- Decho, A. W. Microbial biofilms in intertidal systems: an overview. Cont. Shelf Res. 20, 1257-1273 (2000).
- Dupraz, C., Visscher, P. T. Microbial lithification in marine stromatolites and hypersaline mats. Trends Microbiol. 13, 429-438 (2005).
- Steffan, R. J., Goksoyr, J., Boj, A. K., Atlas, R. M. Recovery of DNA from soils and sediments. Appl. Environ. Microbiol. 54, 2908-2915 (1988).
- Lee, Y. K., Kim, H. W., Liu, C. L., Lee, H. K. A simple method for DNA extraction from marine bacteria that produce extracellular materials. J. Microbiol. Methods. 52, 245-250 (2003).
- Roose-Amsaleg, C. L., Garnier-Sillam, E., Harry, M. Extraction and Purification of Microbial DNA from Soil and Sediment Samples. Appl. Soil Ecol. 18, 47-60 (2001).
- de Lipthay, J. R., Enzinger, C., Johnsen, K., Aamand, J., Sørensen, S. J. Impact of DNA extraction method on bacterial community composition measured by denaturing gradient gel electrophoresis. Soil Biol. Biochem. 36, 1607-1614 (2004).
- Bey, B. S., Fichot, E. B., Dayama, G., Decho, A. W., Norman, R. S. Extraction of High Molecular Weight DNA from Microbial Mats. Biotechniques. 49, 631-640 (2010).
- Kakirde, S. K., Parsley, L. C., Liles, M. R. Size does matter: Application-driven approaches for soil metagenomics. Soil Biology & Biochemistry. 42, 1911-1923 (2010).
- Rodon, M. R., August, P. R., Bettermann, A. D., Brady, S. F., Grossman, T. H., Liles, M. R., Loiacono, K. A., Lynch, B. A., MacNeil, I. A., Minor, C., Tiong, C. L., Gilman, M., Osburne, M. S., Clardy, J., Handelsman, J., Goodman, R. M. Cloning the Soil Metagenomic: A Strategy for Accessing the Genetic and Functional Diversity of Uncultured Microorganisms. Appl. Environ. Microbiol. 66, 2541-2547 (2000).
- Beja, O., Aravind, L., Koonin, E. V., Suzuki, M. T., Hadd, A., Nguyen, L. P., Jovanovich, S. B., Gates, C. M., Feldman, R. A., Spudich, J. L., Spudich, E. N., DeLong, E. F. Bacterial Rhodopsin: Evidence for a New Type of Phototrophy in the Sea. Science. 289, 1902-1906 (2000).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유