Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Mikrobiyal Paspas Yüksek Moleküler Ağırlıklı DNA çıkarımı
Bu Makalede
Özet
Biz hypersaline mikrobiyal paspaslar yüksek molekül ağırlıklı DNA ayıklamak için geliştirilmiş bir protokol sağlar. Mikrobiyal hücrelerin DNA ekstraksiyonu ve saflaştırma mat matris önce ayrılır. Bu konsantrasyonları, kalite ve DNA boyutunu artırır. Bu protokol, diğer refrakter numuneler için kullanılabilir.
Özet
Metagenomic veri Başarılı ve doğru analiz ve yorumlama, yüksek kaliteli, yüksek molekül ağırlığı (HMW) topluluk DNA verimli çıkarma bağımlı. Ancak, çevre mat örnekleri, genellikle yüksek kalite, HMW DNA yüksek konsantrasyonlarda elde etmek için zorluklar teşkil. Hypersaline mikrobiyal paspaslar, yüksek miktarda ekstraselüler polimerik maddelerin (EPS) 1. ve çıkarılan DNA alt uygulamalar inhibe edebilir tuzları içerir. Doğrudan ve sert yöntemleri genellikle refrakter örneklerinden DNA ekstraksiyonu kullanılır. Bu yöntemler, paspaslar, yapışkan bir matris, EPS direkt lizis sırasında DNA 2,3 bağlar, çünkü genellikle kullanılır . Sert ekstraksiyon yöntemlerinin bir sonucu olarak, küçük boyutları 4,5,6 parçalanmış DNA olur.
DNA, bu nedenle büyük eklemek vektör klonlama için uygun olur. Bu sınırlamaları aşmak için, biz hypersaline mikrobiyal paspaslar iyi kalite ve kantite HMW DNA ayıklamak için geliştirilmiş bir yöntem rapor. Biz karıştırma ve diferansiyel santrifüj yoluyla arka plan mat matris mikrobiyal hücrelerin ayrılması içeren dolaylı bir yöntem kullandı. Ayıklanan mikrobiyal hücrelerin DNA özü ve arındırmak için mekanik ve kimyasal işlemler bir arada kullanıldı. Bizim protokol 1,6 A 260/280 oranı, HMW DNA (35-50 kb) mat örnek gram başına yaklaşık 2 mikrogram verir. Ayrıca, 16S rRNA genleri 7 amplifikasyonu protokol bulaşanların herhangi bir inhibitör etkilerini en aza indirmek veya ortadan kaldırmak mümkün olduğunu göstermektedir . Bizim sonuçlarımız Fonksiyonel metagenomic çalışmalar için mikrobiyal paspaslar HMW DNA ekstraksiyonu için uygun bir yöntem sağlar ve diğer çevre örnekleri DNA ekstraksiyonu zorlu olduğu için geçerli olabilir.
Protokol
1. Mikrobiyal Hücre Ekstraksiyon:
- Mikrobiyal paspaslar steril bir taşlama havaneli ile iyice karıştırılarak homojenize. Yaklaşık 1 dakika süreyle homojenize mat malzeme steril kap içine Waring blender bütün 30 g (ıslak ağırlık), yaklaşık 100 mL 1 M NaCl (ya da örnek kullanım için belirli bir konsantrasyon) eklemek, üç kez ve orta hızda karışımı yerleştirin. 1 dakika boyunca -20 ° C derin dondurucuda aralıklı soğutma. Bulamaç 250 ml santrifüj şişeye aktarın ve kalan boş hacim 1 M NaCl ile doldurun. Otoklava DI su NaCl çözeltisi hazırlayın ve sterilize filtre.
- Ayrıca 30 dakika oda sıcaklığında bir vortexer (Vortex-Genie R 2) (150 rpm) sallayarak matris mikrobiyal hücrelerin yerinden.
- 15 dakika 4 ° C düşük hızlı (500x g) Santrifüj Tortu minimum rahatsızlık ile hafifçe bir balona süpernatantı aktarın.
- Pelet tortu kullanarak 1.2 ve 1.3, 1.3 adım sonunda her tortu pelet taze NaCl ilavesi ile dört kez karıştırma ve adımları tekrarlayın. Her NaCl çıkartılmasından süpernatant taze bir balona transfer edilir.
- 15 dakika yüksek hızda (25.000 xg) 200 ml hacimde 5 hücre ekstraksiyon ve santrifüj süpernatantlar birleştirin 4 ° C Süpernatantı atın, 10 ml% 2 sodyum hexametaphosphate her hücre pelletini tekrar süspansiyon, pelet birleştirmek ve% 2 sodyum hexametaphosphate ile 200 mL birimi oluşturacak. 30 dakika oda sıcaklığında (150 rpm) sallayarak hücreleri yıkayın. Bu adımın sonunda bir sonraki adıma geçmek için hücrelerin sadece bir tüp olmalıdır.
- 15 dakika 4 ° C yüksek hızda (25.000 xg) Santrifüj TE (50 mM EDTA), 200 ml supernatant, tekrar süspansiyon hücre pelletini atın ve 10 dakika oda sıcaklığında (150 rpm) sallayarak hücreleri yıkayın.
- 4 az 15 dakika yüksek hızda (25.000 xg) Santrifüj ° C, 15 ml TE (10 mM EDTA) tekrar süspansiyon süpernatant, atmak ve mağaza 200 mcL alikotları -80 ° C kadar DNA ekstraksiyon için gerekli.
2. DNA Ayırma ve Saflaştırma:
- 200 mcL 5 M NaCl, 200 mcL% 10 SDS ve 200 mcL mikrobiyal hücre kısım 100 mcL 14.3 M β-mercaptoethanol ekleyin. Tersini 4-6 kez hafifçe karıştırın.
- , 2 dakika, 5 dakika boyunca 65 ° C su banyosu içinde çözülme takip sıvı nitrojen içinde daldırarak donma-çözülme 3 tur Konu karışımı. Son 10 dakika çözülme uzatın.
- 200 mcL 5 M potasyum asetat (pH 5.5) ve 10 dakika süreyle buz yer ekleyin. Santrifüj 10,000 xg'de 10 dakika 4 ° C ve geniş çaplı bir pipet kullanarak yeni bir tüp süpernatantı aktarın. 1hr RNaz 37 A ve inkübe ° C 3 mcL ekleyin.
- Eşit miktarda kloroform, inversiyon kısaca sallamak ve oda sıcaklığında 10 dakika 15.000 xg'de santrifüj.
- Süpernatant üst tabakası, geniş çaplı bir pipet kullanarak yeni bir tüp içine dikkatlice aktarın. Üst ve alt katmanları arasında yer alan beyaz arabirim katmanı pipetleme kaçının. Kloroform-çıkarma prosedürünü tekrarlayın.
- Soğutulmuş, eşit hacimde (-20 ° C) çökelti DNA 30 dakika buz üzerinde inkübe supernatant ve izopropanol.
- 4 az 10 dakika süreyle maksimum hızı (20,800 xg) Santrifüj ° C pelet DNA.
- 1 ml soğuk (-20 ° C)% 70 etanol ile süpernatant ve yıkama DNA pelet atın.
- Maksimum hız (20,800 xg) 10 dakika 4 ° C santrifüjleyin, süpernatantı atmak, 10 dakika süreyle% 70 etanol yıkama ve kuru hava DNA pelet tekrarlayın. % 70 etanol yıkama tekrarlayın.
- 25 mcL TE (10 mM EDTA) yeniden süspanse DNA, 65 yaşında sıcak ° C 5 dakika boyunca birden fazla tüpler varsa bir tüp içine birleştirmek ve TE 500 mcL hacim makyaj.
- Ekle 500 mcL buz (veya 4 ° C)% 20 polietilen glikol (PEG) (1.2 M NaCl hazırlanan). Pelet DNA 4, 10 dakika süreyle maksimum hızda 10 dakika ve santrifüj buz üzerinde inkübe inversiyon ° C ile hafifçe karıştırın. Süpernatantı çıkartın ve atın ve 1 ml soğutulmuş% 70 etanol, 10 dakika süreyle kurumaya ile pelet DNA yıkayın ve 30-50 mcL TE veya moleküler sınıf su tekrar süspansiyon haline getirin. 65 yaşında DNA tabanda ısınmanın kolaylaştırılması ° -80 ° C arasında 5 dakika ve Mağaza DNA
3. DNA Saflık, Konsantrasyon ve Boyut Belirlenmesi:
- A 260/280 ve 260/230 oranları Nanodrop 1000 spektrofotometre veya herhangi bir başka uygun enstrümantasyon kullanarak ölçerek DNA kalitesini belirlemek .
- Üreticinin talimatlarına göre Quant-iT dsDNA Assay kiti kullanılarak DNA konsantrasyonu belirleyin.
- Darbe alan jel elektroforezi kullanılarak DNA boyutunu belirlemek. 0.5x TBE% 1 agaroz jel hazırlayın ve aşağıdaki parametreleri CHEF Mapper XA Sistemi kullanmak; ilk geçiş süresi 0.35 saniye, 7.67 s son bir geçiş süresi ve 120 ° 6.0 V / cm bir degrade açısı doğrusal rfaktör amping. 30 dakika için 1 x SYBR Green I ile jel Leke. 700-1000 ng toplam DNA jel arasındaki yükleyin. Bkz: Şekil. 3 tam protokol yoğunlaştırılmış bir form için.
Temsilcisi Sonuçlar:
Hücre çıkarma:
Sıralı mikrobiyal hücre özleri (süpernatantlar) özler bulanıklık ekstraksiyon sayısı artış olarak azaldığını göstermiştir. Bu, birbirini izleyen her hücre çıkarma, hücre sayısında bir azalma göstermektedir. Burada dikkat edilmesi gereken önemli noktalar her ek hücre çıkarma adım kirletici tanıtımı için fırsat sağlar, hücre ekstraksiyon sayısı son hücre pelletini genel mikrobiyal topluluğun temsilcisi böylece minimize olması gerektiğini. Diğer kirlenme kaynakları yeterli steril laboratuar teknikleri benimseyerek kontrol altına alındı. Örneğin, çözümler otoklavlanmış DI su hazırlanır ve steril filtre. Konteynerler, alkol ile sterilize otoklava ve UV ışığı ve ultraviyole çapraz bağlayıcı altında tedavi edildi.
DNA konsantrasyonu ve kalite belirlenmesi:
1,6 A 260/280 oranı ile protokol mat örnek gram başına yaklaşık 2 mikrogram HMW DNA (35-50 kb) (Şekil 4) vermiştir ve 0,7 A 260/230 oranı (Tablo 1). A 260/230 oranının düşük olması, 16S rRNA genleri ayrı bir çalışmada 7 gözlendi PCR amplifikasyon olarak mansap moleküler tabanlı uygulama inhibisyonu gibi görünse de. EPS ve tuzları; hypersaline paspaslar DNA'nın iki ana kaynakları kirlenme dikkat etmek önemlidir. Bu nedenle bu kirleticilerin izlerini aşağı akışı uygulamaları üzerindeki etkilerini azaltmak için büyük çabalara rağmen A 260/230 oranları etkileyen olabilir.
DNA büyüklükleri belirlenmesi:
Darbe alan jel elektroforezi Şekil 3'te gösterildiği gibi bir DNA smear sonucu aralıklı yönlü anahtarları ile zonklayan akım kullanır. Protokolü, yaklaşık 35-50 kb HMW DNA vermiştir. Bazı DNA boyutu 30 kb daha küçük olsa da, fosmid klonlama ~ 40 kb DNA ekler gerektirir ve daha büyük DNA parçalarının sağlam biyosentetik yollar için daha fazla erişim sağlayan 35 kb Yukarıdaki smear parçası olması önemlidir. Çalışmalarda, 35 kb Yukarıdaki DNA smear eksize edildi ve büyük takın vektör klonlama ve diğer moleküler uygulamalar için saflaştırılmış.

Şekil 1: Eleuthera, Bahamalar üzerinde bulunan Hypersaline mikrobiyal mat örnekleme sitesi (Big Pond).

Şekil 2'de bu çalışmada kullanılan hypersaline mikrobiyal mat bir kesit. Mikrobiyal mat Eleuthera, Bahamalar üzerinde bulunan bir hypersaline gölet elde edildi.
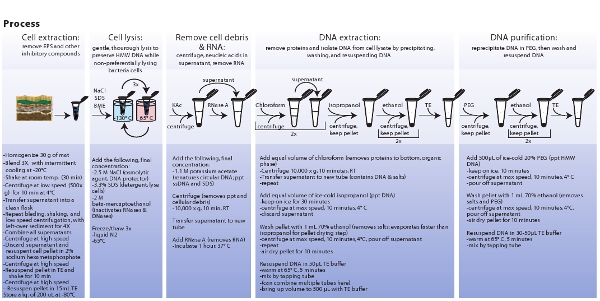
Şekil 3 şematik mikrobiyal hücre çıkarma, hücre parçalama, ekstraksiyon, metagenomic DNA saflaştırılması ve ilgili prosedürlerin daha büyük bir resim görmek için buraya tıklayın.

Şekil 4 darbe alan jel elektroforezi kullanarak çıkarılan DNA Molekül ağırlığı karakterizasyonu. Lane M HMW işaretleyici, 1 ve 2 şeritli Eleuthera hypersaline paspas yukarıdaki protokolü kullanılarak çıkarılan metagenomic DNA çoğaltır.
| Ekstraksiyon Metodu | Konsantrasyon ng / g | A 260/280 | A 260/230 | |
| PEG | Tecrübe 1 | 1806,1 | 1,64 | 0,76 |
| Tecrübe 2 | 2010,8 | 1,61 | 0,75 |
Tablo 1. DNA konsantrasyonu ve kalite ölçümü mikrobiyal hypersaline mat ayıklanır.
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Given that total cell removal from complex and highly diverse microbial mat samples is not practical, the primary concern is how well the extracted cells represent the overall microbial mat community. In a previous study, PCR-DGGE analysis of microbial 16S rRNA genes showed that the five cell removal steps used in this protocol extracts cells that are representative of the overall microbial mat community7. The actual number of cell extraction steps required to provide a cell pellet that is representative of th...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Teşekkürler
Bu çalışma, Ulusal Bilim Vakfı Çevre Genomik Programı (Hibe No EF-0.723.707) tarafından finanse edildi.
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Reaktif Adı | Şirket | Katalog numarası | Yorumlar |
|---|---|---|---|
| ß-mercaptoethanol | Sigma-Aldrich | M3148 | |
| Polietilen glikol 8000 | Promega | V3011 | 1.2 M NaCl içinde% 20 |
| Potasyum asetat | Fisher Scientific | Fisher Scientific | |
| Quant-iT dsDNA Assay kiti | Invitrogen | Q33130 | |
| RNaz | Epicentre | MRNA092 | |
| Sodyum klorür | BDH Kimyasallar | BDH8014 | Uygun kons. |
| Sodyum dodesil Sülfat | Fisher Scientific | 03-500-509 | % 10 su |
| sodyum hexametaphosphate | Merck Kimyasallar | SX0583-3 | Su içinde% 2 |
| TBE | Fisher Scientific | BP1333-1 | |
| CHEF Mapper XA Sistemi | Bio-Rad Laboratories | 170-3670 | |
| NanoDrop 1000 spektrofotometre | Thermo Scientific | ND-1000 | |
| Vortexer | Bilimsel Industries Inc. | ||
| Ultraviyole Çapraz | UVP | ||
| Waring blender | Waring laboratuvar | LB10S |
Referanslar
- Decho, A. W. Microbial biofilms in intertidal systems: an overview. Cont. Shelf Res. 20, 1257-1273 (2000).
- Dupraz, C., Visscher, P. T. Microbial lithification in marine stromatolites and hypersaline mats. Trends Microbiol. 13, 429-438 (2005).
- Steffan, R. J., Goksoyr, J., Boj, A. K., Atlas, R. M. Recovery of DNA from soils and sediments. Appl. Environ. Microbiol. 54, 2908-2915 (1988).
- Lee, Y. K., Kim, H. W., Liu, C. L., Lee, H. K. A simple method for DNA extraction from marine bacteria that produce extracellular materials. J. Microbiol. Methods. 52, 245-250 (2003).
- Roose-Amsaleg, C. L., Garnier-Sillam, E., Harry, M. Extraction and Purification of Microbial DNA from Soil and Sediment Samples. Appl. Soil Ecol. 18, 47-60 (2001).
- de Lipthay, J. R., Enzinger, C., Johnsen, K., Aamand, J., Sørensen, S. J. Impact of DNA extraction method on bacterial community composition measured by denaturing gradient gel electrophoresis. Soil Biol. Biochem. 36, 1607-1614 (2004).
- Bey, B. S., Fichot, E. B., Dayama, G., Decho, A. W., Norman, R. S. Extraction of High Molecular Weight DNA from Microbial Mats. Biotechniques. 49, 631-640 (2010).
- Kakirde, S. K., Parsley, L. C., Liles, M. R. Size does matter: Application-driven approaches for soil metagenomics. Soil Biology & Biochemistry. 42, 1911-1923 (2010).
- Rodon, M. R., August, P. R., Bettermann, A. D., Brady, S. F., Grossman, T. H., Liles, M. R., Loiacono, K. A., Lynch, B. A., MacNeil, I. A., Minor, C., Tiong, C. L., Gilman, M., Osburne, M. S., Clardy, J., Handelsman, J., Goodman, R. M. Cloning the Soil Metagenomic: A Strategy for Accessing the Genetic and Functional Diversity of Uncultured Microorganisms. Appl. Environ. Microbiol. 66, 2541-2547 (2000).
- Beja, O., Aravind, L., Koonin, E. V., Suzuki, M. T., Hadd, A., Nguyen, L. P., Jovanovich, S. B., Gates, C. M., Feldman, R. A., Spudich, J. L., Spudich, E. N., DeLong, E. F. Bacterial Rhodopsin: Evidence for a New Type of Phototrophy in the Sea. Science. 289, 1902-1906 (2000).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır