Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Entwicklung sulfidogenen Schlamm aus marinen Sedimenten und Trichlorethylen Reduction in einer Upflow Anaerobic Sludge Blanket Reactor
In diesem Artikel
Zusammenfassung
Microbial sulfate reduction is a process of great importance in environmental biotechnology. The success of the sulfidogenic reactors depends among other factors on the microbial composition of the sludge. Here, we present a protocol to develop sulfidogenic sludge from hydrothermal vents sediments in a UASB reactor for reductive dechlorination purposes.
Zusammenfassung
The importance of microbial sulfate reduction relies on the various applications that it offers in environmental biotechnology. Engineered sulfate reduction is used in industrial wastewater treatment to remove large concentrations of sulfate along with the chemical oxygen demand (COD) and heavy metals. The most common approach to the process is with anaerobic bioreactors in which sulfidogenic sludge is obtained through adaptation of predominantly methanogenic granular sludge to sulfidogenesis. This process may take a long time and does not always eliminate the competition for substrate due to the presence of methanogens in the sludge. In this work, we propose a novel approach to obtain sulfidogenic sludge in which hydrothermal vents sediments are the original source of microorganisms. The microbial community developed in the presence of sulfate and volatile fatty acids is wide enough to sustain sulfate reduction over a long period of time without exhibiting inhibition due to sulfide.
This protocol describes the procedure to generate the sludge from the sediments in an upflow anaerobic sludge blanket (UASB) type of reactor. Furthermore, the protocol presents the procedure to demonstrate the capability of the sludge to remove by reductive dechlorination a model of a highly toxic organic pollutant such as trichloroethylene (TCE). The protocol is divided in three stages: (1) the formation of the sludge and the determination of its sulfate reducing activity in the UASB, (2) the experiment to remove the TCE by the sludge, and (3) the identification of microorganisms in the sludge after the TCE reduction. Although in this case the sediments were taken from a site located in Mexico, the generation of a sulfidogenic sludge by using this procedure may work if a different source of sediments is taken since marine sediments are a natural pool of microorganisms that may be enriched in sulfate reducing bacteria.
Einleitung
Einer der wichtigsten Beiträge zur Umweltbiotechnologie war die Gestaltung von Bioreaktoren, in denen die verwendeten (Inokulum) Schlamm konnte unter Sulfat reduzierenden Bedingungen durchzuführen. Sulfat-Reduktion (SR) ermöglicht die Behandlung von Abwasserströmen, die hohe Konzentrationen an Sulfat zusätzlich zur gleichzeitigen Entfernung von COD, Schwermetallen und organischen Schadstoffen, eine Tatsache, SR eine wünschenswerte Eigenschaft des Schlamms 1 ermöglicht, enthalten. Einige Beispiele von Abwässern mit Sulfat verunreinigt aus Gerberei, Papier, pharmazeutischen und chemischen Fertigungsindustrie 1 gekommen. , Bezieht sich jedoch die meisten von der Literatur zur Schlamm sulfidogenen bei methanogenen körnigen Schlamm zu sulfidogenesis 2 angepasst. Diese Anpassung wird üblicherweise durch Manipulieren des COD / SO 4 2- Verhältnis in dem Bioreaktor und die Zugabe von Chemikalien zu methanogens im Schlamm 2,3 Hemmung erreicht. Zusätzlich zu der langen Zeit, daß may erfordern die Bildung der sulfidogenen Granulate werden die Konkurrenz zwischen Methanogenen und sulfatreduzierenden und der Toleranz des Schlamms auf hohe Konzentrationen von Schwefelwasserstoff einige der wichtigsten Probleme, die entstehen können, wenn die sulfidogenen Schlamm in den Bioreaktor verwendet wird, aus der Anpassung erhalten vorwiegend methan Schlamm zu Sulfat reduzierenden Bedingungen. In dieser Arbeit, die Vorgehensweise, um eine überwiegend sulfidogenen Schlamm aus hydrothermalen Quellen Sedimente (Punta Mita, Nayarit, Mexiko) in einer Aufwärtsströmung anaeroben Schlammreaktor (UASB) zu erhalten, beschreiben wir, dann bewerten wir dessen Sulfat reduzierenden Aktivität im Laufe der Zeit und führen ein Experiment zu ihrer Anwendung auf reduzierende Dechlorierung zu bewerten. Die Lage der Sedimente wurde gewählt, weil es wurde berichtet, daß in diesem Ort gibt es die Bildung von Sulfiden durch die Sulfat-reduzierenden Aktivität der mikrobiellen Gemeinschaft bewohnen diese bestimmte Stelle 4 gezeigt.
Es gibt several Vorteile bei der Beschaffung dieser sulfidogenen Schlamm aus Sedimenten über die Anpassung methanogenen körnigen Schlamm sulfidogenesis. Einige dieser Vorteile sind: (1) Es ist nicht notwendig, um ein Granulat zu bilden für den Bioreaktor zu funktionieren, (2) der Schlamm toleriert relativ hohe Konzentrationen an Schwefelwasserstoff im Vergleich zu anderen UASB, die mit angepassten methanogene Schlamm arbeiten, und (3) wird keine Konkurrenz Substrats mit methanogenen selbst wenn Ester wird in der Mischung von flüchtigen Fettsäuren, die in dem Kulturmedium enthalten ist, um die Bildung des Schlammes zu fördern verwendet.
Dieses Verfahren wurde durchgeführt, um zu fördern, weil sulfidogenesis marinen Sedimenten sind eine natürliche Pool einer Vielzahl von Mikroorganismen, wie sulfatreduzierende Bakterien, fermentierenden Bakterien und Dehalogenierungsbakterien um nur einige zu nennen 5,6. Die Art des Konsortiums mit diesem Protokoll aus marinen Sedimenten entwickelt kann Effizienz in der Sulfatreduktion und damit hohen s aufweisen ulfate Reduktionsaktivität im Laufe der Zeit und eine höhere Toleranz gegenüber Sulfid in Konzentrationen höher als die berichtete, wie giftig für methanogens und sulfatreduzierenden Bakterien. Auf der anderen Seite, ist es wahrscheinlich, dass die Dehalogenierungsmittel Fähigkeit wird auch in den Sedimenten, indem Sie das Protokoll hier vorgeschlagene, aber es kann auf dem ursprünglichen mikrobiellen Gemeinschaft hängen gezeigt. Diese Annahme basiert auf der Tatsache, dass die reduktive Dechlorierung kann entweder durch Atmung oder Cometabolismus auftreten erfolgen beide Bedingungen, die im Meeresmikrobengemeinschaft 7 gefördert werden kann. Die Kultivierung der Sedimente, um den Schlamm zu erhalten, wurde unter Verwendung einer Mischung aus Acetat, Propionat und Butyrat als Substrat, da diese flüchtigen Fettsäuren werden durch verschiedene Stämme von sulfatreduzierenden Bakterien verwendet wurde. Diese Säuren sind auch die Art der Kohlenstoffverbindungen in marinen Sedimenten häufig gefunden, nach mehreren Berichten in der Literatur für kohlenstoffhaltiges Material in Meeressedimenten 5,6.
content "> Schließlich sind einige der am stärksten toxische Verbindungen, die im Grundwasser und andere Gewässer auf der ganzen Welt zu finden sind, chlorierte Lösungsmittel, wie Trichlorethylen (TCE) oder Perchlorethylen (PCE). Diese Verbindungen toxisch sind nicht nur für den Menschen, aber auch auf Mikroorganismen, insbesondere TCE, das noch in den USA als eine Priorität Schadstoff durch die Environmental Protection Agency 8. In dieser Arbeit wurde ein Experiment, in dem die sulfidogenen Schlamm wird auf seine Fähigkeit zur Reduzierung TCE bei Konzentrationen getestet, die in der sind wir vorgeschlagen, Bereich für chlorierte Verbindungen biologischen Abbau unter Methanogenese Bedingungen 9,10 berichtet. Es ist erwähnenswert, dass die meisten Forschungen auf biologischer Abbau von chlorierten Verbindungen wurde unter methanogenen Bedingungen 9,10 durchgeführt wert. Wir glauben, dass der Versuch mit TCE in diesem Protokoll vorgeschlagen wird ein Beispiel für die Einsatzmöglichkeiten des Schlamms. Das Ziel dieses Experiments war es, eine Ebewerten die Toleranz der Schlamm an den TCE und dem TCE Wirkung auf die Sulfat-reduzierenden Aktivität. Berücksichtigt man, dass die meisten Forschungen auf biologischer Abbau von chlorierten Verbindungen wird unter methanogenen Bedingungen, schlägt dieses Protokoll die Bildung eines Schlamms verwendet werden, um gleichzeitig: (1) Entfernen Sulfat, (2) Entfernen COD und (3) zu entfernen chlorierten Verbindungen. Ein weiterer Schritt könnte, um den Schlamm auf die gleichzeitige Entfernung von TCE und Schwermetalle zu bewerten (zusätzlich zu Sulfat und CSB), zwei Bedingungen, die nicht unter methanogenen Bedingungen ausgewertet werden kann.Protokoll

Abbildung 1. Schema für die Schritte des Protokolls. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
1. Sammeln marinen Sedimenten für die Bildung des Schlamm
- Identifiziert eine zugängliche Unterseebereich entweder nahe hydrothermale (aufgrund der Anwesenheit von Sulfiden, die eine höhere sulfatreduzierende Aktivität anzeigen kann) oder an einem Bereich, wo Ablagerungen von organischen Stoffen sind nachweisbar.
- Für den Zweck dieser Arbeit, nehmen Sie ca. 3 oder 4 kg Sediment und lassen Sie das Wasser aus den Proben. Legen Sie die Proben in dunklen Plastiktüten. Keine Kühlung benötigt wird.
- Einmal im Labor, halten die Taschen mit den Proben in den Kühlschrank, wenn sie nicht sofort verwendet werden. Für den Zweck dieser Arbeit, samples kann im Kühlschrank für Wochen oder Monate, bevor Sie sie.
- Nehmen Sie einen großen Teil der Sedimentprobe (dh 1 oder 2 kg) und verwenden ein geeignetes mesh (0,2 cm), um aus den Sedimenten der große Ablagerungen von kohlenstoffhaltigem Material, die gefunden werden können, oder einige Felsen, die vorhanden sein können, zu beseitigen.
Anmerkung: In diesem Fall ist ein Gitter von 0,20 cm Durchmesser (0,0767 Zoll) wurde verwendet, aber es kann eine unterschiedliche Größe entsprechend der Größe der Teilchen in der Probe.- Nach dem Passieren der Sedimente durch die Maschen, mischen die ausgewählt werden, um zu fördern, dass der Anteil homogen Teil.
- Nehmen getrennt kleinere Proben (dh 2 bis 3 g) zur Entfernung der flüchtigen suspendierten Feststoffen (VSS) Gehalt nach den Standardverfahren 11 zu bestimmen.
Hinweis: Siehe Abbildung 2 für die Schritte 1,2 bis 1,4.

Abbildung 2. Fotografien der Sedimentproben.(A) Sedimentproben unmittelbar nach der (B) Sedimentprobe nach dem Passieren durch das Gitter gemacht.. (C) Probe zum Wiegen vor der flüchtigen suspendierten Feststoffen (VSS) Bestimmung entnommen. Die Petrischale muss nicht sterilisiert werden. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
2. Bioreaktor Set Up
- Für den Zweck dieser Arbeit, verwenden Sie ein UASB-Glasreaktor mit einem Gesamtarbeitsvolumen von 3 L. Alternativ können Sie einen 1 oder 2 L Volumen Glasreaktor.
- Basierend auf dem VSS-Gehalt der Sedimente Berechnung der Menge an Sediment als Inoculum verwendet, um 5 g VSS in 1 L. erhalten
- Bitte berücksichtigen, dass, wenn die Menge des Sediments nach Berechnung zu groß ist, dann etwa 25% bis 30% des Volumens des Bioreaktors sollte durch die Sedimente anstelle besetzt werden.
- Notieren Sie die VSS-Gehalt, da eswird sich ändern, wenn der mikrobiellen Gemeinschaft wird in dem Bioreaktor bereichert. Der VSS-Gehalt ist für die Berechnungen der Sulfat-reduzierenden Aktivität im Bioreaktor notwendig.
- Sicherzustellen, dass die endgültige Konzentration des Grundmediums und der Pufferlösung in dem Bioreaktor ist ähnlich zu der von Guerrero-Barajas et al. (2014) 12.
- Sicherzustellen, dass die endgültige Volumen des Sediments, Basalmedium, Pufferlösungen und flüchtige Fettsäuren gleich der endgültigen Arbeitsvolumen des Reaktors. Das Basismedium Rezept 12 enthält die geeigneten Konzentrationen für die Spurenmetallen und Vitaminen Lösung.
- Vorbereitung einer Stammlösung Grundmedium und der Pufferlösung in einer geeigneten Konzentration für das Arbeitsvolumen des Reaktors verwendet (dh, 2, 3 oder 4-fach konzentrierter als die in Schritt 2.4 angegeben), ist es, sicherzustellen, dass, wenn es verdünnt ist, bei der Konzentration von Guerrero-Barajas et al. (2014)12).
Anmerkung: Die Stammlösung für das Basismedium ist immer notwendig, jedoch wird die Pufferlösung nur am Anfang bis notwendig. Es ist nicht notwendig, Pufferlösung, nach dieser Zeit hinzuzufügen. - Bereiten Sie eine Stammlösung von flüchtigen Fettsäuren: Acetat, Propionat und Butyrat in einem 2,5: 1: 1 COD Anteil. Berücksichtigen Sie für die Berechnungen das Natriumacetat in dem Basismedium enthalten. Die endgültige CSB-Konzentration im Reaktor müssen 2,7 g / l liegen.
Achtung: Diese Lösung ist in einem Abzug. Tragen Nitril Handschuhe und Schutzbrille für die Herstellung von dieser Lösung. Berücksichtigen die Stöchiometrie der Reaktionen von Sulfat mit den flüchtigen Fettsäuren, die in 3 gezeigt wird. - Vorbereitung einer Stammlösung von Natriumsulfat (Na 2 SO 4) in einer geeigneten Konzentration, um den Reaktor zu liefern eine Endkonzentration von 4,000 mg / l Sulfationen (SO 4 2-). Alternativ sind the Menge an Sulfat in dem Basalmedium anstatt sie aus einer Stammlösung, solange der endgültige Sulfat erforderlich (SO 4 2-) Konzentration richtig ist.
- Legen die Sedimente im Reaktor mit einem Teil des Grundmedium gemischt wird, um sicherzustellen, dass sie den unteren Teil des Reaktors erreichen.
- Das restliche Basismedium und der Pufferlösung mit der flüchtigen Fettsäuren Lösung und der Sulfatlösung vermischt. Sicherzustellen, dass die Lösung von flüchtigen Fettsäuren in die Flüssigkeit geschüttet. Hinweis: Führen Sie diesen Schritt in einem Abzug.
- Setzen die Verbindungen und Rohrleitungen aus dem Reaktor zur Rückführpumpe. Stellen Sie den Recyclingstrom bei 60 ml / min. Gesetzt den Bioreaktor in der Temperaturkammer bei 34 ° C. Überprüfen Sie regelmäßig, dass die Temperaturschwankungen sind kleine (dh 34 ± 1,7 ° C)
- Stellen Sie die Verbindungen zur Gaspendel Spalte.
Hinweis: Siehe Abbildung 4 für die Schritte 2.1 bis 2.5.
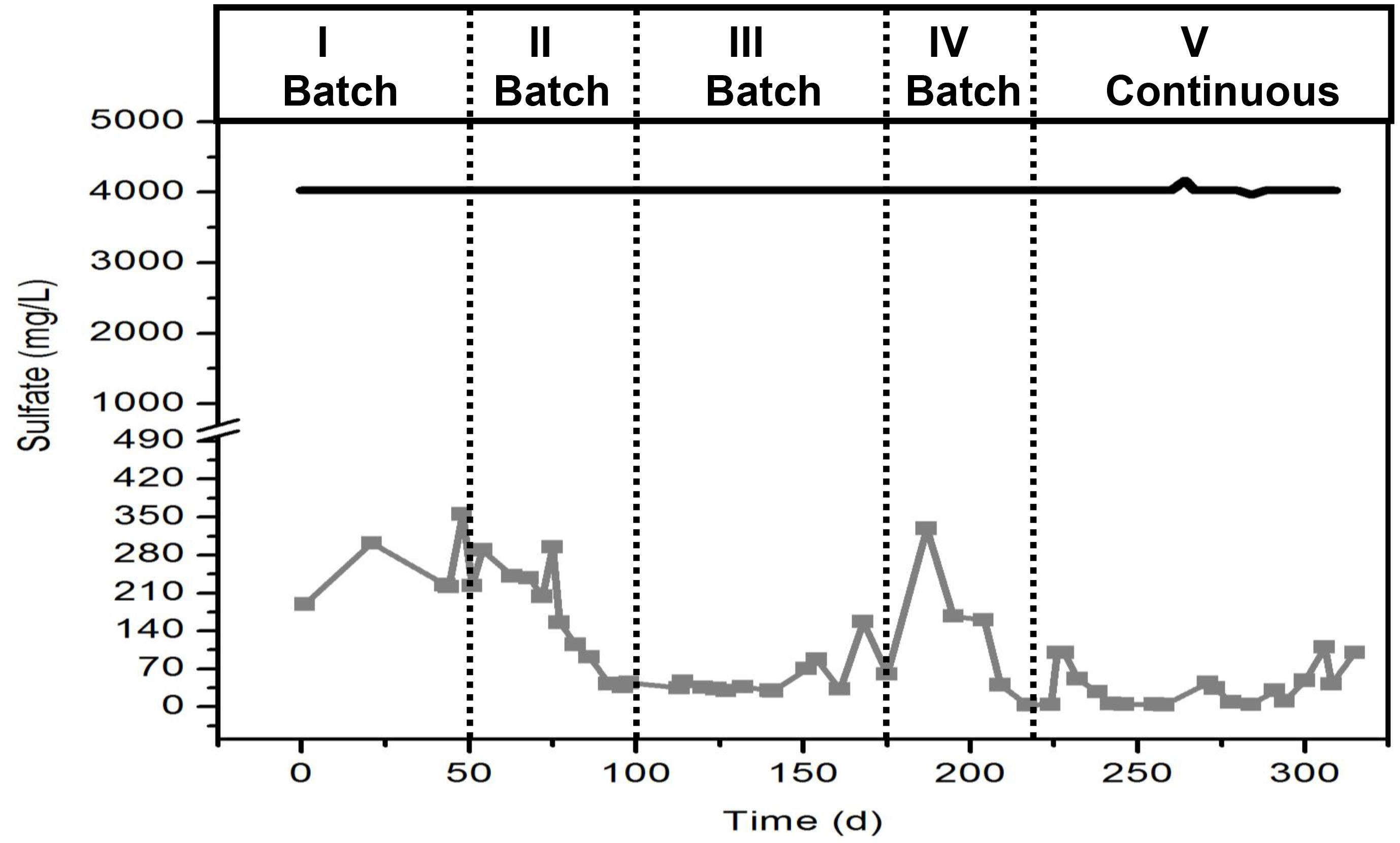
Abbildung 3. Stöchiometrie der Sulfat-Reduktion mit VFA (Acetat, Propionat und Butyrat). Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Abbildung 4. UASB-Reaktor. (A) Anfangszeit. (B) Continuous-Regime nach 300 Betriebstagen. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.3. Betrieb des Reaktors auf Sulfidogenesis und Wachstum der Mikroorganismen zu fördern
Hinweis: Lassen Sie für das Inokulum, das v verbrauchenolatile Fettsäuren und Sulfat. Dazu warten, für eine Woche für die Durchführung der ersten Analyse für Sulfat, Sulfid und CSB Verbrauch.
- Nach einer Woche Inkubation eine Probe von 5-7 ml der Flüssigkeit zur Analyse COD zuführen, Sulfat und Sulfid-Gehalt und pH folgenden Standardmethoden 11, 13.
- Analysieren Sulfid in der Flüssigkeit spektrophotometrisch (bei einer Wellenlänge (λ) von 670 nm), indem Sie die Methylenblau-Methode 13.
- Platzieren 5 ml einer Zinkacetat-Lösung (2% w / w) in einem 25 ml Messkolben übergeführt, schnell 200 ul der Probe auf die Zinkacetat-Lösung.
- Hinzufügen von 2,5 ml einer N, N-Dimethyl-p-phenylendiamin-Oxalat (DMP) -Lösung (0,2% w / w in 20% H 2 SO 4) und 125 ul des Eisen (III) Ammoniumsulfatlösung (10% w / w in 2% H 2 SO 4) und vollständig mit destilliertem Wasser die 25 ml im Messkolben. Warten Sie 30 Minuten für die Reaktionauftritt, Zeit, bei der die blaue Farbe stabilisiert ist. 13.
Hinweis: Warten mindestens 15 min, jedoch nicht mehr als 60 Minuten, um die Proben in das Spektrophotometer zu testen. Leiten das Lesen der blauen Endlösung im Spektrophotometer.
- Analyse Sulfat nach Standardverfahren 11. Hier quantifizieren Sulfat als Bariumsulfat unter Verwendung eines turbidimetrischen Methode.
- Zeigen 5 ml einer Konditionierungslösung (Salzsäure HCl 1: 1) in einem Messkolben von 25 ml, 1 ml der zuvor zentrifugiert Probe (bei 11.320 × g), führen Sie die 25 ml Messkolben mit destilliertem Wasser und Add 1 g Bariumchlorid.
- Mischen Sie die Lösung für 1 min in einem Wirbel. Warten für 4 min für die Bariumsulfat zu bilden und zu lesen, die Probe im Spektralphotometer bei einer Wellenlänge (λ) von 420 nm. 11
- Analyse COD nach Standardverfahren 11. Alternativ können Sie einen COD determination-Kit.
- Vor der CSB-Bestimmung, zentrifugieren Sie die Probe gründlich (bei 11.320 xg), um das verbleibende Sulfid, die bei der Bestimmung beeinträchtigen können, zu entfernen. Falls erforderlich, Zentrifugen zweimal: das erste Mal, unmittelbar nach der Entnahme der Probe und das zweite Mal warten, 6 oder 8 Stunden und dann führen die COD-Analyse.
- 2 ml Probe in ein Reaktionsfläschchen der CSB-Bestimmung Kit, verschließen Sie das Fläschchen und homogenisieren die Mischung durch leichtes Schütteln. Bereiten Sie eine leere durch Zugabe von 2 ml destilliertem Wasser in ein anderes Reaktionsgefäß und die Mischung zu homogenisieren.
- Die Gefäße im Faulungsreaktor bei 150 ° C für 2 Std. Entfernen Sie die Durchstechflaschen und lassen Sie sie abkühlen, in der Dunkelheit. Sie die Ablesung der Fläschchen im Spektrophotometer bei einer Wellenlänge von 620 nm.
- Besorgen Sie sich die Gasmenge aus der Gaspendel Spalte.
- Analysieren Sulfid in der Flüssigkeit spektrophotometrisch (bei einer Wellenlänge (λ) von 670 nm), indem Sie die Methylenblau-Methode 13.
- Warten, bis weitere 5 bis 7 Tage, bis das Sulfat verbraucht wird. Sulfate und CSB muß in ein verzehrt werdenpproximately 85% bis 90%, bevor eine neue Fed-Batch gestartet wird.
- Sobald Sulfat (und COD) verbraucht werden, komplett wiederholen Sie Schritt 2.4. Liefern frisches Medium und neue Nährstoffe für jede Charge.
- Wiederholen Sie die Schritte 3.1 und 3.2. An dieser Stelle sollte jede Charge zwischen 7 und 10 Tagen.
- Bei 3 bis 4 Chargen abgeschlossen sind, wiederholen Sie Schritt 2.4, sondern erhöhen die CSB-Konzentration bis 4 g / L.
- Wiederholen Sie Schritt 3.1 und Schritt 3.2.
- Wiederholen Sie Schritt 3.3, sondern erhöhen die CSB-Konzentration bis 6 g / L.
- Wiederholen 3.6 und 3.6.1 allmählich zunehmende CSB-Konzentration, bis es 10 g / l.
Hinweis: Stellen Sie die Grafik, die die Sulfatkonzentration (mg / l) gegen die Zeit (d) präsentiert.
- Wenn Sulfat Verbrauch liegt bei über 80% in weniger als 24 Stunden und für mehr als eine Woche in diesem Fall, schalten Sie den Betrieb des Reaktors zur kontinuierlichen Modus. Für den Dauerbetrieb stellen Sie die hydraulische Verweilzeit (HRT) bei 24 Stunden und pflegen die Sulfatkonzentration auf 4 g / l und CSBbei 10 g / L.
Hinweis: Im Laufe der Zeit das Sulfat Verbrauch sein sollte schneller.
4. Sulfat-reduzierenden Aktivitätstest
- Vor dieser Prüfung stellen Sie sicher, dass der Bioreaktor unter Dauerregime stellt weniger als 10% Veränderung der übrigen Sulfatkonzentration.
- An jedem beliebigen Tag, stoppen Sie den Reaktor nach einer HRT Zyklus und Durchführung Schritt 2.4. Für Schritt 2.4.3 verwenden eine CSB-Konzentration von 10 g / L.
- Sobald der Bioreaktor zugeführt wird, dauert es 5 bis 7 ml-Proben der Flüssigkeit und Analysen durchzuführen für CSB, Sulfat, Sulfid (Schritt 3.1) und pH jede Stunde. Notieren Sie sich die erzeugte Gasvolumen.
- Errechnen Sulfat reduzierender Aktivität nach der Literatur 14.

SRA = Sulfat-reduzierenden Aktivität (mg CSB-H 2 S) / gVSS * d
m H 2 S = Sulfidkonzentration, ausgedrückt als mg CSB-H 2 S
VSS = flüchtige suspendierte Feststoffkonzentration
t = Zeit (d oder h)
- Nehmen Sie die entsprechenden Diagramme, die den Prozentsatz der Sulfat Verbrauch gegenüber Sulfidkonzentration im Laufe der Zeit in mg / L zu zeigen. Nehmen Sie die Grafiken, die Prozentsatz der COD Verbrauch im Laufe der Zeit zeigen. Nehmen Sie die Grafiken, die pH-Variation über die Zeit zeigen.
5. Trichlorethylen (TCE) Reduktionstest
- Vor dieser Prüfung stellen Sie sicher, dass der Bioreaktor unter Dauerregime arbeiten und stellt weniger als 10% Veränderung der übrigen Sulfatkonzentration. Haben Sie diesen Test nicht starten, wenn die Sulfatreduktion im Bioreaktor weniger als 90% ist.
- Wird eine Stammlösung von Trichlorethylen (TCE) unter Berücksichtigung, daß die Endkonzentration dieser Verbindung in der Flüssigphase des Bioreaktors ist 300 um. Betrachten Sie die partitioning der Verbindung in den Kopfraum unter Verwendung der Henrys Gesetz dimensionslose Konstante (H') zum TCE bei 34 ° C. H'at 34ºC TCE ist 0,4722.
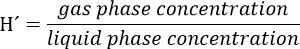
Achtung: Bereiten Sie diese Lösung in einem Abzug und tragen Sie Handschuhe und Schutzbrille.- Zum Beispiel für eine 5.000 uM Stammlösung, berechnet wie folgt:
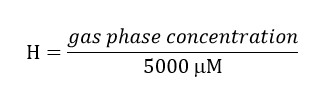
TCE Gasphasenkonzentration = (0,4722) * (5.000) = 2.139 & mgr; M. Umfassen diese Konzentration bei der Herstellung der Vorratslösung, da diese Menge des TCE in dem Kopfraum ist.
Dann in der Flüssigkeit (Wasser) der Stammlösung wird die tatsächliche TCE-Konzentration sein: 5000 + 2139 = 7139 & mgr; M. TCE Dichte = 1,43 g / ml. Konvertieren Sie die 7139 & mgr; M in mg und dann unter Verwendung der Dichte von TCE berechnen das Volumen der TCE für die Stammlösung.
Hinweis: Die Konzentration des TCE-Stammlösung may niedriger als 5,000 & mgr; M, das heißt 3.000 oder 1.000 & mgr; M, dies davon abhängig, wieviel Volumen dieser Lösung kann dem Bioreaktor nach seinem flüssigen Phasenvolumen geliefert werden.
- Zum Beispiel für eine 5.000 uM Stammlösung, berechnet wie folgt:
- Bereiten Standardkurven in den Gaschromatographen für TCE, cis-1,2-Dichlorethylen, trans-1,2-Dichlorethylen, Vinylchlorid und Ethen. Bereiten Sie die cis-1,2-Dichlorethylen und trans-1,2-Dichlorethylen Standardkurven aus einer Stammlösung aus diesen Verbindungen, indem Sie in 5.2 für die TCE-Stammlösung der gleichen Vorgehensweise. Bereiten Sie die Standardkurven für Vinylchlorid und Ethen durch Verdünnen der Konzentration jedes Gas aus den Normen (Gasflaschen).
- Vorbereitung der Standardkurven dieser Verbindungen in einem Bereich von 20 bis 300 um. Verwenden Sie die Methode von Guerrero-Barajas et al. (2011) 15 für die Analyse dieser Verbindungen in den Gaschromatographen.
Achtung: Bereiten Sie diese stehenard-Lösungen in einem Abzug und tragen Sie Handschuhe und Schutzbrille.
- Vorbereitung der Standardkurven dieser Verbindungen in einem Bereich von 20 bis 300 um. Verwenden Sie die Methode von Guerrero-Barajas et al. (2011) 15 für die Analyse dieser Verbindungen in den Gaschromatographen.
- An jedem beliebigen Tag, stoppen Sie den Reaktor nach einer HRT Zyklus und Durchführung Schritt 2.4. Für Schritt 2.4.3 verwenden eine CSB-Konzentration von 10 g / L.
- Sobald der Bioreaktor zugeführt wird, fügen die TCE direkt an das Medium im Bioreaktor aus der in 5.2 hergestellten Stammlösung, muss der letzte TCE-Konzentration in der Flüssigphase des Bioreaktors 300 um sein. Stellen Sie die HRT zu 12 Std.
- Am Ende eines Zyklus HRT nehmen Proben der Flüssigkeit (500 bis 1.000 & mgr; l) und Verhaltensanalyse für CSB, Sulfat und Sulfid (Schritte 3.1.1, 3.1.2 und 3.1.3). Probenahmen aus dem Gasraum (100 bis 250 ul) und Durchführung von Untersuchungen zum TCE, cis -1,2-Dichlorethylen, trans-1,2-Dichlorethylen, Vinylchlorid und Ethen in den Gaschromatographen.
- Wiederholen Sie Schritt 2.4. Für Schritt 2.4.3 verwenden eine CSB-Konzentration von 10 g / L.
- Jede TCE Reduktionstest nicht zu wiederholen, bis der Bioreaktor stellt mehr als 90% Sulfatreduktion und weniger von 10% Variation in beiden, Sulfatreduktion und Sulfat in den Bioreaktor verbleiben.
- Wiederholen 5.4, 5.5 und 5.6 zwei oder drei Mal mehr.
- Nehmen Sedimentproben (0,5 g), um die Identifizierung der Mikroorganismen nur nach einer TCE-Reduktionstest beendet hat durchzuführen. Tun Sie dies nach 2 oder 3 TCE Reduktion Tests.
6. sulfatreduzierenden Aktivitätstest nach TCE Reduction Experiment
- Wiederholen Sie Schritt 4 vollständig.
7. Ermittlung der Mikroorganismen
- Nehmen Sie Proben von Schlamm von etwa 0,5 g je und führen insgesamt RNA-Extraktion nach Standardverfahren 12.
- Amplifikation der 16S rRNA-Gens durch reverse Transkription und Durchführung der Polymerase-Kettenreaktion (RT-PCR) einen Schritt 12.
- Entwerfen der Primer zur Amplifikation oder zur Verwendung als erste Annäherung die, die in der Literatur 11 vorgeschlagen. Folgen Sie dem Amplification Verfahren vorgeschlagen, in der Literatur 12.
- Konstruieren Sie die 16S-rRNA-Bibliotheken. PCR-Produkte können durch Verwendung von Klonierungstechniken-kit 11 kloniert werden. In der Regel können 10 Kolonien von jeder Platte (jede Kolonie, die einen PCR-Produkt) geklont werden. Vorbereitung der Plasmid-DNA zur Sequenzierung nach dem Verfahren in der Literatur 12 vorgeschlagen.
- Führen Sie die Sequenzierung von Fragmenten. Wieder amplifizieren etwa 1.400 bp der PCR-Produkte mit dem externen Protokoll für die PCR-Amplifikation zuvor beschrieben (Schritt 7.4), und Klon gemß dem Verfahren in der Literatur 12 vorgeschlagen. Isolieren Sie das rekombinante Plasmid von E. coli-Kolonien wie in der Literatur 12 vorgeschlagen. Den Teilverfahren zur Sequenzierung mit M13 universelle Primer 12 durchzuführen.
- Führen Sie die Sequenzen Analyse. Ausrichten der Nucleotidsequenzen unter Verwendung des Clustal X und manuell in die Texteditor einzustellen. Führen Sie BLAST-Suchen der NCBI database. (http://www.ncbi.nlm.nih.gov/BLAST/Blast.cgi) 12.
- Besorgen Sie sich die Nukleotidsequenz Zugangsnummern. Abzuscheiden die Nukleotidsequenzen der in der EMBL Nukleotidsequenzdatenbank (Gen-Banken / EMBL / DDBJ) unter den entsprechenden Zugangsnummern (dh JQ713915eJQ713925 nach Sequenzen von Amplicons) 12 identifizierten Klone.
Ergebnisse
Ein typisches Verhalten des Sulfat-Reduktion in dem Bioreaktor ist in 5 gezeigt. Es ist wichtig zu bemerken, dass während der ersten Wochen des Betriebs Sulfatreduktion langsam. Jedoch langsam, zeigt den Verbrauch von mehr als 90% Sulfat mit der Zeit, dass das Inokulum Entwicklung einer mikrobiellen Gemeinschaft zu reduzieren vermag Sulfat und daher in sulfatreduzierenden Bakterien angereichert. Die verschiedenen Perioden in der Figur zeigen, dass die Sulfatreduktion wurde Erhöhung der Geschwindigkeit...
Diskussion
Es gibt mehrere Anwendungen sulfidogenesis in Umweltbiotechnologie, einer der am häufigsten verwendeten Anwendungen des Stoffwechsels von sulfatreduzierenden Bakterien in Konsortien mit gärenden Bakterien in der Abwasserbehandlung. UASB Reaktoren zählen zu den Haupt entwickelt Ansätze zur industriellen Abwasserbehandlung mit hoher Sulfatkonzentrationen. In dieser Arbeit präsentieren wir ein Protokoll, um sulfidogenen Schlamm aus marinen Sedimenten in einem UASB-Reaktor erhalten. Die kritischen Schritte in dem Proto...
Offenlegungen
The authors Selene Montserrat García-Solares, Claudio Garibay-Orijel and Claudia Guerrero Barajas submitted (as inventors) in 2012 the application to obtain a patent entitled: “Process for the anaerobic treatment of industrial wastewater with high content of sulfate and chlorinated compounds”. This submission has been approved to the stage of the formatting. The submission has Instituto Politécnico Nacional as the rights owner.
Danksagungen
The authors are grateful for the financial support provided by Instituto Politécnico Nacional grants 20120110, 20130399 and 20140239 SIP and also by Instituto de Ciencia y Tecnología del Distrito Federal Mexico (PICS 08-79, ICYT-DF, 2009-2012). Thanks also to CONACYT – Mexico for the graduate scholarship (225806) awarded to Selene Montserrat García-Solares and for the financial support provided by grant 82627.
Materialien
| Name | Company | Catalog Number | Comments |
| trichloroethylene | sigma Aldrich | 251402 | |
| cis-1,2-dichlorotehylene | sigma Aldrich | ||
| trans-1,2-dichloroethylene | sigma Aldrich | D-62209 | |
| vinyl chloride scotty standard | supelco | 1,000 ppm v/v in nitrogen | |
| ethene scotty standard | supelco | 99% purity | |
| pump | Masterflex | Model 7553-75 | |
| spectrophotometer | any | ||
| microcentrifuge | any | ||
| gas tight syringes | any | 100 and 200 microliters | |
| UASB glass reactor | any | under design | |
| gas chromatograph | any | FID detector | |
| capillary column SPB-624 | supelco | ||
| pH meter | any | ||
| viton tubing | Masterflex | ||
| basal medium reagents | any | ||
| trace metals reagents | any | ||
| vitamins solution reagents | any | ||
| sodium sulfate | any | ||
| volatile fatty acids | any | ||
| COD determination kit | HACH | range 0-15,000 mg/L | |
| TOPO-TA cloning kit pCR®4.0 | Invitrogen, US | ||
| S.N.A.P. TM Miniprep Kit | Invitrogen, UK | ||
| Pure link TM Quick Plasmid Miniprep kit | Invitrogen |
Referenzen
- Lens, P., Esposito, M. V. G., Zandvoort, M. Perspectives of sulfate reducing bioreactors in environmental biotechnology. ReViews Environmental Science and Biotechnology. 1 (4), 311-325 (2002).
- Omil, F., Lens, P., Hulshoff, P., Lettinga, G. Characterization of biomass from a sulfidogenic, volatile fatty acid-degrading granular sludge reactor. Enzyme and MicrobialTechnology. 20, 229-236 (1997).
- Lopes, S. I. C., Wang, X., Capela, M. I., Lens, P. N. L. Sulfate reduction during the acidification of sucrose at pH 5 under thermophilic (55 °C) conditions.II: Effect of sulfide and COD/SO4-2 ratio. Bioresource Technology. 101, 4278-4284 (2010).
- Alfonso, P., Prol-Ledesma, R. M., Canet, C., Melgarejo, J. C., Fallick, A. E. Sulfur isotope geochemistry of the submarine hydrothermal coastal vents of Punta Mita, Mexico. Journal of Geochemical Exploration. 78-79, 301-304 (2003).
- Valdemarsen, T., Kristensen, E. Degradation of dissolved organic monomers and short chain fatty acids in sandy marine sediment by fermentation and sulfate reduction. Geochimica et Cosmochimica Acta. 74, 1593-1605 (2010).
- Quistad, S. D., Valentine, D. L. Anaerobic propane oxidation in marine hydrocarbon seep sediments. Geochimica et Cosmochimica Acta. 75, 2159-2169 (2011).
- Futagami, T., Morono, Y., Terada, T., Kaksonen, A. H., Inagaki, F. Dehalogenation activities and distribution of reductive dehalogenase homologous genes in marine subsurface sediments. Applied and Environmental Microbiology. 75 (21), 6905-6909 (2009).
- U.S. Environmental Protection Agency. List of priority pollutants. Clean Water Methods. , (2014).
- Ozdemir, C., Dursun, S., Karatas, M., Sen, N., Sahinkaya, S. Removal of trichloroethylene (TCE) in upFlow anaerobic sludge blanket reactors (UASB). Biotechnology and Biotechnological Equipment. 21 (1), 107-112 (2007).
- Zhang, Y., Wang, X., Hu, M., Li, P. Effect of hydraulic retention time (HRT) on the biodegradation of trichloroethylene wastewater and anaerobic bacterial community in the UASB reactor. Applied Microbiology and Biotechnology. 99, 1977-1987 (2015).
- . . Standard Methods for the Examination of Water and Wastewater. , (1998).
- Guerrero-Barajas, C., et al. Enhanced sulfate reduction and trichloroethylene (TCE) biodegradation in a UASB reactor operated with sludge developed from hydrothermal vents sediments: process and microbial ecology. International Biodeterioration and Biodegradation. 94, 182-191 (2014).
- Trüper, H. G., Schlegel, H. G. Sulphur metabolism in Thiorhodaceae I. Quantitative measurements on growing cells of Chromatium okenii. Antoine van Leeuwenhoek. 30, 225-238 (1964).
- Gallegos-García, M. G. . Biological processes of sulfate reduction in biofilms for metals precipitation [Ph D thesis]. , (2009).
- Guerrero-Barajas, C., Garibay-Orijel, C., Rosas-Rocha, L. E. Sulfate reduction and trichloroethylene biodegradation by a marine microbial community from hydrothermal vents sediments. International Biodeterioration and Biodegradation. 65, 116-123 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten