É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Desenvolvimento de sulfidogenico Sludge de sedimentos marinhos e Redução Trichloroethylene em um Reator Anaeróbio de Manta de Lodo
Neste Artigo
Resumo
Microbial sulfate reduction is a process of great importance in environmental biotechnology. The success of the sulfidogenic reactors depends among other factors on the microbial composition of the sludge. Here, we present a protocol to develop sulfidogenic sludge from hydrothermal vents sediments in a UASB reactor for reductive dechlorination purposes.
Resumo
The importance of microbial sulfate reduction relies on the various applications that it offers in environmental biotechnology. Engineered sulfate reduction is used in industrial wastewater treatment to remove large concentrations of sulfate along with the chemical oxygen demand (COD) and heavy metals. The most common approach to the process is with anaerobic bioreactors in which sulfidogenic sludge is obtained through adaptation of predominantly methanogenic granular sludge to sulfidogenesis. This process may take a long time and does not always eliminate the competition for substrate due to the presence of methanogens in the sludge. In this work, we propose a novel approach to obtain sulfidogenic sludge in which hydrothermal vents sediments are the original source of microorganisms. The microbial community developed in the presence of sulfate and volatile fatty acids is wide enough to sustain sulfate reduction over a long period of time without exhibiting inhibition due to sulfide.
This protocol describes the procedure to generate the sludge from the sediments in an upflow anaerobic sludge blanket (UASB) type of reactor. Furthermore, the protocol presents the procedure to demonstrate the capability of the sludge to remove by reductive dechlorination a model of a highly toxic organic pollutant such as trichloroethylene (TCE). The protocol is divided in three stages: (1) the formation of the sludge and the determination of its sulfate reducing activity in the UASB, (2) the experiment to remove the TCE by the sludge, and (3) the identification of microorganisms in the sludge after the TCE reduction. Although in this case the sediments were taken from a site located in Mexico, the generation of a sulfidogenic sludge by using this procedure may work if a different source of sediments is taken since marine sediments are a natural pool of microorganisms that may be enriched in sulfate reducing bacteria.
Introdução
Uma das contribuições mais importantes para a biotecnologia ambiental foi o projeto de biorreatores em que o lodo utilizado (inóculo) foi capaz de realizar em condições redutoras de sulfato. Redução do sulfato (SR) permite que o tratamento de fluxos de águas residuais que contêm elevadas concentrações de sulfato de para além da remoção simultânea de COD, metais pesados e poluentes orgânicos, um facto que torna SR uma característica desejável das lamas 1. Alguns exemplos de efluentes contaminados com sulfato de vir de curtumes, papel, farmacêutica e manufatura química indústrias 1. No entanto, a maioria da literatura refere-se a sulfidogenico lamas quando lodo granular metanogênica foi adaptado para sulfidogenesis 2. Esta adaptação é normalmente alcançada através da manipulação do COD / SO 4 2- ratio no biorreactor e adição de produtos químicos para inibir metanógenos na 2,3 lamas. Em adição ao longo tempo que may requerem a formação dos grânulos sulfidogenico, a competição entre os metanogenos e redutores de sulfato e a tolerância da lama a altas concentrações de sulfureto são alguns dos principais problemas que podem surgir se a lama sulfidogenico utilizado no bioreactor é obtido a partir da adaptação de predominantemente lamas methanogenic a sulfato condições redutoras. Neste trabalho, descrevemos o procedimento para obter uma lama predominantemente sulfidogenico de fontes hidrotermais sedimentos (Punta Mita, Nayarit, México) em um reator anaeróbio de manta de lodo (UASB), em seguida, avaliar a sua sulfato de reduzir a atividade ao longo do tempo e realizar um experimento para avaliar a sua aplicação em descloração redutiva. A localização dos sedimentos foi escolhido porque ele tem sido relatado que, nesse local, há formação de sulfuretos, devido à actividade de redução de sulfato exibida pela comunidade microbiana que habitam em particular lugar 4.
Há Severvantagens al na obtenção deste lodo sulfidogenico de sedimentos sobre a adaptação do lodo granular methanogenic para sulfidogenesis. Algumas destas vantagens são: (1) não é necessária para formar os grânulos para o biorreactor de operar, (2) as lamas tolera concentrações relativamente elevadas de sulfureto quando comparado a outros UASB que operam com lamas metanogênica adaptado, e (3) não está nenhuma competição para substrato com metanógenos mesmo de etilo é usado na mistura de ácidos gordos voláteis que está incluído no meio de cultura para promover a formação do lodo.
Este procedimento foi seguido para promover sulfidogenesis porque sedimentos marinhos é uma associação natural de uma grande variedade de microorganismos, tais como bactérias redutoras de sulfato, da fermentação de bactérias e bactérias desalogenação apenas para mencionar alguns 5,6. O tipo de consórcio desenvolvido a partir de sedimentos marinhos utilizando este protocolo pode apresentar eficiência na redução de sulfato e, portanto, altas s ulfate redução da atividade ao longo do tempo e maior tolerância ao sulfeto em concentrações mais elevadas do que o reportado como tóxico para methanogens e bactérias redutoras de sulfato. Por outro lado, é provável que a capacidade de desalogenação também é mostrado nos sedimentos, seguindo o protocolo proposto aqui, mas pode depender da comunidade microbiana inicial. Esta suposição é feita com base no fato de que descloração redutiva pode ocorrer pela respiração ou co-metabolismo, ambas as condições que podem ser promovidos na comunidade microbiana marinha 7. O cultivo dos sedimentos para obter a lama foi realizada usando uma mistura de acetato, propionato e butirato como substrato, porque estes ácidos gordos voláteis são utilizados por várias estirpes de bactérias redutoras de sulfato. Estes ácidos também são o tipo de compostos de carbono frequentemente encontrados em sedimentos marinhos, de acordo com vários relatos na literatura sobre material carbonáceo em sedimentos marinhos 5,6.
conteúdo "> Finalmente, alguns dos compostos mais tóxicas que estão presentes nas massas de água subterrânea e outra de água em todo o mundo são os solventes clorados tais como tricloroetileno (TCE) ou tetracloroetileno (PCE). Estes compostos são tóxicos, não só para o ser humano, mas também para microorganismos, particularmente TCE, que ainda é considerado um poluente prioritária pela Agência de Proteção Ambiental em os EUA 8. Neste trabalho propôs um experimento no qual a lama sulfidogenico é testado em sua capacidade de reduzir TCE em concentrações que estão na faixa relatada por compostos clorados biodegradação sob condições metanogênicas 9,10. Vale ressaltar que a maioria das pesquisas sobre a biodegradação de compostos clorados tem sido conduzida sob condições metanogênicas 9,10. Consideramos que a experiência com TCE proposto neste protocolo é um bom exemplo das aplicações potenciais do lodo. O objetivo deste experimento foi para eavaliar a tolerância das lamas ao TCE e o efeito sobre o sulfato de TCE redução da actividade. Tendo em conta que a maior parte da investigação sobre a biodegradação de compostos clorados é levada a cabo sob condições metanogénicas, este protocolo sugere a formação de uma lama pode ser utilizada para, simultaneamente: (1) remover o sulfato, (2) remover o COD e (3) remover compostos clorados. Um passo adicional pode ser a de avaliar a lama sobre a remoção simultânea de TCE e metais pesados (em adição ao sulfato e COD), duas condições que não podem ser avaliadas sob condições metanogénicas.Protocolo

Figura 1. Esquema para os passos do protocolo. Por favor clique aqui para ver uma versão maior desta figura.
1. Recolha sedimentos marinhos para a Formação do Sludge
- Identificar uma área submarina acessível quer perto de fontes hidrotermais (devido à presença de sulfuretos, o que pode indicar uma maior actividade redutora de sulfato) ou para uma área em que os restos de matéria orgânica são detectáveis.
- Para o propósito deste trabalho, levar cerca de 3 ou 4 kg de sedimentos e drenar a água fora das amostras. Colocar as amostras em sacos plásticos escuros. Não é necessária nenhuma refrigeração.
- Uma vez no laboratório, manter os sacos com as amostras na geladeira, se eles não estão indo para ser utilizado de imediato. Para os fins deste trabalho, SAMPles pode estar no frigorífico durante semanas ou meses antes de os utilizar.
- Tomar uma grande porção da amostra de sedimento (isto é, 1 ou 2 kg) e utilizar uma malha adequada (0,2 cm) para eliminar os sedimentos a partir de grandes fragmentos de material carbonado que pode ser encontrado ou algumas pedras que podem estar presentes.
Nota: Neste caso, uma malha de 0,20 cm de diâmetro (0,0767 in) foi utilizado, mas pode ser de um tamanho diferente de acordo com o tamanho das partículas na amostra.- Depois de passar o sedimento através da malha, misturar a porção seleccionada para promover que a porção é homogénea.
- Retirar amostras mais pequenas separadas (isto é, 2 a 3 g) para determinar os sólidos suspensos voláteis conteúdo (VSS), seguindo os métodos padrão 11.
Nota: Consulte a Figura 2 para obter os passos 1.2 a 1.4.

Figura 2. Fotografias das amostras de sedimentos.As amostras (A) de sedimentos apenas depois de ter sido feita. Amostra (B) de sedimentos depois de passar através da malha. (C) Amostra colhida para a pesagem antes de sólidos suspensos voláteis (VSS) determinação. A placa de Petri não precisam ser esterilizados. Por favor clique aqui para ver uma versão maior desta figura.
2. Bioreactor Set Up
- Para os fins deste trabalho, usar um reactor de vidro de UASB com um volume de trabalho total de 3 L. Em alternativa, usar um 1 ou 2 L de volume do reactor de vidro.
- Com base no conteúdo do VSS dos sedimentos calcular a quantidade de sedimento para ser usada como inoculo para se obter 5 g de VSS no 1 L.
- Levar em conta que, se a quantidade de sedimento após o cálculo é demasiado grande, o volume, em seguida, cerca de 25% a 30% do bioreactor deveria ser ocupada por os sedimentos em vez disso.
- Grave o conteúdo VSS, uma vez quevai mudar quando a comunidade microbiana é enriquecido no biorreator. O conteúdo VSS é necessária para os cálculos de sulfato de redução da atividade no biorreator.
- Assegure-se que a concentração final da solução de meio de tampão basal e no biorreactor é similar ao relatado por Guerrero-Barajas et ai. (2014) 12.
- Assegurar que os volumes finais dos sedimentos, meio basal, solução tampão e ácidos gordos voláteis são iguais ao volume de trabalho final do reactor. O meio basal 12 receita contém as concentrações apropriadas para a solução de traços de metais e vitaminas.
- Prepara-se uma solução stock de meio basal e solução tampão numa concentração apropriada para o volume de trabalho do reactor utilizado (ou seja, 2, 3 ou 4 vezes mais concentrado do que a relatada no passo 2.4) para garantir que, quando é diluída, é na concentração relatado por Guerrero-Barajas et ai. (2014)12).
Nota: A solução-mãe para o meio basal é sempre necessária, contudo, a solução tampão é necessário apenas no arranque. Não é necessário adicionar uma solução tampão depois de este tempo. - Prepara-se uma solução estoque de ácidos gordos voláteis: acetato, propionato e butirato de 2,5: COD proporção 1: 1. Leve em conta para os cálculos do acetato de sódio incluídos no meio basal. A concentração de COD final no reactor deve ser de 2,7 g / L.
Cuidado: Prepare esta solução em um exaustor. Use luvas de borracha nitrílica e óculos de proteção para a preparação desta solução. Ter em conta a estequiometria das reacções de sulfato com os ácidos gordos voláteis que é mostrado na Figura 3. - Prepara-se uma solução estoque de sulfato de sódio (Na 2 SO 4) em uma concentração apropriada para fornecer ao reactor uma concentração final de 4,000 mg / L do ião sulfato (SO 4 2-). Alternativamente, incluem the quantidade de sulfato necessário no meio basal em vez de adicionar-lo a partir de uma solução estoque, enquanto o sulfato de final (SO 4 2-) a concentração é direita.
- Colocar os sedimentos no reactor misturado com uma porção do meio de base para se certificar de que elas cheguem ao fundo do reactor.
- Adicionar o resto da solução de meio de tampão basal e misturado com a solução de ácidos gordos voláteis e da solução de sulfato. Certifique-se de que a solução de ácidos gordos voláteis é vertida para dentro do líquido. Nota: Realize esta etapa em um exaustor.
- Defina as conexões e tubulações do reator para a bomba de reciclagem. Definir a taxa de fluxo de reciclagem em 60 ml / min. Definir o biorreactor na câmara de temperatura de 34 ° C. Verifique regularmente se as variações de temperatura são pequenos (ou seja, 34 ± 1,7 ° C)
- Defina as conexões com a coluna de deslocamento de gás.
Nota: Consulte a Figura 4 para passos 2.1 a 2.5.
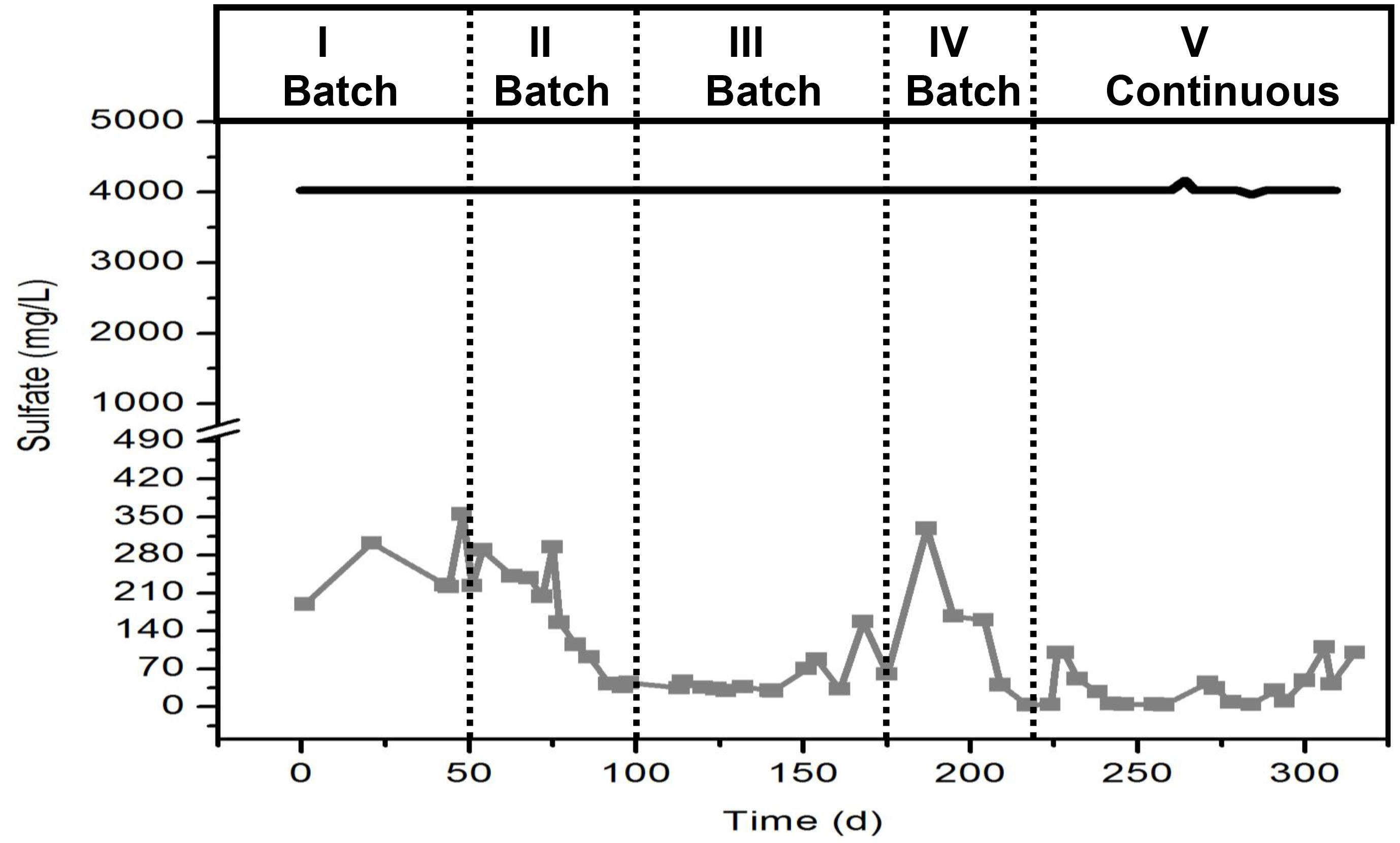
Figura 3. A estequiometria de redução de sulfato com VFA (acetato, propionato e butirato). Por favor clique aqui para ver uma versão maior desta figura.
Figura 4. reator UASB. (A) de tempo inicial. (B) regime contínuo depois de 300 dias de operação. Por favor clique aqui para ver uma versão maior desta figura.3. O funcionamento do reactor para promover Sulfidogenesis e crescimento dos microrganismos
Nota: Permitir para o inóculo para consumir a volatile ácidos gordos e sulfato. Para este efeito, esperar durante uma semana para efectuar a primeira análise para o sulfato, sulfeto e consumo de COD.
- Após uma semana de incubação tomar uma amostra de 5 a 7 ml de líquido para realizar a análise de CQO, sulfato e teor em sulfureto e de pH seguindo métodos convencionais 11, 13.
- Analise sulfureto no espectrofotometricamente líquido (no comprimento de onda (λ) de 670 nm), seguindo o método de azul de metileno 13.
- Colocar 5 ml de uma solução de acetato de zinco (2% w / w) num balão volumétrico de 25 ml, adicionar rapidamente 200 ul da amostra com a solução de acetato de zinco.
- Adicionar 2,5 ml de solução de uma N, N-dimetil-p-fenilenodiamina oxalato (DMP) (0,2% w / w em 20% de H 2 SO 4) e 125 ul de sulfato de ferro (III), solução de sulfato de amónio (10% w / w em 2% de H 2 SO 4) e completa com água destilada a 25 ml em frasco volumétrico. Aguarde 30 minutos para a reaçãoa ocorrer, o tempo em que a cor azul é estabilizada 13..
Nota: Espere pelo menos 15 minutos, mas não mais do que 60 minutos para testar as amostras no espectrofotômetro. Realizar a leitura da solução final azul no espectrofotómetro.
- Analise sulfato de acordo com métodos padrão 11. Aqui, quantificar sulfato como sulfato de bário, usando um método turbidimétrico.
- Colocar 5 ml de uma solução de condicionamento (ácido clorídrico HCl 1: 1) num balão volumétrico de 25 ml, adicionar 1 ml de amostra, previamente centrifugado (a 11.320 x g), para terminar a 25 ml do frasco volumétrico com água destilada e adicionar 1 g de cloreto de bário.
- Misture a solução durante 1 min num vórtice. Espere por 4 min para o sulfato de bário para formar e ler a amostra no espectrofotômetro em um comprimento de onda (λ) de 420 nm 11.
- Analise COD de acordo com métodos padrão 11. Como alternativa, use um COD Determinakit ção.
- Antes da determinação de COD, centrifugar a amostra cuidadosamente (11.320 xg a) para remover o sulfureto remanescente que pode interferir na determinação. Se necessário, centrifugar duas vezes: a primeira vez imediatamente após a colheita da amostra ea segunda vez esperar 6 ou 8 horas e, em seguida, realizar a análise COD.
- Adicionar 2 ml de amostra para um frasco de reacção do kit de determinação de COD, selar o frasco e homogeneizar a mistura por agitação suave. Prepara-se uma placa através da adição de 2 ml de água destilada para um outro frasco de reacção e homogeneizar a mistura.
- Colocar os tubos no reactor de digestão a 150 ° C durante 2 horas. Remover os frascos e deixe-os arrefecer no escuro. Tome as leituras dos frascos no espectrofotômetro em um comprimento de onda de 620 nm.
- Obter o volume de gás a partir da coluna de deslocamento de gás.
- Analise sulfureto no espectrofotometricamente líquido (no comprimento de onda (λ) de 670 nm), seguindo o método de azul de metileno 13.
- Aguarde até mais 5 a 7 dias até que o sulfato é consumido. Sulfato de COD e deve ser consumido numaproximadamente 85% a 90% antes de uma nova fornada alimentada é iniciado.
- Uma vez sulfato (e COD) são consumidos, completamente repita o passo 2.4. Fornecer meio fresco e novos nutrientes para cada lote.
- Repita os passos 3.1 e 3.2. Neste ponto, cada lote deve durar entre 7 e 10 dias.
- Quando 3 a 4 lotes foram concluídos, repita a etapa 2.4, mas aumentar a concentração de CQO a 4 g / L.
- Repita o passo 3.1 e 3.2 passo.
- Repetir o passo 3.3, mas aumentar a concentração de CQO a 6 g / L.
- Repetir 3.6 e 3.6.1 aumentando gradualmente até concentração de CQO é 10 g / L.
Nota: Adicione o gráfico que apresenta a concentração de sulfato (mg / L) em função do tempo (d).
- Quando o consumo de sulfato é superior a 80% em menos de 24 h e isto ocorre durante mais de uma semana, mudar o funcionamento do reactor de modo contínuo. Para o modo contínuo definir o tempo de retenção hidráulica (TRH) em 24 horas e manter a concentração de sulfato a 4 g / L e o CODem 10 g / L.
Nota: Com o tempo o consumo deve ser sulfato mais rápido.
4. redutoras de sulfato atividade de teste
- Antes deste teste se certificar de que o biorreactor em regime contínuo apresenta variação inferior a 10% na concentração de sulfato restante.
- Em um determinado dia, parar o reactor após um ciclo HRT e conduta passo 2.4. Para o passo 2.4.3 utilizar uma concentração de CQO de 10 g / L.
- Uma vez que o bioreactor é alimentado, levar de 5 a 7 ml de amostras do líquido e para executar a análise COD, sulfato, sulfeto (passo 3.1) e o pH a cada hora. Anote o volume de gás produzido.
- Calcule o sulfato de reduzir a atividade de acordo com a literatura 14.

SRA = sulfato de redução da atividade (mg COD-H 2 S) / gVSS * d
m H 2 S = concentração, expressa em mg de sulfureto de COD-H 2 S
VSS = volátil concentração de sólidos em suspensão
t = tempo (d ou h)
- Adicione os gráficos correspondentes que mostram a percentagem de consumo de sulfato versus concentração de sulfureto ao longo do tempo, em mg / L. Faça os gráficos que mostram percentagem de consumo de COD ao longo do tempo. Adicione os gráficos que mostram a variação do pH ao longo do tempo.
5. O tricloroetileno (TCE) Teste de Redução
- Antes deste teste se certificar de que o bioreactor está a trabalhar em regime contínuo e mostra uma variação inferior a 10% na concentração de sulfato restante. Não inicie esse teste se a redução de sulfato em biorreator é inferior a 90%.
- Prepara-se uma solução estoque de tricloroetileno (TCE), tendo em conta que a concentração final desse composto na fase líquida do bioreactor deve ser de 300 uM. Considere o partitioning do composto para o espaço de topo, utilizando a constante adimensional Lei Henry's (H') de TCE a 34 ° C. H'at 34 ° C durante TCE é 0,4722.
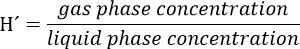
Cuidado: Prepare esta solução num fumehood e usar luvas e óculos de proteção.- Por exemplo, para uma solução stock de 5.000 mM, calculada como segue:
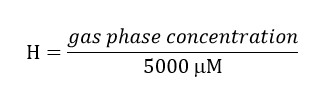
TCE concentração da fase gasosa = (0,4722) * (5.000) = 2.139 M. Inclua essa concentração na preparação da solução de estoque uma vez que esta quantidade de TCE será no espaço de topo.
Em seguida, no líquido (água) da solução de reserva, a concentração de TCE real será: 5,000 + 2,139 = 7,139 uM. Densidade TCE = 1,43 g / ml. Converter a 7139 uM de Mg e, em seguida, utilizando a densidade de TCE calcular o volume de TCE para a solução estoque.
Nota: A concentração da solução-mãe de TCE may ser inferior a 5,000 uM, isto é, 3000 ou 1000 uM, este depende da quantidade de volume desta solução pode ser entregue para o bioreactor de acordo com o seu volume de fase líquida.
- Por exemplo, para uma solução stock de 5.000 mM, calculada como segue:
- Prepare curvas padrão no cromatógrafo a gás para TCE, cis 1,2-dicloroetileno, trans -1,2-dicloroetileno, cloreto de vinilo e etileno. Prepare os cis -1,2-dicloroetileno e trans curvas padrão -1,2-dicloroetileno a partir de uma solução estoque de estes compostos, seguindo o mesmo procedimento descrito no ponto 5.2 para a solução estoque TCE. Preparar as curvas padrão para o cloreto de vinilo e etileno, diluindo a concentração de cada um dos gases a partir dos padrões (cilindros de gás).
- Preparar as curvas padrão destes compostos em um intervalo de 20 a 300 | iM. Usar o método relatado por Guerrero-Barajas et ai. (2011) 15, para a análise destes compostos no cromatógrafo de gás.
Cuidado: Prepare estes sesoluções ard em um fumehood e usar luvas e óculos de proteção.
- Preparar as curvas padrão destes compostos em um intervalo de 20 a 300 | iM. Usar o método relatado por Guerrero-Barajas et ai. (2011) 15, para a análise destes compostos no cromatógrafo de gás.
- Em um determinado dia, parar o reactor após um ciclo HRT e conduta passo 2.4. Para o passo 2.4.3 utilizar uma concentração de CQO de 10 g / L.
- Uma vez que o bioreactor é alimentado, adicionar o TCE directamente para o líquido no bioreactor a partir da solução de reserva preparada no ponto 5.2, a concentração de TCE final na fase líquida do bioreactor deve ser de 300 uM. Defina a HRT a 12 hr.
- No final de um ciclo de HRT tomar amostras do líquido (500 a 1.000 ul) e análise de conduta para CQO, de sulfato e de sulfureto (passos 3.1.1, 3.1.2 e 3.1.3). Tome amostras do headspace (100 a 250 ul) e realizar análise de TCE, cis 1,2-dicloroetileno, trans -1,2-dicloroetileno, cloreto de vinilo e eteno no cromatógrafo a gás.
- Repita o passo 2.4. Para o passo 2.4.3 utilizar uma concentração de CQO de 10 g / L.
- Não repetir qualquer teste de redução TCE até o biorreator apresenta mais de 90% de redução de sulfato e menos variação de 10% em ambos, a redução do sulfato e sulfato restante no bioreactor.
- Repetir 5.4, 5.5 e 5.6 de duas ou três vezes mais.
- Retirar amostras de sedimentos (0,5 g) para realizar a identificação dos microrganismos apenas depois de um teste de redução de TCE tenha terminado. Faça isso depois de testes de redução do TCE 2 ou 3.
6. redutoras de sulfato atividade de teste após a redução TCE Experiment
- Repita o passo 4 completamente.
7. Identificação dos microorganismos
- Recolher amostras de lodo de cerca de 0,5 g cada e realizar a extração de RNA total acordo com o método padrão de 12.
- Amplificar o gene do rRNA 16S com a transcrição reversa e conduzir a reacção em cadeia da polimerase (RT-PCR) um passo 12.
- Desenhar os primers para amplificar ou usar como uma primeira abordagem os sugeridos na literatura 11. Siga o amplificação procedimento sugerido na literatura 12.
- Construir as bibliotecas 16S rRNA. Amplicons de PCR pode ser clonado usando um kit de clonagem-11. Tipicamente, 10 colónias de cada placa (cada colónia representa um produto de PCR) pode ser clonado. Prepara-se o ADN de plasmídeo para sequenciação de acordo com o procedimento sugerido na literatura 12.
- Realizar o sequenciamento de fragmentos. Re-amplificar aproximadamente 1400 pb com os produtos de PCR externos com o protocolo para a amplificação por PCR como descrito anteriormente (passo 7.4) e o clone de acordo com o procedimento sugerido na literatura 12. Isolar o plasmídeo recombinante a partir de E. colônias coli como sugerido na literatura 12. Não realizar o procedimento parcial para o seqüenciamento com primers universais M13 12.
- Conduzir a análise de sequências. Alinhe as sequências de nucleótidos utilizando o Clustal X e ajustar manualmente no editor de texto. Executar pesquisas BLAST das databas NCBIe. (http://www.ncbi.nlm.nih.gov/BLAST/Blast.cgi) 12.
- Obter os números de adesão sequência de nucleótidos. Deposite as sequências de nucleótidos dos clones identificados na base de dados EMBL sequência de nucleótidos (Gen-Bank / EMBL / DDBJ) sob os números de acesso correspondentes (ou seja, JQ713915eJQ713925 para sequências de amplicons) 12.
Resultados
Um comportamento típico da redução do sulfato no bioreactor é mostrado na Figura 5. É importante notar que, durante as primeiras semanas de redução de sulfato de operação será lenta. No entanto lento, o consumo de mais de 90% de sulfato de ao longo do tempo indica que o inoculo é o desenvolvimento de uma comunidade microbiana capaz de reduzir e, portanto, sulfato, enriquecida em bactérias redutoras de sulfato. Os diferentes períodos na figura indicam que a redução de sulf...
Discussão
Existem várias aplicações de sulfidogenesis em biotecnologia ambiental, uma das aplicações mais utilizadas do metabolismo de bactérias redutoras de sulfato em consórcios com bactérias fermentadoras está em tratamento de águas residuais. Reatores UASB estão entre as principais abordagens de engenharia para tratamento de efluentes industriais com elevadas concentrações de sulfato. Neste trabalho, apresentamos um protocolo para obter lodo sulfidogenico de sedimentos marinhos em um reator UASB. Os passos críti...
Divulgações
The authors Selene Montserrat García-Solares, Claudio Garibay-Orijel and Claudia Guerrero Barajas submitted (as inventors) in 2012 the application to obtain a patent entitled: “Process for the anaerobic treatment of industrial wastewater with high content of sulfate and chlorinated compounds”. This submission has been approved to the stage of the formatting. The submission has Instituto Politécnico Nacional as the rights owner.
Agradecimentos
The authors are grateful for the financial support provided by Instituto Politécnico Nacional grants 20120110, 20130399 and 20140239 SIP and also by Instituto de Ciencia y Tecnología del Distrito Federal Mexico (PICS 08-79, ICYT-DF, 2009-2012). Thanks also to CONACYT – Mexico for the graduate scholarship (225806) awarded to Selene Montserrat García-Solares and for the financial support provided by grant 82627.
Materiais
| Name | Company | Catalog Number | Comments |
| trichloroethylene | sigma Aldrich | 251402 | |
| cis-1,2-dichlorotehylene | sigma Aldrich | ||
| trans-1,2-dichloroethylene | sigma Aldrich | D-62209 | |
| vinyl chloride scotty standard | supelco | 1,000 ppm v/v in nitrogen | |
| ethene scotty standard | supelco | 99% purity | |
| pump | Masterflex | Model 7553-75 | |
| spectrophotometer | any | ||
| microcentrifuge | any | ||
| gas tight syringes | any | 100 and 200 microliters | |
| UASB glass reactor | any | under design | |
| gas chromatograph | any | FID detector | |
| capillary column SPB-624 | supelco | ||
| pH meter | any | ||
| viton tubing | Masterflex | ||
| basal medium reagents | any | ||
| trace metals reagents | any | ||
| vitamins solution reagents | any | ||
| sodium sulfate | any | ||
| volatile fatty acids | any | ||
| COD determination kit | HACH | range 0-15,000 mg/L | |
| TOPO-TA cloning kit pCR®4.0 | Invitrogen, US | ||
| S.N.A.P. TM Miniprep Kit | Invitrogen, UK | ||
| Pure link TM Quick Plasmid Miniprep kit | Invitrogen |
Referências
- Lens, P., Esposito, M. V. G., Zandvoort, M. Perspectives of sulfate reducing bioreactors in environmental biotechnology. ReViews Environmental Science and Biotechnology. 1 (4), 311-325 (2002).
- Omil, F., Lens, P., Hulshoff, P., Lettinga, G. Characterization of biomass from a sulfidogenic, volatile fatty acid-degrading granular sludge reactor. Enzyme and MicrobialTechnology. 20, 229-236 (1997).
- Lopes, S. I. C., Wang, X., Capela, M. I., Lens, P. N. L. Sulfate reduction during the acidification of sucrose at pH 5 under thermophilic (55 °C) conditions.II: Effect of sulfide and COD/SO4-2 ratio. Bioresource Technology. 101, 4278-4284 (2010).
- Alfonso, P., Prol-Ledesma, R. M., Canet, C., Melgarejo, J. C., Fallick, A. E. Sulfur isotope geochemistry of the submarine hydrothermal coastal vents of Punta Mita, Mexico. Journal of Geochemical Exploration. 78-79, 301-304 (2003).
- Valdemarsen, T., Kristensen, E. Degradation of dissolved organic monomers and short chain fatty acids in sandy marine sediment by fermentation and sulfate reduction. Geochimica et Cosmochimica Acta. 74, 1593-1605 (2010).
- Quistad, S. D., Valentine, D. L. Anaerobic propane oxidation in marine hydrocarbon seep sediments. Geochimica et Cosmochimica Acta. 75, 2159-2169 (2011).
- Futagami, T., Morono, Y., Terada, T., Kaksonen, A. H., Inagaki, F. Dehalogenation activities and distribution of reductive dehalogenase homologous genes in marine subsurface sediments. Applied and Environmental Microbiology. 75 (21), 6905-6909 (2009).
- U.S. Environmental Protection Agency. List of priority pollutants. Clean Water Methods. , (2014).
- Ozdemir, C., Dursun, S., Karatas, M., Sen, N., Sahinkaya, S. Removal of trichloroethylene (TCE) in upFlow anaerobic sludge blanket reactors (UASB). Biotechnology and Biotechnological Equipment. 21 (1), 107-112 (2007).
- Zhang, Y., Wang, X., Hu, M., Li, P. Effect of hydraulic retention time (HRT) on the biodegradation of trichloroethylene wastewater and anaerobic bacterial community in the UASB reactor. Applied Microbiology and Biotechnology. 99, 1977-1987 (2015).
- . . Standard Methods for the Examination of Water and Wastewater. , (1998).
- Guerrero-Barajas, C., et al. Enhanced sulfate reduction and trichloroethylene (TCE) biodegradation in a UASB reactor operated with sludge developed from hydrothermal vents sediments: process and microbial ecology. International Biodeterioration and Biodegradation. 94, 182-191 (2014).
- Trüper, H. G., Schlegel, H. G. Sulphur metabolism in Thiorhodaceae I. Quantitative measurements on growing cells of Chromatium okenii. Antoine van Leeuwenhoek. 30, 225-238 (1964).
- Gallegos-García, M. G. . Biological processes of sulfate reduction in biofilms for metals precipitation [Ph D thesis]. , (2009).
- Guerrero-Barajas, C., Garibay-Orijel, C., Rosas-Rocha, L. E. Sulfate reduction and trichloroethylene biodegradation by a marine microbial community from hydrothermal vents sediments. International Biodeterioration and Biodegradation. 65, 116-123 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados