Method Article
Warum Quantifizierung von Bedeutung: Charakterisierung von Phänotypen bei der
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Morphology, size and location of intracellular organelles are evolutionarily conserved and appear to directly affect their function. Understanding the molecular mechanisms underlying these processes has become an important goal of modern biology. Here we show how these studies can be facilitated by the application of quantitative techniques.
Zusammenfassung
Die meisten Studien über Morphogenese verlassen sich auf qualitative Beschreibungen, wie anatomische Merkmale durch die Unterbrechung von spezifischen Genen und genetischen Pfade betroffen sind. Quantitative Beschreibungen werden selten durchgeführt, obwohl genetische Manipulationen eine Reihe von phänotypischen Effekte erzeugen und Variationen sind auch unter den Individuen innerhalb der Kontrollgruppe beobachtet. Beweise Schwellen zeigt, dass die Morphologie, Größe und Lage von Organellen eine bisher unterschätzten, aber fundamentale Rolle in der Zellfunktion und das Überleben spielen. Hier stellen wir Schritt- für -Schritt - Anleitungen zur Durchführung von quantitativen Analysen von Phänotypen an der Drosophila Larven neuromuskulären Synapse (NMJ). Wir verwenden mehrere zuverlässige immunhistochemischen Marker mit Bio-Imaging-Techniken kombiniert und morphologisch analysiert die Auswirkungen von genetischen Mutationen auf spezifische zelluläre Prozesse zu untersuchen. Insbesondere konzentrieren wir uns auf die quantitative Analyse von Phänotypen Morphologie, Größe und Position von n zu beeinflussenuclei innerhalb der quergestreiften Muskeln von Drosophila - Larven. Die Drosophila Larven NMJ ist eine wertvolle experimentellen Modell die zugrunde liegenden molekularen Mechanismen , die die Struktur und die Funktion des neuromuskulären Systems, sowohl bei Gesundheit und Krankheit zu untersuchen. die Methoden wir hier beschreiben, können jedoch auch auf andere Systeme ausgedehnt werden.
Einleitung
Qualitative analysis restricts the focus of most experimental studies to the examination of genetic manipulations leading to large phenotypic effects or to phenotypes that are not amenable to quantification, e.g., absence/presence. Quantification of phenotypes is not usually performed and variations present within members of a phenotypic group are not taken into consideration. Additionally, without mathematical descriptions of morphologies, it may be difficult to determine whether fine scale phenotypic changes are the result of genetically induced alterations or whether observed changes are simply due to random fluctuations.
We propose that for an accurate and unbiased analysis of phenotypic defects due to gene disruption, quantitative methodologies should accompany more traditional qualitative approaches. Quantitative evaluation of phenotypes is particularly beneficial for structures such as synapses that present a high degree of variability due to their intrinsic morphological and functional plasticity. We have selected examples of quantitative analyses applied to areas of investigation with which we are most familiar, namely the Drosophila larval neuromuscular junctions (NMJs). However, concepts and principles apply equally well to other experimental systems.
The larval NMJ is an excellent model system to study synaptic development and function because of the highly stereotyped nature of its structure. Each hemi-segment of the larval neuromuscular system contains 32 identifiable motor neurons forming synaptic contacts with their postsynaptic target muscle. Every hemi-segment also contains a fixed number of muscles visible as multinucleated fibers attached to the internal surface of the cuticle1. Another advantage of using the larval neuromuscular system is the power and versatility of Drosophila genetics that easily allows the generation of a number of mutant alleles and the possibility to modify gene expression in a time- and tissue-restricted manner. Finally, 75% of the human genes causing a disease have an evolutionarily conserved orthologue in Drosophila2. Indeed, entire genetic pathways are conserved between flies and humans. Because of this, the Drosophila larval neuromuscular system is a very popular experimental model to elucidate the molecular mechanisms underlying a number of human diseases including amyotrophic lateral sclerosis (ALS)1.
Here we show that the availability of several reliable immuno-histochemical markers, combined with bio-imaging techniques and accurate morphometric analyses can describe anatomical traits that are likely to play an important functional role3,4,5. Among the cellular processes that are amenable to quantitative analyses, we focus on changes in shape, size and position of intracellular structures such as the nuclei. All these are processes that we know very little about.
The challenge for molecular geneticists in the coming decades will be to extend our current knowledge by analyzing the effect of genetic mutations that produce very subtle phenotypic defects. Quantitative methodologies that allow researchers to meticulously explore the effects of genetic mutations can provide a more comprehensive understanding of how genotypes relate to phenotypes, especially for poorly understood cellular processes.
Protokoll
1. Experimentelle Vorbereitung
Hinweis: Dissektionen und Immunhistochemie Verfahren in den Abschnitten 2 und 3 werden nach Referenzen durchgeführt 3-6, aber mit Modifikationen.
- Vorbereitung 1x phosphatgepufferter Kochsalzlösung (PBS), und PBS enthaltend 0,1% Triton-X 100 (PBT). Halten Sie sie auf Eis.
- Bereiten Sie Bouin-Fixiermittel (15 Pikrinsäure: 10 Formaldehyd: 1 Eisessig). Machen Sie dieses Reagenz frisch.
- Wählen Sie sauber Edelstahl minutien Pins und einer feinen Pinzette.
- Bereiten Sie Dissektion Platten eine Sylgard Scheibe in einer 5 cm Petrischale enthält.
2. Dissektion der im dritten Häutungsstadium Larval NMJs
- Pick-Wanderung im dritten Häutungsstadium Larven aus einem Fläschchen oder einer Flasche mit einem feinen Pinsel und legen Sie sie in eine 2 cm Petrischale mit 4 ° C PBS die restliche Nahrung weg zu waschen.
- Legen Sie eine Larve auf der Oberseite der Sylgard Oberfläche der Dissektion Platte und stellen Sie sicher, dass es positi istoned mit seiner dorsalen Seite, so dass die beiden Längs Trachealtuben auf der Oberseite sichtbar sind.
- die Zange Mit dem Stift, Pin die Larve nach unten an seinem vorderen Ende, direkt unter dem Mund Haken zu halten. Strecken Sie die Larve aus so viel wie möglich und Stift ihrem hinteren Ende nach unten.
- Fügen Sie genug PBS Salz die Wände der Platte zu erreichen und tauchen vollständig die Larve.
- Wiederholen Sie die Schritte 2.1 bis 2.3 für andere Larven des gleichen Genotyps. Eine einzige 5 cm Petrischale Dissektion Platte kann bis zu 8 Larven leicht unterbringen.
- Mit Hilfe der Mikro-Dissektion Schere leicht anheben, die dorsalen Kutikula und machen einen kleinen horizontalen Schnitt am hinteren Ende in der Nähe der Stift.
- Legen einer Schere in den Einschnitt, schneiden Sie die Larve den ganzen Weg bis zum vorderen Ende entlang der Mittellinie zwischen den beiden Längsstriche der Trachea. Stellen Sie sicher, dass die Mittellinie schneidet oberflächlich genug, nur die Kutikula durchlaufen und zu vermeiden, durch die Muskeln Schneiden aufder Bauchseite.
- An jedem Ende geschnitten, um zwei Kerben an der linken und der rechten Seite.
- Öffnen des Kehl indem zwei Stifte auf beiden Seiten des vorderen Einschnitts. Wiederholen Sie das gleiche mit dem hinteren Ende. Wenn die Stifte platzieren, stellen Sie sicher auseinander, um die Körperwand zu verbreiten.
- Reinigen Sie die inneren Organe mit einer Pinzette und PBS Salzlösung. Lassen Sie das zentrale Nervensystem intakt. dehnen Sie vorsichtig die Larve mit Eckpunkten, bis sie vollständig gestreckt wird, aber stellen Sie sicher, dass die Muskeln nicht während dieses Prozesses zerrissen.
- Wiederholen Sie die gleiche Präparation Vorgang für die andere Larven auf der gleichen Präparation Platte.
- Waschen mit PBS Salzlösung dreimal alle inneren Organe zu entfernen.
- Ersetzen Sie die PBS mit Bouin-Fixiermittel und lassen für 10 Minuten bei Raumtemperatur.
- Waschen Sie mehrmals mit PBT.
- Entfernen Sie die Stifte sorgfältig und übertragen alle Vorbereitungen, die jetzt ziemlich starr sind, in ein 1,5 ml Mikrozentrifugenröhrchen für Immunfärbung.
3. immunhistochemischen Anfärbung von Drosophila NMJs mit spezifischen Antikörpern für Muskeln und myonuclei
- Schnell die Larven Filets in PBT spülen.
- Blockieren Sie die Vorbereitungen mit 10% normalem Ziegenserum inkubiert (NGS) in PBT für 2 Stunden unter ständigem Rühren.
- Inkubieren eines Satzes von sezierten NMJs in PBT 5% NGS und ein Kaninchen-anti-lamin-Antikörper (bei einer Konzentration von 1: 500) enthält, und mit einem Meerschweinchen anti-DVAP Antikörper (bei einer Konzentration von 1: 500) für 2 Std bei Raumtemperatur oder über Nacht bei 4 ºC. Die Anti-DVAP Antikörper färbt die quergestreifte Muskulatur, während die anti-Lamin Färbung der Kontur des myonuclei erkennt.
- Waschen Sie einmal schnell in PBT, die Antikörper im Überschuss zu entfernen. 2 h in PBT Waschen durch die PBT-Puffer alle 15 min zu ändern.
- Inkubieren der Proben in PBT 5% NGS und fluoreszenzmarkierten Sekundärantikörper bei 1 enthält: 500-Verdünnung für 2 h bei Raumtemperatur. Die gleichen Proben ceine unterzogen werden, mit mehreren Antikörpern gegen Färbung zur gleichen Zeit, wenn Sekundärantikörper konjugiert ist mit unterschiedlichen Chromophoren verwendet werden.
- Entfernen Sekundärantikörper und waschen in PBT für 2 Stunden durch die PBT alle 15 min Puffer zu ändern.
- Färben das Innere des myonuclei die Proben dreimal mit PBS waschen und gehen Sie wie folgt vor:
- Fügen Sie den Kernmarker TO-PRO-3 in einer Verdünnung von 1: 1000 in PBS und Inkubation für 20 Minuten unter ständigem Rühren. Dieser Marker wird in dieser Studie aber jede andere im Handel erhältliche Kernmarker können ebenfalls verwendet werden.
- Waschen Sie schnell dreimal in PBS vor der Montage.
4. Montage Proben auf dem Objektträger
- Wählen Sie die Proben mit einer Pinzette aus der 1,5-ml-Mikrozentrifugenröhrchen und legen sie auf einer Verarbeitungs nach unten rutschen.
- Durch die Verwendung von Mikro-Dissektion Schere, schneiden Sie den Kopf und den Schwanz der Filets und halten ihre innere Oberfläche auf.
- bereiten Sie the Montagegleitzone durch Umwickeln drei Streifen aus Zellstoffband auf jeder Seite eines sauberen Objektträger in einem Abstand von etwa 1 cm voneinander. Sobald das Deckglas auf der Oberseite der beiden Streifen positioniert ist, wird eine Lücke erzeugt, die von den Proben Abflachung vermeiden. Dies ist entscheidend, wenn dreidimensionale Volumen Renderings von Strukturen gemacht werden.
- Setzen Sie einen kleinen Tropfen von etwa 20 & mgr; l des Eindeckmedium in der Mitte des Aufnahmeschlitten zwischen den drei Cellulosebandstreifen.
- Das Eindeckmedium mit sauberen Pinzette Nach dem Verteilen, ziehen Sie die seziert Larven an der Befestigungsschieber in die Montagemedium, die Innenfläche nach oben zu halten. Versuchen Sie, sie in Reihen von vier oder fünf zu montieren.
- Drop vorsichtig mit einem Deckglas auf der Oberseite des Befestigungsschlitten und stellen Sie sicher, dass keine Luftblasen erzeugt werden. Siegel die Folie mit transparentem Nagellack. Lassen Sie die Proben für mindestens 10 min vor der Bebilderung trocknen.
5. Die konfokale Einstellungen für Imaging
Hinweis: Bilder in dieser Studie vorgestellt werden, damit eine Nikon A1R konfokalen Einheit mit integriert auf einem Ti: E inverses Mikroskop. Jedoch kann jedes konfokalen Mikroskop mit einem Minimum von 3 Lasereinheiten in den Wellenlängenbereichen von 488 nm, 561 nm und 642 nm und einem 3-Kanal-Detektionssystem ist für diesen Zweck geeignet.
- Schalten Sie den Laser, Detektoreinheit, Quecksilber-Lampe, Tischsteuerung, Mikroskop und den PC. Die Steuersoftware starten und sichern Sie den Schlitten auf der Bühne Halter.
- Um Bildgebung schneller, wählen und alle Regionen von Interesse (ROI) auf die Probe zu markieren, indem ein 20x-Objektiv verwendet wird.
- Vorsichtig schwenken die Mundstück auf die 60X höhere Vergrößerung Objektivlinse (60X Plan Apo VC / NA 1.4 OIL).
- Geben Sie einen Tropfen des Eintauchens Öl auf die Objektivlinse und wählen Sie eine der markierten ROI von XYZ-Übersichtsfenster auf dem Computer.
- Starten Sie Bildgebung durch die folgenden optischen Einstellungen: Wählen Sie die erste dichroitische Spiegel: 405/488/561/640. Wählen 488 nm-Laser mit Emissionsfilter 525/50 nm in Kanal 1, 561 nm-Lasers mit 595/50-Emissionsfilter in Kanal 2 und 642 nm-Laser mit Langpassfilter 650 nm in Kanal 3.
- Verwenden Sie die folgenden Scan-Einstellungen vor der Bildaufnahme starten: Wählen Sie Galvano-Scanner; Scan-Richtung: ein Weg; Scangeschwindigkeit: 0,5 Bilder pro Sekunde.
- Wählen Sie Channel-Serie Kanaldurchbluten zu vermeiden.
- Stellen Sie die Lochgröße auf 1 luftigen Einheit. Scannen und stellen Sie die Laserleistung, Detektions Verstärkungs- und Offset in geeigneter Weise für jeden Kanal Pixelsättigung und Hintergrundpegel zu vermeiden.
- Erwerben z-Stapel von Voxel Größe unter Verwendung von 0,2 x 0,2 x 0,5 um 3 für alle ROIs und der Objektträger - Präparate. Wenn die Bilder auf Entfaltungs unterworfen werden setzt dann die Voxel - Größe bis 0,06 x 0,06 x 0,15 um 3.
- Speichern von Bildern in .nd2 Dateiformat oder ics-Format.
6. Berechnung des Abstandes zwischen Nuclei innerhalb Striated60; Muskeln durch die Methode der Analyse nächstgelegener Nachbar
- Für diese Analyse verwenden Konfokalbilder Larvenkörperwand Muskeln gefärbt mit DVAP Antikörper Darstellung der Muskeln und mit Lamin-Antikörpern und einem Kern Marker sichtbar zu machen, die Kerne zu markieren.
- Um die nächste Abstand zwischen den Kernen abschätzen zu können , verwenden Messpunkte innerhalb des MeasurementPro Modul der Bildanalyse - Software (zB Imaris). Andere ähnliche Softwareanwendungen für die Bildanalyse kann für die gleichen Zwecke verwendet werden.
- Öffnen Sie die konfokale Bilder. So starten Sie einen Doppelklick auf das Software-Symbol. Drag & Drop die konfokale z-Stapel-Bilder in die Arena.
- Klicken Sie doppelt auf die Bilder um sie zu öffnen automatisch in der Ansicht Surpass, unter dem Menüwerkzeugleiste Symbol
 . Die Surpass View verfügt über drei Hauptarbeitsplatten: Blick Region, Objektliste und Objekteigenschaften Bereich.
. Die Surpass View verfügt über drei Hauptarbeitsplatten: Blick Region, Objektliste und Objekteigenschaften Bereich. - Erstellen Sie ein Volumen gemacht trei Kanal Bild von 3D-Ansicht auf das Menü-Symbol klicken
 .
. - Klicken Sie auf das Hinzufügen neuer Messpunkte Symbol
 von dem Symbolleiste Objekte und die Erstellung Assistenten, die im Objekteigenschaften Bereich angezeigt wird.
von dem Symbolleiste Objekte und die Erstellung Assistenten, die im Objekteigenschaften Bereich angezeigt wird. - Vom Assistenten zum Erstellen von auf die Registerkarte Bearbeiten auswählen und dann bestimmten Kanal. Wählen Sie entweder die Kernmarker oder den Lamin-Kanal um die Kerne zu markieren.
- Stellen Sie den Zeiger auf den Select-Modus, indem Sie die Esc-Taste Tab auf der Tastatur.
- Passen Sie die Größe des 3D-Cursor-Box mit dem Mausrad einen gegebenen Kern im Bild enthalten. Fügen Sie einen Messpunkt durch die Shift-Taste und der linken Maustaste auf dem gleichen Kern zu halten.
- Fügen Sie den zweiten Punkt auf einem nahe gelegenen Kern auf dem gleichen Muskel durch die vorherigen Schritte wiederholen. Eine Linie wird zwischen den zwei Punkten und dem gemessenen Abstand automatisch gezogen zwischen die beiden Kerne wird angezeigt. Der Abstand zwischen den beiden Kernen ist nun als statistische Variable im Objekteigenschaften-Bereich unter der Registerkarte Statistik> Detaillierte> Entfernung Daten aufgezeichnet.
- Wiederholen Sie die Schritte 6,6-6,10 für alle Kerne eines gegebenen Kern umgibt.
- Aus Statistiken> Detail> Entfernung Daten alle erfassten Messpunkte angezeigt werden, wählen Sie den kürzesten Abstand.
- Mit einem Klick auf den Export Statistik Tab Anzeige in Datei
 unter dem Objekteigenschaften Bereich zur Verfügung, werden die Daten auf einer Tabelle gespeichert.
unter dem Objekteigenschaften Bereich zur Verfügung, werden die Daten auf einer Tabelle gespeichert. - Wiederholen Sie den Vorgang für die anderen umgebenden Kerne und für eine ausgewählte Anzahl von Muskelfasern pro Genotyp.
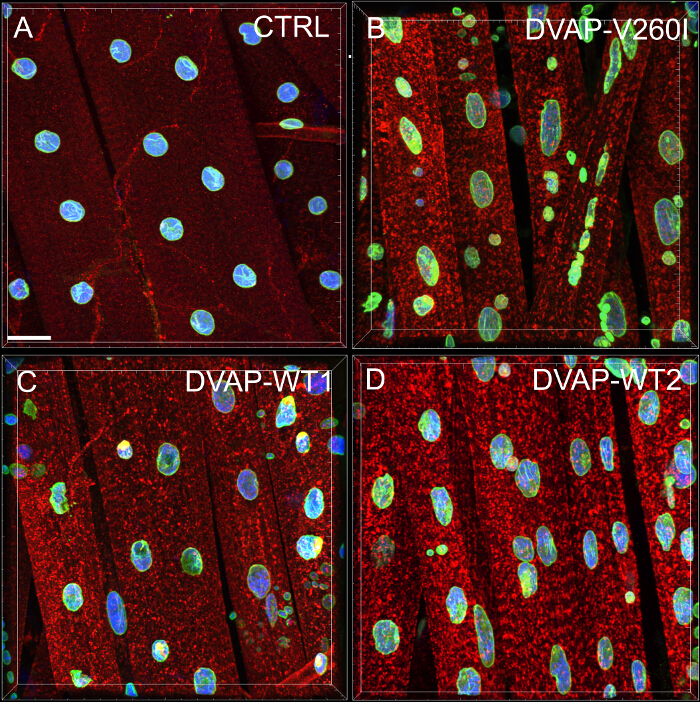
- Mit Hilfe der in ein Tabellenkalkulationsdatei exportiert Daten, die Berechnung der durchschnittlichen kürzesten Abstand (D ave) für alle M Anzahl von Muskeln , um die folgende Gleichung:
OAD / 53821 / 53821eq1.jpg "/>
Di ist der kürzeste Abstand zum benachbarten Zellkern für einen gegebenen Kern i, wo i von 0 bis N variiert und N die Anzahl der Kerne pro Muskel analysiert. Die Summierung von Werten von j = 0 bis j = m bezeichnet die Anzahl M der untersuchten Muskeln. - Alternativ verwenden Spots Assistent für die Erstellung und Spots zu Spots geringste Abstand der durchschnittlichen Entfernung zum nächstgelegenen Nachbar Kern zu schätzen. Dazu wird ein Matlab-Erweiterungsmodul erforderlich.
- Klicken Sie doppelt auf ein bestimmtes Bild auf der Arena und einem 3D-Volumenbild wird im Anzeigebereich angezeigt werden. Klicken Sie auf das Objekterstellung Symbol und New Spots
 aus der Symbolleiste Objekte.
aus der Symbolleiste Objekte. - Vom Assistenten zum Erstellen im Objekteigenschaften-Bereich, klicken Sie auf die Option Überspringen Automatische Erstellung, Bearbeitung manuell.
- Wählen Sie entweder den Lamin oder Kernmarker-Kanal die Kerne anzuzeigen.
- Umschalttaste und klicken Sie auf left Maustaste auf alle Kerne eines bestimmten Muskels in dem Bild. Ein Fleck auf jedem Kern erscheint.
- Wählen Sie Spots zu Spots Closest Entfernung zu finden unter der Registerkarte Extras im Objekteigenschaften-Area. Wählen Sie Spots Statistiken unter dem Ergebnis Modus und einem Matlab-Fenster erscheint.
- Unter der Registerkarte Statistik, wählen Sie eine detaillierte und dann bestimmte Werte. Klicken Sie auf das Distmin und die Werte der Mindestabstände für jeden Kern wird angezeigt.
- Exportieren Sie die Daten in ein Tabellenkalkulationsdatei und berechnen den Mittelwert dieser Abstände pro Muskel. Wiederholen Sie den Vorgang für alle Muskeln eines gegebenen Genotyps.
- Klicken Sie doppelt auf ein bestimmtes Bild auf der Arena und einem 3D-Volumenbild wird im Anzeigebereich angezeigt werden. Klicken Sie auf das Objekterstellung Symbol und New Spots
7. Bestimmung der Form von Nuclei innerhalb der Körperwand Muskeln von Drosophila - Larven
- Für diese Analyse verwenden konfokale Bilder der Körperwand Muskeln gefärbt mit Lamin und einem Kernmarker die Kerne sichtbar zu machen.
- Um die Form der myonuclei, messen Sphärizität (definiert als das Verhältnis von der Auswertungs surGesichtsbereichs einer Kugel mit dem gleichen Volumen wie dem Kern gegeben, um die Oberfläche des Kerns) oder Elliptizität (unterscheidet zwischen abgeplatteten / prolate Ellipsoide und Sphäroide).
- Öffnen das Bild wie zuvor beschrieben. Klicken Sie auf die Symbolleiste Objekte Symbol hinzufügen Neue Oberflächen
 .
. - In der Assistent für die Erstellung, die im Objekteigenschaften-Gebiet erscheint, wählen Sie die Kernmarker-Färbung als Quellkanal die Kerne anzuzeigen.
- Setzen Sie die Option Absolute Intensität als die Schwelle. Stellen Sie sicher, dass die meisten der Kerne eine glatte und nicht überlastet Rendering zeigen, indem sie den Wert auf der Schwellenkurve ändern. Zur gleichen Zeit, zu vermeiden, das Vorhandensein von Löchern oder unvollständige Masken einem Kern die gleiche Kurve verwendet wird.
- Verwenden Sie die Filter-Tool jedes Geräusch in der Oberflächendarstellung auszuschließen. Unter der Registerkarte Bearbeiten der neu geschaffenen Oberflächenschicht, Split oder die Kerne Oberflächen zusammenführen, die falsch sind neundered.
- Export in eine Tabelle, die Elliptizität und Rundheitswerte der Oberfläche gemacht Kerne-Datei, die unter der Registerkarte Statistik zur Verfügung stehen.
- Alternativ verwenden ImageJ oder Fiji Software, um die Kreisförmigkeit der Kerne zu messen, wo die Kreisförmigkeit (C) als C definiert ist,
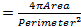 . Ein Wert von eins entspricht einen perfekten Kreis, während ein Wert gegen Null eine zunehmend längliche Form anzeigt.
. Ein Wert von eins entspricht einen perfekten Kreis, während ein Wert gegen Null eine zunehmend längliche Form anzeigt. - Erstellen Sie maximale Intensität Projektionen der z-Stacks aus dem Menü Bilder> Stacks> Z-Projekt. Stellen Sie Projektionstyp Max Intensität.
- Teilen Sie die Kanäle und wählen Sie den Kernmarker-Kanal.
- Aus dem Hauptmenü wählen Sie Bild> Anpassen> Schwellenwert.
- Segment die Kerne durch die Intensitätsschwelle eingestellt wird. Wenn die in der Nähe Kerne als eine einzige Einheit segmentiert sind, klicken Sie auf Prozess> Binary> Watershed Werkzeug, um die Kerne zu trennen.
- Aus dem Hauptmenü wählen Sie Bearbeiten> SWahl> erstellen Auswahl.
- Fügen Sie alle ausgewählten ROIs auf die ROI-Manager durch Analysieren auf Menü klicken> Tools> ROI-Manager. Im ROI-Manager-Fenster klicken Sie auf Hinzufügen. Im gleichen Fenster wählen Sie Mehr> Split.
- Wählen Sie Formendeskriptoren in Analysieren> Set Messungen. Im ROI-Manager-Fenster auf die Registerkarte klicken Messen. Dadurch wird eine Liste von Kreisförmigkeitswerte aller ausgewählten Kerne anzuzeigen.
- Darüber hinaus das Kernvolumen zu messen, Oberflächen Assistent für die Erstellung mit Hilfe der Kernmarker Färbung Kanal folgen, wie 7,3-7,7 in den Unterabschnitten beschrieben.
8. 3D Volume Renderings von ausgewählten Kerne innerhalb der Drosophila Larven Körperwand Muskeln die Intranukleäre Lokalisierung eines spezifischen Proteins zur Bewertung
- Verwenden Sie Konfokalbilder Körperwand Muskeln mit einem Kernmarker und mit Antikörpern, die spezifisch an Lamin und DVAP gefärbt berichten.
- Öffnen Sie die Bilder durch die Software zu initiieren und select die spezifische Zellkern untersucht werden. Dies kann durch die Auswahl des Hauptmenüpunkt Bearbeiten> Zuschneiden 3D erfolgen.
- Folgen Sie Oberfläche Assistent für die Erstellung mit Lamin-Kanal als 7,3-7,7 im Unterabschnitt beschrieben.
- Sobald die Oberfläche erstellt wird, klicken Sie auf Bearbeiten im Objekteigenschaftenbereich und dann auf Alle wählen Maske das Signal innerhalb des Kerns zu isolieren. Dadurch entsteht ein neues Fenster.
- Wählen Sie DVAP Signal aus dem Kanal wählen im Dropdown-Menü. Wählen Sie das DVAP Immunreaktivität Signal im Kern durch Klicken auf die Option Set Voxel außerhalb der Fläche auf Null. Eine neue maskierte Kanal erstellt und ist in der Anzeige Einstellungsfenster zur Auswahl.
- Zur Visualisierung erstellen, das Vorhandensein des Signals innerhalb des Kerns eine Konturebene durch Klicken auf das Symbol New Clippingebene
 aus der Symbolleiste Objekte.
aus der Symbolleiste Objekte. - Interaktives stellen Sie den Winkel des ClippingFlugzeug und seine Position um die Verteilung des Signals innerhalb des Kerns zu visualisieren.
Ergebnisse
ALS ist eine degenerative Erkrankung spezifisch motorischen Neuronen beeinflussen 7 auf eine progressive und tödliche Lähmung der quergestreiften Muskulatur führt. Missense - Mutationen im menschlichen VAMP-Associated Protein B (hVAPB) verursachen eine Reihe von Motoneuron - Erkrankungen einschließlich ALS Typ 8-12 August. A Missense - Mutation (V234I) im hVAPB Gen wurde in einem Fall von typischen ALS beim Menschen 13 kürzlich identifiziert. Um sein pathogenes Potential beurteilen zu können , haben wir einen transgenen Fliegen , die hVAPB Drosophila Ortholog DVAP Expression der krankheitsverursachenden Mutation tragen (DVAP-V260I). Mit der UAS / GAL4 - System und den muskelspezifischen Treiber BG57-Gal4 14,15 Die Expression dieses Transgen in die Muskeln gezielt. Die Wirkung von DVAP-V260I transgenen Expression wurde verglichen und die der beiden anderen Transgenen (DVAP-WT1 und DVAP-WT2) gegenübergestellt , die 16 - Protein verschiedenen Ebenen des Wildtyp DVAP auszudrücken. More gesagt, ist die Zunahme der DVAP Immunreaktivität höherer 2,2fach als bei den Kontrollen für die DVAP-WT2 Linie während DVAP-V260I und DVAP-WT1 zeigen vergleichbare und unteren Ebenen des gleichen Signals 16.
Nuclear Veränderungen haben mit dem Altern und neurodegenerativen Erkrankungen , einschließlich der Parkinson-Krankheit 17,18 in Verbindung gebracht worden. Um zu beurteilen , ob unsere Fliegenmodell für ALS8 zeigt Änderungen in der Kern Architektur, Lage und Größe, gefärbt wir Kerne innerhalb quergestreiften Muskulatur geeigneter Genotypen mit einem nuklearen Marker und dem anti-Lamin Antikörper 19-22, die die Kernhülle visualisiert. Um die Muskeln zu markieren, ein DVAP spezifischen Antikörper wurde auch mit den gleichen Proben (Figur 1) gegeben. Konfokale Bilder wurden gesammelt und detaillierte morphometrischen Analysen wurden unter Verwendung einer Bildanalysesoftware durchgeführt. In Kontroll Muskeln wurden Kerne gefunden entlang der Muskulatur gleichmäßig verteilt werdenFasern , während in DVAP-V260I und DVAP-WT Muskeln ausdrückt, Kerne eine Tendenz aufweisen , eng verbunden Cluster (Abbildung 1) zu verteilen , in.
Wir führten eine Nächste-Nachbar-Analyse eine quantitative Auswertung der Verteilung der Kerne entlang der Muskelfasern jedes Genotyps durchzuführen. Eine nächste Nachbar-Analyse identifiziert zuerst den nächsten Nachbarn für jeden Kern, der durch den Abstand zwischen der Mitte eines gegebenen Kern und Mittelpunkt jeder anderen umgebenden Kern zu messen. Dieses Verfahren wird für alle anderen Kerne entlang der Muskelfasern dann wiederholt. Schließlich wird der kürzeste Abstand zwischen den Kernen innerhalb eines bestimmten Muskels, berechnet durch die kürzesten Abstände von jedem Kern und seinen nächsten Nachbarn gemittelt. (2A - C). Im Vergleich zu den Kontrollen, die Muskeln, die entweder die DVAP-V260I Transgen oder einen der Transgene, die das Wildtyp-Protein überexprimierenpräsentieren, eine drastische Reduzierung der durchschnittlichen kürzesten Abstand zwischen den Kernen und, als Folge, Kerne scheinen eng in Clustern zugeordnet werden. Die Wirkung des ALS - Allel verursacht DVAP-V260I ist schwerer als die mit der Überexpression des Wildtyp - Proteins verbunden ist , selbst wenn die stärksten DVAP-WT2 Transgen verwendet wird (Figur 1 und Figur 2D).
Überexpression von entweder DVAP-V260I oder DVAP-WT Transgenen zeigt auch eine erhebliche Verschlechterung des Kernarchitektur wodurch verformte Kerne mit einer länglichen Struktur (Abbildung 1). Diese strukturelle Aberration wurde unter Verwendung der ImageJ Software quantifiziert, in dem Zirkularität durch die Formel C definiert ist,  , Was die Breite zu Länge-Verhältnis von jedem Kern mit C = 1 repräsentiert einen perfekten Kreis und C = 0 ein unendlich verlängerte Polygon messen. in control Kerne eine deutliche runde Form aufweisen, ist C gleich 1 , während in den transgenen Mutanten eine Änderung der Form mit damit verbundenen Verlust der Zirkularität, eine signifikante Abweichung von diesem Wert (Abbildung 1 und Abbildung 3) verursacht.
, Was die Breite zu Länge-Verhältnis von jedem Kern mit C = 1 repräsentiert einen perfekten Kreis und C = 0 ein unendlich verlängerte Polygon messen. in control Kerne eine deutliche runde Form aufweisen, ist C gleich 1 , während in den transgenen Mutanten eine Änderung der Form mit damit verbundenen Verlust der Zirkularität, eine signifikante Abweichung von diesem Wert (Abbildung 1 und Abbildung 3) verursacht.
Wir fanden auch in Muskeln, die die gleichen Transgene exprimieren, Kerne eine deutliche vergrößerte Kernvolumen angezeigt werden Kontrollen im Vergleich zu, obwohl die ALS verursachen Allel effizienter in diesen Phänotyp zu induzieren , verglichen mit den DVAP-WT Transgene (Abbildung 4) zu sein scheint.
Fast alle neurodegenerativen Erkrankungen werden durch die intrazelluläre Akkumulation von Aggregaten gekennzeichnet das pathogene Protein enthält. Wir haben 3D-Rekonstruktionen und Volumen-Renderings von Kernen und wir fanden heraus, dass in den Muskeln des mutierten Transgen exprimieren, oder das Wildtyp-Protein überexprimieren, DVAP Immunreaktivität Cluster bildeten einend dass einige von ihnen auch in die Kerne lokalisiert wurden (Abbildung 5). Umgekehrt wird in Steuer NMJs wird DVAP Immunreaktivität schwach ganzen Muskelfaser dispergiert und aus dem Kern 16 ausgeschlossen.
. Abbildung 1: Die konfokale Bilder von myonuclei in gestreifter Muskeln entweder die DVAP-WT oder die DVAP-260i Transgene (A) BG57-Gal4 / + Steuerung, (B) BG57 zum Ausdruck bringen; DVAP-V260I, (C) BG57; DVAP-WT1 und (D) BG57; DVAP-WT2 Muskeln die angegebenen Transgene exprimieren , werden mit Antikörpern , die spezifisch für DVAP (rotes Signal) gefärbt, Lamin (grünes Signal) und mit einem Kern spezifischer Marker , die Kerne (blau - Signal) zu visualisieren. Maßstabsbalken = 30 & mgr; m Bitte hier klicken , um anzuzeigenGrößere Version der Figur.
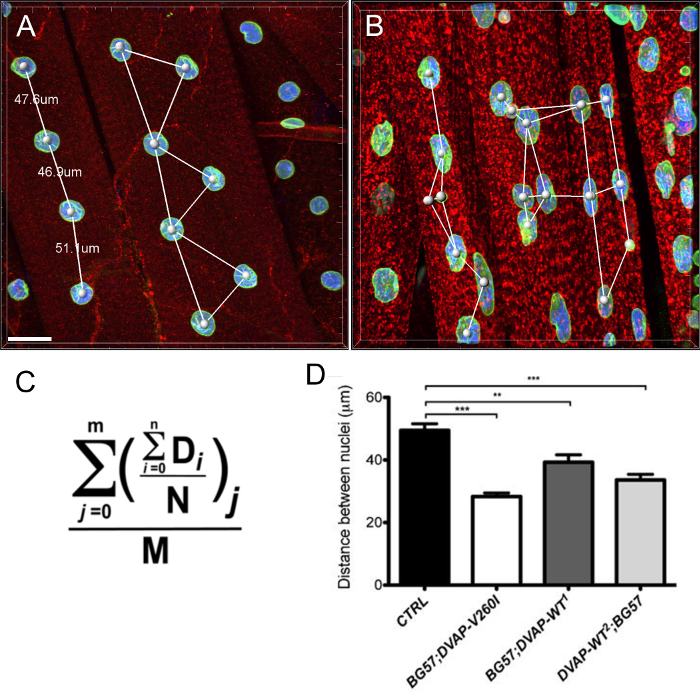
Abbildung 2:. Nächster - Nachbar - Analyse der durchschnittliche Abstand zwischen einem Kern und seinem einzigen nächsten Nachbarn (B) Repräsentative Ergebnisse zeigen veränderte Kern Positionierung in Muskeln Überexpression des DVAP-WT2 Transgen , um zu bestimmen , im Vergleich zu den Kontrollen in (A). Durchschnittliche Atomabstand in Muskeln der angegebenen Genotypen wurde unter Verwendung der Formel in (C) geschätzt , und die Daten werden in (D) angegeben. Larval NMJs sind mit Antikörpern, die spezifisch für DVAP (rotes Signal), Lamin (grün) und mit einem Kernmarker (blaues Signal) gefärbt. Sternchen bezeichnen statistische Signifikanz. *** P <0,001, ** P <0,01. Für die statistische Analyse dieses Experiment und alle berichteten Experimente unter einen Einweg-ANOVA-Test wurde verwendet, und ein Tukey-multiple-Vergleichstest wurde als Post-hoc-Test angewendet, wenn die Unterschiede zwischen den Genotypen durch den ANOVA-Test als signifikant befunden wurden. Die Fehlerbalken stellen SEM. Maßstabsbalken = 30 & mgr; m Bitte klicken Sie hier um eine größere Version dieser Figur zu sehen.
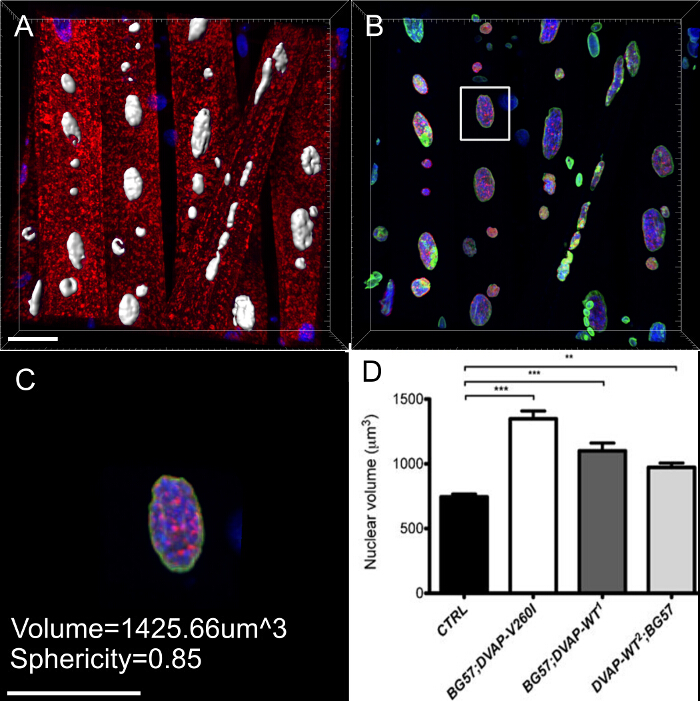
Abbildung 3:. Bilder zeigen repräsentative Schritte bei der Berechnung des Kernvolumens (A) Ein repräsentatives Bild zeigt segmentierte Kerne der Oberfläche Erstellungsassistenten. Nuclei an der Grenze der Bilder wurden ignoriert. (B) Bild der Kern DVAP Signal nach der umgebenden DVAP Färbung zeigt wurde durch die Verwendung der Oberfläche im Kernmarkierungskanal geschaffen maskiert. (C) Oberflächenschicht enthält Informationen von zusätzlichen Parameterneinschließlich des Kernvolumens und der Rundheitsgrad. (D) Daten über die Kernvolumen verschiedener Genotypen. Sternchen bezeichnen statistische Signifikanz. Dissected NMJs wurden mit anti-DVAP Antikörper (rotes Signal), Anti-Lamin Antikörper (grünes Signal) und einem Kernmarker (blaues Signal) gefärbt. *** P <0,001, ** P <0,01. Die Fehlerbalken stellen SEM. Maßstabsbalken = 30 & mgr; m Bitte klicken Sie hier um eine größere Version dieser Figur zu sehen.
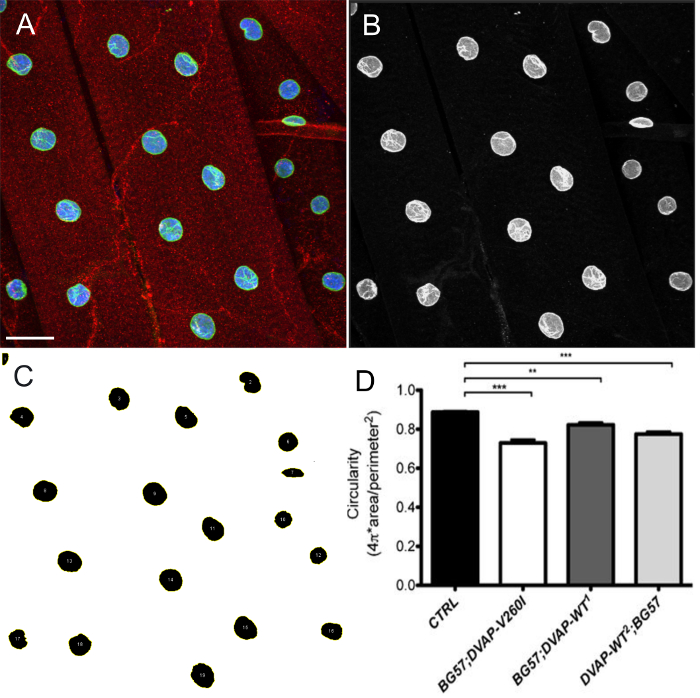
Abbildung 4: Bilder zeigen repräsentative Schritte bei der Schätzung der Kernform von ImageJ. Maximale Intensität Projektion von Bildern wurden mit ImageJ analysiert Zirkularität der Kerne in den Muskeln zu schätzen. (A) Ein repräsentatives Beispiel des Intensitätsprojektionsdes Dreikanalbild, wie in Schritt 7.9 des Protokolls. (B) Bild zeigt Schritt 7.10 des Protokolls , in dem Kanäle aufgeteilt sind und die Kernmarker - Kanal ausgewählt ist. (C) Ein repräsentatives Bild zeigt , dass Intensitätsschwelle zu segmentieren nach dem Auftragen der Kerne und ROI - Manager - Plugin in ImageJ, alle Kerne von Interesse ausgewählt und deren Form gemessen durch Form - Deskriptoren werden kann (Schritte 7,11-7,15). (D) Die Quantifizierung der Zirkularität von verschiedenen Genotypen. Auf den Larven NMJs zeigt das rote Signal DVAP Färbung, während die grüne Kerne umreißt und entspricht der Lamin-Färbung. Das Innere jeder Kern mit einem Kernmarker aufgrund der Färbung in blau markiert. Sternchen bezeichnen statistische Signifikanz. *** P <0,001, ** P <0,01. Die Fehlerbalken stellen SEM. Maßstabsbalken = 30 & mgr; m Bitte klicken siee eine größere Version dieser Figur zu sehen.
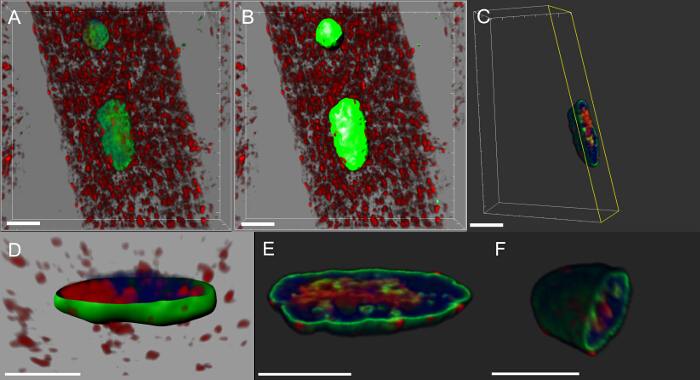
Abbildung 5: Bilder konkrete Schritte bei der Erstellung von Volumen - Renderings von myonuclei zeigt. (A) Bild 3D - Intensität zeigt , gemischt Ansicht von Muskeln gefärbt mit DVAP Protein (rot), die Lamin (grün) und die DNA - Marker (blau). (B) Bild , das eine Oberflächenschicht unter Verwendung des Lamin - Kanal - Segment die Kerne erzeugt darstellt. (C) Bild repräsentiert einen Kern , in dem die Oberflächenschicht verwendet wurde , außerhalb des ausgewählten Kerns die DVAP Signal zu maskieren. Hervorgehoben in gelb ist eine Clipping-Ebene, die dem Bild hinzugefügt wurde. Dessen Blickwinkel und die Position kann interaktiv die Verteilung des Signals innerhalb des Kerns zu visualisieren eingestellt werden. (D) Ein Bild einer Querschnittsansicht der Schicht der Kernoberfläche erzeugt usi Berichterstattungng der Lamin Kanal fusionierte mit unmaskierten DVAP und Kernmarkierungssignale. (E und F) weiteren Schnittvolumen Renderings des gleichen Kerns. Maßstabsbalken = 10 & mgr; m Bitte klicken Sie hier um eine größere Version dieser Figur zu sehen.
Diskussion
In der Vergangenheit morphologischen Variationen innerhalb und zwischen den Versuchsgruppen wurden nur selten berücksichtigt. Jedoch ist die Anwendung quantitativer Verfahren jetzt immer die Norm in vergleichenden Untersuchungen der Morphologie und mathematische Beschreibung der anatomischen Formen berechnet. Die Verwendung von quantitativen Analysen in die Auswirkungen von genetischen Manipulationen auf spezifische zelluläre Prozesse der Beurteilung halten Versprechen bei der Verbesserung unserer Fähigkeit morphologischen Veränderungen zu erfassen und die Genauigkeit zu verbessern, mit der diese Veränderungen beschrieben sind. Darüber hinaus ermöglicht die statistische Analyse von quantitativen Daten uns ob die beobachteten Unterschiede zwischen den Phänotypen signifikant zu bewerten sind.
In quergestreiften Muskulatur weisen Kerne eine deutliche gerundete Struktur und sind entlang der Muskelfasern gleichmäßig verteilt. Obwohl die molekularen Mechanismen, Einführung und Aufrechterhaltung von Größe, Form und Architektur der Kerne nicht bekannt sind, sind diese Kernfunktionen wahrscheinlich to spielen eine fundamentale Rolle Muskelfunktion bei der Kontrolle. Tatsächlich sind mehrere Myopathien durch Mutationen in Genen verursacht Morphologie und Position der Kerne in den Muskeln zu regulieren. Die funktionale Bedeutung der Form und Verteilung von Kernen innerhalb einer Zelle auf Muskeln beschränkt. Akkumulieren Beweise zeigen , dass die Kern Defekte werden auch mit neurodegenerativen Erkrankungen wie Morbus Parkinson 18,24 verbunden. Darüber hinaus beginnen wir, dass die Morphologie, Größe und intrazelluläre Verteilung von anderen Organellen einschließlich endoplasmatischen Retikulum und Mitochondrien zu schätzen funktionelle Auswirkungen haben. Zum Beispiel 25 Veränderungen in der mitochondrialen Morphologie mit neurologischen Erkrankungen wie Optikusatrophie Typ-1 (OPA1) und Charcot-Marie-Tooth - Typ 2A Neuropathie in Verbindung gebracht.
Zur Unterstützung im Prozess der molekularen Mechanismen der Aufklärung diese wichtigen Prozesse zugrunde liegen, schlagen wir eine hohe Auflösung der konfokalen zu kombinierenDaten mit Bildbearbeitungssoftware und morphometrische Analysen quantitativ zu bewerten, wie genetische Manipulationen Form beeinflussen können, Größe und Lage der Kerne innerhalb Muskelfasern. Die Leistungsfähigkeit und Vielseitigkeit der Drosophila Genetik zusammen mit dem sehr klischee Natur des neuromuskulären Systems in Drosophila - Larven machen die Larven NMJ ein experimentelles Modell besonders geeignet für diese Art von Analysen. An den Larven NMJs können phänotypische Analyse bei einer einzelnen Synapse Auflösung durchgeführt werden , um eine genaue morphometrische Analyse ermöglicht , wo eine Anzahl von NMJs kann innerhalb derselben Fliege und sogar die gleiche identifizierbare NMJ zwischen Fliegen verschiedener Genotypen werden 3,4 im Vergleich untersucht werden.
Phänotypischen Charakterisierung von Kern Position, Form und Größe an der Drosophila Larven NMJ beginnt durch Immunfärbung von seziert NMJs mit Antikörpern durchführen , die Muskeln und Kerne in den Muskeln hervorheben. Im Protokoll thi skizziertes Papier, myonuclei wurden mit polyklonalen Antikörpern gegen lamin, einem Marker der Kernhülle gebeizt, mit einem nuklearen Marker Hervorhebung der Kerninnen und mit Antikörpern, die spezifisch an DVAP die gesamte Muskel zu färben. Die lamin Antikörper in diesen Experimenten verwendet wurden von Paul Fisher freundlicherweise 19-22 aber alternative Quellen von anti-lamin Antikörper können verwendet werden. Darüber hinaus sind eine Reihe anderer Antikörper, die spezifisch für die Kernhülle im Handel erhältlich. Schließlich sind Kern Marker, wie DAPI und Propidiumiodid, auch während Muskeln durch Färbung mit anti-Aktin oder anti-Tubulin-Antikörpern sichtbar gemacht werden konnten. Wenn andere Antikörper als die in diesem Versuchsverfahren verwendet, eingesetzt werden, wird die Immunfärbungsprotokoll erfordern Extraschritte, bei denen Fixierungsbedingungen und Arbeitskonzentrationen für die neuen Antikörper müssen optimiert werden. Ein wichtiger Schritt in diesem Protokoll, vor allem, wenn Volumen Renderings analysiert werden müssen, ist the Montage der Proben auf der Folie. In diesem Fall ist es wichtig, Abstandshalter zwischen dem Objektträger und dem Deckglas aufzunehmen, so dass die Proben werden nicht zerquetscht. Drei Bänder aus Celluloseband um die Folie auf beiden Seiten des Deckglases gewickelt stellen eine einfache Möglichkeit, Abstandhalter zu machen.
Während ImageJ für 2D-Bilder verwendet wurde, analysiert die meisten 3D-Multi-Channel-Bild in diesem Papier, wurden unter Verwendung von Imaris getan wegen seiner im Haus vorhanden sind. Jedoch kann jede andere ähnliche kommerzielle Software-Paket kann für diese Anwendungen verwendet werden.
Es gibt mehrere Open-Source (zB ImageJ, CellProfiler, Vaa3D, Icy, KNIME und andere) und kommerzielle Software-Plattformen zur Verfügung für die Analyse von konfokalen Bildern. ImageJ 26, die freie Software von der NIH oder seine erweiterte Version, wie FiJi bekannt 27, hat eine große Anzahl von Import - Filter, Makros und Plugins für die weltweite Kommunika - Bildgebungty. Die meisten dieser Plugins sind auf die Verarbeitung der Informationen auf einer Scheibe-für-Scheibe Weise fokussiert. Es gibt auch Plugins für die Visualisierung und Analyse von mehrkanaligen 3D-Bildern. Allerdings sind sie oft für eine bestimmte Aufgabe entwickelt und Benutzer müssen diese Plugins, um ihre eigenen Bedürfnisse zu erweitern oder anpassen. Auf der anderen Seite, Ziel kommerziellen Plattformen relativ unerfahrene Anwender und werden oft konzentrierte sich auf einfache zu bedienende, breite Abdeckung von Bildverarbeitungsaufgaben mit einer unglaublichen Geschwindigkeit.
Das experimentelle Verfahren zusammen mit der quantitativen in diesem Protokoll beschriebenen phänotypische Analyse kann helfen, die molekularen Mechanismen bei der Aufklärung Organell Morphologie zu steuern und deren Verteilung innerhalb einer Zelle. Jedoch hat dieser Ansatz die offensichtliche Einschränkung dieser Prozesse zu einem bestimmten Endpunkt-Analyse. Das Verfahren Morphologie und Verteilung von Organellen des Steuerns ist wahrscheinlich sehr dynamisch sein und variieren nicht nur zwischen verschiedenen Zell types, sondern auch innerhalb der gleichen Zelle auf der Entwicklungs-oder physiologischen Zustand abhängig. Eine weitere Anwendung dieser Analyse würde durch Zeitrafferabbildungs dargestellt werden, die Änderungen in Organell Morphologie und Position ermöglicht über die Zeit überwacht werden.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
We are grateful to Dr. Andrea Chai for her insightful comments on the manuscript. This work was supported by the Wellcome Trust (grant number: Pennetta8920) and by the Motor Neuron Disease Association (grant number: Pennetta6231).
Materialien
| Name | Company | Catalog Number | Comments |
| Micro-Forceps 0.3 mm x 0.25 mm | Fine Science Tools | 11030-12 | |
| Sylgard dissection plates | SIGMA-ALDRICH | 76103 | Mix the pre-weighed elastomer base with curing agent. Poor the mixture into a 5 cm Petri dish. Let cure it at 60 °C for at least 24 hr |
| Stainless/Steel Minutien Pins 0.1 mm diameter | Fine Science Tools | 26002-10 | |
| Microdissection scissors (ultra-fine) | Fine Science Tools | 15200-00 | |
| 1x PBS (Phosphate Buffered Saline). Composition: 3 mM NaH2PO4, 7 mM Na2HPO4, 130 mM NaCl, pH 7 | NaH2PO4 (SIGMA-S8282), Na2HPO4 (SIGMA-S7907), NaCl (SIGMA-S7653) | ||
| Bouin's Solution. Composition: Picric Acid, Formaldehyde, Acetic Acid (15:10:1) | Picric Acid (SIGMA 197378), Formaldehyde (F8775), Acetic Acid (SIGMA-1005706) | ||
| 1x PBT (Phosphate Buffered Saline with Triton). 1x PBS + 0.1% Triton | Triton-X100. SIGMA-T8787 | ||
| Normal Goat Serum | SIGMA | G9023 | |
| Guinea Pig anti-DVAP antibody | Provided by Dr. Giuseppa Pennetta (University of Edinburgh, UK). Use at 1:200 dilution in 5% NGS | ||
| Rabbit anti-HRP (Horseradish peroxidase) | Jackson ImmunoResearch | 123-065-021 | Use at 1:500 dilution in PBT containing 5% NGS |
| Rabbit anti-Lamin antibody | Provided by Dr. Paul Fisher (State Univeristy of New York at Stony Brook). Use at 1:500 dilution in PBT containing 5% NGS | ||
| TO-PRO-3 | Molecular Probes | T3605 | |
| Goat anti-rabbit antibody, Alexa Fluor488, conjugated | Jackson ImmunoResearch | bs-295G-A555-BSS | Use at 1:500 dilution in PBT containing 5% NGS |
| Goat anti-guinea pig IgG antibody, Cy3, conjugated | Jackson ImmunoResearch | bs-0358G-Cy3-BSS | Use at 1:500 dilution in PBT containing 5% NGS |
| Vectashield mounting medium for fluorescence | Vector laboratories | H-1000 |
Referenzen
- Collins, C. A., DiAntonio, A. Synaptic development: insights from Drosophila. Curr. Opin. Neurobiol. 17 (1), 35-42 (2007).
- Fortini, M. E., Skupski, M. P., Boguski, M. S., Hariharan, I. K. A survey of human disease gene counterparts in the Drosophila genome. J. Cell Biol. 150 (2), 23-30 (2000).
- Ramachandran, P., Budnik, V. Dissection of Drosophila larval body-wall muscles. Cold Spring Harb Protoc. (8), (2010).
- Brent, J. R., Werner, K. M., McCabe, B. D. Drosophila larval NMJ dissection. JoVE. (24), (2009).
- Brent, J., Werner, K., McCabe, B. D. Drosophila larval NMJ immunohistochemistry. JoVE. (25), (2009).
- Ramachandran, P., Budnik, V. Immunocytochemical staining of Drosophila larval body-wall muscles. Cold Spring Harb Protoc. (8), (2010).
- Ling, S. C., Polymenidou, M., Cleveland, D. W. Converging mechanisms in ALS and FTD: disrupted RNA and protein homeostasis. Neuron. 79 (3), 416-438 (2013).
- Nishimura, A. L., et al. A mutation in the vesicle-trafficking protein VAPB causes late-onset spinal muscular atrophy and amyotrophic lateral sclerosis. Am J Hum Genet. 75 (5), 822-831 (2004).
- Chen, H. J., et al. Characterization of the properties of a novel mutation in VAPB in familial amyotrophic lateral sclerosis. J Biol Chem. 285 (51), 40266-40281 (2010).
- Funke, A. D., et al. The P56S mutation in the VAPB gene is not due to a single founder: the first European case. Clin Genet. 77 (3), 302-303 (2010).
- Millecamps, S., et al. SOD1,ANG,VAPB,TARDBP, and FUS mutations in familial amyotrophic lateral sclerosis: genotype-phenotype correlations. J Med. Genet. 47 (8), 554-560 (2010).
- Landers, J. E., et al. New VAPB deletion variant and exclusion of VAPB mutations in familial ALS. Neurology. 70 (14), 1179-1185 (2008).
- Van Blitterswijk, M., et al. VAPB and C9orf72 mutations in 1 familial amyotrophic lateral sclerosis patient. Neurobiol Aging. 33 (12), 2951-2954 (2012).
- Brand, A. H., Perrimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118 (2), 401-415 (1993).
- Budnik, V., et al. Regulation of synapse structure and function by the Drosophila tumor suppressor gene dlg. Neuron. 17 (4), 627-640 (1996).
- Sanhueza, M., Zechini, L., Gillespie, T., Pennetta, G. Gain-of-function mutations in the ALS8 causative gene VAPB have detrimental effects on neurons and muscles. Biol Open. 3 (1), 59-71 (2014).
- Worman, H. J., Ostlund, C., Wang, Y. Diseases of the nuclear envelope. Cold Spring Harb Perspect Biol. 2 (2), a000760 (2010).
- Liu, G. H., et al. Progressive degeneration of human neural stem cells caused by pathogenic LRRK2. Nature. 491 (7425), 603-607 (2012).
- Pennetta, G., Hiesinger, P. R., Fabian-Fine, R., Meinertzhagen, I. A., Bellen, H. J. Drosophila VAP-33A directs bouton formation at neuromuscular junctions in a dosage-dependent manner. Neuron. 35 (2), 291-306 (2002).
- Fisher, P. A., Berrios, M., Blobel, G. Isolation and characterization of a proteinaceous subnuclear fraction composed of nuclear matrix, peripheral lamina, and nuclear pore complexes from embryos of Drosophila melanogaster. J Cell Biol. 92 (3), 674-686 (1982).
- Smith, D. E., Fisher, P. A. Identification, developmental regulation, and response to heat shock of two antigenically related forms of a major nuclear envelope protein in Drosophila embryos: application of an improved method for affinity purification of antibodies using polypeptides immobilized on nitrocellulose blots. J Cell Biol. 99 (1), 20-28 (1984).
- Smith, D. E., Gruenbaum, Y., Berrios, M., Fisher, P. A. Biosynthesis and interconversion of Drosophila nuclear lamin isoforms during normal growth and in response to heat shock. J Cell Biol. 105 (2), 771-790 (1987).
- Puckelwartz, M. J., et al. Disruption of nesprin-1 produces an Emery Dreifuss muscular dystrophy-like phenotype in mice. Hum Mol Genet. 18 (4), 607-620 (2009).
- Tsujikawa, M., Omori, Y., Biyanwila, J., Malicki, J. Mechanism of positioning the cell nucleus in vertebrate photoreceptors. Proc Natl Acad Sci USA. 104 (37), 14819-14824 (2007).
- Westermann, B. Mitochondrial fusion and fission in cell life and death. Nat Rev Mol Cell Biol. 11 (12), 872-884 (2010).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten