Method Article
Einzelmolekül-Super-Resolution Imaging von Phosphatidylinositol 4,5-bisphosphat in der Plasmamembran mit neuartigen Fluoreszenzsonden
In diesem Artikel
Zusammenfassung
PI (4,5) P 2 regelt verschiedene Zellfunktionen, aber die nanoskaligen Organisation in der Zellplasmamembran ist wenig bekannt. Durch Markierung PI (4,5) P 2 mit einer zweifarbige Fluoreszenzsonde zur Domäne Pleckstrin Homology fusioniert beschreiben wir einen neuen Ansatz der PI (4,5) P 2 in der räumlichen Verteilung der Plasmamembran im Nanometerbereich zu studieren .
Zusammenfassung
Phosphoinositides in the cell membrane are signaling lipids with multiple cellular functions. Phosphatidylinositol 4,5-bisphosphate (PI(4,5)P2) is a determinant phosphoinositide of the plasma membrane (PM), and it is required to modulate ion channels, actin dynamics, exocytosis, endocytosis, intracellular signaling, and many other cellular processes. However, the spatial organization of PI(4,5)P2 in the PM is controversial, and its nanoscale distribution is poorly understood due to the technical limitations of research approaches. Here by utilizing single molecule localization microscopy and the Pleckstrin Homology (PH) domain based dual color fluorescent probes, we describe a novel method to visualize the nanoscale distribution of PI(4,5)P2 in the PM in fixed membrane sheets as well as live cells.
Einleitung
Phosphoinositiden tragen zu einem geringen Teil der gesamten Membranlipiden, sondern in einer Vielzahl von zellulären Prozessen eine entscheidende Rolle spielen. Sie umfassen sieben Mitglieder abgeleitet von reversible Phosphorylierung oder Dephosphorylierung des inositol Ringe auf der 3., 4. und 5. Positionen 1. Phosphatidylinositol - 4-phosphat (PI4P) und Phosphatidylinositol - 4,5-bisphosphat (PI (4,5) P 2) sind zwei wichtige Phosphoinositiden , die relativ unabhängig wie die Determinante Lipide der Zellplasmamembran (PM) 2,3 funktionieren. Phosphatidylinositol (3,4,5) -trisphosphate (PIP 3) viel weniger reichlich als PI4P und PI (4,5) P 2, aber es hat einzigartige Funktionen in verschiedenen zellulären Prozessen , einschließlich Krebs und Diabetes 4 5. Diese Lipide haben komplexe molekulare Interaktionen mit ihren Effektoren und viele andere Proteine. Daher ist es entscheidend, die räumliche Organisation dieser pho zu verstehensphoinositides im PM auf der Nanometer-Skala.
Montage Beweise hat gezeigt , dass Proteinkomplexe oder Molekülcluster in den engen PM Bereichen als Signal Hotspots 6 dienen kann. Zum Beispiel Syntaxin 1a, ein Schlüsselprotein , das die Membranfusion 7-9, zeigt Cluster - Organisation in der PM reguliert. Unterscheidet sich von der Konsensusansicht der Clusterorganisation von Syntaxin 1a, die räumliche Verteilung von Phosphoinositiden in der PM ist umstritten. PI (4,5) P 2 Verteilungsmuster reichen von Uniform 10-12, große Flecken 13,14, zu dichten Büscheln 14-18, je nach Zelltypen und experimentelle Methoden verwendet. Die räumliche Organisation von PI (4,5) P 2 mit höherer Auflösung ist auch widersprüchlich. Eine Studie unter Verwendung von stimulierten Emission-Depletion (STED) Mikroskopie 19 hat eine große Anzahl von dicht PI (4,5) P 2 Nanocluster (~ 73 nm Durchmesser) in den PM Blätter PC-12 - Zellen 20 ergab . Dieses Ergebnis unterscheidet sich von Studien unter Verwendung von Schnellfroster Elektronenmikroskopie (EM) 21,22, ein Ansatz, der die intakte PM Struktur von lebenden Zellen viel besser als chemische Fixierung bewahrt. Diese zeigten einen deutlichen Pools von PI (4,5) P 2; relativ konzentrierten PI (4,5) P 2 in Caveolae und beschichtet sowie eine gleichmäßige Verteilung auf der flachen Region PM Pits. Darüber hinaus nanoskaligen PI (4,5) P 2 Organisation in Membranfolien können in lebenden und fixierten Zellen unterscheiden. Unsere jüngsten Arbeiten untersucht dieses Problem in stationären und leben INS-1 - Zellen Einzelmolekül - Lokalisationsmikroskopie (SMLM) 23 verwendet wird .
SMLM basiert auf stochastisch Einschalten nur eine kleine Teilmenge von Fluorophoren zu einem bestimmten Zeitpunkt, so dass einzelne Fluorophore mit hoher Genauigkeit lokalisiert werden. Viele Super-Resolution Imaging-Ansätze wurden unter Verwendung ähnlicher Prinzipien entwickelt, um die Beugungsgrenze der konventionellen Lichtmikroskopie zu überwinden, wie eins Photoaktivierungslokalisationsmikroskopie (PALM) 24, Fluoreszenzphotoaktivierungslokalisationsmikroskopie (FPALM) 25, stochastische optische Rekonstruktionsmikroskopie (STORM) 26,27 und direkte STORM (dSTORM) 28. Mit photoschaltbare oder photoaktivierbaren Fluorophore (Farbstoffe oder fluoreszierende Proteine), erlauben SMLM Techniken Wissenschaftler Bild biologischen Strukturen in Nanometer - Auflösung 24,29,30 mit Video-Rate in lebenden Zellen 31,32.
Mit PI (4,5) P 2 als Beispiel, haben wir das SMLM Ansatz die nanoskalige Verteilung von Phosphoinositid auf dem PM zu studieren. Die PH - Domäne von PLCδ1 (Phospholipase C δ1) , die PI bindet spezifisch an (4,5) P 2 eine gut etablierte Sonde zur Bildgebung PI (4,5) P 2 subzelluläre Verteilung und Dynamik 33,34 in der PM . Wir haben genetisch diese Domäne mit zwei fluoreszierenden Proteinen markiert, PAmCherry1 35 und 36 iRFP , um eine Zweifarben - Fusionsprotein (iRFP-PAmCherry1-PH PLCδ1) (1A-B). PAmCherry1 dient als photoaktivierbare Sonde von PH - Domäne für PALM und iRFP dient als allgemeiner Indikator zu transfizierten Zellen zu identifizieren , bevor PALM Erwerb . Wir wenden diese zweifarbige Fluoreszenzsonde für SMLM Bildgebung in den festen Membranfolien. Für Live - PALM - Bildgebung, getaggt wir mEOS3 37 anstelle von PAmCherry1 an die PH PLCδ1 Domäne der mEOS3.1-PH PLCδ1 Sonde für seine bessere Photoneneffizienz und Helligkeit (1C-D) zu erzeugen.
SMLM Bildgebung mit dieser neuartigen Sonden in der PM von insulinsezernierenden INS-1 - Zellen 38 hat homogene Markierung von PI (4,5) P 2 in der Mehrzahl der PM Regionen sowie konzentrierte PI (4,5) P 2 freigelegt Mikrodomänen , die in der Wohnung PM und einige Filopodien artige Strukturen 23 dünn miteinander vermischt werden. Dann einnoScale Verteilung von PI (4,5) P 2 eine strukturelle Basis für ein Umdenken , wie es funktioniert in lebenden Zellen.
Protokoll
1. Membranblatt Vorbereitung und Fixierung
- Bereiten Membranblätter markiert mit iRFP-PAmCherry1-PH PLC δ1
- Bereiten Sie die DNA - Plasmid - Codierung iRFP-PAmCherry1-PH PLC 23 molekularen Standardbiologische Techniken mit & dgr; 1.
- Kultur INS-1 - Zellen auf # 1.5 18 mm runden Deck vorbeschichtet mit 30 ug / ml Fibronektin zu 50-70% Konfluenz folgenden Standard INS-1 - Zellkulturprotokolle 38,39. Transfizieren einen Tag, um die Zellen nach 50 bis 70% Konfluenz erreicht ist.
- Transfizieren Zellen mit iRFP-PAmCherry1-PH PLC & dgr; 1 der Protokolle folgenden Hersteller Liposom Transfektionsreagenz verwenden. Nach der Transfektion erlauben, die Zellen für 48 Stunden wachsen.
- Am Tag des Experiments coat Deckgläser (auf einer Seite) mit ~ 0,5-1 ml von 500 ug / ml Poly-D-Lysine; für 1-2 h (PDL in dH 2 O verdünnt). Dann PDL Drain durch ein Tissue-Papier Platzierung aufder Rand des Deckglases. Platzieren Sie die Deckgläser auf einem vorgekühlten (4 ° C) Metallplatte zur späteren Verwendung.
- Waschen vorge transfizierten INS-1-Zellen wachsen auf Deckgläser mit ~ 0,5-1 ml eiskalter phosphatgepufferter Kochsalzlösung (PBS), enthaltend 1 mM EGTA. Wiederholen Sie die Wäsche dreimal und Drain-PBS.
- Platzieren Sie die Deckgläser mit kultivierten Zellen (cell-Seite nach unten) auf den PDL-beschichtete Deckgläser auf einem vorgekühlten Metallplatte mit einer Pinzette. Verlassen , um die Platte in einem Kühlschrank (4 ° C) für 7 bis 10 min Zellen zu ermöglichen, den PDL-beschichtete Deckgläser (Abbildung 2) zu befestigen.
HINWEIS: Die Inkubationszeit in dem Bereich von 7 sein sollte - 10 min für eine bessere Befestigung der Zellen an der PDL-beschichteten Oberfläche. Der Kühlschrank Umgebung ist trocken und Inkubation sollte nicht zu lang sein. - Entfernen Sie die Metallplatte aus dem Kühlschrank. schälen Sie vorsichtig das Deckglas die Pre-transfizierten Zellen mit einer Pinzette enthält. Dies erzeugt eine dünne Schicht der Zellmembran Blatt auf derPDL-beschichtete Deckglas. Waschen Sie sich leicht die Membranblätter mit ~ 0,5-1 ml eiskaltem PBS und dann fix mit ~ 0,5-1 ml eiskaltem 4% Paraformaldehyd (PFA) + 0,2% Glutaraldehyd (GA) in PBS für 15 min bei 4 ° C.
Achtung: Paraformaldehyd und Glutaraldehyd sind giftig. Behandeln Sie sie in einem Abzug mit der Haut und Augenschutz. - 1 ml PBS bei 4 ° C - Nach der Fixierung Bild sofort die Membranfolien oder lagern in ~ 0.5 (siehe Abschnitt 3).
HINWEIS: Nachdem das Membranblatt Fixierung, wird es kein Gleichgewicht sein Bindung von pH - Sonden von Cytosol PI (4,5) P2. Die gebundenen pH-Sonden werden nach und nach aus dem Membranblatt dissoziieren und diffundieren in die Lösung (obwohl es Tage bis Wochen dauern kann). Daher ist es die festen Proben direkt nach der Probenvorbereitung auf Bild empfohlen.
- Bereiten Membranblätter für PI (4,5) P 2 - spezifische Antikörper - Markierung
- Kultur INS-1-Zellen auf # 1.5, 18 mm rund Deck vorbeschichtet mit 30 &# 181; g / ml Fibronektin zu 50% -70% Konfluenz. Transfizieren Zellen mit Plasmid-DNA, am nächsten Tag als 1.1.3 in Schritt beschrieben.
- Wiederholen Sie die Schritte von 1.1.4 bis 1.1.7, wie oben beschrieben.
ANMERKUNG: Führen Sie die folgenden Verfahren, bei 4 ° C. - Waschen Sie die Deckgläser befestigt PM Blätter enthält , dreimal mit ~ 0,5 bis 1 ml eiskaltem PBS 50 mM NH 4 Cl enthält. Quenche die Blätter mit ~ 0,5-1 ml 0,1% Natriumborhydrid in PBS für 7 min, und wasche mit ~ 0,5-1 ml PBS (ohne 50 mM NH 4 Cl).
- Blockieren der Proben mit ~ 0,5-1 ml Blockierungslösung (PBS - Lösung , enthaltend 5% (v / v) normales Ziegenserum, 5% (v / v) Rinderserumalbumin und 50 mM NH 4 Cl) für 45 min. Dann inkubieren mit ~ 0,5-1 ml primären PI (4,5) P 2 - Antikörper (1: 300 Verdünnung) in Lösung für 1 h blockiert. Waschen dreimal (je 10 min) mit ~ 0,5-1 ml PBS 50 mM NH 4 Cl enthält.
- Inkubieren Proben mit ~ 0,5-1 ml Sekundär F (ab ') 2-Ziege-Anti-MausAntikörper mit Fluorophoren konjugiert (1: 300) Puffer für 1 h bei der Blockierung. Waschen dreimal mit ~ 0,5-1 ml PBS.
- Postfix-Proben mit 4% PFA + 0.2% GA für 15 min, dann waschen Proben dreimal mit PBS (7 min jedesmal). Fahren Sie mit Abschnitt 3 für die Bildgebung oder lagern in ~ 0.5 ~ 1 ml PBS bei 4 ° C für die spätere Verwendung.
HINWEIS: Bereiten Proben bei 4 ° C. Verwenden Sie 4% PFA + 0,2% GA Fixiermittel die physiologische Verteilung von Phosphoinositid in der PM zu erhalten. RT Vorbereitung oder Fixierung mit 4% PFA allein kann erhebliche Artefakte (Abbildung 4).
2. Zellkultur Vorbereitung für das Live-Cell - Imaging mit mEOS3.1-PH PLC δ1
- Bereiten Sie die DNA - Plasmid , das die mEOS3.1-PH PLC δ1 wie zuvor 23 beschrieben.
- Kultur INS-1-Zellen auf # 1.5 18 mm runden Deckgläschen vorbeschichtet mit 30 ug / ml Fibronektin, bis sie erreichen 50% -70% confluency folgende Standard - INS-1 - Zellkulturprotokolle 38,39.
- Am folgenden Tag, transfizieren INS-1 - Zellen mit dem mEOS3.1-PH PLC & dgr; 1 - DNA - Plasmid - Liposom - Transfektionsreagenz unter Verwendung des Protokolls nach der Herstellung. Inkubieren für 48 Stunden vor der Bildgebung.
3. PALM Image Acquisition von Membranblätter und lebenden Zellen
- PALM Bildaufnahme von Membranfolien
HINWEIS: Hier wird eine interne Totalreflexion Fluoreszenz (TIRF) Mikroskop auf Basis SMLM System (100X Öl, APO, NA = 1,49, WD 0,12 mm) 23 wird für alle Bildaufnahme verwendet.- Vor Bildgebung, verdünnen 1 ul fluoreszierenden Beadlösung in 10 ml PBS + 50 mM MgCl 2. In 200 ul verdünnte Lösung in die Abbildungskammer enthält, Zellproben (aus den Schritten 1.1.8 / 1.2.6) für 10 min. Dann waschen mit PBS dreimal.
- Starten Sie das PALM-Bildgebungssystem. In der zugehörigen Imaging-Software, wählen Sie den "iRFP Kanal &# 34; Taste (642 nm Laseranregung, 700/75 nm-Emission). Finden Sie die Zellmembran PH Sonden im iRFP Kanal exprimieren.
HINWEIS: Die Membranfolie eine fluoreszierende Perle in der Nähe haben. Dies wird später für Driftkorrektur erforderlich. - Verwenden Sie die "Aufnahme" Taste ein herkömmliches TIRF Bild von der Zellmembran in iRFP Kanal als Referenz für zukünftige Bildrekonstruktion zu sammeln. Stellen Sie einen normalen TIRF Winkel (dh nach den kritischen Winkel erreichen, biegen Sie weitere 1,5 Grad) von in 2140 in den TIRF Winkel Eingabe für die Bildgebung zu setzen. Verwenden Sie diesen Winkel für alle Schritte einstellen, sofern nicht anders vermerkt.
HINWEIS: In der TIRF hier beschriebenen System 3500 ist die vertikale up Winkel, 2200 ist der TIRF kritische Winkel, und 2140 ist der normale TIRF Winkel, der 1,5 Grad mehr nach TIRF kritische Winkel. Der schmale TIRF Winkel unten erwähnt in 3.2.3 ist 2120, das ist ein weiterer 2 Grad nach TIRF kritischen Winkel erreicht. - Wechseln Sie in den PAmCherry1 Kanal durch th klickene RFP Kanaltaste (561 nm Anregung, 600/50 nm Emissionsfilter). Stellen Sie die Bildlaufleiste in der "AOTF" pad den ganzen Weg auf der rechten Seite für 10-20 sec volle Leistung Beleuchtung von 561-nm-Laser zu haben, um die Hintergrund Membran Fluoreszenz bleichen.
- Stellen Sie die optimale Kameraeinstellung (2x2 Binning) im Register "Format" und die schnelle Akquisitionsprotokoll in "ND Sequenzerfassung" Register (typischerweise 10.000-40.000 Bilder bei 20 Hz).
- Starten Sie Erwerb von PAmCherry1 Bilder (561-nm-Laser bei voller Leistung, 50 ms / Frame) bei gleichzeitiger 405 nm Laseraktivierung auf einem niedrigen Niveau (0,1% - 1%), indem Sie auf den "Jetzt ausführen" klicken. Passen Sie die Intensität von 405 nm Laser, so dass räumlich getrennt einzelne Punkte in jedem Frame leicht identifizierbar sind.
HINWEIS: Die Anzahl der aktivierbaren PAmCherry1 Moleküle nimmt allmählich während der Erfassung. 405 nm Laserintensität allmählich erhöht werden sollte, um die optimale Dichte der einzelnen Molekül Signale in jedem zu haltenRahmen. - Weiter Bilder zu erwerben, bis kein PAmCherry1 einzelnes Molekül Signal aktiviert wird.
- PALM-Bildgebung in lebenden Zellen
- Vor der Bebilderung, verdünntem fluoreszierenden Kügelchen in die Abbildungskammer für 10 min, wie in Schritt 3.1.1 beschrieben. PALM Bildgebung durch Öffnen der zugehörigen Imaging-Software Starten Sie dann.
HINWEIS: Verwenden Sie eine magnetische Schnellspann Imaging-Kammer für Live-Cell-Imaging. Aufrechterhaltung der Mitte des Abbildungsfeldes bei 35 ° C mit einem Temperaturregler unter konstanter Perfusion mit extrazellulären Puffer (extrazelluläre Lösung: 135 mM NaCl, 5,6 mM KCl, 2,6 mM CaCl 2, 1,2 mM MgCl 2, 3 mM Glucose und 20 mM HEPES; pH = 7,3). - Wählen Sie die GFP-Kanal-Taste (525/50 nm-Emission). Identifizieren Sie die Zellmembran fluoreszierende Sonden auf Basis von grün fluoreszierenden von mEOS3 unter 488 nm Laseranregung ausdrückt. Stellen Sie sicher, dass fluoreszierende Kügelchen in der Nähe auch während der Bildgebung wie diese enthalten sind später für die Offline-dri erforderlich istft Korrektur.
- Klicken Sie auf die "Aufnahme" Taste, um ein TIRF Bild von der Zellmembran in mEOS3 (grün) Kanal als Referenzbild zu sammeln. Verwenden Sie eine flache abklingende Welle Anregungsbeleuchtung (nach den kritischen Winkel erreichen, biegen Sie weitere 2 Grad) von in 2120 in der TIRF Winkel eingeben Einstellung für Live-Cell-Imaging.
ANMERKUNG: Dies minimiert die Fluoreszenz von dem ungebundenen mEos3-PH im Cytosol angrenzend an die Plasmamembran. - Wechseln Sie in den "RFP-Kanal" Taste (561 nm Anregung, 600/50 nm-Emission). Verwenden Sie die volle Leistung 561 nm Laser-Hintergrundfluoreszenz der Membranblätter zu bleichen für 10 ~ 20 sec mit dem Scrollbar in der "AOTF pad" (siehe 3.1.4).
- Starten der Bildaufnahme die "Run Now" durch Klicken auf die Taste im roten Kanal (561 nm volle Leistung, eingestellt bei 10 ms / Frame in der "Kamera" Einstellung Tab) bei gleichzeitiger 405 nm Laseraktivierung. Stellen Sie die 405-nm-Laserintensität zu aktivieren räumlich getrennten Punktenin jedem Rahmen.
HINWEIS: Erwerb Erlös, un-umgewandelt mEOS3.1-PH PLC δ1 Molekülzahl langsam abnimmt. Erhöhen Sie die 405 nm Laserleistung des einzelnen Moleküls Signal zu optimieren. Die Abbildungsakquisitionslänge ist abhängig von der experimentellen Zweck. Erwerben Sie Bilder kontinuierlich für 5 min unter normalen Bedingungen. Wenn eine längere Akquisition erforderlich ist, sammeln mehrere Bildstapel statt eines einzelnen, großen Bildstapel. Die Dateigröße der einzelnen Bildstapel sollte nicht mehr als 4 GB oder es wird später Software-Analyse beeinflussen.
- Vor der Bebilderung, verdünntem fluoreszierenden Kügelchen in die Abbildungskammer für 10 min, wie in Schritt 3.1.1 beschrieben. PALM Bildgebung durch Öffnen der zugehörigen Imaging-Software Starten Sie dann.
4. SMLM Bildverarbeitung und Wiederaufbau
- Übertragen Sie die Bilddateien (TIF-Bildstapel) zu einer Bildanalysestation. Starten Sie die Bildwiederaufbauprogramm (speziell geschriebenen) in Matlab wie zuvor 37 beschrieben und laden Sie die Bildstapel durch die Registerkarte "Datei" im Hauptmenü klicken, dann auf "öffnen", dann "neue Datei".
- Identifizieren und locaLize einzelnen molekularen Ereignisse aus jedem Rahmen. Stellen Filterung Intensitätsschwelle mit einer Schwellenzahl (1-10) in der "Wavelet". Überprüfen Sie die optimalen Parametereinstellungen für Punkterkennung visuell vor dem Umbau. Dann gehen Sie auf die "PALM" und klicken Sie "einen Schritt Prozess" Bildrekonstruktion zu starten.
HINWEIS: Intensitäts-Grenzwert für Punkterkennung ist willkürlich und hängt von persönlichen Erfahrungen. Mehrere Studien kann eine optimale Detektionsschwelle zu erzeugen, erforderlich sein, dass weder zu viele unqualifizierte aufgreift (dim) Punkte noch filtert zu viele qualifizierte (hell) Punkte aus. Stellen Sie andere Parameter Datenanalyse zu optimieren. Hier Nachbarschaft Abstand (Fluoreszenzpunkte in den benachbarten Rahmen in einem Abstand kombiniert werden) wird als 65 nm (1/2 Pixelbreite) und als ein einzelnes Molekül eingebaut. Gap-Frames (Einzelmolekül Ereignisse, die innerhalb dieser Rahmen und Nachbarschaft Entfernung auftraten, wurden kombiniert und als ein einzelnes Molekül Ereignis vorhanden) istSet als 26 Frames (1,3 sec) für PAmCherry1 Bildgebung 40. Stellen Sie diese Parameter entsprechend den Eigenschaften der Einzelmolekülsonden und Erfassungsrate zu vermeiden 40 in SMLM Über zählen. Anwenden Driftkorrektur mit fluoreszierenden Kügelchen während der Rekonstruktionen.- Für die Abbildung der festen Membranblätter gekennzeichnet durch iRFP-PAmCherry1-PH PLC δ1, rekonstruieren die ganze Bildstapel aus der gleichen Membranfolie in einem einzigen Super-Resolution Bild 37.
- Für lebende Zelle markierten Proben von mEOS3.1-PH PLC δ1, den gesamten Bildstapel in mehrere kleinere Stapel von 1.000 Frames trennen , so dass 1000-konsekutiven Rahmen Stapel wird als ein einzelnes Superauflösungsbild rekonstruiert, die eine zeitliche Auflösung hat von 10 sec. Am Ende vereinen alle Zeitreihen - Bilder in der letzten Zeitreihenstapel 23.
HINWEIS: In lebenden Zellen PALM-Bildgebung kann eine kleine Anzahl der aktivierten Sonden bewegen oder trennenvon PI (4,5) P 2 in der PM vor dem Bleichen. Um der Überabtastungs der Sonden verbinden einzelnen Molekül Signale in den benachbarten Einzelbildern innerhalb von 130 nm (anstelle von 65 nm) in einem einzigen Emissionsereignis während der Bildanalyse.
- Nachdem die rekonstruierten Bilder zu erhalten, führen Sie weitere Bildanalyse mit dem speziell geschriebenen Programm in Matlab Moleküldichte, Mikrodomänendichte / Größe und Clusteranalyse mit paar Korrelation 41 zu quantifizieren.
Ergebnisse
Die Lokalisierungsunsicherheit (σ) unserer Super-Resolution - System ist 14,73 nm 23. Direkte Vergleiche zwischen TIRF und PALM Bilder zeigten eine signifikante Verbesserung der räumlichen Auflösung. 3A-B zeigt die repräsentative PI (4,5) P 2 TIRF Bilder markiert mit PI (4,5) P 2 - Antikörper und iRFP-PAmCherry1-PH PLC δ1 in typischen Membranfolien und lebenden Zellen. Die Bilder mit herkömmlichen TIRF - Mikroskopie in Membranblätter bemerkenswert ähnlich sind zu denjenigen in intakten lebenden mit dem enhanced green fluorescence protein markierten Zellen (EGFP) tagged PH Domänen (EGFP-PH PLCδ1) (3C). Alle Proben zeigten eine gleichmäßige Verteilung von Sonden. Im Gegensatz dazu führte nicht optimale Fixierung der Proben in scharfen dichten PI (4,5) P 2 -Clustern und eine Abnahme der Signalintensität (Abbildung 4). Unter optimalen Fixierbedingungen, ter super-aufgelöste Bilder von PI (4,5) P 2 in fixierten Zellen (Figur 5) zeigte eine homogene Verteilung von Sonden in einem erheblichen Teil des PM nur begrenzten Konzentrationsgradienten. Einige Membranflecken angereichert mit PI (4,5) P2 - Sonden wurden dünn verteilt und hatten verschiedene Größen. Live Cell PALM Bilder zeigen eine ähnliche räumliche Verteilung als feste Zellen (Abbildung 6). Eine detaillierte Analyse von PI (4,5) P2 Signale über die Zeit resultiert in schnelle Dynamik in lokalen Bereichen, ohne wesentliche Änderungen ihrer Fülle in weiten Bereichen.
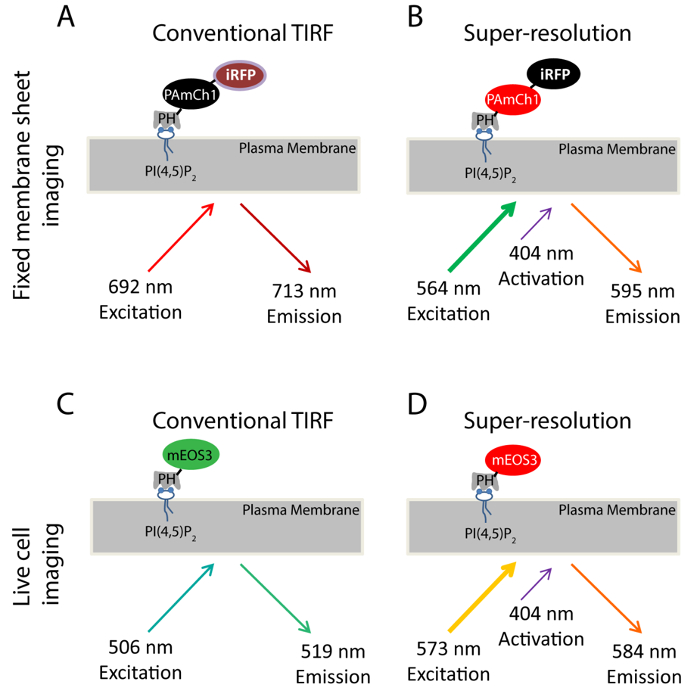
Abbildung 1. Schema für Fluoreszenzsonden in dieser Studie verwendet. (AB) iRFP-PAmCherry1-PH PLC δ1 Sonde in den festen Membranfolie Experimente verwendet. Während herkömmliche TIRF imaging (A), eine 640 nm Laser wird verwendet, um die iRFP zu erregen (Ex: 692 nm; Em: 713 nm). TIRF Bild in diesem Zustand genommen dient als TIRF Referenzbild für die superauflösende Bildgebung von PALM - Bildgebung (B) erhalten. A 405-nm-Laser verwendet wird Photo aktivieren Sie die PAmCherry1 Fluorophor und ein 561-nm-Laser verwendet wird PAmCherry1 zu erregen (Ex: 564 nm; Em: 595 nm) für PALM-Bildgebung. (CD) mEos3.1-PH PLCδ1 Sonde in den lebenden Zellen Experimente verwendet. (C) Bei der konventionellen TIRF Bildgebung, ein 488 nm - Laser verwendet mEOS3.1 (Ex: 506 nm; Em: 519 nm) anzuregen. (D) Nach dem Photokonversion von einem 405 - nm - Laser, wird mEos3.1 in die rote Form (Beispiel: 573 nm; Em: 584 nm). Und einem 561 - nm - Laser wird für PALM Akquisitionen Bitte hier klicken , um eine größere Version zu sehen diese Figur.
s / ftp_upload / 54466 / 54466fig2.jpg "/>
Abbildung 2. Schema für Membranblatt Vorbereitung von INS-1 - Zellen. (A) Legen Sie das Deckglas mit kultivierten Zellen nach unten auf einen PDL-beschichtete Deckglas und warten auf 7 ~ 10 min bei 4 ° C Zellbindung an die PDL- zu ermöglichen beschichtete Deckglas. (B) die obere Deckglas mit einer Pinzette abziehen und fixieren Sie die Folienmembran auf die vorbeschichteten Deck PDL angebracht. (C) Bild der Proben mit TIRFM und PALM. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
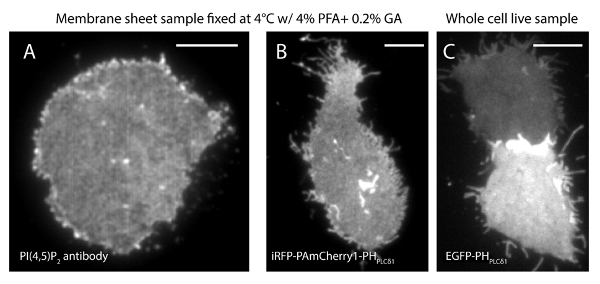
Abbildung 3. PI (4,5) P 2 räumliche Organisation ist ähnlich zwischen den Membranblättern und intakten lebenden Zellen unter konventionellen TIRF - Mikroskop. (A) Atypische TIRF Bildbögen Membran bei 4 ° C mit 4% PFA und 0,2% GA fixiert. PI (4,5) P 2 wurde mit PI (4,5) P 2 spezifische Antikörper markiert. (B) ein Membranblatt von der INS-1 - Zellen iRFP-PAmCherry1-PH PLC δ1 exprimieren. (C) TIRF - Bild von zwei intakten lebenden Zellen EGFP-PH PLCδ1 auf verschiedenen Ebenen. Maßstabsbalken: (A): 3 & mgr; m; (B) und (C):. 5 & mgr; m Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
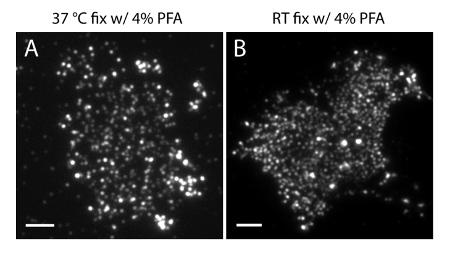
Abbildung 4. PI (4,5) P 2 räumliche Organisation in Membranblätter ist empfindlich gegen gemeinsamen Fixierbedingungen. (A) Membranblätter bei 37 ° C mit 4% PFA alleinund PI (4,5) P 2 - spezifischen Antikörpers (wie in 3A) markiert. (B) Membranfolie bei RT mit PFA fixiert allein und mit PI (4,5) P 2 spezifische Antikörper markiert. Man beachte , dass die dichten Cluster von PI (4,5) P 2 Sonden unter TIRF Mikroskop deutlich sichtbar sind, im Gegensatz zu den viel gleichmäßiger Fluoreszenzbilder in 3A gezeigt. Maßstabsbalken: AB:. 3 & mgr; m Bitte klicken Sie hier um eine größere Version dieser Figur zu sehen.
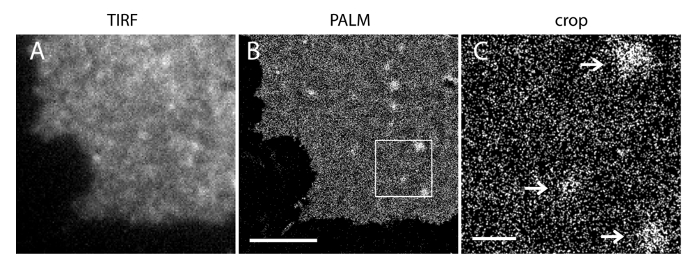
Abbildung 5. PALM Bildgebung von PI (4,5) P 2 Sonden zeigt ihre Nanometer - Skala Verteilung in einem INS-1 - Zellen Uhr. (A) iRFP TIRF Bild einer Membranfolie aus einer INS-1 - Zellen exprimiert iRFP-PAmCherry1-PH PLCδ1. (B) entsprechende PALM Bild von PM in der gleichen Region basierend auf PAmCherry1 Signalrekonstruktion. Beachten Sie die homogene PI (4,5) P 2 räumliche Verteilung in großen PM Regionen und mehrere PI (4,5) P 2 Mikrodomänen. (C) eine vergrößerte Ansicht des eingerahmten Bereichs in (B). Pfeile zeigen dünn mit PI (4,5) P 2 Sonden angereichert PM Mikrodomänen verteilt. Maßstabsbalken: A und B: 3 um; C: 500 nm. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
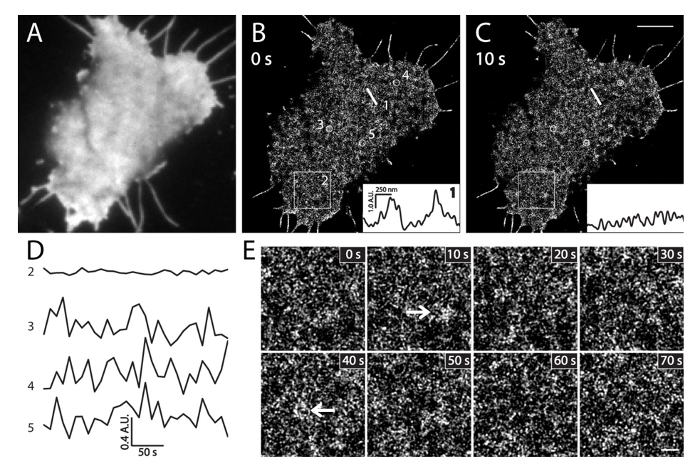
Abbildung 6. PALM Bildgebung in der Live - INS-1 - Zellen. (A) TIRF Bild von PI (4,5) P 2 in einer Live - INS-1 - Zellen mEos3.1-PH PLC exprimierenδ1. Das Bild wurde in den grünen Kanal vor PALM Erwerb (35 ° C) schnell erworben. (B) Sequential lebenden Zellen PALM Bilder in 10 Sekunden - Intervall. Einschübe zeigen die Intensitätsprofile des lokalen PI (4,5) P 2 Dichte entlang der gleichen Position gerade Linie 1 zu verschiedenen Zeitpunkten in (B) und (C). Beachten Sie ihre großen lokalen Intensitätsänderungen innerhalb von 10 Sekunden. (D) Zeitverlauf der durchschnittlichen Intensitätsänderungen von PI (4,5) P 2 in der großen Fläche (box2, 3x3 um) und kleine Kreise (3, 4, und 5, 500 nm Durchmesser) in (B) während 5 min von PALM-Bildgebung (Rahmen / 10 sec). Beachten Sie die schnelle Intensitätsschwankungen der lokalen PI (4,5) P 2 Sonden (Kreis 3, 4 und 5) im Vergleich zu sehr kleine Veränderungen in der breiten Fläche (Kasten 2). (E) Vergrößerte PALM Bilder der box2 Region in (B) zu den angegebenen Zeiten. Die Pfeile zeigen PI (4,5) P 2 angereicherte Membranflecken unter physiological Bedingungen. Maßstabsbalken: C: 3 um; E: 500 nm. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
Diskussion
Zur Fehlersuche, müssen zwei Prozesse besondere Aufmerksamkeit: Membranfolie Produktion und Probenfixierung. Wie in dem Protokoll beschrieben, ist die Inkubationszeit von den Deckgläsern in Schritt 1.1.6 wichtig für die Membranfolie Produktion. Optimale Inkubationszeit unter unseren experimentellen Bedingungen ist 7-10 min (Abbildung 2). Länger als 10 Minuten Inkubation werden intakte Zellen produzieren anstelle der Membranblätter auf PDL Deck und kürzere Inkubation wird weniger oder kein Membranblätter auf den PDL-beschichtete Deckgläser führen. Wie in dem Protokoll beschrieben, sind die Fixiermittel und Temperatur während der Fixierung der kritischen PI (4,5) P 2 -Verteilung im PM aufrechtzuerhalten. Die Fixierung bei RT oder die Verwendung von 4% -PFA allein könnte die normale Lipidverteilung in der PM verzerren.
Durch die Anwendung PALM - Mikroskopie Lipidforschung an der Membran, sind wir in der Lage , die Nanometer - Skala Verteilung von PI (4,5) P2, ein Schlüssel Phosphoinositid , die m vermittelt zu beobachtenalle grundlegenden zellulären Aktivitäten. Diese räumliche Verteilung von PI (4,5) P2 mit begrenzter Konzentrationsgradienten in INS-1 - Zellen bietet einen Rahmen Lipid-Protein - Wechselwirkungen und lokale Signalereignisse von PI (4,5) P 2 in diesen Zellen für ein Umdenken. Darüber hinaus sind die in dieser Arbeit entwickelten Methoden können auch mit der richtigen Sonden, wodurch bietet neue Werkzeuge zum Studium Phosphoinositiden in biologischen Prozessen zu anderen Membran Phospholipid Forschung angewendet werden.
Die Verwendung von Membranfolien in dieser Arbeit umgeht zwei wichtige Anliegen in Phospholipid morphologische Studien: Detergens-Behandlung und mögliche Signal Kontamination aus dem Cytosol. Waschmittel verursachen oft Clustering und einen erheblichen Verlust von PM Phospholipid-Signal. Die Kontamination von zytosolischen Signal ist besonders problematisch im Fall von geringer Häufigkeit Phosphoinositiden auf der PM 23, wie beispielsweise PI (3,4,5) P 3 und PI (3,4) P 2. Membranfolienproben sind in der Lage Circumvent diese Probleme ohne wesentliche Strukturen mit der Plasmamembran, wie kortikale Aktin meshwork und Clathrin-beschichtete Pits 42,43 zugeordnet stören. Die relative homogene Verteilung von PI (4,5) P2 in der PM ist in guter Übereinstimmung mit anderen schnellen Einfrieren EM - Studien unter Verwendung von GST-PH PLC δ1 Sonden in der Fibroblasten - Membran 44.
Es ist wichtig zu beachten, dass unsachgemäßer Probenverarbeitungsbedingungen irreführende Ergebnisse erzeugen kann. Erstens ist es wichtig, die Befestigungsschritte bei einer niedrigeren Temperatur (4 ° C) und Verwendung der Fixiermittel GA für Membranfolienherstellung durchzuführen. Wie in 3 gezeigt, warme Temperatur und PFA Fixierung ohne GA ist nicht ausreichend Phosphoinositid in Zelle PM zu beheben. Dies könnte die intakten PI (4,5) P 2 Verteilung verzerren und scharfe Cluster erzeugen , die in lebenden Zellen unter physiologischen Bedingungen beobachtet werden , nicht. Zweitens ist die Verwendung von PAmCherry1 alsdie SMLM Sonde, eher als andere Sonden ist, ausschlaggebend für die quantitative PALM-Bildgebung. Der Vorteil der PAmCherry1 Anwendung kommt von seiner gut charakterisierten einzelnen molekularen photophysikalischen Eigenschaften 35,40,45, wie Helligkeit, hohe Photoaktivierung Effizienz und vor allem sehr begrenzt Photo Blinken. Diese Eigenschaften ermöglichen es uns , potenzielle Cluster Artefakte aus Photo Blinken zu beseitigen und quantitativ die molekulare Dichte der Membran PI analysieren (4,5) P 2.
Dieser Ansatz hat auch seine Grenzen. Erstens verwendet das Membranblatt Verfahren in dieser Studie nicht vollständig kann die physiologische Verteilung von PI (4,5) P 2 nachahmen , da die Zelle vor der Abbildung gestört wird. Aber unsere Live - PALM - Bildgebung zeigt ähnliche relativ homogene Verteilung von PI (4,5) P2, die Ergebnisse beobachtet , die mit Membranfolie Proben unterstützen. Zweitens, wie wir in unserer bisherigen Arbeit 23 diskutiert, erfordert SMLM Imaging Expertise und extra antention Bildartefakte zu vermeiden, die von verschiedenen Prozessen, einschließlich der verwendeten Sonden, Probenvorbereitung und Fixierung, Bild Probenahme und Wiederaufbau entstehen könnten. Schließlich, obwohl die PH - Domäne basierte Sonden und Antikörper sind weit verbreitet in der Phosphoinositid Studien 46,47 verwendet worden bleibt es möglich , dass nicht alle PI (4,5) P2 in der Membran kann durch diesen Ansatz nachgewiesen werden. Beispielsweise PI (4,5) P 2 durch andere endogene Proteine gebunden sind, können nicht auf pH - Sonden oder Antikörper zugänglich sein, und dies kann eine Unterschätzung von PI (4,5) P 2 aufgrund der Raum Hinderung der Sonden selbst verursachen. Eine alternative Möglichkeit zur Etikettierung PI (4,5) P 2 würde auf den Schwanz des ursprünglichen Top-Fluor PI (4,5) P 2 48, a pre-markierten PI (4,5) P 2 Analogon mit einer Modifikation verwendet werden , PI (4,5) P 2. Allerdings kann es schnell in andere Phosphoinositid-Subtypen durch schnelle Live-Zellstoffwechsel umgewandelt werden, da seine Inositolrings die gleiche wie endogenou ists PI (4,5) P 2. Damit stellt sich die Sorge , ob diese voretikettierten PI (4,5) P 2 analoge in lebenden Zellen treu PI (4,5) P 2 nicht seine Stoffwechselprodukte darstellen kann. Daher ist trotz einiger Einschränkungen, PH - Domäne basierte Sonden sind immer noch zu den besten Sonden , die häufig verwendet wurden PI (4,5) P 2 Verteilung und Dynamik auf dem PM von Zellen zu überwachen.
Die künftige Anwendung dieser Methodik kann auf andere Phosphoinositid Studien erweitert werden, wie beispielsweise PI (3,4,5) P 3 und PI (3,4) P 2. Zusammenfassend verwendet die neuartige SMLM Ansatz eröffnet hier neue Wege Phosphoinositid in Zellen zu untersuchen. Verwendung von PI (4,5) P 2 als Beispiel zeigen wir die einzigartigen Eigenschaften von PALM Bildgebung in die morphologische und quantitative Untersuchung der Zellmembran - Moleküle sowie ihre Nachteile. Dieser Ansatz kann an andere Moleküle von Interessen angepasst werden und breite Anwendungen in der Zellbiologie haben.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
This work is supported by the National Institutes of Health (NIH) grants R01DK093953 (X.L.), P30NS069271, BRFSG-2014-07 (X.L.). C.J. is partially supported by American Heart Associate pre-doctoral fellowship (14PRE20380168, to C.J.).
Materialien
| Name | Company | Catalog Number | Comments |
| Microscope | Nikon | Ti-U | |
| sCMOS camera | Andor | Neo | |
| Spinning disk | Yokogawa | CSU X-1, 10,000 rpm | |
| High Power Monolithic Laser Combiner SP with 405, 488, 561 and 642 nm lasers. | Agilent | MLC400 | laser original powers & powers at the end of optical fiber) 405 nm: 15 mW & 13 mW; 488 nm: 45 mW & 42 mW; 561 nm: 45 mW & 40 mW; 640 nm: 35 mW & 16 mW. |
| 100X Objective | Nikon | APO 100X Oil, NA 1.49, WD 0.12 mm | |
| Nikon acquisition and analysis software (NIS Element) | Nikon | ||
| Matlab | MathWorks | ||
| Coverslip | Warner | 64-0714 | Round 18 mm coverslip, #1.5 |
| Fibronectin | Millipore | FC010-5MG | |
| Lipofectamin 3000 (transfection reagent) | Invitrogen | L3000-008 | |
| Glutaraldehyde | Electron Microscopy Science | #16120 | |
| PI(4,5)P2 1st antibody | Santa Cruz | sc-53412 | |
| secondary antibody F(ab’)2-goat-anti-mouse Alexa Fluor 647 | Invitrogen | A-21237 | |
| Tetraspeck beads | Invitrogen | T7279 | |
| magnetic quick release imaging chamber | Warner Instruments | Cat#641994 | |
| temperature controller | Warner Instruments | TC-344C | |
| Poly-D-Lysine | Sigma | P6407 | |
| Phosphate buffer saline | Sigma | P4417-100TAB | |
| EGTA | Sigma | 3780 | |
| NH4Cl | Sigma | A0171 | |
| Goat serum | Sigma | G9023 | |
| bovine serum albumin | Sigma | A7906 | |
| NaCl | Sigma | S5886 | |
| KCl | Sigma | P5405 | |
| CaCl2•2H2O | Sigma | 223506 | |
| MgCl2•6H2O | Sigma | M9272 | |
| Glucose | Sigma | G7021 | |
| HEPES | Sigma | H4034 | |
| Paraformaldyhyde | Sigma | P6148 |
Referenzen
- Di Paolo, G., De Camilli, P. Phosphoinositides in cell regulation and membrane dynamics. Nature. 443, 651-657 (2006).
- Hammond, G. R. V., et al. PI4P And PI(4,5)P(2) Are Essential But Independent Lipid Determinants Of Membrane Identity. Science. 337, 727-730 (2012).
- Nakatsu, F., et al. PtdIns4P synthesis by PI4KIIIalpha at the plasma membrane and its impact on plasma membrane identity. J Cell Biol. 199, 1003-1016 (2012).
- Cantley, L. C. The phosphoinositide 3-kinase pathway. Science. 296, 1655-1657 (2002).
- Czech, M. P. Dynamics of phosphoinositides in membrane retrieval and insertion. Annu Rev Physiol. 65, 791-815 (2003).
- Spira, F., et al. Patchwork organization of the yeast plasma membrane into numerous coexisting domains. Nat Cell Biol. 14, 640-648 (2012).
- Barg, S., Knowles, M. K., Chen, X., Midorikawa, M., Almers, W. Syntaxin clusters assemble reversibly at sites of secretory granules in live cells. Proc Natl Acad Sci U S A. 107, 20804-20809 (2010).
- Sieber, J. J., et al. Anatomy and dynamics of a supramolecular membrane protein cluster. Science. 317, 1072-1076 (2007).
- Knowles, M. K., et al. Single secretory granules of live cells recruit syntaxin-1 and synaptosomal associated protein 25 (SNAP-25) in large copy numbers. Proc Natl Acad Sci U S A. 107, 20810-20815 (2010).
- Milosevic, I., et al. Plasmalemmal phosphatidylinositol-4,5-bisphosphate level regulates the releasable vesicle pool size in chromaffin cells. J Neurosci. 25, 2557-2565 (2005).
- van Rheenen, J., Jalink, K. Agonist-induced PIP(2) hydrolysis inhibits cortical actin dynamics: regulation at a global but not at a micrometer scale. Mol Biol Cell. 13 (2), 3257-3267 (2002).
- Hammond, G., Schiavo, G., Irvine, R. Immunocytochemical techniques reveal multiple, distinct cellular pools of PtdIns4P and PtdIns (4, 5) P2. Biochem J. 422, 23-35 (2009).
- Huang, S., et al. Phosphatidylinositol-4,5-bisphosphate-rich plasma membrane patches organize active zones of endocytosis and ruffling in cultured adipocytes. Mol Cell Biol. 24, 9102-9123 (2004).
- James, D. J., Khodthong, C., Kowalchyk, J. A., Martin, T. F. Phosphatidylinositol 4,5-bisphosphate regulates SNARE-dependent membrane fusion. J Cell Biol. 182, 355-366 (2008).
- Laux, T., et al. GAP43, MARCKS, CAP23 modulate PI(4,5)P(2) at plasmalemmal rafts, and regulate cell cortex actin dynamics through a common mechanism. J Cell Biol. 149, 1455-1472 (2000).
- Aoyagi, K., et al. The activation of exocytotic sites by the formation of phosphatidylinositol 4,5-bisphosphate microdomains at syntaxin clusters. J Biol Chem. 280, 17346-17352 (2005).
- Kabachinski, G., Yamaga, M., Kielar-Grevstad, D. M., Bruinsma, S., Martin, T. F. CAPS and Munc13 utilize distinct PIP2-linked mechanisms to promote vesicle exocytosis. Mol Biol Cell. 25, 508-521 (2014).
- Wang, J., Richards, D. A. Segregation of PIP2 and PIP3 into distinct nanoscale regions within the plasma membrane. Biol Open. 1, 857-862 (2012).
- Hell, S. W., Wichmann, J. Breaking the diffraction resolution limit by stimulated emission: stimulated-emission-depletion fluorescence microscopy. Opt Lett. 19, 780-782 (1994).
- van den Bogaart, G., et al. Membrane protein sequestering by ionic protein-lipid interactions. Nature. 479, 552-555 (2011).
- van Rheenen, J., Achame, E. M., Janssen, H., Calafat, J., Jalink, K. PIP2 signaling in lipid domains: a critical re-evaluation. Embo J. 24, 1664-1673 (2005).
- Sato, K., et al. Differential requirements for clathrin in receptor-mediated endocytosis and maintenance of synaptic vesicle pools. Proc Natl Acad Sci U S A. 106, 1139-1144 (2009).
- Ji, C., Zhang, Y., Xu, P., Xu, T., Lou, X. Nanoscale Landscape of Phosphoinositides Revealed by Specific Pleckstrin Homology (PH) Domains Using Single-molecule Superresolution Imaging in the Plasma Membrane. J of Biol Chem. 290, 26978-26993 (2015).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313, 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophys J. 91, 4258-4272 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Methods. 3, 793-795 (2006).
- Xu, K., Zhong, G., Zhuang, X. Actin, spectrin, and associated proteins form a periodic cytoskeletal structure in axons. Science. 339, 452-456 (2013).
- Wombacher, R., et al. Live-cell super-resolution imaging with trimethoprim conjugates. Nat Methods. 7, 717-719 (2010).
- Szymborska, A., et al. Nuclear pore scaffold structure analyzed by super-resolution microscopy and particle averaging. Science. 341, 655-658 (2013).
- Pertsinidis, A., et al. Ultrahigh-resolution imaging reveals formation of neuronal SNARE/Munc18 complexes in situ. Proc Natl Acad Sci U S A. 110, 2812-2820 (2013).
- Shim, S. -. H., et al. Super-resolution fluorescence imaging of organelles in live cells with photoswitchable membrane probes. Proc Natl Acad Sci. 109, 13978-13983 (2012).
- Huang, F., et al. Video-rate nanoscopy using sCMOS camera-specific single-molecule localization algorithms. Nat Methods. 10, 653-658 (2013).
- Balla, T., Várnai, P. Visualization of Cellular Phosphoinositide Pools with GFP-Fused Protein-Domains. Curr Protoc Cell Biol. 24, 24 (2009).
- Xu, C., Watras, J., Loew, L. M. Kinetic analysis of receptor-activated phosphoinositide turnover. J Cell Biol. 161, 779-791 (2003).
- Subach, F. V., et al. Photoactivatable mCherry for high-resolution two-color fluorescence microscopy. Nat Methods. 6, 153-159 (2009).
- Filonov, G. S., et al. Bright and stable near infra-red fluorescent protein for in vivo imaging. Nature Biotechnol. 29, 757-761 (2011).
- Zhang, M., et al. Rational design of true monomeric and bright photoactivatable fluorescent proteins. Nat Methods. 9, 727-729 (2012).
- Hohmeier, H. E., et al. Isolation of INS-1-derived cell lines with robust ATP-sensitive K+ channel-dependent and -independent glucose-stimulated insulin secretion. Diabetes. 49, 424-430 (2000).
- Dyachok, O., Isakov, Y., Sågetorp, J., Tengholm, A. Oscillations of cyclic AMP in hormone-stimulated insulin-secreting β-cells. Nature. 439, 349-352 (2006).
- Nan, X., et al. Single-molecule superresolution imaging allows quantitative analysis of RAF multimer formation and signaling. Proc Natl Acad Sci U S A. 110, 18519-18524 (2013).
- Sengupta, P., et al. Probing protein heterogeneity in the plasma membrane using PALM and pair correlation analysis. Nat Methods. 8, 969-975 (2011).
- Morone, N., et al. Three-dimensional reconstruction of the membrane skeleton at the plasma membrane interface by electron tomography. J Cell Biol. 174, 851-862 (2006).
- Peters, K. R., Carley, W. W., Palade, G. E. Endothelial plasmalemmal vesicles have a characteristic striped bipolar surface structure. J Cell Biol. 101, 2233-2238 (1985).
- Fujita, A., Cheng, J., Tauchi-Sato, K., Takenawa, T., Fujimoto, T. A distinct pool of phosphatidylinositol 4,5-bisphosphate in caveolae revealed by a nanoscale labeling technique. Proc Natl Acad Sci U S A. 106, 9256-9261 (2009).
- Durisic, N., Laparra-Cuervo, L., Sandoval-Álvarez, &. #. 1. 9. 3. ;., Borbely, J. S., Lakadamyali, M. Single-molecule evaluation of fluorescent protein photoactivation efficiency using an in vivo nanotemplate. Nat Methods. 11, 156-162 (2014).
- Thomas, C. L., Steel, J., Prestwich, G. D., Schiavo, G. Generation of phosphatidylinositol-specific antibodies and their characterization. Biochem Soc Trans. 27, 648-652 (1999).
- Várnai, P., Balla, T. Live cell imaging of phosphoinositides with expressed inositide binding protein domains. Methods. 46, 167-176 (2008).
- Honigmann, A., et al. Phosphatidylinositol 4,5-bisphosphate clusters act as molecular beacons for vesicle recruitment. Nature Struct Mol Biol. 20, 679-686 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten