Method Article
Hohe Auflösung Quantitative Synaptische Proteome Profilieren von Maus Hirnregionen Nach Auditory Diskriminierung Lernen
In diesem Artikel
Zusammenfassung
The identification of molecules and pathways controlling synaptic plasticity and memory is still a major challenge in neuroscience. Here, a workflow is described addressing the relative quantification of synaptic proteins supposedly involved in the molecular reorganization of synapses during learning and memory consolidation in an auditory learning paradigm.
Zusammenfassung
The molecular synaptic mechanisms underlying auditory learning and memory remain largely unknown. Here, the workflow of a proteomic study on auditory discrimination learning in mice is described. In this learning paradigm, mice are trained in a shuttle box Go/NoGo-task to discriminate between rising and falling frequency-modulated tones in order to avoid a mild electric foot-shock. The protocol involves the enrichment of synaptosomes from four brain areas, namely the auditory cortex, frontal cortex, hippocampus, and striatum, at different stages of training. Synaptic protein expression patterns obtained from trained mice are compared to naïve controls using a proteomic approach. To achieve sufficient analytical depth, samples are fractionated in three different ways prior to mass spectrometry, namely 1D SDS-PAGE/in-gel digestion, in-solution digestion and phospho-peptide enrichment.
High-resolution proteomic analysis on a mass spectrometer and label-free quantification are used to examine synaptic protein profiles in phospho-peptide-depleted and phospho-peptide-enriched fractions of synaptosomal protein samples. A commercial software package is utilized to reveal proteins and phospho-peptides with significantly regulated relative synaptic abundance levels (trained/naïve controls). Common and differential regulation modes for the synaptic proteome in the investigated brain regions of mice after training were observed. Subsequently, meta-analyses utilizing several databases are employed to identify underlying cellular functions and biological pathways.
Einleitung
Lernen beruht auf der Bildung von Speicherspuren und deren Wartung. Es ist weithin anerkannt, dass eine zugrundeliegende Mechanismus eine aktivitätsabhängige Bildung von neuen und / oder Umordnung von bestehenden synaptischen Kontakte zwischen Neuronen darstellen. Auf molekularer Ebene werden verschiedene Proteinmodifikationen, subzellulären relocalizations und Veränderungen in den Umsatz der synaptischen Proteinen 1-4 (Lamprecht 2004 # 8) beschrieben worden. Jedoch so weit meisten Studien auf ausgewählte Proteine konzentriert und nicht auf der globalen, aber komplexen synaptischen Proteom Zusammensetzung. Der vorliegende Ansatz erlaubt eine objektive Screening für die synaptische Proteom Veränderungen in der Maus Hirnregionen nach einem Lernexperiment. Es ist geeignet, zeitpunktabhängige molekulare Momentaufnahmen des Lern-induzierten Reorganisation der synaptischen Architektur zu machen. Die beschriebene Workflow erfordert eine besondere Zusammenspiel von verschiedenen Spezialisten in Tierverhalten, Proteinbiochemie, Massenspektrometrie und bioinformatics.
Die gewählte Lernparadigma, das heißt frequenzmodulierten Ton Diskriminierung (FMTD), ist eine gut charakterisierte auditorischen Unterscheidungsaufgabe bei Nagern 5. Lernen und Langzeitgedächtnisbildung in diesem Shuttle-Box Go / No-Go-Aufgabe umfasst Mechanismen in Abhängigkeit von erhöhten kortikalen Dopamin-Signalisierung und die Proteinsynthese. Dementsprechend offenbarte kürzlich proteomic Studien über Gerbils und Mäuse Dopamin- und Lerninduzierten plastischen Umlagerungen von synaptischen Komponenten in kortikalen, sondern auch in mehr basal Hirnregionen , die vermutlich während 6-8 FMTD Lernen und Gedächtnis in Wechselwirkung treten. Dies zeigt, dass die Gedächtnisbildung ein komplexes Zusammenspiel verschiedener Gehirnregionen umfasst und somit könnten differentiell innerhalb dieser Regionen auf der Proteom-Ebene geregelt werden. Daher wird Präparation ausgewählter kortikalen und subkortikalen Maus Hirnregionen im Workflow enthalten.
Darüber hinaus ist die zuverlässige characterizatiauf auch schwacher Veränderungen der synaptischen Protein - Zusammensetzung erfordert eine Anreicherung von prä- und postsynaptischen Kompartimente anstatt die Analyse von Homogenaten oder rohe Membranfraktionen 9. Daher wird die Herstellung von Synaptosomen Unter Verwendung etablierter Protokolle vor Proteomanalyse beschrieben , um 10,11 der Erfassungspegel und den Dynamikbereich für die Synapse-spezifische Proteine zu erhöhen.
Eine wesentliche Voraussetzung markierungsfreie hochauflösende Massenspektrometrie für quantitative Zwecke zu verwenden, ist ein hohes Maß an Ähnlichkeit der Proteinproben. Als eher geringfügige Veränderungen der synaptischen Protein-Zusammensetzung wird erwartet, dass nach dem Lernen stattfindet, eine markierungsfreie Ansatz angemessen sein wird, entsprechende Proteinproben aus ausgebildeten und naiven Mäusen zu vergleichen. Alternativ Zustand spezifische Label Strategien Proteine / Peptide mit stabilen Isotopen (zB TMT, iTRAQ, ICPL und SILAC) sowie MS2-Label kostenlos quantification (SWATH) sind nützlich, aber sie sind teurer als die gewählte markierungsfreien Ansatz oder spezielle Hardware massenspektrometrischen benötigen.
Da oft Proteom-Screenings komplexe Datensätze ergeben, bioinformatischen Verarbeitung wird für geeignete Interpretation der Daten empfohlen. Zusätzliche Meta-Analysen kann ein besseres Verständnis der potenziellen zugrunde liegenden molekularen Mechanismen Paradigma bedingten Veränderungen und die Identifizierung der beteiligten Schlüssel zelluläre Prozesse und Signalwege zu unterstützen. Geeignete Methoden sind auch im Folgenden beschrieben.
Protokoll
Alle Verfahren, einschließlich Tierversuchspersonen wurden in Übereinstimmung mit den Vorschriften des Bundesgesetzes, die entsprechenden EU-Verordnungen und NIH-Richtlinien durchgeführt und wurden von der Ethikkommission der Landesverwaltungsamt Sachsen / Anhalt (42502-2-1102 IfN) genehmigt.
1. Auditory Lernen
- Auditory Diskriminierung Lernen in der Shuttle - Box (FMTD Paradigma) Hinweis: Immer Handschuhe tragen , während die Mäuse Handhabung.
- Haus C57Bl6 / J-Mäuse in Gruppen von drei oder vier mit freiem Zugang zu Futter-Pellets und Leitungswasser in klarem Polycarbonat Käfigen. Pflegen Sie eine 12 Stunden Licht: Dunkel-Zyklus in der Tierhaltung. Wenn Tiere aus einem anderen Labor oder von einem Unternehmen ermöglichen, mindestens eine Woche von Akklimatisierung und Eingewöhnung empfangen werden.
- Führen Sie eine Shuttle-Box Trainingseinheit pro Tag.
- Nehmen Sie die Maus aus seinem Käfig in der Tierhaltung und legen Sie sie in einem schwach beleuchteten Shuttle-Box in einer schalldichten Kammer.
- Verwenden Sie einen vollständig computergesteuerte Lernplan für Gehör Diskriminierung Lernen. Beginnen Sie mit einer Eingewöhnungszeit von 3 min der Stille, und starten Sie die Trainingseinheit.
- Verwenden Sequenzen des ansteigenden Ton (4 - 8 kHz, CS +) als Go-Stimulus während Go-Studien: Das Tier hat die Hürde innerhalb von 6 Sekunden von Ton-Präsentation zu überqueren (richtige Antwort, getroffen). Bestrafen einen Fehl von einem milden Fuß-Schock von 50 bis 300 & mgr; A, über den Gitterboden des Shuttle - Box geliefert.
- Verwenden Sequenzen des fallenden Ton (8 - 4 kHz, CS-) als No-Go-Stimulus während No-Go-Studien: Das Tier im aktuellen Raum des Shuttle-Box während der 6 Sekunden von Tonpräsentation bleiben muss. Bestrafen einen Fehlalarm von einem milden Fuß-Schock von 50 bis 300 & mgr; A, über den Gitterboden des Shuttle-Box geliefert.
- Verwenden Sie intertrial Intervallen von 20 5 Sekunden.
- Führen Sie 30 Go-Studien und 30 No-Go-Versuche pro Sitzung in einer pseudo-randomisierten Reihenfolge, so dass man sichssion besteht aus 60 Studien und dauert etwa 25 min.
- Legen Sie die ausgebildete Tier wieder in seine Heimat Käfig in der Tierhaltung.
- Gehirnsezierung
- Euthanize das Tier in der gewünschten Zeitpunkt nach einer gewünschten Anzahl von Trainingseinheiten zervikale Dislokation (zB 24 h nach Abschluss der ersten Sitzung). Enthaupten das Tier.
- Sezieren schnell das Gehirn über die folgenden Schritte: Schneiden Sie zuerst die Haut und dann den Schädel mit geraden Schere entlang der Sutura sagittalis. vollständig zu entfernen, die Teile des Knochens, der das Hirngewebe mit starken Zange abdecken. Nehmen Sie das Gehirn mit einem Spatel aus.
- Für Dissektion, legen Gehirn auf eine Petrischale mit Eis gefüllt. Präparieren Sie die auditorischen Kortex, den frontalen Cortex, Striatum und Hippocampus unter einem Stereomikroskop mit einem Skalpell und Nadel.
- Localize den auditorischen Cortex mit visuellen Sehenswürdigkeiten auf das Gehirn sberfl wie Blutgefäße und der Form der Oberfläche (Bregma -2,06 bis -3,4, Größe rostrokaudalen 2 mm, dorsoventral 1.3 mm) und sezieren bilateral als Block rechteckigen Gewebe mit der Dicke des Kortex.
- Präparieren Sie die frontalen Kortex als Hirnschnitt zwischen Bregma 3,56 und 1,54 die Chiasma opticum als Meilenstein Verwendung findet und Gewebe von Bulbus olfactorius.
- Präparieren Sie die Striatum als Hirnschnitt zwischen Bregma 1,54 und 0,5 und sorgfältig Rindengewebe zu entfernen.
- Sezieren den Hippocampus durch das Gehirn mit der Nadel durch das Cerebellum Fixieren und die Hirnrinde Abwickeln an der Hinterhauptslappen beginnen.
- Shock-freeze seziert Gehirnproben in flüssigem Stickstoff und bei -80 ° C.
2. Herstellung von Synaptosomen oder alternativ eine postsynaptische Dichte (PSD) -angereicherten Fraction
HINWEIS: Bei allen Verfahren, halten Proben und Puffer bei 0-4 ° C.Puffer enthalten frisch Protease-Inhibitor-Cocktails verdünnt, um den proteolytischen Abbau von Proteinen zu verhindern. Wenn Proteinphosphorylierung ebenfalls untersucht wird, haben Phosphatase-Inhibitor-Cocktails zugegeben werden. Alle g-Werte angegeben werden als g (Durchschnitt) während des gesamten Protokoll gegeben.
- Herstellung eines rohen Membranfraktion (3A)
- Transfer seziert Hirngewebe in ein Gefäß Homogenisierung mit 1 ml eiskaltem A (5 mM HEPES, 320 mM Saccharose, pH 7,4) Puffer und Gewebe bei 900 Umdrehungen pro Minute mit 12 Schlägen homogenisieren.
- Zentrifugieren Sie die Proben bei 1000 × g für 10 min. Halten Sie die Überstände.
- Wieder homogenisieren Pellets bei den gleichen Bedingungen in dem gleichen Volumen Homogenisierungspuffer wie zuvor und Zentrifuge Proben wieder bei 1.000 xg für 10 min. Kombinieren entsprechenden Überstände. Entsorgen Sie die Pellets P1, die hauptsächlich Kerne und Zelltrümmer enthalten.
- Dreh die vereinigten Überstände für 20 Minuten bei 12.000 x g. Verwerfen supernanern oder Verwendung für weitere Fraktionierung 11.
- Resuspendieren Pellets in dem gleichen Volumen Homogenisierungspuffer nach wie vor den Homogenisator mit 6 Hüben bei 900 Upm und Schleudern bei 12.000 xg für 20 min verwendet wird. Verwerfen Stände. Die Pellets P2 die rohen Membranfraktionen darstellen.
- Reinigung von Synaptosomen aus rohen Hirnmembranfraktionen (3A)
HINWEIS: Crude Gehirnmembranfraktionen getrennt in Myelin werden kann, leichte Membranen, Synaptosomen und Mitochondrien Saccharose Dichte Schritt Ultrazentrifugation verwendet wird. Für diese 5 mM Tris / HCl pH 8,1 Puffer Sucrose bei entweder 0,32 M, 1,0 M oder 1,2 M Konzentration enthalten, sind erforderlich.- Während der Zentrifugation Durchführung der P2-Fraktionen zu erzeugen, Saccharose Stufengradienten in den Ultrazentrifugenröhrchen vorbereiten. Beginnen Sie mit 2,5 ml 1,0 M Saccharosepuffer und Unterschicht mit 1,5 ml 1,2 M Saccharose-Puffer mit einem Glas Pasteur Pipette.
- Re-homogenisieren P2-Fraktionenin 0,5 ml 0,32 M Saccharosepuffer manuell mit 6 Hübe und Last auf der Oberseite des Gradienten.
- Spin bei 85.000 × g für 2 Stunden in einer Ultrazentrifuge eine Schwingbecherrotor mit.
- Entsorgen Sie die oben 0,32 M Saccharose Schicht, die das Material an der Schnittstelle zu den 1,0 M Saccharosepuffer (Myelin, leichte Membranen). Sammeln Sie Synaptosomen im 1,0 / 1,2 M Saccharosepuffer-Schnittstelle. Das Pellet am Boden des Röhrchens enthält Mitochondrien.
- In 0,32 M Saccharosepuffer zur synaptosomaler Fraktion bei 1: 1-Verhältnis, sorgfältig zu mischen und Spin bei 150.000 g für 1 Std. Synaptosomen sind im Pellet und können nun in einer für die Weiterverarbeitung erforderlichen Puffer werden.
- Herstellung einer PSD-angereicherte Fraktion (3B)
- Homogenisieren jede spezifische Gehirnbereich von einem einzelnen Tier in 100 ul Extraktionspuffer (5 mM Tris / HCl pH 8,1, 0,5% Triton X-100) in einem 200 & mgr; l Ultrazentrifugenröhrchen mit einer PTFE (Polytetrafluorethylen) Pistillbei 2.000 Umdrehungen pro Minute mit 12 Schlägen.
- Je 100 & mgr; l Extraktionspuffer, mischen und für 1 Stunde bei 4 ° C inkubiert. Spin down bei 100.000 g für 1 Stunde und sammeln den Überstand S1 vorsichtig mit einem 200 ul-Pipette.
- Re-homogenisieren Pellet P1 im gleichen Röhrchen mit 100 ul Extraktionspuffer wieder mit einem PTFE Pistill bei 2.000 rpm mit 12 Schlägen.
- 100 l Extraktionspuffer und gut mischen mit einer Pipette und Spin bei 100.000 g für 1 Std.
- Kombinieren der Überstand S2 mit S1 zu der löslichen Proteinfraktion. Diese Fraktion enthält cytosolischen Proteine, 0,5% Triton X-100 löslichen Membranproteine und extrazelluläre Matrixmoleküle.
- Resuspendieren verbleibende Pellet in 50 & mgr; l 5 mM Tris / HCl pH 8.1. Diese Fraktion enthält PSDs, Detergenz-resistente Membranen, unlösliche Cytoskelettelemente, Mitochondrien und Zelltrümmer einschließlich Kerne. Es wird in PSDs angereichert, die den Kern der postsynaptischen Strukturen, sondern auch wichtige Teile der präsynaptischen bildencytomatrix an der aktiven Zone. Der Faktor für die Anreicherung von PSDs ist etwa 4, und die Anreicherung von PSD Komponenten zuvor gezeigt worden. 12
3. Probenvorbereitung für die Massenspektrometrie
- Lysis und Proben Normalisierung
HINWEIS: Proben Normalisierung hinsichtlich der Proteinkonzentration ist ein sehr wichtiger Schritt, um endlich verlässliche quantitative Daten auch bei schwachen synaptische Protein-Expression Veränderungen erzielen.- Löse Synaptosomen oder PSD angereicherte Präparate jedes Hirnareal eines Tieres in 20 bis 50 & mgr; l (abhängig von der Gesamtmenge des Materials: für auditorischen Kortex mit 5 bis 15 mg Gewebe Einsatz 20 ul) von 8 M Harnstoff und Inkubation auf Eis für 1 h im Ultraschallbad.
- Für In-Gel verdauen, lösen sich Synaptosomen direkt in der SDS-Probenpuffer. Sorgfältig berechnen die geladene Menge Überlastung des Gels zu vermeiden. Bedenken Sie, dass in diesem Fall das hohe reichlich Gerüst proteins während der Gelelektrophorese und in-Gel-digest verloren.
- Verdünnt mit 1% eines abnehmbaren Reinigungsmittel in einer Endkonzentration von 2 M Harnstoff zu gewährleisten. Vermeiden jede Temperatur höher als 30 ° C Protein Carbamylierung zu vermeiden.
- Führen SDS-PAGE mit einem Aliquot (beispielsweise 10 & mgr; l) der Probe gemß Standardverfahren 13,14.
- Fleck des Gels mit Coomassie-Blau Herstellerprotokoll nach. Das Verfahren kombiniert die Fixier- und Färbeschritt mit Methanol und Essigsäure.
- Bestimmung der optischen Dichte jeder Probe für die gesamte Spur mit einem kalibrierten Gelscanner im Transmissionsmodus und berechnet die relative Proteinmenge.
- Normalisieren der Proben dieser Berechnungen nach.
- Split jede Probe in zwei verschiedene Teile. Verwenden Sie ein Drittel für die in-Gel zu verdauen und zu zwei Dritteln für die in-Lösung verdauen.
- Löse Synaptosomen oder PSD angereicherte Präparate jedes Hirnareal eines Tieres in 20 bis 50 & mgr; l (abhängig von der Gesamtmenge des Materials: für auditorischen Kortex mit 5 bis 15 mg Gewebe Einsatz 20 ul) von 8 M Harnstoff und Inkubation auf Eis für 1 h im Ultraschallbad.
- In-Gel - Verdauung
- Gel Trennung
- Führen Sie eine zweite SDS-PAGE, um die eingestellten Konzentration Proben verwendet. Fleck und quantifizieren die Gele zum zweiten Mal die Normalisierung Qualität zu überprüfen.
- Schneiden Sie jede Spur einer Probe aus innerhalb des Gels in verschiedenen Bereichen (8 / Spur), aber das Molekulargewicht im Bereich oberhalb von 170 kDa auszuschließen. Übertragen Sie die Gelstücke in getrennte Röhren.
- Geschnitten, um die Bereiche in kleinere Stücke (ca.. 1 x 1 mm) mit einem scharfen Skalpell in-Gel-Verdauung Wirksamkeit zu erleichtern.
- Digest 15
- Waschen Sie die Gelstücke mehrfach (auf Färbungsintensität abhängig) während 10 min mit 50 bis 150 ul eines Puffers , bestehend aus 50% Acetonitril (ACN) und 50 mM Ammoniumhydrogencarbonat (NH 4 HCO 3).
- Entfernen Stände. Decken Sie die Gelstücke mit ACN und Inkubation bei 20 ° C bis Gelstücke weiß werden und schrumpfen.
- Entfernen Sie die ACN und rehydratisieren die Gelstücke für 5min mit 50 ul 0,1 M NH 4 HCO 3 das gleiche Volumen an ACN hinzufügen und für weitere 15 min bei 37 ° C inkubieren.
- Entfernen und entsorgen vollständig flüssig. Trocknen der Gelstücke in einer Vakuumzentrifuge.
- Gelstücke in 50 & mgr; l NH 4 HCO 3 , enthaltend 10 mM Dithiothreitol (DTT) und Wärme Proben für 45 min bei 56 ° C rehydriert Cysteinreste zu reduzieren.
- Entfernen Überstand und 50 & mgr; l NH 4 HCO 3 , die 55 mM Iodacetamid (IAA) für 30 min im Dunkeln reduzierten Cysteinen zu carbamidomethylate.
- Entfernen und entsorgen Sie alle Flüssigkeit über den Gelstücke und waschen Sie sie zweimal mit 50 & mgr; l NH 4 HCO 3 und ACN (1: 1) für 10 Minuten restliche IAA zu entfernen. Trockenen Proben in einer Vakuumzentrifuge.
- Für begrenzte Verdauung von Proteinen hinzufügen 25 mM NH 4 HCO 3 12,5 ng / ul Trypsin enthält. Das erforderliche Volumen ist abhängig von Größe und Menge des Gels pieces. Inkubieren für ein paar Minuten und prüfen Sie, ob der Puffer absorbiert wird. Fügen Sie mehr Puffer, wenn nötig, Gelstücke sollte vollständig abgedeckt werden. Inkubieren bei 37 ° C über Nacht (min. 12 h).
- Peptid - Extraktion
- Overlay Gelstücke mit 10 - 20 & mgr; l von 25 mM NH 4 HCO 3 und das gleiche Volumen an ACN hinzuzufügen. Inkubieren für 10 Minuten auf Eis mit Ultraschallbad. Danach entfernen und die Überstände zu sammeln, die die meisten der erzeugten Peptide enthalten.
- Füge 100 & mgr; l Extraktionspuffer, enthaltend 30% ACN / 0,1% Trifluoressigsäure (TFA) in die Gelstücke. Wiederholen Sie die Inkubation in einem Ultraschallbad und sorgfältig diese Überstand zu sammeln.
- Wiederholen Sie die letzten Extraktionsschritte durch die ACN-Konzentration auf 50% zu erhöhen. Nach 10 min Ultraschallbad Abschaltens und Stände sammeln.
- Kombinieren Sie alle drei entsprechenden Überstände der Extraktionsschritte und trocknen Sie sie in einer Vakuumzentrifuge. Beachten Sie, dassals Ergebnis der Geltrennung die 8 Bereiche pro Spur / Probe eine Probe wieder in diesem Schritt kombiniert werden.
- Gel Trennung
- In-Lösung verdauen
- Verdauen
- Verwenden , um die berechnete Menge (beispielsweise 100 & mgr; l einer 150 & mgr; l Lysat, hängt von der Menge des Materials und dem Volumen , die für die Resuspension einer Probe aus einem bestimmten Bereich des Gehirns) der normalisierten Abtastwerte , um eine ausreichende Ausgangsmaterial erhalten mindestens drei technische Replikate zu markierungsfreie Massenspektrometrie durchführen.
- In 2 mM DTT in 25 mM NH 4 HCO 3 und vorsichtig auf die Probe vortexen. Reduzieren Sie die Proben für 45 min bei 20 ° C.
- In 10 mM IAA die Cysteinreste carbamidomethylate. Mischen und Inkubation für 30 min im Dunkeln bei 20 ° C.
- Schließlich fügen 1 ul einer Trypsin-Stammlösung (1 ug / ul Trypsin in 25 mM Essigsäure) und für 12 Stunden bei 20 ° C inkubieren.
- Festphasenextraktion (SPE) -Purification
- die Säure spaltbare Detergens, stellen Proben auf eine Endkonzentration von 1% TFA und Inkubation für 1 h bei 20 ° C zu entfernen.
- Zentrifugieren Sie die Proben bei 16.000 × g für 10 min und sorgfältig sammeln Stände.
- Legen Sie die SPE-Säule in einem Rack und ins Gleichgewicht der Matrix mit 2 ml Methanol. Wasche zweimal mit 2 ml 0,1% TFA in Wasser (Puffer B).
- 2 ml Puffer B und der Probe laden. Waschen Sie noch dreimal.
- Eluieren der Peptide durch Zugabe von 200 ul 70% ACN / 0,1% TFA. Wiederholen Sie diesen Schritt.
- Pool beide Eluate und trocknen Sie sie in einer Vakuumzentrifuge nach unten.
- Verdauen
- Phospho-Peptid-Anreicherung von TiO 2 Chromatographie 16
- Auflösen Peptide durch in-Gel hergestellt oder in-Lösung verdauen in 150 & mgr; l von 80% ACN / 2,5% TFA (Puffer C) äquilibriert und ~ 2 mg der TiO 2 -Kugeln in 50ul Puffer C.
- Beads an die Probe und inkubiere in einer Drehvorrichtung für 1 h bei 20 ° C. Danach Spin Perlen nach unten (16.000 · g, 1 min) und die Überstände zu sammeln.
- Waschen Sie die Perlen dreimal mit 100 ul Puffer C durch vorsichtiges Mischen und Spinnen nach 5 min nach unten. Sammeln Sie Stände. Wiederholen Sie diesen Schritt dreimal mit 100 & mgr; l von 80% ACN / 0,1% TFA, gefolgt von drei Wäschen mit 100 ul 0,1% TFA (ohne ACN), respectively.
- Kombinieren Sie alle zehn Stände, trocknen Sie sie in einer Vakuumzentrifuge und behandeln sie als das Phospho-Peptid-abgereicherte Fraktion zur weiteren Reinigung gemäß Schritt 3.5.
- Eluieren der gebundenen Phospho-Peptide mit 20 ul 400 mM NH 4 OH / 30% ACN von den Kügelchen. Wiederholen Sie diesen Schritt dreimal und sammeln Sie alle Stände nach den Perlen Spinnen nach unten.
- Kombinieren Sie die Eluate der in-Gel verdauen und der in-Lösung verdauen einer Probe und behandeln sie als die phospho-peptide angereicherte Fraktion. Trocknen Sie sie in einer Vakuumzentrifuge zu einem Endvolumen von 4 bis 8 & mgr; l.
- Konzentriert und Entsalzung von Phospho-Peptid-verarmten Fraktionen , die durch Mikro-SPE
- Auflösen der getrockneten Peptide in 20 ul 0,1% TFA.
- Äquilibrieren der festen C 18 -Matrix von 20 ul Zeichnung der Spitze ist ACN zu. Waschen der Matrix mit 0,1% TFA in Wasser in die Spitze zu ziehen. Wiederholen Sie den Vorgang dreimal.
- Langsam angesäuerten Probe in die Spitze zu laden (Wiederholen Sie diesen Schritt dreimal).
- Waschen Sie die C 18 -Matrix dreimal mit je 20 & mgr; l 0,1% TFA in Wasser und entsorgen Sie die Waschlösung.
- Eluieren Peptide aus der Pipettenspitze wiederholt (3 mal) 20 & mgr; l von 70% ACN / 0,1% TFA Zeichnung und sammeln diese Elutionslösung in einem separaten Röhrchen.
- Kombinieren Sie die Eluate einer Probe und trocknen Sie sie in einer Vakuumzentrifuge.
4. Proteomanalyse
Hinweis: Die Proteomanalyse erfolgt auf einem Hybrid-Zweidruck linearen Ionenfalle / Orbitrap-Massenspektrometer, ausgestattet mit einer ultra HPLC. Die HPLC besteht aus einem gekühlten Autosampler mit einer 20 & mgr; l-Injektionsschleife, eine binäre Ladepumpe (ul Strömungsbereich), eine binäre nano Strömungsseparationspumpe, eine Säulenheizung mit zwei Mikroventilen und ein Entgaser Schalt. Proben werden zunächst einer Trapping - Säule (beispielsweise 100 um x 2 cm) bei einer Strömungsgeschwindigkeit von 7 durch Trennung auf einer Säule (beispielsweise 75 um x 25 cm) bei 250 nl / min , gefolgt ul / min unterzogen. Die Trennsäule Auslaß ist direkt gekoppelt an eine beschichtete Spitze Pico Emitter in einer Nanospray-Grenzfläche an der Massenspektrometers Ionisationsquelle positioniert.
- Nano-Flüssig - Chromatographie und Tandem - Massenspektrometrie
- Auflösen Peptidproben in 12 ul 2% ACN / 0,1% TFA für mindestens 30 min. Spin down für 15 Sekunden und Transfer 11 ul Überstand Autosamplervials (konisch, reduziert diameter).
- Richten Sie eine automatische Regelung für die Beispielanwendung, chromatographische Trennung und Tandem - Massenspektrometrie bei Controlling - Software (zB Xcalibur) wie folgt.
- Verwenden Sie die folgenden für Temperatur: Autosampler: 5 ° C; Säulenofen: 45 ° C.
- Verwenden Sie die folgenden für die Injektion: Volumen: 10 & mgr; l; Fließgeschwindigkeit: 7 & mgr; l / min (2% ACN, 0,1% TFA); Zeit: 8 min; Ventileinstellung: Trap-Säule - Abfall; Massenspek Erwerb: off.
- Verwenden Sie die folgenden für die Trennung: Fließgeschwindigkeit: 250 nl / min Ventileinstellung: Trap-Säule-Trennsäule; Massenspek Erwerb: on.
0 min - 100 min: 2% ACN, 0,1% Ameisensäure - 40% ACN, 0,1% Ameisensäure
100 min - 105 min: 40% ACN, 0,1% Ameisensäure - 95% ACN, 0,1% Ameisensäure
105 min - 109 min: 95% ACN, 0,1% Ameisensäure
109 min - 120 min: 2% ACN, 0,1% Ameisensäure - Verwenden Sie die folgenden für die Massenspektrometrie Einstellungen: Volle MS: FTMS; Auflösung 60.000; m / z-Bereich 400 - 2000; FRAU/MS: Linear Iontrap; Mindestsignalschwelle 500; Isolationsbreite 2 Da; dynamische Ausschlusszeit 30 Sekunden einstellen; einfach geladene Ionen werden von der Auswahl ausgeschlossen; normalisierten Kollisionsenergie wird auf 35% festgelegt, und Aktivierungszeit wurde auf 10 ms.
HINWEIS: Eine vollständige MS Scan bis zu 15 LTQ MS / MS Läufe mit kollisionsinduzierte Dissoziation (CID) der am reichlichsten detektiert Peptidionen folgt.
- Führen Sie drei technische Replikate für alle Proben.
- Proteinidentifizierung und markierungsfreie Quantifizierung
- Prozess massenspektrometrischen Rohdaten auf Proteinidentifikation und markierungsfreie Quantifizierung eine kommerzielle Software - Suite nutzen (zB SPITZEN Studio). Im Gegensatz zu den meisten anderen Proteom - Software - Pakete diese spezielle Software verwendet eine De - novo - -sequencing Algorithmus vor der Protein - Datenbank Ausrichtungen. Allerdings kann dieser Schritt leicht durch andere populäre Softwarepakete ersetzt werden.
- Verwenden essential Einstellungen in Tabelle 2 aufgeführt.
- Phospho-Proteomik
HINWEIS: Effiziente und zuverlässige Phospho-Peptid Akquisition erfordert einige wesentliche Änderungen des Proteom-Workflow-Setup.- Nach Phospho-Peptid-Anreicherung, nie trocken Proben vollständig. Halten Sie immer Proben gelöst.
HINWEIS: Der Phospho-Esterbindung von phosphoryliert Threonine oder Serine ist sehr zerbrechlich. Während kollisionsinduzierte Fragmentierung innerhalb der Ionenfalle führt dies zu einem Neutralverlust von Phosphat. Dies verhindert jede weitere Fragmentierung des Peptids, das wiederum für die Identifizierung erforderlich. Zulässige Breitband-Aktivierung in der Massenspektrometrie Aufbau ermöglicht die Fragmentierung von Phospho-Peptide auch nach einem neutralen Verlust der Phosphatgruppe. Es führt eine Zeit "Pseudo-MS 3" zu speichern. Phospho-Ort-Bestimmung in MS / MS-Daten erfordert eine besondere Prüfung und Bewertung und kann von Phosphore 3 durchgeführt werden.0.
- Nach Phospho-Peptid-Anreicherung, nie trocken Proben vollständig. Halten Sie immer Proben gelöst.
5. Bioinformatik - Meta-Analyse
HINWEIS: Vor dem Funktionsannotation und Netzwerk-Analyse durchgeführt, die Proteinlisten vorverarbeitet werden müssen. Zunächst verschmelzen die Listen der regulierten Proteine und Phospho-Peptide für jede Gehirnregion getrennt. Dann entfernen Sie alle für jede Fraktion UniProt-IDs duplizieren um Fehlinterpretationen zu vermeiden.
- Singuläre Anreicherung Analyse mit GeneCodis 17
- Öffnen Sie das webbasierte Tool von GeneCodis (http://genecodis.cnb.csic.es)
- Wählen Sie "Mus musculus" als Organismus und "GO Biologisches Verfahren" als Anmerkung.
- Fügen Sie eine Liste von UniProt-IDs von einer bestimmten Fraktion. Senden und warten, bis die Analyse durchgeführt wird. Klicken Sie auf "Singuläre Enrichment Analyse von GO Biologisches Verfahren" und die Ergebnisse anzuzeigen.
- Wiederholen Sie Schritt 5.1.3 für die anderen drei Fraktionen.
- Um zu sehen, keine Doppelungen und Überschneidungen zwischen den Ergebnislistenverwenden, um eine Skriptsprache wie Perl oder Python zum Filtern der Daten benötigt wird. Ähnliche Werkzeuge für eine einzigartige Bereicherung Analyse sind DAVID (https://david.ncifcrf.gov/) und Cytoscape (http://www.cytoscape.org/) mit dem Plug-Ins-BINGO (http://apps.cytoscape.org/ apps / Bingo) und ClueGO (http://apps.cytoscape.org/apps/cluego).
- Erzeugen einer Kraft basierend Graph aus GeneCodis Daten mit Gephi (https://gephi.org/)
HINWEIS: Die Daten für die Graphen muss vom Benutzer zur Verfügung gestellt werden, entweder in einer Grafik-Format (.gexf, .graphml, .dot, .gv, .gml) oder von Hand eingegeben.- Generieren der Graphknoten
- Mit der Hand: Open Gephi und klicken Sie auf "Data Laboratory". Erstellen Sie Knoten. Klicken Sie auf "Knoten" auf die auf den "Knoten" Tabelle zu wechseln links. Klicken Sie auf "Hinzufügen Knoten". Geben Sie den Namen des Wortes. Klicken Sie auf "OK" / Drücken Sie die Eingabetaste.
- Alternative: Speichern GeneCodis führen zu PC. Öffnen Sie die TXT mit einem Tabellenkalkulationsprogramm. Löschen Sie alle Zeilen except von "Item_Details" (Begriff Namen). Ändern header "Item_Details" zu "Label". Tabelle speichern als ".csv". Jetzt in Gephi, klicken Sie auf "Import-Tabelle". Wählen Sie Tabelle aus dem Datei-Browser von Gephi. Klicken Sie auf "Weiter". Klicken Sie auf "Fertig stellen".
- Verbindungsknoten über Kanten.
- Klicken Sie auf "Edges" auf der linken Seite auf die "Kanten" Tabelle zu wechseln. Für jeden Knoten (Term): nachschlagen Gen Namen in anderen Bedingungen. Wenn ein oder mehrere Gene gemeinsam genutzt werden -> erstellen Rand.
- Klicken Sie auf "Hinzufügen Edge". Wählen Sie "Undirected". Wählen Sie Quell- und Zielknoten aus Dropdown-Listen. Klicken Sie auf "OK" / Drücken Sie die Eingabetaste. Wenn mehr als ein Gen freigegeben ist, geben reichlich in "Gewicht" (Tabelle).
- Force-basierte grafische Layout.
- Open Graph-Datendatei, den Diagrammtyp auf "ungerichtet" oder die Daten verwenden, wie von Hand eingegeben haben, klicken Sie auf "Übersicht", wenn nicht bereits selected.
- Resize Knoten in Abhängigkeit von der Fülle von Verbindungen. Klicken Sie auf Statistiken, laufen entweder "Average Degree" (ungewichtet Kanten) oder "Avg. Die gewichteten Degree" (gewichtete Kanten) unter "Netzwerkübersicht". In "Erscheinungsbild", klicken Sie auf "Knoten", dann auf die Größe Knopf neben wählen Sie "Eigenschaften" und die Attribute Parameter auf "Avg. Die gewichteten Degree" oder "Average Degree". Klicken Sie auf Anwenden.
- Schließlich: Wählen Sie "Force-Atlas" in "Layout" und führen; ändern "Repulsion Stärke", wenn Knoten kollidieren.
- Export in Bild.
- Screenshot-Funktion: Klicken Sie auf "Übersicht", Änderung Graph Layout, Randdicke, Etikettengröße und Skalierung mit dem Menü am unteren Rand des "Graph" -Fenster. Klicken Sie auf die Kamera nach links-Taste und Bild speichern.
- Export-Funktion von "Preview": Klicken Sie auf "Vorschau". Ändern Sie Presets auf "Default gerade". Die Umgebung wechselns entsprechend der gewählten Einstellungen und klicken Sie auf "SVG / PDF / PNG" zu exportieren.
- Generieren der Graphknoten
Ergebnisse
Abbildung 1 fasst den kompletten Workflow von quantitativen synaptischen Proteom Profilierung der Maus Hirnregionen nach Gehör Diskriminierung Lernen. Es beginnt mit dem Tiertraining in einem Shuttle-Box. In dem Beispiel in Abbildung 2 gezeigt, begann Mäuse signifikant FM Ton Diskriminierung im 4. Training zu zeigen, effizientes Lernen anzeigt. Die Tiere werden zu ausgewählten Zeitpunkten für Gehirnbereich Dissektion geopfert. Die erforderliche Anreicherung von Synapsen kann entweder durch die Herstellung von Synaptosomen oder alternativ durch Herstellung eines PSD-angereicherte Fraktion erreicht werden, sowohl im einzelnen in Figur 3 beschrieben , die PSD-Anreicherungsmethode wurde für geringe Gewebemengen, beispielsweise 1 entwickelt worden - 2 hippocampal slices aus Rattenhirn 12, 18. Es erfordert kleine Rohre, PTFE-Pistill passend zu diesen Rohren und ein Labor Bohrantrieb für den Stößel antreibt.
Aufgrund der besonderen Proteinzusammensetzung von Synaptosomen, ist es nur sehr empfehlen die Probenvorbereitung in zwei verschiedenen, aber komplementären Weise durchzuführen. Scaffolds der PSDs sind oft sehr hochmolekulare Proteine in hoher Stöchiometrie auftreten. In-Lösung verdauen ist der beste Weg, sie effizient zu extrahieren, sondern zu einem Überabtastungs des erzeugten Peptidmischung führen kann. Die in-Gel-Verdau von derselben Probe in parallel ausgeführt werden, können diese hochmolekularen Proteine auszuschließen und die Analyse von Proteinen mit mittlerer und niedrigerem Molekulargewicht begünstigen. Für eine umfassende Analyse werden beide Arten von proteolytischen Verdaus empfohlen.
Die unterschiedlichen Mengen von Gewebe der Hirnareale untersucht erfordern eine Anpassung des aufgetragenen Materials zum besseren Vergleich. Innerhalb der vier untersuchten Hirnarealen ist die auditorischen Kortex im Allgemeinen die Begrenzung der Tatoder. Das Material aller anderen Gehirnbereichen sollten sorgfältig auf die Menge des auditorischen Cortex nach der Herstellung von Synaptosomen oder PSD-angereicherten Fraktionen eingestellt werden (siehe 3.1.1.). Typische Gewichte von frisch hergestelltem Hirnarealen von Mäusen sind, wie folgend: auditorischen Cortex (AC): ~ 50 mg; Hippocampus (HIP): ~ 90 mg; Striatum (STR): ~ 120 mg und frontalen Kortex (FC): ~ 100 mg.
Die PSD-Anreicherungsverfahren in Abschnitt 2.3 beschriebenen erlaubte die Identifizierung von ca. 1500 verschiedene Proteine und etwa 250 verschiedene Phospho-Peptide pro Hirnregion auf der Ebene eines einzelnen Tier (Tabelle 1). Proteomanalyse 24 h nach der ersten Trainingseinheit ergab , dass 7,3% der identifizierten Proteine und 5,8% der Phospho-Peptide zeigten eine signifikante (p <0,05) quantitative Veränderungen in ihrer synaptischen Expression im Vergleich zu naiven Kontrollen (Tabelle 1). Eine auffällige Tendenz nach unten regulation der synaptischen Gerüste zu einer ausgeprägten Umlagerung der synaptischen Architektur während der frühen Stadien der FMTD Lernen hinweisen. Die überwiegende Mehrheit der regulierten Proteine wurden in einer Hirnregion-spezifische Weise verändert, wohingegen nur 22% gefunden wurden, in zwei oder mehr Hirnregionen zu regeln. Sechs ausgewählten Beispiele sind in Abbildung 4 dargestellt.
Meta-Analyse der komplexen Ergebnisse von IPA liefert den Beweis für die besondere Beteiligung / Manipulation der folgenden kanonische Wege: "Clathrin-vermittelte Endozytose Signaling", "Axonal Guidance Signaling", "Calcium-Signale", "RhoA Signaling", "Notch-Signal "," Remodeling von Epithelial Adhärenzverbindungen "," Glutamate Receptor Signaling "," GABA-Rezeptor-Signal "," Dopamine Receptor Signaling "und" Synaptische Langzeitpotenzierung ".
Single Anreicherungsanalyse ergab signifikante biologische Prozesse überrepräsentiert im frontalen Kortex in Bezug auf den Proteintransport, Zelladhäsion, Phosphorylierung, Endozytose, Vesikel-vermittelten Transport, forebrain Entwicklung und axonogenesis (Abbildung 5). In den auditorischen Kortex Prozesse biologischen einschließlich Ionentransport, Übersetzung, mRNA Transport, Proteintransport und Lernen waren spürbar. Die Analyse der Proteinfraktion des Hippocampus detektiert wesentlich angereicherten Prozesse im Zusammenhang mit Ionentransport, Zellzyklus, Translation, Phosphorylierung und Entwicklung des Nervensystems. Im Striatum, überrepräsentiert biologische Prozesse einschließlich mRNA Transport, Vesikel-vermittelten Transport, axonogenesis, Proteolyse, Proteintransport und der Endozytose gefunden wurden.
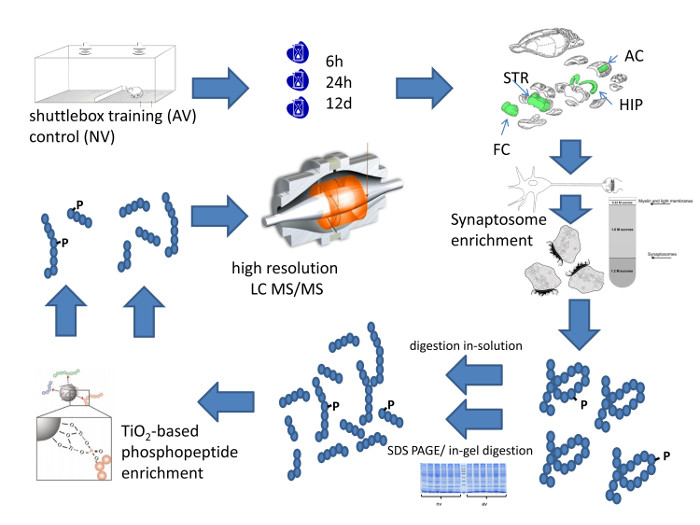
Abbildung 1: Systematische WorkFlow des methodischen Ansatzes. Diese Zahl fasst schematisch den Arbeitsablauf von hochauflösenden quantitative Profilierung von Gehirnbereich spezifischen synaptischen Protein-Zusammensetzung. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.

Abbildung 2: Beispiel für die Leistung der Mäuse in der FM - Ton - Diskriminierung Aufgabe. Tiere zeigen eine steigende Rate von Treffern (blaue Kurve) und eine abnehmende Rate von Fehlalarmen (schwarze Kurve) im Laufe des Trainings. Erhebliche Diskriminierung tritt aus der vierten Sitzung. Die Fehlerbalken sind als SEM zur Verfügung gestellt. Bitte klicken Sie hier , um eine lar anzuzeigenger-Version dieser Figur.

Abbildung 3: Herstellung der Synaptosomen und dem PSD-angereicherte Fraktion. A: Synaptosomen Vorbereitung. B: PSD-angereicherte Fraktion Zubereitung. Beide Figuren erläutern die detaillierten Workflow zur Herstellung von Synaptosomen oder alternativ PSD-angereicherten Fraktionen von Hirngewebe. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
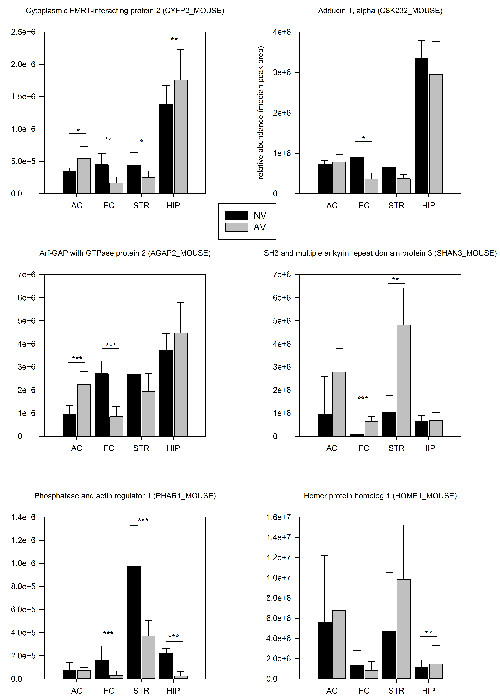
Abbildung 4: Ausgewählte Quantitative Proteomik Ergebnisse. Die relativen synaptischen Häufigkeiten ausgewählter Proteine werden zwischen Mäusen tra verglichenined auf der FMTD Aufgabe (AV, n = 6) und naiven Kontrollmäuse (NV, n = 6), 24 Stunden nach der ersten Trainingseinheit. Die Fülle Werte wurden als Mittelwert der Peakflächen der drei intensivsten Peptide eines Proteins berechnet. Die Proteine mit signifikanten Änderungen Fülle (AV / NV; t-Test) werden innerhalb der Parzellen gekennzeichnet: * p <0,05, ** p <0,01, *** p <0,005. Die Fehlerbalken sind als SD zur Verfügung gestellt. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.

Abbildung 5: Visualisierung der biologischen Pathways für Frontal Cortex von GeneCodis / Gephi. Nur wesentliche Bestimmungen der Gene Ontology (GO) Datenbank (http://geneontology.org) über "Biologische Verfahren" mit einer minimalen Anzahl von Protein drei werden hier gezeigt. Knoten stellen GO Begriffe, die Größe des Knotens aus, die Linienbreite und die Anzahl von Verbindungen von einem bestimmten Knoten zeigen die Anzahl von Proteinen, die diese GO Begriff mit anderen Knoten teilen. Aufgrund der "Force-Atlas" Methode der Gephi werden Clustering verwandten Knoten eng zusammen. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
| Gehirn - Region | AC | FC | HÜFTE | 000 "face =" Calibri "size =" 3 "> STR | Σ |
| identifizierten Proteine | 1435 | 1758 | 1572 | 1507 | 6272 |
| regulierte Proteine (p <0,05) | 59 | 130 | 162 | 108 | face = "Calibri" size = "3"> 459 |
| ↑ AV / NV | 8 | 4 | 76 | 35 | 123 |
| ↓ AV / NV | 51 | 126 | 86 | 73 | 336 |
| identifiziert phosphomoti fs | 197 | 361 | 273 | 278 | 1109 |
| geregelten phosphomotifs (p <0,05) | 8 | 22 | 21 | 14 | 65 |
| ↑ AV / NV | 4 | 00000 "face =" Calibri "size =" 3 "> 17 | 5 | 9 | 35 |
| ↓ AV / NV | 4 | 5 | 16 | 5 | 30 |
Tabelle 1: Zusammenfassung der Proteom - Ergebnis. In dieser Tabelle sind eine repräsentative Proteom-Experiment von ausgebildeten Mäuse (AV, n = 6) 24 Stunden nach der ersten Trainingseinheit im Vergleich zu ihren naiven Kontrollen (NV, n = 6). DasSumme von 459 regulierten Proteine enthält überlappende Vorschriften. 283 unterschiedliche Regelungen wurden als Gehirn spezifisch bestimmt. Im Einzelnen 57 Proteine reguliert werden in zwei Hirnregionen, 18 Protein Vorschriften wurden in drei Gehirnregionen erkannt und nur 2-Proteine sind in allen vier untersuchten Gehirnbereichen geregelt.
| Fehlertoleranzen | |
| Vorläufermasse (Fourier-Transformation-Massenspektrometrie) | 10 ppm |
| Fragmentionenmasse (linearen Ionenfalle) | 0,6 Da |
| Maximale verpassten cleavages pro Peptid | 3 |
| Feste Änderungen | |
| für in-Gel-verdauten Proben | Carbamidomethylierung von Cysteine |
| für in-Lösung-verdauten Proben | Methylthiolation von Cysteine |
| Variable Änderungen | Die Oxidation von Methionin |
| Deamidierungen von Asparagin und / oder Glutamin | |
| Datenbank | Uniprot / Sprot |
| Taxonomy | Maus |
| Statistische Identifikationsannahme Einstellungen | |
| de novo durchschnittliche örtliche Vertrauen (ALC) | > 50% |
| Peptid-False Discovery Rate (FDR, bezogen auf est. Köder-Fusion) | <1% |
| Protein Signifikanz (-10logP, auf Basis von modifizierten T-Test) | > 20 |
| einzigartige Peptide / Protein | ≥ 1 |
| Die Quantifizierung Einstellungen: | |
| Peptide für die Quantifizierung verwendet, wenn: | |
| Peptid Bedeutung (-10logP) | > 30 |
| Peptid-Identifikation in | ≥ 50% der Proben |
| Peptidsignalqualität | > 1 |
| Peptid durchschnittliche Fläche | > 1E5 |
| Peptid-Retentionszeit Toleranz | <5 min |
| Normalisierung | von Gesamtionenstrom (TIC) |
Tabelle 2: Einstellungen für die Proteinidentifikation (Schritt 4.2.2).
Diskussion
Die Studie stellt einen methodischen Workflow für eine genaue quantitative Profilierung der synaptischen Protein-Expression Änderungen optimiert während des Lernens und der Gedächtniskonsolidierung in verschiedenen Hirnarealen von Mäusen. Das Setup bietet die Möglichkeit, die Protein-Expression auf der Ebene eines einzelnen Tieres zu studieren trotz der erforderlichen Anwendung von mindestens drei technische Replikate pro Probe für die massenspektrometrische Analyse.
Die Methodik berücksichtigt die speziellen Proteinzusammensetzung der Prä- und Postsynapse bestehend aus hochmolekularen Gerüstproteine aber auch Proteine wichtiger Mediator Medium oder niedrigere Molekulargewichte tragen. Die in-solution Verdaus von synaptosomalen Zubereitungen ergeben eine effiziente Erzeugung und damit eine Überrepräsentation von Gerüst abgeleitete Peptide. Dies wiederum kann die Analyse kleinerer oder weniger reichlich Proteine unterdrücken. Die vorgeschlagene Herstellung von SDS-PAGE-Fraktionen von einerAliquot jeder mit einem in-Gel-Aufschlussverfahren parallel kombiniert Probe erleichtert die Analyse von mittleren und niedrigen Fülle Proteine und stellt eine hoch ergänzende Methode zu empfehlen. Nach separate Anwendung massenspektrometrischer aller Fraktionen , die von einer Probe abgeleitet (zB in-Lösung zu verdauen, in-Gel verdauen, vereinigten Fraktionen phospho angereichert) können die entsprechenden MS / MS - Datensätzen kombiniert werden und weiter für die Proteinidentifikation und Quantifizierung von SPITZEN berechnet Software oder alternative beliebte Software-Pakete.
Alternativ kann die individuelle Anwendung von in-Gel-Digestion-abgeleitete Fraktionen einer Probe (separat verarbeitet Gel-Bereiche einer Probe lane) und Fraktionen der in-Lösung digeriert Probe erzeugt (zB durch Ionenaustauschchromatographie) Massenspektrometrie kann zunehmen die analytische Tiefe. Dies erhöht jedoch erweiterten Workflow dramatisch die erforderliche Zeit für die LS-MS / MS-Datenerfassung. Für genen einer detaillierten molekularen Sequenz von synaptischen Protein Umlagerungen während der Lern- und Gedächtnisbildung einer spezifizierten Zeitverlauf der Proteom-Profilierung ist nicht erforderlich. Dieses Mal kann natürlich unmittelbar nach oder sogar während der ersten Trainingssitzung starten und deckt ein engmaschiges Zeitrahmen, bis die Tiere "Leistung die asymptotische Niveau der Lernkurve nach ca. erreicht. 8 - 10 Tage Training (Abbildung 2 für Details).
Die Analyse der Phosphorylierung Veränderungen der synaptischen Proteinen erfordert einen besonderen Fokus auf den ausgewählten Zeitrahmen während FMTD Lernen. Zum einen Signalkaskaden synaptischen Protein Umlagerungen bekannt Einleitung von Proteinphosphorylierungen und dephosphorylations ausgelöst werden sollen, in sehr frühen Stadien der Tiertraining erwartet. Auf der anderen Seite gibt es langfristiger Änderungen mehrerer phosphorylierten synaptischen Proteinen bekannt, die die Verbindung und Montage innerhalb der s regulierenynaptic Architektur 19, 20. Diese posttranslationale Modifikationen sind auch zu späteren Zeitpunkten der Gedächtniskonsolidierung erwartet.
Die komplexen Datensätze dieses Proteom Workflow generiert erfordern bioinformatischen Verarbeitung molekularen Wege und Schlüsselmoleküle zu identifizieren, teilnehmen. Meta-Analyse zeigt signifikante repräsentierter Wege, die eine Rolle bei Lern- und Gedächtnisprozessen spielen.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
We wish to thank Yvonne Ducho and Kathrin Pohlmann for excellent technical assistance. This work was supported by the Deutsche Forschungsgemeinschaft (SFB 779) and by the State Saxony-Anhalt / European Regional Development Fund (ERDF) via the Center for Behavioral Brain Sciences (CBBS).
Materialien
| Name | Company | Catalog Number | Comments |
| 3 M Empore Solid Phase Extraction- Filter | 3M Bioanalytical Technologies | 4245SD | 7 mm/3 ml |
| Acclaim PepMap 100 | Dionex/Thermo Scientific | 164564 | 100 µm x 2 cm, C18 |
| Acclaim PepMap 100 | Dionex/Thermo Scientific | 164569 | 75 µm x 25 cm, C18 |
| Acetic acid | Carl Roth GmbH | 3738.1 | |
| Acetonitrile (ACN) | Carl Roth GmbH | AE70.2 | |
| Acrylamide (30%) | AppliChem | A0951 | |
| Ammonium hydrogen carbonate | Fluka | 9830 | |
| Ammonium hydroxide | Fluka | 44273 | |
| Ammonium persulfate (APS) | AppliChem | A2941 | |
| Biofuge pico | Heraeus GmbH | 75003280 | |
| Blue R-250 | SERVA Electrophoresis GmbH | 17525 | |
| Bromophenol Blue | Pharmacia Biotech | 17132901 | |
| C57BL/6J mice | Charles River | ||
| Cantharidin | Carl Roth GmbH | 3322.1 | |
| Centrifuge tubes for MLS-50 | Beckman Coulter | 344057 | |
| Centrifuge tubes for TLA 100.1 rotor | Beckman Coulter | 343776 | |
| Dithiothreitol (DTT) | AppliChem | A1101 | |
| Eppendorf 5417R centrifuge | VWR | 22636138 | |
| Eppendorf A-8-11 rotor | VWR | 5407000317 | |
| Formic acid | Fluka | 14265 | |
| GeneCodis | http://genecodis.cnb.csic.es/ | ||
| Gephi | https://gephi.org/ | ||
| Glycerol | AppliChem | A1123 | |
| Glycine | AppliChem | A1067 | |
| HALT Phosphatase Inhibitor Cocktail | Pierce /Thermo Scientific | 78420 | |
| HEPES Buffer solution | PAA Laboratories GmbH | S11-001 | |
| Homogenization vessel 2 ml | Sartorius AG | 854 2252 | |
| Hydrochloric acid | Sigma-Aldrich | H1758 | |
| Imidazole | Sigma-Aldrich | I2399 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide (IAA) | Sigma-Aldrich | I1149 | |
| Laboratory drilling drive K-ControlTLC 4957 | Kaltenbach & Vogt GmbH | 182997 | |
| LTQ Tune Plus 2.7.0.1112 SP2 | Thermo Scientific | ||
| LTQ Orbitrap Velos Pro | Thermo Scientific | ||
| Macs-mix tube rotator | Miltenyi Biotech | 130-090-753 | |
| Magic Scan 4.71 | UMAX | ||
| Methanol | Carl Roth GmbH | AE71.2 | |
| MLS-50 rotor | Beckman Coulter | 367280 | |
| Optima MAX Ultracentrifuge | Beckman Coulter | 364300 | |
| PageRuler Prestained Protein Ladder | Thermo Scientific | 26616 | |
| PEAKS 7.5 | Bioinformatic Solutions | ||
| Phosphatase Inhibitor Cocktail 3 | Sigma-Aldrich | P0044 | |
| PhosphoRS 3.1 | IMP/IMBA/GMI | ||
| PhosSTOP | Roche | 4906845001 | |
| Plunger/pestle made of PTFE | Sartorius AG | 854 2651 | |
| PotterS homogenizer | Sartorius AG | 853 3024 | |
| Protease Inhibitor complete mini | Roche | 4693159001 | |
| Quantity One 4.5.1 | BioRad | ||
| RapiGest | Waters | 186002122 | |
| Shuttle box | Coulbourne Instruments | ||
| Sodium dodecylsulfate (SDS) | AppliChem | A1112 | |
| Sodium molybdate | Carl Roth GmbH | 274.2 | |
| Sodium tartrate dihydrate | Sigma-Aldrich | 228729 | |
| SONOREX RK 156 Ultrasonic Bath | BANDELIN electronic GmbH & Co. KG | 305 | |
| Soundproof chamber | Industrial Acoustics Company | ||
| Sucrose | Carl Roth GmbH | 4621.2 | |
| Tetramethyl ethylene -1,2-diamine (TEMED) | Sigma-Aldrich | T9281 | |
| Thermomixer basic | CallMedia | 111000 | |
| Titansphere TiO 5µm | GL Sciences Inc. Japan | 502075000 | |
| TLA 100.1 rotor | Beckman Coulter | 343840 | |
| Trifluoro acetic acid (TFA) | Sigma-Aldrich | T6508 | |
| Tris ( hydroxymethyl) aminomethane (TRIS) | AppliChem | A1086 | |
| Triton X-100 | Sigma-Aldrich | T8532 | |
| Trypsin Gold | Promega | V5280 | |
| Ultimate 3000 Ultra HPLC | Dionex/Thermo Scientific | ||
| Ultracentrifuge tube | Beckman Coulter | 343776 | |
| Unijet II Refrigerated Aspirator | Uniequip Laborgeräte- und Vertriebs GmbH | ||
| UNIVAPO 100 H Concentrator Centrifuge | Uniequip Laborgeräte- und Vertriebs GmbH | ||
| Urea | AppliChem | A1049 | |
| Water (high quality purifed) | Resistivity: > 18.2 MΩ*cm at 25 °C Pyrogens: < 0.02 EU/ml TOC: < 10 ppb | ||
| Xcalibur 3.0.63 | Thermo Scientific | ||
| ZipTipC18 Pipette Tips | MILLIPORE | ZTC18S960 |
Referenzen
- Lamprecht, R., LeDoux, J. Structural plasticity and memory. Nat Rev Neurosci. 5 (1), 45-54 (2004).
- Bingol, B., Schuman, E. M. Synaptic protein degradation by the ubiquitin proteasome system. Curr Opin Neurobiol. 15 (5), 536-541 (2005).
- Richter, J. D., Klann, E. Making synaptic plasticity and memory last: mechanisms of translational regulation. Genes Dev. 23 (1), 1-11 (2009).
- Rosenberg, T., et al. The roles of protein expression in synaptic plasticity and memory consolidation. Front Mol Neurosci. 7, 86 (2014).
- Scheich, H., et al. Behavioral semantics of learning and crossmodal processing in auditory cortex: the semantic processor concept. Hear Res. 271 (1-2), 3-15 (2011).
- Kähne, T., et al. Synaptic proteome changes in mouse brain regions upon auditory discrimination learning. Proteomics. 12 (15-16), 2433-2444 (2012).
- Reichenbach, N., et al. Differential effects of dopamine signalling on long-term memory formation and consolidation in rodent brain. Proteome Sci. 13, 13 (2015).
- Kähne, T., et al. Proteome rearrangements after auditory learning: high-resolution profiling of synapse-enriched protein fractions from mouse brain. J Neurochem. , (2016).
- Li, K., et al. Organelle proteomics of rat synaptic proteins: correlation-profiling by isotope-coded affinity tagging in conjunction with liquid chromatography-tandem mass spectrometry to reveal post-synaptic density specific proteins. J Proteome Res. 4 (3), 725-733 (2005).
- Carlin, R. K., Grab, D. J., Cohen, R. S., Siekevitz, P. Isolation and characterization of postsynaptic densities from various brain regions: enrichment of different types of postsynaptic densities. J Cell Biol. 86 (3), 831-845 (1980).
- Smalla, K. H., Klemmer, P., Wyneken, U., Dermietzel, R. . The Cytoskeleton - Imaging, Isolation, and Interaction. , 265-282 (2012).
- Smalla, K. H., et al. The synaptic glycoprotein neuroplastin is involved in long-term potentiation at hippocampal CA1 synapses. Proc Natl Acad Sci U S A. 97 (8), 4327-4332 (2000).
- Shapiro, A. L., Vinuela, E., Maizel, J. V. Molecular weight estimation of polypeptide chains by electrophoresis in SDS-polyacrylamide gels. Biochem Biophys Res Commun. 28 (5), 815-820 (1967).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Shevchenko, A., Wilm, M., Vorm, O., Mann, M. Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. Anal Chem. 68 (5), 850-858 (1996).
- Thingholm, T. E., Larsen, M. R., Ingrell, C. R., Kassem, M., Jensen, O. N. TiO(2)-based phosphoproteomic analysis of the plasma membrane and the effects of phosphatase inhibitor treatment. J Proteome Res. 7 (8), 3304-3313 (2008).
- Carmona-Saez, P., Chagoyen, M., Tirado, F., Carazo, J. M., Pascual-Montano, A. GENECODIS: a web-based tool for finding significant concurrent annotations in gene lists. Genome Biol. 8 (1), 3 (2007).
- Bonn, S., Seeburg, P. H., Schwarz, M. K. Combinatorial expression of alpha- and gamma-protocadherins alters their presenilin-dependent processing. Mol Cell Biol. 27 (11), 4121-4132 (2007).
- Tweedie-Cullen, R. Y., Reck, J. M., Mansuy, I. M. Comprehensive mapping of post-translational modifications on synaptic, nuclear, and histone proteins in the adult mouse brain. J Proteome Res. 8 (11), 4966-4982 (2009).
- Li, L., et al. The differential hippocampal phosphoproteome of Apodemus sylvaticus paralleling spatial memory retrieval in the Barnes maze. Behav Brain Res. 264, 126-134 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten