Method Article
청각 차별 학습 한 후 마우스 뇌 영역의 높은 해상도 양적 시냅틱 프로테옴 프로파일
요약
The identification of molecules and pathways controlling synaptic plasticity and memory is still a major challenge in neuroscience. Here, a workflow is described addressing the relative quantification of synaptic proteins supposedly involved in the molecular reorganization of synapses during learning and memory consolidation in an auditory learning paradigm.
초록
The molecular synaptic mechanisms underlying auditory learning and memory remain largely unknown. Here, the workflow of a proteomic study on auditory discrimination learning in mice is described. In this learning paradigm, mice are trained in a shuttle box Go/NoGo-task to discriminate between rising and falling frequency-modulated tones in order to avoid a mild electric foot-shock. The protocol involves the enrichment of synaptosomes from four brain areas, namely the auditory cortex, frontal cortex, hippocampus, and striatum, at different stages of training. Synaptic protein expression patterns obtained from trained mice are compared to naïve controls using a proteomic approach. To achieve sufficient analytical depth, samples are fractionated in three different ways prior to mass spectrometry, namely 1D SDS-PAGE/in-gel digestion, in-solution digestion and phospho-peptide enrichment.
High-resolution proteomic analysis on a mass spectrometer and label-free quantification are used to examine synaptic protein profiles in phospho-peptide-depleted and phospho-peptide-enriched fractions of synaptosomal protein samples. A commercial software package is utilized to reveal proteins and phospho-peptides with significantly regulated relative synaptic abundance levels (trained/naïve controls). Common and differential regulation modes for the synaptic proteome in the investigated brain regions of mice after training were observed. Subsequently, meta-analyses utilizing several databases are employed to identify underlying cellular functions and biological pathways.
서문
학습은 메모리 트레이스 및 유지 보수의 형성에 기초한다. 널리 하나의 기본 메커니즘은 신경 세포 사이에 존재하는 시냅스 접촉의 새로운 및 / 또는 재배치의 활동에 의존 형성을 나타낼 수 있음을 인정한다. 분자 수준에서 다양한 단백질 변형, 세포 내 relocalizations과 시냅스 단백질의 회전율의 변화 1-4 (LAMPRECHT을 2004 년 # 8) 설명 하였다. 그러나 대부분의 연구는 지금까지 오히려 글로벌 그러나 복잡한 시냅스 프로테옴 조성에보다 선택 단백질에 초점을 맞추었다. 본 방법은 학습 실험 후 마우스 뇌 영역에서 시냅스 프로테옴의 변화에 대한 공정한 심사를 할 수 있습니다. 이 시냅스 아키텍처의 학습에 의한 구조 조정의 시간 - 포인트 의존 분자 스냅 샷을 렌더링하기에 적합합니다. 설명 된 워크 플로는 동물 행동, 단백질 생화학, 질량 분석 및 bioi 다른 전문가의 특정 팀워크를 필요nformatics.
선택된 학습 패러다임, 즉 주파수 변조 톤 차별 (FMTD)는 설치류 5에서 잘 특징 청각 차별 작업입니다. 이 셔틀 박스로 이동 / 학습과 장기 기억 형성 없음 고우 태스크 증가 피질 도파민 시그널링 및 단백질 합성에 따라 메커니즘을 포함한다. 따라서, 게르 빌 루스 쥐와 쥐에 최근 단백체 연구는 도파민과 대뇌 피질에서 시냅스 구성 요소의 학습에 의한 플라스틱 재 배열뿐만 아니라, 가정 FMTD의 학습과 기억 6 ~ 8시 상호 작용을 더 기저 뇌 영역에 보여집니다. 이 기억 형성 다양한 뇌 영역의 복잡한 상호 작용을 포함하므로, 차분 프로테옴 수준에서 이러한 영역들 내에서 조정될 수 있음을 나타낸다. 따라서, 선택 피질 및 피질 마우스 뇌 영역의 해부는 워크 플로에 포함되어 있습니다.
또한, 신뢰성 characterizati심지어 시냅스 단백질 성분에 약한 변화에 사전 및 시냅스 구획의 농축이 아닌 균질 또는 원유 막 분수 (9)의 분석을 필요로한다. 종래 단백질체 분석에 따라서, 시냅 토좀 (synaptosome)의 제조에 이용하는 단계 확립 프로토콜 검출 레벨과 시냅스 특정 단백질 10,11위한 동적 범위를 증가시키기 위해 설명된다.
정량적 목적 라벨없는 고해상도 질량 분석을 사용하는 필수 전제 조건은 단백질 샘플의 높은 유사도이다. 로 시냅스 단백질 조성이 아니라 사소한 변경, 라벨없는 접근 방식은 훈련과 순진 생쥐에서 얻은 단백질 샘플을 대응하는 비교하는 것이 적합 할 것이다 학습 후에 발생할 것으로 예상된다. 또한, 안정 동위 원소 (예 : TMT, iTRAQ, ICPL 및 SILAC)과 MS2 기반 라벨 무료 ...로서를 사용하여 단백질 / 펩티드의 조건 - 특정 레이블 전략ntification (SWATH)이 유용하지만, 이들은 선택된 라벨없는 접근법보다 더 비싼 특수 질량 분석 하드웨어가 필요하다.
단백체 검사는 종종 복잡한 데이터 세트를 생성하기 때문에, 생물 정보학 처리가 적절한 데이터 해석을 권장합니다. 추가 메타 분석은 패러다임 관련 변경 사항 및 관련 핵심 세포 프로세스 및 신호 전달 경로의 식별을 기본 잠재적 인 분자 메커니즘의 더 나은 이해를 지원할 수있다. 적절한 방법은 아래에 설명되어 있습니다.
프로토콜
동물 과목을 포함한 모든 절차는 독일 연방 법률, 각각의 EU 규정 및 NIH 지침의 규정에 따라 수행하고, 그 Landesverwaltungsamt 작센 / 안할 트 (42502-2-1102 IFN)의 윤리위원회에 의해 승인되었습니다.
1. 청각 학습
- 셔틀 상자 (FMTD 패러다임) 주에서 청각 차별 학습 : 마우스를 처리하는 동안 항상 장갑을 착용하십시오.
- 명확한 폴리 카보네이트 케이지에서 무료 음식 펠릿에 대한 액세스 및 수돗물 서너 그룹으로 집 C57BL6 / J 마우스. 동물 시설에서 어두운주기 : 12 시간 빛을 유지한다. 동물이 다른 실험실 또는 회사로부터 수신되는 경우 적응 적어도 일주일 허용 및 정착.
- 하루에 한 셔틀 상자 훈련을 수행합니다.
- 동물 시설에서 홈 케이지에서 마우스를 가지고 사운드 증거 챔버 내에서 어렴풋이 조명 셔틀 상자에 넣습니다.
- 청각 차별 학습을위한 완벽한 컴퓨터 제어 학습 일정을 사용합니다. 침묵의 3 분의 습관화 기간이 시작하고 훈련을 시작합니다.
- 상승 톤의 사용 순서 - 이동 - 시험 동안 이동-자극 등 (4 8 kHz에서, CS +) : 동물은 (올바른 응답이 히트) 톤 프리젠 테이션의 6 초 내에 장애물을 통과해야한다. 셔틀 상자의 그리드 바닥을 통해 전달 300 μA, - 50의 가벼운 발 충격에 의해 미스를 처벌.
- 동물은 톤 프리젠 테이션의 6 초 동안 셔틀 상자의 현재 구획에 남아있다 : 아니 - 이동 - 시험 동안 무 이동-자극으로 - (4 kHz에서, CS-8) 떨어지는 톤의 시퀀스를 사용합니다. 셔틀 상자의 그리드 바닥을 통해 전달 300 μA, - 50의 가벼운 발 충격에 의해 허위 경보를 처벌.
- 20 5 초 intertrial 간격을 사용합니다.
- , 의사 무작위 순서로 세션 당 30 이동 - 시련과 30 무 이동-시험을 수행되도록 한 자체ssion 60 시험으로 구성되어 있으며 약 25 분 동안 지속됩니다.
- 동물 시설에서 홈 케이지에 다시 훈련 된 동물을 넣습니다.
- 뇌 해부
- 경추 탈구 (첫 번째 세션 종료 후 24 시간 등)를 사용하여 훈련 원하는 수 후 원하는 시점에서, 동물을 안락사. 동물의 목을 벨.
- 잘라 먼저 피부와 다음 Sutura의 sagittalis을 따라 직선 가위로 두개골 : 신속하게 다음 단계를 통해 뇌를 해부하다. 완전 강력한 집게를 사용하여 뇌 조직 커버 뼈의 일부를 제거한다. spattle으로 뇌를 꺼내.
- 해부를 들어, 얼음을 가득 페트리 접시에 두뇌를 놓습니다. 청각 피질, 전두엽 피질, 선조체와 메스와 바늘을 사용하여 실체 현미경 하에서 해마를 해부하다.
- 뇌의에 시각적 랜드 마크를 사용하여 청각 피질을 현지화이러한 혈관 표면의 형상 (-3.4에 브레 그마 -2.06, 크기 rostrocaudal 2mm, dorsoventral 1.3 mm) 및 양측 피질의 두께와 직사각형 조직 블록으로 해부로 urface.
- 랜드 마크로서 chiasma의 opticum를 사용 bulbus의 olfactorius에서 조직을 제외 브레 그마 3.56과 1.54 사이에 뇌 슬라이스로 전두엽 피질을 해부하다.
- 브레 그마 1.54과 0.5 사이의 뇌 조각으로 선조체을 해부 조심스럽게 대뇌 피질의 조직을 제거합니다.
- 소뇌를 통해 바늘로 뇌를 고정하고 후두엽에서 시작 피질을 uncoiling에 의해 해마를 해부하다.
- 충격 동결은 -80 ° C에서 액체 질소와 저장소에 뇌 샘플을 해부.
2. 시냅 토좀 (synaptosome)의 제조 또는 대안 시냅스 밀도 (PSD) 풍성 분수
참고 : - 4 ℃로 모든 절차 동안, 0에서 샘플 버퍼를 유지합니다.버퍼 갓 단백질의 단백질 가수 분해를 방지하기 위해, 프로테아제 저해제 칵테일을 포함 희석. 단백질의 인산화는 또한 조사되면 포스파타제 억제제 칵테일을 첨가해야한다. 표시된 모든 g 값은 전체 프로토콜에 걸쳐 g (평균)로 제공됩니다.
- 조 막 분획의 제조 (도 3a)
- 1 ㎖의 얼음을 함유하는 균질기에 전송 해부 뇌 조직은 A (5 mM의 HEPES, 320 mM의 수크로오스, pH 7.4의)를 버퍼 (12) 선을 900 rpm으로 조직을 균질화 콜드.
- 원심 분리기 샘플을 10 분 동안 1,000 XG에. 상층 액을 유지합니다.
- 다시 균질화 10 분 동안 1,000 XG에서 다시 같은 이전과 균질화 버퍼의 동일한 볼륨에서 조건 원심 분리기 샘플에서 알약을. 해당 상층 액을 결합합니다. 주로 핵 및 세포 파편을 포함하는 펠렛의 P1을 폐기하십시오.
- 12,000 x g에서 20 분 동안 결합 상청액 스핀. 취소 superna더 분별 11 tants 또는 사용.
- 20 분 동안 12,000 XG에서 900 rpm으로 회전에서 6 스트로크와 균질를 사용하여 이전과 균질화 버퍼의 동일한 볼륨을 Resuspend 펠릿. 취소 상층 액. 펠릿 조 막 분획을 나타내는 P2.
- 조질 뇌 세포막 분획으로부터 정제 시냅 토좀 (synaptosome) (도 3a)
주 : 원유 뇌 세포막 분획 미엘린, 자당 밀도 단계 구배 초 원심 분리를 이용하여 광 막, 시냅 토좀 (synaptosome) 및 미토콘드리아로 분리 될 수있다. 이 5 mM 트리스 / 염산의 pH를 들어 중 0.32 M, 1.0 M 1.2 M 농도로 자당을 함유하는 8.1 버퍼가 필요합니다.- P2 분획을 생산하기 위해 원심 분리를 수행하는 동안 초 원심 분리 튜브에 자당 단계 구배를 준비한다. 1.5 ml의 1.2 M 자당 버퍼가 유리 파스퇴르 피펫을 사용하여 2.5 ml의 1.0 M 자당 버퍼와 계층 시작합니다.
- 다시 균질화 P2의 분수를그라디언트의 상단에 0.32 M 자당 수동 6 스트로크 버퍼와 부하의 0.5 ml를한다.
- 스윙 버킷 로터를 사용하여 초 원심 분리기에서 2 시간 동안 85,000 XG에 스핀.
- 1.0 M 수 크로즈 완충액 (미엘린 광 막)의 계면에서의 재료를 포함하는 상부 0.32 M 수크로오스 층을 버린다. 1.0 / 1.2 M 자당 버퍼 인터페이스에서 시냅 토좀 (synaptosome)를 수집합니다. 튜브의 하단에있는 펠렛은 미토콘드리아가 포함되어 있습니다.
- 1 비율로 1 시간 동안 150,000 XG에 신중하고 스핀 혼합 : 1의 synaptosomal 분획에 0.32 M 자당 버퍼를 추가합니다. 시냅 토좀 (synaptosome) 펠릿에있는 더 이상의 처리를 위해 요구되는 완충액에 재현 탁 될 수있다.
- PSD 농축 분획의 제조 (그림 3B)
- 100 ㎕의 추출 완충액 한 동물에서 각각의 특정 뇌 영역 균질화 (5 mM 트리스 / 염산 pH를 8.1에서 0.5 % 트리톤 X-100)은 PTFE (폴리 테트라 플루오로 에틸렌) 봉과 함께 200 ㎕의 초 원심 분리 튜브에서12 스트로크 2,000 rpm에서.
- 100 μL 추출 버퍼를 추가 혼합하여 4 ℃에서 1 시간 동안 배양한다. 1 시간 동안 100,000 XG에 스핀 다운과 200 μL 피펫 조심스럽게 상층 액 S1를 수집합니다.
- 12 스트로크 2,000 rpm에서 PTFE 유봉 다시 100 μL 추출 버퍼와 같은 튜브에 펠릿 P1을 다시 균질화.
- 100 μL 추출 버퍼를 추가하고 1 시간 동안 100,000 XG에 피펫 스핀과 잘 섞는다.
- 가용성 단백질 분획에 S1과 S2 상청액을 결합한다. 이 부분은 세포질 단백질, 0.5 % 트리톤 X-100 수용성 막 단백질 및 세포 외 기질 분자가 포함되어 있습니다.
- 50 μl의 5 mM 트리스 / 염산의 pH 8.1의 나머지 펠렛을 재현 탁. 이 분수의 PSD, 세제 저항 막, 불용성 세포 골격 요소, 미토콘드리아 및 핵을 포함한 세포 파편이 포함되어 있습니다. 이것은 시냅스 구조의 코어를 형성 개의 PSD 또한 시냅스의 중요한 부분에 충실활성 영역에서 cytomatrix. 의 PSD의 농축에 대한 요인은 약 4이며, PSD 성분의 농축는 이전에 증명되었다. (12)
질량 분석 3. 샘플 준비
- 용해 및 샘플 정상화
주 : 단백질 농도에 관한 샘플 정규화 마지막에도 약한 시냅스 단백질 발현 변화를 정량적 데이터 신뢰성을 달성하기 위해 매우 중요한 단계이다.- (: 5 청각 피질에 대한 - 15 mg의 조직 사용 20 μL 물질의 총량에 따라 다름) 8 M 요소의 1 얼음에 품어 50 μL - 시냅 토좀 (synaptosome) 또는 (20) 동물의 각 뇌 영역의 PSD 풍부한 준비를 녹여 초음파 조에서 HR.
- 겔에서 소화를 들어, SDS 샘플 버퍼에 직접 시냅 토좀 (synaptosome)을 녹인다. 조심 겔의 과부하를 방지하기 위해로드 크기를 계산한다. 이 경우 그 고려, 높은 풍부한 발판 PROTEINS는 전기 영동시 잃어버린 겔에서 소화 될 것이다.
- 2 M 우레아의 최종 농도를 위해 분리형 세정제 1 %로 희석. 단백질의 carbamylation을 방지하기 위해 30 ° C보다 높은 온도하지 마십시오.
- 표준 절차 (13, 14)에 따라 나누어지는로 샘플 (예를 들면 10 μl를) SDS-PAGE를 수행합니다.
- 쿠마 블루 제조 업체의 프로토콜에 따라 함께 젤 얼룩. 과정은, 메탄올과 아세트산과 고정 및 염색 공정을 결합한다.
- 송신 모드에서 교정 겔 스캐너 전체 차선에 대한 각 샘플의 광학 밀도를 결정하고, 상대 단백질 량을 계산한다.
- 이러한 계산에 따라 샘플들을 정규화.
- 두 개의 서로 다른 부분으로 각 샘플을 분할합니다. 인 - 솔루션 다이제스트에 대한 인 - 젤 소화에 대한 삼분의 3 분의 2를 사용합니다.
- (: 5 청각 피질에 대한 - 15 mg의 조직 사용 20 μL 물질의 총량에 따라 다름) 8 M 요소의 1 얼음에 품어 50 μL - 시냅 토좀 (synaptosome) 또는 (20) 동물의 각 뇌 영역의 PSD 풍부한 준비를 녹여 초음파 조에서 HR.
- 에서 젤 소화
- 젤 분리
- 농도 조정 된 샘플을 이용하여 두 번째 SDS-PAGE를 수행합니다. 얼룩과 정규화 품질을 확인하기위한 두 번째 시간 동안 젤을 정량화.
- 다른 지역 (8 / 레인)에서 겔 내에서 샘플의 각 레인을 잘라하지만 170 kDa의 위의 분자량 범위를 제외합니다. 별도의 튜브에 겔 조각을 전송합니다.
- 에 - 겔 소화 효능을 용이하게하기 위해 날카로운 메스로 작은 조각의 영역 (약. 1 × 1 mm)를 잘라.
- 다이제스트 (15)
- 50 % 아세토 니트릴 (ACN) 및 50mM의 탄산 수소 암모늄 (NH 4 HCO 3)로 이루어지는 완충액 150 μL - 수회 50 10 분 (염색 강도에 따라) 겔 조각을 씻는다.
- 상층 액을 제거합니다. ACN과 겔 조각을 덮고 젤 조각은 흰색이되고 축소 될 때까지 20 ° C에서 품어.
- ACN을 제거하고 5 겔 조각을 재수0.1 M NH 4 HCO (3)의 50 μL와 분 ACN의 동일한 볼륨을 추가하고 37 ° C에서 추가로 15 분 동안 품어.
- 제거하고 완전히 액체 버린다. 진공 원심 분리기에서 겔 조각을 건조.
- 시스테인 잔기를 줄이기 위해 56 ℃에서 NH 45 분 동안 10 mM의 디티 오 트레이 톨 (DTT) 및 열 샘플들을 포함하는 4 HCO 3 50 μL에 겔 조각을 재수.
- 상층 액을 제거하고 감소 시스테인을 carbamidomethylate하기 위해 어둠 속에서 30 분 동안 55 mM의 요오도 아세트 아미드 (IAA)를 포함하는 50 μl의 NH 4 HCO 3을 추가합니다.
- 제거하고 젤 조각 위의 모든 액체를 폐기하고 50 μl의 HCO 3 NH 4 ACN로 두 번 씻어 (1 : 1) 10 분 동안 남아있는 IAA를 제거합니다. 진공 원심 분리기에서 건조 샘플.
- 단백질의 제한 소화 25 mM의 NH 4 HCO 3 트립신의 12.5 NG / μl를 포함하는 추가합니다. 필요한 양의 겔 (P)의 크기와 양에 따라 달라ieces. 몇 분 동안 품어 버퍼가 흡수되어 있는지 확인. 필요한 젤 조각이 완전히 적용 할 필요가있는 경우 더 많은 버퍼를 추가합니다. 37 ° C 하룻밤 (분. 12 시간)에 품어.
- 펩타이드 추출
- (10)와 오버레이 젤 조각 - 25 mM의 NH 4 HCO 3의 20 μL 및 ACN의 동일한 볼륨을 추가합니다. 초음파 목욕을 사용하여 얼음에 10 분 동안 품어. 그 후 제거하고 생성 된 펩티드의 대부분을 포함하는 상층 액을 수집합니다.
- 겔 조각을 30 % ACN / 0.1 % 트리 플루오로 아세트산 (TFA)을 함유하는 추출 완충액 100 ㎕를 추가한다. 초음파 조에서 배양을 반복하고 신중하게 뜨는를 수집합니다.
- 50 %까지 ACN 농도를 증가시킴으로써, 마지막 추출 단계를 반복한다. 초음파 목욕 10 분 후 스핀 다운 및 상층 액을 수집합니다.
- 추출 단계의 세 해당 상청액을 합하고, 진공 원심 분리기에서 건조한다. 그 주레인 / 샘플 당 8 개의 영역이 단계에서 다시 하나의 샘플에 결합 된 겔의 분리의 결과.
- 젤 분리
- 인 - 솔루션 소화
- 요람
- 계산 된 양을 사용하는 적어도 세 개의 기술 복제 충분한 출발 물질을 얻기 위해 표준화 된 샘플들 (예, 150 μl의 용 해물 100 ㎕를, 특정 뇌 영역에서 시료의 부유에 필요한 재료의 양 및 체적에 따라) 라벨이없는 질량 분석을 수행합니다.
- 25 mM의 NH 4 HCO 3 2 mM의 DTT를 추가하고 부드럽게 샘플을 소용돌이. 20 ° C에서 45 분 동안 샘플을 줄입니다.
- 시스테인 잔기를 carbamidomethylate 10 mM의 IAA를 추가합니다. 혼합하고 20 ° C에서 어둠 속에서 30 분 동안 품어.
- 마지막으로, (25 mM의 아세트산에 1 μg의 / μL 트립신) 트립신 주식 솔루션 1 μl를 추가하고 12 시간 동안 20 ° C에서 품어.
- 고체 상 추출 (SPE) -Purification
- 산 분해성 세제를 제거하고, 1 % TFA의 최종 농도로 시료를 조정하고 20 ℃에서 1 시간 동안 배양한다.
- 원심 분리기 샘플을 10 분 동안 16,000 XG에 조심스럽게 상층 액을 수집합니다.
- 랙에 SPE 칼럼을 넣고 2 ml의 메탄올로 행렬을 평형화. 물에 0.1 % TFA 2 ㎖ (버퍼 B)로 2 회 씻는다.
- 버퍼 B 2 ㎖를 추가하고 샘플을로드합니다. 다른 세 번 씻으십시오.
- 200 ㎕의 70 % ACN / 0.1 % TFA를 첨가하여 펩티드를 용출시켰다. 이 단계를 반복합니다.
- 모두 용출액을 수영장과 진공 원심 분리기에 그들을 건조.
- 요람
- 이산화 티탄 크로마토 그래피 (16)에 의해 포스 포 펩타이드 농축
- (50)에 이산화 티탄 구슬의 2 ~ 80 % ACN / 2.5 % TFA (버퍼 C)의 150 μL에 소화하고 평형 겔에서 또는-솔루션 밀리그램에 의해 생성 된 펩티드를 녹여버퍼 C의 μL
- 20 ° C에서 1 시간 동안 회전 장치에 시료에 구슬을 추가하고 품어. 그 후, 스핀 다운 비즈 (16,000 XG, 1 분) 상층 액을 수집합니다.
- 부드럽게 혼합하고 5 분 후에 아래로 회전하여 구슬을 버퍼 C 100 ㎕로 3 회 씻는다. 상층 액을 수집합니다. 각각 (ACN 제외) 0.1 % TFA 100 ㎕ 세 세척 한 다음 80 % ACN / 0.1 % TFA 100 ㎕로이 단계를 세 번 반복합니다.
- 모든 열 상층 액을 결합 진공 원심 분리기를 건조 단계 3.5에 따라 추가의 정제에 대한 포스 포 펩타이드가 고갈 된 분수로 처리합니다.
- 비드를 400 mM의 NH 4 OH / 30 % ACN 20 μL와 결합 포스 포 펩티드를 용출시켰다. 이 단계를 세 번 반복 구슬을 아래로 회전 후 모든 상층 액을 수집합니다.
- 겔에서 소화의 용출액을 결합 시료의 용액 인 다이제스트와하면 포스 P로 처리분수를 eptide 강화. 8 μL - (4)의 최종 부피로 진공 원심 분리기에서 건조한다.
- 농축 마이크로 SPE에 의해 포스 포 펩티드 결핍 분획을 탈염
- 0.1 % TFA의 20 μL에 건조 된 펩티드를 녹여.
- 끝으로 ACN 20 μl를 그림으로써 고정 C (18) -matrix 평형. 끝으로 물에 0.1 % TFA을 그려 행렬을 씻으십시오. 과정을 세 번 반복합니다.
- 천천히 끝으로 산성화 샘플을로드 (이 단계를 세 번 반복).
- 물에 20 μL와 0.1 % TFA를 C (18) -matrix 세 번 세척하고, 세척 용액을 버린다.
- 반복하여 (3 회) TFA 70 % ACN 20 μL / 0.1 % 그려서 피펫 팁에서 펩티드를 용출하고, 분리 관이 용출 용액을 수집한다.
- 시료의 용출액을 합하고, 진공 원심 분리기에서 건조한다.
4. 프로테옴 분석
참고 프로테옴 분석 울트라 HPLC를 구비 한 하이브리드 이중 압력 선형 이온 트랩 / orbitrap 질량 분광계상에서 수행된다. HPLC를가 20 μL 주입 루프 냉각 오토 샘플러로 구성되는 이진 로딩 펌프 (μL 유량 범위) 이진 나노 유동 분리 펌프, 밸브 및 탈기 스위칭 개의 마이크로있는 열 히터. 시료는 먼저 250 NL / 분 열 (예를 들면 75 ㎛의 X 25cm)에 분리 하였다 7 μL / 분의 유속으로 트래핑 컬럼 (예를 들면 100 μm의 X 2cm)을 실시한다. 분리 컬럼의 유출구 직접 질량 분광계 이온화 소스에서 나노 분무 계면에 위치하는 코팅 피코 방출기 팁에 연결된다.
- 나노 액체 크로마토 그래피 탠덤 질량 분석
- 적어도 30 분 동안 12 μl의 2 % ACN / 0.1 % TFA에서 펩타이드 샘플을 녹인다. 15 초 동안 스핀 다운 및 튜브 (원뿔 감소 diamete를 자동 시료 주입기하는 11 μl의 상층 액을 전송아르 자형).
- 다음과 같이 제어 소프트웨어 (예를 들어, Xcalibur)에서 샘플 응용 프로그램, 크로마토 그래피 분리와 탠덤 질량 분석을위한 자동화 된 정권을 설정합니다.
- 자동 시료 주입기 : 다음 온도에 대해 다음 사용 5 ° C를; 열 오븐 : 45 ° C.
- 볼륨 : 주입에 대해 다음 사용 10 μl를; 유속 : 7 μL / 분 (2 % ACN 0.1 % TFA)를; 시간 : 8 분; 밸브 설정 : 트랩 열 - 폐기물; 질량 분석 획득 : 끕니다.
- 유속 : 250 NL / 분 밸브 설정 : 분리에 대해 다음을 사용하여 트랩 열 분리 컬럼을; 질량 분석 획득 :에.
0 분 - 100 분 : 2 % ACN, 0.1 % 포름산 - 40 % ACN, 0.1 % 포름산
100 분 - 105 분, 40 % ACN 0.1 % 포름산 - 95 % ACN 0.1 % 포름산
105 분 - 109 분, 95 % ACN 0.1 % 포름산
109 분 - 120 분 : 2 % ACN, 0.1 % 포름산 - 전체 MS : FTMS, 질량 분석 설정에 대해 다음을 사용하여 해상도 60000; m / z 범위 400 - 2000; MS /MS : 선형 Iontrap; 최소 신호 임계 값 (500); 절연 폭이 다; 30 초를 설정 동적 배제 시간; 단독으로 충전 된 이온은 선택에서 제외됩니다; 정규화 충돌 에너지는 10 밀리 초 시간을 35 %로 설정하고, 활성화된다.
참고 : 전체 MS 스캔이 가장 풍부하게 검출 펩타이드 이온의 충돌 유도 해리 (CID)를 사용하여 실행 최대 15 LTQ MS / MS옵니다.
- 모든 샘플에 대한 세 가지 기술 복제를 실행합니다.
- 단백질 식별 및 라벨 무료 정량
- 단백질 식별 및 라벨없는 정량 (예, PEAKS 스튜디오) 상용 소프트웨어 스위트를 사용하는 방향으로 공정 질량 분석 원시 데이터. 대부분 프로테옴 소프트웨어 패키지 달리 특정 소프트웨어를 사용하는 드 노보 -sequencing 알고리즘 단백질 데이터베이스 정렬에 앞서. 그러나,이 단계는 쉽게 다른 대중적인 소프트웨어 패키지에 의해 치환 될 수있다.
- 사용 ESS표 2에 나열된 ential 설정.
- 포스 프로테오믹스
참고 : 효율적이고 신뢰할 수있는 포스 포 펩타이드 인수는 단백체 워크 플로 설정의 몇 가지 중요한 변화가 필요합니다.- 포스 포 펩타이드 농축 한 후, 결코 건조 샘플 완전히. 항상 용해 샘플을 유지합니다.
참고 : 인산화 트레오닌 또는 세린의 포스 포 에스테르 결합은 매우 취약하다. 이 인산염의 중성 손실을 초래 이온 트랩 내에서 충돌 유도 분열하는 동안. 이것은 차례로 식별에 필요한 펩티드의 더 단편화를 방지한다. 질량 분석 설정에서 허용 광대역 활성화에도 인산염 그룹의 중립 손실 후 포스 포 펩타이드의 분열을 할 수 있습니다. 이는 시간 "의사 - MS (3)"을 저장을 수행한다. MS / MS 데이터 내의 포스 위치 결정은 특정 인증 및 평가를 필요로하며, 형광체 (3)에 의해 수행 될 수있다.0.
- 포스 포 펩타이드 농축 한 후, 결코 건조 샘플 완전히. 항상 용해 샘플을 유지합니다.
5. 생물 정보학 - 메타 분석
주 : 특수 기능 및 네트워크 분석을 수행하기 전에, 단백질의 목록을 사전 처리해야한다. 제 별도로 각 뇌 영역에 대한 조절 단백질 및 포스 포 펩티드의 목록을 병합. 그런 다음 모든 오해를 방지하기 위해 각 부분에 대한 UniProt-ID를 중복 제거합니다.
- GeneCodis 17 단수 농축 분석
- GeneCodis의 웹 기반 도구를 엽니 다 (http://genecodis.cnb.csic.es)
- 주석으로 "생물학적 프로세스를 GO"유기체로 "뮤스의 musculus"를 선택합니다.
- 특정 부분의 UniProt-ID 목록을 붙여 넣습니다. 제출 및 분석이 수행 될 때까지 기다립니다. "GO 생물 공정의 단수 농축 분석"을 클릭하고 결과를 볼 수 있습니다.
- 다른 세 분수 단계를 반복 5.1.3.
- 결과 목록 사이의 중복과 교차를 참조하십시오필요한 데이터를 필터링하는 펄 파이썬 같은 스크립팅 언어를 사용한다. 단수 농축 분석을위한 유사 도구는 플러그인 빙고 (http://apps.cytoscape.org/와 DAVID (https://david.ncifcrf.gov/) 및 Cytoscape (http://www.cytoscape.org/)입니다 앱 / 빙고) 및 ClueGO (http://apps.cytoscape.org/apps/cluego).
- Gephi와 GeneCodis 데이터 중 힘을 기반으로 그래프를 생성 (https://gephi.org/)
주의 : 그래프의 데이터는 그래프 형식 (.gexf, .graphml, .DOT, .gv, .gml) 또는 손으로 입력에서든 사용자가 제공되어야한다.- 그래프의 노드를 생성
- 손으로 : 열기 Gephi "데이터 연구소"를 클릭합니다. 노드를 만듭니다. (가)은 "노드"테이블로 전환 왼쪽의 "노드"를 클릭합니다. "노드 추가"를 클릭합니다. 계약 기간의 이름을 입력합니다. "확인"을 클릭 / Enter 키를 누르십시오.
- 대안 : 저장 GeneCodis는 PC에 결과. 스프레드 시트 프로그램으로이 .txt를 엽니 다. 모든 행의 exce 삭제"Item_Details"(용어의 이름)에서 태평양 표준시. 변경 헤더 "라벨"을 "Item_Details". ".CSV"과 같은 스프레드 시트를 저장합니다. 지금 Gephi에서 "스프레드 시트 가져 오기"를 클릭합니다. Gephi의 파일 브라우저에서 스프레드 시트를 선택합니다. "다음"을 클릭합니다. "마침"을 클릭합니다.
- 가장자리를 통해 노드를 연결합니다.
- 은 "가장자리"테이블로 전환 왼쪽의 "가장자리"를 클릭합니다. 모든 노드 (기간)의 경우 : 다른 조건에서 유전자 이름을 찾아보십시오. 하나 이상의 유전자를 공유하는 경우 -> 가장자리를 만듭니다.
- "에지 추가"를 클릭합니다. "방향성이"를 선택합니다. 소스 선택 및 드롭 다운 목록 중 대상 노드. "확인"을 클릭 / Enter 키를 누르십시오. 하나 이상의 유전자를 공유하는 경우, "무게"(테이블)에 풍요를 입력합니다.
- 강제 그래픽 레이아웃을 기반으로.
- 손으로 입력 한 오픈 그래프 데이터 파일, 그래프 타입 "무향"또는 데이터를 사용 설정, 이미 SELECTE하지 않을 경우 "개요"를 클릭디.
- 상호의 풍부에 따라 노드의 크기를 조정합니다. , 통계를 클릭하면 "네트워크 개요"에서 "평균 정도"(비가 가장자리) 또는 "평균. 가중 과정"(가중 가장자리)를 실행합니다. "모양"에서 "속성"을 선택 다음, 크기 버튼을 누른 다음, "노드"를 클릭하고 속성 파라미터를 "매력에 빠져보십시오. 가중 과정"또는 "평균 정도"로 설정하십시오. 적용을 클릭합니다.
- 마지막으로 "레이아웃"에서 "강제 아틀라스"를 선택하고 실행; 노드가 충돌하는 경우 "반발 강도"로 변경합니다.
- 사진을 내보내기.
- 스크린 샷 기능 : "개요", 변화 그래프 레이아웃, 엣지 두께, 라벨 크기와 "그래프"창 하단의 메뉴 확장을 클릭합니다. 카메라 왼쪽 버튼을 클릭하고 사진을 저장합니다.
- '미리보기'의 내보내기 기능은 "미리보기"를 클릭합니다. "바로 기본"을 사전 설정을 변경합니다. 설정 변경의 선택된 환경 설정에 따라 및 수출 "SVG / PDF / PNG"을 클릭합니다.
- 그래프의 노드를 생성
결과
그림 1은 청각 차별 학습 후 마우스 뇌 영역의 양적 시냅스 프로테옴 프로파일의 전체 흐름을 요약 한 것입니다. 그것은 셔틀 상자의 동물 훈련을 시작합니다. 도 2에 도시 된 예에서, 마우스는 효율적인 학습을 나타내는 네 번째 훈련 상당한 FM 톤 구별을 표시하기 시작했다. 동물의 뇌 영역 해부에 대한 선택 시점에서 희생된다. 시냅스 필요한 농축 어느 시냅 토좀 (synaptosome)의 제조 방법에 의해 달성 될 수 있거나 PSD 농축 방법은 낮은 조직 양을 위해 개발 된 대안 PSD - 풍부 분획의 제조에 의해, 모두도 3에 상세히 설명 예 1 - 쥐의 뇌 (12), (18)에서 2 해마 조각. 그것은 작은 튜브,이 튜브 피팅 PTFE의 방앗와 유 봉에 전력을 공급하기위한 실험실 드릴링 드라이브가 필요합니다.
인해 시냅 토좀 (synaptosome)의 특정 단백질 조성물에 강력히 개의 서로 다르지만 상보적인 방식으로 샘플 준비를 수행 할 것을 권장한다. 의 PSD의 골격은 종종 높은 화학 양론에서 발생하는 매우 높은 분자량의 단백질이다. 인 - 솔루션 다이제스트 효율적으로 추출 할 수 있지만, 생성 된 펩타이드 혼합물의 오버 샘플링으로 이어질 수있는 가장 좋은 방법입니다. 는 겔에 그 고 분자량 단백질을 제외하고 중간 및 낮은 분자량의 단백질의 분석을 선호 할 수 병렬로 동일한 샘플의 수행 다이제스트. 포괄적 인 분석을 위해 단백질 분해 소화의 두 종류가 권장됩니다.
조사 뇌 영역의 조직의 서로 다른 양 더 나은 비교를 위해 적용 재료의 조정이 필요합니다. 네 조사 뇌 영역에서 청각 피질은 일반적으로 제한적인 사실또는. 다른 뇌 영역의 재료 신중 시냅 토좀 (synaptosome) 또는 PSD 풍부한 분획을 제조 한 후, 청각 피질의 양을 조정한다 (3.1.1 참조.). 다음과 같은 마우스에서 새로 제조 된 뇌 영역의 일반적인 무게는 다음과 같습니다 청각 피질 (AC) : ~ 50 mg의; 해마 (HIP) ~ 90 mg의; 선조체 (STR) : ~ 120 mg의 전두엽 피질 (FC) : ~ 100 mg의.
2.3 절에 설명 된 PSD 농축 방법은 약 1,500 가지 단백질 번의 동물 (표 1)의 수준에서의 뇌 영역 당 약 250 가지 포스 포 펩티드의 식별을 허용했다. 24 시간 첫 훈련 후 단백체 분석은 확인 된 단백질의 7.3 % 및 포스 포 펩타이드의 5.8 %가 유의 한 (P <0.05) 순진한 컨트롤 (표 1)에 비해 자신의 시냅스 식의 양적 변화를 보였다 것으로 나타났습니다. 다운 REGULA에 대한 눈에 띄는 경향시냅스 비계의 기는 FMTD 학습의 초기 단계 시냅스 아키텍처의 발음 재 배열을 가리 수 있습니다. 단지 22 %가 두 개 이상의 뇌 영역에서 조절되는 것으로 밝혀졌다 반면 조절 단백질의 대부분이 뇌 영역 특정 방법으로 변경 하였다. 여섯 선택된 예는도 4에 도시되어있다.
"에서 RhoA 시그널링", "노치 신호 전달"클라 매개 엔도 시토 시스 시그널링 ","축삭 지침 시그널링 ","칼슘 신호 전달 "IPA에 의해 복잡한 결과를 메타 분석은 다음과 같은 정식 경로의 특정 참여 / 조작에 대한 증거를 제공 ","상피 Adherens 접합의 리모델링 ","글루타메이트 수용체 신호 전달 ","GABA 수용체 신호 전달 ","도파민 수용체 신호 전달 '과'시냅스 장기 증강 ".
단일 농축 분석 단백질 수송, 세포 부착, 인산화, 엔도 시토 시스, 소포 매개 교통, 전뇌 개발 및 axonogenesis (그림 5)와 관련된 전두엽 피질이 크게 부풀려 생물학적 과정을 밝혔다. 이온 수송, 번역, mRNA의 전송을 포함하는 청각 피질 생물학적 과정에서 단백질 수송 및 학습에 띄게되었다. 해마의 단백질 분획의 분석은 상당히 이온 전달, 세포주기, 번역, 인산화 및 신경계 발달과 관련된 농축 처리를 검출한다. 선조체에서 mRNA의 운송, 소포 매개 교통, axonogenesis, 단백질 분해, 단백질 수송 및 세포 내 이입을 포함한 생물학적 과정이 발견되었다 부풀려.
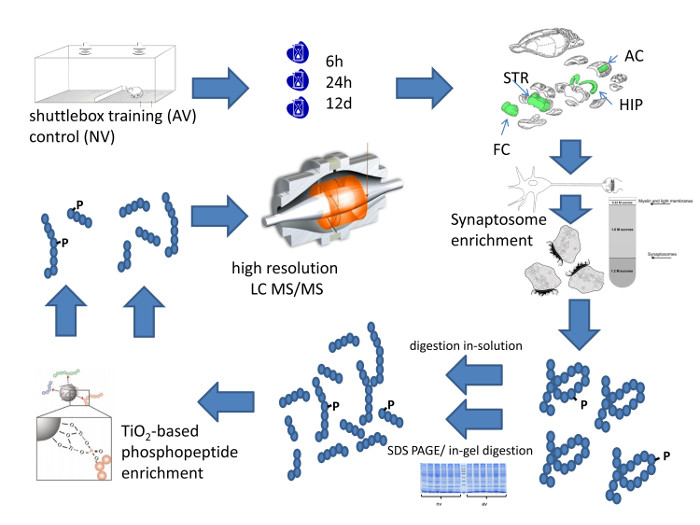
그림 1 : 체계적인 Workflo방법 론적 접근의 w 이 그림은 개략적으로 뇌 영역 특정 시냅스 단백질 조성물의 높은 해상도 정량 프로파일의 흐름을 요약 한 것입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2 : FM 톤 차별 작업에서 마우스의 성능의 예. 동물 안타의 증가 속도 (파란색 곡선)과 훈련의 과정에서 잘못된 경보의 감소 속도 (검정 곡선)을 보여줍니다. 중요한 판별 네번째 세션에서 일어난다. 오차 막대는 SEM로 제공됩니다. 고맙다을 보려면 여기를 클릭하십시오이 그림의 게르 버전입니다.

그림 3 : 시냅 및 PSD 풍부한 분수의 준비. A : 시냅 준비. B : PSD 농축 분획 제제. 두 그림은 뇌 조직에서 시냅 토좀 (synaptosome) 또는 대안 PSD 농축 분획 제제의 상세한 워크 플로우를 설명합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
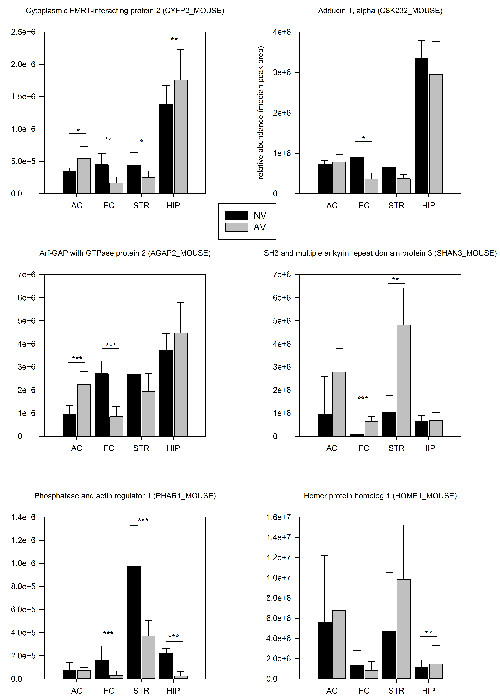
그림 4 : 선정 된 양적 프로테옴 결과. 선택된 단백질의 상대적 시냅스 존재비는 마우스 TRA 사이 비교(AV N = 6) FMTD 태스크와 나이브 대조군 마우스 (NV, N = 6) 24 시간 후 1 트레이닝 세션 리트. 풍부한 값은 단백질의 세 가장 강렬한 펩티드의 피크 면적의 중앙값으로서 계산 하였다. 중요한 풍부 변경 (AV / NV; t 테스트)와 단백질은 플롯 내에서 표시됩니다 : * P <0.05, ** p <0.01, *** p <0.005. 오차 막대는 SD로 제공됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 5 : GeneCodis / Gephi에 의해 정면 코어 텍스에 대한 생물 진학의 시각화. 최소 3 단백질 번호 "생물학적 과정"에 관련된 유전자 온톨로지 (GO) 데이터베이스 (http://geneontology.org)의 중요한 용어 여기에 표시됩니다. 노드 GO 조건을 대표하는 노드의 크기, 특정 노드의 연결 라인 폭 및 개수는 다른 노드와이 GO 용어 공유 단백질의 수를 도시한다. 때문에 Gephi의 "강제 아틀라스"방법, 관련 노드는 서로 밀접하게 클러스터링됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 뇌 영역 | AC | FC | 잘 알고 있기 | 000 "얼굴 ="돋움 "크기 ="3 "> STR | Σ |
| 확인 된 단백질 | 1,435 | 1,758 | 1,572 | 1,507 | 6272 |
| 규제 단백질 (P <0.05) | 59 | (130) | (162) | (108) | 얼굴 = "돋움"크기 = "3"> (459) |
| ↑ AV / NV | 8 | 4 | (76) | (35) | (123) |
| ↓의 AV / NV | (51) | (126) | (86) | 73 | (336) |
| 확인 phosphomoti FS | 197 | (361) | (273) | (278) | 1,109 |
| 규제 phosphomotifs (P <0.05) | 8 | (22) | (21) | (14) | (65) |
| ↑ AV / NV | 4 | 00000 "얼굴 ="돋움 "크기 ="3 "> (17) | (5) | 9 | (35) |
| ↓의 AV / NV | 4 | (5) | (16) | (5) | (30) |
표 1 : 단백체 결과의 요약. 이 표는 훈련 된 쥐의 대표 단백체 실험 요약 (AV를 N = 6) 자신의 순진한 컨트롤에 비해 첫 훈련 후 24 시간 (NV, N = 6). 그만큼459 조절 단백질의 합은 중복 규정을 포함한다. 283 다른 규정은 뇌의 특정로 결정 하였다. 구체적으로, 57 단백질이 뇌 영역에서 규제, 18 단백질 규정은 세 가지 뇌 영역에서 발견 된 만 2 단백질은 네 개의 연구 뇌 영역에서 조절된다.
| 오류 허용 오차 | |
| 전구체 질량 (푸리에 변환 질량 분석) | 10 ppm의 |
| 조각 이온 질량 (선형 이온 트랩) | 0.6 다 |
| 펩타이드 당 최대 놓친 분열 | 삼 |
| 수정 수정 | |
| 용의 젤 소화 샘플 | 시스테인의 Carbamidomethylation |
| 대한 샘플에-솔루션 소화 | Methylthiolatio시스테인의 N |
| 변수 수정 | 메티오닌의 산화 |
| 아스파라긴 및 / 또는 글루타민의 Deamidations | |
| 데이터 베이스 | Uniprot / Sprot |
| 분류 | 쥐 |
| 통계 식별-허용 설정 | |
| 드 노보 평균 지방 신뢰 (ALC) | > 50 % |
| 펩타이드 거짓 검색 속도 (FDR, 동부 표준시에 따라. 디코이 융합) | <1 % |
| 단백질 유의성 (-10logP는 변형 T 테스트 기준) | > (20) |
| 독특한 펩타이드 / 단백질 | ≥ 1 |
| 정량 설정 : | |
| 정량하는 경우에 사용되는 펩티드 : | |
| 펩타이드의 의미 (-10logP) | > (30) |
| 의 펩타이드 식별 | 샘플의 ≥ 50 % |
| 펩타이드 신호 품질 | > 1 |
| 펩타이드 평균 면적 | > 1E5 |
| 펩타이드 보유 시간 허용 | <5 분 |
| 표준화 | 총 이온 전류 (TIC) |
표 2 : 단백질 식별을위한 설정 (단계 4.2.2).
토론
이 연구는 학습과 생쥐의 다른 뇌 영역 메모리 통합 동안 시냅스 단백질 발현 변화의 정확한 정량 프로파일에 최적화 된 방법론 워크 플로우를 제공합니다. 설치는 질량 분석 분석을위한 샘플 당 적어도 세 기술 복제의 필요한 응용 프로그램의에도 불구하고 단일 동물의 수준에서 단백질 발현을 연구 할 수있는 기회를 제공합니다.
방법론이 고려 고분자 지지체 단백질로 이루어진 사전 및 postsynapse의 특정 단백질 조성물을 얻어 아니라 중간 또는 낮은 분자량 담지 중요한 매개체 단백질. synaptosomal 준비의에서-솔루션 다이제스트 효율적인 세대가 발생하고, 따라서 발판 유래 펩티드의 과잉 표현입니다. 이는 차례로, 더 작은 또는 저급 풍부한 단백질의 분석을 억제 할 수있다. 에서 SDS-PAGE 분획의 제안 준비평행 인 - 겔 소화 과정과 함께 각각의 샘플의 분취 량 중저 풍부한 단백질의 분석을 용이하게하고, 추천 상보적인 방법을 나타낸다. 샘플 유래의 모든 분획의 분리 질량 분석 도포 후 (인 용액 다이제스트 예에서 겔 결합 포스 - 풍부 분획 다이제스트)에 대응하는 MS / MS 데이터 세트 합하고 상기 PEAKS 의한 단백질 확인 및 정량을 위해 계산 될 수있다 소프트웨어 나 다른 인기있는 소프트웨어 패키지.
대안 적으로, 인 용액 절단 샘플의 생성에 겔 소화 유래 시료의 분획 (시료 차선 별도로 처리 겔 부분) 및 분획의 개별 애플리케이션이 증가 할 수 질량 스펙트럼 (이온 교환 크로마토 그래피에 의해 예) 분석 깊이. 그러나,이 확장 된 워크 플로우를 극적으로 LS-MS / MS 데이터 수집에 필요한 시간을 증가시킨다. generatio에 대한프로테옴 프로파일의 지정된 시간 코스를 학습과 기억 형성 동안 시냅스 단백질의 재 배열의 상세한 분자 시퀀스의 N이 필요합니다. 동물의 성능 후 약 학습 곡선의 점근 적 수준에 도달 할 때까지이 시간 코스는 후 또는 첫 번째 훈련 세션 동안 즉시 시작하고 확대 메시 시간 프레임을 커버 할 수있다. 8 - 훈련 10 일 (자세한 내용은 그림 2 참조).
시냅스 단백질의 인산화 변화의 분석은 FMTD 학습하는 동안 선택된 시간 프레임에서 특정 초점을 필요로한다. 단백질 인산화와 dephosphorylations에 의해 유발되는 것으로 알려져 시냅스 단백질의 재 배열을 개시 한 손으로 신호 폭포에서 동물 훈련의 초기 단계에서 예상된다. 한편, (S) 내에 연결 및 조립을 조절 알려진 여러 시냅스 인산화 단백질의 장기적인 변형이 존재ynaptic 건축 19, 20. 이러한 번역 후 변형에도 메모리 통합 이후 시점에서 예상된다.
이 단백체 워크 플로우에 의해 생성 된 복잡한 데이터 세트 분자 경로 및 주요 분자를 참여 식별하기 위해 생물 정보학 처리를 필요로한다. 메타 분석은 학습과 기억 과정에 중요한 역할을 크게 부풀려 경로를 보여줍니다.
공개
The authors have nothing to disclose.
감사의 말
We wish to thank Yvonne Ducho and Kathrin Pohlmann for excellent technical assistance. This work was supported by the Deutsche Forschungsgemeinschaft (SFB 779) and by the State Saxony-Anhalt / European Regional Development Fund (ERDF) via the Center for Behavioral Brain Sciences (CBBS).
자료
| Name | Company | Catalog Number | Comments |
| 3 M Empore Solid Phase Extraction- Filter | 3M Bioanalytical Technologies | 4245SD | 7 mm/3 ml |
| Acclaim PepMap 100 | Dionex/Thermo Scientific | 164564 | 100 µm x 2 cm, C18 |
| Acclaim PepMap 100 | Dionex/Thermo Scientific | 164569 | 75 µm x 25 cm, C18 |
| Acetic acid | Carl Roth GmbH | 3738.1 | |
| Acetonitrile (ACN) | Carl Roth GmbH | AE70.2 | |
| Acrylamide (30%) | AppliChem | A0951 | |
| Ammonium hydrogen carbonate | Fluka | 9830 | |
| Ammonium hydroxide | Fluka | 44273 | |
| Ammonium persulfate (APS) | AppliChem | A2941 | |
| Biofuge pico | Heraeus GmbH | 75003280 | |
| Blue R-250 | SERVA Electrophoresis GmbH | 17525 | |
| Bromophenol Blue | Pharmacia Biotech | 17132901 | |
| C57BL/6J mice | Charles River | ||
| Cantharidin | Carl Roth GmbH | 3322.1 | |
| Centrifuge tubes for MLS-50 | Beckman Coulter | 344057 | |
| Centrifuge tubes for TLA 100.1 rotor | Beckman Coulter | 343776 | |
| Dithiothreitol (DTT) | AppliChem | A1101 | |
| Eppendorf 5417R centrifuge | VWR | 22636138 | |
| Eppendorf A-8-11 rotor | VWR | 5407000317 | |
| Formic acid | Fluka | 14265 | |
| GeneCodis | http://genecodis.cnb.csic.es/ | ||
| Gephi | https://gephi.org/ | ||
| Glycerol | AppliChem | A1123 | |
| Glycine | AppliChem | A1067 | |
| HALT Phosphatase Inhibitor Cocktail | Pierce /Thermo Scientific | 78420 | |
| HEPES Buffer solution | PAA Laboratories GmbH | S11-001 | |
| Homogenization vessel 2 ml | Sartorius AG | 854 2252 | |
| Hydrochloric acid | Sigma-Aldrich | H1758 | |
| Imidazole | Sigma-Aldrich | I2399 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide (IAA) | Sigma-Aldrich | I1149 | |
| Laboratory drilling drive K-ControlTLC 4957 | Kaltenbach & Vogt GmbH | 182997 | |
| LTQ Tune Plus 2.7.0.1112 SP2 | Thermo Scientific | ||
| LTQ Orbitrap Velos Pro | Thermo Scientific | ||
| Macs-mix tube rotator | Miltenyi Biotech | 130-090-753 | |
| Magic Scan 4.71 | UMAX | ||
| Methanol | Carl Roth GmbH | AE71.2 | |
| MLS-50 rotor | Beckman Coulter | 367280 | |
| Optima MAX Ultracentrifuge | Beckman Coulter | 364300 | |
| PageRuler Prestained Protein Ladder | Thermo Scientific | 26616 | |
| PEAKS 7.5 | Bioinformatic Solutions | ||
| Phosphatase Inhibitor Cocktail 3 | Sigma-Aldrich | P0044 | |
| PhosphoRS 3.1 | IMP/IMBA/GMI | ||
| PhosSTOP | Roche | 4906845001 | |
| Plunger/pestle made of PTFE | Sartorius AG | 854 2651 | |
| PotterS homogenizer | Sartorius AG | 853 3024 | |
| Protease Inhibitor complete mini | Roche | 4693159001 | |
| Quantity One 4.5.1 | BioRad | ||
| RapiGest | Waters | 186002122 | |
| Shuttle box | Coulbourne Instruments | ||
| Sodium dodecylsulfate (SDS) | AppliChem | A1112 | |
| Sodium molybdate | Carl Roth GmbH | 274.2 | |
| Sodium tartrate dihydrate | Sigma-Aldrich | 228729 | |
| SONOREX RK 156 Ultrasonic Bath | BANDELIN electronic GmbH & Co. KG | 305 | |
| Soundproof chamber | Industrial Acoustics Company | ||
| Sucrose | Carl Roth GmbH | 4621.2 | |
| Tetramethyl ethylene -1,2-diamine (TEMED) | Sigma-Aldrich | T9281 | |
| Thermomixer basic | CallMedia | 111000 | |
| Titansphere TiO 5µm | GL Sciences Inc. Japan | 502075000 | |
| TLA 100.1 rotor | Beckman Coulter | 343840 | |
| Trifluoro acetic acid (TFA) | Sigma-Aldrich | T6508 | |
| Tris ( hydroxymethyl) aminomethane (TRIS) | AppliChem | A1086 | |
| Triton X-100 | Sigma-Aldrich | T8532 | |
| Trypsin Gold | Promega | V5280 | |
| Ultimate 3000 Ultra HPLC | Dionex/Thermo Scientific | ||
| Ultracentrifuge tube | Beckman Coulter | 343776 | |
| Unijet II Refrigerated Aspirator | Uniequip Laborgeräte- und Vertriebs GmbH | ||
| UNIVAPO 100 H Concentrator Centrifuge | Uniequip Laborgeräte- und Vertriebs GmbH | ||
| Urea | AppliChem | A1049 | |
| Water (high quality purifed) | Resistivity: > 18.2 MΩ*cm at 25 °C Pyrogens: < 0.02 EU/ml TOC: < 10 ppb | ||
| Xcalibur 3.0.63 | Thermo Scientific | ||
| ZipTipC18 Pipette Tips | MILLIPORE | ZTC18S960 |
참고문헌
- Lamprecht, R., LeDoux, J. Structural plasticity and memory. Nat Rev Neurosci. 5 (1), 45-54 (2004).
- Bingol, B., Schuman, E. M. Synaptic protein degradation by the ubiquitin proteasome system. Curr Opin Neurobiol. 15 (5), 536-541 (2005).
- Richter, J. D., Klann, E. Making synaptic plasticity and memory last: mechanisms of translational regulation. Genes Dev. 23 (1), 1-11 (2009).
- Rosenberg, T., et al. The roles of protein expression in synaptic plasticity and memory consolidation. Front Mol Neurosci. 7, 86(2014).
- Scheich, H., et al. Behavioral semantics of learning and crossmodal processing in auditory cortex: the semantic processor concept. Hear Res. 271 (1-2), 3-15 (2011).
- Kähne, T., et al. Synaptic proteome changes in mouse brain regions upon auditory discrimination learning. Proteomics. 12 (15-16), 2433-2444 (2012).
- Reichenbach, N., et al. Differential effects of dopamine signalling on long-term memory formation and consolidation in rodent brain. Proteome Sci. 13, 13(2015).
- Kähne, T., et al. Proteome rearrangements after auditory learning: high-resolution profiling of synapse-enriched protein fractions from mouse brain. J Neurochem. , (2016).
- Li, K., et al. Organelle proteomics of rat synaptic proteins: correlation-profiling by isotope-coded affinity tagging in conjunction with liquid chromatography-tandem mass spectrometry to reveal post-synaptic density specific proteins. J Proteome Res. 4 (3), 725-733 (2005).
- Carlin, R. K., Grab, D. J., Cohen, R. S., Siekevitz, P. Isolation and characterization of postsynaptic densities from various brain regions: enrichment of different types of postsynaptic densities. J Cell Biol. 86 (3), 831-845 (1980).
- Smalla, K. H., Klemmer, P., Wyneken, U. The Cytoskeleton - Imaging, Isolation, and Interaction. Dermietzel, R. , Humana Press. 265-282 (2012).
- Smalla, K. H., et al. The synaptic glycoprotein neuroplastin is involved in long-term potentiation at hippocampal CA1 synapses. Proc Natl Acad Sci U S A. 97 (8), 4327-4332 (2000).
- Shapiro, A. L., Vinuela, E., Maizel, J. V. Molecular weight estimation of polypeptide chains by electrophoresis in SDS-polyacrylamide gels. Biochem Biophys Res Commun. 28 (5), 815-820 (1967).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Shevchenko, A., Wilm, M., Vorm, O., Mann, M. Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. Anal Chem. 68 (5), 850-858 (1996).
- Thingholm, T. E., Larsen, M. R., Ingrell, C. R., Kassem, M., Jensen, O. N. TiO(2)-based phosphoproteomic analysis of the plasma membrane and the effects of phosphatase inhibitor treatment. J Proteome Res. 7 (8), 3304-3313 (2008).
- Carmona-Saez, P., Chagoyen, M., Tirado, F., Carazo, J. M., Pascual-Montano, A. GENECODIS: a web-based tool for finding significant concurrent annotations in gene lists. Genome Biol. 8 (1), 3(2007).
- Bonn, S., Seeburg, P. H., Schwarz, M. K. Combinatorial expression of alpha- and gamma-protocadherins alters their presenilin-dependent processing. Mol Cell Biol. 27 (11), 4121-4132 (2007).
- Tweedie-Cullen, R. Y., Reck, J. M., Mansuy, I. M. Comprehensive mapping of post-translational modifications on synaptic, nuclear, and histone proteins in the adult mouse brain. J Proteome Res. 8 (11), 4966-4982 (2009).
- Li, L., et al. The differential hippocampal phosphoproteome of Apodemus sylvaticus paralleling spatial memory retrieval in the Barnes maze. Behav Brain Res. 264, 126-134 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유