Method Article
Résolution quantitative Haute Synaptic Proteome Profiling des souris régions cérébrales Après Auditory Discrimination apprentissage
Dans cet article
Résumé
The identification of molecules and pathways controlling synaptic plasticity and memory is still a major challenge in neuroscience. Here, a workflow is described addressing the relative quantification of synaptic proteins supposedly involved in the molecular reorganization of synapses during learning and memory consolidation in an auditory learning paradigm.
Résumé
The molecular synaptic mechanisms underlying auditory learning and memory remain largely unknown. Here, the workflow of a proteomic study on auditory discrimination learning in mice is described. In this learning paradigm, mice are trained in a shuttle box Go/NoGo-task to discriminate between rising and falling frequency-modulated tones in order to avoid a mild electric foot-shock. The protocol involves the enrichment of synaptosomes from four brain areas, namely the auditory cortex, frontal cortex, hippocampus, and striatum, at different stages of training. Synaptic protein expression patterns obtained from trained mice are compared to naïve controls using a proteomic approach. To achieve sufficient analytical depth, samples are fractionated in three different ways prior to mass spectrometry, namely 1D SDS-PAGE/in-gel digestion, in-solution digestion and phospho-peptide enrichment.
High-resolution proteomic analysis on a mass spectrometer and label-free quantification are used to examine synaptic protein profiles in phospho-peptide-depleted and phospho-peptide-enriched fractions of synaptosomal protein samples. A commercial software package is utilized to reveal proteins and phospho-peptides with significantly regulated relative synaptic abundance levels (trained/naïve controls). Common and differential regulation modes for the synaptic proteome in the investigated brain regions of mice after training were observed. Subsequently, meta-analyses utilizing several databases are employed to identify underlying cellular functions and biological pathways.
Introduction
L'apprentissage est basé sur la formation de traces de la mémoire et de leur entretien. Il est largement admis que l'un mécanisme sous-jacent peut représenter une formation dépendant de l'activité de la nouvelle et / ou le réarrangement des contacts synaptiques entre les neurones existants. Au niveau moléculaire, diverses modifications de protéines, relocalizations subcellulaires et des changements dans le chiffre d' affaires des protéines synaptiques ont été décrites 1-4 (Lamprecht, 2004 N ° 8). Cependant, la plupart des études ont porté jusqu'ici sur des protéines sélectionnées plutôt que sur la composition globale, mais protéome synaptique complexe. La présente approche permet un examen impartial des changements protéomiques synaptiques dans les régions du cerveau de la souris après une expérience d'apprentissage. Il convient de rendre le temps points instantanés moléculaires dépendant de la réorganisation induite par l'apprentissage de l'architecture synaptique. Le workflow décrit nécessite un travail d'équipe particulière de différents spécialistes du comportement animal, la biochimie des protéines, spectrométrie de masse et Biolnformatics.
Le paradigme d'apprentissage choisi, soit modulé en fréquence ton discrimination (FMTD), est une tâche de discrimination auditive bien caractérisé chez les rongeurs 5. Apprentissage et formation de la mémoire à long terme dans cette boîte de navette Go / No-Go-tâche implique des mécanismes en fonction de l'augmentation de signalisation de la dopamine corticale et la synthèse des protéines. Par conséquent, des études récentes sur protéomiques gerbilles et de souris ont révélé la dopamine et de réarrangements en plastique induite par l' apprentissage des composantes-synaptiques corticales, mais aussi dans les régions du cerveau qui interagissent basales plus supposés pendant FMTD apprentissage et la mémoire 6-8. Cela montre que la formation de la mémoire implique une interaction complexe de diverses régions du cerveau et ainsi, peut-être différemment réglementée dans ces régions au niveau du protéome. Par conséquent, la dissection de régions du cerveau de la souris corticales et sous-corticales sélectionnés est inclus dans le flux de travail.
En outre, le characterizati fiablemême sur des changements faibles dans la composition des protéines synaptiques nécessite un enrichissement de compartiments avant et post - synaptiques plutôt que l'analyse des broyats ou des fractions membranaires brutes 9. Protocoles Par conséquent, la préparation de synaptosomes utilisant établie avant l'analyse protéomique est décrite afin d'augmenter le niveau de détection et de la plage dynamique pour des protéines spécifiques synapse 10,11.
Une condition indispensable d'utiliser sans étiquette haute résolution spectrométrie de masse à des fins quantitatives est un haut degré de similitude des échantillons de protéines. Comme il est prévu des changements plutôt mineures dans la composition des protéines synaptiques à se produire après avoir appris, une approche sans étiquette sera approprié de comparer les échantillons de protéines obtenus à partir de souris formés et naïfs correspondant. Alternativement, les stratégies de protéines / peptides en utilisant des isotopes stables (par exemple TMT, iTRAQ, ICPL et SILAC), ainsi que l' étiquette à base de MS2 libre qua étiquette spécifique conditionnéentification (COULOIR) sont utiles, mais ils sont plus chers que l'approche sans étiquette choisie ou besoin de matériel de spectrométrie de masse spéciale.
Depuis des projections protéomiques donnent souvent des ensembles de données complexes, le traitement bioinformatique est recommandé pour l'interprétation des données appropriées. méta-analyses supplémentaires peuvent favoriser une meilleure compréhension des mécanismes moléculaires sous-jacents potentiels changements liés paradigmes et l'identification des processus cellulaires clés impliqués et voies de signalisation. Des méthodes appropriées sont également décrites ci-dessous.
Protocole
Toutes les procédures, y compris des sujets animaux ont été effectuées en conformité avec les règlements de la loi fédérale allemande, les réglementations européennes respectives et les lignes directrices du NIH, et ont été approuvés par le comité d'éthique de l'Landesverwaltungsamt Sachsen / Anhalt (42502-2-1102 IfN).
1. Apprentissage Auditory
- Auditory apprentissage discrimination dans la boîte de navette (FMTD de paradigme) Note: Toujours porter des gants lors de la manipulation des souris.
- Maison C57BL6 / J souris en groupes de trois ou quatre avec libre accès aux boulettes de nourriture et de l'eau du robinet dans des cages en polycarbonate transparent. Maintenir une lumière 12 h: cycle d'obscurité dans l'animalerie. Si les animaux sont reçus d'un autre laboratoire ou d'une entreprise permettra au moins une semaine d'acclimatation et l'emménagement.
- Effectuer une session de formation de boîte à navettes par jour.
- Prenez la souris de sa cage dans l'animalerie et le placer dans une boîte de navette faiblement éclairée dans un bruit chambre preuve.
- Utilisez un calendrier entièrement contrôlé par ordinateur pour l'apprentissage auditif apprentissage de la discrimination. Commencez par une période d'habituation de 3 min de silence, puis commencer la session de formation.
- Utilisez des séquences du ton montant (4-8 kHz, CS +) comme Go-stimulus lors de Go-essais: L'animal doit franchir l'obstacle dans les 6 secondes de la présentation de la tonalité (réponse correcte, a frappé). Punir un échec par un pied-léger choc de 50 - 300 pA, délivré par le plancher de la grille de la boîte de navette.
- Utilisez des séquences du ton descendant (8-4 kHz, CS-) que le No-Go-stimulus pendant No-Go-essais: L'animal doit rester dans le compartiment en cours de la boîte de navette pendant 6 sec de la présentation de ton. Punir une fausse alarme par un pied-léger choc de 50 - 300 pA, délivré par le plancher de la grille de la boîte de navette.
- Utilisez des intervalles de 20 intertrial 5 sec.
- Effectuer 30 Go-essais et 30 No-Go-essais par session dans un ordre pseudo-aléatoire, de sorte que l'on session se compose de 60 essais et dure environ 25 min.
- Mettez l'animal formé de nouveau dans sa cage à domicile dans l'animalerie.
- Dissection du cerveau
- Euthanasier l'animal au point de temps désiré après un nombre souhaité de sessions de formation à l' aide de la dislocation cervicale (par exemple , 24 heures après la fin de la première session). Décapitez l'animal.
- Disséquer rapidement le cerveau via les étapes suivantes: Coupez d' abord la peau, puis le crâne avec des ciseaux droits le long des sagittalis Sutura. Retirez complètement les parties de l'os qui couvrent les tissus du cerveau en utilisant des pinces solides. Sortez le cerveau avec une spatule.
- Pour la dissection, placez le cerveau sur une boîte de Pétri remplie de glace. Disséquer le cortex auditif, le cortex frontal, le striatum et l'hippocampe sous un stéréomicroscope à l'aide d'un scalpel et d'une aiguille.
- Localisez le cortex auditif en utilisant des repères visuels sur le cerveau surface tels que les vaisseaux sanguins et la forme de la surface (bregma -2,06 à -3,4, la taille rostrocaudale 2 mm, 1,3 mm) dorso-ventral et bilatéralement disséquer un bloc de tissu rectangulaire à l'épaisseur du cortex.
- Disséquer le cortex frontal comme une tranche de cerveau entre bregma 3,56 et 1,54 en utilisant le opticum de chiasma comme un point de repère et à l' exclusion des tissus de bulbus olfactorius.
- Disséquer le striatum comme une tranche de cerveau entre bregma 1,54 et 0,5 et retirer délicatement le tissu cortical.
- Disséquer l'hippocampe, en fixant le cerveau avec l'aiguille à travers le cervelet et le cortex débobinage à partir du lobe occipital.
- Choc-gel disséquée échantillons de cerveau dans de l'azote liquide et conserver à -80 ° C.
2. Préparation de synaptosomes ou encore un Postsynaptique densité (PSD) Fraction enrichie
REMARQUE: Pendant toutes les procédures, garder des échantillons et des tampons à 0 - 4 ° C.Tampons contiennent fraîchement dilués cocktails d'inhibiteurs de la protéase afin de prévenir la dégradation protéolytique de protéines. Si la phosphorylation des protéines est également étudiée, un cocktail d'inhibiteurs de phosphatases doivent être ajoutés. Tous les g-valeurs indiquées sont données à titre g (moyenne) à travers le protocole entier.
- Préparation d'une fraction membranaire brute (figure 3A)
- Transfert disséqué du tissu cérébral dans une cuve d'homogénéisation contenant 1 ml de tampon A refroidi à la glace (5 mM HEPES, 320 mM de saccharose, pH 7,4) et on homogénéise le tissu à 900 tours par minute avec 12 coups.
- Centrifuger les échantillons à 1000 g pendant 10 min. Gardez les surnageants.
- Ré-homogénéiser les pastilles dans les mêmes conditions dans le même volume de tampon d'homogénéisation comme précédemment et les échantillons à centrifuger de nouveau à 1000 g pendant 10 min. Combinez surnageants correspondants. Jeter le pastilles P1, qui contiennent essentiellement les noyaux et les débris cellulaires.
- Faites tourner les surnageants combinés pendant 20 min à 12 000 x g. superna Jeterhabi- ou de l' utilisation pour un fractionnement supplémentaire 11.
- Granulés Remettre en suspension dans le même volume de tampon d'homogénéisation comme précédemment à l'aide de l'homogénéisateur à 6 coups à 900 tours par minute et centrifuger à 12 000 xg pendant 20 min. surnageants Jeter. Les pastilles P2 représentent les fractions de membranes brutes.
- Purification de synaptosomes à partir de fractions de membranes brutes de cerveau (figure 3A)
REMARQUE: Les fractions de membranes brutes de cerveau peuvent être divisés en myéline, la lumière, les membranes et les mitochondries en utilisant des synaptosomes de saccharose à gradient de densité de pas ultracentrifugation. Pour ce pH Tris 5 mM / HCl 8,1 tampons contenant du saccharose à soit 0,32 M, 1,0 M ou 1,2 M de concentration sont nécessaires.- Tout en effectuant la centrifugation pour produire des fractions de P2, de préparer des gradients de saccharose étape dans les tubes d'ultracentrifugation. Commencez avec 2,5 ml 1,0 M tampon de saccharose et sous-couche avec 1,5 ml 1,2 M tampon de saccharose à l'aide d'une pipette Pasteur en verre.
- Re-homogénéiser les fractions P2dans 0,5 ml de tampon 0,32 M de sucrose manuellement avec 6 coups et la charge sur le dessus du gradient.
- Spin à 85.000 xg pendant 2 heures dans une ultracentrifugation en utilisant un rotor à godet oscillant.
- Jeter la couche supérieure à 0,32 M de saccharose, comprenant le matériau à l'interface avec le tampon de saccharose 1,0 M (myéline, des membranes de lumière). Collecter synaptosomes à l'interface 1.0 / 1.2 M tampon de saccharose. Le culot au fond du tube contient des mitochondries.
- Ajouter un tampon 0,32 M de saccharose à la fraction de synaptosomes au rapport 1: 1, mélanger soigneusement et essorage à 150 000 xg pendant 1 h. Synaptosomes sont dans le culot et peuvent maintenant être remises en suspension dans un tampon requise pour un traitement ultérieur.
- Préparation d'une fraction enrichie en PSD (figure 3B)
- Homogénéiser chaque zone spécifique du cerveau à partir d'un seul animal dans un tampon d'extraction 100 pi (5 mM de Tris / HCl pH 8,1, 0,5% de Triton X-100) dans un tube d'ultracentrifugation 200 ul avec un PTFE (polytétrafluoréthylène) pilonà 2000 tpm avec 12 coups.
- Ajouter un tampon d'extraction de 100 pi, mélanger et incuber pendant 1 heure à 4 ° C. Isoler à 100.000 xg pendant 1 h et recueillir le surnageant S1 soigneusement avec une pipette de 200 pi.
- Ré-homogénéiser les pastilles P1 dans le même tube avec 100 ul de tampon d'extraction de nouveau avec un pilon en PTFE à 2000 tours par minute avec 12 coups.
- Ajouter 100 ul de tampon d'extraction et bien mélanger avec une pipette et de spin à 100.000 xg pendant 1 h.
- Combiner le surnageant avec S2 S1 à la fraction de protéine soluble. Cette fraction contient des protéines cytosoliques, 0,5% de Triton X-100 protéines membranaires solubles et des molécules de la matrice extracellulaire.
- Remettre en suspension le culot restant dans 50 ul de 5 mM de Tris / HCl, pH 8,1. Cette fraction contient PSDs, des membranes résistant aux détergents, des éléments du cytosquelette insolubles, les mitochondries et les débris cellulaires, y compris les noyaux. Il est enrichi en PSDs qui forment le noyau des structures post-synaptiques, mais aussi des éléments importants du présynaptiquecytomatrix à la zone active. Le facteur d'enrichissement de PSDs est d'environ 4 et l'enrichissement des composants PSD a été démontré précédemment. 12
3. Préparation d'échantillons pour la spectrométrie de masse
- La lyse et la normalisation échantillon
NOTE: la normalisation échantillon concernant la concentration en protéines est une étape très cruciale pour enfin obtenir des données quantitatives fiables, même pour de faibles changements d'expression de protéines synaptiques.- Dissoudre synaptosomes ou préparations PSD enrichies de chaque zone du cerveau d'un animal dans 20-50 pi (en fonction de la quantité totale de matière: pour cortex auditif avec 5-15 mg utilisation de tissus de 20 pi) d'urée 8 M et incuber sur la glace pendant 1 h dans un bain à ultrasons.
- Pour en gel digérer, dissoudre synaptosomes directement dans le tampon SDS-échantillon. calculer soigneusement le montant chargé pour éviter la surcharge du gel. Considérez que, dans ce cas, l'échafaud protei abondante hautens seront perdues lors de l'électrophorèse sur gel et en le gel digérer.
- On dilue avec 1% d'un détergent amovible pour assurer une concentration finale de 2 M d'urée. Éviter toute température supérieure à 30 ° C pour éviter la carbamylation des protéines.
- Effectuer une SDS-PAGE avec une aliquote (par exemple 10 pi) de l'échantillon selon des procédures standard 13,14.
- Colorer le gel avec du bleu de Coomassie, selon le protocole du fabricant. Le procédé combine l'étape de fixation et coloration avec du methanol et de l'acide acétique.
- Déterminer la densité optique de chaque échantillon pour l'ensemble de la voie avec un scanner de gel calibrée en mode de transmission et calculer la quantité relative de protéine.
- Normaliser les échantillons en fonction de ces calculs.
- Diviser chaque échantillon en deux parties différentes. Utilisez un tiers pour le in-gel digérer et deux tiers pour l'in-solution digest.
- Dissoudre synaptosomes ou préparations PSD enrichies de chaque zone du cerveau d'un animal dans 20-50 pi (en fonction de la quantité totale de matière: pour cortex auditif avec 5-15 mg utilisation de tissus de 20 pi) d'urée 8 M et incuber sur la glace pendant 1 h dans un bain à ultrasons.
- Digérer dans le gel
- séparation de gel
- Effectuer une deuxième SDS-PAGE en utilisant les échantillons de concentration ajustée. Colorer et quantifier les gels pour une deuxième fois pour vérifier la qualité de la normalisation.
- Découper chaque voie d'un échantillon dans le gel dans différentes zones (8 / voie), mais exclut la plage de poids moléculaire supérieur à 170 kDa. Transférer les morceaux de gel dans des tubes séparés.
- Couper les zones en petits morceaux (env. 1 x 1 mm) avec un scalpel pour faciliter dans le gel digestion d'efficacité.
- Digest 15
- Laver les morceaux de gel à plusieurs reprises ( en fonction de l' intensité de coloration) pendant 10 minutes avec 50 - 150 ul d'un tampon constitué de 50% d' acétonitrile (ACN) et 50 mM de carbonate acide d'ammonium (NH 4 HCO 3).
- Retirer surnageants. Recouvrir les morceaux de gel avec ACN et incuber à 20 ° C jusqu'à ce que des morceaux de gel deviennent blancs et se rétrécir.
- Retirez le ACN et réhydrater les morceaux de gel pour 5min avec 50 ul de 0,1 M NH 4 HCO 3. Ajouter le même volume d'ACN et incuber pendant 15 min à 37 ° C.
- Retirer et jeter le liquide complètement. Sécher les morceaux de gel dans une centrifugeuse sous vide.
- Réhydrater morceaux de gel dans 50 pl de NH 4 HCO 3 , contenant 10 mM de dithiothréitol (DTT) et des échantillons de chaleur pendant 45 minutes à 56 ° C pour réduire les résidus de cysteine.
- Retirer surnageants et ajouter 50 ul NH 4 HCO 3 contenant 55 mM d' iodoacétamide (IAA) pendant 30 minutes dans l'obscurité à carbamidomethylate cysteines réduits.
- Retirer et jeter tout le liquide au- dessus des morceaux de gel et les laver deux fois avec 50 ul NH 4 HCO 3 et ACN (1: 1) pendant 10 minutes pour éliminer toute IAA résiduel. sécher les échantillons dans une centrifugeuse sous vide.
- Pour la digestion limitée de protéines ajouter 25 mM NH 4 HCO 3 contenant 12,5 ng / ul de trypsine. Le volume requis dépend de la taille et de la quantité du gel pieces. Incuber pendant quelques minutes et vérifier si le tampon est absorbé. Ajouter plus de mémoire tampon si nécessaire, des morceaux de gel doivent être entièrement recouverts. Incuber à 37 ° C pendant une nuit (min. 12 heures).
- Extraction du peptide
- Morceaux de gel Overlay avec 10 - 20 ul de 25 mM NH 4 HCO 3 et ajouter le même volume d'ACN. Incuber pendant 10 min sur la glace à l'aide de bain à ultrasons. Ensuite, enlever et recueillir les surnageants qui contiennent la plupart des peptides générés.
- Ajouter 100 ul de tampon d'extraction contenant 30% d'acide ACN / 0,1% d'acide trifluoroacétique (TFA) pour les morceaux de gel. Répétez l'incubation dans un bain à ultrasons et recueillir soigneusement ce surnageant.
- Répéter les dernières étapes d'extraction en augmentant la concentration d'ACN à 50%. Après 10 min de bain à ultrasons tourner vers le bas et recueillir des surnageants.
- Combiner les trois surnageants correspondants des étapes d'extraction et les sécher dans une centrifugeuse sous vide. Notez queà la suite de la séparation sur gel des 8 zones par voie / échantillon sont combinés à un échantillon à nouveau dans cette étape.
- séparation de gel
- En solution à digérer
- Digérer
- Utilisez le montant calculé (par exemple 100 pl d'un lysat 150 pi, dépend de la quantité de matière et le volume requis pour la remise en suspension d'un échantillon d'une zone spécifique du cerveau) des échantillons normalisés pour obtenir un matériau de départ suffisant pour au moins trois répétitions techniques à effectuer sans étiquette de spectrométrie de masse.
- Ajouter 2 mM de DTT dans 25 mM NH 4 HCO 3 et vortex doucement l'échantillon. Réduire les échantillons pendant 45 minutes à 20 ° C.
- Ajouter 10 mM IAA à carbamidomethylate les résidus de cystéine. Mélanger et laisser incuber pendant 30 minutes dans l'obscurité à 20 ° C.
- Enfin, ajouter 1 pl d'une solution de trypsine de stock (1 pg / trypsine ul dans 25 mM d'acide acétique) et incuber à 20 ° C pendant 12 heures.
- Extraction en phase solide (SPE) -Purification
- Pour éliminer l'acide détergent clivable, ajuster les échantillons à une concentration finale de 1% de TFA et incuber pendant 1 heure à 20 ° C.
- Centrifuger les échantillons à 16000 xg pendant 10 min et soigneusement recueillir surnageants.
- Placer la colonne SPE dans un rack et équilibrer la matrice avec 2 ml de methanol. Laver deux fois avec 2 ml de 0,1% de TFA dans de l'eau (tampon B).
- Ajouter 2 ml de tampon B et charger l'échantillon. Lavez encore trois fois.
- Éluer les peptides par addition de 200 ul de 70% d'ACN / TFA à 0,1%. Répétez cette étape.
- Réunir les deux éluats et les sécher dans une centrifugeuse sous vide.
- Digérer
- Phospho-peptide enrichissement par chromatographie en TiO 2 16
- Dissoudre des peptides produits par des produits en gel ou en solution à digérer dans 150 ul de 80% d' ACN / TFA à 2,5% (tampon C) et d' équilibrer ~ 2 mg des perles en TiO 2 50pi de tampon C.
- Ajouter des perles à l'échantillon et incuber dans un dispositif rotatif pendant 1 heure à 20 ° C. Ensuite, le spin perles vers le bas (16 000 xg, 1 min) et de recueillir des surnageants.
- Laver les billes trois fois avec 100 pi de tampon C en mélangeant doucement et le filage au bout de 5 min. Collecter les surnageants. Répéter cette étape trois fois avec 100 ul de 80% d'ACN / TFA à 0,1%, suivi par trois lavages avec 100 ul de TFA à 0,1% (sans ACN), respectivement.
- Mélanger tous les dix surnageants, les sécher dans une centrifugeuse à vide et les traiter comme la fraction phospho-peptide appauvri pour une purification supplémentaire selon l' étape 3.5.
- Eluer les phospho-peptides liés avec 20 pi de 400 mM NH 4 OH / 30% d' ACN des perles. Répétez cette étape trois fois et de recueillir tous les surnageants après le filage en bas des perles.
- Combiner les éluats de l'en-gel et la digestion de la solution en produit de digestion d'un échantillon et de les manipuler comme phospho-peptide fraction enrichie. Les sécher dans une centrifugeuse sous vide jusqu'à un volume final de 4 - 8 ul.
- Concentrer et dessalage des fractions phospho-peptide appauvri par des micro-SPE
- Dissoudre les peptides séchés dans 20 ul de TFA à 0,1%.
- Equilibrer fixe C 18 -MATRIX en tirant 20 pi ACN dans la pointe. Laver la matrice en tirant 0,1% de TFA dans de l'eau dans la pointe. Répétez le processus à trois reprises.
- charger lentement échantillon acidifié dans la pointe (répétez cette étape trois fois).
- Laver les temps C 18 -MATRIX trois avec 20 ul de 0,1% de TFA dans de l' eau et jeter la solution de lavage.
- Eluer peptides de la pointe de la pipette par plusieurs reprises (3 fois) dessin 20 pi de 70% ACN / TFA à 0,1% et de recueillir cette solution d'élution dans un tube séparé.
- Combiner les éluats d'un échantillon et les sécher dans une centrifugeuse sous vide.
4. Proteome Analysis
NOTE: Proteome analyse est effectuée sur une double pression piège linéaire ionique hybride / spectromètre de masse Orbitrap équipé d'un HPLC ultra. HPLC est composée d'un auto-échantillonneur refroidi avec une boucle d'injection de 20 ul, une pompe binaire de chargement (plage de débit ul), une pompe de séparation de flux nano binaire, un dispositif de chauffage de la colonne avec deux micro-vannes de commutation et un dégazeur. Les échantillons sont tout d' abord soumis à une colonne de piégeage (par exemple 100 um x 2 cm) à un débit de 7 ul / min , suivie d' une séparation sur une colonne (par exemple , 75 pm x 25 cm) à 250 nl / min. La sortie de la colonne de séparation est directement couplée à une pointe d'émetteur Pico revêtu positionné dans une interface nano-spray à la source spectromètre de masse à ionisation.
- Spectrométrie Nano-chromatographie liquide et masse en tandem
- Dissoudre les échantillons peptidiques dans 12 ul de 2% d'ACN / TFA à 0,1% pendant au moins 30 min. Isoler pendant 15 secondes et transférer 11 ul surnageant à échantillonneur automatique des flacons (conique, diamete réduiter).
- Mettre en place un régime automatisé pour l' application de l' échantillon, la séparation chromatographique et spectrométrie de masse tandem au logiciel de commande (par exemple, Xcalibur) comme suit.
- Utilisez les éléments suivants pour Température: Autosampler: 5 ° C; Colonne four: 45 ° C.
- Utilisez ce qui suit pour injection: Volume: 10 pi; Débit: 7 pi / min (2% ACN, 0,1% de TFA); Durée: 8 min; réglage de la vanne: colonne de piège - déchets; acquisition de spectrométrie de masse: off.
- Utilisez les éléments suivants pour la séparation: Débit: 250 nl / réglage min Valve: colonne colonne de séparation piège; acquisition de spectrométrie de masse: sur.
0 min - 100 min: 2% d'ACN, 0,1% d'acide formique - 40% d'ACN, 0,1% d'acide formique
min 100-105 min: 40% d'ACN, 0,1% d'acide formique - 95% d'ACN, 0,1% d'acide formique
105 min - 109 min: 95% d'ACN, de l'acide formique à 0,1%
109 min - 120 min: 2% d'ACN, de l'acide formique à 0,1% - Utilisez les éléments suivants pour les paramètres de spectrométrie de masse: FTMS;: Full MS résolution 60.000; m / z gamme de 400 - 2000; MME/MS: Iontrap linéaire; seuil minimal de signal 500; largeur d'isolement 2 Da; Temps d'exclusion dynamique réglage 30 sec; ions seuls chargés sont exclus de la sélection; énergie de collision normalisée est réglée à 35% et le temps d'activation de 10 msec.
NOTE: Une analyse complète MS est suivie jusqu'à 15 LTQ MS / MS exécute à l'aide induite par collision de dissociation (CID) des ions peptidiques plus abondamment détectés.
- Exécutez trois répétitions techniques pour tous les échantillons.
- L' identification des protéines et de l' étiquette sans quantification
- Processus données brutes spectrométrie de masse vers l' identification des protéines et la quantification sans étiquette en utilisant une suite commerciale de logiciels (par exemple, PEAKS Studio). Contrairement à la plupart des autres logiciels de protéome ce logiciel particulier utilise un algorithme de novo -sequencing avant alignements de bases de données de protéines. Toutefois, cette étape peut être facilement remplacé par d'autres logiciels populaires.
- Utilisez essparamètres tiels énumérés dans le tableau 2.
- Phospho-protéomique
REMARQUE: l'acquisition de phospho-peptide efficace et fiable nécessite quelques changements essentiels de la configuration du flux de travail protéomique.- Après enrichissement phospho-peptide, les échantillons ne sèche complètement. Toujours garder les échantillons dissous.
NOTE: La liaison phospho-ester de threonines ou sérines phosphorylées est très fragile. Au cours de la fragmentation induite par collision dans le piège à ions cela se traduit par une perte de neutre de phosphate. Cela empêche toute nouvelle fragmentation du peptide, ce qui est nécessaire pour l'identification. Autorisé à large bande-activation dans la configuration de la spectrométrie de masse permet la fragmentation des phospho-peptides , même après une perte neutre du groupe phosphate. Il effectue un gain de "pseudo-MS 3" temps. détermination Phospho-site dans les données MS / MS exige une vérification et une évaluation particulière et peut être effectuée par des luminophores 3.0.
- Après enrichissement phospho-peptide, les échantillons ne sèche complètement. Toujours garder les échantillons dissous.
5. Bioinformatics - Méta-analyse
REMARQUE: Avant d'effectuer l'annotation fonctionnelle et l'analyse de réseau, les listes de protéines doivent être prétraités. Tout d'abord fusionner les listes des protéines régulées et phospho-peptides pour chaque région du cerveau séparément. Retirez ensuite tous les dupliquer UniProt-ID pour chaque fraction pour éviter une mauvaise interprétation.
- Analyse de l' enrichissement Singular avec GeneCodis 17
- Ouvrez l'outil Web de GeneCodis (http://genecodis.cnb.csic.es)
- Sélectionnez "Mus musculus" comme organisme et "GO Processus biologique» comme annotation.
- Coller une liste de UniProt-ID d'une certaine fraction. Soumettre et attendre jusqu'à ce que l'analyse est effectuée. Cliquez sur "Singular Analyse Enrichissement du processus GO biologique» et afficher les résultats.
- Répétez l'étape 5.1.3 pour les trois autres fractions.
- Pour voir toutes les duplications et les intersections entre les listes de résultatsutiliser un langage de script comme Perl ou Python pour filtrer les données nécessaires. Des outils similaires pour une analyse d'enrichissement singulier sont DAVID (https://david.ncifcrf.gov/) et Cytoscape (http://www.cytoscape.org/) avec le PlugIns BINGO (http://apps.cytoscape.org/ apps / bingo) et ClueGO (http://apps.cytoscape.org/apps/cluego).
- Générer un graphique à base de la force sur les données GeneCodis avec Gephi (https://gephi.org/)
NOTE: Les données pour les graphiques doivent être fournies par l'utilisateur, soit dans un format graphique (.gexf, .graphml, .dot, .gv, .gml) ou entré manuellement.- Génération des noeuds du graphe
- A la main: Open Gephi et cliquez sur "des données de laboratoire". Créer des nœuds. Cliquez sur "nœuds" sur la gauche pour passer à la "Nodes" table. Cliquez sur "Ajouter noeud". Entrez le nom du terme. Cliquez sur "OK" / Appuyez sur Entrée.
- Alternative: Save GeneCodis résultat pour PC. Ouvrez le .txt avec un programme de feuille de calcul. Supprimer toutes les lignes except de «Item_Details» (noms de terme). Changer de tête "Item_Details" à "étiquette". Enregistrer la feuille de calcul comme ".csv". Maintenant en Gephi, cliquez sur "Importer une feuille de calcul". Choisissez une feuille de calcul à partir du navigateur de fichier de Gephi. Cliquez sur Suivant". Cliquez sur "Terminer".
- Connexion via les nœuds bords.
- Cliquez sur "Edges" sur la gauche pour passer à la "Edges" table. Pour chaque noeud (Term): rechercher des noms de gènes dans d'autres conditions. Si un ou plusieurs gènes sont partagés -> créer bord.
- Cliquez sur "Ajouter Edge". Sélectionnez "non dirigé". Sélectionnez la source et le noeud cible de listes déroulantes. Cliquez sur "OK" / Appuyez sur Entrée. Si plus d'un gène est partagée, entrez abondance dans "Poids" (tableau).
- Force de base mise en page graphique.
- Ouvrir le fichier de données de graphe, définissez le type de «undirected» ou utiliser les données graphique comme entrée à la main, cliquez sur "Aperçu" sinon Selecte déjàré.
- Redimensionner nœuds en fonction de l'abondance des interconnexions. Cliquez sur les statistiques, exécutez soit "degré moyen" (bords non pondérés) ou "Avg. Degré pondérée" (bords pondérés) sous la rubrique «Vue d'ensemble du réseau". Dans "Apparence", cliquez sur "nœuds", puis sur le bouton de taille, suivant, sélectionnez "Attributs" et définissez le paramètre Attributs pour "Avg. Degree pondérée" ou "degré moyen". Cliquez sur Appliquer.
- Enfin: Sélectionnez "Atlas Force" dans "Layout" et exécuter; changer "la force de Répulsion" si les noeuds sont en collision.
- Exporter vers l'image.
- fonction Capture d'écran: Cliquez sur "Aperçu", le changement graphique mise en page, l'épaisseur de la bordure, la taille de l'étiquette et mise à l'échelle avec le menu en bas de la fenêtre "Graph". Cliquez sur le bouton de la caméra à gauche, et enregistrer l'image.
- fonction d'exportation de "Preview": Cliquez sur "Aperçu". Changer Presets pour "défaut droite". Modifier les paramètress selon les préférences choisies et cliquez sur "SVG / PDF / PNG" à exporter.
- Génération des noeuds du graphe
Résultats
La figure 1 résume le flux de travail complet de quantitative synaptique protéome profilage des régions du cerveau de la souris après auditive apprentissage discrimination. Il commence avec la formation de l'animal dans une boîte de navette. Dans l'exemple illustré à la figure 2, les souris ont commencé à montrer significative la discrimination sonore FM à la session de formation de 4 ème, ce qui indique un apprentissage efficace. Les animaux sont sacrifiés à des moments sélectionnés pour la zone de dissection du cerveau. L'enrichissement requis de synapses peut soit être réalisé par la préparation de synaptosomes , ou encore par la préparation d'une fraction de densité PSD enrichi, tous deux décrits en détail dans la figure 3. Le procédé PSD d' enrichissement a été développé pour des quantités de tissus faible, par exemple 1 - 2 tranches hippocampiques de cerveau de rat 12, 18. Elle exige de petits tubes, des pilons de PTFE raccord à ces tubes, et un laboratoire de forage d'entraînement pour alimenter le pilon.
En raison de la composition de protéine particulière de synaptosomes, il est fortement recommandé d'effectuer la préparation des échantillons de deux manières différentes mais complémentaires. Scellants des PSD sont souvent des protéines de très haut poids moléculaire se produisant en haute stoechiométrie. En solution de digestion est la meilleure façon de les extraire efficacement, mais peut conduire à un suréchantillonnage du mélange de peptide généré. Le gel dans digest réalisée du même échantillon en parallèle peut exclure ces protéines de poids moléculaire élevé et de favoriser l'analyse de protéines avec du milieu et de bas poids moléculaire. Pour une analyse complète des deux types de digestions protéolytiques sont recommandés.
Les différentes quantités de tissus des régions cérébrales étudiées nécessitent un ajustement du matériau appliqué pour une meilleure comparaison. Dans les quatre régions du cerveau étudiées le cortex auditif est généralement le fait de limiterou. Le matériau de toutes les autres régions du cerveau doit être soigneusement ajustée à la quantité du cortex auditif après la préparation de synaptosomes ou de fractions enrichies en PSD (voir 3.1.1.). poids typiques des zones du cerveau fraîchement préparées à partir de souris sont les suivantes: cortex auditif (AC): ~ 50 mg; hippocampe (HIP): ~ 90 mg; striatum (STR): ~ 120 mg et le cortex frontal (FC): ~ 100 mg.
La méthode PSD enrichissement décrite dans la section 2.3 a permis d'identifier environ 1500 différentes protéines et environ 250 différentes phospho-peptides par région du cerveau au niveau d'un seul animal (tableau 1). L' analyse protéomique 24 h après la première session de formation a révélé que 7,3% des protéines identifiées et 5,8% des phospho-peptides ont montré significative (p <0,05) des changements quantitatifs dans leur expression synaptique par rapport aux témoins naïfs (tableau 1). Une tendance remarquable pour regula vers le bastion des échafauds synaptiques peut pointer vers un réarrangement prononcé de l'architecture synaptique au cours des premières étapes de l'apprentissage FMTD. La grande majorité des protéines régulées ont été modifiés d'une manière spécifique à la région du cerveau, alors que seulement 22% ont été trouvés être réglementées dans deux ou plusieurs zones du cerveau. Six exemples choisis sont représentés sur la figure 4.
La méta-analyse des résultats complexes par l'IPA fournit la preuve pour notamment la participation / manipulation des voies canoniques suivantes: "clathrine endocytose Signaling", "axonale Guidance Signaling", "Signalisation de calcium", "RhoA Signaling", "Notch Signaling "," Le remodelage de épithéliale jonctions adhérentes "," glutamate Receptor Signaling "," GABA Receptor Signaling "," dopamine Receptor Signaling »et« Synaptic à long terme Potentialisation ".
Simple analyse de l' enrichissement a révélé des processus biologiques surreprésentés significatifs dans le cortex frontal concernant le transport des protéines, l' adhésion cellulaire, la phosphorylation, l' endocytose, le transport des vésicules médiée, le développement du cerveau antérieur et axonogenesis (Figure 5). Dans les processus biologiques du cortex auditif, y compris le transport des ions, la traduction, le transport de l'ARNm, le transport et l'apprentissage des protéines sont perceptibles. L'analyse de la fraction protéique de l'hippocampe détecte de façon significative les processus enrichis liés au transport d'ions, cycle cellulaire, la traduction, la phosphorylation et le développement du système nerveux. Dans le striatum, surreprésentés processus biologiques, y compris le transport de l'ARNm, le transport vésiculaire à médiation axonogenesis, la protéolyse, le transport de la protéine et ont été trouvés endocytose.
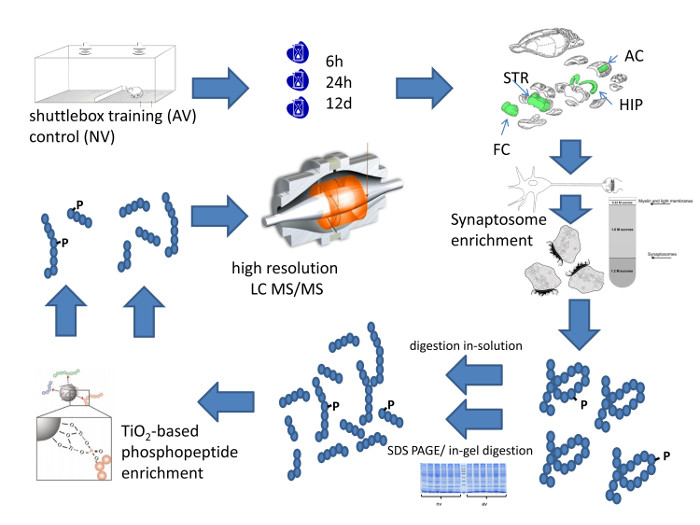
Figure 1: Systematic WorkFlow de l'approche méthodologique. Ce chiffre résume schématiquement le flux de travail de haute résolution profilage quantitative de la composition des protéines synaptiques spécifiques de la zone du cerveau. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 2: Exemple de la performance des souris dans le Tone FM Discrimination Task. Les animaux montrent une augmentation du taux de succès (courbe bleue) et un taux décroissant de fausses alarmes (courbe noire) dans le cadre de sessions de formation. discrimination significative se produit à partir de la quatrième session. Les barres d'erreur sont fournis à titre SEM. S'il vous plaît cliquer ici pour voir une larger version de ce chiffre.

Figure 3: Préparation des synaptosomes et la fraction enrichie en PSD. A: préparation synaptosomes. B: préparation de la fraction PSD enrichi. Les deux chiffres expliquent le flux de travail détaillé de préparation de synaptosomes ou fractions alternativement PSD enrichies de tissus cérébraux. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
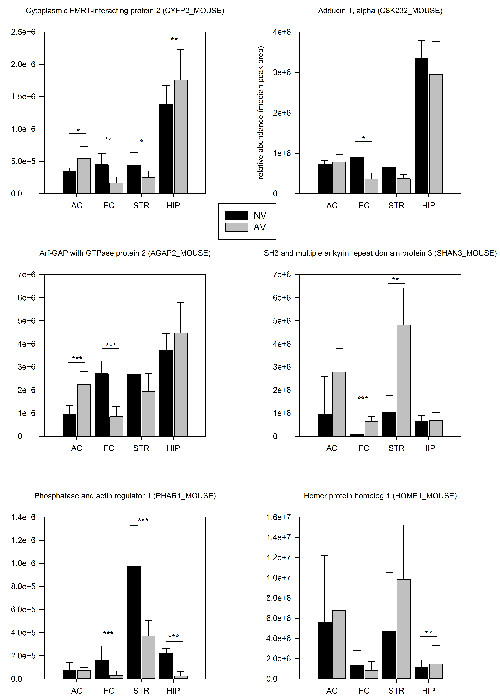
Figure 4: Résultats choisis protéomique quantitative. Les abondances synaptiques relatifs des protéines sélectionnées sont comparées entre tra sourisiné sur la tâche FMTD (AV, n = 6) et les souris témoins naïfs (NV, n = 6) 24 heures après la première session de formation. Les valeurs d'abondance ont été calculés comme médiane des surfaces de pic des trois peptides les plus intenses d'une protéine. Les protéines avec des modifications importantes de l'abondance (AV / NV; t-test) sont marqués dans les parcelles: * p <0,05, ** p <0,01, *** p <0,005. Les barres d'erreur sont fournis à titre SD. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 5: Visualisation des voies biologiques pour Cortex Frontal par GeneCodis / Gephi. Seuls les termes importants de la base de données (http://geneontology.org) liée à "processus biologique" avec un certain nombre de protéines minimum de trois Gene Ontology (GO) sont présentés ici. Les nœuds représentent des termes GO, la taille du noeud, la largeur de la ligne et le nombre de connexions d'un certain noeud représentent le nombre de protéines, qui partagent ce terme GO avec d'autres nœuds. En raison de la méthode "Atlas Force" de Gephi, nœuds associés se regroupent en étroite collaboration. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
| Région du cerveau | AC | FC | HANCHE | 000 "face =" "size =" Calibri 3 "> STR | Σ |
| protéines identifiées | 1435 | 1758 | 1572 | 1507 | 6272 |
| des protéines régulées (p <0,05) | 59 | 130 | 162 | 108 | face = "Calibri" size = "3"> 459 |
| ↑ AV / NV | 8 | 4 | 76 | 35 | 123 |
| ↓ AV / NV | 51 | 126 | 86 | 73 | 336 |
| phosphomoti identifié fs | 197 | 361 | 273 | 278 | 1109 |
| phosphomotifs régulés (p <0,05) | 8 | 22 | 21 | 14 | 65 |
| ↑ AV / NV | 4 | 00000 "face =" "size =" Calibri 3 "> 17 | 5 | 9 | 35 |
| ↓ AV / NV | 4 | 5 | 16 | 5 | 30 |
Tableau 1: Résumé d'un Résultat protéomique. Ce tableau résume une expérience protéomique représentant de souris formés (AV, n = 6) 24 heures après la première session de formation par rapport à leurs contrôles naïfs (NV, n = 6). lesomme de 459 protéines réglementées comprend des règlements qui se chevauchent. 283 règlements différents ont été déterminés comme spécifique du cerveau. Dans le détail, 57 protéines sont réglementées dans deux régions du cerveau, 18 règlements de protéines ont été détectées dans trois régions du cerveau et seulement 2 protéines sont réglementées dans les quatre régions du cerveau étudiées.
| Tolérances d'erreur | |
| masse précurseur (transformation de Fourier spectrométrie de masse) | 10 ppm |
| masse fragment d'ions (piège à ions linéaire) | 0,6 Da |
| Maximum manqués par clivages peptide | 3 |
| modifications fixes | |
| pour en gel digéré des échantillons | Carbamidomethylation de Cystéine |
| pour les échantillons dans la solution digérée | MethylthiolatioN Cystéine |
| modifications variables | L'oxydation de la methionine |
| Déamidations de l'asparagine et / ou Glutamine | |
| Base de données | Uniprot / Sprot |
| Taxonomie | Souris |
| Paramètres statistiques identification-acceptation | |
| de novo confiance moyenne locale (ALC) | > 50% |
| Peptide-faux taux de découverte (FDR, sur la base est. Leurre-fusion) | <1% |
| Protein importance (-10logP, basée sur T-test modifié) | > 20 |
| peptides uniques / protéines | ≥ 1 |
| Paramètres de quantification: | |
| Peptides utilisés pour la quantification si: | |
| Peptide signification (-10logP) | > 30 |
| l'identification des peptides dans | ≥ 50% des échantillons |
| Peptide qualité du signal | > 1 |
| superficie moyenne Peptide | > 1E5 |
| Peptide tolérance au temps de rétention | <5 min |
| Normalisation | par le courant ionique total (CIT) |
Tableau 2: Réglages pour la protéine d' identification (étape 4.2.2).
Discussion
L'étude présente un flux de travail optimisé méthodologique pour un profilage quantitative précise des changements d'expression des protéines synaptiques lors de l'apprentissage et de consolidation de la mémoire dans les différentes zones du cerveau de souris. La configuration offre la possibilité d'étudier l'expression de la protéine au niveau d'un seul animal en dépit de l'application requise d'au moins trois répétitions techniques par échantillon pour analyse par spectrométrie de masse.
La méthodologie tient compte de la composition protéique particulière de la pré- et postsynapse constitué par les protéines de haut poids moléculaire, mais aussi d'échafaudage de protéines de médiateur important portant des masses moléculaires moyennes ou inférieures. Les digestions en solution de préparations synaptosomiques se traduisent par une production efficace et, par conséquent, une sur-représentation des peptides d'échafaudage dérivés. Ceci, à son tour, peut supprimer l'analyse des protéines abondantes plus petites ou plus faibles. La préparation suggérée de fractions SDS-PAGE à partir d'unaliquote de chaque échantillon combiné avec un procédé de digestion dans le gel en parallèle facilite l'analyse des protéines d'abondance moyenne et faible et représente une méthode complémentaire fortement recommandée. Après l' application de la spectrométrie de masse distincte de toutes les fractions provenant d'un échantillon (par exemple en solution de digestion, en gel digestion, les fractions phospho enrichi combinés) , les ensembles de données MS / MS correspondantes peuvent être combinées et en outre calculés pour l' identification des protéines et à la quantification par des pics logiciels ou progiciels populaires alternatives.
En variante, l'application individuelle des fractions en gel digestion dérivé d'un échantillon (gel-zones traitées séparément d'un échantillon voie) et les fractions générées de l'échantillon en solution digérée (par exemple , par chromatographie par échange d'ions) à la spectrométrie de masse peut accroître la profondeur d'analyse. Cependant, ce flux de travail prolongée augmente considérablement le temps requis pour l'acquisition de données LS-MS / MS. Pour generation d'une séquence de réarrangements moléculaire détaillée de protéines synaptiques lors de l'apprentissage et de formation de la mémoire un décours temporel spécifié du profil protéomique est nécessaire. Ce cours de temps peut commencer immédiatement après ou même pendant la première session de formation et couvre un laps de temps à mailles serrées jusqu'à ce que la performance des animaux a atteint le niveau asymptotique de la courbe d'apprentissage après env. 8 - 10 jours de formation (voir la figure 2 pour plus de détails).
L'analyse des changements de phosphorylation de protéines synaptiques nécessite un accent particulier sur les délais sélectionnés lors de l'apprentissage FMTD. Sur les cascades de signalisation d'une part, initiant des réarrangements de protéines synaptiques connues pour être déclenché par phosphorylations de protéines et déphosphorylations sont attendus à un stade très précoce de la formation des animaux. D'autre part, il y a des modifications durables de multiples protéines synaptiques phosphorylés connues qui régulent la connectivité et l'assemblage dans les sl' architecture ynaptic 19, 20. Ces modifications post-traductionnelles sont attendus même aux points de temps ultérieurs de consolidation de la mémoire.
Les ensembles de données complexes générées par ce flux de travail protéomique nécessitent un traitement bioinformatique pour identifier les participants voies moléculaires et des molécules clés. La méta-analyse montre les voies surreprésentés importantes, qui jouent un rôle dans les processus d'apprentissage et de mémoire.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
We wish to thank Yvonne Ducho and Kathrin Pohlmann for excellent technical assistance. This work was supported by the Deutsche Forschungsgemeinschaft (SFB 779) and by the State Saxony-Anhalt / European Regional Development Fund (ERDF) via the Center for Behavioral Brain Sciences (CBBS).
matériels
| Name | Company | Catalog Number | Comments |
| 3 M Empore Solid Phase Extraction- Filter | 3M Bioanalytical Technologies | 4245SD | 7 mm/3 ml |
| Acclaim PepMap 100 | Dionex/Thermo Scientific | 164564 | 100 µm x 2 cm, C18 |
| Acclaim PepMap 100 | Dionex/Thermo Scientific | 164569 | 75 µm x 25 cm, C18 |
| Acetic acid | Carl Roth GmbH | 3738.1 | |
| Acetonitrile (ACN) | Carl Roth GmbH | AE70.2 | |
| Acrylamide (30%) | AppliChem | A0951 | |
| Ammonium hydrogen carbonate | Fluka | 9830 | |
| Ammonium hydroxide | Fluka | 44273 | |
| Ammonium persulfate (APS) | AppliChem | A2941 | |
| Biofuge pico | Heraeus GmbH | 75003280 | |
| Blue R-250 | SERVA Electrophoresis GmbH | 17525 | |
| Bromophenol Blue | Pharmacia Biotech | 17132901 | |
| C57BL/6J mice | Charles River | ||
| Cantharidin | Carl Roth GmbH | 3322.1 | |
| Centrifuge tubes for MLS-50 | Beckman Coulter | 344057 | |
| Centrifuge tubes for TLA 100.1 rotor | Beckman Coulter | 343776 | |
| Dithiothreitol (DTT) | AppliChem | A1101 | |
| Eppendorf 5417R centrifuge | VWR | 22636138 | |
| Eppendorf A-8-11 rotor | VWR | 5407000317 | |
| Formic acid | Fluka | 14265 | |
| GeneCodis | http://genecodis.cnb.csic.es/ | ||
| Gephi | https://gephi.org/ | ||
| Glycerol | AppliChem | A1123 | |
| Glycine | AppliChem | A1067 | |
| HALT Phosphatase Inhibitor Cocktail | Pierce /Thermo Scientific | 78420 | |
| HEPES Buffer solution | PAA Laboratories GmbH | S11-001 | |
| Homogenization vessel 2 ml | Sartorius AG | 854 2252 | |
| Hydrochloric acid | Sigma-Aldrich | H1758 | |
| Imidazole | Sigma-Aldrich | I2399 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide (IAA) | Sigma-Aldrich | I1149 | |
| Laboratory drilling drive K-ControlTLC 4957 | Kaltenbach & Vogt GmbH | 182997 | |
| LTQ Tune Plus 2.7.0.1112 SP2 | Thermo Scientific | ||
| LTQ Orbitrap Velos Pro | Thermo Scientific | ||
| Macs-mix tube rotator | Miltenyi Biotech | 130-090-753 | |
| Magic Scan 4.71 | UMAX | ||
| Methanol | Carl Roth GmbH | AE71.2 | |
| MLS-50 rotor | Beckman Coulter | 367280 | |
| Optima MAX Ultracentrifuge | Beckman Coulter | 364300 | |
| PageRuler Prestained Protein Ladder | Thermo Scientific | 26616 | |
| PEAKS 7.5 | Bioinformatic Solutions | ||
| Phosphatase Inhibitor Cocktail 3 | Sigma-Aldrich | P0044 | |
| PhosphoRS 3.1 | IMP/IMBA/GMI | ||
| PhosSTOP | Roche | 4906845001 | |
| Plunger/pestle made of PTFE | Sartorius AG | 854 2651 | |
| PotterS homogenizer | Sartorius AG | 853 3024 | |
| Protease Inhibitor complete mini | Roche | 4693159001 | |
| Quantity One 4.5.1 | BioRad | ||
| RapiGest | Waters | 186002122 | |
| Shuttle box | Coulbourne Instruments | ||
| Sodium dodecylsulfate (SDS) | AppliChem | A1112 | |
| Sodium molybdate | Carl Roth GmbH | 274.2 | |
| Sodium tartrate dihydrate | Sigma-Aldrich | 228729 | |
| SONOREX RK 156 Ultrasonic Bath | BANDELIN electronic GmbH & Co. KG | 305 | |
| Soundproof chamber | Industrial Acoustics Company | ||
| Sucrose | Carl Roth GmbH | 4621.2 | |
| Tetramethyl ethylene -1,2-diamine (TEMED) | Sigma-Aldrich | T9281 | |
| Thermomixer basic | CallMedia | 111000 | |
| Titansphere TiO 5µm | GL Sciences Inc. Japan | 502075000 | |
| TLA 100.1 rotor | Beckman Coulter | 343840 | |
| Trifluoro acetic acid (TFA) | Sigma-Aldrich | T6508 | |
| Tris ( hydroxymethyl) aminomethane (TRIS) | AppliChem | A1086 | |
| Triton X-100 | Sigma-Aldrich | T8532 | |
| Trypsin Gold | Promega | V5280 | |
| Ultimate 3000 Ultra HPLC | Dionex/Thermo Scientific | ||
| Ultracentrifuge tube | Beckman Coulter | 343776 | |
| Unijet II Refrigerated Aspirator | Uniequip Laborgeräte- und Vertriebs GmbH | ||
| UNIVAPO 100 H Concentrator Centrifuge | Uniequip Laborgeräte- und Vertriebs GmbH | ||
| Urea | AppliChem | A1049 | |
| Water (high quality purifed) | Resistivity: > 18.2 MΩ*cm at 25 °C Pyrogens: < 0.02 EU/ml TOC: < 10 ppb | ||
| Xcalibur 3.0.63 | Thermo Scientific | ||
| ZipTipC18 Pipette Tips | MILLIPORE | ZTC18S960 |
Références
- Lamprecht, R., LeDoux, J. Structural plasticity and memory. Nat Rev Neurosci. 5 (1), 45-54 (2004).
- Bingol, B., Schuman, E. M. Synaptic protein degradation by the ubiquitin proteasome system. Curr Opin Neurobiol. 15 (5), 536-541 (2005).
- Richter, J. D., Klann, E. Making synaptic plasticity and memory last: mechanisms of translational regulation. Genes Dev. 23 (1), 1-11 (2009).
- Rosenberg, T., et al. The roles of protein expression in synaptic plasticity and memory consolidation. Front Mol Neurosci. 7, 86 (2014).
- Scheich, H., et al. Behavioral semantics of learning and crossmodal processing in auditory cortex: the semantic processor concept. Hear Res. 271 (1-2), 3-15 (2011).
- Kähne, T., et al. Synaptic proteome changes in mouse brain regions upon auditory discrimination learning. Proteomics. 12 (15-16), 2433-2444 (2012).
- Reichenbach, N., et al. Differential effects of dopamine signalling on long-term memory formation and consolidation in rodent brain. Proteome Sci. 13, 13 (2015).
- Kähne, T., et al. Proteome rearrangements after auditory learning: high-resolution profiling of synapse-enriched protein fractions from mouse brain. J Neurochem. , (2016).
- Li, K., et al. Organelle proteomics of rat synaptic proteins: correlation-profiling by isotope-coded affinity tagging in conjunction with liquid chromatography-tandem mass spectrometry to reveal post-synaptic density specific proteins. J Proteome Res. 4 (3), 725-733 (2005).
- Carlin, R. K., Grab, D. J., Cohen, R. S., Siekevitz, P. Isolation and characterization of postsynaptic densities from various brain regions: enrichment of different types of postsynaptic densities. J Cell Biol. 86 (3), 831-845 (1980).
- Smalla, K. H., Klemmer, P., Wyneken, U., Dermietzel, R. . The Cytoskeleton - Imaging, Isolation, and Interaction. , 265-282 (2012).
- Smalla, K. H., et al. The synaptic glycoprotein neuroplastin is involved in long-term potentiation at hippocampal CA1 synapses. Proc Natl Acad Sci U S A. 97 (8), 4327-4332 (2000).
- Shapiro, A. L., Vinuela, E., Maizel, J. V. Molecular weight estimation of polypeptide chains by electrophoresis in SDS-polyacrylamide gels. Biochem Biophys Res Commun. 28 (5), 815-820 (1967).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Shevchenko, A., Wilm, M., Vorm, O., Mann, M. Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. Anal Chem. 68 (5), 850-858 (1996).
- Thingholm, T. E., Larsen, M. R., Ingrell, C. R., Kassem, M., Jensen, O. N. TiO(2)-based phosphoproteomic analysis of the plasma membrane and the effects of phosphatase inhibitor treatment. J Proteome Res. 7 (8), 3304-3313 (2008).
- Carmona-Saez, P., Chagoyen, M., Tirado, F., Carazo, J. M., Pascual-Montano, A. GENECODIS: a web-based tool for finding significant concurrent annotations in gene lists. Genome Biol. 8 (1), 3 (2007).
- Bonn, S., Seeburg, P. H., Schwarz, M. K. Combinatorial expression of alpha- and gamma-protocadherins alters their presenilin-dependent processing. Mol Cell Biol. 27 (11), 4121-4132 (2007).
- Tweedie-Cullen, R. Y., Reck, J. M., Mansuy, I. M. Comprehensive mapping of post-translational modifications on synaptic, nuclear, and histone proteins in the adult mouse brain. J Proteome Res. 8 (11), 4966-4982 (2009).
- Li, L., et al. The differential hippocampal phosphoproteome of Apodemus sylvaticus paralleling spatial memory retrieval in the Barnes maze. Behav Brain Res. 264, 126-134 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon