Method Article
Alta resolução quantitativa Synaptic Proteome Profiling de rato regiões cerebrais Depois Auditivo Discriminação Aprendizagem
Neste Artigo
Resumo
The identification of molecules and pathways controlling synaptic plasticity and memory is still a major challenge in neuroscience. Here, a workflow is described addressing the relative quantification of synaptic proteins supposedly involved in the molecular reorganization of synapses during learning and memory consolidation in an auditory learning paradigm.
Resumo
The molecular synaptic mechanisms underlying auditory learning and memory remain largely unknown. Here, the workflow of a proteomic study on auditory discrimination learning in mice is described. In this learning paradigm, mice are trained in a shuttle box Go/NoGo-task to discriminate between rising and falling frequency-modulated tones in order to avoid a mild electric foot-shock. The protocol involves the enrichment of synaptosomes from four brain areas, namely the auditory cortex, frontal cortex, hippocampus, and striatum, at different stages of training. Synaptic protein expression patterns obtained from trained mice are compared to naïve controls using a proteomic approach. To achieve sufficient analytical depth, samples are fractionated in three different ways prior to mass spectrometry, namely 1D SDS-PAGE/in-gel digestion, in-solution digestion and phospho-peptide enrichment.
High-resolution proteomic analysis on a mass spectrometer and label-free quantification are used to examine synaptic protein profiles in phospho-peptide-depleted and phospho-peptide-enriched fractions of synaptosomal protein samples. A commercial software package is utilized to reveal proteins and phospho-peptides with significantly regulated relative synaptic abundance levels (trained/naïve controls). Common and differential regulation modes for the synaptic proteome in the investigated brain regions of mice after training were observed. Subsequently, meta-analyses utilizing several databases are employed to identify underlying cellular functions and biological pathways.
Introdução
A aprendizagem é baseado na formação de traços de memória e a sua manutenção. É amplamente aceito que um mecanismo subjacente pode representar uma formação de novo e / ou rearranjo dos contatos sinápticos existentes entre os neurônios dependente de atividade. Ao nível molecular, várias modificações de proteínas, relocalizações subcelulares e mudanças no volume das proteínas sinápticas foram descritos 1-4 (Lamprecht, 2004 # 8). No entanto, a maioria dos estudos até agora focada em proteínas selecionados, em vez de sobre a composição proteoma sináptica global, mas complexa. A presente abordagem permite uma seleção imparcial por mudanças proteoma sinápticas em regiões do cérebro do rato após uma experiência de aprendizagem. Ele é adequado para tornar ponto de tempo instantâneos moleculares dependentes da reorganização induzida-learning da arquitetura sináptica. O fluxo de trabalho descrito requer um trabalho em equipe específica de diversos especialistas em comportamento animal, bioquímica de proteínas, espectrometria de massa e bioinformatics.
O paradigma de aprendizagem escolhida, ou seja, tom de discriminação de frequência modulada (FMTD), é uma tarefa de discriminação auditiva bem caracterizado em roedores 5. Aprendizagem e formação da memória de longo prazo nesta caixa de transporte Go / No-Go-tarefa envolve mecanismos dependendo aumentou sinalização de dopamina cortical e síntese de proteínas. Assim, estudos recentes proteômicas sobre gerbils e ratos revelaram dopamina e rearranjos de plástico induzida por aprendizagem dos componentes sinápticas em cortical, mas também em regiões cerebrais mais basais que supostamente interagir durante a aprendizagem e memória FMTD 6-8. Isto ilustra que a formação da memória envolve uma complexa interacção de várias regiões do cérebro e, assim, podem ser regulados diferencialmente dentro destas regiões sobre o nível do proteoma. Portanto, o esvaziamento das regiões do cérebro do rato corticais e subcorticais seleccionadas incluído no fluxo de trabalho.
Além disso, o characterizati fiávelno mesmo de alterações fracas na composição de proteína sináptica requer um enriquecimento de compartimentos pré e pós-sinápticos, ao invés da análise de homogenatos de membrana em bruto ou fracções 9. Protocolos Portanto, a preparação de sinaptossomas utilizando estabelecida antes de proteomic análise é descrito, a fim de aumentar o nível de detecção e a faixa dinâmica para proteínas específicas de sinapse 10,11.
Um pré-requisito essencial para utilizar a espectrometria de massa de alta resolução livre de marcador para fins quantitativos é um elevado grau de semelhança das amostras de proteína. Como são esperadas mudanças em vez menores na composição de proteína sináptica a ocorrer depois de saber, uma abordagem livre de etiqueta será adequado para comparar amostras de proteínas obtidos a partir de ratos treinados e ingênuo correspondente. Alternativamente, as estratégias de etiquetas de condições específicas de proteínas / peptídeos usando isótopos estáveis (por exemplo, TMT, iTRAQ, ICPL e SILAC), bem como qua livre rótulo baseado em MS2ntification (SWATH) são úteis, mas eles são mais caros do que a abordagem sem rótulo escolhido ou precisa de hardware especial de espectrometria de massa.
Desde rastreios proteômicas, muitas vezes produzir conjuntos de dados complexos, processamento de bioinformática é recomendado para interpretação de dados apropriado. meta-análises adicionais podem apoiar uma melhor compreensão dos mecanismos moleculares subjacentes potenciais alterações relacionadas com o paradigma e a identificação de processos celulares chave envolvidas e vias de sinalização. metodologias adequadas também são descritos abaixo.
Protocolo
Todos os procedimentos, incluindo indivíduos animais foram realizados de acordo com os regulamentos da Lei Federal alemão, os respectivos regulamentos da UE e as diretrizes do NIH, e ter sido aprovado pelo comitê de ética da Landesverwaltungsamt Sachsen / Anhalt (42502-2-1102 IFN).
1. Aprendizagem Auditivo
- Aprendizagem discriminação auditiva na caixa de transporte (FMTD paradigma) Nota: Use sempre luvas ao manusear os ratos.
- camundongos Casa C57BL6 / J em grupos de três ou quatro com livre acesso a pelotas de alimentos e água da torneira em gaiolas de policarbonato transparente. Manter uma luz de 12 horas: ciclo escuro no biotério. Se os animais forem recebidos de um outro laboratório ou de uma empresa de permitir que pelo menos uma semana de aclimatação e estabelecendo-se em.
- Execute uma sessão de treinamento caixa de shuttle por dia.
- Leve o mouse de sua gaiola no biotério e colocá-lo em uma caixa de transporte mal iluminada dentro de um som câmara de prova.
- Use uma agenda de aprendizado totalmente controlado por computador para a aprendizagem discriminação auditiva. Comece com um período de habituação de 3 min de silêncio, e então começar a sessão de treino.
- Utilize sequências do tom crescente (4-8 kHz, CS +) como o Go-estímulo durante a Go-ensaios: O animal tem de atravessar a barreira dentro de 6 segundos de apresentação de tom (resposta correta, hit). Punir uma falta por um pé-leve choque de 50 - 300 mA, entregues através do chão de grade da caixa de shuttle.
- Use sequências da queda tom (8-4 kHz, CS-) como o No-Go-estímulo durante a no-go-julgamentos: O animal tem de permanecer no compartimento atual da caixa de shuttle durante a 6 segundos de apresentação tom. Punir um alarme falso por um pé-leve choque de 50 - 300 mA, entregues através do chão de grade da caixa de shuttle.
- Use intervalos entre tentativas de 20 5 seg.
- Execute 30 Vão-julgamentos e 30 no-go-julgamentos por sessão em uma ordem pseudo-randomizados, de modo que uma session consiste de 60 ensaios e dura cerca de 25 min.
- Coloque o animal treinado para trás em sua gaiola no biotério.
- dissecção do cérebro
- Eutanásia o animal no ponto de tempo desejado, após um número desejado de sessões de treino utilizando deslocamento cervical (por exemplo, 24 horas após a conclusão da primeira sessão). Decapitar o animal.
- Dissecar rapidamente o cérebro através dos seguintes passos: primeiro corte na pele e, em seguida, o crânio com uma tesoura reta ao longo da sagittalis Sutura. Remover completamente as partes do osso que cobre o tecido cerebral usando fórceps fortes. Retire o cérebro com uma spattle.
- Por dissecção, colocar cérebro para uma placa de Petri cheio de gelo. Dissecar o córtex auditivo, o córtex frontal, do corpo estriado e hipocampo sob um microscópio estereoscópico, usando um bisturi e uma agulha.
- Localizar o córtex auditivo usando marcos visuais na s cérebrourface, tais como os vasos sanguíneos e a forma da superfície (bregma -2,06 a -3,4, tamanho rostrocaudal 2 mm, dorsoventral 1,3 mm) e bilateralmente dissecar como um bloco rectangular de tecido com a espessura do córtex.
- Dissecar o córtex frontal como uma fatia do cérebro entre bregma 3,56 e 1,54 usando o opticum chiasma como um marco e excluindo tecido do olfactorius bulbo.
- Dissecar o corpo estriado como uma fatia do cérebro entre bregma 1,54 e 0,5 e remova cuidadosamente tecido cortical.
- Dissecar o hipocampo, pela fixação do cérebro com a agulha através do cerebelo e desenrolar-se o córtex começando no lobo occipital.
- Shock-congelamento dissecados amostras de cérebro em azoto líquido e armazenar a -80 ° C.
2. Preparação de sinaptossomas ou, alternativamente, um pós-sináptico densidade (PSD) Fracção enriquecida
NOTA: Durante todos os procedimentos, manter as amostras e tampões a 0-4 ° C.Os tampões contêm cocktails de inibidores da protease diluída de fresco, a fim de evitar a degradação proteolítica de proteínas. Se a fosforilação da proteína é também estudada, cocktail inibidor de fosfatase tem que ser adicionado. Todos os valores de G indicada são dados como g (médio) durante todo o protocolo.
- Preparação de uma fracção de membrana em bruto (Figura 3A)
- Transferência dissecados de tecido cerebral dentro de um recipiente de homogeneização contendo 1 mL de gelo frio de tampão A (HEPES a 5 mM, sacarose 320 mM, pH 7,4) e homogeneizar o tecido a 900 rpm com 12 pancadas.
- Centrifugar as amostras a 1000 xg durante 10 min. Mantenha os sobrenadantes.
- Re-homogeneizar peletes nas mesmas condições, para o mesmo volume de tampão de homogeneização como antes e as amostras Centrifugar novamente a 1000 xg durante 10 min. Combinar os sobrenadantes correspondentes. Descartar o P1 pelotas, que contêm principalmente os núcleos e os resíduos celulares.
- Girar os sobrenadantes combinados durante 20 min a 12.000 x g. superna Rejeitartes ou utilização para posterior fracionamento 11.
- Ressuspender as pelotas no mesmo volume de tampão de homogeneização como anteriormente usando o homogeneizador com 6 batidas a 900 rpm e centrifugação a 12.000 xg durante 20 min. sobrenadantes de descarte. Os peletes P2 representam as fracções de membrana em bruto.
- A purificação de fracções de membrana a partir de sinaptossomas de cérebro em bruto (Figura 3A)
Nota: As fracções de membrana de cérebro em bruto pode ser separado em mielina, membranas leves, sinaptossomas e mitocôndrias utilizando gradiente de densidade de sacarose passo de ultracentrifugação. Para este valor de pH de Tris 5 mM / HCl a 8,1 são necessários tampões contendo sacarose a 0,32 M tanto, a concentração de 1,0 M ou 1,2 M.- Durante a realização da centrifugação para produzir as fracções P2, preparar passos de gradientes de sacarose nos tubos de ultracentrífuga. Comece com 2,5 ml de 1,0 M de tampão de sacarose e subcamada com 1,5 ml de 1,2 M de tampão de sacarose utilizando uma pipeta de Pasteur de vidro.
- Re-homogeneizar as fracções P2em 0,5 ml de tampão de 0,32 M de sacarose com 6 manualmente golpes e a carga no topo do gradiente.
- Giram a 85.000 xg durante 2 horas em uma ultracentrífuga usando um rotor basculante.
- Rejeitar a camada de topo de sacarose 0,32 M, incluindo o material na interface para o tampão a 1,0 M de sacarose (mielina, membranas leves). Recolhe sinaptossomas na interface tampão de sacarose 1,0 / 1,2 M. O sedimento no fundo do tubo contém mitocôndrias.
- Adicionar tampão de 0,32 M de sacarose para a fracção sinaptosomal em proporção de 1: 1, mistura-se cuidadosamente e centrifugação a 150000 xg durante 1 h. Os sinaptossomas são em pelete e agora pode ser ressuspensa num tampão necessária para posterior processamento.
- Preparação de uma fracção enriquecida em PSD (Figura 3B)
- Homogeneizar cada área específica do cérebro a partir de um único animal, em 100 ul de tampão de extracção (5 mM Tris / HCl pH 8,1, 0,5% de Triton X-100) em um tubo de ultracentrífuga 200 ul com um (politetrafluoretileno) pilão de PTFEa 2.000 rpm com 12 pancadas.
- Adicionar 100 uL de tampão de extracção, misturar e incubar durante 1 hora a 4 ° C. Girar a 100.000 xg durante 1 hora e recolher o sobrenadante S1 cuidadosamente com uma pipeta de 200 uL.
- Re-homogeneizar sedimento P1 no mesmo tubo com 100 ul de tampão de extracção de novo com um pilão de PTFE a 2.000 rpm com 12 pancadas.
- Adicionar 100 ul de tampão de extracção e misturar bem com uma pipeta e centrifugação a 100.000 xg durante 1 h.
- Combinar o sobrenadante S2 com S1 para a fracção de proteína solúvel. Esta fracção contém proteínas citosólicas, proteínas de membrana solúvel em 0,5% de Triton X-100 e as moléculas da matriz extracelular.
- Ressuspender o sedimento restante em 50 ul de Tris 5 / HCl mM pH 8,1. Esta fracção contém PSDs, membranas resistentes a detergente, elementos do citoesqueleto insolúveis, mitocôndrias e detritos celulares incluindo núcleos. É enriquecida em PSDs que formam o núcleo de estruturas pós-sinápticas, mas também de partes importantes do pré-sinápticacytomatrix na zona ativa. O factor de enriquecimento de PSDs é de cerca de 4 e o enriquecimento dos componentes do PSD foi demonstrada anteriormente. 12
3. Preparação de Amostras para Espectrometria de Massa
- Lise e normalização de exemplo
NOTA: a normalização da amostra relativa à concentração de proteína é um passo muito importante para alcançar, finalmente, os dados quantitativos confiáveis, mesmo para fracas mudanças de expressão de proteínas sinápticas.- Dissolve-se sinaptossomas ou preparações PSD-enriquecidos de cada área do cérebro de um animal em 20-50 ul (dependente da quantidade total de material: para córtex auditivo com 5-15 mg de utilização de tecido de 20 ul) de ureia 8 M e incubar em gelo durante 1 h num banho de ultra-sons.
- Para digerir em gel, dissolver sinaptosomas directamente no tampão de amostra SDS. calcular cuidadosamente a quantidade carregada para evitar a sobrecarga do gel. Considere-se que, neste caso, o elevado protei andaime abundanteNS serão perdidos durante a electroforese em gel e em gel de digerir.
- Dilui-se com 1% de um detergente removível para assegurar uma concentração final de 2 M de ureia. Evitar qualquer temperatura superior a 30 ° C para evitar carbamila�o proteína.
- Realizar SDS-PAGE com uma alíquota (por exemplo, 10 ul) da amostra de acordo com os procedimentos padrão 13,14.
- Corar o gel com Azul de Coomassie de acordo com o protocolo do fabricante. O procedimento combina o passo de fixação e coloração com metanol e ácido acético.
- Determinar a densidade óptica de cada amostra para toda a pista com um leitor de gel calibrada em modo de transmissão e calcular a quantidade relativa de proteína.
- Normalizar as amostras de acordo com esses cálculos.
- Dividir cada amostra em duas partes diferentes. Use um terceiro para a in-gel digerir e dois terços para a in-solução para análise.
- Dissolve-se sinaptossomas ou preparações PSD-enriquecidos de cada área do cérebro de um animal em 20-50 ul (dependente da quantidade total de material: para córtex auditivo com 5-15 mg de utilização de tecido de 20 ul) de ureia 8 M e incubar em gelo durante 1 h num banho de ultra-sons.
- Em gel digerir
- separação Gel
- Executar uma segunda SDS-PAGE utilizando as amostras de concentração ajustada ao. Mancha e quantificar os géis para uma segunda vez para verificar a qualidade de normalização.
- Cortar cada pista de uma amostra no gel em diferentes áreas (8 / pista), mas exclui a faixa de peso molecular acima de 170 kDa. Transferir os pedaços de gel em tubos separados.
- Corte as áreas em pedaços mais pequenos (aprox. 1 x 1 mm) com um bisturi afiado para facilitar a eficácia de digestão em gel.
- Digest 15
- Lavam-se as peças de gel várias vezes (dependendo da intensidade de coloração), durante 10 min com 50 - 150 ul de um tampão que consiste em 50% de acetonitrilo (ACN) e hidrogenocarbonato de amónio 50 mM (NH 4 HCO 3).
- Remover sobrenadantes. Cobrir os pedaços do gel com ACN e incubar a 20 ° C até se tornar branco pedaços de gel e encolher.
- Remova o ACN e hidratar os pedaços de gel para 5min com 50 ul de 0,1 M de NH 4 HCO 3. Adicionar o mesmo volume de ACN e incubar durante mais 15 minutos a 37 ° C.
- Retirar e descartar o líquido completamente. Secam-se os pedaços de gel em uma centrífuga de vácuo.
- Rehidratar pedaços de gel em 50 ul de NH 4 HCO 3 contendo ditiotreitol 10 mM (DTT) e amostras de calor durante 45 min a 56 ° C para reduzir os resíduos de cisteína.
- Remover os sobrenadantes e adicionar 50 ul de NH 4 HCO 3 contendo 55 mM de iodoacetamida (IAA) durante 30 minutos no escuro para carbamidomethylate cisteinas reduzidas.
- Remover e rejeitar todo o líquido acima dos pedaços de gel e lava-se duas vezes com 50 ul de NH 4 HCO 3 e ACN (1: 1) durante 10 min para remover qualquer IAA residual. As amostras secas numa centrífuga de vácuo.
- Para a digestão limitada de proteínas adicionar 25 mM NH 4 HCO 3 contendo 12,5 ng / mL de tripsina. O volume requerido depende do tamanho e quantidade do gel de pieces. Incubar por alguns minutos e verifique se o tampão é absorvido. Adicione mais tampão, se necessário, pedaços de gel deve ser completamente coberto. Incubar a 37 ° C durante a noite (min. 12 h).
- extração de peptídeo
- Pedaços de gel de sobreposição com 10 - 20 ul de 25 mM NH 4 HCO 3 e adicionar o mesmo volume de ACN. Incubar durante 10 min em gelo utilizando um banho de ultra-sons. Depois remover e recolher os sobrenadantes que contêm a maioria dos péptidos gerados.
- Adicionar 100 ul de tampão de extracção contendo 30% / ácido trifluoroacético 0,1% de ACN (TFA) até os pedaços de gel. Repita incubação num banho de ultra-sons e recolher atentamente este sobrenadante.
- Repita os últimos passos de extracção, aumentando a concentração de ACN a 50%. Após 10 min de banho ultra-sônico spin down e recolher os sobrenadantes.
- Combinar todos os três sobrenadantes correspondentes dos passos de extracção e secá-las numa centrífuga de vácuo. Observe quecomo um resultado da separação em gel as áreas 8 por pista / amostra são combinados para uma amostra novamente neste passo.
- separação Gel
- In-solução para a análise
- Digerir
- Utilize a quantidade calculada (por exemplo, 100 ul de um ligado de 150 ul, depende da quantidade de material e o volume requerido para a ressuspensão de uma amostra a partir de uma área específica do cérebro) de amostras normalizadas para se obter o material inicial suficiente para que, pelo menos, três repetições técnicas realizar a espectrometria de massa livre de etiqueta.
- Adicionar DTT 2 mM em 25 mM NH 4 HCO 3 e gentilmente vortex a amostra. Reduzir as amostras durante 45 min a 20 ° C.
- Adicionar IAA 10 mM a carbamidomethylate os resíduos de cisteína. Misturar e incubar durante 30 min no escuro a 20 ° C.
- Finalmente, adicionar 1 ml de uma solução estoque de tripsina (1 ug / uL de tripsina em 25 mM de ácido acético) e incubar a 20 ° C durante 12 h.
- Extracção em fase sólida (SPE) -Purification
- Para remover o detergente ácido clivável, ajustar as amostras a uma concentração final de 1% de TFA e incubar durante 1 hora a 20 ° C.
- Centrifugar as amostras a 16.000 xg por 10 min e com cuidado recolher sobrenadantes.
- Colocar a coluna de SPE numa cremalheira e equilibrar a matriz com 2 ml de metanol. Lavar duas vezes com 2 ml de TFA a 0,1% em água (tampão B).
- Adicionar 2 ml de tampão B e carregar a amostra. Lavar mais três vezes.
- Elui-se os péptidos através da adição de 200 ul de 70% ACN / TFA a 0,1%. Repita este passo.
- Piscina ambos os eluatos e secá-las numa centrífuga de vácuo.
- Digerir
- A fosfo-péptido-enriquecimento de TiO 2 16 cromatografia
- Dissolve-se os péptidos produzidos por em gel ou em solução para a análise em 150 ul de 80% ACN / TFA a 2,5% (tampão C) e equilibrar ~ 2 mg dos grânulos de TiO 2 em 50ul de tampão C.
- Adicionar grânulos à amostra e incubar num dispositivo rotativo durante 1 hora a 20 ° C. Depois, rotação grânulos para baixo (16.000 xg, 1 min) e recolher os sobrenadantes.
- Lave as contas três vezes com 100 ul de tampão C por mistura e girando para baixo depois de 5 min suavemente. Recolha sobrenadantes. Repetir esta etapa, três vezes com 100 ul de 80% ACN / TFA a 0,1%, seguido de três lavagens com 100 uL de 0,1% de TFA (sem ACN), respectivamente.
- Combinar todos os dez sobrenadantes, secá-las numa centrífuga de vácuo e tratá-los como a fracção de péptido-fosfo-empobrecido para posterior purificação de acordo com o passo 3.5.
- Eluir os fosfo-péptidos ligados com 20 ul de mM de NH4OH / 30% de ACN 400 a partir dos grânulos. Repita este passo três vezes e recolher todos os sobrenadantes após girar para baixo as contas.
- Combinar os eluatos da digest em gel e da solução em-digest de uma amostra e tratá-los como a fosfo-peptide fracção enriquecida. Secá-las numa centrífuga de vácuo até um volume final de 4 - 8 ul.
- Concentrar e dessalinização das fracções fosfo-empobrecido-péptido por micro-SPE
- Dissolve-se os péptidos secos em 20 ul de 0,1% de TFA.
- Equilibrar a C 18 -matrix fixada pelo desenho 20 l ACN na ponta. Lava-se a matriz de desenho por 0,1% de TFA em água para a ponta. Repetir o processo três vezes.
- carregar lentamente amostra acidificada na ponta (repita este passo três vezes).
- Lavam-se as 18 C -matrix três vezes com 20 uL de 0,1% de TFA em água e descartar a solução de lavagem.
- Eluir os péptidos a partir da ponta de pipeta por várias vezes (3 vezes) de estiramento 20 ul de 70% ACN / TFA a 0,1%, e recolher esse solução de eluição em um tubo separado.
- Combinar os eluatos de uma amostra e secá-las numa centrífuga de vácuo.
4. Análise Proteome
NOTA: Proteome análise é realizada em uma pressão dupla ion trap linear híbrida / Orbitrap espectrômetro de massa equipado com um HPLC de ultra. A HPLC é composto por um auto-amostrador refrigerado com uma ansa de injecção de 20 ul, uma bomba de binário de carga (intervalo de fluxo ul), uma bomba de separação de fluxo nano binário, um aquecedor de coluna, com dois micro válvulas comutadoras e um desgaseificador. As amostras são, em primeiro lugar submetido a uma coluna de armadilhagem (por exemplo, 100 mm x 2 cm) com um caudal de 7 mL / min, seguida por separação numa coluna (por exemplo, 75 um x 25 cm) em 250 nl / min. A saída da coluna de separação é directamente acoplado a uma ponta revestida emissor Pico posicionada num interface de nano-pulverização na fonte espectrómetro de massa de ionização.
- Espectrometria de nano-líquido e cromatografia de massa em tandem
- Dissolve-se amostras de péptido em 12 ul de 2% de ACN / TFA a 0,1% durante pelo menos 30 min. Spin down por 15 segundos e transferir 11 ul sobrenadante para amostrador automático frascos (cônica, reduzida diameter).
- Defina-se um regime automatizado para aplicação da amostra, a separação cromatográf ica e de espectrometria de massa em tandem no software de controlo (por exemplo, Xcalibur) como se segue.
- Use o seguinte para Temperatura: Autosampler: 5 ° C; forno da coluna: 45 ° C.
- Use o seguinte para injeção: Volume: 10 ml; Caudal: 7 mL / min (2% de ACN, 0,1% de TFA); Time: 8 min; regulagem da válvula: Coluna trap - resíduos; aquisição de espectro de massa: off.
- Use o seguinte para a separação: Caudal: 250 nl / definição min Válvula: Armadilha coluna coluna de separação; aquisição de espectro de massa: na.
0 min - 100 minutos: 2% de ACN, 0,1% de ácido fórmico - 40% de ACN, 0,1% ácido fórmico
100 min - 105 min: 40% de ACN, 0,1% de ácido fórmico - 95% de ACN, 0,1% ácido fórmico
105 min - 109 min: 95% de ACN, 0,1% ácido fórmico
109 min - 120 minutos: 2% de ACN, 0,1% ácido fórmico - Use as seguintes configurações para a espectrometria de massa: MS completa: FTMS; resolução 60.000; m / z gama 400 - 2.000; SENHORA/MS: Linear Iontrap; limiar mínimo de sinal 500; largura de isolamento 2 Da; tempo de exclusão dinâmica definindo 30 seg; íons individualmente carregados são excluídos da selecção; energia de colisão normalizada é definida como 35%, e a activação de tempo de 10 mseg.
NOTA: Uma verificação completa MS é seguido por até 15 LTQ MS / MS é executado usando induzida por colisão-dissociação (CID) dos íons peptídeo mais abundante detectados.
- Execute três repetições técnicas para todas as amostras.
- Identificação de proteínas e quantificação livre rótulo
- Dados de espectrometria de massa de processo matérias para a identificação de proteínas e quantificação livre de etiqueta utilizando um conjunto comercial de software (por exemplo, picos Studio). Em contraste com a maioria dos outros pacotes de software proteoma este software especial usa um algoritmo de novo -sequencing antes de alinhamentos de banco de dados de proteínas. No entanto, este passo pode ser facilmente substituído por outros pacotes de software populares.
- Use essconfigurações cial listados na Tabela 2.
- Phospho-proteômica
NOTA: aquisição fosfo-peptídeo eficiente e confiável requer algumas mudanças essenciais da configuração do fluxo de trabalho proteômica.- Após o enriquecimento fosfo-péptidos, as amostras não secar completamente. Sempre mantenha amostras dissolvidas.
NOTA: A ligação de éster fosfo de treoninas fosforilada ou serines é muito frágil. Durante a fragmentação induzida por colisão no interior da armadilha de iões isto resulta em uma perda de neutro de fosfato. Isto impede que qualquer outra fragmentação do péptido, o que por sua vez, é necessária para a identificação. Permitido de banda larga-activação na configuração de espectrometria de massa permite a fragmentação de fosfo-péptidos, mesmo depois de uma perda de neutro do grupo fosfato. Ele executa uma economia de "pseudo-MS 3" tempo. determinação fosfo local em dados MS / MS requer uma verificação e de avaliação em particular e pode ser realizada por 3 fósforos.0.
- Após o enriquecimento fosfo-péptidos, as amostras não secar completamente. Sempre mantenha amostras dissolvidas.
5. Bioinformatics - Meta-Analysis
NOTA: Antes de executar anotação funcional e análise de rede, as listas de proteínas têm de ser pré-processado. Primeiro fundir as listas de proteínas reguladas e fosfo-peptídeos para cada região do cérebro separadamente. Em seguida, remova todos os duplicados UniProt-IDs para cada fracção para evitar erros de interpretação.
- Análise de Enriquecimento de singular com 17 GeneCodis
- Abra a ferramenta de web-based de GeneCodis (http://genecodis.cnb.csic.es)
- Selecione "Mus musculus" como organismo e "GO Processo Biológico" como anotação.
- Cole uma lista de UniProt-IDs de uma certa fração. Enviar e aguarde até que a análise é realizada. Clique em "Singular Análise Enriquecimento de Processo GO Biológica" e ver os resultados.
- Repita o passo 5.1.3 para os outros três fracções.
- Para ver todas as duplicações e intersecções entre as listas de resultadosusar uma linguagem de script como Perl ou Python para filtrar os dados necessários. ferramentas similares para uma análise enriquecimento singular são DAVID (https://david.ncifcrf.gov/) e Cytoscape (http://www.cytoscape.org/) com os plugins do bingo (http://apps.cytoscape.org/ apps / bingo) e ClueGO (http://apps.cytoscape.org/apps/cluego).
- Gerando um gráfico baseado vigor a partir de dados GeneCodis com Gephi (https://gephi.org/)
NOTA: Os dados para os gráficos tem que ser fornecida pelo usuário, seja em um formato gráfico (.gexf, .graphml, .dot, .gv, .gml) ou inseridos manualmente.- Gerando os nós do grafo
- À mão: Open Gephi e clique em "Laboratório de dados". Criar nós. Clique em "nós" à esquerda para mudar para a tabela de "Nodes". Clique em "Adicionar nó". Digite o nome do termo. Clique em "OK" / Pressione Enter.
- Alternativas: Salve GeneCodis resultar em PC. Abra o .txt com um programa de planilha. Excluir todas as linhas except da "Item_Details" (nomes prazo). Mudança de cabeçalho "Item_Details" a "etiqueta". Salvar planilha como ".csv". Agora em Gephi, clique em "Importar planilha". Escolha planilha a partir do navegador de arquivos de Gephi. Clique em "Next". Clique em "Finish".
- A ligação dos nós via bordas.
- Clique em "bordas" da esquerda para mudar para a tabela de "Edges". Para cada nó (Term): procurar nomes de genes em outros termos. Se um ou mais genes são compartilhados -> criar borda.
- Clique em "Adicionar Edge". Selecione "sem direção". Seleccione a fonte e nó de destino fora de listas suspensas. Clique em "OK" / Pressione Enter. Se mais do que um gene é compartilhada, entrar em abundância "peso" (tabela).
- Força baseada layout gráfico.
- Abrir arquivo de dados do gráfico, defina o tipo de gráfico para "não dirigida" ou utilizar os dados como digitado manualmente, clique em "Visão Geral" se ainda não estiver selected.
- Redimensionar nodos dependendo da abundância de interligações. Clique em Estatística, execute um "grau médio" (arestas não ponderadas) ou "médio. Grau Weighted" (bordas ponderada) em "Rede Overview". Em "Aparência", clique em "nós", em seguida, no botão Tamanho, próxima escolha "atributos" e definir o parâmetro Atributos para "médio. Grau ponderada" ou "grau médio". Clique em Aplicar.
- Finalmente: Selecione "Força Atlas" em "Layout" e correr; mudar "força Repulsion" se os nós estão colidindo.
- Exportar para foto.
- Screenshot recurso: Clique em "Visão Geral", o layout gráfico de mudança, espessura de borda, tamanho de etiqueta e dimensionamento com o menu na parte inferior da janela "Gráfico". Clique o botão da câmera para a esquerda, e salvar imagem.
- O recurso de exportação de "Preview": Clique em "Preview". Alterar Presets para "Default reta". Alterar configuraçãos de acordo com as preferências escolhidas e clique em "SVG / PDF / PNG" para exportar.
- Gerando os nós do grafo
Resultados
A Figura 1 resume o fluxo de trabalho completa do quantitativo de perfis proteoma sináptica de regiões do cérebro do rato depois de saber discriminação auditiva. Ele começa com o treinamento de animais em uma caixa de transporte. No exemplo mostrado na Figura 2, os murganhos começaram a mostrar significativa discriminação de tom de FM na sessão de treino 4 th, indicando aprendizagem eficiente. Os animais são sacrificados em momentos seleccionados para dissecção área do cérebro. O enriquecimento requerido de sinapses pode ser conseguido por a preparação de sinaptossomas ou, alternativamente, através da preparação de uma fracção enriquecida em PSD, ambos descritos em pormenor na Figura 3. O método PSD-enriquecimento foi desenvolvido para baixas quantidades de tecido, por exemplo, 1 - 2 fatias de hipocampo de cérebro de rato 12, 18. Ela exige pequenos tubos, pilões PTFE de montagem para estes tubos, e uma unidade de perfuração de laboratório para alimentar o pilão.
Devido à composição de proteína particular de sinaptossomas, é altamente recomendável para realizar a preparação da amostra de duas maneiras diferentes mas complementares. Os andaimes de os PSDs são muitas vezes as proteínas de peso molecular muito elevado que ocorrem em alta estequiometria. Em resumo-solução é a melhor maneira de extrair de forma eficiente, mas pode levar a uma sobre-amostragem da mistura de péptidos gerada. A digest em gel realizada da mesma amostra em paralelo pode excluir essas proteínas de elevado peso molecular e favorecer a análise de proteínas com peso molecular médio e inferior. Para uma análise abrangente são recomendados os dois tipos de digestões proteolíticas.
Os diferentes quantidades de tecidos de as áreas do cérebro investigadas requerem um ajustamento do material aplicado para uma melhor comparação. Dentro das quatro áreas do cérebro investigaram o córtex auditivo é geralmente o fato limitanteou. O material de todas as outras áreas do cérebro deve ser cuidadosamente ajustada com a quantidade de córtex auditivo após a preparação de sinaptossomas ou fracções enriquecidas-PSD (ver 3.1.1.). pesos típicos de áreas cerebrais recentemente preparadas a partir de ratinhos são como se segue: córtex auditivo (AC): ~ 50 mg; hipocampo (HIP): ~ 90 mg; striatum (STR): ~ 120 mg e córtex frontal (FC): ~ 100 mg.
O método PSD-enriquecimento descrito na seção 2.3 permitiram a identificação de cerca de 1500 proteínas diferentes e cerca de 250 diferentes fosfo-péptidos por região do cérebro sobre o nível de um único animal (Tabela 1). Análise proteomic 24 h depois da primeira sessão de treino revelou que 7,3% das proteínas identificadas e 5,8% dos fosfo-péptidos mostraram aumentos significativos (p <0,05) alterações quantitativas da sua expressão sináptica em comparação com controlos sem tratamento prévio (tabela 1). Uma tendência evidente para regula para baixoção de andaimes sinápticas podem apontar para um rearranjo pronunciada da arquitetura sináptica durante as fases iniciais da aprendizagem FMTD. A vasta maioria das proteínas reguladas foram alteradas de uma maneira específica para uma região do cérebro, ao passo que apenas 22% foram encontrados para ser regulada em duas ou mais áreas do cérebro. Seis exemplos seleccionados são mostrados na Figura 4.
Meta-análise dos resultados complexos por IPA fornece evidência para a especial, a participação / manipulação das seguintes vias canônicas: "mediada por Clathrin Endocytosis sinalização", "axonal Orientação de sinalização", "Sinalização de Cálcio", "RhoA sinalização", "Sinalização Notch "," Reforma de epitelial junções aderentes "," O glutamato Receptor Signaling "," GABA Receptor Signaling "," receptor de dopamina sinalização "e" Synaptic potenciação de longa duração ".
análise de enriquecimento individuais revelou processos biológicos significativos sobre-representados no córtex frontal em matéria de transporte de proteínas, a adesão celular, a fosforilação, a endocitose, transporte mediado por vesícula, desenvolvimento do cérebro anterior e axonogenesis (Figura 5). Nos processos biológicos córtex auditivo, incluindo o transporte de íons, tradução, transporte mRNA, transporte de proteínas e aprendizagem eram perceptíveis. A análise da fracção de proteína do hipocampo detecta significativamente enriquecidos processos relacionados com o transporte de iões, do ciclo celular, a tradução, a fosforilação e desenvolvimento do sistema nervoso. No estriado, sobre-representados foram encontrados processos biológicos, incluindo o transporte de mRNA, transporte mediado por vesícula, axonogenesis, proteólise, transporte de proteínas e endocitose.
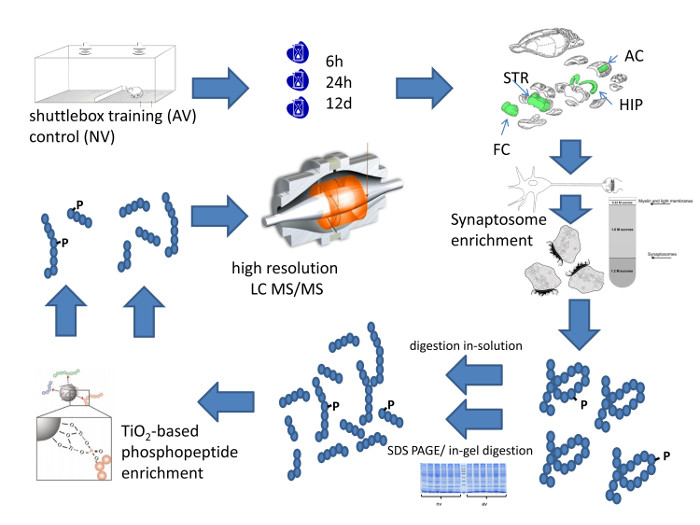
Figura 1: Systematic Workflow da abordagem metodológica. Esta figura resume esquematicamente o fluxo de trabalho de alta resolução profiling quantitativa da composição de proteína sináptica área específica do cérebro. Por favor clique aqui para ver uma versão maior desta figura.

Figura 2: Exemplo do desempenho de ratos no tom FM Discriminação de tarefas. Animais mostram uma taxa crescente de visitas (curva azul) e uma taxa decrescente de falsos alarmes (curva preta) no decurso das sessões de treinamento. discriminação significativa ocorre a partir da quarta sessão. As barras de erro são fornecidos como SEM. Por favor clique aqui para ver um larversão ger desta figura.

Figura 3: Preparação da Fracção de sinaptosoma e o PSD-enriquecida. A: preparação de sinaptossomas. B: Preparação fracção enriquecida em PSD. Ambas as figuras explicar o fluxo de trabalho detalhado de preparação de sinaptossomas ou frações alternativamente PSD-enriquecido a partir de tecidos cerebrais. Por favor clique aqui para ver uma versão maior desta figura.
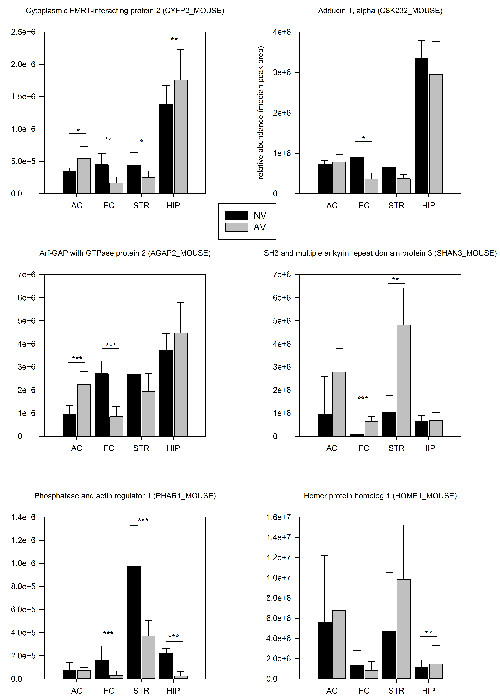
Figura 4: Selecionado resultados quantitativos proteômica. As abundâncias sinápticas relativas de proteínas selecionadas são comparados entre tra ratosinada na tarefa FMTD (AV, n = 6) e ratinhos de controlo sem tratamento prévio (NV, n = 6) 24 h após a primeira sessão de treino. Os valores de abundância foram calculados como média das áreas dos picos dos três mais intensos péptidos de uma proteína. Proteínas com alterações significativas abundância (AV / NV; t-teste) são marcadas dentro dos lotes: * p <0,05, ** p <0,01, *** p <0,005. As barras de erro são fornecidos como SD. Por favor clique aqui para ver uma versão maior desta figura.

Figura 5: Visualização de vias biológicas para córtex frontal por GeneCodis / Gephi. Somente os termos significativos do banco de dados Gene Ontology (GO) (http://geneontology.org) relacionadas com "Processo biológico" com um número de proteínas mínimo de três são mostradas aqui. Nós representam termos GO, o tamanho do nó, a largura de linha e o número de ligações de um determinado nó representam o número de proteínas, que partilham este termo GO com outros nós. Devido ao método de "Força Atlas" de Gephi, nós relacionados se aglomeram em conjunto. Por favor clique aqui para ver uma versão maior desta figura.
| região do cérebro | CA | FC | QUADRIL | 000 "face =" Calibri "size =" 3 "> STR | Σ |
| proteínas identificadas | 1435 | 1758 | 1572 | 1507 | 6272 |
| proteínas reguladas (p <0,05) | 59 | 130 | 162 | 108 | face = "Calibri" size = "3"> 459 |
| ↑ AV / NV | 8 | 4 | 76 | 35 | 123 |
| ↓ AV / NV | 51 | 126 | 86 | 73 | 336 |
| phosphomoti identificados fs | 197 | 361 | 273 | 278 | 1109 |
| phosphomotifs regulamentados (p <0,05) | 8 | 22 | 21 | 14 | 65 |
| ↑ AV / NV | 4 | 00000 "face =" Calibri "size =" 3 "> 17 | 5 | 9 | 35 |
| ↓ AV / NV | 4 | 5 | 16 | 5 | 30 |
Tabela 1: Resumo de um resultado Proteomic. Esta tabela resume uma experiência representativa de proteómica ratos treinados (AV, n = 6) 24 h após a primeira sessão de treino em relação aos seus controlos naive (NV, n = 6). osoma de 459 proteínas reguladas inclui regulamentos que se sobrepõem. 283 regulamentos diferentes foram determinadas como específica do cérebro. Em detalhe, 57 proteínas são regulados em duas regiões cerebrais, 18 regulamentos proteínas foram detectadas em três regiões do cérebro e apenas 2 proteínas são regulados em todas as quatro áreas do cérebro investigadas.
| tolerâncias de erro | |
| massa precursor (Fourier massa transformação espectrometria) | 10 ppm |
| massa fragmento de íons (ion trap linear) | 0.6 Da |
| Clivagens máximas perdidas por peptídeo | 3 |
| modificações fixos | |
| no digerido-gel amostras | Carbamidomethylation de cisteína |
| para as amostras em solução digerido | MethylthiolatioN de cisteína |
| modificações variáveis | A oxidação de metionina |
| Deamidations de asparagina e / ou glutamina | |
| Banco de dados | UniProt / Sprot |
| Taxonomia | rato |
| Ajustes estatísticos de identificação de aceitação | |
| de novo médio de confiança local (ALC) | > 50% |
| Peptide-falsa taxa de detecção (FDR, com base na est. Chamariz-fusion) | <1% |
| A proteína de significância (-10logP, baseado em t-teste modificado) | > 20 |
| péptidos únicos / proteína | 1 ≥ |
| Configurações de quantificação: | |
| Os péptidos utilizados para a quantificação se: | |
| Peptide significância (-10logP) | > 30 |
| identificação péptido em | ≥ 50% das amostras |
| qualidade de sinal Péptido | > 1 |
| área média Peptide | > 1E5 |
| tolerância tempo de retenção Péptido | <5 min |
| Normalização | pela corrente de íons total (TIC) |
Tabela 2: Definições para a identificação de proteínas (passo 4.2.2).
Discussão
O estudo apresenta um fluxo de trabalho metodológico otimizado para um perfil quantitativa precisa de mudanças de expressão de proteínas sinápticas durante a aprendizagem e consolidação da memória em diferentes áreas cerebrais de camundongos. A configuração oferece a oportunidade para estudar a expressão de proteína ao nível de um único animal, apesar de a aplicação desejada de pelo menos três replicados por amostra de técnicas para a análise por espectrometria de massa.
A metodologia leva em conta a composição de proteína em particular do pré e postsynapse consistindo de proteínas de elevado peso molecular de andaime, mas também de proteínas importante mediador rolamento pesos moleculares médios ou baixos. Os digeridos em solução de preparações sinaptosomais resultar numa geração eficiente e, portanto, uma sobre-representação de péptidos derivados de andaime. Este, por sua vez, pode suprimir a análise de proteínas abundantes menores ou inferiores. A preparação sugerido de frações de SDS-PAGE de umalíquota de cada amostra combinada com um procedimento de digestão em gel em paralelo facilita a análise de proteínas de abundância médio e baixo e representa um método complementar altamente recomendado. Após a aplicação de espectrometria de massa separada de todas as fracções derivadas a partir de uma amostra (por exemplo, em solução digerir, em gel de digerir, fracções enriquecidas-fosfo combinados) os correspondentes conjuntos de dados de MS / MS pode ser combinado e ainda calculadas para a identificação de proteínas e quantificação de picos software ou pacotes de software alternativos populares.
Alternativamente, a aplicação individual das fracções em gel derivado de digestão de uma amostra (gel-áreas processados separadamente de uma pista de amostra) e fracções geradas da amostra digerida em solução (por exemplo, por cromatografia de permuta iónica) para espectrometria de massa pode aumentar a profundidade analítica. No entanto, este fluxo de trabalho prolongado aumenta drasticamente o tempo necessário para LS-MS / MS de aquisição de dados. para generatioN de uma sequência molecular detalhada de rearranjos de proteínas sinápticas durante a aprendizagem e a formação da memória de um curso de tempo especificado do perfilamento proteomic é necessária. Este curso tempo pode começar imediatamente depois ou mesmo durante a primeira sessão de treinamento e abrange um período de tempo do fim-de malha até que o desempenho dos animais atingiu o nível assintótica da curva de aprendizagem após aprox. 8 - 10 dias de treinamento (veja a Figura 2 para obter detalhes).
A análise das alterações de fosforilação de proteínas sinápticas requer um foco particular sobre os prazos selecionados durante a aprendizagem FMTD. Sobre as cascatas de sinalização um lado, iniciando rearranjos de proteínas sinápticas conhecidos por ser desencadeada por fosfolarização proteína e dephosphorylations são esperados nos primeiros estágios de treinamento animal. Por outro lado, existem longos modificações duradouras de múltiplas proteínas fosforiladas sinápticas conhecidas que regulam a conectividade e a montagem dentro dos sarquitetura ynaptic 19, 20. Essas modificações pós-translacionais são esperados, mesmo em momentos posteriores da consolidação da memória.
Os conjuntos de dados complexos gerados por este fluxo de trabalho proteômica requerem processamento de bioinformática para identificar participar vias moleculares e moléculas-chave. Meta-análise mostra caminhos sobre-representados significativas, que desempenham um papel nos processos de aprendizagem e memória.
Divulgações
The authors have nothing to disclose.
Agradecimentos
We wish to thank Yvonne Ducho and Kathrin Pohlmann for excellent technical assistance. This work was supported by the Deutsche Forschungsgemeinschaft (SFB 779) and by the State Saxony-Anhalt / European Regional Development Fund (ERDF) via the Center for Behavioral Brain Sciences (CBBS).
Materiais
| Name | Company | Catalog Number | Comments |
| 3 M Empore Solid Phase Extraction- Filter | 3M Bioanalytical Technologies | 4245SD | 7 mm/3 ml |
| Acclaim PepMap 100 | Dionex/Thermo Scientific | 164564 | 100 µm x 2 cm, C18 |
| Acclaim PepMap 100 | Dionex/Thermo Scientific | 164569 | 75 µm x 25 cm, C18 |
| Acetic acid | Carl Roth GmbH | 3738.1 | |
| Acetonitrile (ACN) | Carl Roth GmbH | AE70.2 | |
| Acrylamide (30%) | AppliChem | A0951 | |
| Ammonium hydrogen carbonate | Fluka | 9830 | |
| Ammonium hydroxide | Fluka | 44273 | |
| Ammonium persulfate (APS) | AppliChem | A2941 | |
| Biofuge pico | Heraeus GmbH | 75003280 | |
| Blue R-250 | SERVA Electrophoresis GmbH | 17525 | |
| Bromophenol Blue | Pharmacia Biotech | 17132901 | |
| C57BL/6J mice | Charles River | ||
| Cantharidin | Carl Roth GmbH | 3322.1 | |
| Centrifuge tubes for MLS-50 | Beckman Coulter | 344057 | |
| Centrifuge tubes for TLA 100.1 rotor | Beckman Coulter | 343776 | |
| Dithiothreitol (DTT) | AppliChem | A1101 | |
| Eppendorf 5417R centrifuge | VWR | 22636138 | |
| Eppendorf A-8-11 rotor | VWR | 5407000317 | |
| Formic acid | Fluka | 14265 | |
| GeneCodis | http://genecodis.cnb.csic.es/ | ||
| Gephi | https://gephi.org/ | ||
| Glycerol | AppliChem | A1123 | |
| Glycine | AppliChem | A1067 | |
| HALT Phosphatase Inhibitor Cocktail | Pierce /Thermo Scientific | 78420 | |
| HEPES Buffer solution | PAA Laboratories GmbH | S11-001 | |
| Homogenization vessel 2 ml | Sartorius AG | 854 2252 | |
| Hydrochloric acid | Sigma-Aldrich | H1758 | |
| Imidazole | Sigma-Aldrich | I2399 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide (IAA) | Sigma-Aldrich | I1149 | |
| Laboratory drilling drive K-ControlTLC 4957 | Kaltenbach & Vogt GmbH | 182997 | |
| LTQ Tune Plus 2.7.0.1112 SP2 | Thermo Scientific | ||
| LTQ Orbitrap Velos Pro | Thermo Scientific | ||
| Macs-mix tube rotator | Miltenyi Biotech | 130-090-753 | |
| Magic Scan 4.71 | UMAX | ||
| Methanol | Carl Roth GmbH | AE71.2 | |
| MLS-50 rotor | Beckman Coulter | 367280 | |
| Optima MAX Ultracentrifuge | Beckman Coulter | 364300 | |
| PageRuler Prestained Protein Ladder | Thermo Scientific | 26616 | |
| PEAKS 7.5 | Bioinformatic Solutions | ||
| Phosphatase Inhibitor Cocktail 3 | Sigma-Aldrich | P0044 | |
| PhosphoRS 3.1 | IMP/IMBA/GMI | ||
| PhosSTOP | Roche | 4906845001 | |
| Plunger/pestle made of PTFE | Sartorius AG | 854 2651 | |
| PotterS homogenizer | Sartorius AG | 853 3024 | |
| Protease Inhibitor complete mini | Roche | 4693159001 | |
| Quantity One 4.5.1 | BioRad | ||
| RapiGest | Waters | 186002122 | |
| Shuttle box | Coulbourne Instruments | ||
| Sodium dodecylsulfate (SDS) | AppliChem | A1112 | |
| Sodium molybdate | Carl Roth GmbH | 274.2 | |
| Sodium tartrate dihydrate | Sigma-Aldrich | 228729 | |
| SONOREX RK 156 Ultrasonic Bath | BANDELIN electronic GmbH & Co. KG | 305 | |
| Soundproof chamber | Industrial Acoustics Company | ||
| Sucrose | Carl Roth GmbH | 4621.2 | |
| Tetramethyl ethylene -1,2-diamine (TEMED) | Sigma-Aldrich | T9281 | |
| Thermomixer basic | CallMedia | 111000 | |
| Titansphere TiO 5µm | GL Sciences Inc. Japan | 502075000 | |
| TLA 100.1 rotor | Beckman Coulter | 343840 | |
| Trifluoro acetic acid (TFA) | Sigma-Aldrich | T6508 | |
| Tris ( hydroxymethyl) aminomethane (TRIS) | AppliChem | A1086 | |
| Triton X-100 | Sigma-Aldrich | T8532 | |
| Trypsin Gold | Promega | V5280 | |
| Ultimate 3000 Ultra HPLC | Dionex/Thermo Scientific | ||
| Ultracentrifuge tube | Beckman Coulter | 343776 | |
| Unijet II Refrigerated Aspirator | Uniequip Laborgeräte- und Vertriebs GmbH | ||
| UNIVAPO 100 H Concentrator Centrifuge | Uniequip Laborgeräte- und Vertriebs GmbH | ||
| Urea | AppliChem | A1049 | |
| Water (high quality purifed) | Resistivity: > 18.2 MΩ*cm at 25 °C Pyrogens: < 0.02 EU/ml TOC: < 10 ppb | ||
| Xcalibur 3.0.63 | Thermo Scientific | ||
| ZipTipC18 Pipette Tips | MILLIPORE | ZTC18S960 |
Referências
- Lamprecht, R., LeDoux, J. Structural plasticity and memory. Nat Rev Neurosci. 5 (1), 45-54 (2004).
- Bingol, B., Schuman, E. M. Synaptic protein degradation by the ubiquitin proteasome system. Curr Opin Neurobiol. 15 (5), 536-541 (2005).
- Richter, J. D., Klann, E. Making synaptic plasticity and memory last: mechanisms of translational regulation. Genes Dev. 23 (1), 1-11 (2009).
- Rosenberg, T., et al. The roles of protein expression in synaptic plasticity and memory consolidation. Front Mol Neurosci. 7, 86(2014).
- Scheich, H., et al. Behavioral semantics of learning and crossmodal processing in auditory cortex: the semantic processor concept. Hear Res. 271 (1-2), 3-15 (2011).
- Kähne, T., et al. Synaptic proteome changes in mouse brain regions upon auditory discrimination learning. Proteomics. 12 (15-16), 2433-2444 (2012).
- Reichenbach, N., et al. Differential effects of dopamine signalling on long-term memory formation and consolidation in rodent brain. Proteome Sci. 13, 13(2015).
- Kähne, T., et al. Proteome rearrangements after auditory learning: high-resolution profiling of synapse-enriched protein fractions from mouse brain. J Neurochem. , (2016).
- Li, K., et al. Organelle proteomics of rat synaptic proteins: correlation-profiling by isotope-coded affinity tagging in conjunction with liquid chromatography-tandem mass spectrometry to reveal post-synaptic density specific proteins. J Proteome Res. 4 (3), 725-733 (2005).
- Carlin, R. K., Grab, D. J., Cohen, R. S., Siekevitz, P. Isolation and characterization of postsynaptic densities from various brain regions: enrichment of different types of postsynaptic densities. J Cell Biol. 86 (3), 831-845 (1980).
- Smalla, K. H., Klemmer, P., Wyneken, U. The Cytoskeleton - Imaging, Isolation, and Interaction. Dermietzel, R. , Humana Press. 265-282 (2012).
- Smalla, K. H., et al. The synaptic glycoprotein neuroplastin is involved in long-term potentiation at hippocampal CA1 synapses. Proc Natl Acad Sci U S A. 97 (8), 4327-4332 (2000).
- Shapiro, A. L., Vinuela, E., Maizel, J. V. Molecular weight estimation of polypeptide chains by electrophoresis in SDS-polyacrylamide gels. Biochem Biophys Res Commun. 28 (5), 815-820 (1967).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Shevchenko, A., Wilm, M., Vorm, O., Mann, M. Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. Anal Chem. 68 (5), 850-858 (1996).
- Thingholm, T. E., Larsen, M. R., Ingrell, C. R., Kassem, M., Jensen, O. N. TiO(2)-based phosphoproteomic analysis of the plasma membrane and the effects of phosphatase inhibitor treatment. J Proteome Res. 7 (8), 3304-3313 (2008).
- Carmona-Saez, P., Chagoyen, M., Tirado, F., Carazo, J. M., Pascual-Montano, A. GENECODIS: a web-based tool for finding significant concurrent annotations in gene lists. Genome Biol. 8 (1), 3(2007).
- Bonn, S., Seeburg, P. H., Schwarz, M. K. Combinatorial expression of alpha- and gamma-protocadherins alters their presenilin-dependent processing. Mol Cell Biol. 27 (11), 4121-4132 (2007).
- Tweedie-Cullen, R. Y., Reck, J. M., Mansuy, I. M. Comprehensive mapping of post-translational modifications on synaptic, nuclear, and histone proteins in the adult mouse brain. J Proteome Res. 8 (11), 4966-4982 (2009).
- Li, L., et al. The differential hippocampal phosphoproteome of Apodemus sylvaticus paralleling spatial memory retrieval in the Barnes maze. Behav Brain Res. 264, 126-134 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados