Method Article
Echtzeitüberwachung der Aktivierung der Aurora-Kinase A mit konformationellen FRET-Biosensoren in lebenden Zellen
In diesem Artikel
Zusammenfassung
Die Aktivierung der multifunktionalen Ser/Thr-Kinase AURKA zeichnet sich durch ihre Autophosphorylierung auf Thr288 aus. Fluoreszierende Sonden, die auf FRET setzen, können zwischen inaktivem und aktiviertem Zustand unterscheiden. Hier veranschaulichen wir einige Strategien für das Sonden-Engineering, zusammen mit einem schnellen FRET-Protokoll, um die Kinase-Aktivierung während der mitose zu verfolgen.
Zusammenfassung
Epithelkarzinome sind oft durch die Überexpression der Ser/Thr-Kinase Aurora A/AURKA gekennzeichnet. AURKA ist ein multifunktionales Protein, das bei seiner Autophosphorylierung auf Thr288 aktiviert wird. AURKA-Häufigkeit erreicht ihren Höhepunkt in der Mitose, wo es die Stabilität und die Treue der mitotischen Spindel und die Gesamteffizienz der Mitose steuert. Obwohl auf struktureller Ebene gut charakterisiert, fehlt eine konsistente Überwachung der Aktivierung von AURKA während des gesamten Zellzyklus. Eine mögliche Lösung besteht darin, mit hilfe von genetisch kodierten Förster's Resonance Energy Transfer (FRET) Biosensoren Einblicke in die Autophosphorylierung von AURKA mit ausreichender raumzeitlicher Auflösung zu gewinnen. Hier beschreiben wir ein Protokoll zur Entwicklung von FRET-Biosensoren, die die Thr288-Autophosphorylierung erkennen, und wie diese Modifikation während der Mitose zu verfolgen ist. Zunächst geben wir einen Überblick über mögliche Donor/Akzeptor-FRET-Paare und zeigen mögliche Klonierungs- und Insertionsmethoden von AURKA FRET-Biosensoren in Säugetierzellen auf. Anschließend bieten wir eine Schritt-für-Schritt-Analyse für schnelle FRET-Messungen durch Fluoreszenz-Lifetime-Imaging-Mikroskopie (FLIM) in einem speziell angefertigten Setup. Dieses Protokoll ist jedoch auch auf alternative kommerzielle Lösungen anwendbar. Abschließend betrachten wir die am besten geeigneten FRET-Kontrollen für einen AURKA-basierten Biosensor und heben mögliche zukünftige Verbesserungen hervor, um die Empfindlichkeit dieses Werkzeugs weiter zu erhöhen.
Einleitung
Die Aurora-Kinase A/AURKA ist eine multifunktionale Serin/Threonin-Kinase, die während des gesamten Zellzyklus und in verschiedenen subzellulären Kompartimenten aktiv ist1. Das Verständnis seiner breiten räumlich-zeitlichen Aktivierung ist bei Krebs besonders wichtig, da AURKA bei epithelialen und hämatologischen Malignomen häufig überexprimiert wird und die Patienten schlecht auf die derzeit verfügbaren Therapien ansprechen2.
Strukturstudien zeigten, dass AURKA zwei Schritte durchläuft, um sich von einer inaktiven in eine aktive Kinase umzuwandeln. Erstens verändert die Autophosphorylierung von Thr288 die Konformation der kinetischen Tasche der Kinase und aktiviert sie3,4,5,6. Dieser Schritt erhöht die katalytische Aktivität von AURKA in menschlichen Zellen und in Xenopus laevis3,6,7 und bereitet die Kinase auf volle Aktivität vor. Einmal aktiviert, induziert die Interaktion von AURKA mit dem Zielprotein für Xklp2 (TPX2) eine zweite Konformationsänderung5. Diese weitere Modifikation ermöglicht es AURKA, die volle enzymatische Aktivität gegenüber seinen Substraten in der Zelle zu erreichen5,8,9,10.
Fast zwei Jahrzehnte lang wurden Erkenntnisse über die Aktivierung und Aktivität von AURKA hauptsächlich durch eine Kombination biochemischer Ansätze gewonnen. Dazu gehören der Nachweis von phosphoryliertem Thr288 in Zellen oder in vivo als Kennzeichen der AURKA-Aktivierung, kristallographische Analysen und In-vitro- oder In-Cellulo-Kinase-Assays zur Untersuchung der Aktivität von AURKA1. Die räumlich-zeitliche Auflösung dieser Ansätze ist jedoch schlecht oder fehlt, und es waren neuartige Lösungen erforderlich, um das Wissen über die Dynamik dieser beiden Ereignisse zu erweitern.
Die Entwicklung von Fluoreszenzsonden in den letzten Jahren erleichterte die Überwachung von AURKA in lebenden Zellen, so dass die Aktivierung mit größerer raumzeitlicher Auflösung erfolgen konnte. Die bisher speziellsten Sensoren für AURKA beruhen auf dem FRET-Prinzip (Förster's Resonance Energy Transfer)11, um zwischen inaktivem und aktivem AURKA zu unterscheiden. Der erste entwickelte Sensor war ein substratbasierter Biosensor der AURKA-Kinase-Aktivität. Substratbasierte Biosensoren werden durch eine kurze aminoskalische Sequenz konstituiert, die von einer bestimmten Kinase zur Phosphorylierung angegriffen wird, und in ein Donor/Akzeptor-FRET-Paar und eine Bindungsdomäne eingefügt, die den phosphorylierten Rückstand erkennt, was die Faltung des Biosensors für einen effizienten FRET-Prozess unterstützt12. Im Fall von AURKA wurde ein 14-Aminosäure-Fragment von KIF2C, das durch Phosphorylierung angegriffen wurde, zwischen ein CFP-YFP-Donor/Akzeptor-Paar eingefügt13. Dieser Sensor hat jedoch einige große Nachteile. Erstens kann die in dieser Sonde verwendete KIF2C-Sequenz sowohl von AURKA als auch von der eng verwandten Kinase AURKB anvisiert werden, wodurch die Spezifität dieses Biosensors verringert wird. Zweitens verlässt sich der Sensor auf die endogene Kinase für die Phosphorylierung. Daher kann die FRET-Effizienz nicht nachweisbar oder nicht signifikant sein, wenn die Mengen der Kinase begrenzt sind (z. B. in subzellulären Kompartimenten oder Zellzyklusphasen). Um diese Einschränkungen zu überwinden, wurde eine neue Klasse von AURKA-Sensoren entwickelt, die als "Konformationssensoren" bekannt sind. In diesen Sonden wurde die AURKA-Sequenz in voller Länge in ein Donorfluorophor am N-Terminus und ein Akzeptorfluorophor am C-Terminus eingefügt. Inaktives AURKA präsentiert eine "offene" Konformation, die die N- und C-Termini der Kinase voneinander wegbringt. Mit einem solchen Abstand zwischen den beiden Termini (> 10 nm) befindet sich das Donor/Akzeptor-Paar in einer nicht-permissiven Konfiguration für FRET. Im Gegenteil, autophosphoryliertes AURKA nimmt eine "geschlossene" Konformation an, wobei die beiden Proteintermini und die beiden Fluorophore in der Nähe liegen. Es wurde gezeigt, dass dies eine FRET zwischen dem Spender und dem Akzeptor ermöglicht, die anhand der Variationen in der Spenderlebensdauer gemessen werden kann14,15. Solche Sonden bieten mehrere Vorteile. Erstens sind sie genetisch kodiert und können verwendet werden, um die endogene Kinase in der Zelle zu ersetzen. Zweitens retten sie die Phänotypen, die durch den Knockdown von AURKA induziert werden, was darauf hindeutet, dass sie in der Zelle funktionsfähig sind. Drittens ermöglichen sie es, die Aktivierung der Kinase in verschiedenen subzellulären Kompartimenten und während des gesamten Zellzyklus zu verfolgen. Die Sonden erkannten die Aktivierung von AURKA an Stellen, an denen die Kinase bekanntermaßen aktiviert ist (d.h. Zentrosomen und die mitotische Spindel), und waren auch an der Entdeckung der Aktivierung von AURKA an den Mitochondrien beteiligt16. Schließlich ermöglichten diese Sensoren High-Content-Screenings auf der Basis von FRET/FLIM, bei denen die Konformationsänderungen von AURKA zur Identifizierung neuartiger pharmakologischer Inhibitoren verwendet wurden17.
In der vorliegenden Arbeit beschreiben wir ein Verfahren zur Visualisierung der AURKA-Aktivierung in kultivierten Zellen. Zunächst werden wir einen Einblick in mögliche Fluorophorpaare für FRET geben. Die Wahl des am besten geeigneten Spender/Akzeptor-Paares erfolgt entsprechend dem verfügbaren Mikroskopaufbau oder einer bestimmten nachgeschalteten Anwendung als Multiplex FRET18,19. Dann schlagen wir eine Pipeline vor, um das Verhalten der Biosensoren zu untersuchen, die in einem schnellen FRET/FLIM-Mikroskopaufbau ausgewählt wurden. Diese Pipeline wird sich von Zellkultur- und Synchronisationsverfahren bis hin zur FLIM-Erfassung und Datenanalyse erstrecken. Schließlich werden wir die potenziellen Vorteile dieses Protokolls diskutieren, da eine analoge Strategie für das Biosensordesign auf andere Kinasen angewendet werden könnte und auch mit anderen FRET-basierten Bildgebungssystemen verwendet werden kann.
Protokoll
HINWEIS: U2OS-Zellen, die in diesem Protokoll verwendet werden, wurden von der American Type Culture Collection (ATCC, HTB-96) gekauft und frei von Mykoplasmen getestet. Die Schritte 2.1 bis 2.7 sollten unter einer Laminar-Flow-Haube durchgeführt werden, um Zellen und Reagenzien steril zu halten.
1. Auswahl des FRET-Paares Spender/Akzeptor
- Die Auswahl der am besten geeigneten Spender/Akzeptor-FRET-Paare finden Sie in der Literatur. Nützliche Beispiele finden sich in20,21,22,23,24, obwohl die endgültige Wahl nach den Eigenschaften des FRET/FLIM-Setups (verfügbare Laserlinien, Filter usw.) getroffen werden muss. Im Folgenden finden Sie einige Überlegungen zur Auswahl eines Spender/Akzeptor-Paares.
- Auswahl des Donors: Beziehen Sie sich auf die FP-Base (https://www.fpbase.org/) für einen vollständigen Satz von Informationen über die verfügbaren fluoreszierenden Proteine. Diese Datenbank wird ständig mit allen neu entwickelten Fluorophoren aktualisiert.
- Weitere Informationen zu den bereits in der Literatur verfügbaren Biosensoren und den jeweils verwendeten fluoreszierenden Proteinen finden Sie in der Fluorescent Biosensor Database (https://biosensordb.ucsd.edu/index.php).
- Wählen Sie als allgemeinen Ausgangspunkt ein helles Spenderfluorophor. Gute Kandidaten sind cyan fluoreszierende Proteine wie mTFP1 oder ECFP oder GFP-Varianten wie EGFP oder mEGFP.
- Oligomere können die Proteinlokalisation und/oder -funktion beeinflussen25. Erwägen Sie die Verwendung monomerer Mutanten von CFP als mTurquoise226 oder Aquamarin27,28. Diese Varianten haben auch gute Quantenausbeute und Extinktionskoeffizienten, was sie zu guten Kandidaten als FRET-Donatoren macht.
- Bevorzugen Sie fluoreszierende Proteine (sowohl als Donor als auch als Akzeptoren), die unempfindlich gegen Umweltveränderungen wie intrazelluläres pH23 sind, oder photobleichen25, da die FRET-Effizienz durch diese Parameter stark beeinflusst werden kann29. Heutzutage werden Fluorophore wie mTurquoise2 oder mTFP1 dank ihrer guten Photostabilität ausgiebig als Spender verwendet22,25,26.
- Wahl des Akzeptors: Cyan-Spender werden oft mit YFP-Varianten (Yellow Fluorescent Protein) gepaart, wie mVenus, Citrin und YPet20,21,22,30. Es sollte jedoch beachtet werden, dass diese Proteine eine viel größere Empfindlichkeit gegenüber dem pH-Wert aufweisen und weltweit eine schlechte Photostabilität aufweisen.
- Erwägen Sie die Verwendung neu entwickelter, pH-unempfindlicher gelber Varianten von YFP als pH-Lemon31, grüner Fluorophore als mNeonGreen23 oder roter Fluorophore als mScarlet-I23,32 als zuvor validierte Fluoreszenzakzeptoren für mTurquoise2.
- Alternativ können Sie nicht-fluoreszierende / dunkle Derivate von YFP wie ShadowG33 oder ShadowY34 verwenden, die sich in FRET / FLIM-Experimenten als gute Akzeptoren für Cyan-Fluoreszenz-Donatoren erwiesen haben.
- Wenn Sie mEGFP als Donor verwenden, sollten Sie die Verwendung von monomeren roten Akzeptoren als mCherry in Betracht ziehen.
- Überprüfen Sie die spektralen Eigenschaften des ausgewählten Donor/Akzeptor-Paares mit den auf der FP-Basis-Website verfügbaren Tools. In Abbildung 1 finden Sie ein Beispiel für das mEGFP/mCherry-Paar.
- Wählen Sie auf der FP-Basis-Website das Dropdown-Menü Extras und dann Spectra viewer aus.
- Geben Sie in den Dropdown-Menüs den Namen des zu visualisierenden Fluorophorpaares ein (z. B. mEGFP und mCherry).
- Simulieren Sie die Eigenschaften des Donor/Akzeptor-Paares mit einer bestimmten Lichtquelle, indem Sie einen bestimmten Laser im Dropdown-Menü auswählen. Alternativ können Sie eine bestimmte Laserwellenlänge eingeben, indem Sie Laser hinzufügen auswählen. Klicken Sie auf die Option Emission normalisieren auf diese Option, um die Fluorophorspektren auf die gewünschte Wellenlänge einzustellen. Hier liegt die Wellenlänge, die zur Anregung von GFP verwendet wird, bei 480 ± 10 nm.
- Klonen Sie das ausgewählte Donor/Akzeptor-Paar, indem Sie einen Fluorophor am N-Terminus der AURKA-Sequenz in voller Länge und einen am C-Terminus hinzufügen. Befolgen Sie die Richtlinien der bevorzugten Klonierungsmethode, um dieses Konstrukt in einen Säugetierexpressionsvektor ihrer Wahl einzufügen.
2. Zellkultur, Transfektion und Synchronisation
- TAG 1. Kulturmedium für U2OS-Zellen vorbereiten: Verwenden Sie Dulbeccos Modified Eagle Medium (DMEM), ergänzt mit 10% Fetal Bovine Serum (FBS), 1% Penicillin-Streptomycin und 1% L-Glutamin (von nun an Complete Growth Media). Alternativ kann auch DMEM mit vorergänztem L-Glutamin verwendet werden.
- Wenn Zellen eingefroren sind, eine Durchstechflasche mindestens 8 Tage vor den Experimenten auftauen (d. h. ab Punkt 2.5).
- Züchtung von Zellen in einem Inkubator für Säugetierzellkulturen bei 37 °C und mit 5% CO2. Reinigen und sterilisieren Sie den Inkubator regelmäßig, um Kontaminationen zu vermeiden.
- Wenn die Zellen ~ 80% Konfluenz erreichen:
- Waschen Sie sie kurz mit steriler 1x Phosphatpuffer-Kochsalzlösung (PBS) ohne Ca2+ und Mg2+.
- Trypsinisieren Sie Zellen mit sterilen 0,05% Trypsin-EDTA gemäß Herstellerprotokoll und indem Sie die Zellen für 1-3 min in den Inkubator legen.
- Inaktivieren Sie Trypsin, indem Sie das doppelte Volumen an vollständigen Wachstumsmedien hinzufügen; gut mischen.
- Zentrifugieren Sie die Zellsuspension bei 800 x g für 3-5 min.
- Zählen Sie die Zellen mit einem Hämozytometer und berechnen Sie die geeignete Verdünnung, damit sie am Tag danach bei ~ 70-80% Konfluenz in Kammerobjektträgern liegen. Alternativ können auch ähnliche Träger für die Lebendzellbildgebung verwendet werden.
- Pipettieren Sie das entsprechende Zellvolumen in der gewählten Lebendzellbildgebungsunterstützung und legen Sie die Zellen bis zum Tag danach wieder in den Inkubator.
- TAG 2. Fahren Sie mit der Transfektion fort. Befolgen Sie die Richtlinien der bevorzugten transienten Transfektionsmethode(n), um eine optimale Transfektionseffizienz (~ 50/80%) zu erhalten. Es ist keine spezifische Transfektionsmethode erforderlich. Beachten Sie, dass die Transfektionseffizienz je nach verwendeter Zelllinie variieren kann. Inkubieren Sie für 48 h.
HINWEIS: Produzieren Sie stabile Klone, die jeden der drei Vektoren enthalten, um die Notwendigkeit transienter Transfektionen zu umgehen. Zu diesem Zeitpunkt sollten zwei Arten von Kontrollen geplant werden. Erstens ist eine "Donor-only"-Kontrolle erforderlich, um zu überprüfen, ob das Vorhandensein von AURKA in voller Länge die Lebensdauer von mTurquoise2 an sich nicht stört. Zweitens sollte ein Biosensor, der eine Kinase-tote Mutation trägt, als Negativkontrolle verwendet werden, bei der die FRET aufgehoben oder signifikant gesenkt wird. Alternativ zu einer Kinase-toten Mutante kann ein chemischer Inhibitor der AURKA-Aktivierung als ATP-Analogon MLN8237 als Negativkontrolle eingesetzt werden.- Planen Sie drei Transfektionsbedingungen im Voraus, jede davon in einem unabhängigen Bohrloch:
Der "Donor-only"-Vektor (z. B. AURKA-mTurquoise2)
Der "Biosensor" (z.B. superYFP-AURKA-mTurquoise2)
Ein Kinase-dead/"K162M"-Biosensor (z. B. superYFP-AURKA K162M-mTurquoise2) oder alternativ ein Inhibitor der AURKA-Aktivierung (z. B. MLN8237) - Führen Sie drei Bedingungen für jedes unabhängige Spender/Akzeptor-Paar aus, um es zu vergleichen.
- Erwägen Sie, die Anzahl der transfizierten Vertiefungen zu verdoppeln, wenn Sie unsynchronisierte und G2/M-synchronisierte Zellen vergleichen (siehe Schritt 2.6).
- Planen Sie drei Transfektionsbedingungen im Voraus, jede davon in einem unabhängigen Bohrloch:
- TAG 3. Synchronisieren Sie die Zellen in G2 / M. Geben Sie 100 ng / ml nocodazol gelöst in DMSO zu jeder transfizierten Vertiefung hinzu, vermeiden Sie Lichteinwirkung und inkubieren Sie für 16 h (vorzugsweise über Nacht). Wenn Sie unsynchronisierte und G2/M-synchronisierte Zellen vergleichen, behandeln Sie jede Transfektionsbedingung mit Nocodazol oder mit einem gleichen Volumen von DMSO. Für eine bessere Synchronisationseffizienz Einweg-Aliquots von Nocodazol in DMSO herstellen, bei -20 °C lagern und nach Gebrauch verwerfen.
HINWEIS: Die Effizienz der Zellsynchronisation kann zwischen den Zelllinien variieren. Die optimale Konzentration von Nocodazol und seine Inkubationszeit sollten vor FRET/FLIM-Experimenten experimentell durch Durchflusszytometrie-Ansätze bestimmt werden. Für statistisch relevante FRET/FLIM-Analysen empfehlen wir eine Synchronisationseffizienz in G2/M von mindestens 50% der gesamten Zellpopulation. - TAG 4. Nocodazol-Auswaschung und FRET/FLIM-Bildgebung auf mitotischen Zellen
- Entfernen Sie das Kulturmedium mit einer Pipette und ersetzen Sie es durch vorgewärmtes, steriles PBS. Vermeiden Sie nach Möglichkeit Lichteinwirkung. Den Teller vorsichtig schaukeln.
- Wiederholen Sie den Waschvorgang und vermeiden Sie immer Lichteinwirkung.
- Entfernen Sie die zweite PBS-Wäsche und ersetzen Sie sie durch vorgewärmtes, steriles Leibovitz L-15 Medium, ergänzt mit 20% Fetal Bovine Serum (FBS) und 1% Penicillin-Streptomycin (von nun an bildgebende Medien).
HINWEIS: Bildmedien sollten ohne pH-Indikatoren (z. B. Phenolrot) und mittlere Komponenten wie Riboflavin erworben werden. Diese Substanzen sind eine Quelle der Autofluoreszenz, die die Lebenszeitwerte stören könnte. - Fahren Sie mit der FRET/FLIM-Bildgebung fort. Minimieren Sie schnelle Temperaturänderungen und fahren Sie so schnell wie möglich mit dem Bildgebungsschritt (Schritt 3) fort. Erwägen Sie, die Probe vor Licht zu schützen, während Sie sie zum Mikroskopaufbau transportieren (z. B. indem Sie sie in eine Aluminiumfolie wickeln oder in eine Schachtel legen).
3. Übernahmen von FRET/FLIM
HINWEIS: DIE FRET/FLIM-Erfassungen in diesem Protokoll wurden in einem benutzerdefinierten Setup durchgeführt, das in35 beschrieben und mit einer Steuerungslösung wie in15,17 (Abbildung 2) ausgestattet ist. Das Setup wird jetzt von Inscoper kommerzialisiert und besteht aus einem rotierenden Scheibenmikroskop mit einem weißen Laser für gepulste Anregung und einem hochgeschwindigkeitsgesteuerten Zeitverstärker vor der Kamera. Zeitliche Gatter von 2 ns in einem Zeitfenster von 10 ns werden sequentiell verwendet, um einen Stapel von fünf zeitgesteuerten Bildern zu erhalten. Diese Bilder werden dann verwendet, um die mittlere Fluoreszenzlebensdauer Pixel für Pixel gemäß der folgenden Gleichung zu berechnen: τ = ΣΔti • Ιi/ΣΙi, wobei Δti der Verzögerungszeit des i-ten Gatters entspricht, während I die pixelweise zeitgesteuerte Intensität angibt Bild35,36 . Diese Methode gewährleistet schnelle FLIM-Messungen: Es sind keine Montage- oder Binning-Schritte erforderlich, und die Lebensdauer kann im Online-Modus mit minimalem Photonenbudget berechnet werden. Das System bietet auch eine benutzerfreundliche Softwareoberfläche. Das gleiche Experiment kann jedoch unter jedem anderen kommerziellen Mikroskop-Setup durchgeführt werden, das für FLIM-Messungen ausgestattet ist.
- Um eine optimale Freisetzung von Zellen aus dem G2/M-Block in die Mitose zu gewährleisten, führen Sie Experimente bei 37 °C durch. Wenn möglich, führen Sie FRET/FLIM-Experimente mit Mikroskop-Setups durch, die mit einer Thermostatkammer ausgestattet sind.
- Schalten Sie die Thermostatkammer des Mikroskops mindestens 30 Minuten bis 1 Stunde vor dem Experiment ein.
- Schalten Sie den Laser, die Kamera, das Mikroskop-Setup und die Bildgebungssoftware ein (Abbildung 2).
- Wählen Sie die geeigneten Anregungs- und Emissionswellenlängen für das Donorfluorophor aus. Praktische Wellenlängenoptionen sind: λex 440/10 nm und λem 483/35 nm für mTurquoise2 (Abbildung 3A); λex 488/10 nm und λem 525/50 nm für GFP) (Abbildung 3B).
- Stellen Sie die Belichtungszeit ein, in der Regel zwischen 30 und 100 ms (Abbildung 3). Beachten Sie, dass übermäßige Laserleistung zu induzierten phototoxischen Effekten wie Photobleichen führen kann, was wiederum die Fluoreszenzlebensdauer verändern könnte37. Validieren Sie im Setup das Fehlen von Photobleichereignissen, indem Sie die Fluoreszenzintensität des ersten Tores während der Zeitraffererfassung überwachen. Wenn Schwankungen der Fluoreszenzintensität beobachtbar sind, verwerfen Sie die Erfassung und passen Sie die Laserleistung an.
HINWEIS: Wählen Sie im Setup hier eine Belichtungszeit aus, die mindestens 3000 Graustufen im ersten Tor zulässt. Andernfalls berechnet die Software nicht die Spenderlebensdauer. Dieser Grauwert entspricht dem minimalen Photonenbudget, das notwendig ist, um relevante Lebensdauerwerte zu erhalten. - Stellen Sie vor dem Start von FRET/ FLIM-Akquisitionen sicher, dass die Zellen in die Mitose eingetreten sind, indem Sie bis zum Auftreten der bipolaren Spindel warten (~ 20/30 min in U2OS-Zellen). Da das mitotische AURKA hauptsächlich an dieser Struktur lokalisiert ist, sollte die mitotische Progression überprüft werden, indem die Bildung der Spindel in Zellen direkt unter dem Mikroskop mit einer externen Lichtquelle abgeschirmt wird (Abbildung 1). Beachten Sie, dass die zeit, die für das mitotische Fortschreiten benötigt wird, je nach verwendeter Zelllinie variieren kann.
- Wenn eine Behandlung mit MLN8237 geplant ist, legen Sie die Zellen unter das Mikroskop und lassen Sie sie die Metaphase erreichen (ca. 20 min nach Nocodazol-Auswaschung). Fügen Sie 250 nM MLN8237, gelöst in DMSO, sowohl zu Zellen, die das reine Spenderkonstrukt exprimieren, als auch zu Zellen, die den Biosensor exprimieren, hinzu.
- Kontrollieren Sie diesen Zustand auf Zellen, die wie oben transfiziert werden, und inkubieren Sie mit einem gleichen Volumen an DMSO. Für eine bessere AURKA-Hemmung Einweg-Aliquots von MLN8237 in DMSO herstellen und bei -80 °C lagern. Tauen Sie sie auf, indem Sie die Aliquots auf Eis legen, und entsorgen Sie sie nach Gebrauch.
- 10 Min. inkubieren. Nach dieser Zeit schrumpft die mitotische Spindel und es bleibt nur noch ein einziger, intensiver Punkt übrig. Ein ähnlicher Phänotyp wird beobachtet, wenn K162M-Mutanten verwendet werden.
- Für eine bessere Auflösung der mitotischen Spindel verwenden Sie mindestens ein 63-faches Objektiv (Abbildung 3).
- Nachdem Sie eine Zelle in der Metaphase gefunden haben (siehe Abbildung 4 als Beispiel für eine Zelle in der Metaphase), passen Sie die xyz-Koordinaten an, um sie in der Mitte des Sichtfelds zu platzieren.
- Für schnellere Bilder wählen Sie eine einzelne Z-Ebene aus. Wählen Sie die Ebene, auf der die mitotische Spindel sichtbarer oder intensiver ist.
- Starten Sie die Aufzeichnung. Die Erfassungszeit kann je nach verwendetem FLIM-Setup variieren (von wenigen Sekunden bis zu minuten). Die Mehrheit der auf dem Markt erhältlichen kommerziellen Setups wird sowohl die Fluoreszenzmikroskopie als auch die Pixel-für-Pixel-Lebensdauerkarte ausarbeiten. Speichern Sie beide Bilder.
- Erfassen Sie mindestens 10 unabhängige Bilder von jeder Transfektion und / oder Behandlungsbedingung.
4. Berechnung der ΔLifetime und Vergleich der FLIM-Werte zwischen Donor/Akzeptor-Paaren
- Extrahieren Sie Lebensdauerwerte aus der gesamten Pixel-für-Pixel-Lebensdauerkarte (d. h. der gesamten mitotischen Spindel) oder wählen Sie Regions of Interest (ROIs) aus, die bestimmten Teilbereichen entsprechen.
HINWEIS: Je nach verwendetem FRET/FLIM-Setup können Lebensdauerberechnungen direkt auf der Erfassungssoftware durchgeführt oder mit generischen Bildverarbeitungslösungen extrahiert werden (z. B. Fiji/ImageJ: https://fiji.sc/). Software, die Die Lebensdauerwerte direkt berechnet (auch online-Modus genannt), bietet eine benutzerfreundlichere Lösung, die für Anfänger und für Mikroskopie-Anwender geeignet ist, die mit FRET/FLIM nicht vollständig vertraut sind. Im Gegenteil, die Extraktion von Lebenszeitwerten nach der Erfassung erfordert oft ein anpassungsfähiges Verfahren. Diese Option ist für Anfänger weniger zugänglich, da einige Vorkenntnisse in mathematischen Anpassungsmodellen erforderlich sind. - Berechnen Sie nach der Visualisierung oder Extraktion die mittlere Lebensdauer der Zellen, die den "Nur-Spender"-Vektor exprimieren (z. B. AURKA-mTurquoise2), hiermit die mittlere Spenderlebensdauer.
- Subtrahieren Sie jeden unabhängigen Lebenszeitwert, der in Schritt 4.1 berechnet wurde, von der mittleren Spenderlebensdauer. Die Wiederholung dieses Schritts für alle Zellen unter allen analysierten Bedingungen ergibt die ΔLifetime für jede Bedingung.
- Vergleichen Sie die ΔLifetime-Werte für die Bedingungen "nur Donor", "Biosensor" und "K162M" oder DMSO und MLN8237.
HINWEIS: Für die Bedingung "nur Spender" sollte ΔLifetime zu Werten nahe Null führen, die den experimentellen Fluktuationen der Lebenszeitwerte entsprechen. Für die Bedingung "Biosensor" sollten ΔLifetime-Werte die Nettodifferenz zwischen den beiden Konstrukten ergeben (siehe Abbildung 4 für ein illustriertes Beispiel). - Vergleichen Sie die ΔLifetime-Werte zwischen verschiedenen Donor/Akzeptor-Paaren.
- Vergleichen Sie die "Biosensor"-Bedingungen: Zeigen sie eine ähnliche ΔLifetime?
- Zeigen die "K162M" oder die MLN8237-Bedingungen eine ähnliche ΔLifetime unter ihnen? Ähnelt ihre ΔLifetime der "Donor-only"-Bedingung?
Ergebnisse
Wir haben das oben beschriebene Verfahren befolgt, um die Autophosphorylierung von AURKA auf Thr288 mit zwei Biosensoren mit unterschiedlichen spektralen Eigenschaften aufzuzeichnen. Wir haben die ursprüngliche GFP-AURKa-mCherry Sonde14 mit zwei Biosensoren mit unterschiedlichen spektralen Eigenschaften verglichen. Diese beiden Sonden stützen sich in einem Fall auf den fluoreszierenden Donor mTurquoise2 und auf einen nicht-fluoreszierenden Akzeptor (ShadowG) oder in einem zweiten Fall auf einen gelben Akzeptor (superYFP). Wir haben dann die AURKA-Sequenz in voller Länge in jedes Spender-/Akzeptorpaar eingefügt. Um eine Negativkontrolle für die AURKA-Aktivierung zu haben, können zwei Strategien verfolgt werden. Erstens stört die Verwendung eines kleinen ATP-Analogons (MLN8237) die Bindung von ATP in der kinetischen Tasche der Kinase und verhindert deren Aktivierung38. Zweitens erzeugt die Mutation von Lys162 in Met (K162M) eine Kinase-tote Version jedes Biosensors, die nicht in der Lage ist, zu aktivieren14,15,39. Diese Mutation induziert die Störung einer Salzbrücke, die normalerweise zwischen Lys162 und Glu181 aufgebaut ist, was zu einer stabilen Öffnung der kinetischen Tasche der Kinase führt und ihre allgemeine Inaktivierung auslöst40. Als Negativkontrolle für FRET verwendeten wir ein akzeptorfreies Konstrukt (GFP-AURKA oder AURKA-mTurquoise2).
Nachdem wir Zellen in G2/M synchronisiert und in die Mitose freigesetzt hatten, haben wir die Lebensdauer aller transfizierten Konstrukte an der mitotischen Spindel gemessen (Abbildung 4). Bemerkenswert ist, dass diese Struktur als Ganzes betrachtet wurde und keine ROIs innerhalb der Spindel analysiert wurden. Wir haben dann ΔLifetime für alle Bedingungen berechnet. Wie erwartet, lag die Lebensdauer von GFP-AURKA oder AURKA-mTurquoise2 (die "Donor-only"-Bedingungen) nahe 0, was darauf hindeutet, dass die für diese Konstrukte gemessenen Werte um den Mittelwert schwankten (Abbildung 4A,4B). Umgekehrt unterschieden sich die ΔLifetime-Werte für GFP-AURKA-mCherry statistisch von der reinen Donor-Bedingung, wobei die ΔLifetime um ~130 ps zunahm (Abbildung 4A). Ähnliche Beobachtungen wurden für shadowG-AURKA-mTurquoise2 und für superYFP-AURKA-mTurquoise2 gemacht, wobei die ΔLifetime gegenüber der reinen Spenderbedingung um ~150 bzw. ~220 ps zunahm (Abbildung 4B,4C). Diese Daten können einfach in einzelnen Zellen mit einer pseudofarbigen Lookup Table (LUT) visualisiert werden. In diesem Fall sind Werte von ΔLifetime um 0 pseudogefärbt gelb, während signifikantere Unterschiede pseudogefärbt rot/violett sind. Tatsächlich war die Pixel-für-Pixel-LUT in Zellen, die die reinen Donor-Konstrukte exprimierten, näher an Gelb, während sie in Zellen, die einen der beiden Biosensoren exprimierten, eher im rot/violetten Spektrum lag (Abbildung 4A,4B). Dies wurde auch beobachtet, als der GFP-AURKA-mCherry-Biosensor mit dem pharmakologischen Inhibitor MLN8237 behandelt wurde.
Wir haben dann die ΔLifetime von Kinase-toten Biosensoren analysiert. Diese Konstrukte zeigten mittlere ΔLifetime-Werte: ΔLifetime war im Vergleich zur reinen Donor-Bedingung signifikant höher (Abbildung 4B,4C), aber auch signifikant niedriger als ihre normalen Gegenstücke (Abbildung 4B,4D). Die Vergleiche mit Zellen, die mit MLN8237 oder exprimierenden Kinase-toten Biosensoren behandelt wurden, sind notwendig, um abzuschätzen, ob ΔLifetime-Variationen für jedes Donor/Akzeptor-Paar ausschließlich mit der Aktivierung von AURKA zusammenhängen. Im Falle von GFP-AURKA-mCherry werden ΔLifetime-Variationen aufgehoben, wenn ein AURKA-spezifischer Inhibitor verwendet wird. Umgekehrt sind ΔLifetime-Variationen bei shadowG-AURKA-mTurquoise2 und bei superYFP-AURKA-mTurquoise2 meist, aber nicht ausschließlich mit der AURKA-Aktivierung verbunden.
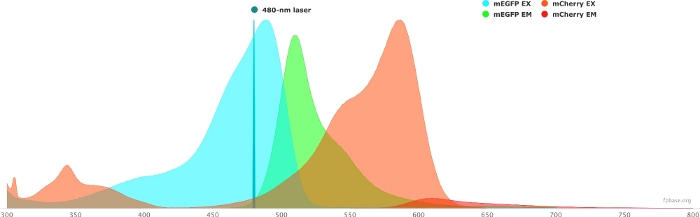
Abbildung 1: GFP ( Donor) und mCherry (Akzeptor) Anregungs- und Emissionsspektren.
Spektren wurden von der FP-Basis-Website (https://www.fpbase.org/) erhalten und angepasst und an eine 480 nm-Laseranregung angepasst. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Abbildung des experimentellen Arbeitsbereichs.
(1) Die Kontrolllösung; (2) weiße Laserquelle; (3) CCD-Kamera; (4) Aufbau des Mikroskops; (5) externe Lichtquelle/Lampe für die Augenabschirmung der Probe. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
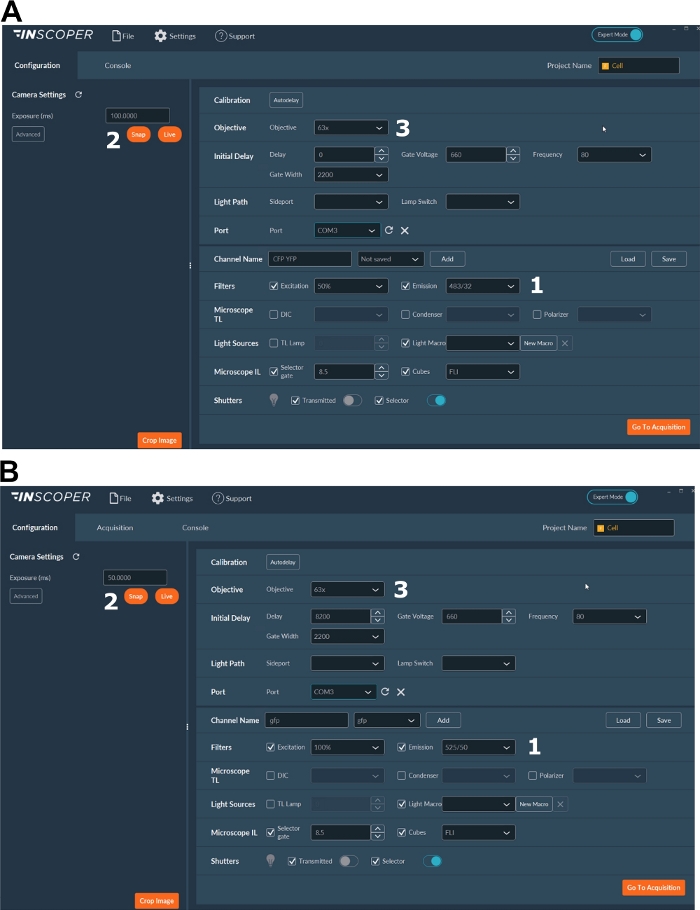
Abbildung 3: Repräsentative Bilder der Software für die FLIM-Erfassung.
(A, B) (1) Anregungs- und Emissionsparameter für den Donor (CFP in A oder GFP in B); (2) Expositionszeit; (3) Auswahl des Ziels. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
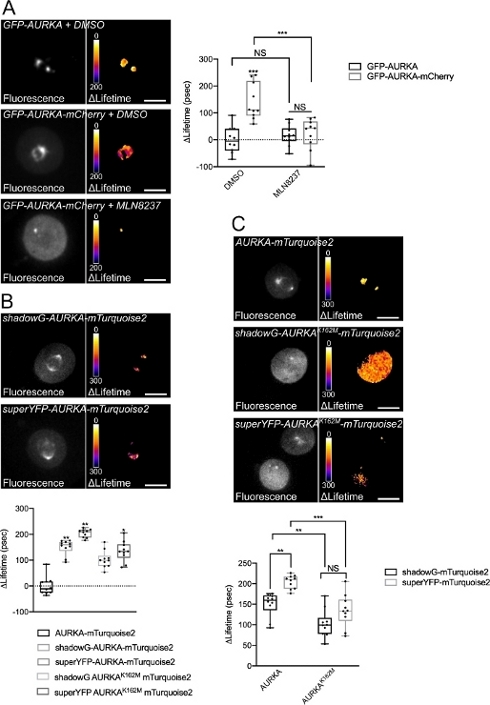
Abbildung 4: Repräsentative Bilder von AURKA FRET Biosensoren und deren Negativkontrollen.
(A) (Mikroaufnahmen) Fluoreszenz (grüner Kanal) und entsprechende Pixel-für-Pixel-ΔLifetime (nur Donor – Biosensor) von U2OS-Zellen, die GFP-AURKA oder GFP-AURKA-mCherry exprimieren, synchronisiert bei G2/M, werden freigesetzt, bis die bipolare Spindel sichtbar ist und dann mit DMSO oder mit MLN8237 behandelt. ΔLifetime wird mit einer Pseudofarbskala illustriert ("Fire"-Nachschlagetabelle). (Grafik) Entsprechende Quantifizierung und Zwei-Wege-ANOVA-Analyse für die angegebenen Bedingungen. (B) (Mikroaufnahmen) Fluoreszenz (cyanfarbener Kanal) und entsprechende Pixel-für-Pixel-ΔLifetime (nur Donor – Biosensor) von U2OS-Zellen, die SchattenG-AURKA-mTurquoise2 (oberes Bild) oder superYFP-AURKA-mTurquoise2 (unteres Panel) exprimieren, synchronisiert bei G2/M und freigesetzt, bis die bipolare Spindel sichtbar ist. ΔLifetime wird mit einer Pseudofarbskala illustriert ("Fire"-Nachschlagetabelle). (Grafik) Entsprechende Quantifizierung und Einweg-ANOVA-Analyse der in den obigen Mikroaufnahmen dargestellten Bedingungen. (C) (Mikroaufnahmen) Bilder von AURKA-mTurquoise2 (oberes Bild), shadowG-AURKA K162M-mTurquoise2 (mittleres Bild) und superYFP-AURKA K162M-mTurquoise2 aufgenommen und dargestellt wie in den Mikroaufnahmen. (Grafik) Zwei-Wege-ANOVA-Analyse für die angegebenen Transfektionsbedingungen. Der Balken in Boxplots stellt den Median dar; Schnurrhaare erstrecken sich von der min bis zur max. n = 10 Zellen pro Bedingung eines repräsentativen Experiments (von drei). Einzelne Werte werden als Punkte dargestellt. Maßstabsbalken: 10 μm. *P < 0,05, **P < 0,01, ***P < 0,001 gegen jede angegebene Bedingung in (A) die "AURKA-mTurquoise2" -Bedingung in (B) und gegen jede angegebene Bedingung in (C). NS: nicht signifikant. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Genetisch kodierte FRET-Biosensoren sind zuverlässige Werkzeuge, um die Aktivierung einzelner Proteine oder ganzer Signalwege zu messen41. Insbesondere der AURKA FRET Biosensor stellt eine bevorzugte Möglichkeit dar, die Aktivierung der Kinase in Zeit und Raum zu erforschen. Einige Elemente verdienen jedoch besondere Aufmerksamkeit bei der Entwicklung oder Optimierung eines FRET-Biosensors, nicht nur allgemein, sondern speziell für AURKA.
Erstens können die Art und die relative Position des Donor/Akzeptor-FRET-Paares für spezifische Funktionen dieser Kinase angepasst werden. AURKA ist während der Mitose an der mitotischen Spindel stark angereichert, kommt aber während des gesamten Zellzyklus und an verschiedenen subzellulären Stellen (z. B. Zentrosomen, Kern und Mitochondrien) vor)1,2. Wenn der Biosensor in bestimmten Kompartimenten wie Mitochondrien eingesetzt werden soll, die einen sauren pH-Wert erreichen können, sollte die Wahl eines pH-insensitiven Donor-Akzeptor-FRET-Paares als mTurquoise2/shadowG getroffen werden. Darüber hinaus könnte die Platzierung des FRET-Donors am C-Terminus eine bessere Visualisierung des Biosensors in diesem subzellulären Kompartiment ermöglichen und möglicherweise sogar den FRET-Nachweis optimieren, da gezeigt wurde, dass AURKA N-Terminus an Mitochondrien teilweise abspaltet16,42.
Zweitens würde eine noch unerforschte Methode zur Optimierung des AURKA FRET-Biosensors ein sorgfältigeres Design der Linker zwischen AURKA in voller Länge und dem Donor/Akzeptor-Paar erfordern. Nicht nur der Abstand zwischen dem Fluoreszenzpaar, sondern auch die Eigenschaften des Linkers selbst erwiesen sich als Schlüsselfaktoren zur Verbesserung der FRET-Effizienz43,44,45,46. Vor diesem Hintergrund könnte eine Erhöhung der Steifigkeit oder Flexibilität des Linkers entweder die FRET-Effizienz beeinträchtigen oder weiter verbessern.
Drittens ist bekannt, dass die Überexpression von AURKA bei einem signifikanten Anteil der Zellen mitotische Spindelanomalien induziert2. Es wäre interessant, die ΔLifetime zu vergleichen, die durch die Expression des gleichen FRET-Konstrukts unter einem starken Promotor wie dem Cytomegalovirus (CMV) – einem der häufigsten Promotoren in Säugetierexpressionsvektoren gefunden wird – oder unter der AURKA-Minimalpromotorsequenz (CTTCCGG)14,47. Es wurde zuvor gezeigt, dass dieser Promotor monopolare oder multipolare Spindeln rettet, die nach dem Abschlag der Kinase entstehen, und seine Verwendung induzierte per se keine Störungen des Zellzyklus14,47. Obwohl FLIM unempfindlich gegen Proteinexpressionsniveaus und relative Konzentrationen in der Zelle ist11, würde ein gründlicher Vergleich der beiden Promotoren auf demselben Biosensor-Setup das Verständnis des Pools von aktiviertem AURKA an einem bestimmten Ort erweitern. Darüber hinaus würde es neue Erkenntnisse darüber liefern, wie sich die AURKA-Aktivierung bei Überexpression ändern kann, was für epitheliale und hämatologische Krebsparadigmen relevant ist.
Schließlich sollte auch die nachgelagerte FRET-Anwendung berücksichtigt werden. Eine Zukunftsperspektive im Bereich AURKA wäre die Kumulierung des Kinase-Konformations-Biosensors mit einem substratbasierten Biosensor. Die gleichzeitige Analyse des FRET-Verhaltens von zwei Biosensoren – ein Prozess, der als Multiplex-FRET bezeichnet wird – erfordert einen dunklen Akzeptor auf dem ersten Biosensor, um ein spektrales Durchbluten im zweiten Donorkanal zu vermeiden. Im Kontext von AURKA würde dies die aufregende neue Perspektive eröffnen, die Aktivierung der Kinase mit dem ersten Biosensor und ihre enzymatische Aktivität gegenüber einem bestimmten Substrat mit dem zweiten zu erkennen. Jüngste Entwicklungen im Multiplexing erlauben es nun, bis zu drei Biosensoren gleichzeitig zu kumulieren48. Die Anwendung einer ähnlichen Methode im Kontext von AURKA könnte eine sehr vielversprechende Strategie darstellen, um nicht nur das Aktivierungs-Aktivitäts-Zusammenspiel der Kinase zu testen, sondern auch AURKA-Signalkaskaden mit beispielloser raumzeitlicher Auflösung zu erforschen.
Zusammenfassend lässt sich sagen, dass FRET/FLIM eine bequeme Möglichkeit ist, das Wissen über die Proteinaktivität zu vertiefen. Auf der einen Seite ermöglicht es, die Lokalisation eines bestimmten Proteins in lebenden Zellen zu visualisieren, dank mindestens einer fluoreszierenden Einheit. Auf der anderen Seite kann es Proteinkonformationsänderungen entwirren, die für die Proteinaktivierung und / oder -aktivität informativ sein könnten. Daher haben FRET/FLIM- und konformationale FRET-Biosensoren das Potenzial, zu weit verbreiteten Methoden zu werden, um Signalwege in lebenden Zellen und mit exquisiter raumzeitlicher Auflösung zu verfolgen.
Offenlegungen
G.B. führte die Experimente durch, schrieb und überprüfte das Manuskript und stellte die Finanzierung zur Verfügung, M.T. überprüfte das Manuskript und bot Unterstützung an. M.T. ist wissenschaftlicher Berater und Gesellschafter der Firma Inscoper (Frankreich), die die in diesem Manuskript gezeigten Lösungen für schnelle FLIM-Messungen herstellt. Inscoper unterstützte teilweise die Open-Access-Publikation des Manuskripts. Inscoper war weder am experimentellen Design, an der Datenverarbeitung noch am Schreiben des Manuskripts beteiligt.
Danksagungen
Wir danken den Ingenieuren des Microscopy-Rennes Imaging Center (MRic, BIOSIT, Rennes, Frankreich) für Rat und Hilfe, insbesondere X. Pinson für die kritische Lektüre des Manuskripts. MRic ist Mitglied der nationalen Infrastruktur France-BioImaging, die von der französischen Nationalen Forschungsagentur (ANR-10-INBS-04) unterstützt wird. Diese Arbeit wurde vom Centre National de la Recherche Scientifique (CNRS), der Ligue Contre le Cancer Comités d'Ille et Vilaine, des Côtes d'Armor et du Finistère und der Association pour la Recherche Contre le Cancer (ARC) zu G.B unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Alisertib (MLN8237) | SelleckChem | S1133 | Use at a 250 nM final dilution |
| Dulbecco's Modified Eagle Medium (DMEM) | ThermoFischer Scientific Brand: Gibco | 41966052 | High glucose + L-glutamine + Sodium pyruvate |
| Fetal Bovine Serum (FBS) | ThermoFischer Scientific Brand: Gibco | 10270106 | |
| L15 | ThermoFischer Scientific Brand: Gibco | 21083027 | Leibovitz's L15 medium + L-glutamine, no Phenol red |
| LabTek | Nunc | 2515380 | |
| Nocodazole | Merck Brand: Sigma-Aldrich | M1404 | Use at a 100 ng/mL final dilution |
| Penicillin/Streptomycin | ThermoFischer Scientific Brand: Gibco | 15140122 | Penicillin-Streptomycin 10,000 U/mL (100x) |
| Phosphate Buffer Saline (PBS) | ThermoFischer Scientific Brand: Gibco | 14190169 | DPBS, no calcium, no magnesium |
| Trypsin/EDTA | ThermoFischer Scientific Brand: Gibco | 25300096 | Trypsin-EDTA 0.05%, Phenol Red (1x) |
Referenzen
- Bertolin, G., Tramier, M. Insights into the non-mitotic functions of Aurora kinase A: more than just cell division. Cellular and Molecular Life Sciences. , (2019).
- Nikonova, A. S., Astsaturov, I., Serebriiskii, I. G., Dunbrack, R. L., Golemis, E. A. Aurora A kinase (AURKA) in normal and pathological cell division. Cellular and Molecular Life Sciences. 70 (4), 661-687 (2013).
- Walter, A. O., Seghezzi, W., Korver, W., Sheung, J., Lees, E. The mitotic serine/threonine kinase Aurora2/AIK is regulated by phosphorylation and degradation. Oncogene. 19 (42), 4906-4916 (2000).
- Cheetham, G. M. T. Crystal Structure of Aurora-2, an Oncogenic Serine/Threonine Kinase. Journal of Biological Chemistry. 277 (45), 42419-42422 (2002).
- Bayliss, R., Sardon, T., Vernos, I., Conti, E. Structural basis of Aurora-A activation by TPX2 at the mitotic spindle. Molecular Cell. 12 (4), 851-862 (2003).
- Zhang, Y., et al. Identification of the auto-inhibitory domains of Aurora-A kinase. Biochemical and Biophysical Research Communications. 357 (2), 347-352 (2007).
- Littlepage, L. E., Wu, H., Andresson, T., Deanehan, J. K., Amundadottir, L. T., Ruderman, J. V. Identification of phosphorylated residues that affect the activity of the mitotic kinase Aurora-A. Proceedings of the National Academy of Sciences of the United States of America. 99 (24), 15440-15445 (2002).
- Kufer, T. A., et al. Human TPX2 is required for targeting Aurora-A kinase to the spindle. The Journal of Cell Biology. 158 (4), 617-623 (2002).
- Eyers, P. A., Erikson, E., Chen, L. G., Maller, J. L. A novel mechanism for activation of the protein kinase Aurora A. Current Biology. 13 (8), 691-697 (2003).
- Brunet, S., et al. Characterization of the TPX2 Domains Involved in Microtubule Nucleation and Spindle Assembly in Xenopus Egg Extracts. Molecular Biology of the Cell. 15 (12), 5318-5328 (2004).
- Padilla-Parra, S., Tramier, M. FRET microscopy in the living cell: Different approaches, strengths and weaknesses. BioEssays. 34 (5), 369-376 (2012).
- Aoki, K., Kamioka, Y., Matsuda, M. Fluorescence resonance energy transfer imaging of cell signaling from in vitro to in vivo: Basis of biosensor construction, live imaging, and image processing. Development, Growth & Differentiation. 55 (4), 515-522 (2013).
- Fuller, B. G., et al. Midzone activation of aurora B in anaphase produces an intracellular phosphorylation gradient. Nature. 453 (7198), 1132-1136 (2008).
- Bertolin, G., et al. A FRET biosensor reveals spatiotemporal activation and functions of aurora kinase A in living cells. Nature Communications. 7, 12674 (2016).
- Bertolin, G., et al. Optimized FRET Pairs and Quantification Approaches To Detect the Activation of Aurora Kinase A at Mitosis. ACS Sensors. 4 (8), 2018-2027 (2019).
- Bertolin, G., et al. Aurora kinase A localises to mitochondria to control organelle dynamics and energy production. eLife. 7, (2018).
- Sizaire, F., Le Marchand, G., Pécréaux, J., Bouchareb, O., Tramier, M. Automated screening of AURKA activity based on a genetically encoded FRET biosensor using fluorescence lifetime imaging microscopy. Methods and Applications in Fluorescence. 8 (2), 024006 (2020).
- Demeautis, C., et al. Multiplexing PKA and ERK1&2 kinases FRET biosensors in living cells using single excitation wavelength dual colour FLIM. Scientific Reports. 7, 41026 (2017).
- Ringer, P., et al. Multiplexing molecular tension sensors reveals piconewton force gradient across talin-1. Nature Methods. 14 (11), 1090-1096 (2017).
- Nagai, T., Yamada, S., Tominaga, T., Ichikawa, M., Miyawaki, A. Expanded dynamic range of fluorescent indicators for Ca(2+) by circularly permuted yellow fluorescent proteins. Proceedings of the National Academy of Sciences of the United States of America. 101 (29), 10554-10559 (2004).
- Klarenbeek, J., Goedhart, J., van Batenburg, A., Groenewald, D., Jalink, K. Fourth-Generation Epac-Based FRET Sensors for cAMP Feature Exceptional Brightness, Photostability and Dynamic Range: Characterization of Dedicated Sensors for FLIM, for Ratiometry and with High Affinity. PLOS ONE. 10 (4), 0122513 (2015).
- Fritz, R. D., et al. A Versatile Toolkit to Produce Sensitive FRET Biosensors to Visualize Signaling in Time and Space. Science Signaling. 6 (285), (2013).
- Mastop, M., et al. Characterization of a spectrally diverse set of fluorescent proteins as FRET acceptors for mTurquoise2. Scientific Reports. 7 (1), 11999 (2017).
- vander Krogt, G. N. M., Ogink, J., Ponsioen, B., Jalink, K. A Comparison of Donor-Acceptor Pairs for Genetically Encoded FRET Sensors: Application to the Epac cAMP Sensor as an Example. PLoS ONE. 3 (4), 1916 (2008).
- Cranfill, P. J., et al. Quantitative assessment of fluorescent proteins. Nature Methods. 13 (7), 557-562 (2016).
- Goedhart, J., et al. Structure-guided evolution of cyan fluorescent proteins towards a quantum yield of 93. Nature Communications. 3 (1), (2012).
- Mérola, F., et al. Newly engineered cyan fluorescent proteins with enhanced performances for live cell FRET imaging. Biotechnology Journal. 9 (2), 180-191 (2014).
- Erard, M., et al. Minimum set of mutations needed to optimize cyan fluorescent proteins for live cell imaging. Molecular BioSystems. 9 (2), 258-267 (2013).
- Tramier, M., Zahid, M., Mevel, J. C., Masse, M. J., Coppey-Moisan, M. Sensitivity of CFP/YFP and GFP/mCherry pairs to donor photobleaching on FRET determination by fluorescence lifetime imaging microscopy in living cells. Microscopy Research and Technique. 69 (11), 933-939 (2006).
- Padilla-Parra, S., et al. Quantitative Comparison of Different Fluorescent Protein Couples for Fast FRET-FLIM Acquisition. Biophysical Journal. 97 (8), 2368-2376 (2009).
- Burgstaller, S., et al. pH-Lemon, a Fluorescent Protein-Based pH Reporter for Acidic Compartments. ACS Sensors. , (2019).
- Bindels, D. S., et al. mScarlet: a bright monomeric red fluorescent protein for cellular imaging. Nature Methods. 14 (1), 53-56 (2016).
- Murakoshi, H., Shibata, A. C. E., Nakahata, Y., Nabekura, J. A dark green fluorescent protein as an acceptor for measurement of Förster resonance energy transfer. Scientific Reports. 5, 15334 (2015).
- Murakoshi, H., Shibata, A. C. E. ShadowY: a dark yellow fluorescent protein for FLIM-based FRET measurement. Scientific Reports. 7 (1), 6791 (2017).
- Leray, A., Padilla-Parra, S., Roul, J., Héliot, L., Tramier, M. Spatio-Temporal Quantification of FRET in living cells by fast time-domain FLIM: a comparative study of non-fitting methods [corrected]. PloS One. 8 (7), 69335 (2013).
- Padilla-Parra, S., Audugé, N., Coppey-Moisan, M., Tramier, M. Quantitative FRET analysis by fast acquisition time domain FLIM at high spatial resolution in living cells. Biophysical Journal. 95 (6), 2976-2988 (2008).
- Song, L., Hennink, E. J., Young, I. T., Tanke, H. J. Photobleaching kinetics of fluorescein in quantitative fluorescence microscopy. Biophysical Journal. 68 (6), 2588-2600 (1995).
- Manfredi, M. G., et al. Characterization of Alisertib (MLN8237), an investigational small-molecule inhibitor of aurora A kinase using novel in vivo pharmacodynamic assays. Clinical Cancer Research: An Official Journal of the American Association for Cancer Research. 17 (24), 7614-7624 (2011).
- Katayama, H., et al. Phosphorylation by aurora kinase A induces Mdm2-mediated destabilization and inhibition of p53. Nature Genetics. 36 (1), 55-62 (2004).
- Nowakowski, J., et al. Structures of the Cancer-Related Aurora-A, FAK, and EphA2 Protein Kinases from Nanovolume Crystallography. Structure. 10 (12), 1659-1667 (2002).
- Palmer, A. E., Qin, Y., Park, J. G., McCombs, J. E. Design and application of genetically encoded biosensors. Trends in Biotechnology. 29 (3), 144-152 (2011).
- Grant, R., et al. Constitutive regulation of mitochondrial morphology by Aurora A kinase depends on a predicted cryptic targeting sequence at the N-terminus. Open Biology. 8 (6), 170272 (2018).
- Shimozono, S., Miyawaki, A. Engineering FRET Constructs Using CFP and YFP. Methods in Cell Biology. 85, 381-393 (2008).
- Komatsu, N., et al. Development of an optimized backbone of FRET biosensors for kinases and GTPases. Molecular Biology of the Cell. 22 (23), 4647-4656 (2011).
- Schifferer, M., Griesbeck, O. A Dynamic FRET Reporter of Gene Expression Improved by Functional Screening. Journal of the American Chemical Society. 134 (37), 15185-15188 (2012).
- Peroza, E. A., Boumezbeur, A. H., Zamboni, N. Rapid, randomized development of genetically encoded FRET sensors for small molecules. Analyst. 140 (13), 4540-4548 (2015).
- Reboutier, D., et al. Aurora A is involved in central spindle assembly through phosphorylation of Ser 19 in P150Glued. The Journal of Cell Biology. 201 (1), 65-79 (2013).
- Mo, G. C. H., Posner, C., Rodriguez, E. A., Sun, T., Zhang, J. A rationally enhanced red fluorescent protein expands the utility of FRET biosensors. Nature Communications. 11 (1), 1848 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten