Method Article
Monitoraggio in tempo reale dell'attivazione dell'aurora chinasi A utilizzando biosensori FRET conformazionali in cellule vive
In questo articolo
Riepilogo
L'attivazione della Ser/Thr chinasi multifunzionale AURKA è caratterizzata dalla sua autofosforilazione su Thr288. Le sonde fluorescenti che si basano su FRET possono discriminare tra i suoi stati inattivi e attivati. Qui, illustriamo alcune strategie per l'ingegneria della sonda, insieme a un protocollo FRET rapido per seguire l'attivazione della chinasi durante la mitosi.
Abstract
I tumori epiteliali sono spesso caratterizzati dalla sovraespressione della Ser/Thr chinasi Aurora A/AURKA. AURKA è una proteina multifunzionale che si attiva con la sua autofosforilazione su Thr288. L'abbondanza di AURKA raggiunge il picco nella mitosi, dove controlla la stabilità e la fedeltà del fuso mitotico e l'efficienza complessiva della mitosi. Sebbene ben caratterizzato a livello strutturale, manca un monitoraggio coerente dell'attivazione di AURKA durante tutto il ciclo cellulare. Una possibile soluzione consiste nell'utilizzare biosensori FRET (Resonance Energy Transfer) di Förster codificati geneticamente per ottenere informazioni sull'autofosforilazione di AURKA con sufficiente risoluzione spaziotemporale. Qui, descriviamo un protocollo per progettare biosensori FRET che rilevano l'autofosforilazione Thr288 e come seguire questa modifica durante la mitosi. In primo luogo, forniamo una panoramica delle possibili coppie FRET donatore/accettore e mostriamo possibili metodi di clonazione e inserimento di biosensori AURKA FRET in cellule di mammifero. Quindi, forniamo un'analisi passo-passo per misurazioni FRET rapide mediante microscopia di imaging a fluorescenza (FLIM) su una configurazione personalizzata. Tuttavia, questo protocollo è applicabile anche alle soluzioni commerciali alternative disponibili. Concludiamo considerando i controlli FRET più appropriati per un biosensore basato su AURKA e evidenziando potenziali miglioramenti futuri per aumentare ulteriormente la sensibilità di questo strumento.
Introduzione
Aurora chinasi A/AURKA è una serina/treonina chinasi multifunzionale, attiva durante tutto il ciclo cellulare e in diversi compartimenti subcellulari1. Comprendere la sua ampia attivazione spaziotemporale è particolarmente importante nel cancro, poiché l'AURKA è spesso sovraespresso nelle neoplasie epiteliali ed ematologiche, con pazienti che mostrano scarsa reattività alle terapie attualmente disponibili2.
Studi strutturali hanno rivelato che AURKA subisce due fasi per convertire da una chinasi inattiva a una attiva. In primo luogo, l'autofosforilazione di Thr288 cambia la conformazione della tasca cinetica della chinasi e la attiva3,4,5,6. Questo passaggio aumenta l'attività catalitica di AURKA nelle cellule umane e in Xenopus laevis3,6,7, innescando la chinasi per la piena attività. Una volta attivato, l'interazione di AURKA con la proteina bersaglio di Xklp2 (TPX2) induce un secondo cambiamento conformazionale5. Questa ulteriore modifica consente ad AURKA di raggiungere la piena attività enzimatica verso i suoi substrati nella cellula5,8,9,10.
Per quasi due decenni, le intuizioni sull'attivazione e l'attività di AURKA sono state ottenute principalmente attraverso una combinazione di approcci biochimici. Questi includono la rilevazione di Thr288 fosforilato nelle cellule o in vivo come segno distintivo dell'attivazione di AURKA, analisi cristallografiche e saggi in vitro o in cellulo chinasi per sondare l'attività di AURKA1. Tuttavia, la risoluzione spazio-temporale di questi approcci è scarsa o assente, e sono state necessarie nuove soluzioni per ampliare la conoscenza delle dinamiche di questi due eventi.
Lo sviluppo di sonde fluorescenti negli ultimi anni ha facilitato il monitoraggio di AURKA in cellule vive, permettendo il seguito della sua attivazione con maggiore risoluzione spaziotemporale. I sensori più specifici per AURKA sviluppati finora si basano sul principio FRET (Förster's Resonance Energy Transfer)11 per discriminare tra AURKA inattivo e attivo. Il primo sensore sviluppato è stato un biosensore basato su substrato dell'attività chinasica AURKA. I biosensori basati su substrato sono costituiti da una breve sequenza aminoacidica mirata da una data chinasi per la fosforilazione, e inserita all'interno di una coppia FRET donatore/accettore e da un dominio legante che riconosce il residuo fosforilato, che aiuta il ripiegamento del biosensore per un efficiente processo FRET12. Nel caso di AURKA, un frammento di 14-amminoacidi di KIF2C bersaglio della fosforilazione è stato inserito tra una coppia donatore/accettore CFP-YFP13. Tuttavia, questo sensore presenta alcuni importanti inconvenienti. In primo luogo, la sequenza KIF2C utilizzata in questa sonda può essere presa di mira sia da AURKA che dalla chinasi AURKB strettamente correlata, diminuendo così la specificità di questo biosensore. In secondo luogo, il sensore si basa sulla chinasi endogena per la fosforilazione. Pertanto, l'efficienza FRET può essere non rilevabile o non significativa se le quantità della chinasi sono limitanti (ad esempio, nei compartimenti subcellulari o nelle fasi del ciclo cellulare). Per superare queste limitazioni, è stata creata una nuova classe di sensori AURKA nota come "sensori conformazionali". In queste sonde, la sequenza a lunghezza intera di AURKA è stata inserita all'interno di un fluoroforo donatore al capolinea N e un fluoroforo accettore al C-terminus. L'AURKA inattivo presenta una conformazione "aperta", che allontana l'uno dall'altro i termini N- e C della chinasi. Con tale distanza tra i due termini (> 10 nm), la coppia donatore/accettore è in una configurazione non permissiva per FRET. Al contrario, l'AURKA autofosforilato adotta una conformazione "chiusa", con i due termini proteici e i due fluorofori in prossimità. Ciò ha dimostrato di consentire il FRET tra il donatore e l'accettore, che può essere misurato utilizzando le variazioni nella vita del donatore14,15. Tali sonde presentano diversi vantaggi. In primo luogo, sono geneticamente codificati e possono essere utilizzati per sostituire la chinasi endogena nella cellula. In secondo luogo, salvano i fenotipi indotti dall'abbattimento di AURKA, indicando che sono funzionali nella cellula. In terzo luogo, consentono di seguire l'attivazione della chinasi in diversi compartimenti subcellulari e durante tutto il ciclo cellulare. Le sonde hanno rilevato l'attivazione di AURKA in luoghi in cui è nota l'attivazione della chinasi (cioè centrosomi e fuso mitotico) e hanno anche partecipato alla scoperta dell'attivazione di AURKA nei mitocondri16. Infine, questi sensori hanno permesso screening ad alto contenuto basati su FRET/FLIM, in cui i cambiamenti conformazionali di AURKA sono stati utilizzati per identificare nuovi inibitori farmacologici17.
Nel presente lavoro, descriviamo una procedura per visualizzare l'attivazione di AURKA in cellule in coltura. In primo luogo, faremo un'idea delle potenziali coppie di fluorofori per FRET. La scelta della coppia donatore/accettore più adatta sarà effettuata in base alla configurazione del microscopio disponibile, o ad una particolare applicazione a valle come multiplex FRET18,19. Quindi, proponiamo una pipeline per esplorare il comportamento dei biosensori scelti in una rapida configurazione del microscopio FRET / FLIM. Questa pipeline si estenderà dalle procedure di coltura cellulare e sincronizzazione all'acquisizione FLIM e all'analisi dei dati. Infine, discuteremo i potenziali vantaggi di questo protocollo, in quanto una strategia analoga per la progettazione di biosensori potrebbe essere applicata ad altre chinasi e potrebbe anche essere utilizzata con altri sistemi di imaging basati su FRET.
Protocollo
NOTA: le cellule U2OS utilizzate in questo protocollo sono state acquistate da American Type Culture Collection (ATCC, HTB-96) e sono state testate prive di micoplasma. I passaggi da 2.1 a 2.7 devono essere eseguiti sotto un cappuccio a flusso laminare per mantenere sterili cellule e reagenti.
1. Scelta della coppia DONATORE/ACCETTORE FRET
- Fare riferimento alla letteratura per la scelta delle coppie FRET donatore/accettore più adatte. Esempi utili si possono trovare in20,21,22,23,24, anche se la scelta finale deve essere fatta in base alle caratteristiche del setup FRET/FLIM (linee laser disponibili, filtri, ecc.). Di seguito sono riportate alcune considerazioni su come selezionare una coppia donatore/accettore.
- Scelta del donatore: fare riferimento alla base FP (https://www.fpbase.org/) per una serie completa di informazioni sulle proteine fluorescenti disponibili. Questo database è costantemente aggiornato con tutti i fluorofori di nuova concezione.
- Fare riferimento al Fluorescent Biosensor Database (https://biosensordb.ucsd.edu/index.php) per maggiori informazioni sui biosensori già disponibili in letteratura, insieme alle rispettive proteine fluorescenti utilizzate.
- Come punto di partenza generale, scegli un fluoroforo donatore luminoso. Buoni candidati sono proteine fluorescenti ciano come mTFP1 o ECFP, o varianti GFP come EGFP o mEGFP.
- Gli oligomeri possono influenzare la localizzazione e/o la funzione delle proteine25. Considerare l'utilizzo di mutanti monomerici di CFP come mTurquoise226 o Aquamarine27,28. Queste varianti hanno anche una buona resa quantistica e coefficienti di estinzione, che li rendono buoni candidati come donatori fret.
- Dare la preferenza alle proteine fluorescenti (sia come donatori che come accettori) insensibili ai cambiamenti ambientali come il pH intracellulare23, o al fotosbiancamento25, poiché l'efficienza fret può essere fortemente influenzata da questi parametri29. Al giorno d'oggi, fluorofori come mTurquoise2 o mTFP1 sono ampiamente utilizzati come donatori, grazie alla loro buona fotostabilità22,25,26.
- Scelta dell'accettore: i donatori di ciano sono spesso abbinati a varianti di proteina fluorescente gialla (YFP), come mVenus, Citrino e YPet20,21,22,30. Tuttavia, va notato che queste proteine hanno una sensibilità molto maggiore al pH e mostrano globalmente una scarsa fotostabilità.
- Prendere in considerazione l'utilizzo di varianti gialle di YFP di nuova concezione e insensibili al pH come pH-Lemon31, fluorofori verdi come mNeonGreen23 o fluorofori rossi come mScarlet-I23,32 come accettori fluorescenti precedentemente convalidati per mTurquoise2.
- In alternativa, considera l'utilizzo di derivati non fluorescenti / scuri di YFP come ShadowG33 o ShadowY34, che hanno dimostrato di comportarsi come buoni accettori per i donatori fluorescenti di ciano negli esperimenti FRET / FLIM.
- Se si utilizza mEGFP come donatore, prendere in considerazione l'utilizzo di accettori rossi monomerici come mCherry.
- Verificare le proprietà spettrali della coppia donatore/accettore scelta utilizzando gli strumenti disponibili sul sito Web di base FP. Vedere la Figura 1 per un esempio della coppia mEGFP/mCherry.
- Sul sito Web di base FP, selezionare il menu a discesa Strumenti e selezionare Visualizzatore Spectra.
- Nei menu a discesa, inserisci il nome della coppia di fluorofori da visualizzare (ad esempio, mEGFP e mCherry).
- Simulare le proprietà della coppia donatore/accettore con una specifica sorgente luminosa selezionando un determinato laser nel menu a discesa. In alternativa, immettere una lunghezza d'onda laser specifica selezionando Aggiungi laser. Fare clic su Normalizza emissione a questa opzione per regolare gli spettri del fluoroforo alla lunghezza d'onda desiderata. Qui, la lunghezza d'onda utilizzata per eccitare la GFP è a 480 ± 10 nm.
- Clonare la coppia donatore/accettore selezionata aggiungendo un fluoroforo al capolinea N della sequenza a lunghezza intera di AURKA e uno al capolinea C. Seguire le linee guida del metodo di clonazione preferito per inserire questo costrutto in un vettore di espressione mammifero di scelta.
2. Coltura cellulare, trasfezione e sincronizzazione
- GIORNO 1. Preparare il terreno di coltura per le cellule U2OS: utilizzare il Modified Eagle Medium (DMEM) di Dulbecco integrato con il 10% di siero bovino fetale (FBS), l'1% di penicillina-streptomicina e l'1% di L-glutammina (da qui in poi, mezzi di crescita completi). In alternativa, DMEM con L-Glutammina pre-integrata può anche essere utilizzato.
- Se le cellule sono congelate, scongelare un flaconcino almeno 8 giorni prima degli esperimenti (cioè dal punto 2.5 in poi).
- Coltiva cellule in un incubatore dedicato alle colture cellulari di mammiferi a 37 °C e con il 5% di CO2. Pulire e sterilizzare regolarmente l'incubatore per evitare contaminazioni.
- Quando le cellule raggiungono ~ 80% di confluenza:
- Lavarli brevemente con 1x tampone fosfato sterile soluzione salina (PBS) senza Ca2+ e Mg2+.
- Tripsinizzare le cellule con psisina-EDTA sterile allo 0,05% secondo il protocollo del produttore e posizionando le cellule nell'incubatore per 1-3 minuti.
- Inattivare la tripsina aggiungendo il doppio del volume dei mezzi di crescita completi; mescolare bene.
- Centrifugare la sospensione cellulare a 800 x g per 3-5 min.
- Contare le cellule usando un emocitometro e calcolare la diluizione appropriata per loro di essere a ~ 70-80% di confluenza nelle diapositive della camera il giorno dopo. In alternativa, è possibile utilizzare anche supporti simili per l'imaging di cellule vive.
- Pipettare il volume corrispondente di cellule nel supporto di imaging delle cellule vive scelto e riposizionare le cellule nell'incubatrice fino al giorno successivo.
- GIORNO 2. Procedere con la trasfezione. Seguire le linee guida dei metodi di trasfezione transitoria preferiti per ottenere un'efficienza di trasfezione ottimale (~ 50/80%). Non è richiesto alcun metodo di trasfezione specifico. Si noti che l'efficienza di trasfezione può variare in base alla linea cellulare utilizzata. Incubare per 48 h.
NOTA: Produrre cloni stabili contenenti ciascuno dei tre vettori per bypassare la necessità di trasfezioni transitorie. In questa fase, dovrebbero essere pianificati due tipi di controlli. In primo luogo, è necessario un controllo "solo donatore" per verificare che la presenza di AURKA a lunghezza intera non perturbi la durata di mTurquoise2 di per sé. In secondo luogo, un biosensore portatore di una mutazione chinasi-morte dovrebbe essere usato come controllo negativo in cui il FRET viene abolito o significativamente abbassato. In alternativa a un mutante morto di chinasi, un inibitore chimico dell'attivazione di AURKA come l'analogo ATP MLN8237 può essere usato come controllo negativo.- Pianifica in anticipo tre condizioni di trasfezione, ognuna delle quali in un pozzo indipendente:
Il vettore "solo donatore" (ad esempio, AURKA-mTurquoise2)
Il "biosensore" (ad esempio, superYFP-AURKA-mTurquoise2)
Un biosensore chinasi-morto/"K162M" (ad esempio, superYFP-AURKA K162M-mTurquoise2) o, in alternativa, un inibitore dell'attivazione di AURKA (ad esempio, MLN8237) - Eseguire tre condizioni per ogni coppia donatore/accettore indipendente da confrontare.
- Considerare la possibilità di raddoppiare il numero di pozzetti trasfettati se si confrontano celle non sincronizzate e sincronizzate con G2/M (vedere il passaggio 2.6).
- Pianifica in anticipo tre condizioni di trasfezione, ognuna delle quali in un pozzo indipendente:
- GIORNO 3. Sincronizzare le cellule in G2/M. Aggiungere 100 ng/mL di nocodazolo disciolto in DMSO a ciascun pozzo trasfettato evitando l'esposizione alla luce e incubare per 16 ore (preferibilmente durante la notte). Se si confrontano cellule non sincronizzate e sincronizzate con G2/M, trattare ogni condizione di trasfezione con nocodazolo o con un volume uguale di DMSO. Per una migliore efficienza di sincronizzazione, preparare aliquote monouso di nocodazolo in DMSO, conservarle a -20 °C ed eliminarle dopo l'uso.
NOTA: l'efficienza della sincronizzazione delle celle può variare tra le linee cellulari. La concentrazione ottimale di nocodazolo e il suo tempo di incubazione devono essere determinati sperimentalmente mediante approcci di citometria a flusso prima degli esperimenti FRET/FLIM. Per le analisi FRET/FLIM statisticamente rilevanti, raccomandiamo un'efficienza di sincronizzazione in G2/M di almeno il 50% della popolazione cellulare complessiva. - GIORNO 4. Washout di nocodazolo e imaging FRET/FLIM su cellule mitotiche
- Rimuovere il terreno di coltura con una pipetta e sostituirlo con PBS sterile preriscaldato. Evitare l'esposizione alla luce, se possibile. Scuotere delicatamente il piatto.
- Ripetere la procedura di lavaggio, evitando sempre l'esposizione alla luce.
- Rimuovere il secondo lavaggio PBS e sostituirlo con un mezzo Leibovitz L-15 sterile preriscaldato, integrato con il 20% di siero bovino fetale (FBS) e l'1% di penicillina-streptomicina (da qui in poi, mezzi di imaging).
NOTA: i supporti di imaging devono essere acquistati senza indicatori di pH (ad esempio, rosso fenolo) e componenti medi come riboflavina. Queste sostanze sono una fonte di autofluorescenza che potrebbe perturbare i valori di vita. - Procedere con l'imaging FRET/FLIM. Riduci al minimo i rapidi cambiamenti di temperatura e procedi alla fase di imaging (passaggio 3) il più rapidamente possibile. Prendi in considerazione la possibilità di proteggere il campione dalla luce mentre lo trasporti al microscopio (ad esempio, avvolgendolo in un foglio di alluminio o mettendolo in una scatola).
3. Acquisizioni FRET/FLIM
NOTA: le acquisizioni FRET/FLIM in questo protocollo sono state eseguite su una configurazione personalizzata, descritta in35 e dotata di una soluzione di controllo come in15,17 (Figura 2). La configurazione è ora commercializzata da Inscoper ed è costituita da un microscopio a disco rotante con un laser bianco per l'eccitazione pulsata e un intensificatore time-gated ad alta velocità davanti alla telecamera. Le porte temporali di 2 ns in una finestra temporale di 10 ns vengono utilizzate in sequenza per ottenere una pila di cinque immagini time-gated. Queste immagini vengono quindi utilizzate per calcolare la durata media di fluorescenza pixel per pixel secondo la seguente equazione: τ = ΣΔti • Ιi/ΣΙi, dove Δti corrisponde al tempo di ritardo dell'i-esima porta mentre I indica l'immagine di intensità tempo-dipendente pixel per pixel35,36 . Questo metodo garantisce misurazioni FLIM rapide: non sono necessarie fasi di montaggio o di binning e la durata può essere calcolata in modalità online, con un budget minimo di fotoni. Il sistema presenta anche un'interfaccia software user-friendly. Tuttavia, lo stesso esperimento può essere eseguito con qualsiasi altra configurazione di microscopio commerciale equipaggiata per misurazioni FLIM.
- Per garantire un rilascio ottimale delle cellule dal blocco G2/M nella mitosi, eseguire esperimenti a 37 °C. Se possibile, eseguire esperimenti FRET/FLIM con configurazioni di microscopio dotate di camera termostatica.
- Accendere la camera termostatica del microscopio almeno 30 minuti a 1 ora prima dell'esperimento.
- Accendere il laser, la fotocamera, la configurazione del microscopio e il software di imaging (Figura 2).
- Selezionare le lunghezze d'onda di eccitazione ed emissione appropriate per il fluoroforo donatore. Le opzioni di lunghezze d'onda convenienti sono: λex 440/10 nm e λem 483/35 nm per mTurquoise2 (Figura 3A); λex 488/10 nm e λem 525/50 nm per GFP) (Figura 3B).
- Impostare il tempo di esposizione, generalmente compreso tra 30 e 100 ms (Figura 3). Attenzione che un'eccessiva potenza del laser può provocare effetti fototossici indotti come il fotosbiancamento, che potrebbe a sua volta modificare la durata della fluorescenza37. Durante il setup, convalidare l'assenza di eventi di fotosbiancamento monitorando l'intensità di fluorescenza del primo gate durante le acquisizioni time-lapse. Se sono osservabili variazioni nell'intensità della fluorescenza, scartare l'acquisizione e regolare la potenza del laser.
NOTA: nella configurazione qui, selezionare un tempo di esposizione che consenta almeno 3000 livelli di grigio nel primo gate; in caso contrario, il software non calcolerà la durata del donatore. Questo valore del livello di grigio corrisponde al budget minimo di fotoni necessario per ottenere valori di vita rilevanti. - Prima di avviare le acquisizioni FRET/FLIM, assicurarsi che le cellule siano entrate nella mitosi aspettando fino alla comparsa del fuso bipolare (~20/30 min nelle cellule U2OS). Poiché l'AURKA mitotico si localizza principalmente in questa struttura, verificare la progressione mitotica schermando la formazione del fuso nelle cellule direttamente al microscopio, con una sorgente luminosa esterna (Figura 1). Si noti che il tempo necessario per la progressione mitotica potrebbe variare in base alla linea cellulare utilizzata.
- Se è previsto un trattamento con MLN8237, posizionare le cellule al microscopio e consentire loro di raggiungere la metafase (circa 20 minuti dopo il washout del nocodazolo). Aggiungere 250 nM MLN8237 disciolti in DMSO sia alle cellule che esprimono il costrutto solo donatore che alle cellule che esprimono il biosensore.
- Controllare questa condizione sulle cellule trasfettate come sopra e incubare con un volume uguale di DMSO. Per una migliore inibizione dell'AURKA, preparare aliquote monouso di MLN8237 in DMSO, conservarle a -80 °C. Scongelarli mettendo le aliquote sul ghiaccio e scartarle dopo l'uso.
- Incubare per 10 min. Dopo questo periodo, il fuso mitotico si restringerà e rimarrà solo un singolo punto intenso. Un fenotipo simile si osserva quando vengono utilizzati mutanti K162M.
- Per una migliore risoluzione del fuso mitotico, utilizzare almeno un obiettivo 63x (Figura 3).
- Una volta trovata una cella in metafase (vedi Figura 4 come esempio di cella in metafase), regola le coordinate xyz per posizionarla al centro del campo visivo.
- Per immagini più veloci, selezionare un singolo piano z . Scegli il piano in cui il fuso mitotico è più visibile o intenso.
- Avviare la registrazione. Il tempo di acquisizione può variare in base alla configurazione FLIM utilizzata (da pochi secondi a min). La maggior parte delle configurazioni commerciali disponibili sul mercato elaborerà sia la micrografia a fluorescenza che la mappa della durata pixel per pixel. Salvare entrambe le immagini.
- Acquisire almeno 10 immagini indipendenti da ogni trasfezione e/o condizione di trattamento.
4. Calcolo di ΔLifetime e confronto dei valori FLIM tra coppie donatore/accettore
- Estrai i valori di durata dall'intera mappa della durata pixel per pixel (cioè l'intero fuso mitotico) o seleziona le regioni di interesse (ROI) corrispondenti a sottoregioni specifiche.
NOTA: in base alla configurazione FRET/FLIM utilizzata, i calcoli della durata possono essere eseguiti direttamente sul software di acquisizione o estratti con soluzioni generiche di elaborazione delle immagini (ad esempio, Fiji/ImageJ: https://fiji.sc/). Il software che calcola direttamente i valori di durata (noto anche come modalità online) offre una soluzione più user-friendly, adatta ai principianti e agli utenti di microscopia che non hanno piena familiarità con FRET / FLIM. Al contrario, l'estrazione dei valori di vita dopo l'acquisizione richiede spesso una procedura appropriata. Questa opzione è meno accessibile ai principianti, in quanto sono necessarie alcune conoscenze precedenti sui modelli matematici di adattamento. - Una volta visualizzato o estratto, calcola la vita media delle cellule che esprimono il vettore "solo donatore" (ad esempio, AURKA-mTurquoise2), con la presente significa la durata del donatore.
- Sottrarre ogni lifetime value indipendente calcolato nel punto 4.1 dalla vita media del donatore. Ripetendo questo passaggio per tutte le celle in tutte le condizioni analizzate si otterrà il ΔLifetime per ogni condizione.
- Confrontare i valori di ΔLifetime per le condizioni "solo donatore", "biosensore" e "K162M", o DMSO e MLN8237.
NOTA: Per la condizione "solo donatore", ΔLifetime dovrebbe generare valori prossimi allo zero e corrispondenti alle fluttuazioni sperimentali dei valori di vita. Per la condizione "biosensore", i valori di ΔLifetime dovrebbero produrre la differenza netta tra i due costrutti (vedere la Figura 4 per un esempio illustrato). - Confronta i valori di ΔLifetime tra diverse coppie donatore/accettore.
- Confronta le condizioni dei "biosensori": mostrano ΔLifetime simili?
- Le condizioni "K162M" o MLN8237 mostrano un ΔLifetime simile tra loro? Il loro ΔLifetime è simile alla condizione "solo donatore"?
Risultati
Abbiamo seguito la procedura sopra descritta per registrare l'autofosforilazione di AURKA su Thr288 utilizzando due biosensori con proprietà spettrali diverse. Abbiamo confrontato la sonda iniziale GFP-AURKa-mCherry14 con due biosensori con proprietà spettrali diverse. Queste due sonde si basano sul donatore fluorescente mTurquoise2 e su un accettore non fluorescente (ShadowG) in un caso, o un accettore giallo (superYFP) in un secondo caso. Abbiamo quindi inserito la sequenza completa di AURKA all'interno di ogni coppia donatore/accettore. Per avere un controllo negativo per l'attivazione di AURKA, è possibile perseguire due strategie. In primo luogo, l'uso di un piccolo analogo ATP (MLN8237) interferisce con il legame dell'ATP nella tasca cinetica della chinasi e ne impedisce l'attivazione38. In secondo luogo, la mutazione di Lys162 in Met (K162M), crea una versione morta chinasi di ciascun biosensore incapace di attivare14,15,39. Questa mutazione induce la rottura di un ponte salino normalmente stabilito tra Lys162 e Glu181, che si traduce in un'apertura stabile della tasca cinetica della chinasi e innesca la sua inattivazione complessiva40. Come controllo negativo per FRET, abbiamo usato un costrutto accettore-vuoto (GFP-AURKA o AURKA-mTurquoise2).
Dopo aver sincronizzato le cellule in G2/M e averle rilasciate in mitosi, abbiamo misurato la durata di tutti i costrutti trasfettati nel fuso mitotico (Figura 4). Da notare, questa struttura è stata considerata nel suo insieme e non sono stati analizzati ROI all'interno del mandrino. Abbiamo quindi calcolato ΔLifetime per tutte le condizioni. Come previsto, la durata di vita di GFP-AURKA o AURKA-mTurquoise2 (le condizioni "solo donatore") era vicina a 0, indicando che i valori misurati per questi costrutti oscillavano intorno al valore medio (Figura 4A,4B). Al contrario, i valori di ΔLifetime per GFP-AURKA-mCherry erano statisticamente diversi dalla condizione di solo donatore, con ΔLifetime in aumento di ~ 130 ps (Figura 4A). Osservazioni simili sono state fatte per shadowG-AURKA-mTurquoise2 e per superYFP-AURKA-mTurquoise2, con ΔLifetime che aumenta rispettivamente di ~150 e ~220 ps dalla condizione di solo donatore (Figura 4B,4C). Questi dati possono essere facilmente visualizzati in singole celle con una tabella di ricerca (LUT) pseudocolore. In questo caso, i valori di ΔLifetime intorno a 0 sono pseudocolori di giallo, mentre le differenze più significative sono pseudocolori rosso/viola. In effetti, la LUT pixel-by-pixel era più vicina al giallo nelle cellule che esprimevano i costrutti del solo donatore, mentre era più nello spettro rosso / viola nelle cellule che esprimono entrambi i biosensori (Figura 4A,4B). Ciò è stato osservato anche quando il biosensore GFP-AURKA-mCherry è stato trattato con l'inibitore farmacologico MLN8237.
Abbiamo quindi analizzato il ΔLifetime dei biosensori chinasi-morti. Questi costrutti mostravano valori intermedi di ΔLifetime: ΔLifetime era significativamente più alto rispetto alla condizione di solo donatore (Figura 4B,4C), ma era anche significativamente inferiore rispetto alle loro controparti normali (Figura 4B,4D). I confronti con cellule trattate con MLN8237 o biosensori che esprimono chinasi-morte sono necessari per stimare se le variazioni di ΔLifetime per ciascuna coppia donatore/accettore siano legate esclusivamente all'attivazione di AURKA. Nel caso di GFP-AURKA-mCherry, le variazioni ΔLifetime sono abolite quando viene utilizzato un inibitore specifico di AURKA. Al contrario, le variazioni ΔLifetime sono per lo più, ma non esclusivamente legate all'attivazione di AURKA nel caso di shadowG-AURKA-mTurquoise2 e di superYFP-AURKA-mTurquoise2.
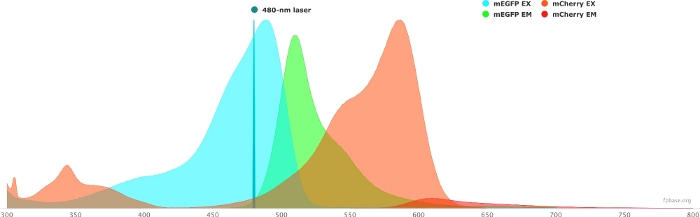
Figura 1: Spettri di eccitazione ed emissione GFP (donatore) e mCherry (accettore).
Gli spettri sono stati ottenuti e adattati dal sito web di base FP (https://www.fpbase.org/) e adattati a un'eccitazione laser a 480 nm. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Immagine dell'area di lavoro sperimentale.
(1) La soluzione di controllo; (2) sorgente laser bianca; (3) Telecamera CCD; (4) configurazione del microscopio; (5) sorgente luminosa/lampada esterna per lo screening oculare del campione. Fare clic qui per visualizzare una versione più grande di questa figura.
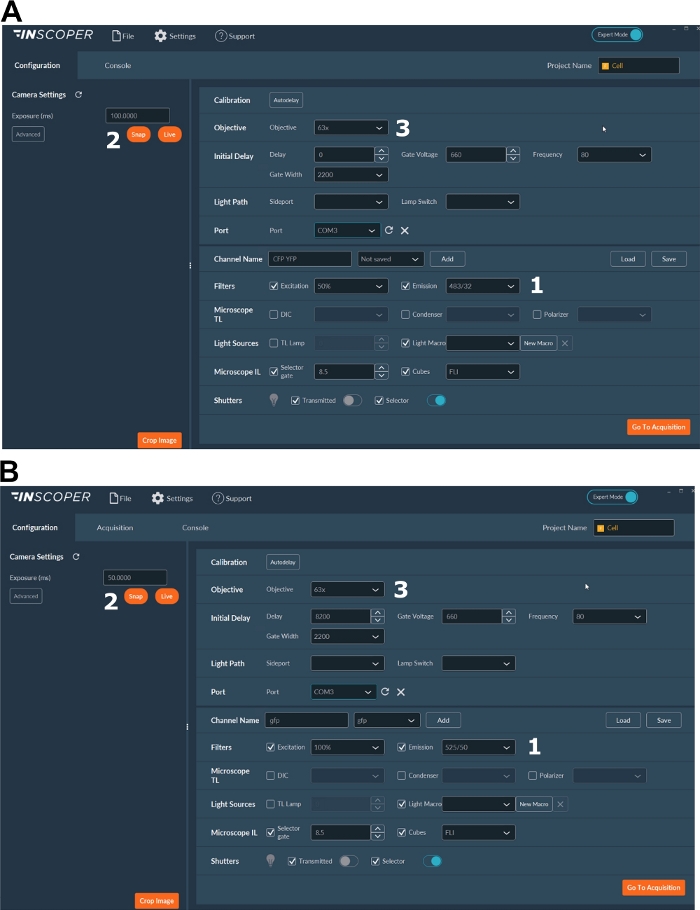
Figura 3: Immagini rappresentative del software per l'acquisizione FLIM.
(A, B) (1) parametri di eccitazione ed emissione per il donatore (CFP in A o GFP in B); (2) tempo di esposizione; (3) selezione dell'obiettivo. Fare clic qui per visualizzare una versione più grande di questa figura.
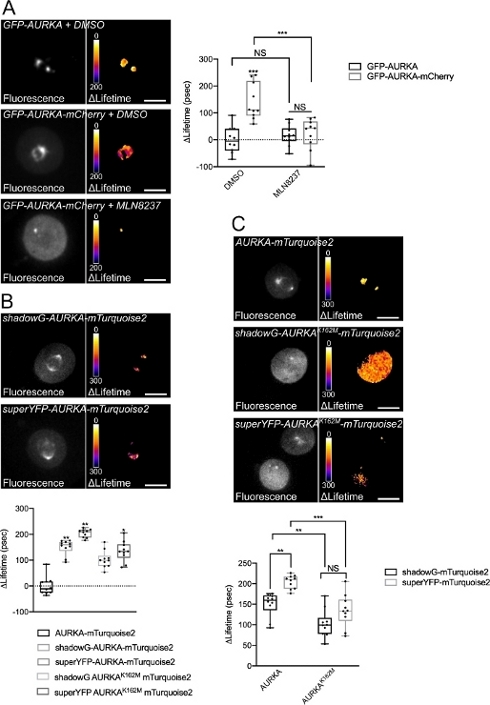
Figura 4: Immagini rappresentative dei biosensori AURKA FRET e dei loro controlli negativi.
(A) (Micrografie) Fluorescenza (canale verde) e corrispondente pixel per pixel ΔLifetime (solo donatore – biosensore) di cellule U2OS che esprimono GFP-AURKA o GFP-AURKA-mCherry, sincronizzate a G2/M, rilasciate fino a quando il fuso bipolare è visibile e quindi trattate con DMSO o con MLN8237. ΔLifetime è illustrato con una scala di pseudocolori (tabella di ricerca "Fire"). (Grafico) Quantificazione corrispondente e analisi ANOVA bidirezionale per le condizioni indicate. (B) (Micrografie) Fluorescenza (canale ciano) e corrispondente pixel per pixel ΔLifetime (solo donatore – biosensore) di cellule U2OS che esprimono shadowG-AURKA-mTurquoise2 (pannello superiore) o superYFP-AURKA-mTurquoise2 (pannello inferiore), sincronizzate a G2/M e rilasciate fino a quando il mandrino bipolare è visibile. ΔLifetime è illustrato con una scala di pseudocolori (tabella di ricerca "Fire"). (Grafico) Quantificazione corrispondente e analisi ANOVA unidirezionale delle condizioni rappresentate nelle micrografie di cui sopra. (C) (Micrografie) Immagini di AURKA-mTurquoise2 (pannello superiore), shadowG-AURKA K162M-mTurquoise2 (pannello centrale) e superYFP-AURKA K162M-mTurquoise2 acquisite e rappresentate come nelle micrografie. (Grafico) Analisi ANOVA bidirezionale per le condizioni di trasfezione indicate. La barra nei boxplots rappresenta la mediana; i baffi si estendono dal min al max. n = 10 cellule per condizione di un esperimento rappresentativo (di tre). I singoli valori sono rappresentati come punti. Barra della scala: 10 μm. *P < 0,05, **P < 0,01, ***P < 0,001 contro ogni condizione indicata in (A) la condizione "AURKA-mTurquoise2" in (B) e contro ogni condizione indicata in (C). NS: non significativo. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
I biosensori FRET geneticamente codificati sono strumenti affidabili per misurare l'attivazione di singole proteine o di intere vie di segnalazione41. In particolare, il biosensore AURKA FRET costituisce un modo preferenziale per esplorare l'attivazione della chinasi nel tempo e nello spazio. Tuttavia, alcuni elementi meritano un'attenzione particolare quando si progetta o si ottimizza un biosensore FRET, non solo in termini generali ma più specificamente per AURKA.
In primo luogo, la natura e la posizione relativa della coppia FRET donatore/accettore possono essere adattate a funzioni specifiche di questa chinasi. AURKA è notevolmente arricchito nel fuso mitotico durante la mitosi, ma è presente in tutto il ciclo cellulare e in diverse posizioni subcellulari (ad esempio, centrosomi, nucleo e mitocondri)1,2. Se il biosensore deve essere utilizzato in compartimenti specifici come i mitocondri, che possono raggiungere un pH acido, deve essere effettuata la scelta di una coppia FRET donatore-accettore insensibile al pH come mTurquoise2/shadowG. Inoltre, posizionare il donatore FRET al C-terminus potrebbe consentire una migliore visualizzazione del biosensore in questo compartimento subcellulare e potenzialmente anche ottimizzare il rilevamento FRET dato che AURKA N-terminus ha dimostrato di staccarsi parzialmente ai mitocondri16,42.
In secondo luogo, un modo ancora inesplorato di ottimizzare il biosensore AURKA FRET richiederebbe una progettazione più attenta dei linker tra AURKA a lunghezza intera e la coppia donatore/accettore. Non solo la distanza tra la coppia fluorescente, ma anche le proprietà del linker stesso si sono dimostrate fattori chiave per migliorare l'efficienza fret43,44,45,46. In quest'ottica, aumentare la rigidità o la flessibilità del linker potrebbe essere dannoso per l'efficienza fret o migliorarla ulteriormente.
In terzo luogo, è noto che la sovraespressione di AURKA induce anomalie mitotiche del fuso in una percentuale significativa di cellule2. Sarebbe interessante confrontare ΔLifetime ottenuto esprimendo lo stesso costrutto FRET sotto un promotore forte come il citomegalovirus (CMV) – uno dei promotori più comuni trovati nei vettori di espressione dei mammiferi – o sotto la sequenza del promotore minimo AURKA (CTTCCGG)14,47. Questo promotore ha precedentemente dimostrato di salvare fusi monopolari o multipolari che si verificano dopo l'abbattimento della chinasi e il suo uso non ha indotto perturbazioni del ciclo cellulare di per sé14,47. Sebbene FLIM sia insensibile ai livelli di espressione proteica e alle concentrazioni relative nella cellula11, beneficiare di un confronto approfondito dei due promotori sulla stessa configurazione del biosensore amplierebbe la comprensione del pool di AURKA attivato in una data posizione. Inoltre, fornirebbe nuove intuizioni su come l'attivazione di AURKA può cambiare in caso di sovraespressione, che è rilevante per i paradigmi del cancro epiteliale ed ematologico.
Infine, dovrebbe essere presa in considerazione anche l'applicazione FRET a valle. Una prospettiva futura nel campo dell'AURKA sarebbe quella di cumulare il biosensore conformazionale chinasico con un biosensore basato su substrato. L'analisi del comportamento FRET di due biosensori contemporaneamente – un processo noto come multiplex FRET – richiede un accettore scuro sul primo biosensore per evitare il sanguinamento spettrale nel secondo canale donatore. Nel contesto di AURKA, questo aprirebbe la nuova eccitante prospettiva di rilevare l'attivazione della chinasi con il primo biosensore e la sua attività enzimatica verso un dato substrato con il secondo. I recenti sviluppi nel multiplexing consentono ora di accumulare fino a tre biosensori alla volta48. L'applicazione di un metodo simile nel contesto di AURKA potrebbe rappresentare una strategia molto promettente non solo per testare l'interazione attivazione-attività della chinasi, ma anche per esplorare cascate di segnalazione AURKA con risoluzione spaziotemporale senza precedenti.
In conclusione, FRET/FLIM è un modo conveniente per approfondire le conoscenze sull'attività proteica. Da un lato, permette di visualizzare la localizzazione di una data proteina in cellule vive, grazie ad almeno una porzione fluorescente. D'altra parte, può svelare i cambiamenti conformazionali delle proteine, che potrebbero essere informativi sull'attivazione e / o l'attività delle proteine. Pertanto, FRET / FLIM e biosensori FRET conformazionali hanno il potenziale di diventare metodi diffusi per seguire percorsi di segnalazione in cellule vive e con squisita risoluzione spaziotemporale.
Divulgazioni
G.B. eseguì gli esperimenti, scrisse e revisionò il manoscritto e fornì finanziamenti, M.T. recensì il manoscritto e fornì supporto. M.T. è consulente scientifico e azionista della società Inscoper (Francia), che produce le soluzioni per misurazioni rapide FLIM mostrate in questo manoscritto. Inscoper ha parzialmente supportato la pubblicazione Open Access del manoscritto. Inscoper non è stato coinvolto nella progettazione sperimentale, nella gestione dei dati, né nella scrittura del manoscritto.
Riconoscimenti
Ringraziamo gli ingegneri del Microscopy-Rennes Imaging Center (MRic, BIOSIT, Rennes, Francia) per i consigli e l'aiuto, e in particolare X. Pinson per la lettura critica del manoscritto. MRic è membro dell'infrastruttura nazionale France-BioImaging sostenuta dall'Agenzia Nazionale Francese per la Ricerca (ANR-10-INBS-04). Questo lavoro è stato sostenuto dal Centre National de la Recherche Scientifique (CNRS), dalla Ligue Contre le Cancer Comités d'Ille et Vilaine, des Côtes d'Armor et du Finistère e dall'Association pour la Recherche Contre le Cancer (ARC) a G.B.
Materiali
| Name | Company | Catalog Number | Comments |
| Alisertib (MLN8237) | SelleckChem | S1133 | Use at a 250 nM final dilution |
| Dulbecco's Modified Eagle Medium (DMEM) | ThermoFischer Scientific Brand: Gibco | 41966052 | High glucose + L-glutamine + Sodium pyruvate |
| Fetal Bovine Serum (FBS) | ThermoFischer Scientific Brand: Gibco | 10270106 | |
| L15 | ThermoFischer Scientific Brand: Gibco | 21083027 | Leibovitz's L15 medium + L-glutamine, no Phenol red |
| LabTek | Nunc | 2515380 | |
| Nocodazole | Merck Brand: Sigma-Aldrich | M1404 | Use at a 100 ng/mL final dilution |
| Penicillin/Streptomycin | ThermoFischer Scientific Brand: Gibco | 15140122 | Penicillin-Streptomycin 10,000 U/mL (100x) |
| Phosphate Buffer Saline (PBS) | ThermoFischer Scientific Brand: Gibco | 14190169 | DPBS, no calcium, no magnesium |
| Trypsin/EDTA | ThermoFischer Scientific Brand: Gibco | 25300096 | Trypsin-EDTA 0.05%, Phenol Red (1x) |
Riferimenti
- Bertolin, G., Tramier, M. Insights into the non-mitotic functions of Aurora kinase A: more than just cell division. Cellular and Molecular Life Sciences. , (2019).
- Nikonova, A. S., Astsaturov, I., Serebriiskii, I. G., Dunbrack, R. L., Golemis, E. A. Aurora A kinase (AURKA) in normal and pathological cell division. Cellular and Molecular Life Sciences. 70 (4), 661-687 (2013).
- Walter, A. O., Seghezzi, W., Korver, W., Sheung, J., Lees, E. The mitotic serine/threonine kinase Aurora2/AIK is regulated by phosphorylation and degradation. Oncogene. 19 (42), 4906-4916 (2000).
- Cheetham, G. M. T. Crystal Structure of Aurora-2, an Oncogenic Serine/Threonine Kinase. Journal of Biological Chemistry. 277 (45), 42419-42422 (2002).
- Bayliss, R., Sardon, T., Vernos, I., Conti, E. Structural basis of Aurora-A activation by TPX2 at the mitotic spindle. Molecular Cell. 12 (4), 851-862 (2003).
- Zhang, Y., et al. Identification of the auto-inhibitory domains of Aurora-A kinase. Biochemical and Biophysical Research Communications. 357 (2), 347-352 (2007).
- Littlepage, L. E., Wu, H., Andresson, T., Deanehan, J. K., Amundadottir, L. T., Ruderman, J. V. Identification of phosphorylated residues that affect the activity of the mitotic kinase Aurora-A. Proceedings of the National Academy of Sciences of the United States of America. 99 (24), 15440-15445 (2002).
- Kufer, T. A., et al. Human TPX2 is required for targeting Aurora-A kinase to the spindle. The Journal of Cell Biology. 158 (4), 617-623 (2002).
- Eyers, P. A., Erikson, E., Chen, L. G., Maller, J. L. A novel mechanism for activation of the protein kinase Aurora A. Current Biology. 13 (8), 691-697 (2003).
- Brunet, S., et al. Characterization of the TPX2 Domains Involved in Microtubule Nucleation and Spindle Assembly in Xenopus Egg Extracts. Molecular Biology of the Cell. 15 (12), 5318-5328 (2004).
- Padilla-Parra, S., Tramier, M. FRET microscopy in the living cell: Different approaches, strengths and weaknesses. BioEssays. 34 (5), 369-376 (2012).
- Aoki, K., Kamioka, Y., Matsuda, M. Fluorescence resonance energy transfer imaging of cell signaling from in vitro to in vivo: Basis of biosensor construction, live imaging, and image processing. Development, Growth & Differentiation. 55 (4), 515-522 (2013).
- Fuller, B. G., et al. Midzone activation of aurora B in anaphase produces an intracellular phosphorylation gradient. Nature. 453 (7198), 1132-1136 (2008).
- Bertolin, G., et al. A FRET biosensor reveals spatiotemporal activation and functions of aurora kinase A in living cells. Nature Communications. 7, 12674 (2016).
- Bertolin, G., et al. Optimized FRET Pairs and Quantification Approaches To Detect the Activation of Aurora Kinase A at Mitosis. ACS Sensors. 4 (8), 2018-2027 (2019).
- Bertolin, G., et al. Aurora kinase A localises to mitochondria to control organelle dynamics and energy production. eLife. 7, (2018).
- Sizaire, F., Le Marchand, G., Pécréaux, J., Bouchareb, O., Tramier, M. Automated screening of AURKA activity based on a genetically encoded FRET biosensor using fluorescence lifetime imaging microscopy. Methods and Applications in Fluorescence. 8 (2), 024006 (2020).
- Demeautis, C., et al. Multiplexing PKA and ERK1&2 kinases FRET biosensors in living cells using single excitation wavelength dual colour FLIM. Scientific Reports. 7, 41026 (2017).
- Ringer, P., et al. Multiplexing molecular tension sensors reveals piconewton force gradient across talin-1. Nature Methods. 14 (11), 1090-1096 (2017).
- Nagai, T., Yamada, S., Tominaga, T., Ichikawa, M., Miyawaki, A. Expanded dynamic range of fluorescent indicators for Ca(2+) by circularly permuted yellow fluorescent proteins. Proceedings of the National Academy of Sciences of the United States of America. 101 (29), 10554-10559 (2004).
- Klarenbeek, J., Goedhart, J., van Batenburg, A., Groenewald, D., Jalink, K. Fourth-Generation Epac-Based FRET Sensors for cAMP Feature Exceptional Brightness, Photostability and Dynamic Range: Characterization of Dedicated Sensors for FLIM, for Ratiometry and with High Affinity. PLOS ONE. 10 (4), 0122513 (2015).
- Fritz, R. D., et al. A Versatile Toolkit to Produce Sensitive FRET Biosensors to Visualize Signaling in Time and Space. Science Signaling. 6 (285), (2013).
- Mastop, M., et al. Characterization of a spectrally diverse set of fluorescent proteins as FRET acceptors for mTurquoise2. Scientific Reports. 7 (1), 11999 (2017).
- vander Krogt, G. N. M., Ogink, J., Ponsioen, B., Jalink, K. A Comparison of Donor-Acceptor Pairs for Genetically Encoded FRET Sensors: Application to the Epac cAMP Sensor as an Example. PLoS ONE. 3 (4), 1916 (2008).
- Cranfill, P. J., et al. Quantitative assessment of fluorescent proteins. Nature Methods. 13 (7), 557-562 (2016).
- Goedhart, J., et al. Structure-guided evolution of cyan fluorescent proteins towards a quantum yield of 93. Nature Communications. 3 (1), (2012).
- Mérola, F., et al. Newly engineered cyan fluorescent proteins with enhanced performances for live cell FRET imaging. Biotechnology Journal. 9 (2), 180-191 (2014).
- Erard, M., et al. Minimum set of mutations needed to optimize cyan fluorescent proteins for live cell imaging. Molecular BioSystems. 9 (2), 258-267 (2013).
- Tramier, M., Zahid, M., Mevel, J. C., Masse, M. J., Coppey-Moisan, M. Sensitivity of CFP/YFP and GFP/mCherry pairs to donor photobleaching on FRET determination by fluorescence lifetime imaging microscopy in living cells. Microscopy Research and Technique. 69 (11), 933-939 (2006).
- Padilla-Parra, S., et al. Quantitative Comparison of Different Fluorescent Protein Couples for Fast FRET-FLIM Acquisition. Biophysical Journal. 97 (8), 2368-2376 (2009).
- Burgstaller, S., et al. pH-Lemon, a Fluorescent Protein-Based pH Reporter for Acidic Compartments. ACS Sensors. , (2019).
- Bindels, D. S., et al. mScarlet: a bright monomeric red fluorescent protein for cellular imaging. Nature Methods. 14 (1), 53-56 (2016).
- Murakoshi, H., Shibata, A. C. E., Nakahata, Y., Nabekura, J. A dark green fluorescent protein as an acceptor for measurement of Förster resonance energy transfer. Scientific Reports. 5, 15334 (2015).
- Murakoshi, H., Shibata, A. C. E. ShadowY: a dark yellow fluorescent protein for FLIM-based FRET measurement. Scientific Reports. 7 (1), 6791 (2017).
- Leray, A., Padilla-Parra, S., Roul, J., Héliot, L., Tramier, M. Spatio-Temporal Quantification of FRET in living cells by fast time-domain FLIM: a comparative study of non-fitting methods [corrected]. PloS One. 8 (7), 69335 (2013).
- Padilla-Parra, S., Audugé, N., Coppey-Moisan, M., Tramier, M. Quantitative FRET analysis by fast acquisition time domain FLIM at high spatial resolution in living cells. Biophysical Journal. 95 (6), 2976-2988 (2008).
- Song, L., Hennink, E. J., Young, I. T., Tanke, H. J. Photobleaching kinetics of fluorescein in quantitative fluorescence microscopy. Biophysical Journal. 68 (6), 2588-2600 (1995).
- Manfredi, M. G., et al. Characterization of Alisertib (MLN8237), an investigational small-molecule inhibitor of aurora A kinase using novel in vivo pharmacodynamic assays. Clinical Cancer Research: An Official Journal of the American Association for Cancer Research. 17 (24), 7614-7624 (2011).
- Katayama, H., et al. Phosphorylation by aurora kinase A induces Mdm2-mediated destabilization and inhibition of p53. Nature Genetics. 36 (1), 55-62 (2004).
- Nowakowski, J., et al. Structures of the Cancer-Related Aurora-A, FAK, and EphA2 Protein Kinases from Nanovolume Crystallography. Structure. 10 (12), 1659-1667 (2002).
- Palmer, A. E., Qin, Y., Park, J. G., McCombs, J. E. Design and application of genetically encoded biosensors. Trends in Biotechnology. 29 (3), 144-152 (2011).
- Grant, R., et al. Constitutive regulation of mitochondrial morphology by Aurora A kinase depends on a predicted cryptic targeting sequence at the N-terminus. Open Biology. 8 (6), 170272 (2018).
- Shimozono, S., Miyawaki, A. Engineering FRET Constructs Using CFP and YFP. Methods in Cell Biology. 85, 381-393 (2008).
- Komatsu, N., et al. Development of an optimized backbone of FRET biosensors for kinases and GTPases. Molecular Biology of the Cell. 22 (23), 4647-4656 (2011).
- Schifferer, M., Griesbeck, O. A Dynamic FRET Reporter of Gene Expression Improved by Functional Screening. Journal of the American Chemical Society. 134 (37), 15185-15188 (2012).
- Peroza, E. A., Boumezbeur, A. H., Zamboni, N. Rapid, randomized development of genetically encoded FRET sensors for small molecules. Analyst. 140 (13), 4540-4548 (2015).
- Reboutier, D., et al. Aurora A is involved in central spindle assembly through phosphorylation of Ser 19 in P150Glued. The Journal of Cell Biology. 201 (1), 65-79 (2013).
- Mo, G. C. H., Posner, C., Rodriguez, E. A., Sun, T., Zhang, J. A rationally enhanced red fluorescent protein expands the utility of FRET biosensors. Nature Communications. 11 (1), 1848 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon