Method Article
Künstliche Intelligenz Ansätze zur Beurteilung primärer Zilien
In diesem Artikel
Zusammenfassung
Der Einsatz von künstlicher Intelligenz (Ai) zur Analyse von Bildern entwickelt sich im Vergleich zu häufig verwendeten Methoden zu einem leistungsfähigen, weniger voreingenommenen und schnellen Ansatz. Hier trainierten wir Ai, eine zelluläre Organelle, primäre Zilien zu erkennen und Eigenschaften wie Länge und Färbeintensität rigoros und reproduzierbar zu analysieren.
Zusammenfassung
Zilien sind auf Mikrotubuli basierende zelluläre Anhängsel, die als Signalzentren für eine Vielzahl von Signalwegen in vielen Säugetierzelltypen fungieren. Die Zilienlänge ist stark konserviert, streng reguliert und variiert zwischen verschiedenen Zelltypen und Geweben und wurde direkt mit deren Signalfähigkeit in Verbindung gebracht. Zum Beispiel wurde gezeigt, dass Zilien ihre Länge als Reaktion auf die Aktivierung von Ziliar-G-Protein-gekoppelten Rezeptoren verändern. Die genaue und reproduzierbare Messung der Längen zahlreicher Zilien ist jedoch ein zeit- und arbeitsintensiver Vorgang. Aktuelle Ansätze sind auch fehler- und verzerrungsanfällig. Programme für künstliche Intelligenz (Ai) können verwendet werden, um viele dieser Herausforderungen aufgrund von Fähigkeiten zu bewältigen, die die Assimilation, Manipulation und Optimierung umfangreicher Datensätze ermöglichen. Hier zeigen wir, dass ein Ai-Modul trainiert werden kann, um Zilien in Bildern von in vivo- und in vitro-Proben zu erkennen. Nachdem wir das trainierte Ai zur Identifizierung von Zilien verwendet haben, sind wir in der Lage, Anwendungen zu entwerfen und schnell zu nutzen, die Hunderte von Zilien in einer einzigen Probe auf Länge, Fluoreszenzintensität und Co-Lokalisation analysieren. Dieser unvoreingenommene Ansatz erhöhte unser Selbstvertrauen und unsere Strenge beim Vergleich von Proben aus verschiedenen primären neuronalen Preps in vitro sowie über verschiedene Gehirnregionen innerhalb eines Tieres und zwischen Tieren. Darüber hinaus kann diese Technik verwendet werden, um die Ziliendynamik aus jedem Zelltyp und Gewebe mit hohem Durchsatz über mehrere Proben und Behandlungsgruppen hinweg zuverlässig zu analysieren. Letztendlich werden Ki-basierte Ansätze wahrscheinlich zum Standard werden, da sich die meisten Bereiche in Richtung weniger voreingenommener und reproduzierbarerer Ansätze für die Bildaufnahme und -analyse bewegen.
Einleitung
Primäre Zilien sind Sinnesorganellen, die aus den meisten Säugetierzelltypen1,2,3,4herausragen. Sie sind im Allgemeinen einzelgängerische Anhängsel, die für die Koordination verschiedener Zellsignalwege durch die Integration extrazellulärer Signale5,6,7entscheidend sind. Primäre Zilien spielen eine wichtige Rolle während der Embryonalentwicklung und der Homöostase des erwachsenen Gewebes, und eine Störung ihrer Funktion oder Morphologie ist mit mehreren genetischen Störungen verbunden, die zusammen als Ziliopathien bezeichnet werden. Aufgrund der nahezu allgegenwärtigen Natur von Zilien sind Ziliopathien mit einer Vielzahl von klinischen Merkmalen verbunden, die alle Organsysteme beeinflussen können8,9,10,11,12. In Tiermodellen von Ziliopathien manifestiert sich der Verlust der Ziliarstruktur oder Signalfähigkeit in mehreren klinisch relevanten Phänotypen, einschließlich Hyperphagie-assoziierter Adipositas3,13,14,15. In vielen Modellsystemen wurde gezeigt, dass Änderungen der Zilienlänge ihre Signalfähigkeit und Funktionenbeeinflussen 16,17,18,19. Es gibt jedoch mehrere zeitaufwändige und technische Herausforderungen, die mit der genauen und reproduzierbaren Beurteilung der Länge und Zusammensetzung der Zilien verbunden sind.
Das zentralnervöse System (ZNS) der erwachsenen Säugetiere ist ein biologischer Kontext, der eine Herausforderung für das Verständnis der Morphologie und Funktion der Zilien darstellt. Während es scheint, dass Neuronen und Zellen im gesamten ZNS Zilien besitzen, bleibt aufgrund der begrenzten Werkzeuge und Fähigkeiten, diese Zilien zu beobachten und zu analysieren, ein Verständnis ihrer Funktionen schwer fassbar20. Zum Beispiel markiert der prototypische Zilienmarker, acetyliertes α-Tubulin, keine neuronalen Zilien20. Die Schwierigkeit, diese Zilien zu untersuchen, wurde teilweise durch die Entdeckung mehrerer G-Protein-gekoppelter Rezeptoren (GPCR), Signalmaschinen und membranassoziierter Proteine gelöst, die auf der Membran neuronaler Zilien angereichert sind21,22. All diese einfachen Grundbeobachtungen deuten auf die Bedeutung und Vielfalt der ZNS-Zilien hin, die bisher von anderen Geweben beispiellos zu sein scheint. Zum Beispiel kann eine Variation der Zilienlänge und der GPCR-Lokalisation im gesamten Gehirn beobachtet werden, wobei die Längen in bestimmten neuronalen Kernen im Vergleich zu anderen Kernen unterschiedlich sind19,23. In ähnlicher Weise zeigen ihr GPCR-Gehalt und ihre Signalmaschinerie eine Vielfalt basierend auf der neuroanatomischen Lage und dem neuronalen Typ2,24,25,26,27,28,29. Diese einfachen Beobachtungen zeigen, dass die Länge und Zusammensetzung der ZILIEN bei Säugetieren streng reguliert ist, genau wie in Modellorganismen wie Chlamydomonas reinhardtii, aber der Einfluss dieser Längenunterschiede auf die Zilienfunktion, die Signalgebung und letztendlich das Verhalten bleibt unklar16,30,31,32.
Die genaue Messung der Länge und Zusammensetzung der Zilien erweist sich als technische Herausforderung, die anfällig für Benutzerfehler und Irreproduzierbarkeit ist. Derzeit werden Zilien in vivo und in vitro am häufigsten mit immunfluoreszierenden Ansätzen identifiziert, die Ziliarproteine oder mit Zilien angereicherte fluoreszierende Reporterallele33,34,35markieren. Die Längen dieser fluoreszenzmarkierten Zilien werden dann aus einem 2-dimensionalen (2D) Bild mit Linienmesswerkzeugen in Bildanalyseprogrammen wie ImageJ36gemessen. Dieser Prozess ist nicht nur mühsam und arbeitsintensiv, sondern auch anfällig für Verzerrungen und Fehler. Dieselben Hindernisse treten bei der Messung der Zilienintensitäten auf, die auf Veränderungen in der Zilienstruktur hinweisen37. Um die Inkonsistenzen bei diesen Arten von Bildanalysen zu minimieren, werden Programme der künstlichen Intelligenz (AI) immer häufiger und erschwingliche Optionen38.
Ai ist die Weiterentwicklung von Computersystemen, die den Vorteil von Computeralgorithmen und Programmierung nutzen, um Aufgaben auszuführen, die normalerweise menschliche Intelligenz erfordern würden39. KI-Geräten wird beigebracht, wiederkehrende Muster, Parameter und Merkmale wahrzunehmen und Maßnahmen zu ergreifen, um die Chancen auf erfolgreiche Ergebnisse zu maximieren. Ai ist vielseitig und kann trainiert werden, um bestimmte Objekte oder Strukturen von Interesse, wie z. B. Zilien, zu erkennen und dann so programmiert zu werden, dass eine Vielzahl von Analysen an den identifizierten Objekten durchgeführt werden. Daher können komplexe Bilddaten durch Ai38schnell und reproduzierbar erzeugt werden. Automatisierung und KI-Analyse der aufgenommenen Bilder erhöhen die Effizienz und Effizienz und begrenzen gleichzeitig potenzielle menschliche Fehler undVerzerrungen 39. Die Etablierung einer Ai-basierten Methodik zur Identifizierung von Zilien schafft eine konsistente Möglichkeit für alle Forschungsgruppen, Ziliendaten zu analysieren und zu interpretieren.
Hier verwenden wir ein Ai-Modul, um Zilien sowohl in vivo als auch in vitro auf 2D-Bildern zu identifizieren. Anhand einer Reihe von Beispielbildern wird die Ai darauf trainiert, Zilien zu identifizieren. Sobald das Training abgeschlossen ist, wird das designierte Ai verwendet, um eine binäre Maske über Ai identifizierte Zilien in einem Bild anzuwenden. Die von der AI angebrachten Binärdateien können bei Bedarf geändert werden, um sicherzustellen, dass alle Zilien in den Bildern ordnungsgemäß identifiziert werden und eine unspezifische Identifizierung ausgeschlossen wird. Nach der Verwendung der Ai zur Identifizierung von Zilien werden maßgeschneiderte allgemeine Analyseprogramme (GA) verwendet, um verschiedene Analysen wie die Messung der Zilienlänge und der Fluoreszenzintensität durchzuführen. Die gesammelten Daten werden in eine Tabelle exportiert, die leicht gelesen, interpretiert und für statistische Analysen verwendet werden kann (Abbildung 1). Der Einsatz von automatisierter Technologie und Ai zur Identifizierung von Zilien und zur Erzielung spezifischer Messungen zwischen experimentellen Gruppen wird zukünftige Studien unterstützen, die darauf abzielen, den Einfluss der ZNS-Zilienfunktion und -morphologie auf die Zell-Zell-Kommunikation und das Zellverhalten zu verstehen.
Protokoll
1. Raw-Bilder erfassen
- Fixieren und immunomarkieren Sie Proben nach Bedarf20.
- Bildzilien mit einem konfokalen Mikroskop bei maximaler Bittiefe bei gleicher Pixelgröße mit Nyquist-Auflösung.
- Exportieren Sie Bilder als monochrome Tagged Image Format (.tif)-Dateien.
HINWEIS: Dieses Protokoll beschreibt, wie das Ai-Modul speziell in der NIS Elements-Software verwendet wird. Wenn Bilder als .nd2-Dateien aufgenommen wurden, ist es nicht erforderlich, Bilder als .tif Dateien zu exportieren, und der Benutzer kann direkt mit Schritt 2.3 fortfahren. Wenn Bilder auf einem anderen System erworben wurden, kann eine NIS Elements-Lizenz separat erworben werden und .tif Dateien können wie in den nächsten Schritten beschrieben konvertiert werden.
2. Trainiere Ai, um Zilien zu identifizieren
- Öffnen Sie das Trainingsdataset.
- Wählen Sie ca. 50 Beispielbilder mit mindestens einem Cilium pro Frame aus, um die Software zu trainieren und in einen einzigen Ordner zu kopieren. Dieser Ordner wird verwendet, um die Software beim Öffnen der Bilder zu leiten. Öffnen Sie diese 50 Frames innerhalb eines einzelnen ND2-Dokuments mit mindestens einem Cilium pro Frame. Wählen Sie Datei > Importieren/Exportieren > ND-Datei aus Dateisequenz erstellen.
- Wählen Sie den Ordner aus, der das Trainingsdataset enthält. Dadurch wird die Liste der Dateien in der Mitte des Dialogfensters geöffnet. Definieren Sie die Organisation der Dateien manuell mit mindestens einer Option im Dropdown-Menü oben. Die Optionen sind Multipoint (für mehrere maximale Projektionsdateien), Z-Serie (für ein Z-Stapelbild), Zeit (für ein Zeitrafferbild) und Wellenlänge (für Dateien aus mehreren Kanälen).
- Geben Sie die entsprechenden numerischen Werte unter jeder ausgewählten Option ein. Wählen Sie Keine aus, wenn die Optionen nicht ausgewählt sind. Klicken Sie auf Konvertieren, um das ND-Dokument zu öffnen.
- Kalibrieren Sie Bilder.
- Geben Sie die Pixelgröße in die untere linke Ecke des Bildes ein: Klicken Sie mit der rechten Maustaste auf Unkalibrierte > Dokument > Pixelgröße kalibrieren.
- Identifizieren Sie Zilien.
- Identifizieren Sie die Zilien von Hand, indem Sie einzelne Ziliarstrukturen auf allen geöffneten Frames mitHilfe von AutoDetect oder Draw Object in der Binären Symbolleistepräzise nachzeichnen. Dadurch werden binäre Masken auf die Objekte von Interesse gezogen. Diese Binärdateien werden als Beispielobjekte dienen, um die Software zu trainieren, Zilien anhand pixelbasierter Merkmale in zukünftigen experimentellen Bildanalysen zu identifizieren. Wählen Sie > Analysesteuerelemente > binären Symbolleiste > Objekt zeichnen aus.
HINWEIS: Entfernen Sie alle Frames, die keine Binärdateien haben, da die Software nicht mit dem Training beginnt, es sei denn, sie ist in der Lage, Binärdateien auf allen geöffneten Frames zu erkennen.
- Identifizieren Sie die Zilien von Hand, indem Sie einzelne Ziliarstrukturen auf allen geöffneten Frames mitHilfe von AutoDetect oder Draw Object in der Binären Symbolleistepräzise nachzeichnen. Dadurch werden binäre Masken auf die Objekte von Interesse gezogen. Diese Binärdateien werden als Beispielobjekte dienen, um die Software zu trainieren, Zilien anhand pixelbasierter Merkmale in zukünftigen experimentellen Bildanalysen zu identifizieren. Wählen Sie > Analysesteuerelemente > binären Symbolleiste > Objekt zeichnen aus.
- Zug Ai.
- Beginnen Sie mit dem Training der Software. Dadurch wird die Segment.ai-Box des Zuges geöffnet. Wählen Sie NIS.ai > Train Segment.aiaus.
- Wählen Sie im Feld Segment.ai trainieren den Quellkanal aus, der für das Training verwendet werden soll. Wenn Dateien aus mehreren Kanälen geöffnet sind, wählen Sie nur einen Kanal als Quellkanal aus. Wählen Sie dann die entsprechenden Ground-Truth-Binärdateien aus, auf denen Sie die Ai trainieren möchten. Wählen Sie schließlich die Anzahl der Iterationen aus, die zum Trainieren der AI erforderlich sind, abhängig von der Größe und Verteilung der Binärdateien.
HINWEIS: Wenn die Binärdateien in der Umgebung leicht erkennbar und gut über das Bild verteilt sind, benötigt die Software möglicherweise weniger als 1000 Iterationen, um zur Identifizierung der Bilder trainiert zu werden. Wenn die Bilder ein niedriges Signal-Rausch-Verhältnis aufweisen, ist es ideal, während des Trainings mindestens 1000 Iterationen auszuführen, damit die Ai Zilien in Testproben mit hoher Zuverlässigkeit identifizieren kann. - Wählen Sie den Zielordner aus, um die trainierte AI-Datei (.sai) zu speichern, und klicken Sie auf Trainieren, um die Software zu trainieren. Die Software trainiert nun selbst, um Zilien anhand der verfolgten Binärdateien zu identifizieren. Dieser Vorgang dauert mehrere Stunden.
HINWEIS: Während des Trainings zeigt die Software ein Diagramm an, das den Trainingsverlust anzeigt. Die Grafik zeigt zunächst eine Spitze, bevor sie sich auf idealerweise etwa 1% Verlust verjüngt, wo sie für den Rest des Trainings ein Plateau erreicht. Speichern Sie das Diagramm zur späteren Bezugnahme, indem Sie das Kontrollkästchen Diagramm-Screenshot speichern im Feld Train Segment.ai aktivieren (Ergänzende Abbildung 1). - Wenn eine weitere Verfeinerung des Trainings erforderlich ist, setzen Sie das Training mit demselben Datensatz fort. Alternativ können Sie an einem neuen Datensatz mit genau denselben Parametern trainieren. Es ist nicht ratsam, eine bereits trainierte Ai an einem neuen Datensatz mit unterschiedlichen Parametern oder unterschiedlichen Objekten von Interesse zu trainieren. Wählen Sie Train segment.ai > Continue Training On > Wählen Sie Trained AI Fileaus.
3. Identifizieren Sie Zilien mit trainiertem Ai
- Öffnen Sie das experimentelle Dataset.
- Öffnen Sie die experimentellen konfokalen Bilder von Zilien in der Software, indem Sie die Beispieldateien .tif in .nd2-Dateien konvertieren, ähnlich wie in Schritt 2.1. Wählen Sie Datei > Importieren/Exportieren > ND-Datei aus Dateisequenz erstellen.
HINWEIS: Die Bilder sollten die gleiche Pixelgröße haben wie diejenigen, die für das Training der Ai verwendet werden. Wenn Bilder bereits im ND2-Format vorliegen, fahren Sie mit Schritt 3.3 fort.
- Öffnen Sie die experimentellen konfokalen Bilder von Zilien in der Software, indem Sie die Beispieldateien .tif in .nd2-Dateien konvertieren, ähnlich wie in Schritt 2.1. Wählen Sie Datei > Importieren/Exportieren > ND-Datei aus Dateisequenz erstellen.
- Kalibrieren Sie Bilder.
- Geben Sie die Pixelgröße in der unteren linken Ecke des Bildes ein. Klicken Sie mit der rechten Maustaste auf unkalibrierte > Dokument > Pixelgröße kalibrieren.
- Führen Sie die trainierte Ai für die geöffneten Dateien aus.
- Beginnen Sie, die Zilien mit Ai zu identifizieren. Die Software zeichnet nun Binärdateien auf den Zilien basierend auf dem Training, das sie im vorherigen Schritt erhalten hat. Dieser Vorgang dauert einige Sekunden. Wählen Sie NIS.ai > Segment.aiaus.
HINWEIS: Die Software fordert Sie auf, den Kanal auszuwählen, wenn mehrere Kanäle geöffnet sind. Kanäle werden hier nach ihren jeweiligen Namen aufgelistet. Wenn nicht, wird das Kästchen "Mono" automatisch ausgewählt.
- Beginnen Sie, die Zilien mit Ai zu identifizieren. Die Software zeichnet nun Binärdateien auf den Zilien basierend auf dem Training, das sie im vorherigen Schritt erhalten hat. Dieser Vorgang dauert einige Sekunden. Wählen Sie NIS.ai > Segment.aiaus.
- Überprüfen Sie Bilder auf falsch identifizierte Binärdateien.
- Sobald die Ai Zilien identifiziert und Binärdateien gezeichnet hat, überprüfen Sie die Bilder auf falsch identifizierte Objekte. Löschen Sie bei Bedarf alle falsch identifizierten Binärdateien manuell. Wählen Sie > Analysesteuerelemente > binären Symbolleiste > Objekt löschen aus.
4. Messung der Länge und Intensität der Zilien
- Erstellen Sie ein neues Allgemeines Analyse 3 (GA3)-Rezept.
- Nachdem die Zilien identifiziert und segmentiert wurden, analysieren Sie verschiedene Parameter der Zilien wie Längen und Intensitäten mit dem GA3-Tool. Dadurch wird ein neues Fenster mit einem leeren Feld in der Mitte geöffnet, in dem die Analyse definiert wird. Wählen Sie Bild > Neues GA3-Rezeptaus.
- Wählen Sie die zu analysierenden Binärdateien aus.
- Da die Zilien bereits mit Segment.ai segmentiert sind, erkennt GA3 automatisch die entsprechend dem Ai gekennzeichneten Binärdateien und schließt den Knoten ein. Wählen Sie'Binaries > Auto Detect_AI' oder 'Binaries > Draw Object_AI'.
- Wählen Sie die für die Analyse erforderlichen Kanäle aus. GA3 erkennt auch automatisch die Kanäle in den Bildern und zeigt ihre Registerkarten unter Kanäle an.
- Entfernen Sie Objekte, die den Rahmen des Rahmens berühren.
- Da die Ai alle Zilien wie Objekte im Rahmen segmentiert, erkennt sie auch unvollständige Zilien entlang der Ränder des Rahmens. Diese Objekte können entweder manuell in Schritt 3.4 oder automatisch in GA3 entfernt werden. Wählen Sie Binärverarbeitung > Objekte entfernen > Rahmen berühren.
- Wählen Sie Parameter aus, um Zilien zu messen.
- Ziehen Sie die zu messenden Parameter wie Zilienlänge (Länge) und Intensitäten (Summe Objektintensität) per Drag & Drop ab. Verbinden Sie die Knoten mit dem entsprechenden Binärknoten (Verbindung A) und Kanalknoten (Verbindung B). Bewegen Sie den Mauszeiger über die Knotenverbindung, um eine QuickInfo anzuzeigen, zu welcher Verbindung der Knoten gehört. Wählen Sie "Messung > Objektgröße > -länge" und "Messung" > "Objektintensität > "Summe Obj-Intensität"aus.
HINWEIS: Die Knotenlänge verbindet sich nur mit dem Binärknoten, während Sum Obj Intensity sowohl mit dem Binär- als auch mit dem Kanalknoten verbunden ist.
- Ziehen Sie die zu messenden Parameter wie Zilienlänge (Länge) und Intensitäten (Summe Objektintensität) per Drag & Drop ab. Verbinden Sie die Knoten mit dem entsprechenden Binärknoten (Verbindung A) und Kanalknoten (Verbindung B). Bewegen Sie den Mauszeiger über die Knotenverbindung, um eine QuickInfo anzuzeigen, zu welcher Verbindung der Knoten gehört. Wählen Sie "Messung > Objektgröße > -länge" und "Messung" > "Objektintensität > "Summe Obj-Intensität"aus.
- Fügen Sie die Maße in einer einzigen Tabelle an.
- Kombinieren Sie alle Messungen in einer einzigen Ausgabetabelle, indem Sie den Knoten ziehen und ablegen Spalte an das Analyseflussdiagramm anhängen und mit den Messknoten Länge und Summe Obj Intensitätverbinden. Wählen Sie Data Management > Spalte Basic > Append aus.
- Messen Sie Zilien.
- Messen Sie Zilien, indem Sie auf Ausführenklicken. Dieser Prozess dauert einige Augenblicke, um alle Zilien in den experimentellen Bildern zu messen. Die Längen und Intensitäten werden in einem neuen Fenster Analyseergebnisse angezeigt.
HINWEIS: Die Tabelle kann manchmal Daten von Masken enthalten, die Ai als Zilien erkannte, aber zu klein waren, um vom menschlichen Auge erkannt und in Schritt 3.4 eliminiert zu werden. Diese Objekte können vor der statistischen Analyse mithilfe eines Filters aus dem Datensatz entfernt werden. Hier wurde ein Filter von 1 μm für In-vitro-Zilienlängenmessungen in Abbildung 2 und 2 μm für In-vivo-Zilien verwendet. Dies kann vor dem Exportieren von Daten über den folgenden Pfad erfolgen. Wählen Sie das Fenster Analyseergebnisse > Filter definieren > Wert eingeben > Filter verwenden.
- Messen Sie Zilien, indem Sie auf Ausführenklicken. Dieser Prozess dauert einige Augenblicke, um alle Zilien in den experimentellen Bildern zu messen. Die Längen und Intensitäten werden in einem neuen Fenster Analyseergebnisse angezeigt.
- Exportieren Sie Daten für statistische Analysen.
5. Kolokalisierungsstudien
HINWEIS: Die Kolokalisationsanalyse kann in dasselbe GA3-Rezept aufgenommen werden, das für Messungen der Zilienlängen- und Intensitätsanalyse verwendet wird. Wenn Sie dasselbe Rezept verwenden, öffnen Sie dateien wie unten beschrieben und messen Sie die Längen und Intensitäten beider Kanäle zusammen mit den Kolokalisierungskoeffizienten in derselben Analysepipeline.
- Öffnen Sie das experimentelle Dataset.
- Öffnen Sie die experimentellen konfokalen Bilder von Zilien in der Software, indem Sie die Beispieldateien .tif in .nd2-Dateien konvertieren. Wählen Sie Datei > Importieren/Exportieren > ND-Datei aus Dateisequenz erstellen.
- Wählen Sie im Popup-Fenster die Monochrom-Dateien mit 16 Bit Tiefe aus allen gewünschten Kanälen aus dem Fenster-Explorer aus, der sich in der ersten Spalte des Popup-Fensters befindet. Wählen Sie Multipoint oder Z Series aus dem ersten Dropdown-Menü und geben Sie einen Wert ein, der der Gesamtzahl der Bilder bzw. Stacks entspricht.
- Wählen Sie im zweiten Dropdown-Feld Wellenlänge aus, und ändern Sie den Wert in die Gesamtzahl der Kanäle im Ordner. Die Software entsperrt automatisch ein Wellenlängenauswahlfenster am unteren rechten Ende des Popup-Fensters. Verwenden Sie das Dropdown-Menü Farbe, um die Farbe der einzelnen Kanäle auszuwählen. Geben Sie jedem Kanal in der Spalte Name einen anderen Namen an. Sobald alle Informationen aktualisiert wurden, klicken Sie auf Konvertieren. Die Software generiert automatisch eine All-Image-Datei, die mit allen einzelnen Bildern aus allen ausgewählten Kanälen überlagert ist.
- Kalibrieren Sie Bilder.
- Geben Sie die Pixelgröße in der unteren linken Ecke des Bildes ein. Klicken Sie mit der rechten Maustaste auf unkalibrierte > Dokument > Pixelgröße kalibrieren.
- Führen Sie die trainierte AI auf dem ersten Kanal aus.
- Beginnen Sie mit der Identifizierung der Zilien auf einem der geöffneten Kanäle (z. B. ACIII; Abbildung 5A) mit Ai. Die Software zeichnet nun Binärdateien auf ACIII-gekennzeichneten Zilien basierend auf dem Training, das sie für diesen Kanal erhalten hat. Dieser Vorgang dauert einige Sekunden. Wählen Sie NIS.ai > Segment.ai > Quellkanäle > ACIIIaus.
- Führen Sie das trainierte Ai auf dem zweiten Kanal aus.
- Beginnen Sie mit der Identifizierung der Zilien auf dem anderen geöffneten Kanal (z. B. MCHR1; Abbildung 5B) mit Ai. Die Software zeichnet nun Binärdateien auf MCHR1-gekennzeichneten Zilien basierend auf dem Training, das sie für diesen Kanal erhalten hat. Dieser Vorgang wird einige Augenblicke in Anspruch nehmen. Wählen Sie NIS.ai > Segment.ai > Quellkanäle > MCHR1aus.
- Überprüfen Sie Bilder auf falsch identifizierte Binärdateien.
- Sobald die Ai Zilien identifiziert und Binärdateien gezeichnet hat, überprüfen Sie die Bilder auf falsch identifizierte Objekte. Löschen Sie bei Bedarf alle falsch identifizierten Binärdateien manuell. Wählen Sie > Analysesteuerelemente > binären Symbolleiste > Objekt löschenaus.
- Erstellen Sie ein neues GA3-Rezept.
- Nachdem die Zilien identifiziert und segmentiert wurden, fahren Sie mit der Kolokalisierungsanalyse mit dem GA3-Tool fort. Dadurch wird ein neues Fenster mit einem leeren Feld in der Mitte geöffnet, in dem die Analyse definiert wird. Ein Fenster mit allen identifizierten Binärdateien und Kanälen wird generiert. Stellen Sie sicher, dass alle gewünschten Kanäle und Binärdateien, die für die Analyse erforderlich sind, vorhanden und ausgewählt sind. Wählen Sie Bild > Neues GA3-Rezeptaus.
- Entfernen Sie Objekte, die den Rahmen des Rahmens berühren.
- Da die Ai alle zilienartigen Objekte im Rahmen segmentiert, erkennt sie auch unvollständige Zilien entlang der Ränder des Rahmens. Diese Objekte können entweder manuell in Schritt 5.5 oder automatisch in GA3 entfernt werden. Wählen Sie Binärverarbeitung > Objekte entfernen > Rahmen berührenaus.
- Richten Sie den Kolokalisierungspfad in GA3 ein.
- Um die Überlappung der beiden Kanäle innerhalb der Zilien zu messen, verwenden Sie manders Koeffizientenkorrelation. Ziehen Sie den Knoten Manders Coefficient per Drag & Drop in den leeren Raum des GA3-Rezepts und verbinden Sie ihn mit der entsprechenden Binärdatei und den entsprechenden Kanälen. Hier verbindet sich "Verbindung A" mit der ACIII-Binärdatei, "Verbindung B" mit dem MCHR1-Kanal und "Verbindung C" mit dem ACIII-Kanal, um die Überlappung von MCHR1 innerhalb der ACIII-Binärdatei zu bestimmen. Wählen Sie "Messung > Objektverhältnismetrie" > "Manders-Koeffizient"aus.
HINWEIS: Die Software ermöglicht die Messung der Kolokalisation unter Verwendung der Pearson-Koeffizienten-Korrelation mit den gleichen Schritten wie in diesem Protokollbeschrieben 40.
- Um die Überlappung der beiden Kanäle innerhalb der Zilien zu messen, verwenden Sie manders Koeffizientenkorrelation. Ziehen Sie den Knoten Manders Coefficient per Drag & Drop in den leeren Raum des GA3-Rezepts und verbinden Sie ihn mit der entsprechenden Binärdatei und den entsprechenden Kanälen. Hier verbindet sich "Verbindung A" mit der ACIII-Binärdatei, "Verbindung B" mit dem MCHR1-Kanal und "Verbindung C" mit dem ACIII-Kanal, um die Überlappung von MCHR1 innerhalb der ACIII-Binärdatei zu bestimmen. Wählen Sie "Messung > Objektverhältnismetrie" > "Manders-Koeffizient"aus.
- Fügen Sie die Maße in einer einzigen Tabelle an.
- Kombinieren Sie alle Messungen in einer einzigen Ausgabetabelle. Wählen Sie Data Management > Spalte Basic > Append aus.
- Messen Sie die Kolokalisierung.
- Messen Sie Zilien, indem Sie auf Ausführenklicken. Dieser Prozess dauert einige Augenblicke, um alle Zilien in den experimentellen Bildern zu messen. Die Daten werden in einem neuen Fenster Analyseergebnisse angezeigt.
- Exportieren Sie Daten für statistische Analysen.
Ergebnisse
Training Ai zur Identifizierung von Zilien
Das Messen und Bewerten der Strukturlänge und -zusammensetzung von Zilien kann ein langwieriger, zeitaufwendiger und fehleranfälliger Prozess sein. Hier verwenden wir Ai, um Zilien aus einem großen Pool von Bildern genau zu segmentieren und ihre Längen und Intensitäten mit einem Analysewerkzeug zu analysieren (Abbildung 1). Alle KI-Ansätze erfordern Trainingsschritte für ihre Umsetzung. Wir haben eine Trainingspipeline zur Erkennung von Zilien eingerichtet, die durch manuelles Auftragen von Binäre masken auf Ziliarstrukturen durchgeführt wurde. Diese Informationen werden dann verwendet, um die Ai basierend auf Pixeleigenschaften unter den angewendeten Binärdateien zu trainieren. Als allgemeine Richtlinie gilt, dass die Software mehrere Iterationen, etwa 1000, durchläuft und als optimal angesehen wird, wenn die Trainingsverlust- oder Fehlerrate weniger als 1% beträgt. Die Anzahl der Iterationen und Fehler im Trainingsprozess kann jedoch je nach den für das Training verwendeten Beispielbildern variieren. Zum Beispiel betrug die Fehlerquote nach unseren Trainingseinheiten mit in vitro neuronalen Zilienbildern 1,378% im Vergleich zu 3,36% für in vivo Gehirnschnittbilder ( ergänzendeAbbildung 1). Sobald das Training abgeschlossen ist, kann Ai verwendet werden, um Zilien aus experimentellen Bildern innerhalb von Sekunden zu segmentieren, und die resultierenden binären Masken werden zur Messung von Strukturparametern verwendet. Dadurch entfällt die Notwendigkeit, Objekte mit der traditionellen Methode der Intensitätsschwellenwerte zu segmentieren, was in Bildern mit hohem Hintergrundrauschen oder wenn sich Objekte in unmittelbarer Nähe zueinander befinden, schwierig sein kann. Ai reduziert auch das Potenzial für Fehler und Verzerrungen, indem es den gleichen Algorithmus auf alle Bilder anwendet, unabhängig vom Benutzer.
Messung der Zilienlänge mit GA3
Die Zilienlänge ist streng reguliert und mit funktionellen Auswirkungen auf die Ziliarsignalisierung verbunden16,19. Hier haben wir Zilienlängen mit einer Analysepipeline innerhalb der NIS Elements-Software namens General Analysis 3 oder GA3 gemessen. GA3 hilft bei der Kombination mehrerer Tools in einem einzigen Workflow, um benutzerdefinierte Routinen für jedes Experiment zu erstellen. Wir begannen mit der Messung der Zilienlängen in einer Zelllinie. Zilien an Zellen des inneren medullären Sammelgangs (IMCD-3) der Maus wurden mit acetyliertem Tubulin immunmarkiert und mit einem konfokalen Mikroskop abgebildet. Wir haben Zilienlängen mit GA3 gemessen, nachdem wir mit segment.ai segmentiert wurden (Ergänzende Abbildung 3A). Während acetyliertes α-Tubulin bevorzugt im primären Cilium vorkommt, kommt es auch in anderen mikrotubulireichen Regionen wie dem Zytoskelett sowie der zytokinetischen Brücke vor. Die trainierte Ai identifizierte Zilien im Bild richtig, aber keine anderen nicht-ziliarischen, acetylierten Tubulin-positiven Strukturen. Zilien auf IMCD-Zellen reichten von 0,5 μm bis 4,5 μm mit einer durchschnittlichen Länge von 1,8 ± 0,04 μm (Abbildung 2A). Als nächstes testeten wir die Fähigkeit von Ai, Zilienlängen in primären neuronalen Kulturen zu messen. Wir kultivierten Neuronen aus dem Hypothalamus und Hippocampus von neonatalen Mäusen für 10 Tage und immunomarkierten sie mit dem Zilienmarker Adenylatcyclase III (ACIII)21,41. Bei der Analyse neuronaler Kulturen fanden wir es nützlich, vor der statistischen Analyse der Längen einen Filter anzuwenden. Aufgrund eines geringeren Signal-Rausch-Verhältnisses wurden mehrere Objekte unter 1 μm identifiziert, die keine Zilien waren. Daher haben wir die Daten gefiltert, um Objekte mit einer Länge von weniger als 1 μm zu eliminieren, um sicherzustellen, dass nur Zilien analysiert wurden. In kultivierten hypothalamischen Neuronen lagen die Zilienlängen zwischen 2 μm und 7 μm mit einer durchschnittlichen Länge von 3,8 ± 0,19 μm (Abbildung 2B). Interessanterweise waren kultivierte hippokampale neuronale Zilien mit einer mittleren Länge von 6,73 ±0,15 μm länger (Abbildung 2C). Es wurde berichtet, dass verschiedene neuronale Kerne innerhalb des Hypothalamus unterschiedliche Zilienlängen aufweisen und dass diese Zilien ihre Länge als Reaktion auf physiologische Veränderungen in einer kernspezifischen Weise verändern19,23. Daher markierten wir auch hypothalamische Hirnschnitte von erwachsenen männlichen C57BL/6J-Mäusen mit ACIII und bildeten den Bogenkern (ARC) und den paraventrikulären Kern (PVN) ab. Mit GA3 zur Messung der Zilienlängen beobachteten wir, dass die hypothalamischen Zilien in vivo länger auftraten als die In-vitro-Zilien. Insbesondere hypothalamische Zilien in vivo reichen von 1 μm bis etwa 15 μm (Abbildung 3). Es gab keine signifikanten Unterschiede zwischen den Längen der Zilien im PVN (5,54 ± 0,0,42 μm) und denen im ARC (6,16 ± 0,27 μm) (Abbildung 3C)23. In ähnlicher Weise weisen Zilien im Cornu Ammonis (CA1)-Bereich des Hippocampus einen engeren Längenbereich von 1 μm bis 10 μm mit einer durchschnittlichen Länge von 5,28 ± 0,33 μm auf (Abbildung 3). In Übereinstimmung mit zuvor veröffentlichten Studien zeigte unsere Analyse mit Ai- und GA3-Tools, dass Zilien aus verschiedenen Hirnregionen eine Vielfalt in der Längeaufweisen 19,23. Darüber hinaus sind wir mit diesem Ai-Ansatz in der Lage, eine große Anzahl von Zilien schnell zu beurteilen.
Messung der Zilienzusammensetzung mit GA3
Das primäre Cilium ist ein Signalknotenpunkt für viele Signalwege, die verschiedene Arten von Proteinen nutzen, um einzigartige Funktionen wie Motorproteine, intraflagellare Transportproteine und GPCRs auszuführen, um nur einige zu nennen3,24,42,43. Die Aufrechterhaltung der entsprechenden Konzentrationen dieser Proteine im Cilium ist wichtig für das reibungslose Funktionieren und scheint oft vom Zellkontext abhängig zu sein. Die fluoreszierende Markierung dieser Proteine hat es uns nicht nur ermöglicht, sie zu visualisieren, sondern auch ihre Intensitäten als Maß für die Menge des markierten Proteins innerhalb des relativ kleinen Kompartiments20zu quantifizieren. Daher versuchten wir, die Intensitäten eines ziliaren GPCR, Melanin Concentrating Hormone Receptor 1 (MCHR1), in vivo sowohl im ARC als auch im PVN des Hypothalamus erwachsener männlicher Mäuse zu bestimmen24,44. Mit Ai und GA3 haben wir die Längen von MCHR1-positiven Zilien zusammen mit Intensitäten gemessen, um sicherzustellen, dass die gezählten Objekte Zilien waren (Ergänzende Abbildung 3A). Wir eliminierten Objekte nach der Analyse, die weniger als 2 μm lang waren, und analysierten die Intensitäten der verbleibenden binären Masken. Interessanterweise fanden wir heraus, dass die Intensität von Ziliar-MCHR1 in PVN signifikant höher ist als die in ARC, was auf eine stärkere Präsenz von Ziliar-MCHR1 in PVN hinweist (Abbildung 4). Weitere Studien sind erforderlich, um die Bedeutung von Ziliar-MCHR1 in diesen neuronalen Schaltkreisen zu bestimmen. Wir haben auch die Intensitäten von Ziliar-MCHR1 in primär kultivierten Neuronen von Hypothalamus und Hippocampus gemessen. Zilien aus beiden Kulturen zeigen eine breite Verteilung von MCHR1-Intensitäten, was auf das Vorhandensein heterogener neuronaler Populationen hindeutet (Ergänzende Abbildung 2). So ermöglicht der Einsatz ausgefeilter Analysewerkzeuge wie Ai und GA3 die Bewertung der Zilienheterogenität innerhalb desselben Gewebes oder zwischen mehreren Geweben. Es wird interessant sein zu sehen, ob andere neuronale GPCRs ähnliche Unterschiede in ihrer Lokalisation innerhalb von Neuronen desselben Gewebes zeigen und ob sich dies als Reaktion auf physiologische Veränderungen ändert.
Kolokalisierung
Während die Messung der Fluoreszenzintensitäten innerhalb eines vollständigen Bildfeldes einen Eindruck von Protein vermitteln kann, liefert sie keine Informationen wie die räumliche Verteilung oder die Nähe zu anderen nahe gelegenen Proteinen und Zellstrukturen. Hier haben wir die Überlappung von MCHR1 mit ACIII als Zilienmarker gemessen, indem wir die Intensitäten von MCHR1 gegen die von ACIII für jede binäre Maske aufgetragen haben (Abbildung 5). Die Grafik zeigt, dass die Mehrheit der Zilien sowohl für ACIII als auch für MCHR1 positiv ist, obwohl einige Zilien eine stärkere Expression eines Kanals gegenüber dem anderen zeigen. Darüber hinaus gibt es einige Zilien, die das Vorhandensein von ACIII oder MCHR1 zeigen, wie aus den Punkten hervorgeht, die direkt auf der x-Achse bzw. der y-Achse liegen. Um diese Überlappung zu quantifizieren, haben wir manders Überlappungskoeffizienten gemessen und das Ausmaß der MCHR1-Expression in neuronalen Zilien der ARC und PVN40verglichen. Interessanterweise ergab unsere Analyse, dass es einen signifikanten Anstieg der Koeffizienten des PVN (0,6382 ± 0,0151) als im ARC (0,5430 ± 0,0181) gab (Abbildung 5C). Dies steht im Einklang mit unseren früheren Daten, bei denen wir höhere MCHR1-Intensitäten in PVN im Vergleich zu ARC beobachtet haben (Abbildung 4). Diese Daten deuten darauf hin, dass das Expressionsmuster von MCHR1 im Ziliarkompartiment wie die Zilienlänge in verschiedenen Regionen des Gehirns variiert. Mit der gleichen Analysepipeline wird es möglich sein zu bestimmen, ob andere ziliarische GPCRs wie Neuropeptid-Y-Rezeptor Typ 2 (NPY2R) und Somatostatin-Rezeptor Typ 3 (SSTR3) eine ähnliche Diversität aufweisen.
Messung des Intensitätsprofils entlang des Ziliums
Sobald Zilien mit segment.ai identifiziert wurden, kann das GA3-Rezept modifiziert werden, um die Zilienanalyse mit der Identifizierung anderer Strukturen von Interesse im Bild zu kombinieren. Zum Beispiel ist die Markierung mit Basalmarkern nützlich, um die Polarität der Zilien zu identifizieren. Um diese Analyse durchzuführen, haben wir hypothalamische Hirnschnitte von P0-Mäusen abgebildet, die ARL13B-mCherry und Centrin2-GFP exprimieren, und die ARC und PVN34 abgebildet. Hier wurden Zilien wie zuvor mit Ai identifiziert, aber jetzt beinhaltet das modifizierte GA3-Rezept die Identifizierung von Centrin2-GFP, einem zentriolaren Protein, das an der Basis von Zilien gefunden wird (Ergänzende Abbildung 3B). Durch die Kennzeichnung Von Centrin2-GFP kann die Basis der Zilien von den Spitzen der ARL13B-mCherry positiven Zilien unterschieden werden (Abbildung 6A). Anstatt dann die Intensität innerhalb der gesamten Zilien zu messen, können wir Änderungen der ARL13B-Intensität entlang der Länge der Zilien messen (Abbildung 6B). Wir können auch Unterschiede in der Intensität von ARL13B zwischen den proximalen Enden und distalen Enden der Zilien vergleichen. Um dies zu tun, haben wir die Ciliumlänge in 1-Mikron-Behälter ausgehend von der Basis aufgeteilt und den ersten Mikron-Behälter als proximales Ende und den letzten Mikrometer-Behälter als distales Ende bezeichnet. Unsere Analyse ergab, dass sowohl in ARC als auch in PVN signifikant mehr ARL13B näher an der Basis als an der Spitze des Ciliums vorhanden ist, und dies stimmt mit zuvor veröffentlichten Studien an menschlichen Chondrozytenüberein 45 (Abbildung 6C). Anstatt bei dieser Art der Analyse einen Längenfilter anzuwenden, um kleine nicht-ziliarische Objekte von der Analyse auszuschließen, werden nur Zilien analysiert, die mit der Centrin2-GFP-Markierung verbunden sind. Dies kann in Situationen von Vorteil sein, in denen genetische Mutationen sehr kurze Zilien verursachen oder wenn Veränderungen in Zilien-Subdomänen wie der Übergangszone oder der Spitze beteiligt sind. Die Identifizierung von Zilien mittels Ai- und GA3-Analyse ist in hohem Maße anpassungsfähig und kann auf eine Vielzahl komplexer Forschungsfragen zugeschnitten werden.
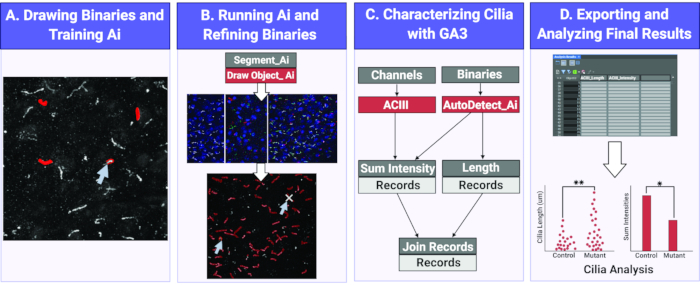
Abbildung 1. Workflow zur Messung von Zilienlänge und -intensität mit Ai. (A) Um die AI zu trainieren, werden Binärdateien um die Objekte von Interesse (Zilien) auf den rohen Trainingsbildern gezeichnet. Anhand der gezeichneten Binärdateien wird Segment Ai darauf trainiert, die Form und Pixelintensität der Zilien zu erkennen. (B) Als nächstes wird das trainierte Segment Ai auf experimentelle Rohbilder angewendet. Es zeichnet Binärdateien auf Objekte, die es als Zilien erkennt. Diese Binärdateien können verfeinert werden, um sicherzustellen, dass alle und nur Zilien analysiert werden. (C) Ein GA3-Programm ist konstruiert, um die Intensität und Länge von Objekten zu analysieren, die von der Ai erkannt werden. (D) Die Datensätze werden in eine Tabelle in der Software importiert. Diese Tabelle kann dann zur weiteren Analyse exportiert werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2. In-vitro-Zilienlängenmessungen. Repräsentative Abbildungen von Zilien in (A) IMCD-Zellen (grünes, acetyliertes Tubulin) (B) primäre hypothalamische Kulturen (grün, ACIII) und (C) Hippocampuskulturen (grün, ACIII). Ein trainiertes Ai wurde verwendet, um Zilien zu erkennen, wie in der binären Maske (Magenta) gezeigt, und dann wurde GA3 verwendet, um die Zilienlänge zu messen. Die Verteilung der Zilienlänge wird als Prozentsatz der Zilien in 0,5- oder 1,0-Mikron-Behältern dargestellt. * zeigt eine zytokinetische Brücke an, die von Ai nicht richtig erkannt wird. n=225 Zilien in IMCD-Zellen aus 3 Replikaten, 54 Zilien in Hypothalamus und 139 Zilien in Hippocampuskulturen aus 3 Tieren. Maßstabsbalken 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
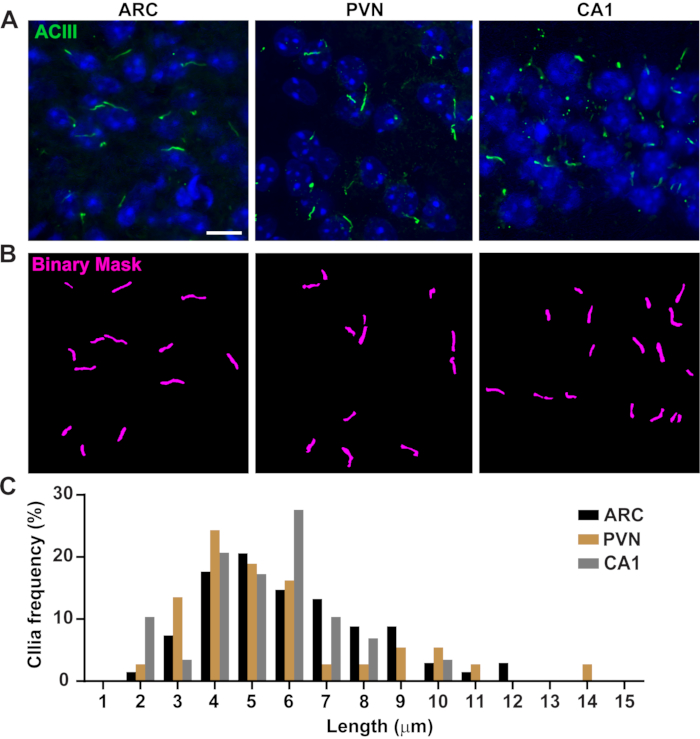
Abbildung 3. In vivo Zilienlängenmessungen. (A) Repräsentative Bilder von Zilien (grün, ACIII) in der ARC, PVN und CA1 von erwachsenen Maushirnschnitten. (B) Ein trainiertes Ai in NIS-Elementen wurde verwendet, um Zilien zu erkennen, wie in der binären Maske (Magenta) gezeigt, und dann wurde GA3 verwendet, um die Zilienlänge zu messen. (C) Die Verteilung der Zilienlänge wird als Prozentsatz der Zilien in Ein-Mikron-Behältern dargestellt. n= 68 Zilien in ARC, 36 in PVN und 29 in CA1 von 3 Tieren. Maßstabsbalken 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
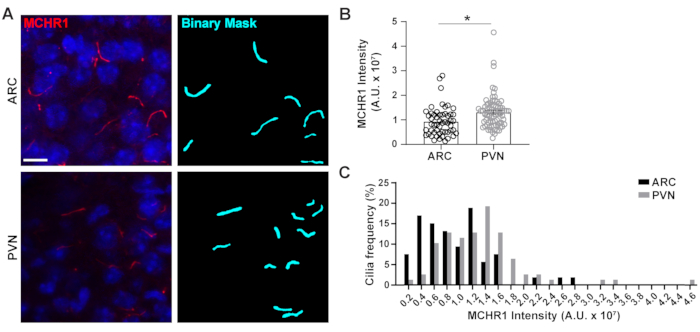
Abbildung 4. Ai-gestützte Zilienfärbung Intensitätsmessungen von hypothalamischen neuronalen Zilien. (A) Repräsentative Bilder von Zilien (MCHR1, rot) in der ARC und PVN von erwachsenen Maushirnschnitten. Ein trainiertes Ai in NIS-Elementen wurde verwendet, um Zilien zu erkennen, wie in der binären Maske (Cyan) gezeigt, und dann wurde GA3 verwendet, um die Intensität der MCHR1-Färbung in Zilien zu messen. (B) McHR1-Intensitäten werden als Durchschnitt ± S.E.M dargestellt. Jeder Punkt stellt ein Cilium dar. * p < 0,05, Student's t-test. (C) Die Verteilung der MCHR1-Intensität wird als Prozentsatz der Zilien in Behältern von 0,2 x 107 beliebigen Einheiten (A. U.) dargestellt. n= 53 Zilien in ARC, 78 in PVN von 3 Tieren. Maßstabsbalken 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
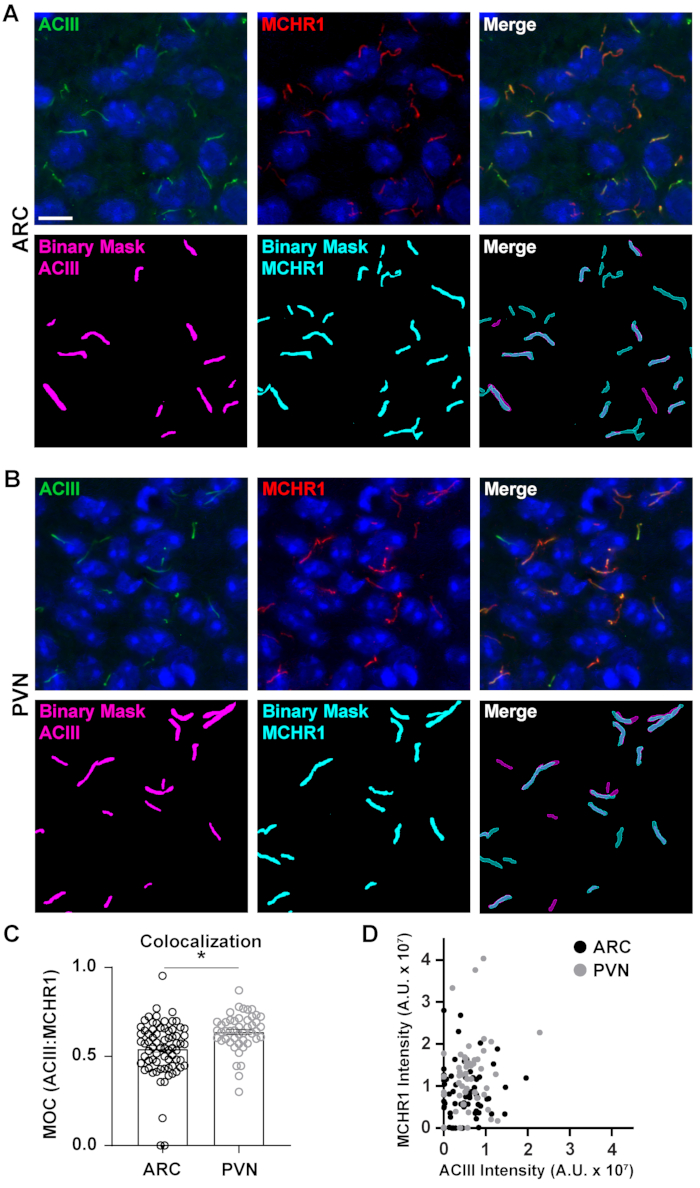
Abbildung 5. Ai-gestützte Zilien-Kolokalisationsanalyse. (A, B) Repräsentative Bilder von Zilien im ARC bzw. PVN. Zilien sind mit ACIII (grün) und MCHR1 (rot) gekennzeichnet. Ein trainiertes Ai in NIS-Elementen wurde verwendet, um Zilien zu erkennen, wie in der binären Maske gezeigt (Magenta für ACIII-markierte Zilien, Cyan für MCHR1 markierte Zilien). GA3 wurde verwendet, um Zilien zu erkennen, die sowohl ACIII als auch MCHR1 enthielten. (C) Die MoC-Werte (Manders Overlap Coefficient) werden als Durchschnitt ± S.E.M dargestellt. Jeder Punkt stellt ein Cilium dar. * p < 0,05, Student's t-test. (D) Streudiagramm der MCHR1-Intensität vs. ACIII-Intensität in ARC und PVN. Jeder Punkt stellt ein Cilium dar. n= 72 Zilien in ARC, 47 in PVN von 3 Tieren. Maßstabsbalken 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
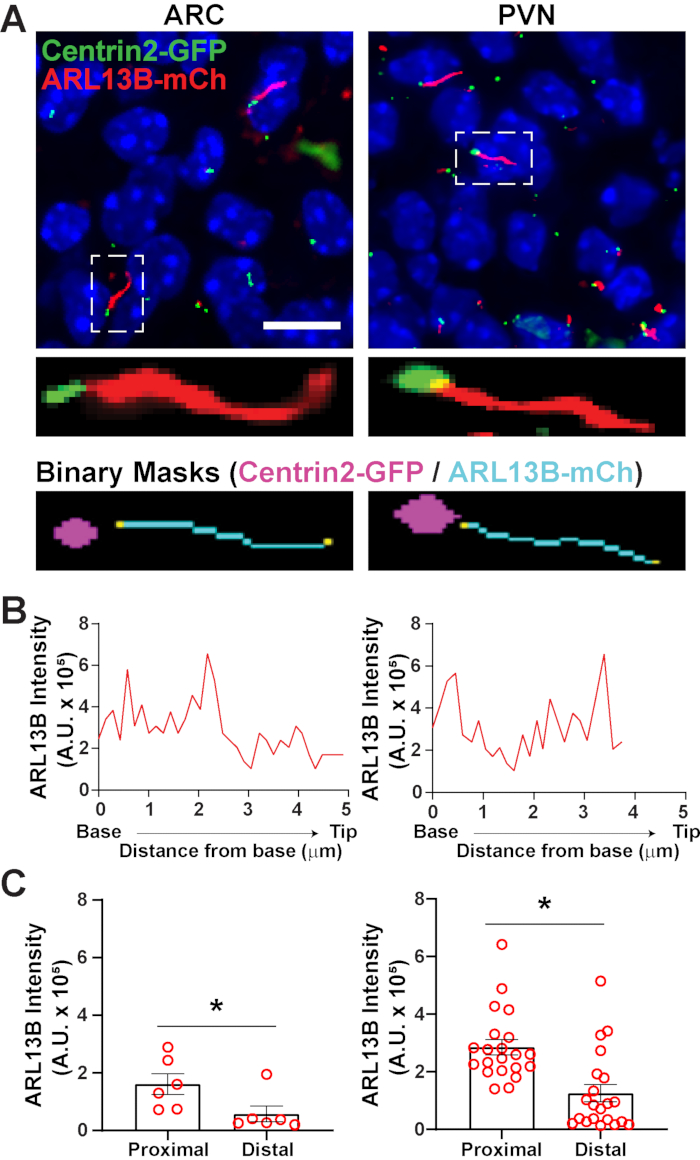
Abbildung 6. Zilien- und Basalanalyse. (A) Repräsentative Bilder von Zilien (rot, ARL13B-mCherry) und Basalmarker (grün, Centrin2-GFP) in der ARC und PVN von P0-Mäusen. Ein trainiertes Ai wurde verwendet, um Zilien zu erkennen, wie in der binären Maske (Cyan) gezeigt. Die binäre Maske für Basalkörper (Magenta) wurde durch Thresholding im GA3-Rezept gezeichnet. (B) Repräsentative Zeilenscanintensität eines Ciliums. (C) ARL13B-Intensitäten an den proximalen und distalen Enden von Ai identifizierten Zilien, die als durchschnittliche ± S.E.M dargestellt wurden. Die proximalen und distalen Enden sind definiert als die Region innerhalb der ersten 1 μm Länge bzw. der letzten 1 μm Länge von der Basis des Ciliums. Jeder Punkt stellt ein Cilium dar. * S. < 0,05. n = 6 Zilien in ARC von 2 Tieren und 21 Zilien in PVN von 3 Tieren. Maßstabsbalken 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1. Ai-Trainingsverlustdiagramme. (A, B) Graphiken, die den Trainingsverlust von segment.ai auf neuronale zilien in vitro bzw. in vivo zeigen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 2. Ai Assisted Cilia Staining Intensität Messungen von in vitro neuronalen Zilien. (A, B) Repräsentative Bilder von Zilien (MCHR1, rot) in primären Hypothalamus- bzw. Hippocampuskulturen. Ein trainiertes Ai in NIS-Elementen wurde verwendet, um Zilien zu erkennen, wie in der binären Maske (Cyan) gezeigt, und dann wurde GA3 verwendet, um die Intensität der MCHR1-Färbung in Zilien zu messen. Die Verteilung der MCHR1-Intensität wird als Prozentsatz der Zilien in 1000 A.E. Behältern für Hypothalamus- und 2000 A. U. Behältern für Hippocampuskulturen grafisch dargestellt. n= 30 Zilien in Hypothalamus und 106 Zilien in Hippocampuskulturen von 3 Tieren. Maßstabsbalken 10 μm. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 3. Allgemeine Analyse 3 Rezepte für die Zilienanalyse. (A) Einfache allgemeine Analyse (GA3) Rezept zur Messung der Zilienlänge, -intensität und des Mander-Koeffizienten. (B) Komplexe GA3-Rezeptur zur Messung der Intensität entlang der Länge des Ciliums unter Verwendung eines Markers für den Basalkörper. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Längen- und Intensitätsmessungen sind gängige Methoden zur Analyse primärer Zilien, es gibt jedoch keine standardisierte konventionelle Methode, die im Feld verwendet wird. Die Identifizierung und Quantifizierung primärer Zilien mit Software wie ImageJ ist zeitaufwendig und anfällig für Benutzerverzerrungen und -fehler. Dies macht es schwierig, große Datensätze genau zu analysieren. Hier zeigen wir, dass die Verwendung eines AI-Programms viele dieser Herausforderungen überwinden kann, so dass eine Hochdurchsatzanalyse der primären Zilien möglich ist. Hierin beschreiben wir das Verfahren zum Trainieren einer Ai-basierten Anwendung zur Erkennung primärer Zilien und skizzieren die Schritte, die zur Analyse von Länge und Intensität erforderlich sind.
Während das anfängliche Training der Ai zur Erkennung von Zilien viel Zeit vom Benutzer erfordert, kann es nach Abschluss für jeden Datensatz verwendet werden, der mit den gleichen Parametern erfasst wurde. Die von der Ai erzeugte Binärmaske ist so veränderbar, dass Fehler korrigiert werden können. Fehler bei der Identifizierung von Zilien sollten dem Benutzer jedoch signalisieren, dass die AI mit zusätzlichen Bildern weiter trainiert werden muss. Ein großer Vorteil dieser Methode ist, dass die Ai trainiert werden kann, Zilien in verschiedenen Probentypen sowohl in 2D als auch in 3D zu erkennen. Frühere Analysemethoden, die in Laboren generiertwurden,haben verschiedene Einschränkungen, einschließlich der Anforderung manueller Schwellenwerte zur Identifizierung und Problemen bei der Identifizierung von Zilien, die aus Gewebeschnitten mit hoher Zelldichte von36,46,47 abgebildetwurden. Diese Methoden sind auch auf die Zilienanalyse spezialisiert, während die Analyse mit der NIS Elements-Software mehrere Aspekte der Bilder gleichzeitig auswerten kann. Da das hier beschriebene Ai Teil des NIS Elements-Softwarepakets ist, können Bilder, die mit einem Nikon-Mikroskop aufgenommen wurden, einfach bis zur Analyse fortgesetzt werden. Für die Verwendung dieser Methode ist jedoch keine Bildgebung mit Nikon erforderlich. Unabhängig vom erfassten Rohdatendateiformat können ".tif"-Dateien von NIS Elements geöffnet werden, um sie in der Ai zu verwenden.
Diese AI-Anwendung innerhalb von NIS Elements ist weit verbreitet und möglicherweise bereits Teil der Bildanalysesoftware, die von Labors verwendet wird, die primäre Zilien untersuchen. Mit der zunehmenden Verbreitung der AI-Technologie können andere Bildgebungssoftware ihre Analyseoptionen um ein ähnliches AI-Modul erweitern. Die Anwendung der Ai-Analyse zur Identifizierung von Zilien kann für verschiedene Aspekte der Zilienanalyse verwendet werden. Während wir Methoden für einige einfache Analysen wie Länge (Abbildung 2 und 3), Intensität (Abbildung 4) und Kolokalisierung (Abbildung 5) skizziert haben, kann dem GA3-Analyse-Workflow wie in Abbildung 6eine ausgefeiltere Analyse hinzugefügt werden. Anstatt beispielsweise die Intensität eines vollständigen Ciliums zu messen, können Intensitätsunterschiede innerhalb einer Subregion eines Ciliums von Interesse sein, um die subziliare Lokalisation zu beurteilen. Intensitätsunterschiede innerhalb einer Subregion eines Ciliums könnten darauf hindeuten, dass sich das Protein an der Spitze oder an der Basis des Ciliums ansammelt, wie z.B. wie Gli-Proteine an der Spitze der Zilien angereichert sind48. Darüber hinaus kann diese Ai-Anwendung verwendet werden, um Unterschiede zwischen Genotypen oder Behandlungsgruppen leicht zu identifizieren. Während unser Labor diese Methode hauptsächlich zur Analyse von Zilien verwendet, die aus Hirnschnitten oder neuronalen Kulturen stammen, kann sie auf Bilder aus verschiedenen Zelllinien oder anderen Gewebetypen angewendet werden. Die Flexibilität des Probentyps, für den diese Anwendung verwendet werden kann, macht diese Analysemethode für viele verschiedene Gruppen wertvoll, die primäre Zilien oder diskrete Organellen untersuchen, die bewertet werden, wie Mitochondrien, Kern oder ER.
Offenlegungen
Co-Autor Wesley Lewis ist ein Mitarbeiter von Nikon. Es gibt keine finanziellen Offenlegungen.
Danksagungen
Diese Arbeit wurde vom National Institute of Diabetes and Digestive and Kidney Diseases R01 DK114008 an NFB und dem American Heart Association Fellowship Grant #18PRE34020122 an RB finanziert. Wir danken Rich Gruskin, General Manager von Nikon Software, Melissa Bentley, Courtney Haycraft und Teresa Mastracci für aufschlussreiche Kommentare zum Manuskript.
Materialien
| Name | Company | Catalog Number | Comments |
| Intel Xeon, 3.6 GHz, 32GB RAM | Intel Corporation | W-2123 | Processor used for running NIS Elements. |
| Nikon Elements Software | Nikon Instruments Inc. | - | Ai and GA3 software |
| Quadro RTX 4000 Graphics card | NVIDIA Corporation | Quadro RTX 4000 | |
| Windows 10 Professional 64-bit | Microsoft Inc. | - | Operating system used for running NIS Elements |
| Workstation | HP Development Company, L.P. | HP Z4G4 | Workstation used for running NIS Elements |
Referenzen
- Wang, L., et al. Ciliary gene RPGRIP1L is required for hypothalamic arcuate neuron development. JCI Insight. 4 (3), (2019).
- Siljee, J. E., et al. Subcellular localization of MC4R with ADCY3 at neuronal primary cilia underlies a common pathway for genetic predisposition to obesity. Nature Genetics. 50 (2), 180-185 (2018).
- Davenport, J. R., et al. Disruption of intraflagellar transport in adult mice leads to obesity and slow-onset cystic kidney disease. Current Biology. 17 (18), 1586-1594 (2007).
- Berbari, N. F., O'Connor, A. K., Haycraft, C. J., Yoder, B. K. The primary cilium as a complex signaling center. Current Biology. 19 (13), 526-535 (2009).
- Walz, G. Role of primary cilia in non-dividing and post-mitotic cells. Cell Tissue Research. 369 (1), 11-25 (2017).
- Nachury, M. V., Mick, D. U. Establishing and regulating the composition of cilia for signal transduction. Nature Reviews. Molecular Cell Biology. 20 (7), 389-405 (2019).
- Goetz, S. C., Anderson, K. V. The primary cilium: a signalling centre during vertebrate development. Nature Reviews Genetics. 11 (5), 331-344 (2010).
- Engle, S. E., Bansal, R., Antonellis, P. J., Berbari, N. F. Cilia signaling and obesity. Seminars in Cell and Developmental Biology. , (2020).
- Reiter, J. F., Leroux, M. R. Genes and molecular pathways underpinning ciliopathies. Nature reviews. Molecular Cell Biology. 18 (9), 533-547 (2017).
- Waters, A. M., Beales, P. L. Ciliopathies: an expanding disease spectrum. Pediatric Nephrology. 26 (7), Berlin, Germany. 1039-1056 (2011).
- Hildebrandt, F., Benzing, T., Katsanis, N. Ciliopathies. New England Journal of Medicine. 364 (16), 1533-1543 (2011).
- Vaisse, C., Reiter, J. F., Berbari, N. F. Cilia and Obesity. Cold Spring Harbor Perspectives in Biology. 9 (7), (2017).
- Berbari, N. F., et al. Leptin resistance is a secondary consequence of the obesity in ciliopathy mutant mice. Proceedings of the National Academy of Sciences of the United States of America. 110 (19), 7796-7801 (2013).
- Jacobs, D. T., et al. Dysfunction of intraflagellar transport-A causes hyperphagia-induced obesity and metabolic syndrome. Disease Models & Mechanisms. 9 (7), 789-798 (2016).
- Arsov, T., et al. Fat aussie--a new Alström syndrome mouse showing a critical role for ALMS1 in obesity, diabetes, and spermatogenesis. Molecular Endocrinology. 20 (7), 1610-1622 (2006).
- Tam, L. W., Ranum, P. T., Lefebvre, P. A. CDKL5 regulates flagellar length and localizes to the base of the flagella in Chlamydomonas. Molecular Biology of the Cell. 24 (5), 588-600 (2013).
- Rajagopalan, V., Subramanian, A., Wilkes, D. E., Pennock, D. G., Asai, D. J. Dynein-2 affects the regulation of ciliary length but is not required for ciliogenesis in Tetrahymena thermophila. Molecular Biology of the Cell. 20 (2), 708-720 (2009).
- Bengs, F., Scholz, A., Kuhn, D., Wiese, M. LmxMPK9, a mitogen-activated protein kinase homologue affects flagellar length in Leishmania mexicana. Molecular Microbiology. 55 (5), 1606-1615 (2005).
- Han, Y. M., et al. Leptin-promoted cilia assembly is critical for normal energy balance. Journal of Clinical Investigation. 124 (5), 2193-2197 (2014).
- Caspary, T., Marazziti, D., Berbari, N. F. Cilia: Methods and Protocols. Satir, P., Tvorup Christensen, S. , Springer. New York. 203-214 (2016).
- Bishop, G. A., Berbari, N. F., Lewis, J., Mykytyn, K. Type III adenylyl cyclase localizes to primary cilia throughout the adult mouse brain. Journal of Comparative Neurology. 505 (5), 562-571 (2007).
- Domire, J. S., Mykytyn, K. Markers for neuronal cilia. Methods in Cell Biology. 91, 111-121 (2009).
- Sun, J. S., et al. Ventromedial hypothalamic primary cilia control energy and skeletal homeostasis. Journal of Clinical Investigation. 131 (1), (2021).
- Berbari, N. F., Johnson, A. D., Lewis, J. S., Askwith, C. C., Mykytyn, K. Identification of ciliary localization sequences within the third intracellular loop of G protein-coupled receptors. Molecular Biology of the Cell. 19 (4), 1540-1547 (2008).
- Brailov, I., et al. Localization of 5-HT(6) receptors at the plasma membrane of neuronal cilia in the rat brain. Brain Research. 872 (1-2), 271-275 (2000).
- Domire, J. S., et al. Dopamine receptor 1 localizes to neuronal cilia in a dynamic process that requires the Bardet-Biedl syndrome proteins. Cellular and Molecular Life Sciences. 68 (17), 2951-2960 (2011).
- Handel, M., et al. Selective targeting of somatostatin receptor 3 to neuronal cilia. Neuroscience. 89 (3), 909-926 (1999).
- Koemeter-Cox, A. I., et al. Primary cilia enhance kisspeptin receptor signaling on gonadotropin-releasing hormone neurons. Proceedings of the National Academy of Sciences of the United States of America. 111 (28), 10335-10340 (2014).
- Mukhopadhyay, S., et al. The ciliary G-protein-coupled receptor Gpr161 negatively regulates the Sonic hedgehog pathway via cAMP signaling. Cell. 152 (1-2), 210-223 (2013).
- Berman, S. A., Wilson, N. F., Haas, N. A., Lefebvre, P. A. A novel MAP kinase regulates flagellar length in Chlamydomonas. Current Biology. 13 (13), 1145-1149 (2003).
- Nguyen, R. L., Tam, L. W., Lefebvre, P. A. The LF1 gene of Chlamydomonas reinhardtii encodes a novel protein required for flagellar length control. Genetics. 169 (3), 1415-1424 (2005).
- Tam, L. W., Wilson, N. F., Lefebvre, P. A. A CDK-related kinase regulates the length and assembly of flagella in Chlamydomonas. Journal of Cell Biology. 176 (6), 819-829 (2007).
- O'Connor, A. K., et al. An inducible CiliaGFP mouse model for in vivo visualization and analysis of cilia in live tissue. Cilia. 2 (1), 8(2013).
- Bangs, F. K., Schrode, N., Hadjantonakis, A. K., Anderson, K. V. Lineage specificity of primary cilia in the mouse embryo. Nature Cell Biology. 17 (2), 113-122 (2015).
- Delling, M., et al. Primary cilia are not calcium-responsive mechanosensors. Nature. 531 (7596), 656-660 (2016).
- Saggese, T., Young, A. A., Huang, C., Braeckmans, K., McGlashan, S. R. Development of a method for the measurement of primary cilia length in 3D. Cilia. 1 (1), 11(2012).
- Kobayashi, Y., Hamamoto, A., Saito, Y. Analysis of ciliary status via G-protein-coupled receptors localized on primary cilia. Microscopy. 69 (5), 277-285 (2020).
- Zhou, L. Q., et al. Artificial intelligence in medical imaging of the liver. World Journal of Gastroenterology. 25 (6), 672-682 (2019).
- Naugler, C., Church, D. L. Automation and artificial intelligence in the clinical laboratory. Critical Reviews in Clinical Laboratory Sciences. 56 (2), 98-110 (2019).
- Dunn, K. W., Kamocka, M. M., McDonald, J. H. A practical guide to evaluating colocalization in biological microscopy. American Journal of Physiology-Cell Physiology. 300 (4), 723-742 (2011).
- Bansal, R., et al. Hedgehog Pathway Activation Alters Ciliary Signaling in Primary Hypothalamic Cultures. Frontiers in Cellular Neuroscience. 13, 266(2019).
- Jin, H., et al. The conserved Bardet-Biedl syndrome proteins assemble a coat that traffics membrane proteins to cilia. Cell. 141 (7), 1208-1219 (2010).
- Liew, G. M., et al. The intraflagellar transport protein IFT27 promotes BBSome exit from cilia through the GTPase ARL6/BBS3. Developmental Cell. 31 (3), 265-278 (2014).
- Engle, S. E., et al. A CreER Mouse to Study Melanin Concentrating Hormone Signaling in the Developing Brain. Genesis. , (2018).
- Thorpe, S. D., et al. Reduced primary cilia length and altered Arl13b expression are associated with deregulated chondrocyte Hedgehog signaling in alkaptonuria. Journal of Cellular Physiology. 232 (9), 2407-2417 (2017).
- Lauring, M. C., et al. New software for automated cilia detection in cells (ACDC). Cilia. 8, 1(2019).
- Dummer, A., Poelma, C., DeRuiter, M. C., Goumans, M. J., Hierck, B. P. Measuring the primary cilium length: improved method for unbiased high-throughput analysis. Cilia. 5, 7(2016).
- Haycraft, C. J., et al. Gli2 and Gli3 localize to cilia and require the intraflagellar transport protein polaris for processing and function. PLoS Genetics. 1 (4), 53(2005).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten