Method Article
Approches de l’intelligence artificielle pour évaluer les cils primaires
Dans cet article
Résumé
L’utilisation de l’intelligence artificielle (IA) pour analyser les images émerge comme une approche puissante, moins biaisée et rapide par rapport aux méthodes couramment utilisées. Ici, nous avons formé Ai à reconnaître un organite cellulaire, des cils primaires, et à analyser des propriétés telles que la longueur et l’intensité de la coloration de manière rigoureuse et reproductible.
Résumé
Les cils sont des appendices cellulaires à base de microtubules qui fonctionnent comme des centres de signalisation pour une diversité de voies de signalisation dans de nombreux types de cellules de mammifères. La longueur des cils est hautement conservée, étroitement régulée et varie entre différents types de cellules et de tissus et a été impliquée dans l’impact direct sur leur capacité de signalisation. Par exemple, il a été démontré que les cils modifient leur longueur en réponse à l’activation des récepteurs couplés à la protéine G ciliaire. Cependant, mesurer avec précision et de manière reproductible les longueurs de nombreux cils est une procédure longue et laborieuse. Les approches actuelles sont également sujettes aux erreurs et aux biais. Les programmes d’intelligence artificielle (IA) peuvent être utilisés pour surmonter bon nombre de ces défis en raison de capacités qui permettent l’assimilation, la manipulation et l’optimisation de vastes ensembles de données. Ici, nous démontrons qu’un module d’IA peut être formé pour reconnaître les cils dans des images provenant d’échantillons in vivo et in vitro. Après avoir utilisé l’IA entraînée pour identifier les cils, nous sommes en mesure de concevoir et d’utiliser rapidement des applications qui analysent des centaines de cils dans un seul échantillon pour la longueur, l’intensité de fluorescence et la colocalisation. Cette approche impartiale a augmenté notre confiance et notre rigueur lors de la comparaison d’échantillons de différentes préparations neuronales primaires in vitro ainsi que dans différentes régions du cerveau au sein d’un animal et entre les animaux. De plus, cette technique peut être utilisée pour analyser de manière fiable la dynamique des cils de n’importe quel type de cellule et de tissu de manière élevée sur plusieurs échantillons et groupes de traitement. En fin de compte, les approches basées sur l’IA deviendront probablement la norme à mesure que la plupart des domaines évolueront vers des approches moins biaisées et plus reproductibles pour l’acquisition et l’analyse d’images.
Introduction
Les cils primaires sont des organites sensoriels dépassant de la plupart des types de cellules demammifères1,2,3,4. Ce sont généralement des appendices solitaires essentiels à la coordination de diverses voies de signalisation cellulaire en intégrant des signaux extracellulaires5,6,7. Les cils primaires jouent un rôle important au cours du développement embryonnaire et de l’homéostasie des tissus adultes, et la perturbation de leur fonction ou de leur morphologie est associée à plusieurs troubles génétiques, collectivement appelés ciliopathies. En raison de la nature presque omniprésente des cils, les ciliopathies sont associées à un large éventail de caractéristiques cliniques qui peuvent avoir un impact sur tous les systèmes d’organes8,9,10,11,12. Dans les modèles animaux de ciliopathies, la perte de la structure ciliaire ou de la capacité de signalisation se manifeste dans plusieurs phénotypes cliniquement pertinents, y compris l’obésité associée à l’hyperphagie3,13,14,15. Dans de nombreux systèmes modèles, il a été démontré que les changements de longueur des cils ont un impact sur leur capacité de signalisation et leurs fonctions16,17 , 18,19. Cependant, il existe plusieurs défis techniques et chronophages associés à l’évaluation précise et reproductible de la longueur et de la composition des cils.
Le système nerveux central (SNC) des mammifères adultes est un contexte biologique qui a posé un défi pour la compréhension de la morphologie et de la fonction des cils. Bien qu’il semble que les neurones et les cellules du SNC possèdent des cils, en raison des outils et des capacités limités pour observer et analyser ces cils, la compréhension de leurs fonctions reste insaisissable20. Par exemple, le marqueur prototypique des cils, la α-tubuline acétylée, ne marque pas les cils neuronaux20. La difficulté d’étudier ces cils a été en partie résolue avec la découverte de plusieurs récepteurs couplés aux protéines G (RCPG), de mécanismes de signalisation et de protéines associées à la membrane qui s’enrichissent sur la membrane des cils neuronaux21,22. Toutes ces observations de base simples font allusion à l’importance et à la diversité des cils du SNC, qui semblent jusqu’à présent inégalés par d’autres tissus. Par exemple, une variation de la longueur des cils et de la localisation des RCPG peut être observée dans tout le cerveau, les longueurs de certains noyaux neuronaux étant différentes de celles des autres noyaux19,23. De même, leur contenu GPCR et leur machinerie de signalisation complètent la diversité basée sur la localisation neuroanatomique et le type neuronal2,24,25,26,27,28,29. Ces observations simples démontrent que la longueur et la composition des cils du SNC des mammifères sont étroitement régulées, tout comme chez les organismes modèles, comme Chlamydomonas reinhardtii,mais l’impact de ces différences de longueur sur la fonction des cils, la signalisation et finalement le comportement reste incertain16,30,31,32.
Mesurer avec précision la longueur et la composition des cils s’avère être un défi technique sujet à l’erreur de l’utilisateur et à l’irreproductibilité. Actuellement, les cils in vivo et in vitro sont le plus souvent identifiés à l’aide d’approches immunofluorescentes qui marquent les protéines ciliaires ou les allèles rapporteurs fluorescents enrichis en cils33,34,35. Les longueurs de ces cils marqués par fluorescence sont ensuite mesurées à partir d’une image en 2 dimensions (2D) à l’aide d’outils de mesure de ligne dans des programmes d’analyse d’images tels que ImageJ36. Ce processus est non seulement fastidieux et laborieux, mais aussi sujet aux biais et aux erreurs. Ces mêmes obstacles surviennent lors de la mesure de l’intensité des cils, ce qui aide à indiquer des changements dans la structure descils 37. Pour minimiser les incohérences dans ces types d’analyses d’images, les programmes d’intelligence artificielle (IA) deviennent de plus en plus répandus et abordables38.
L’IA est l’avancement des systèmes informatiques qui utilisent l’avantage des algorithmes informatiques et de la programmation pour exécuter des tâches qui nécessiteraient généralement une intelligence humaine39. Les dispositifs d’IA apprennent à percevoir des modèles, des paramètres et des caractéristiques récurrents et à prendre des mesures pour maximiser les chances de créer des résultats positifs. L’IA est polyvalente et peut être entraînée à reconnaître des objets ou des structures d’intérêt spécifiques, tels que les cils, puis être programmée pour exécuter une variété d’analyses sur les objets identifiés. Par conséquent, des données d’image complexes peuvent être générées rapidement et de manière reproductible par Ai38. L’automatisation et l’analyse ia des images capturées augmenteront l’efficacité et l’efficience tout en limitant toute erreur et biais humain potentiel39. L’établissement d’une méthodologie basée sur l’IA pour l’identification des cils crée un moyen cohérent pour tous les groupes de recherche d’analyser et d’interpréter les données sur les cils.
Ici, nous utilisons un module Ai pour identifier les cils in vivo et in vitro sur des images 2D. À l’aide d’un ensemble d’exemples d’images, l’IA est entraînée à identifier les cils. Une fois la formation terminée, l’IA désignée est utilisée pour appliquer un masque binaire sur les cils identifiés par l’IA dans une image. Les binaires appliqués par l’Ai sont modifiables, si nécessaire, pour s’assurer que tous les cils dans les images sont correctement identifiés et que l’identification non spécifique est éliminée. Après avoir utilisé l’IA pour identifier les cils, des programmes d’analyse générale (GA) sur mesure sont utilisés pour effectuer différentes analyses telles que la mesure de la longueur des cils et de l’intensité de fluorescence. Les données recueillies sont exportées dans un tableau qui peut être facilement lu, interprété et utilisé pour des analyses statistiques (Figure 1). L’utilisation de la technologie automatisée et de l’IA pour identifier les cils et obtenir des mesures spécifiques entre les groupes expérimentaux aidera dans les futures études visant à comprendre l’impact de la fonction et de la morphologie des cils du SNC sur la communication et le comportement cellule-cellule.
Protocole
1. Acquérir des images brutes
- Fixer et étiqueter les échantillons au besoin20.
- Imagez les cils à l’aide d’un microscope confocal à la profondeur de bits maximale en utilisant la même taille de pixel avec une résolution Nyquist.
- Exportez des images en tant que fichiers monochromes au format .tif (Tagged Image Format).
REMARQUE: Ce protocole explique comment utiliser le module Ai spécifiquement dans le logiciel NIS Elements. Si les images ont été acquises en tant que fichiers .nd2, l’exportation d’images en tant que fichiers .tif n’est pas nécessaire et l’utilisateur peut passer directement à l’étape 2.3. Si les images ont été acquises sur un autre système, une licence NIS Elements peut être achetée séparément et .tif fichiers peuvent être convertis comme indiqué dans les étapes suivantes.
2. Entraînez l’IA à identifier les cils
- Ouvrez le jeu de données de formation.
- Sélectionnez environ 50 exemples d’images avec au moins un ccil par image pour entraîner le logiciel et copiez-les dans un seul dossier. Ce dossier est utilisé pour diriger le logiciel lors de l’ouverture des images. Ouvrez ces 50 images dans un seul document ND2 avec au moins un ciliium par image. Sélectionnez Fichier > Importer/Exporter > Créer un fichier ND à partir de la séquence de fichiers.
- Sélectionnez le dossier contenant le jeu de données de formation. Cela ouvrira la liste des fichiers au centre de la fenêtre de dialogue. Définissez manuellement l’organisation des fichiers à l’aide d’au moins une option dans le menu déroulant ci-dessus. Les options sont multipoint (pour plusieurs fichiers de projection maximum), série Z (pour une image de pile z), temps (pour une image time-lapse) et longueur d’onde (pour les fichiers de plusieurs canaux).
- Entrez les valeurs numériques correspondantes sous chaque option sélectionnée. Sélectionnez Aucun lorsque les options ne sont pas sélectionnées. Cliquez sur Convertir pour ouvrir le document ND.
- Calibrer les images.
- Entrez la taille en pixels dans le coin inférieur gauche de l’image : cliquez avec le bouton droit sur Non étalonné > Calibrer le document > Taille des pixels.
- Identifiez les cils.
- Identifiez manuellement les cils en traçant avec précision des structures ciliaires individuelles sur tous les cadres ouverts à l’aide de la détection automatique ou de l’objet Draw dans la barre d’outils binaire. Cela dessinera des masques binaires sur les objets d’intérêt. Ces binaires serviront d’exemples d’objets pour former le logiciel à identifier les cils sur les caractéristiques basées sur les pixels dans l’analyse expérimentale future d’images. Sélectionnez Afficher > contrôles d’analyse > barre d’outils binaire > Draw Object.
REMARQUE : Supprimez toute image qui n’a pas de fichiers binaires car le logiciel ne commencera pas l’entraînement à moins qu’il ne soit en mesure de détecter les fichiers binaires sur toutes les images ouvertes.
- Identifiez manuellement les cils en traçant avec précision des structures ciliaires individuelles sur tous les cadres ouverts à l’aide de la détection automatique ou de l’objet Draw dans la barre d’outils binaire. Cela dessinera des masques binaires sur les objets d’intérêt. Ces binaires serviront d’exemples d’objets pour former le logiciel à identifier les cils sur les caractéristiques basées sur les pixels dans l’analyse expérimentale future d’images. Sélectionnez Afficher > contrôles d’analyse > barre d’outils binaire > Draw Object.
- Train Ai.
- Commencez à former le logiciel. Cela ouvrira la boîte de Segment.ai du train. Sélectionnez NIS.ai > train Segment.ai.
- Dans la zone Train Segment.ai, sélectionnez le canal source à utiliser pour la formation. Si des fichiers de plusieurs canaux sont ouverts, sélectionnez un seul canal comme canal source. Sélectionnez ensuite les binaires de vérité de terrain appropriés sur lesquels entraîner l’Ai. Enfin, sélectionnez le nombre d’itérations nécessaires pour entraîner l’IA en fonction de la taille et de la distribution des binaires.
REMARQUE: Si les binaires sont facilement détectables depuis l’environnement et bien répartis dans toute l’image, le logiciel peut avoir besoin de moins de 1000 itérations pour être formé à identifier les images. Si les images ont un faible rapport signal/bruit, il est idéal d’exécuter au moins 1000 itérations pendant l’entraînement pour permettre à l’IA d’identifier les cils dans les échantillons de test avec une grande confiance. - Sélectionnez le dossier de destination pour enregistrer le fichier Ai entraîné (.sai) et cliquez sur Train pour entraîner le logiciel. Le logiciel va maintenant s’entraîner à identifier les cils en fonction des binaires tracés. Ce processus prend plusieurs heures.
REMARQUE: Lors de la formation, le logiciel affichera un graphique montrant la perte d’entraînement. Le graphique montrera d’abord un pic avant de diminuer pour atteindre idéalement environ 1% de perte où il plafonne pour le reste de l’entraînement. Enregistrez le graphique pour référence ultérieure en cochant la case Enregistrer la capture d’écran du graphique dans la zone train Segment.ai (Figure supplémentaire 1). - Si d’autres améliorations de la formation sont nécessaires, poursuivez la formation sur le même ensemble de données. Vous pouvez également vous entraîner sur un nouvel ensemble de données avec exactement les mêmes paramètres. Il n’est pas conseillé d’entraîner une IA déjà entraînée sur un nouvel ensemble de données avec différents paramètres ou différents objets d’intérêt. Sélectionnez S’entraîner segment.ai > continuer la formation sur > Sélectionnez Le fichier Ai formé.
3. Identifiez les cils à l’aide d’ia entraînée
- Ouvrez le jeu de données expérimental.
- Ouvrez les images confocales expérimentales de cils dans le logiciel en convertissant l’exemple .tif fichiers en fichiers .nd2, similaires à l’étape 2.1. Sélectionnez Fichier > Importer/Exporter > Créer un fichier ND à partir de la séquence de fichiers.
REMARQUE: Les images doivent être de la même taille de pixel que celles utilisées pour l’entraînement de l’IA. Si les images sont déjà au format ND2, passez à l’étape 3.3.
- Ouvrez les images confocales expérimentales de cils dans le logiciel en convertissant l’exemple .tif fichiers en fichiers .nd2, similaires à l’étape 2.1. Sélectionnez Fichier > Importer/Exporter > Créer un fichier ND à partir de la séquence de fichiers.
- Calibrer les images.
- Entrez la taille des pixels dans le coin inférieur gauche de l’image. Cliquez avec le bouton droit de la souris sur Uncalibrated > Calibrer le document > taille des pixels.
- Exécutez l’Ai entraîné sur les fichiers ouverts.
- Commencez à identifier les cils à l’aide de l’IA. Le logiciel va maintenant dessiner des binaires sur les cils en fonction de la formation qu’il a reçue à l’étape précédente. Ce processus prendra quelques secondes. Sélectionnez NIS.ai > Segment.ai.
REMARQUE: Le logiciel vous invite à sélectionner le canal si plusieurs canaux sont ouverts. Les chaînes sont répertoriées par leurs noms respectifs ici. Sinon, la case marquée 'Mono' sera automatiquement sélectionnée.
- Commencez à identifier les cils à l’aide de l’IA. Le logiciel va maintenant dessiner des binaires sur les cils en fonction de la formation qu’il a reçue à l’étape précédente. Ce processus prendra quelques secondes. Sélectionnez NIS.ai > Segment.ai.
- Vérifiez les images pour les fichiers binaires mal identifiés.
- Une fois que l’IA a identifié les cils et dessiné des binaires, vérifiez les images pour tout objet identifié par erreur. Si vous le souhaitez, supprimez manuellement tous les fichiers binaires mal identifiés. Sélectionnez Afficher > contrôles d’analyse > barre d’outils binaire > Supprimer l’objet.
4. Mesure de la longueur et de l’intensité des cils
- Créez une nouvelle recette d’analyse générale 3 (GA3).
- Maintenant que les cils ont été identifiés et segmentés, procédez à l’analyse de différents paramètres des cils tels que les longueurs et les intensités à l’aide de l’outil GA3. Cela ouvrira une nouvelle fenêtre avec un espace vide au centre où l’analyse sera définie. Sélectionnez Image > nouvelle recette GA3.
- Sélectionnez les fichiers binaires à analyser.
- Étant donné que les cils sont déjà segmentés à l’aide de Segment.ai, GA3 détectera automatiquement les binaires correctement étiquetés en fonction de l’Ai et inclura le nœud. Sélectionnez «Binaires > Detect_AI automatique » ou « Binaires > dessiner Object_AI ».
- Sélectionnez les canaux requis pour l’analyse. GA3 détectera également automatiquement les canaux dans les images et affichera leurs onglets sous Canaux.
- Supprimez les objets qui touchent la bordure du cadre.
- Étant donné que l’Ai segmentera tous les cils comme des objets dans le cadre, il détectera également les cils incomplets le long des bords du cadre. Ces objets peuvent être supprimés manuellement à l’étape 3.4 ou automatiquement dans GA3. Sélectionnez Traitement binaire > Supprimer les objets > Toucher les bordures.
- Sélectionnez les paramètres pour mesurer les cils.
- Faites glisser et déposez les paramètres à mesurer tels que la longueur des cils (Longueur) et les intensités (Somme de l’intensité de l’objet). Connectez les nœuds au nœud binaire approprié (connexion A) et aux nœuds de canal appropriés (connexion B). Passez le curseur de la souris sur la connexion du nœud pour obtenir une info-bulle afin d’indiquer à quelle connexion appartient le nœud. Sélectionnez Mesure > Taille de l’objet > Longueur et Mesure > Intensité de l’objet > Somme Intensité Obj.
REMARQUE: La longueur du nœud se connecte uniquement au nœud binaire, tandis que Sum Obj Intensity se connecte à la fois aux nœuds binaires et aux nœuds de canal.
- Faites glisser et déposez les paramètres à mesurer tels que la longueur des cils (Longueur) et les intensités (Somme de l’intensité de l’objet). Connectez les nœuds au nœud binaire approprié (connexion A) et aux nœuds de canal appropriés (connexion B). Passez le curseur de la souris sur la connexion du nœud pour obtenir une info-bulle afin d’indiquer à quelle connexion appartient le nœud. Sélectionnez Mesure > Taille de l’objet > Longueur et Mesure > Intensité de l’objet > Somme Intensité Obj.
- Ajoutez les mesures dans un seul tableau.
- Combinez toutes les mesures dans une seule table de sortie en faisant glisser et en déposant le nœud Ajouter une colonne à l’organigramme d’analyse et connectez-le aux nœuds de mesure, Longueur et Somme Obj Intensité. Sélectionnez Gestion des données > colonne Basic > Append.
- Mesurez les cils.
- Mesurez les cils en cliquant sur Exécuter. Ce processus prend quelques instants pour mesurer tous les cils dans les images expérimentales. Les longueurs et les intensités apparaîtront dans une nouvelle fenêtre Résultats d’analyse.
REMARQUE: Le tableau peut parfois inclure des données provenant de masques que Ai a reconnus comme des cils, mais qui étaient trop petits pour être détectés par l’œil humain et éliminés à l’étape 3.4. Ces objets peuvent être supprimés de l’ensemble de données à l’aide d’un filtre avant l’analyse statistique. Ici, un filtre de 1 μm a été utilisé pour les mesures in vitro de la longueur des cils dans la figure 2 et de 2 μm pour les cils in vivo. Cela peut être fait avant d’exporter des données en utilisant le chemin ci-dessous. Sélectionnez la fenêtre Résultats de l’analyse > Définir le filtre > Entrer la valeur > Utiliser le filtre.
- Mesurez les cils en cliquant sur Exécuter. Ce processus prend quelques instants pour mesurer tous les cils dans les images expérimentales. Les longueurs et les intensités apparaîtront dans une nouvelle fenêtre Résultats d’analyse.
- Exporter des données à des fins d’analyse statistique.
5. Études de colocalisation
REMARQUE: L’analyse de colocalisation peut être incluse dans la même recette GA3 utilisée pour les mesures de l’analyse de la longueur et de l’intensité des cils. Si vous utilisez la même recette, ouvrez les fichiers comme décrit ci-dessous et mesurez les longueurs et les intensités des deux canaux ainsi que les coefficients de colocalisation dans le même pipeline d’analyse.
- Ouvrez le jeu de données expérimental.
- Ouvrez les images confocales expérimentales de cils dans le logiciel en convertissant les exemples de fichiers .tif en fichiers .nd2. Sélectionnez Fichier > Importer/Exporter > Créer un fichier ND à partir de la séquence de fichiers.
- Dans la fenêtre contextuelle, sélectionnez les fichiers monochromes de profondeur 16 bits parmi tous les canaux d’intérêt dans l’explorateur de fenêtres situé dans la première colonne de la fenêtre contextuelle. Sélectionnez Multipoint ou Série Z dans le premier menu déroulant et entrez une valeur correspondant au nombre total d’images ou de piles, respectivement.
- Dans la deuxième liste déroulante, sélectionnez Longueur d’onde et remplacez la valeur par le nombre total de canaux dans le dossier. Le logiciel déverrouillera automatiquement une fenêtre de sélection de longueur d’onde située en bas à droite de la fenêtre contextuelle. Utilisez le menu déroulant Couleur pour sélectionner la couleur de chaque couche. Indiquez à chaque canal un nom différent sous la colonne Nom. Une fois toutes les informations mises à jour, cliquez sur Convertir. Le logiciel générera automatiquement un fichier d’image entièrement superposé avec toutes les images individuelles de tous les canaux sélectionnés.
- Calibrer les images.
- Entrez la taille des pixels dans le coin inférieur gauche de l’image. Cliquez avec le bouton droit de la souris sur Uncalibrated > Calibrer le document > taille des pixels.
- Exécutez l’IA entraînée sur le premier canal.
- Commencez à identifier les cils sur l’un des canaux ouverts (p. ex., ACIII; Figure 5A) en utilisant Ai. Le logiciel va maintenant dessiner des binaires sur des cils étiquetés ACIII en fonction de la formation qu’il a reçue pour ce canal. Ce processus prendra quelques secondes. Sélectionnez NIS.ai > Segment.ai > Canaux source > ACIII.
- Exécutez l’Ia entraînée sur le deuxième canal.
- Commencez à identifier les cils sur l’autre canal ouvert (p. ex., MCHR1; Figure 5B) en utilisant Ai. Le logiciel va maintenant dessiner des binaires sur des cils étiquetés MCHR1 en fonction de la formation qu’il a reçue pour ce canal. Ce processus prendra quelques instants. Sélectionnez NIS.ai > Segment.ai > Canaux source > MCHR1.
- Vérifiez les images pour les fichiers binaires mal identifiés.
- Une fois que l’IA a identifié les cils et dessiné des binaires, vérifiez les images pour tout objet mal identifié. Supprimez manuellement tous les fichiers binaires mal identifiés si nécessaire. Sélectionnez Afficher > contrôles d’analyse > barre d’outils binaire > Supprimer l’objet.
- Créez une nouvelle recette GA3.
- Maintenant que les cils ont été identifiés et segmentés, passez à l’analyse de colocalisation à l’aide de l’outil GA3. Cela ouvrira une nouvelle fenêtre avec un espace vide au centre où l’analyse sera définie. Une fenêtre avec tous les fichiers binaires et canaux identifiés sera générée. Vérifiez que tous les canaux et fichiers binaires souhaités requis pour l’analyse sont présents et sélectionnés. Sélectionnez Image > nouvelle recette GA3.
- Supprimez les objets qui touchent la bordure du cadre.
- Étant donné que l’Ai segmentera tous les objets ressemblant à des cils dans le cadre, il détectera également les cils incomplets le long des bords du cadre. Ces objets peuvent être supprimés manuellement à l’étape 5.5 ou automatiquement dans GA3. Sélectionnez Traitement binaire > Supprimer les objets > Toucher les bordures.
- Configurez la voie de colocalisation dans GA3.
- Pour mesurer le chevauchement des deux canaux au sein des cils, utilisez la corrélation de coefficient de Mander. Faites glisser et déposez le nœud Coefficient de Manders dans l’espace vide de la recette GA3 et connectez-le au binaire et aux canaux appropriés. Ici, 'connexion A' se connecte avec le binaire ACIII, 'connexion B' avec le canal MCHR1 et 'connexion C’avec le canal ACIII pour déterminer le chevauchement de MCHR1 dans le binaire ACIII. Sélectionnez Measurement > Object Ratiometry > Manders Coefficient.
REMARQUE: Le logiciel permet de mesurer la colocalisation à l’aide de la corrélation de coefficient de Pearson en utilisant les mêmes étapes que celles décrites dans ce protocole40.
- Pour mesurer le chevauchement des deux canaux au sein des cils, utilisez la corrélation de coefficient de Mander. Faites glisser et déposez le nœud Coefficient de Manders dans l’espace vide de la recette GA3 et connectez-le au binaire et aux canaux appropriés. Ici, 'connexion A' se connecte avec le binaire ACIII, 'connexion B' avec le canal MCHR1 et 'connexion C’avec le canal ACIII pour déterminer le chevauchement de MCHR1 dans le binaire ACIII. Sélectionnez Measurement > Object Ratiometry > Manders Coefficient.
- Ajoutez les mesures dans un seul tableau.
- Combinez toutes les mesures dans une seule table de sortie. Sélectionnez Gestion des données > colonne Basic > Append.
- Mesurer la colocalisation.
- Mesurez les cils en cliquant sur Exécuter. Ce processus prend quelques instants pour mesurer tous les cils dans les images expérimentales. Les données apparaîtront dans une nouvelle fenêtre Résultats d’analyse.
- Exporter des données à des fins d’analyse statistique.
Résultats
Former l’IA pour identifier les cils
Mesurer et évaluer la longueur structurelle et la composition des cils peut être un processus fastidieux, long et sujet aux erreurs. Ici, nous utilisons Ai pour segmenter avec précision les cils à partir d’un grand nombre d’images et analyser leurs longueurs et intensités avec un outil d’analyse(Figure 1). Toutes les approches d’IA nécessitent des étapes de formation pour leur mise en œuvre. Nous avons établi un pipeline de formation pour reconnaître les cils, qui a été effectué en appliquant manuellement des masques binaires sur des structures ciliaires. Ces informations sont ensuite utilisées pour entraîner l’IA en fonction des caractéristiques des pixels sous les binaires appliqués. En règle générale, la formation implique que le logiciel passe par plusieurs itérations, environ 1000, et est considérée comme optimale si le taux de perte ou d’erreur de formation est inférieur à 1%. Toutefois, le nombre d’itérations et d’erreurs dans le processus de formation peut varier en fonction des exemples d’images utilisés pour la formation. Par exemple, après nos séances d’entraînement utilisant des images de cils neuronaux in vitro, le taux d’erreur était de 1,378% contre 3,36% pour les images de section cérébrale in vivo (Figure supplémentaire 1). Une fois la formation terminée, l’IA peut ensuite être utilisée pour segmenter les cils à partir d’images expérimentales en quelques secondes et les masques binaires résultants sont utilisés pour mesurer les paramètres structurels. Cela élimine le besoin de segmenter les objets en utilisant la méthode traditionnelle de seuil d’intensité, ce qui peut être difficile dans les images avec un bruit de fond élevé ou lorsque les objets sont à proximité les uns des autres. Ai réduit également le potentiel d’erreur et de biais en appliquant le même algorithme à toutes les images, quel que soit l’utilisateur.
Mesure de la longueur des cils à l’aide de GA3
La longueur des cils est étroitement régulée et est associée à des impacts fonctionnels sur la signalisation ciliaire16,19. Ici, nous avons mesuré les longueurs de cils à l’aide d’un pipeline d’analyse dans le logiciel NIS Elements appelé General Analysis 3 ou GA3. GA3 aide à combiner plusieurs outils dans un seul flux de travail pour créer des routines personnalisées pour chaque expérience. Nous avons commencé par mesurer la longueur des cils dans une lignée cellulaire. Les cils sur les cellules du canal collecteur médullaire interne (IMCD-3) de souris ont été immunomarqués avec de la tubuline acétylée et imagés à l’aide d’un microscope confocal. Nous avons mesuré les longueurs des cils à l’aide de GA3 après segmentation avec segment.ai (Figure supplémentaire 3A). Alors que la α-tubuline acétylée se trouve préférentiellement dans le cilium primaire, on la trouve également dans d’autres régions riches en microtubules telles que le cytosquelette ainsi que le pont cytocinétique. L’IA entraînée a correctement identifié les cils dans l’image, mais pas d’autres structures positives à la tubuline acétylée non ciliaire. Les cils sur cellules IMCD variaient de 0,5 μm à 4,5 μm avec une longueur moyenne de 1,8 ± 0,04 μm(Figure 2A). Nous avons ensuite testé la capacité de l’IA à mesurer la longueur des cils dans les cultures neuronales primaires. Nous avons cultivé des neurones de l’hypothalamus et de l’hippocampe de souris néonatales pendant 10 jours et les avons immunomarqués avec le marqueur des cils adénylate cyclase III (ACIII)21,41. Lors de l’analyse des cultures neuronales, nous avons trouvé utile d’appliquer un filtre avant d’analyser statistiquement les longueurs. En raison d’un rapport signal/bruit plus faible, plusieurs objets de moins de 1 μm qui n’étaient pas des cils ont été identifiés. Par conséquent, nous avons filtré les données pour éliminer tous les objets de moins de 1 μm de longueur afin de nous assurer que seuls les cils étaient analysés. Dans les neurones hypothalamiques cultivés, les longueurs des cils variaient de 2 μm à 7 μm avec une longueur moyenne de 3,8 ± 0,19 μm(Figure 2B). Fait intéressant, les cils neuronaux de l’hippocampe cultivés étaient plus longs avec une longueur moyenne de 6,73 ±0,15 μm(figure 2C). Il a été rapporté que différents noyaux neuronaux dans l’hypothalamus présentent des longueurs de cils distinctes et que ces cils modifient leur longueur en réponse à des changements physiologiques d’une manière spécifique au noyau19,23. Par conséquent, nous avons également marqué des sections cérébrales hypothalamiques de souris mâles adultes C57BL / 6J avec ACIII et imagé le noyau arqué (ARC) et le noyau paraventriculaire (PVN). En utilisant GA3 pour mesurer la longueur des cils, nous avons observé que les cils hypothalamiques in vivo semblaient plus longs que les cils in vitro. Plus précisément, les cils hypothalamiques in vivo vont de 1 μm à environ 15 μm(figure 3). Il n’y avait pas de différences significatives entre les longueurs de cils dans le PVN (5,54 ± 0,0,42 μm) et celles dans l’ARC (6,16 ± 0,27 μm)(Figure 3C)23. De même, les cils de la région cornu ammonis (CA1) de l’hippocampe présentent une plage de longueur plus étroite allant de 1 μm à 10 μm avec une longueur moyenne de 5,28 ± 0,33 μm(Figure 3). Conformément à des études publiées précédemment, notre analyse utilisant des outils Ai et GA3 a montré que les cils de différentes régions du cerveau présentent une diversité de longueur19,23. De plus, en utilisant cette approche d’IA, nous sommes en mesure d’évaluer rapidement un grand nombre de cils.
Mesure de la composition des cils à l’aide de GA3
Le cilium primaire est un centre de signalisation pour de nombreuses voies qui utilisent divers types de protéines pour remplir des fonctions uniques telles que les protéines motrices, les protéines de transport intraflagelaires et les RCPG pour n’en nommer quequelques-unes3,24,42,43. Le maintien des niveaux appropriés de ces protéines dans le cilium est important pour le bon fonctionnement et semble souvent dépendre du contexte cellulaire. Le marquage fluorescent de ces protéines nous a non seulement permis de les visualiser, mais aussi de quantifier leurs intensités en tant que mesure de la quantité de protéine marquée dans le compartiment relativement petit20. Par conséquent, nous avons cherché à déterminer les intensités d’un RCPG ciliaire, le récepteur 1 de l’hormone de concentration de la mélanine (MCHR1), in vivo dans l’ARC et le PVN de l’hypothalamus de souris mâles adultes24,44. En utilisant Ai et GA3, nous avons mesuré les longueurs des cils positifs MCHR1 ainsi que les intensités pour nous assurer que les objets comptés étaient des cils(figure supplémentaire 3A). Nous avons éliminé les objets post-analyse dont la longueur était inférieure à 2 μm et analysé les intensités des masques binaires restants. Fait intéressant, nous avons constaté que l’intensité de MCHR1 ciliaire dans le PVN est significativement plus élevée que celle dans l’ARC, ce qui indique une présence plus forte de MCHR1 ciliaire dans le PVN(Figure 4). D’autres études sont nécessaires pour déterminer l’importance de MCHR1 ciliaire dans ces circuits neuronaux. Nous avons également mesuré les intensités de MCHR1 ciliaire dans les neurones de culture primaire de l’hypothalamus et de l’hippocampe. Les cils des deux cultures présentent une large distribution des intensités MCHR1 suggérant la présence de populations neuronales hétérogènes (Figure supplémentaire 2). Ainsi, l’utilisation d’outils analytiques sophistiqués tels que Ai et GA3 permet d’évaluer l’hétérogénéité des cils au sein d’un même tissu ou entre plusieurs tissus. Il sera intéressant de voir si d’autres RCPG neuronaux présentent des différences similaires dans leur localisation au sein des neurones du même tissu et si cela change en réponse à des changements physiologiques.
Colocalisation
Bien que la mesure des intensités de fluorescence dans un champ d’image complet puisse donner une impression de protéine, elle ne fournit pas d’informations telles que la distribution spatiale ou la proximité d’autres protéines et structures cellulaires voisines. Ici, nous avons mesuré le chevauchement de MCHR1 avec ACIII comme marqueur de cils en traçant les intensités de MCHR1 par rapport à celles d’ACIII pour chaque masque binaire (Figure 5). Le graphique montre que la majorité des cils sont positifs pour les deux, ACIII et MCHR1, bien que certains cils montrent une expression plus forte d’un canal par rapport à l’autre. En outre, il y a quelques cils qui montrent la présence de ACIII ou MCHR1 comme en témoignent les points qui se trouvent directement sur l’axe des x et l’axe des y respectivement. Pour quantifier ce chevauchement, nous avons mesuré le coefficient de chevauchement de Mander et comparé l’étendue de l’expression de MCHR1 dans les cils neuronaux de l’ARC et du PVN40. Fait intéressant, notre analyse a révélé qu’il y avait une augmentation significative des coefficients du PVN (0,6382 ± 0,0151) par rapport à ceux de l’ARC (0,5430 ± 0,0181)(Figure 5C). Ceci est cohérent avec nos données précédentes où nous avons observé des intensités MCHR1 plus élevées dans le PVN par rapport à l’ARC(Figure 4). Ces données suggèrent que, comme la longueur des cils, le modèle d’expression de MCHR1 dans le compartiment ciliaire varie dans différentes régions du cerveau. En utilisant le même pipeline d’analyse, il sera possible de déterminer si d’autres RCPG ciliaires tels que le récepteur du neuropeptide Y de type 2 (NPY2R) et le récepteur de la somatostatine de type 3 (SSTR3) présentent des quantités similaires de diversité.
Mesure du profil d’intensité le long du cilium
Une fois que les cils ont été identifiés à l’aide de segment.ai, la recette GA3 peut être modifiée pour combiner l’analyse des cils avec l’identification d’autres structures d’intérêt dans l’image. Par exemple, l’étiquetage avec des marqueurs du corps basal est utile pour identifier la polarité des cils. Pour effectuer cette analyse, nous avons imagé des sections hypothalamiques du cerveau de souris P0 qui expriment ARL13B-mCherry et Centrin2-GFP et imagé l’ARC et le PVN34. Ici, les cils ont été identifiés en utilisant Ai comme auparavant, mais maintenant la recette GA3 modifiée comprend l’identification de Centrin2-GFP, une protéine centriolaire trouvée à la base des cils (Figure supplémentaire 3B). En étiquetant Centrin2-GFP, la base des cils peut être distinguée des extrémités des cils positifs ARL13B-mCherry (Figure 6A). Ensuite, au lieu de mesurer l’intensité dans l’ensemble des cils, nous sommes en mesure de mesurer les changements d’intensité ARL13B le long de la longueur descils (Figure 6B). Nous pouvons également comparer les différences d’intensité de l’ARL13B entre les extrémités proximales et les extrémités distales des cils. Pour ce faire, nous avons divisé la longueur du ciliium en bacs de 1 micron à partir de la base et désigné le premier bac de microns comme extrémité proximale et le dernier bac de micron comme extrémité distale. Notre analyse a révélé qu’il y a significativement plus d’ARL13B présent plus près de la base que la pointe du cilium dans l’ARC et le PVN, ce qui est cohérent avec les études publiées précédemment dans les chondrocytes humains45 (Figure 6C). Dans ce type d’analyse, au lieu d’appliquer un filtre de longueur pour exclure les petits objets non ciliaires de l’analyse, seuls les cils associés à l’étiquetage Centrin2-GFP sont analysés. Cela peut être avantageux dans les situations où les mutations génétiques rendent les cils très courts, ou si des changements dans les sous-domaines des cils comme la zone de transition ou la pointe ont été impliqués. L’identification des cils à l’aide de l’analyse Ai et GA3 est hautement adaptable et peut être adaptée pour s’adapter à une variété de questions de recherche complexes.
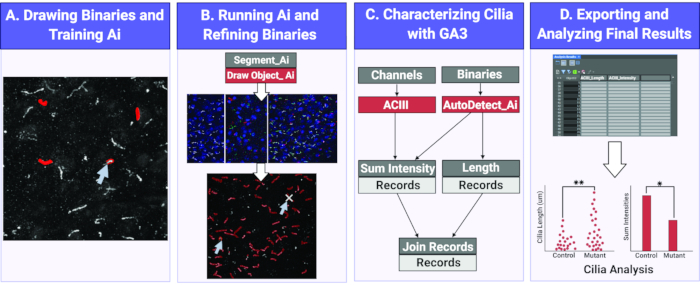
Graphique 1. Flux de travail pour mesurer la longueur et l’intensité des cils à l’aide de l’IA. (A) Pour entraîner l’Ai, des binaires sont dessinés autour des objets d’intérêt (cils) sur les images d’entraînement brutes. À l’aide des binaires dessinés, Segment Ai est entraîné à reconnaître la forme et l’intensité des pixels des cils. (B) Ensuite, le segment Ai entraîné est appliqué à des images expérimentales brutes. Il dessine des binaires sur des objets qu’il reconnaît comme des cils. Ces binaires peuvent être affinés pour s’assurer que tous et seulement les cils sont analysés. (C) Un programme GA3 est construit pour analyser l’intensité et la longueur des objets reconnus par l’Ai. (D) Les enregistrements sont importés dans une table du logiciel. Ce tableau peut ensuite être exporté pour une analyse plus approfondie. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 2. Mesures in vitro de la longueur des cils. Images représentatives des cils dans (A) les cellules IMCD (vert, tubuline acétylée) (B) les cultures hypothalamiques primaires (vert, ACIII) et (C) les cultures hippocampiques (vertes, ACIII). Un Ai entraîné a été utilisé pour reconnaître les cils comme indiqué dans le masque binaire (magenta), puis GA3 a été utilisé pour mesurer la longueur des cils. La distribution de la longueur des cils est représentée graphiquement en pourcentage de cils dans des bacs de 0,5 ou 1,0 micron. * indique un pont cytocinétique correctement non reconnu par Ai. n = 225 cils dans les cellules IMCD de 3 répliques, 54 cils dans l’hypothalamique et 139 cils dans les cultures hippocampiques de 3 animaux. Barres d’échelle 10 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
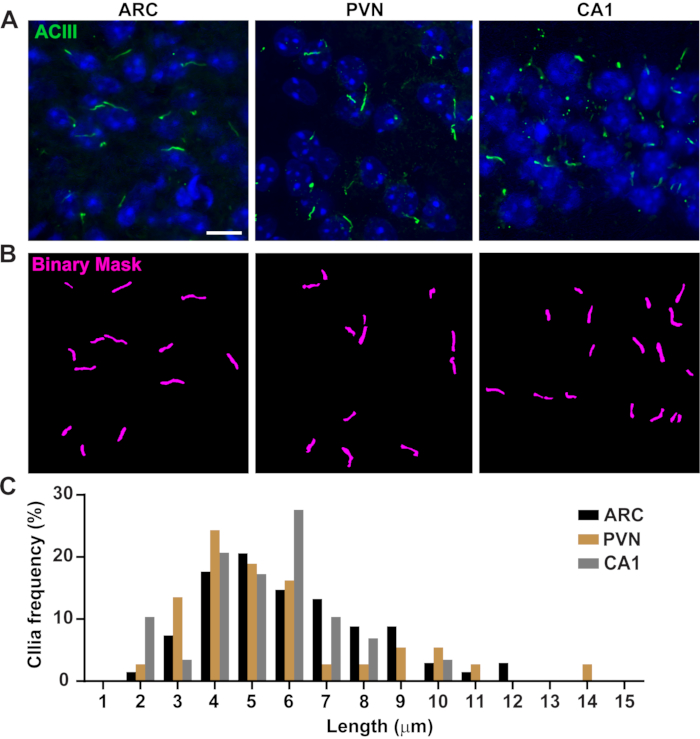
Graphique 3. Mesures in vivo de la longueur des cils. (A) Images représentatives des cils (verts, ACIII) dans l’ARC, le PVN et le CA1 des sections du cerveau de souris adultes. (B) Un Ai entraîné dans les éléments NIS a été utilisé pour reconnaître les cils comme indiqué dans le masque binaire (magenta), puis GA3 a été utilisé pour mesurer la longueur des cils. (C) La distribution de la longueur des cils est représentée graphiquement en pourcentage de cils dans des bacs d’un micron. n= 68 cils en ARC, 36 en PVN et 29 en CA1 de 3 animaux. Barres d’échelle 10 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
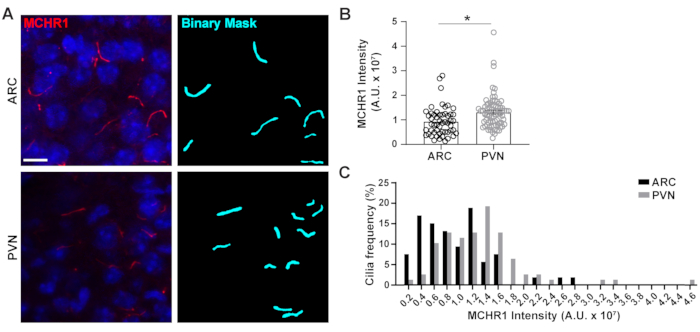
Graphique 4. Ai a aidé les mesures d’intensité de coloration des cils des cils neuronaux hypothalamiques. (A) Images représentatives des cils (MCHR1, rouge) dans l’ARC et le PVN des sections du cerveau de souris adultes. Un Ai entraîné dans les éléments NIS a été utilisé pour reconnaître les cils comme indiqué dans le masque binaire (cyan), puis GA3 a été utilisé pour mesurer l’intensité de la coloration MCHR1 dans les cils. (B) Les intensités MCHR1 sont représentées sous forme de ± S.E.M. Chaque point représente un cilium. * p < 0,05, Test t de Student. (C) La distribution de l’intensité MCHR1 est représentée graphiquement en pourcentage de cils dans des bacs de 0,2 x 107 unités arbitraires (A. U.). n= 53 cils en ARC, 78 en PVN de 3 animaux. Barres d’échelle 10 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
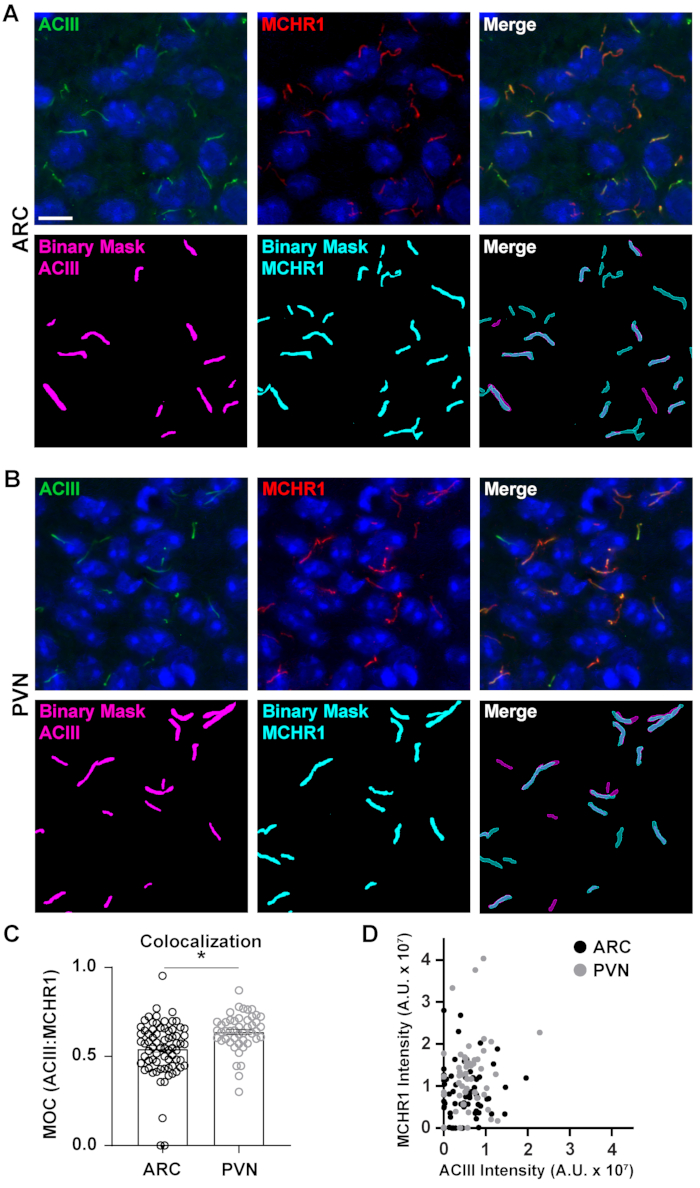
Graphique 5. Ai assisté l’analyse de colocalisation des cils. (A, B) Images représentatives des cils dans l’ARC et le PVN respectivement. Les cils sont étiquetés ACIII (vert) et MCHR1 (rouge). Un Ai entraîné dans les éléments NIS a été utilisé pour reconnaître les cils comme indiqué dans le masque binaire (magenta pour les cils marqués ACIII, cyan pour les cils marqués MCHR1). GA3 a été utilisé pour reconnaître les cils qui contenaient à la fois ACIII et MCHR1. (C) Les valeurs du coefficient de chevauchement (MOC) de Manders sont représentées sous forme de ± S.E.M. Chaque point représente un cilium. * p < 0,05, Test t de Student. (D) Nuage de points de l’intensité MCHR1 par rapport à l’intensité ACIII en ARC et PVN. Chaque point représente un cilium. n= 72 cils en ARC, 47 en PVN de 3 animaux. Barres d’échelle 10 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
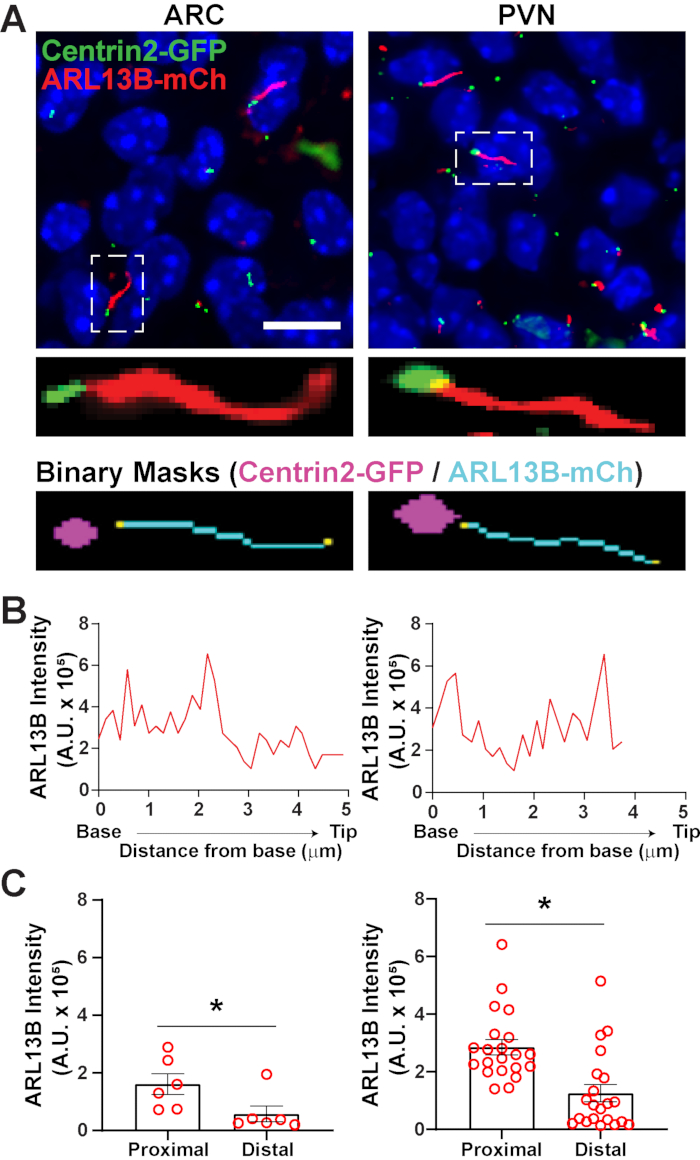
Graphique 6. Analyse des cils et du corps basal. (A) Images représentatives des cils (rouge, ARL13B-mCherry) et du marqueur corporel basal (vert, Centrin2-GFP) dans l’ARC et le PVN de souris P0. Une IA entraînée a été utilisée pour reconnaître les cils comme indiqué dans le masque binaire (cyan). Le masque binaire pour le corps basal (magenta) a été dessiné par seuil dans la recette GA3. (B) Intensité représentative du balayage linéaire d’un ciliium. (C) Intensités ARL13B aux extrémités proximale et distale de l’Ai identifiées comme étant représentées graphiquement comme moyennes ± S.E.M. Les extrémités proximale et distale sont définies comme la région située respectivement dans la première longueur de 1 μm et la dernière longueur de 1 μm à partir de la base du cilium. Chaque point représente un cilium. * p < 0,05. n = 6 cils en ARC de 2 animaux et 21 cils en PVN de 3 animaux. Barres d’échelle 10 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
Figure supplémentaire 1. Graphiques de perte d’entraînement Ai. (A, B) Graphiques montrant la perte d’entraînement des segment.ai sur les cils neuronaux in vitro et in vivo, respectivement. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 2. Ai Assisted Cilia mesures de l’intensité de coloration des cils neuronaux in vitro. (A, B) Images représentatives des cils (MCHR1, rouge) dans les cultures hypothalamique et hippocampique primaires, respectivement. Un Ai entraîné dans les éléments NIS a été utilisé pour reconnaître les cils comme indiqué dans le masque binaire (cyan), puis GA3 a été utilisé pour mesurer l’intensité de la coloration MCHR1 dans les cils. La distribution de l’intensité MCHR1 est représentée graphiquement en pourcentage de cils dans 1000 bacs A.U. pour les bacs hypothalamiques et 2000 bacs A.U. pour les cultures hippocampiques. n= 30 cils en hypothalamique et 106 cils en cultures hippocampiques de 3 animaux. Barres d’échelle 10 μm. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 3. Analyse générale 3 recettes pour l’analyse des cils. (A) Recette d’analyse générale simple (GA3) pour la mesure de la longueur des cils, de l’intensité et du coefficient de Mander. (B) Recette complexe GA3 pour la mesure de l’intensité le long de la longueur du cil à l’aide d’un marqueur pour le corps basal. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Les mesures de longueur et d’intensité sont des moyens courants d’analyse des cils primaires, mais il n’existe pas de méthode conventionnelle normalisée utilisée sur le terrain. L’identification et la quantification des cils primaires à l’aide de logiciels tels qu’ImageJ prennent beaucoup de temps et sont sujettes aux biais et aux erreurs de l’utilisateur. Il est donc difficile d’analyser avec précision de grands ensembles de données. Nous montrons ici que l’utilisation d’un programme d’IA peut surmonter bon nombre de ces défis, ce qui rend l’analyse à haut débit des cils primaires réalisable. Ici, nous décrivons la procédure d’entraînement d’une application basée sur l’IA pour reconnaître les cils primaires et décrivons les étapes nécessaires pour analyser la longueur et l’intensité.
Bien que la formation initiale de l’IA à reconnaître les cils nécessite beaucoup de temps de la part de l’utilisateur, une fois terminée, elle peut être utilisée sur n’importe quel ensemble de données acquis avec les mêmes paramètres. Le masque binaire généré par l’Ai est modifiable de sorte que toute erreur peut être corrigée. Cependant, les erreurs dans l’identification des cils devraient signaler à l’utilisateur que l’IA doit être davantage entraînée avec des images supplémentaires. L’un des principaux avantages de cette méthode est que l’IA peut être entraînée à reconnaître les cils dans différents types d’échantillons en 2D et en 3D. Les méthodes d’analyse antérieures générées dans les laboratoires ont diverses limites, notamment l’exigence d’un seuil manuel pour l’identification et des problèmes d’identification des cils imagés à partir de coupes de tissus où la densité cellulaire est élevée36,46,47. Ces méthodes sont également spécialisées pour l’analyse des cils tandis que l’analyse à l’aide du logiciel NIS Elements peut évaluer plusieurs aspects des images simultanément. Étant donné que l’IA décrit ici fait partie du progiciel NIS Elements, les images acquises à l’aide d’un microscope Nikon peuvent être facilement poursuivies jusqu’à l’analyse. Cependant, l’imagerie avec Nikon n’est pas nécessaire pour l’utilisation de cette méthode. Quel que soit le format de fichier de données brutes capturé, les fichiers « .tif » peuvent être ouverts par NIS Elements pour être utilisés dans l’Ai.
Cette application d’IA au sein de NIS Elements est largement disponible et fait peut-être déjà partie d’un logiciel d’analyse d’images utilisé par les laboratoires étudiant les cils primaires. Avec l’expansion de la prévalence de la technologie de l’IA, d’autres logiciels d’imagerie peuvent élargir leurs options d’analyse pour inclure un module d’IA similaire. L’application de l’analyse de l’IA à l’identification des cils peut être utilisée pour plusieurs aspects différents de l’analyse des cils. Bien que nous ayons décrit des méthodes pour quelques analyses simples telles que la longueur(Figure 2 et 3),l’intensité(Figure 4)et la colocalisation(Figure 5),une analyse plus sophistiquée peut être ajoutée au flux de travail d’analyse GA3 comme dans la Figure 6. Par exemple, au lieu de mesurer l’intensité d’un ciliium complet, les différences d’intensité au sein d’une sous-région d’un cil peuvent être intéressantes pour évaluer la localisation sous-ciliaire. Les différences d’intensité au sein d’une sous-région d’un cilium pourraient indiquer que la protéine s’accumule à la pointe ou à la base du cilium, comme la façon dont les protéines Gli sont enrichies à la pointe descils 48. En outre, cette application d’IA peut être utilisée pour identifier facilement les différences entre les génotypes ou les groupes de traitement. Bien que notre laboratoire utilise principalement cette méthode pour analyser les cils imagés à partir de coupes cérébrales ou de cultures neuronales, elle peut être appliquée à des images acquises à partir de diverses lignées cellulaires ou d’autres types de tissus. La flexibilité du type d’échantillon sur lequel cette application peut être utilisée rend cette méthode d’analyse précieuse pour de nombreux groupes différents étudiant les cils primaires ou tout organite discret évalué tel que les mitochondries, le noyau ou l’ER.
Déclarations de divulgation
Le co-auteur Wesley Lewis est un employé de Nikon. Il n’y a pas de divulgation financière.
Remerciements
Ce travail a été financé par le National Institute of Diabetes and Digestive and Kidney Diseases R01 DK114008 à l’ONF et l’American Heart Association Fellowship Grant #18PRE34020122 à RB. Nous remercions Rich Gruskin, directeur général de Nikon Software, Melissa Bentley, Courtney Haycraft et Teresa Mastracci pour leurs commentaires perspicaces sur le manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| Intel Xeon, 3.6 GHz, 32GB RAM | Intel Corporation | W-2123 | Processor used for running NIS Elements. |
| Nikon Elements Software | Nikon Instruments Inc. | - | Ai and GA3 software |
| Quadro RTX 4000 Graphics card | NVIDIA Corporation | Quadro RTX 4000 | |
| Windows 10 Professional 64-bit | Microsoft Inc. | - | Operating system used for running NIS Elements |
| Workstation | HP Development Company, L.P. | HP Z4G4 | Workstation used for running NIS Elements |
Références
- Wang, L., et al. Ciliary gene RPGRIP1L is required for hypothalamic arcuate neuron development. JCI Insight. 4 (3), (2019).
- Siljee, J. E., et al. Subcellular localization of MC4R with ADCY3 at neuronal primary cilia underlies a common pathway for genetic predisposition to obesity. Nature Genetics. 50 (2), 180-185 (2018).
- Davenport, J. R., et al. Disruption of intraflagellar transport in adult mice leads to obesity and slow-onset cystic kidney disease. Current Biology. 17 (18), 1586-1594 (2007).
- Berbari, N. F., O'Connor, A. K., Haycraft, C. J., Yoder, B. K. The primary cilium as a complex signaling center. Current Biology. 19 (13), 526-535 (2009).
- Walz, G. Role of primary cilia in non-dividing and post-mitotic cells. Cell Tissue Research. 369 (1), 11-25 (2017).
- Nachury, M. V., Mick, D. U. Establishing and regulating the composition of cilia for signal transduction. Nature Reviews. Molecular Cell Biology. 20 (7), 389-405 (2019).
- Goetz, S. C., Anderson, K. V. The primary cilium: a signalling centre during vertebrate development. Nature Reviews Genetics. 11 (5), 331-344 (2010).
- Engle, S. E., Bansal, R., Antonellis, P. J., Berbari, N. F. Cilia signaling and obesity. Seminars in Cell and Developmental Biology. , (2020).
- Reiter, J. F., Leroux, M. R. Genes and molecular pathways underpinning ciliopathies. Nature reviews. Molecular Cell Biology. 18 (9), 533-547 (2017).
- Waters, A. M., Beales, P. L. Ciliopathies: an expanding disease spectrum. Pediatric Nephrology. 26 (7), Berlin, Germany. 1039-1056 (2011).
- Hildebrandt, F., Benzing, T., Katsanis, N. Ciliopathies. New England Journal of Medicine. 364 (16), 1533-1543 (2011).
- Vaisse, C., Reiter, J. F., Berbari, N. F. Cilia and Obesity. Cold Spring Harbor Perspectives in Biology. 9 (7), (2017).
- Berbari, N. F., et al. Leptin resistance is a secondary consequence of the obesity in ciliopathy mutant mice. Proceedings of the National Academy of Sciences of the United States of America. 110 (19), 7796-7801 (2013).
- Jacobs, D. T., et al. Dysfunction of intraflagellar transport-A causes hyperphagia-induced obesity and metabolic syndrome. Disease Models & Mechanisms. 9 (7), 789-798 (2016).
- Arsov, T., et al. Fat aussie--a new Alström syndrome mouse showing a critical role for ALMS1 in obesity, diabetes, and spermatogenesis. Molecular Endocrinology. 20 (7), 1610-1622 (2006).
- Tam, L. W., Ranum, P. T., Lefebvre, P. A. CDKL5 regulates flagellar length and localizes to the base of the flagella in Chlamydomonas. Molecular Biology of the Cell. 24 (5), 588-600 (2013).
- Rajagopalan, V., Subramanian, A., Wilkes, D. E., Pennock, D. G., Asai, D. J. Dynein-2 affects the regulation of ciliary length but is not required for ciliogenesis in Tetrahymena thermophila. Molecular Biology of the Cell. 20 (2), 708-720 (2009).
- Bengs, F., Scholz, A., Kuhn, D., Wiese, M. LmxMPK9, a mitogen-activated protein kinase homologue affects flagellar length in Leishmania mexicana. Molecular Microbiology. 55 (5), 1606-1615 (2005).
- Han, Y. M., et al. Leptin-promoted cilia assembly is critical for normal energy balance. Journal of Clinical Investigation. 124 (5), 2193-2197 (2014).
- Caspary, T., Marazziti, D., Berbari, N. F. Cilia: Methods and Protocols. Satir, P., Tvorup Christensen, S. , Springer. New York. 203-214 (2016).
- Bishop, G. A., Berbari, N. F., Lewis, J., Mykytyn, K. Type III adenylyl cyclase localizes to primary cilia throughout the adult mouse brain. Journal of Comparative Neurology. 505 (5), 562-571 (2007).
- Domire, J. S., Mykytyn, K. Markers for neuronal cilia. Methods in Cell Biology. 91, 111-121 (2009).
- Sun, J. S., et al. Ventromedial hypothalamic primary cilia control energy and skeletal homeostasis. Journal of Clinical Investigation. 131 (1), (2021).
- Berbari, N. F., Johnson, A. D., Lewis, J. S., Askwith, C. C., Mykytyn, K. Identification of ciliary localization sequences within the third intracellular loop of G protein-coupled receptors. Molecular Biology of the Cell. 19 (4), 1540-1547 (2008).
- Brailov, I., et al. Localization of 5-HT(6) receptors at the plasma membrane of neuronal cilia in the rat brain. Brain Research. 872 (1-2), 271-275 (2000).
- Domire, J. S., et al. Dopamine receptor 1 localizes to neuronal cilia in a dynamic process that requires the Bardet-Biedl syndrome proteins. Cellular and Molecular Life Sciences. 68 (17), 2951-2960 (2011).
- Handel, M., et al. Selective targeting of somatostatin receptor 3 to neuronal cilia. Neuroscience. 89 (3), 909-926 (1999).
- Koemeter-Cox, A. I., et al. Primary cilia enhance kisspeptin receptor signaling on gonadotropin-releasing hormone neurons. Proceedings of the National Academy of Sciences of the United States of America. 111 (28), 10335-10340 (2014).
- Mukhopadhyay, S., et al. The ciliary G-protein-coupled receptor Gpr161 negatively regulates the Sonic hedgehog pathway via cAMP signaling. Cell. 152 (1-2), 210-223 (2013).
- Berman, S. A., Wilson, N. F., Haas, N. A., Lefebvre, P. A. A novel MAP kinase regulates flagellar length in Chlamydomonas. Current Biology. 13 (13), 1145-1149 (2003).
- Nguyen, R. L., Tam, L. W., Lefebvre, P. A. The LF1 gene of Chlamydomonas reinhardtii encodes a novel protein required for flagellar length control. Genetics. 169 (3), 1415-1424 (2005).
- Tam, L. W., Wilson, N. F., Lefebvre, P. A. A CDK-related kinase regulates the length and assembly of flagella in Chlamydomonas. Journal of Cell Biology. 176 (6), 819-829 (2007).
- O'Connor, A. K., et al. An inducible CiliaGFP mouse model for in vivo visualization and analysis of cilia in live tissue. Cilia. 2 (1), 8(2013).
- Bangs, F. K., Schrode, N., Hadjantonakis, A. K., Anderson, K. V. Lineage specificity of primary cilia in the mouse embryo. Nature Cell Biology. 17 (2), 113-122 (2015).
- Delling, M., et al. Primary cilia are not calcium-responsive mechanosensors. Nature. 531 (7596), 656-660 (2016).
- Saggese, T., Young, A. A., Huang, C., Braeckmans, K., McGlashan, S. R. Development of a method for the measurement of primary cilia length in 3D. Cilia. 1 (1), 11(2012).
- Kobayashi, Y., Hamamoto, A., Saito, Y. Analysis of ciliary status via G-protein-coupled receptors localized on primary cilia. Microscopy. 69 (5), 277-285 (2020).
- Zhou, L. Q., et al. Artificial intelligence in medical imaging of the liver. World Journal of Gastroenterology. 25 (6), 672-682 (2019).
- Naugler, C., Church, D. L. Automation and artificial intelligence in the clinical laboratory. Critical Reviews in Clinical Laboratory Sciences. 56 (2), 98-110 (2019).
- Dunn, K. W., Kamocka, M. M., McDonald, J. H. A practical guide to evaluating colocalization in biological microscopy. American Journal of Physiology-Cell Physiology. 300 (4), 723-742 (2011).
- Bansal, R., et al. Hedgehog Pathway Activation Alters Ciliary Signaling in Primary Hypothalamic Cultures. Frontiers in Cellular Neuroscience. 13, 266(2019).
- Jin, H., et al. The conserved Bardet-Biedl syndrome proteins assemble a coat that traffics membrane proteins to cilia. Cell. 141 (7), 1208-1219 (2010).
- Liew, G. M., et al. The intraflagellar transport protein IFT27 promotes BBSome exit from cilia through the GTPase ARL6/BBS3. Developmental Cell. 31 (3), 265-278 (2014).
- Engle, S. E., et al. A CreER Mouse to Study Melanin Concentrating Hormone Signaling in the Developing Brain. Genesis. , (2018).
- Thorpe, S. D., et al. Reduced primary cilia length and altered Arl13b expression are associated with deregulated chondrocyte Hedgehog signaling in alkaptonuria. Journal of Cellular Physiology. 232 (9), 2407-2417 (2017).
- Lauring, M. C., et al. New software for automated cilia detection in cells (ACDC). Cilia. 8, 1(2019).
- Dummer, A., Poelma, C., DeRuiter, M. C., Goumans, M. J., Hierck, B. P. Measuring the primary cilium length: improved method for unbiased high-throughput analysis. Cilia. 5, 7(2016).
- Haycraft, C. J., et al. Gli2 and Gli3 localize to cilia and require the intraflagellar transport protein polaris for processing and function. PLoS Genetics. 1 (4), 53(2005).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon