Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Isolierung und Selektion entomopathogener Pilze aus Bodenproben und Bewertung der Pilzvirulenz gegen Schadinsekten
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll vor, das auf dem Mehlwurm (Tenebrio molitor) -Ködersystem basiert, das zur Isolierung und Auswahl entomopathogener Pilze (EPF) aus Bodenproben verwendet wurde. Eine effektive Konidienzahlformel (ECN) wird verwendet, um einen hoch stresstoleranten EPF basierend auf physiologischen Eigenschaften für die mikrobielle Schädlingsbekämpfung im Feld auszuwählen.
Zusammenfassung
Entomopathogene Pilze (EPF) sind eines der mikrobiellen Bekämpfungsmittel für den integrierten Pflanzenschutz. Um lokale oder invasive Schädlinge zu bekämpfen, ist es wichtig, einheimische EPF zu isolieren und auszuwählen. Daher wurde in dieser Studie die Bodenködermethode in Kombination mit dem Insektenködersystem (Mehlwurm, Tenebrio molitor) mit einigen Modifikationen verwendet. Die isolierten EPF wurden dann dem Virulenztest gegen den landwirtschaftlichen Schädling Spodoptera litura unterzogen. Darüber hinaus wurden die potenziellen EPF-Stämme morphologischen und molekularen Identifizierungen unterzogen. Darüber hinaus wurden die Konidienproduktion und der Thermotoleranztest für die vielversprechenden EPF-Stämme durchgeführt und verglichen; Diese Daten wurden weiter in die Formel der effektiven Konidienzahl (ECN) für das Laborranking substituiert. Das Bodenköder-Mehlwurm-System und die ECN-Formel können verbessert werden, indem Insektenarten ersetzt und mehr Stressfaktoren für die Bewertung der Kommerzialisierung und Feldanwendung integriert werden. Dieses Protokoll bietet einen schnellen und effizienten Ansatz für die EPF-Selektion und wird die Forschung an biologischen Bekämpfungsmitteln verbessern.
Einleitung
Derzeit werden entomopathogene Pilze (EPF) häufig bei der mikrobiellen Bekämpfung von landwirtschaftlichen, forstwirtschaftlichen und gartenbaulichen Schädlingen eingesetzt. Die Vorteile von EPF sind seine große Wirtsbreite, gute Umweltanpassungsfähigkeit, umweltfreundliche Natur und dass es mit anderen Chemikalien verwendet werden kann, um den Synergieeffekt für den integrierten Pflanzenschutz zu zeigen1,2. Für die Anwendung als Schädlingsbekämpfungsmittel ist es notwendig, eine große Anzahl von EPF entweder aus erkrankten Insekten oder der natürlichen Umgebung zu isolieren.
Die Probenahme dieser Organismen von ihren Wirten hilft beim Verständnis der geografischen Verteilung und Prävalenzrate von EPF in natürlichen Wirten3,4,5. Die Sammlung von pilzinfizierten Insekten ist jedoch in der Regel durch Umweltfaktoren und Insektenpopulationen im Feld begrenzt4. In Anbetracht der Tatsache, dass Insektenwirte nach einer EPF-Infektion sterben und dann in den Boden fallen, könnte die Isolierung von EPF aus Bodenproben eine stabile Ressource sein3,6. Zum Beispiel sind Saprophyten dafür bekannt, den toten Wirt als ihre Ressource für das Wachstum zu nutzen. Die Bodenköder- und selektiven Mediumsysteme wurden häufig verwendet, um EPF aus dem Boden zu erkennen und zu isolieren3,4,7,8,9,10.
Bei der Methode des selektiven Mediums wird die verdünnte Bodenlösung auf ein Medium plattiert, das Breitbandantibiotika (z. B. Chloramphenicol, Tetracyclin oder Streptomycin) enthält, um das Wachstum von Bakterien zu hemmen2,3,9,11. Es wurde jedoch berichtet, dass diese Methode die Vielfalt und Dichte des Stammes verzerren und zu einer Über- oder Unterschätzung vieler mikrobieller Gemeinschaften führen kann6. Darüber hinaus sind die isolierten Stämme weniger pathogen und konkurrieren während der Isolierung mit Saprophyten. Es ist schwierig, EPF aus der verdünnten Bodenlösung zu isolieren3. Anstatt ein selektives Medium zu verwenden, isoliert die Bodenködermethode EPF von den infizierten toten Insekten, die für 2-3 Wochen gelagert werden können, wodurch eine effizientere und standardisierte EPF-Trennmethode3,4,7,6 bereitgestellt wird. Da die Methode einfach zu bedienen ist, kann man eine Vielzahl von pathogenen Stämmen zu geringen Kosten isolieren4. Daher ist es von vielen Forschern weit verbreitet.
Beim Vergleich der verschiedenen Arten von Insektenködersystemen sind Beauveria bassiana und Metarhizium anisopliae die häufigsten EPF-Arten, die in Insekten der Hemiptera, Lepidoptera, Blattella und Coleoptera gefunden werden6,12,13,14. Unter diesen Insektenködern zeigen Galleria mellonella (Ordnung Lepidoptera) und Tenebrio molitor (Ordnung Coleoptera) im Vergleich zu anderen Insekten höhere Erholungsraten von Beauveria und Metarhizium spp.. Daher werden G. mellonella und T. molitor häufig für Insektenköder verwendet. Im Laufe der Jahre hat das Landwirtschaftsministerium der Vereinigten Staaten (USDA) eine EPF-Bibliothek (Agricultural Research Service Collection of EPF cultures, ARSEF) eingerichtet, die eine Vielzahl von Arten enthält, darunter 4081 Arten von Beauveria spp., 18 Arten von Clonostachys spp., 878 Arten von Cordyceps spp., 2473 Arten von Metarhizium spp., 226 Arten von Purpureocillium spp., und 13 Arten von Pochonia spp. unter anderem15. Eine weitere EPF-Bibliothek wurde vom Entomology Research Laboratory (ERL) der University of Vermont in den Vereinigten Staaten für ca. 30 Jahre gebaut. Es umfasst 1345 Stämme von EPF aus den Vereinigten Staaten, Europa, Asien, Afrika und dem Nahen Osten16.
Um lokale oder Invasionsschädlinge in Taiwan zu kontrollieren, ist die Isolierung und Auswahl der einheimischen EPF erforderlich. Daher haben wir in diesem Protokoll das Verfahren der Bodenködermethode modifiziert und beschrieben und mit dem Insektenködersystem (Mehlwurm, Tenebrio molitor) kombiniert17. Basierend auf diesem Protokoll wurde eine EPF-Bibliothek eingerichtet. Für die vorläufigen EPF-Isolate wurden zwei Screening-Runden (Quantifizierung der Impfung) durchgeführt. EPF-Isolate zeigten Pathogenität gegenüber Insekten. Die potenziellen Stämme wurden morphologischen und molekularen Identifizierungen unterzogen und durch den Thermotoleranz- und Konidialproduktionstest weiter analysiert. Darüber hinaus wurde auch ein Konzept der effektiven Konidienzahl (ECN) vorgeschlagen. Unter Verwendung der ECN-Formel und der Hauptkomponentenanalyse (PCA) wurden die potenziellen Stämme unter simuliertem Umgebungsdruck analysiert, um den Prozess der Einrichtung und des Screenings der EPF-Bibliothek abzuschließen. Anschließend wurde die Pathogenität vielversprechender EPF-Stämme auf den Zielschädling (z.B. Spodoptera litura) getestet. Das aktuelle Protokoll integriert Thermotoleranz- und konidiale Produktionsdaten in die ECN-Formel und PCA-Analyse, die als Standard-Ranking-System für EPF-bezogene Forschung verwendet werden kann.
Protokoll
HINWEIS: Das gesamte Flussdiagramm ist in Abbildung 1 dargestellt.
1. Isolierung und Selektion potenzieller entomopathogener Pilze (EPF)
- Sammeln Sie die Bodenprobe
- Entfernen Sie 1 cm der Oberflächenerde und sammeln Sie dann den Boden innerhalb der 5-10 cm Tiefe mit einer Schaufel von jeder Probenahmestelle.
HINWEIS:Probenahmestellen wären Berg-, Wald- oder dünn besiedelte Gebiete, um die Kontamination künstlich versprühter EPF-Stämme zu vermeiden. Stellen Sie sicher, dass die Bereiche für die Bodenprobenentnahme an der Oberfläche mit Unkraut bedeckt sind. Trockener Boden oder feuchter Boden ist für dieses Experiment nicht geeignet. - Zeichnen Sie die Details jedes Probenahmestandorts auf, einschließlich GPS, Höhe, Art des Feldes, Jahrestemperatur, jährlicher Niederschlag, Erfassungszeit (Saison), Bodentyp und pH-Wert.
- Sammeln Sie 100 g der Bodenprobe in einer Plastiktüte und halten Sie sie bei Raumtemperatur, wenn Sie sie innerhalb von 3 h im Labor dem Pilzisolationsprotokoll unterziehen.
HINWEIS: Wenn die Probe nicht innerhalb von 3 h verwendet werden kann, lagern Sie den Boden bei Raumtemperatur unter dunklen Bedingungen. Wird das Experiment nicht sofort durchgeführt, kann die Bodenprobe 1 Woche lang bis zum Beginn des Protokolls bei 4 °C gelagert werden18,19.
- Entfernen Sie 1 cm der Oberflächenerde und sammeln Sie dann den Boden innerhalb der 5-10 cm Tiefe mit einer Schaufel von jeder Probenahmestelle.
- Köder und Isolierung der EPF mit Mehlwurm (Tenebrio molitor)
- 100 g der Bodenprobe in einen Plastikbecher geben (Kappendurchmesser = 8,5 cm, Höhe = 12,5 cm) und dann 5 Mehlwürmer bei Raumtemperatur im Dunkeln für 2 Wochen auf die Bodenoberfläche legen.
HINWEIS: Andere Arten von Plastikbecherbehältern können ebenfalls verwendet werden. Wenn die Böden zu trocken sind (rissig oder sandig), sprühen Sie sterilisiertes ddH2O (ca. 5-10 ml) auf die Böden. Die Körperlänge ca. 2,5 cm (14. Stadium) von T. molitor-Larven hilft bei Pilzisolationssieben20. - Beobachten und notieren Sie die Larven täglich auf Mortalität und Mykose; Halten Sie die toten Larven bis 2 Wochen in der Tasse für die Pilzisolierung.
HINWEIS: Die Pilz-Conidia-Spore in den Bodenproben wird während des obigen Prozesses an den Mehlwurmlarven anhaften. Die Pilzmykose wird beobachtet, wenn die Hyphen aus der intersegmentalen Membran wachsen, und dann wird der ganze Körper mit dem Myzel bedeckt. Die Sporulation beginnt nach 7 Tagen und die Farbe der Pilzinfektion ändert sich in die Farbe der Konidienmasse. - Übertragen Sie die toten Insekten auf eine saubere Bank und verwenden Sie einen sterilen Zahnstocher, um die Konidien zu sammeln. Streifen Sie sie im Labor auf einer Viertelstärke Sabouraud Dextrose Agar Medium (1/4 SDA) Platte (55 mm)21. Die Kulturplatte bei 25 °C für 7 Tage inkubieren, um die Primärkultur der Pilze zu erhalten.
HINWEIS: Die 1/4 SDA-Platte wird wie folgt zubereitet: Mischen Sie 1,5 g Sabouraud Dextrosebrühe und 3 g Agar in 200 ml H2O und sterilisieren Sie dann für 20 min. Aliquot 1/4 SDA in jede 55 mm Petrischale vor der Erstarrung. Die erstarrten 1/4 SDA-Platten werden bis zum Gebrauch bei 4 °C gelagert. - Streift jeden Primärkulturpilz auf einer 55 mm 1/4 SDA-Platte in einer laminaren Strömung erneut und inkubiert die Kulturplatte bei 25 °C für 7 Tage, um einzelne Pilzkolonien zu erhalten.
- Wiederholen Sie diese erneute Isolierung ~ 2-3 Mal und beobachten Sie unter Lichtmikroskopie, um einzelne und reine morphologische Pilzkolonien zu erhalten.
HINWEIS: Isolieren Sie alle EPF mit T. molitor-bait und lagern Sie sie wie im folgenden Abschnitt beschrieben. Die Trennung, Konservierung, Reinkultur und Streifenbildung müssen in den nachfolgenden Abschnitten in einer laminaren Strömung durchgeführt werden.
- 100 g der Bodenprobe in einen Plastikbecher geben (Kappendurchmesser = 8,5 cm, Höhe = 12,5 cm) und dann 5 Mehlwürmer bei Raumtemperatur im Dunkeln für 2 Wochen auf die Bodenoberfläche legen.
- Lagern der EPF-Isolate
- Schneiden Sie 3 der 5 mm Agarblöcke am Rand jeder isolierten reinen Pilzkulturplatte mit einem Korkbohrer ab und legen Sie sie als ein Replikat in ein 1,5 ml Mikrozentrifugenröhrchen.
HINWEIS: Für die Lagerung der EPF-Stämme nach dem 2. Virulenztest werden drei Replikate für jedes Pilzisolat empfohlen. Die molekulare Identifizierung wird auch empfohlen, wenn der Lagerraum begrenzt ist. - 250 μL 0,03%ige Tensidlösung (Materialtabelle) und 250 μL 60%ige Glycerin unter Verwendung einer Mikropipette in das 1,5 ml Mikrozentrifugenröhrchen geben; dann Wirbel für 10 s.
- Verschließen Sie das 1,5 mL Mikrozentrifugenröhrchen mit Paraffinfolie und kühlen Sie es in einem -20 °C kühlschrank für 24 h vor. Anschließend werden die vorgekühlten Pilzvorräte zur Kryokonservierung in einen -80 °C warmen Kühlschrank überführt.
ANMERKUNG: In Abschnitt 1.4 wurden extra reine Pilzkulturplatten (abgesehen von den kryokonservierten Proben) verwendet.
- Schneiden Sie 3 der 5 mm Agarblöcke am Rand jeder isolierten reinen Pilzkulturplatte mit einem Korkbohrer ab und legen Sie sie als ein Replikat in ein 1,5 ml Mikrozentrifugenröhrchen.
- 1. Pathogenitätsscreening für Pilzisolate
- Fünf T. molitor-Larven direkt auf die Oberfläche jeder reinen Pilzkulturplatte bei 25 °C auftragen.
- Beobachten und notieren Sie die Mykose und Mortalität für 10 Tage. Wählen Sie das Pilzisolat zur weiteren Analyse aus.
HINWEIS: Pilzisolate, die eine Mortalität von 100% verursachen, werden für den 2. Virulenztest ausgewählt, um ihre Virulenz gegenüber T. molitor-Larven zu bestätigen. Alternativ kann der Forscher die Kriterien gemäß seiner eigenen Studie anpassen.
- 2. Virulenztest von Pilzisolaten
HINWEIS: Basierend auf dem 1. Pathogenitätsscreening die ausgewählten Pilzisolate von -80 °C für den 2. Virulenztest wiederherstellen . Der Zweck des 2. Virulenztests besteht darin, die Pathogenität der ausgewählten Pilzisolate nach der 1. Screening-Runde zu quantifizieren.- Ernten Sie Konidien jedes Pilzisolats durch Wirbeln für 1 min und zählen Sie die Anzahl der Konidien mit einem Hämozytometer.
- Die Konidiensuspension wird auf eine Konzentration von 1 x 107 Konidien/ml in einer 0,03%igen Tensidlösung eingestellt (Materialtabelle).
- 10 μL der Pilzsuspension auf 55 mm 1/4 SDA-Platten verteilen und 7 Tage bei 25 °C im Dunkeln wachsen lassen.
- Fünf T. molitor-Larven direkt auf die Oberfläche jeder reinen Pilzkulturplatte (ca. 6 x 107 Konidien) legen. Die Platten mit Paraffinfolie verschließen und bei 25 °C im Dunkeln inkubieren.
- Beobachten und notieren Sie die Mykose und Mortalität für 10 Tage.
- Wiederholen Sie den Test (von Schritt 1.51 bis 1.5.5) in dreifacher Ausfertigung für jedes Pilzisolat.
HINWEIS: Pilzisolate, die eine Mortalität von 100% verursachen, werden für den 3. Virulenztest ausgewählt, um ihre Virulenz gegen den Schädling zu bestätigen.
- 3. Virulenztest von Pilzisolaten für Zielschädlinge (Spodoptera litura als Beispiel)
- Wiederholen Sie die Schritte 1.5.2 bis 1.5.6 mit ausgewählten Isolaten aus der 2. Virulenz, um die Virulenz gegen den Zielschädling zu testen.
- Berechnen Sie den LT50 jedes Pilzisolats22.
HINWEIS: Der LT50 jedes Pilzisolats wurde durch verallgemeinerte lineare Modelle (GLMs) mit R Studio (Version 3.4.1) berechnet; Die quasibinomiale Fehlerverteilung und eine Log-Link-Funktion können verwendet werden, um eine Überdispersion zu berücksichtigen.
2. Molekulare Identifizierung von EPF
- Extraktion von pilzlicher genomischer DNA
- Sammeln Sie ca. 1 cm2 EPF von der 7-tägigen 1/4 SDA-Platte.
- Extrahieren Sie die genomische Pilz-DNA mit einem Pilz-Genom-DNA-Extraktionskit gemäß den Anweisungen des Herstellers23 (Materialtabelle).
- PCR-Amplifikation und DNA-Sequenzierung
- Amplifizieren Sie die Fungal ITS-Region durch PCR der DNA-Probe21 mit dem PCR Master Mix (2x), ITS1F/ITS4R Primer Set24 (Tabelle 1) mit dem folgenden PCR-Programm: 94 °C für 1 min und dann 35 Zyklen von 94 °C für 30 s, 55 °C für 30 s und 72 °C für 1 min, gefolgt von einer 7 min abschließenden Verlängerung bei 72 °C.
HINWEIS: Der ITS1F/ITS4R-Primersatz dient zur Identifizierung auf Gattungsebene. - Sequenzieren Sie die PCR nach kommerziellem Sequenzierungsdienst.
- Verwenden Sie die NCBI BLAST-Suche nach ähnlichen Pilzen in der NCBI-Datenbank und wählen Sie die relative Pilztypart für die phylogenetische Analyse aus.
ANMERKUNG: Die Pilzarten der Gattung Metarhizium oder Beauveria sollten mit tef-983F/tef-2218R primer set25 weiter auf Artebene identifiziert werden (Wiederholungsschritte 2.1.1 bis 2.2.3). Für Pilze, die nicht zu den Gattungen Metarhizium oder Beauveria gehören, können andere molekulare Marker zur Identifizierung der Spezies verwendet werden, einschließlich DNA-Lyase (APN2), Beta-Tubulin (BTUB), RNA-Polymerase II größte Untereinheit (RPB1), RNA-Polymerase II zweitgrößte Untereinheit (RPB2) und 3'-Anteil des Translationsdehnungsfaktors 1 alpha (TEF)25,26 .
- Amplifizieren Sie die Fungal ITS-Region durch PCR der DNA-Probe21 mit dem PCR Master Mix (2x), ITS1F/ITS4R Primer Set24 (Tabelle 1) mit dem folgenden PCR-Programm: 94 °C für 1 min und dann 35 Zyklen von 94 °C für 30 s, 55 °C für 30 s und 72 °C für 1 min, gefolgt von einer 7 min abschließenden Verlängerung bei 72 °C.
- Phylogenetische Analyse
- Verwenden Sie ClustalX 2.1 software27 , um die verschiedenen Sequenzen aus den Schritten 2.2.2 und 2.2.3 auszurichten. Schneiden Sie den konservierten Sequenzbereich manuell mit GeneDoc28 zu.
- Führen Sie die phylogenetische Analyse mit MEGA7 software29 basierend auf den Methoden Minimale Evolution (ME), Neighbor-Joining (NJ) und Maximum Likelihood (ML) durch.
HINWEIS: Die Durchführung aller drei Methoden kann helfen, den Klassifizierungsstatus zu bestätigen und genau abzuschließen. Die pilzlichen Isolate, die durch den 1. Pathogenitätsscreening gescreent werden, werden zur molekularen Identifizierung auf Gattungsebene verwendet. Die durch den 2. Virulenztest gescreenten Pilzisolate werden zur molekularen und morphologischen Identifizierung auf Speziesebene verwendet.
3. Morphologische Identifizierung von EPF
- Beobachtung der Morphologie der Pilzkolonie
- Verwenden Sie eine Kamera, um das Wachstum der Pilzkulturkolonie für 7 Tage zu erfassen, und zeichnen Sie das Wachstum, die Form (flauschig, fest) und die Farbe der Kolonien auf.
- Beobachtung von Konidien und Konidiophoren
- Kratzen Sie Konidien aus der Reinkultur-Pilzkolonie mit einer Impfschleife und übertragen Sie die Sporen auf einen Glasobjektträger mit 0,1% Tween 80-Lösung. Dann mit einem Deckglas für die lichtmikroskopische Beobachtung von Konidien abdecken.
- Verwenden Sie ein Skalpell, um einen 5 mm2 großen Agarblock des Hyphenstrangs am Rand der Pilzkolonie zu schneiden, und übertragen Sie den Agarblock dann auf einen Glasträger.
- Führen Sie die Reinigung wie folgt durch: Fügen Sie die 0,1% Tween 80-Lösung mit einer Kunststofftropfer auf den Agarblock und waschen Sie die meisten überschüssigen Konidien mit einer Pinzette ab. Dann bedecken Sie es mit einem Deckglas für die lichtmikroskopische Beobachtung.
HINWEIS: 0,1% Tween 80 können je nach Pilzart und Hydrophobie durch ein anderes stärkeres Tensid (Materialtabelle) ersetzt werden. - Messen und notieren Sie die Breite und Länge der Konidien und Konidiophoren, um die Unterschiede zwischen verschiedenen Pilzisolaten zu vergleichen.
- Verwenden Sie den Welch's ANOVA-Test und den Games-Howell-Test (Post-hoc-Test), um die Konidialbreite und -länge jedes Stammes mit R Studio (Version 3.4.1) zu analysieren.
HINWEIS: Die Datenanalyse morphologischer Merkmale kann fallweise angepasst werden. Die mit dem 3. Virulenztest gescreenten Pilzisolate werden für die physiologische Charakterisierung und das ECN-Ranking in den Abschnitten 4 und 5 verwendet.
4. Untersuchung der konidialen Produktivität und Thermotoleranz
- Assay der konidialen Produktion
- Kultur des ausgewählten Pilzisolats auf 1/4 SDA-Medium bei 25 ± 1 °C im Dunkeln für 10 Tage.
- 1 mL der konidialen Suspension des Pilzisolats in 0,03%iger Tensidlösung zubereiten und wie oben beschrieben auf 1 x 107 Konidien/ml einstellen.
- Drei Tröpfchen von 10 μL konidialer Suspension auf 1/4 SDA fallen lassen und bei 25 °C im Dunkeln für 7, 10 und 14 Tage inkubieren, um die Sporulation von Pilzen zu zählen.
HINWEIS: 10 μL ist das beste Volumen, um den 5 mm Block mit gleichmäßiger Pilzsporulation nach Pilzwachstum für 7-14 Tage zu sammeln. - Verwenden Sie den Korkbohrer, um den 5 mm Agarblock von der Mitte der Kolonie zu lösen und zu jedem Zeitpunkt in 1 ml 0,03% ige Tensidlösung (Materialtabelle) in einem 1,5 ml Mikrozentrifugenröhrchen zu überführen.
- Wirbeln Sie das Rohr bei 3.000 U / min bei Raumtemperatur für 15 min und verwenden Sie ein Hämozytometer, um die Anzahl der Konidien zu zählen.
HINWEIS: Die für die Zählung verwendete Formel ist die Anzahl der Konidien pro 25 Quadrate der kleinsten Zelle (Größe = 0,025 mm2; Kammertiefe = 0,1 mm):
Gesamtzahl der Konidien in 5 Quadraten ÷ 80 × (4 × 106) - Wiederholen Sie dies dreimal für jedes Isolat.
- Thermotoleranz-Assay
- Kultur des ausgewählten Pilzisolats auf 1/4 SDA-Medium bei 25 ± 1 °C im Dunkeln für 10 Tage.
- 1 ml der konidialen Suspension von Pilzisolat in 0,03%iger Tensidlösung herstellen und wie oben beschrieben auf 1 x 107 Konidien/ml einstellen.
- Die Konidialsuspension aufwirbeln und in einem 45 °C trockenen Bad für 0, 30, 60, 90 und 120 min erhitzen. Drei Tröpfchen von 5 μL der Konidialsuspension zu jedem Zeitpunkt nach der Hitzeexposition auf 55 mm 1/4 SDA-Medium fallen lassen und bei 25 ± 1 °C für 18 h inkubieren.
HINWEIS: Vermeiden Sie es, die Pilztröpfchen zu verteilen, um sich besser auf den Bereich konzentrieren zu können. - Zählen Sie die Anzahl der gekeimten Konidiensporen mit fünf zufällig ausgewählten Feldern unter 200-facher Lichtmikroskopie, um die Keimrate zu bestimmen.
- Führen Sie drei Replikate für jedes Isolat durch.
5. Effektives ECN-Ranking (Conidia Number)
- ECN-Berechnung
ANMERKUNG: Erhalten Sie die Konidialproduktions- und Thermotoleranzdaten jedes potenziellen Pilzstamms, bevor Sie den Gesamt-ECN21 berechnen.- Berechnen Sie den Faltenwechsel (FC) der konidialen Produktion zu jedem Zeitpunkt:
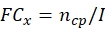
wobei x = Zeitpunkt für die Datenerhebung ist; ncp = Anzahl der Konidien nach jedem Wachstumstag; und I = Anfangszahl der gesetzten Konidien. - Berechnen Sie die Konidienzahl unter der Stressbehandlung zu jedem Zeitpunkt mit der folgenden Formel:

Dabei ist y = die ECN des zu behandelnden Zeitpunkts; TT0 = Keimrate von Konidien, die keiner Hitzebelastung ausgesetzt sind (= Keimrate von 0 min Wärmebehandlung); TTz = Spannungskoeffizient ist die Keimrate der Konidien zu verschiedenen Zeiten der Wärmebehandlung (z). - Berechnen Sie den Gesamt-ECN mit der folgenden Formel:

- Vergleichen Sie den ECN jedes Pilzstamms.
- Berechnen Sie den Faltenwechsel (FC) der konidialen Produktion zu jedem Zeitpunkt:
- Hauptkomponentenanalyse (PCA) von Pilzstämmen
HINWEIS:Die PCA-Analyse bestätigt das Ranking von ECN und hilft beim Verständnis der Korrelation zwischen den physiologischen Charakterwerten. Vergleichen Sie die ECN-Werte und wählen Sie EPF-Isolate mit höheren ECN-Werten aus. - Verwenden Sie die R-Software, um PCA durch Codierung zu erstellen:
#Input PCA-Datendatei
a = read.table("PCA.csv",sep=',',header=T)- # Verarbeitung von Beispieldaten
row.names(a) <- c("NCHU-9","NCHU-11", "NCHU-64", "NCHU-69", "NCHU-95", "NCHU-113")
X=Zeilennamen(a)
df<- a[2:11] - #PCA Berechnung
pca <- prcomp(df, center = TRUE, scale = TRUE)
vars <- (pca$sdev)^2
pc1_percent = vars[1] / sum(vars)
pc2_percent = vars[2] / sum(vars)
Wert = pca$x - #Output PCA-Visualisierungsdatei
png(datei = 'pca.png', höhe = 2000, breite = 2000, res = 300)
HINWEIS: Verwenden Sie 7 bis 14 Tage konidiale Produktion und alle Thermotoleranzdaten, um eine Hauptkomponentenanalyse (PCA) zur Bestätigung des ECN-Rankings durchzuführen.
- # Verarbeitung von Beispieldaten
- Wählen Sie die leistungsstärksten Pilzstämme basierend auf ECN oder PCA aus und führen Sie den Virulenztest von Zielschädlingen für weitere Forschungen durch.
Ergebnisse
Isolierung und Selektion potentieller entomopathogener Pilze (EPF)
Durch die Verwendung der Tenebrio molitor-vermittelten entomopathogenen Pilze (EPF) Bibliotheksbauweise würde die Anzahl der Pilze ohne insektentötende Aktivität ausgeschlossen; somit könnte die Isolationseffizienz und selektion von EPF stark erhöht werden. Bei der Anwendung dieser Methode wurden die Informationen über Probenahmestellen, Bodenproben und die Pilzkeimungsraten erfasst (
Diskussion
Entomopathogene Pilze (EPF) wurden zur Insektenbekämpfung eingesetzt. Es gibt mehrere Methoden zum Isolieren, Auswählen und Identifizieren von EPF30,31,32. Im Vergleich der verschiedenen Arten von Insektenködermethoden wurden Beauveria bassiana und Metarhizium anisopliae häufig in Insektenködern gefunden6,12,13,14.
Offenlegungen
Die Autoren erklären, dass in dieser Arbeit kein Interessenkonflikt besteht.
Danksagungen
Diese Forschung wurde durch Grant 109-2313-B-005-048-MY3 des Ministeriums für Wissenschaft und Technologie (MOST) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Agar Bacteriological grade | BIOMAN SCIENTIFIC Co., Ltd. | AGR001 | Suitable in most cell culture/molecular, biology applications. |
| AGAROSE, Biotechnology Grade | BIOMAN SCIENTIFIC Co., Ltd. | AGA001 | For DNA electrophoresis. |
| BioGreen Safe DNA Gel Buffer | BIOMAN | SDB001T | |
| Brass cork borer | Dogger | D89A-44001 | |
| Canon kiss x2 | Canon | EOS 450D | For record strain colony morphology |
| Constant temperature incubator | Yihder Co., Ltd. | LE-509RD | Fungal keeping. |
| cubee Mini-Centrifuge | GeneReach | MC-CUBEE | |
| DigiGel 10 Digital Gel Image System | TOPBIO | DGIS-12S | |
| Finnpipette F2 0.2 to 2 µL Pipette | Thermo Scientific | 4642010 | |
| Finnpipette F2 1 to 10 µL Pipette | Thermo Scientific | 4642030 | |
| Finnpipette F2 10 to 100 µL Pipette | Thermo Scientific | 4642070 | |
| Finnpipette F2 100 to 1000 µL Pipette | Thermo Scientific | 4642090 | |
| Finnpipette F2 2 to 20 µL Pipette | Thermo Scientific | 4642060 | |
| Finnpipette F2 20 to 200 µL Pipette | Thermo Scientific | 4642080 | |
| GeneAmp PCR System 9700 | Applied Biosystems | 4342718 | |
| GenepHlow Gel/PCR Kit | Geneaid | DFH100 | |
| Genius Dry Bath Incubator | Major Science | MD-01N | |
| Graduated Cylinder Custom A 100mL | SIBATA | SABP-1195906 | Measure the volume of reagents. |
| Hand tally counter | SDI | NO.1055 | |
| Hemocytometer | bioman | AP-0650010 | Calculate the number of spore |
| Inoculating loop | Dogger | D8GA-23000 | |
| lid | IDEAHOUSE | RS92004 | |
| Micro cover glass | MUTO PURE CHEMICALS CO.,LTD | 24241 | |
| Microscope imaging system | SAGE VISION CO.,LTD | SGHD-3.6C | |
| Microscope Slides | DOGGER | DG75001-07105 | |
| Mupid-2plus DNA Gel Electrophoresis | ADVANCE | AD110 | |
| Nikon optical microscope | SAGE VISION CO.,LTD | Eclipse CI-L | |
| Plastic cup | IDEAHOUSE | CS60016 | |
| Presto Mini gDNA Yeast Kit | Geneaid | GYBY300 | Fungal genomic DNA extraction kit |
| Sabouraud Dextrose Broth (Sabouraud Liquid Medium) | HiMedia Leading BioSciences Company | M033 | Used for cultivation of yeasts, moulds and aciduric microorganisms. |
| Scalpel Blade No.23 | Swann-Morton | 310 | |
| Scalpel Handle No.4 | AGARWAL SURGICALS | SSS -FOR-01-91 | |
| Shovel | Save & Safe | A -1580242 -00 | |
| Silwet L-77 | bioman(phytotech) | S7777 | Surfactant |
| Sorvall Legend Micro 17 Microcentrifuge | Thermo Scientific | 75002403 | |
| Steel Tweezers | SIPEL ELECTRONIC SA | GG-SA | |
| Sterile Petri Dish | BIOMAN SCIENTIFIC Co., Ltd. | 1621 | Shallow cylindrical containers with fitted lids, specifically for microbiology or cell culture use. |
| ThermoCell MixingBlock | BIOER | MB-101 | |
| Tween 80 | FUJIFILM Wako Pure Chemical Corporation | 164-21775 | |
| TwinGuard ULT Freezer | Panasonic Healthcare Holdings Co., Ltd. | MDF-DU302VX | -80°C sample stored. |
| Vertical floor type cabinet | Chih Chin | BSC-3 | Fungal operating culturing. |
| Vortex Genie II | Scientific | SIG560 | |
| Zipper storage bags | Save & Safe | A -1248915 -00 | |
| 100 bp DNA Ladder | Geneaid | DL007 | |
| -20°C Freezer | FRIGIDAIRE | Frigidaire FFFU21M1QW | -20°C sample and experimental reagents stored. |
| 2X SuperRed PCR Master Mix | TOOLS | TE-SR01 | |
| 50X TAE Buffer | BIOMAN | TAE501000 |
Referenzen
- Wraight, S. P., Carruthers, R. I. . Biopesticides: use and Delivery. , 233-269 (1999).
- Chase, A., Osborne, L., Ferguson, V. Selective isolation of the entomopathogenic fungi Beauveria bassiana and Metarhizium anisopliae from an artificial potting medium. Florida Entomologist. , 285-292 (1986).
- Meyling, N. V. Methods for isolation of entomopathogenic fungi from the soil environment. University of Copenhagen. , 1-18 (2007).
- Zimmermann, G. The 'Galleria bait method'for detection of entomopathogenic fungi in soil. Journal of applied Entomology. 102 (1-5), 213-215 (1986).
- Schneider, S., Widmer, F., Jacot, K., Kölliker, R., Enkerli, J. Spatial distribution of Metarhizium clade 1 in agricultural landscapes with arable land and different semi-natural habitats. Applied Soil Ecology. 52, 20-28 (2012).
- Hallouti, A., et al. Diversity of entomopathogenic fungi associated with Mediterranean fruit fly (Ceratitis capitata (Diptera: Tephritidae)) in Moroccan Argan forests and nearby area: impact of soil factors on their distribution. BMC Ecology. 20 (1), 1-13 (2020).
- Meyling, N. V., Eilenberg, J. Occurrence and distribution of soil borne entomopathogenic fungi within a single organic agroecosystem. Agriculture, Ecosystems and Environment. 113 (1-4), 336-341 (2006).
- Skalický, A., Bohatá, A., Šimková, J., Osborne, L. S., Landa, Z. Selection of indigenous isolates of entomopathogenic soil fungus Metarhizium anisopliae under laboratory conditions. Folia Microbiologica. 59 (4), 269-276 (2014).
- Veen, K., Ferron, P. A selective medium for the isolation of Beauveria tenella and of Metarrhizium anisopliae. Journal of Invertebrate Pathology. 8 (2), 268-269 (1966).
- Goettel, M., Inglis, D., Lacy, L. . Manual of Techniques in Insect Pathology. , 213-249 (1997).
- Luz, C., Netto, M. C. B., Rocha, L. F. N. In vitro susceptibility to fungicides by invertebrate-pathogenic and saprobic fungi. Mycopathologia. 164 (1), 39-47 (2007).
- Mantzoukas, S., et al. Trapping entomopathogenic fungi from vine terroir soil samples with insect baits for controlling serious pests. Applied Sciences. 10 (10), 3539 (2020).
- Goble, T., Dames, J., Hill, M., Moore, S. The effects of farming system, habitat type and bait type on the isolation of entomopathogenic fungi from citrus soils in the Eastern Cape Province, South Africa. BioControl. 55 (3), 399-412 (2010).
- Nishi, O., Iiyama, K., Yasunaga-Aoki, C., Shimizu, S. Isolation of entomopathogenic fungi from soil by using bait method with termite, Reticulitermes speratus. Enotomotech. 35, 21-26 (2011).
- Castrillo, L. . ARS Collection of Entomopathogenic Fungal Cultures (ARSEF). , (2014).
- Kim, J. C., et al. Tenebrio molitor-mediated entomopathogenic fungal library construction for pest management. Journal of Asia-Pacific Entomology. 21 (1), 196-204 (2018).
- Keyser, C. A., Henrik, H., Steinwender, B. M., Meyling, N. V. Diversity within the entomopathogenic fungal species Metarhizium flavoviride associated with agricultural crops in Denmark. BMC Microbiology. 15 (1), 1-11 (2015).
- Quesada-Moraga, E., Navas-Cortés, J. A., Maranhao, E. A., Ortiz-Urquiza, A., Santiago-Álvarez, C. Factors affecting the occurrence and distribution of entomopathogenic fungi in natural and cultivated soils. Mycological Research. 111 (8), 947-966 (2007).
- Park, J. B., et al. Developmental characteristics of Tenebrio molitor larvae (Coleoptera: Tenebrionidae) in different instars. International Journal of Industrial Entomology. 28 (1), 5-9 (2014).
- Chang, J. -. C., et al. Construction and selection of an entomopathogenic fungal library from soil samples for controlling Spodoptera litura. Frontiers in Sustainable Food Systems. 5, 15 (2021).
- Podder, D., Ghosh, S. K. A new application of Trichoderma asperellum as an anopheline larvicide for eco friendly management in medical science. Scientific reports. 9 (1), 1-15 (2019).
- . Geneaid Biotech Ltd. Presto Mini gDNA Yeast, Ver. 04.27.17 Available from: https://www.geneaid.com/data/files/1605664221308055331.pdf (2021)
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR protocols: A guide to methods and applications. 18 (1), 315-322 (1990).
- Kepler, R. M., Humber, R. A., Bischoff, J. F., Rehner, S. A. Clarification of generic and species boundaries for Metarhizium and related fungi through multigene phylogenetics. Mycologia. 106 (4), 811-829 (2014).
- Kepler, R. M. A phylogenetically-based nomenclature for Cordycipitaceae (Hypocreales). IMA Fungus. 8 (2), 335-353 (2017).
- Thompson, J. D., Gibson, T. J., Plewniak, F., Jeanmougin, F., Higgins, D. G. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research. 25 (24), 4876-4882 (1997).
- Kumar, S., Stecher, G., Tamura, K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution. 33 (7), 1870-1874 (2016).
- Herlinda, S., Mulyati, S. I. Selection of isolates of entomopathogenic fungi and the bioefficacy of their liquid production against Leptocorisa oratorius nymphs. Microbiology Indonesia. 2 (3), 9 (2008).
- Herlinda, S., Irsan, C., Mayasari, R., Septariani, S. Identification and selection of entomopathogenic fungi as biocontrol agents for Aphis gossypii from South Sumatra. Microbiology Indonesia. 4 (3), 137-142 (2010).
- Montes-Bazurto, L. G., Peteche-Yonda, Y., Medina-Cardenas, H. C., Bustillo-Pardey, A. E. Selection of entomopathogenic fungi for the biological control of Demotispa neivai (Coleoptera: Chrysomelidae) in oil palm plantations in Colombia. Journal of Entomological Science. 55 (3), 388-404 (2020).
- Shin, T. -. Y., Choi, J. -. B., Bae, S. -. M., Koo, H. -. N., Woo, S. -. D. Study on selective media for isolation of entomopathogenic fungi. International Journal of Industrial Entomology. 20 (1), 7-12 (2010).
- Sharma, L., Oliveira, I., Torres, L., Marques, G. Entomopathogenic fungi in Portuguese vineyards soils: Suggesting a 'Galleria-Tenebrio-bait method'as bait-insects Galleria and Tenebrio significantly underestimate the respective recoveries of Metarhizium (robertsii) and Beauveria (bassiana). MycoKeys. (38), 1 (2018).
- Rodríguez, M., Gerding, M., France, A. Selección de Hongos Entomopatógenos para el Control de Varroa destructor (Acari: Varroidae). Chilean journal of agricultural research. 69 (4), 534-540 (2009).
- Yang, H., et al. Persistence of Metarhizium (Hypocreales: Clavicipitaceae) and Beauveria bassiana (Hypocreales: Clavicipitaceae) in tobacco soils and potential as biocontrol agents of Spodoptera litura (Lepidoptera: Noctuidae). Environmental entomology. 48 (1), 147-155 (2019).
- Muñiz-Reyes, E., Guzmán-Franco, A. W., Sánchez-Escudero, J., Nieto-Angel, R. Occurrence of entomopathogenic fungi in tejocote (C rataegus mexicana) orchard soils and their pathogenicity against R hagoletis pomonella. Journal of Applied Microbiology. 117 (5), 1450-1462 (2014).
- Lacey, L. A., et al. Goettel Insect pathogens as biological control agents: Back to the future. Journal of Invertebrate Pathology. 132, 1-41 (2015).
- Humber, R. A. . Manual of techniques in insect pathology. , 153-185 (1997).
- Rehner, S. A., Buckley, E. A Beauveria phylogeny inferred from nuclear ITS and EF1-α sequences: evidence for cryptic diversification and links to Cordyceps teleomorphs. Mycologia. 97 (1), 84-98 (2005).
- Quandt, C. A., et al. Phylogenetic-based nomenclatural proposals for Ophiocordycipitaceae (Hypocreales) with new combinations in Tolypocladium. IMA fungus. 5 (1), 121-134 (2014).
- Shah, F. A., Wang, C. S., Butt, T. M. Nutrition influences growth and virulence of the insect-pathogenic fungus Metarhizium anisopliae. FEMS Microbiology Letters. 251 (2), 259-266 (2005).
- Ignoffo, C. Environmental factors affecting persistence of entomopathogens. Florida Entomologist. , 516-525 (1992).
- Rodrigues, I. W., Forim, M., Da Silva, M., Fernandes, J., Batista Filho, A. Effect of ultraviolet radiation on fungi Beauveria bassiana and Metarhizium anisopliae, pure and encapsulated, and bio-insecticide action on Diatraea saccharalis. Advances in Entomology. 4 (3), 151-162 (2016).
- Paula, A. R., Ribeiro, A., Lemos, F. J. A., Silva, C. P., Samuels, R. I. Neem oil increases the persistence of the entomopathogenic fungus Metarhizium anisopliae for the control of Aedes aegypti (Diptera: Culicidae) larvae. Parasites and Vectors. 12 (1), 1-9 (2019).
- Morley-Davies, J., Moore, D., Prior, C. Screening of Metarhizium and Beauveria spp. conidia with exposure to simulated sunlight and a range of temperatures. Mycological Research. 100 (1), 31-38 (1996).
- Rangel, D. E., Braga, G. U., Flint, S. D., Anderson, A. J., Roberts, D. W. Variations in UV-B tolerance and germination speed of Metarhizium anisopliae conidia produced on insects and artificial substrates. Journal of Invertebrate Pathology. 87 (2-3), 77-83 (2004).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten