È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Isolamento e selezione di funghi entomopatogeni da campioni di suolo e valutazione della virulenza fungina contro gli insetti nocivi
In questo articolo
Riepilogo
Qui presentiamo un protocollo basato sul sistema di esca del verme della farina (Tenebrio molitor) che è stato utilizzato per isolare e selezionare funghi entomopatogeni (EPF) da campioni di suolo. Una formula efficace del numero di conidi (ECN) viene utilizzata per selezionare EPF ad alta tolleranza allo stress in base alle caratteristiche fisiologiche per il controllo microbico dei parassiti sul campo.
Abstract
I funghi entomopatogeni (EPF) sono uno degli agenti di controllo microbico per la gestione integrata dei parassiti. Per controllare i parassiti locali o invasivi, è importante isolare e selezionare l'EPF indigeno. Pertanto, il metodo dell'esca del suolo combinato con il sistema di esca per insetti (verme della farina, Tenebrio molitor) è stato utilizzato in questo studio con alcune modifiche. L'EPF isolato è stato quindi sottoposto al test di virulenza contro il parassita agricolo Spodoptera litura. Inoltre, i potenziali ceppi di EPF sono stati sottoposti a identificazioni morfologiche e molecolari. Inoltre, sono stati eseguiti i test di produzione e termotolleranza dei conidi per i promettenti ceppi di EPF e confrontati; questi dati sono stati ulteriormente sostituiti nella formula del numero di conidi effettivi (ECN) per la classificazione di laboratorio. Il sistema esche-vermi della farina del suolo e la formula ECN possono essere migliorati sostituendo le specie di insetti e integrando più fattori di stress per la valutazione della commercializzazione e dell'applicazione sul campo. Questo protocollo fornisce un approccio rapido ed efficiente per la selezione dell'EPF e migliorerà la ricerca sugli agenti di controllo biologico.
Introduzione
Attualmente, i funghi entomopatogeni (EPF) sono ampiamente utilizzati nel controllo microbico di parassiti agricoli, forestali e orticoli. I vantaggi dell'EPF sono le sue ampie gamme di ospiti, la buona adattabilità ambientale, la natura ecocompatibile e il fatto che possa essere utilizzato con altre sostanze chimiche per mostrare l'effetto sinergico per la gestione integrata dei parassiti1,2. Per l'applicazione come agente di controllo dei parassiti, è necessario isolare un gran numero di EPF da insetti malati o dall'ambiente naturale.
Il campionamento di questi organismi dai loro ospiti aiuta a comprendere la distribuzione geografica e il tasso di prevalenza dell'EPF negli ospiti naturali3,4,5. Tuttavia, la raccolta di insetti infetti da funghi è solitamente limitata da fattori ambientali e popolazioni di insetti nel campo4. Considerando che gli ospiti degli insetti moriranno dopo l'infezione da EPF e poi cadranno nel terreno, l'isolamento dell'EPF dai campioni di suolo potrebbe essere una risorsa stabile3,6. Ad esempio, i saprofiti sono noti per utilizzare l'ospite morto come risorsa per la crescita. L'esca del suolo e i sistemi di mezzo selettivo sono stati ampiamente utilizzati per rilevare e isolare l'EPF dal suolo3,4,7,8,9,10.
Nel metodo del mezzo selettivo, la soluzione di terreno diluito viene placcata su un mezzo contenente antibiotici ad ampio spettro (ad esempio, cloramfenicolo, tetraciclina o streptomicina) per inibire la crescita dei batteri2,3,9,11. Tuttavia, è stato riportato che questo metodo può distorcere la diversità e la densità del ceppo e può causare una sovrastima o sottostima di molte comunità microbiche6. Inoltre, i ceppi isolati sono meno patogeni e competono con i saprofiti durante l'isolamento. È difficile isolare l'EPF dalla soluzione di terreno diluito3. Invece di utilizzare un mezzo selettivo, il metodo dell'esca del suolo isola l'EPF dagli insetti morti infetti, che possono essere conservati per 2-3 settimane, fornendo così un metodo di separazione EPF più efficiente e standard3,4,7,6. Poiché il metodo è facile da usare, è possibile isolare una varietà di ceppi patogeni a basso costo4. Pertanto, è ampiamente utilizzato da molti ricercatori.
Confrontando i diversi tipi di sistemi di esche per insetti, Beauveria bassiana e Metarhizium anisopliae sono le specie di EPF più comuni che si trovano negli insetti appartenenti agli Hemiptera, Lepidoptera, Blattella e Coleoptera6,12,13,14. Tra queste esche per insetti, Galleria mellonella (ordine Lepidoptera) e Tenebrio molitor (ordine Coleoptera) mostrano tassi di recupero più elevati di Beauveria e Metarhizium spp., rispetto ad altri insetti. Pertanto, G. mellonella e T. molitor sono comunemente usati per l'esca degli insetti. Nel corso degli anni, il Dipartimento dell'Agricoltura degli Stati Uniti (USDA) ha istituito una biblioteca EPF (Agricultural Research Service Collection of EPF cultures, ARSEF) che contiene un'ampia varietà di specie, tra cui 4081 specie di Beauveria spp., 18 specie di Clonostachys spp., 878 specie di Cordyceps spp., 2473 specie di Metarhizium spp., 226 specie di Purpureocillium spp., e 13 specie di Pochonia spp. tra gli altri15. Un'altra biblioteca EPF è stata costruita dall'Entomology Research Laboratory (ERL) dell'Università del Vermont negli Stati Uniti per circa 30 anni. Comprende 1345 ceppi di EPF provenienti da Stati Uniti, Europa, Asia, Africa e Medio Oriente16.
Per controllare i parassiti locali o di invasione a Taiwan, è necessario l'isolamento e la selezione dell'EPF indigeno. Pertanto, in questo protocollo, abbiamo modificato e descritto la procedura del metodo dell'esca del suolo e l'abbiamo combinata con il sistema di esca per insetti (verme della farina, Tenebrio molitor)17. Sulla base di questo protocollo, è stata istituita una libreria EPF. Sono stati eseguiti due cicli di screening (quantificazione dell'inoculazione) per gli isolati preliminari di EPF. Gli isolati di EPF hanno mostrato patogenicità agli insetti. I ceppi potenziali sono stati sottoposti a identificazioni morfologiche e molecolari e ulteriormente analizzati mediante il test di termotolleranza e produzione conidiale. Inoltre, è stato proposto anche un concetto di numero di conidi effettivi (ECN). Utilizzando la formula ECN e l'analisi dei componenti principali (PCA), i ceppi potenziali sono stati analizzati sotto pressione ambientale simulata per completare il processo di creazione e screening della libreria EPF. Successivamente, la patogenicità di ceppi di EPF promettenti è stata testata per il parassita bersaglio (ad esempio, Spodoptera litura). L'attuale protocollo integra i dati di termotoleranza e produzione conidiale nella formula ECN e nell'analisi PCA, che può essere utilizzata come sistema di classificazione standard per la ricerca relativa all'EPF.
Access restricted. Please log in or start a trial to view this content.
Protocollo
NOTA: l'intero diagramma di flusso è illustrato nella Figura 1.
1. Isolamento e selezione di potenziali funghi entomopatogeni (EPF)
- Raccogli il campione di terreno
- Rimuovere 1 cm del terreno superficiale e quindi raccogliere il terreno entro la profondità di 5-10 cm utilizzando una pala da ciascun sito di campionamento.
NOTA: i siti di campionamento sarebbero una montagna, una foresta o aree scarsamente popolate per evitare la contaminazione di ceppi di EPF spruzzati artificialmente. Assicurarsi che le aree per la raccolta dei campioni di terreno siano coperte con erba sulla superficie. Il terreno asciutto o il terreno umido non sono adatti per questo esperimento. - Registrare i dettagli di ciascun sito di campionamento, inclusi GPS, elevazione, tipo di campo, temperatura annuale, precipitazioni annuali, tempo di raccolta (stagione), tipo di suolo e valore del pH.
- Raccogliere 100 g del campione di terreno in un sacchetto di plastica e mantenerlo a temperatura ambiente se sottoposto al protocollo di isolamento fungino in laboratorio entro 3 ore.
NOTA: Se il campione non può essere utilizzato entro 3 ore, conservare il terreno a temperatura ambiente in condizioni di oscurità. Se l'esperimento non viene eseguito immediatamente, il campione di terreno può essere conservato a 4 °C per 1 settimana fino all'inizio del protocollo18,19.
- Rimuovere 1 cm del terreno superficiale e quindi raccogliere il terreno entro la profondità di 5-10 cm utilizzando una pala da ciascun sito di campionamento.
- Esca e isolare l'EPF con il verme della farina (Tenebrio molitor)
- Posizionare 100 g del campione di terreno in un bicchiere di plastica (diametro del cappuccio = 8,5 cm, altezza = 12,5 cm), quindi posizionare 5 vermi della farina sulla superficie del terreno a temperatura ambiente al buio per 2 settimane.
NOTA: è possibile utilizzare anche altri tipi di contenitori per bicchieri di plastica. Se i terreni sono troppo secchi (screpolati o sabbiosi), spruzzare ddH2O sterilizzato a spruzzo (circa 5-10 ml) sui terreni. La lunghezza del corpo c.a. 2,5 cm (14 ° instar) delle larve di T. molitor aiuta negli schermi di isolamento fungino20. - Osservare e registrare quotidianamente le larve per mortalità e micosi; tenere le larve morte nella tazza fino a 2 settimane per l'isolamento fungino.
NOTA: La spora di conidi fungini nei campioni di terreno si attaccherà alle larve di verme della farina durante il processo di cui sopra. La micosi fungina sarà osservata mentre le ife crescono dalla membrana intersegmentale, e quindi l'intero corpo sarà coperto dal micelio. La sporulazione inizierà dopo 7 giorni e il colore dell'infezione fungina cambierà nel colore della massa dei conidi. - Trasferire gli insetti morti in una panca pulita e utilizzare uno stuzzicadenti sterile per raccogliere i conidi. Strisciarli su un quarto di forza Sabouraud destrosio agar medio (1/4 SDA) piastra (55 mm) in laboratorio21. Incubare la piastra di coltura a 25 °C per 7 giorni per ottenere la coltura primaria di funghi.
NOTA: La piastra 1/4 SDA viene preparata come segue: Mescolare 1,5 g di brodo di destrosio Sabouraud e 3 g di agar in 200 ml di H2O, quindi sterilizzare per 20 minuti. Aliquota 1/4 SDA in ogni capsula di Petri da 55 mm prima della solidificazione. Le piastre solidificate 1/4 SDA vengono conservate a 4 °C fino all'uso. - Ri-strisciare ogni fungo di coltura primaria su una piastra SDA da 55 mm 1/4 in un flusso laminare e incubare la piastra di coltura a 25 °C per 7 giorni per ottenere singole colonie di funghi.
- Ripetere questo re-isolamento ~ 2-3 volte e osservare al microscopio ottico per ottenere colonie fungine morfologiche singole e pure.
NOTA: Isolare tutto l'EPF utilizzando T. molitor-bait e conservare come descritto nella sezione seguente. La separazione, la conservazione, la coltura pura e la striatura devono essere eseguite in un flusso laminare nelle sezioni successive.
- Posizionare 100 g del campione di terreno in un bicchiere di plastica (diametro del cappuccio = 8,5 cm, altezza = 12,5 cm), quindi posizionare 5 vermi della farina sulla superficie del terreno a temperatura ambiente al buio per 2 settimane.
- Conservare gli isolati EPF
- Tagliare 3 dei blocchi di agar da 5 mm sul bordo di ogni piastra isolata di coltura fungina pura con una piralide di sughero e posizionarla in un tubo di microcentrifuga da 1,5 ml come una replica.
NOTA: Tre repliche per ogni isolato fungino sono raccomandate per la conservazione dei ceppi EPF dopo il 2 ° test di virulenza. L'identificazione molecolare è raccomandata anche se lo spazio di archiviazione è limitato. - Aggiungere 250 μL di soluzione di tensioattivo allo 0,03% (Tabella dei materiali) e 250 μL di glicerolo al 60% nel tubo della microcentrghina da 1,5 mL utilizzando una micropipetta; quindi, vortice per 10 s.
- Sigillare il tubo della microcentrghina da 1,5 ml con film di paraffina e preraffreddarlo in un frigorifero a -20 °C per 24 ore. Quindi, trasferire le scorte fungine preraffreddate in un frigorifero a -80 °C per la crioconservazione.
NOTA: nel paragrafo 1.4 sono state utilizzate piastre di coltura fungina extra pure (a parte i campioni crioconservati).
- Tagliare 3 dei blocchi di agar da 5 mm sul bordo di ogni piastra isolata di coltura fungina pura con una piralide di sughero e posizionarla in un tubo di microcentrifuga da 1,5 ml come una replica.
- 1° screening di patogenicità per isolati fungini
- Posizionare cinque larve di T. molitor direttamente sulla superficie di ogni piastra di coltura fungina pura a 25 °C.
- Osservare e registrare la micosi e la mortalità per 10 giorni. Selezionare l'isolato fungino per ulteriori analisi.
NOTA: Gli isolati fungini che causano il 100% di mortalità sono selezionati per il 2 ° test di virulenza per confermare la loro virulenza alle larve di T. molitor . In alternativa, il ricercatore può regolare i criteri secondo il proprio studio.
- 2° test di virulenza di isolati fungini
NOTA: Sulla base del 1° screening di patogenicità, recuperare gli isolati fungini selezionati da -80 °C per il 2° test di virulenza. Lo scopo del 2° test di virulenza è quello di quantificare la patogenicità degli isolati fungini selezionati dopo il 1° ciclo di screening.- Raccogli i conidi di ciascun isolato fungino vorticosamente per 1 minuto e conta il numero di conidi usando un emocitometro.
- Regolare la sospensione di conidi ad una concentrazione di 1 x 107 conidi/mL in una soluzione di tensioattivo allo 0,03% (Tabella dei materiali).
- Distribuire 10 μL della sospensione fungina su piastre SDA da 55 mm 1/4 e crescere per 7 giorni a 25 °C al buio.
- Posizionare cinque larve di T. molitor direttamente sulla superficie di ogni piastra di coltura fungina pura (c.a. 6 x 107 conidi). Sigillare le piastre con pellicola di paraffina e incubare a 25 °C al buio.
- Osservare e registrare la micosi e la mortalità per 10 giorni.
- Ripetere il test (dal passo 1.51 al 1.5.5) in triplice copia per ogni isolato fungino.
NOTA: gli isolati fungini che causano il 100% di mortalità sono selezionati per il 3 ° test di virulenza per confermare la loro virulenza per colpire il parassita.
- 3° test di virulenza di isolati fungini per parassita bersaglio (Spodoptera litura come esempio)
- Ripetere i passaggi da 1.5.2 a 1.5.6 con isolati selezionati dalla 2a virulenza per testare la virulenza contro l'organismo nocivo bersaglio.
- Calcola l'LT50 di ogni isolato fungino22.
NOTA: L'LT50 di ciascun isolato fungino è stato calcolato attraverso modelli lineari generalizzati (GLM) utilizzando R studio (versione 3.4.1); la distribuzione dell'errore quasibinomiale e una funzione di collegamento di registro possono essere utilizzate per tenere conto della sovradispersione.
2. Identificazione molecolare dell'EPF
- Estrazione del DNA genomico fungino
- Raccogliere c.a. 1 cm2 EPF dalla piastra SDA 1/4 di 7 giorni.
- Estrarre il DNA genomico fungino utilizzando un kit di estrazione del DNA genomico fungino secondo le istruzioni del produttore23 (Tabella dei materiali).
- Amplificazione PCR e sequenziamento del DNA
- Amplificare la regione ITS fungina mediante PCR del campione di DNA21 utilizzando il PCR Master Mix (2x), il set di primer ITS1F/ITS4R24 (Tabella 1) con il seguente programma PCR: 94 °C per 1 min, e poi 35 cicli di 94 °C per 30 s, 55 °C per 30 s e 72 °C per 1 min, seguiti da un'estensione finale di 7 min a 72 °C.
NOTA: il set di primer ITS1F/ITS4R è destinato all'identificazione a livello di genere. - Sequenziare la PCR per servizio di sequenziamento commerciale.
- Usa NCBI BLAST per cercare funghi simili nel database NCBI e seleziona le relative specie di tipo fungino per l'analisi filogenetica.
NOTA: Le specie fungine appartenenti al genere Metarhizium o Beauveria devono essere ulteriormente identificate a livello di specie con tef-983F/tef-2218R primer set25 (ripetere i passaggi da 2.1.1 a 2.2.3). Per i funghi che non appartengono ai generi Metarhizium o Beauveria, altri marcatori molecolari possono essere utilizzati per identificare la specie, tra cui DNA liasi (APN2), beta tubulina (BTUB), subunità più grande della RNA polimerasi II (RPB1), RNA polimerasi II seconda subunità più grande (RPB2) e 3' porzione di fattore di allungamento della traduzione 1 alfa (TEF)25,26 .
- Amplificare la regione ITS fungina mediante PCR del campione di DNA21 utilizzando il PCR Master Mix (2x), il set di primer ITS1F/ITS4R24 (Tabella 1) con il seguente programma PCR: 94 °C per 1 min, e poi 35 cicli di 94 °C per 30 s, 55 °C per 30 s e 72 °C per 1 min, seguiti da un'estensione finale di 7 min a 72 °C.
- Analisi filogenetica
- Utilizzare il software ClustalX 2.127 per allineare le sequenze multiple dai passaggi 2.2.2 e 2.2.3. Tagliare manualmente la regione delle sequenze conservate con GeneDoc28.
- Eseguire l'analisi filogenetica con il software MEGA729 in base ai metodi di evoluzione minima (ME), Neighbor-Joining (NJ) e massima probabilità (ML).
NOTA: l'esecuzione di tutti e tre i metodi può aiutare a confermare e concludere con precisione lo stato della classificazione. Gli isolati fungini sottoposti a screening dal 1° screening di patogenicità sono utilizzati per l'identificazione molecolare a livello di genere. Gli isolati fungini sottoposti a screening dal 2° test di virulenza sono utilizzati per l'identificazione molecolare e morfologica a livello di specie.
3. Identificazione morfologica dell'EPF
- Osservazione della morfologia della colonia fungina
- Usa una fotocamera per catturare la crescita della colonia di colture fungine per 7 giorni e registrare la crescita, la forma (soffice, soda) e il colore delle colonie.
- Osservazione di conidi e conidiofori
- Raschiare i conidi dalla colonia fungina di coltura pura con un anello di inoculazione e trasferire le spore in un vetrino con soluzione di Tween 80 allo 0,1%. Quindi, coprire con un coverslip per l'osservazione microscopica leggera dei conidi.
- Utilizzare un bisturi per tagliare un blocco di agar di 5 mm2 del filo ifale sul bordo della colonia fungina, quindi trasferire il blocco di agar su un vetrino.
- Eseguire la pulizia come segue: Aggiungere la soluzione 0,1% Tween 80 sul blocco di agar con un contagocce di plastica e lavare via la maggior parte dei conidi in eccesso usando una pinzetta. Quindi, coprilo con un coverslip per l'osservazione microscopica leggera.
NOTA: 0,1% Tween 80 può essere sostituito con un altro tensioattivo più potente (Tabella dei materiali) a seconda delle specie fungine e dell'idrofobicità. - Misurare e registrare la larghezza e la lunghezza dei conidi e dei conidiofori per confrontare le differenze tra i diversi isolati fungini.
- Usa il test ANOVA di Welch e il test Games-Howell (test post-hoc) per analizzare la larghezza e la lunghezza conidiale di ciascun ceppo usando R studio (versione 3.4.1).
NOTA: L'analisi dei dati dei caratteri morfologici può essere regolata per caso. Gli isolati fungini sottoposti a screening con il 3° test di virulenza sono utilizzati per la caratterizzazione fisiologica e la classificazione ECN nelle sezioni 4 e 5.
4. Studio della produttività conidiale e della termotolleranza
- Saggio di produzione conidiale
- Coltivare l'isolato fungino selezionato su 1/4 di terreno SDA a 25 ± 1 °C al buio per 10 giorni.
- Preparare 1 mL della sospensione conidiale dell'isolato fungino in soluzione tensioattiva allo 0,03% e regolare a 1 x 107 conidi/ml come descritto sopra.
- Rilasciare tre goccioline da 10 μL di sospensione conidiale su 1/4 di SDA e incubare a 25 °C al buio per 7, 10 e 14 giorni per contare la sporulazione dei funghi.
NOTA: 10 μL è il volume migliore per raccogliere il blocco di 5 mm con sporulazione fungina anche dopo la crescita fungina per 7-14 giorni. - Utilizzare la piralide del sughero per staccare il blocco di agar da 5 mm dal centro della colonia e trasferirlo in 1 mL di soluzione di tensioattivo allo 0,03% (Tabella dei materiali) in un tubo di microcentrificazione da 1,5 ml in ogni punto temporale.
- Ruotare il tubo a 3.000 giri / min a temperatura ambiente per 15 minuti e utilizzare un emocitometro per contare il numero di conidi.
NOTA: La formula utilizzata per il conteggio è il numero di conidi per 25 quadrati della cella più piccola (dimensione = 0,025 mm2; profondità della camera = 0,1 mm):
Numero totale di conidi in 5 quadrati ÷ 80 × (4 × 106) - Ripetere tre volte per ogni isolato.
- Saggio di termotolleranza
- Coltivare l'isolato fungino selezionato su 1/4 di terreno SDA a 25 ± 1 °C al buio per 10 giorni.
- Preparare 1 mL della sospensione conidiale di isolato fungino in soluzione tensioattiva allo 0,03% e regolare a 1 x 107 conidi/mL come descritto sopra.
- Vortice della sospensione conidiale e riscaldarla in un bagno secco di 45 °C per 0, 30, 60, 90 e 120 min. Far cadere tre goccioline da 5 μL della sospensione conidiale su un mezzo SDA da 55 mm 1/4 in ogni punto temporale dopo l'esposizione al calore e incubare a 25 ± 1 °C per 18 ore.
NOTA: Evitare di diffondere le goccioline fungine per potersi concentrare meglio sull'area. - Contare il numero di spore di conidi germinate con cinque campi selezionati casualmente sotto microscopia ottica 200x per determinare il tasso di germinazione.
- Eseguire tre repliche per ogni isolato.
5. Classificazione del numero di conidi effettivi (ECN)
- Calcolo ECN
NOTA: Ottenere i dati di produzione conidiale e di termotolleranza di ciascun potenziale ceppo fungino prima di calcolare l'ECN21 totale.- Calcola il cambio di piega (FC) della produzione conidiale in ogni punto temporale:
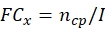
dove, x = punto temporale per la raccolta dei dati; ncp = numero di conidi dopo ogni giorno di crescita; e I = numero iniziale di conidi seminati. - Calcola il numero di conidi sotto il trattamento dello stress in ogni punto temporale usando la seguente formula:

Dove, y = l'ECN del punto temporale in esame; TT0 = il tasso di germinazione dei conidi che non subiscono stress termico (= tasso di germinazione del trattamento termico di 0 minuti); TTz = coefficiente di stress è il tasso di germinazione dei conidi in diversi momenti del trattamento termico (z). - Calcola l'ECN totale utilizzando la seguente formula:

- Confrontare l'ECN di ciascun ceppo fungino.
- Calcola il cambio di piega (FC) della produzione conidiale in ogni punto temporale:
- Analisi dei componenti principali (PCA) di ceppi fungini
NOTA: L'analisi PCA conferma la classifica dell'ECN e aiuta a comprendere la correlazione tra i valori fisiologici del carattere. Confrontare i valori ECN e selezionare gli isolati EPF con valori ECN più elevati. - Utilizzare il software R per creare PCA codificando:
#Input file di dati PCA
a = read.table("PCA.csv",sep=',',header=T)- # Elaborazione dei dati di esempio
row.names(a) <- c("NCHU-9","NCHU-11", "NCHU-64", "NCHU-69", "NCHU-95", "NCHU-113")
X=row.names(a)
df<- a[2:11] - #PCA calcolo
pca <- prcomp(df, center = TRUE, scale = TRUE)
vars <- (pca$sdev)^2
pc1_percent = vars[1] / sum(vars)
pc2_percent = vars[2] / sum(vars)
valore = pca$x - #Output file di visualizzazione PCA
png(file = 'pca.png', altezza = 2000, larghezza = 2000, res = 300)
NOTA: utilizzare una produzione conidiale da 7 a 14 giorni e tutti i dati di termotolleranza per eseguire l'analisi dei componenti principali (PCA) per confermare la classifica ECN.
- # Elaborazione dei dati di esempio
- Seleziona i ceppi fungini più performanti basati su ECN o PCA ed esegui il test di virulenza dei parassiti bersaglio per ulteriori ricerche.
Access restricted. Please log in or start a trial to view this content.
Risultati
Isolamento e selezione di potenziali funghi entomopatogeni (EPF)
Utilizzando il metodo di costruzione della libreria di funghi entomopatogeni mediati da Tenebrio molitor (EPF), il numero di funghi senza attività di uccisione degli insetti sarebbe escluso; pertanto, l'efficienza di isolamento e la selezione dell'EPF potrebbero essere ampiamente aumentate. Durante l'applicazione di questo metodo, sono state registrate le informazioni dei siti di campionamento,...
Access restricted. Please log in or start a trial to view this content.
Discussione
I funghi entomopatogeni (EPF) sono stati utilizzati per il controllo degli insetti. Esistono diversi metodi per isolare, selezionare e identificare EPF30,31,32. Confrontando i diversi tipi di metodi di esca per insetti, Beauveria bassiana e Metarhizium anisopliae sono stati comunemente trovati nelle esche per insetti6,12,13,14.
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori dichiarano che non vi è alcun conflitto di interessi coinvolto in questo lavoro.
Riconoscimenti
Questa ricerca è stata supportata da Grant 109-2313-B-005 -048 -MY3 del Ministero della Scienza e della Tecnologia (MOST).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Agar Bacteriological grade | BIOMAN SCIENTIFIC Co., Ltd. | AGR001 | Suitable in most cell culture/molecular, biology applications. |
| AGAROSE, Biotechnology Grade | BIOMAN SCIENTIFIC Co., Ltd. | AGA001 | For DNA electrophoresis. |
| BioGreen Safe DNA Gel Buffer | BIOMAN | SDB001T | |
| Brass cork borer | Dogger | D89A-44001 | |
| Canon kiss x2 | Canon | EOS 450D | For record strain colony morphology |
| Constant temperature incubator | Yihder Co., Ltd. | LE-509RD | Fungal keeping. |
| cubee Mini-Centrifuge | GeneReach | MC-CUBEE | |
| DigiGel 10 Digital Gel Image System | TOPBIO | DGIS-12S | |
| Finnpipette F2 0.2 to 2 µL Pipette | Thermo Scientific | 4642010 | |
| Finnpipette F2 1 to 10 µL Pipette | Thermo Scientific | 4642030 | |
| Finnpipette F2 10 to 100 µL Pipette | Thermo Scientific | 4642070 | |
| Finnpipette F2 100 to 1000 µL Pipette | Thermo Scientific | 4642090 | |
| Finnpipette F2 2 to 20 µL Pipette | Thermo Scientific | 4642060 | |
| Finnpipette F2 20 to 200 µL Pipette | Thermo Scientific | 4642080 | |
| GeneAmp PCR System 9700 | Applied Biosystems | 4342718 | |
| GenepHlow Gel/PCR Kit | Geneaid | DFH100 | |
| Genius Dry Bath Incubator | Major Science | MD-01N | |
| Graduated Cylinder Custom A 100mL | SIBATA | SABP-1195906 | Measure the volume of reagents. |
| Hand tally counter | SDI | NO.1055 | |
| Hemocytometer | bioman | AP-0650010 | Calculate the number of spore |
| Inoculating loop | Dogger | D8GA-23000 | |
| lid | IDEAHOUSE | RS92004 | |
| Micro cover glass | MUTO PURE CHEMICALS CO.,LTD | 24241 | |
| Microscope imaging system | SAGE VISION CO.,LTD | SGHD-3.6C | |
| Microscope Slides | DOGGER | DG75001-07105 | |
| Mupid-2plus DNA Gel Electrophoresis | ADVANCE | AD110 | |
| Nikon optical microscope | SAGE VISION CO.,LTD | Eclipse CI-L | |
| Plastic cup | IDEAHOUSE | CS60016 | |
| Presto Mini gDNA Yeast Kit | Geneaid | GYBY300 | Fungal genomic DNA extraction kit |
| Sabouraud Dextrose Broth (Sabouraud Liquid Medium) | HiMedia Leading BioSciences Company | M033 | Used for cultivation of yeasts, moulds and aciduric microorganisms. |
| Scalpel Blade No.23 | Swann-Morton | 310 | |
| Scalpel Handle No.4 | AGARWAL SURGICALS | SSS -FOR-01-91 | |
| Shovel | Save & Safe | A -1580242 -00 | |
| Silwet L-77 | bioman(phytotech) | S7777 | Surfactant |
| Sorvall Legend Micro 17 Microcentrifuge | Thermo Scientific | 75002403 | |
| Steel Tweezers | SIPEL ELECTRONIC SA | GG-SA | |
| Sterile Petri Dish | BIOMAN SCIENTIFIC Co., Ltd. | 1621 | Shallow cylindrical containers with fitted lids, specifically for microbiology or cell culture use. |
| ThermoCell MixingBlock | BIOER | MB-101 | |
| Tween 80 | FUJIFILM Wako Pure Chemical Corporation | 164-21775 | |
| TwinGuard ULT Freezer | Panasonic Healthcare Holdings Co., Ltd. | MDF-DU302VX | -80°C sample stored. |
| Vertical floor type cabinet | Chih Chin | BSC-3 | Fungal operating culturing. |
| Vortex Genie II | Scientific | SIG560 | |
| Zipper storage bags | Save & Safe | A -1248915 -00 | |
| 100 bp DNA Ladder | Geneaid | DL007 | |
| -20°C Freezer | FRIGIDAIRE | Frigidaire FFFU21M1QW | -20°C sample and experimental reagents stored. |
| 2X SuperRed PCR Master Mix | TOOLS | TE-SR01 | |
| 50X TAE Buffer | BIOMAN | TAE501000 |
Riferimenti
- Wraight, S. P., Carruthers, R. I. Biopesticides: use and Delivery. , Springer. 233-269 (1999).
- Chase, A., Osborne, L., Ferguson, V. Selective isolation of the entomopathogenic fungi Beauveria bassiana and Metarhizium anisopliae from an artificial potting medium. Florida Entomologist. , 285-292 (1986).
- Meyling, N. V. Methods for isolation of entomopathogenic fungi from the soil environment. University of Copenhagen. , Frederiksberg Denmark. Department of Ecology 1-18 (2007).
- Zimmermann, G. The 'Galleria bait method'for detection of entomopathogenic fungi in soil. Journal of applied Entomology. 102 (1-5), 213-215 (1986).
- Schneider, S., Widmer, F., Jacot, K., Kölliker, R., Enkerli, J. Spatial distribution of Metarhizium clade 1 in agricultural landscapes with arable land and different semi-natural habitats. Applied Soil Ecology. 52, 20-28 (2012).
- Hallouti, A., et al. Diversity of entomopathogenic fungi associated with Mediterranean fruit fly (Ceratitis capitata (Diptera: Tephritidae)) in Moroccan Argan forests and nearby area: impact of soil factors on their distribution. BMC Ecology. 20 (1), 1-13 (2020).
- Meyling, N. V., Eilenberg, J. Occurrence and distribution of soil borne entomopathogenic fungi within a single organic agroecosystem. Agriculture, Ecosystems and Environment. 113 (1-4), 336-341 (2006).
- Skalický, A., Bohatá, A., Šimková, J., Osborne, L. S., Landa, Z. Selection of indigenous isolates of entomopathogenic soil fungus Metarhizium anisopliae under laboratory conditions. Folia Microbiologica. 59 (4), 269-276 (2014).
- Veen, K., Ferron, P. A selective medium for the isolation of Beauveria tenella and of Metarrhizium anisopliae. Journal of Invertebrate Pathology. 8 (2), 268-269 (1966).
- Goettel, M., Inglis, D. Manual of Techniques in Insect Pathology. Lacy, L. , Academic. Amsterdam. 213-249 (1997).
- Luz, C., Netto, M. C. B., Rocha, L. F. N. In vitro susceptibility to fungicides by invertebrate-pathogenic and saprobic fungi. Mycopathologia. 164 (1), 39-47 (2007).
- Mantzoukas, S., et al. Trapping entomopathogenic fungi from vine terroir soil samples with insect baits for controlling serious pests. Applied Sciences. 10 (10), 3539(2020).
- Goble, T., Dames, J., Hill, M., Moore, S. The effects of farming system, habitat type and bait type on the isolation of entomopathogenic fungi from citrus soils in the Eastern Cape Province, South Africa. BioControl. 55 (3), 399-412 (2010).
- Nishi, O., Iiyama, K., Yasunaga-Aoki, C., Shimizu, S. Isolation of entomopathogenic fungi from soil by using bait method with termite, Reticulitermes speratus. Enotomotech. 35, 21-26 (2011).
- Castrillo, L. ARS Collection of Entomopathogenic Fungal Cultures (ARSEF). , (2014).
- Fungal database WorldWide Collection of Entomopathogenic Fungi. University of Vermont. , Available from: http://www.uvm.edu/~entlab/Fungus.html (2019).
- Kim, J. C., et al. Tenebrio molitor-mediated entomopathogenic fungal library construction for pest management. Journal of Asia-Pacific Entomology. 21 (1), 196-204 (2018).
- Keyser, C. A., Henrik, H., Steinwender, B. M., Meyling, N. V. Diversity within the entomopathogenic fungal species Metarhizium flavoviride associated with agricultural crops in Denmark. BMC Microbiology. 15 (1), 1-11 (2015).
- Quesada-Moraga, E., Navas-Cortés, J. A., Maranhao, E. A., Ortiz-Urquiza, A., Santiago-Álvarez, C. Factors affecting the occurrence and distribution of entomopathogenic fungi in natural and cultivated soils. Mycological Research. 111 (8), 947-966 (2007).
- Park, J. B., et al. Developmental characteristics of Tenebrio molitor larvae (Coleoptera: Tenebrionidae) in different instars. International Journal of Industrial Entomology. 28 (1), 5-9 (2014).
- Chang, J. -C., et al. Construction and selection of an entomopathogenic fungal library from soil samples for controlling Spodoptera litura. Frontiers in Sustainable Food Systems. 5, 15(2021).
- Podder, D., Ghosh, S. K. A new application of Trichoderma asperellum as an anopheline larvicide for eco friendly management in medical science. Scientific reports. 9 (1), 1-15 (2019).
- Geneaid Biotech Ltd. Presto Mini gDNA Yeast, Ver. 04.27.17. , Available from: https://www.geneaid.com/data/files/1605664221308055331.pdf (2021).
- White, T. J., Bruns, T., Lee, S., Taylor, J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics. PCR protocols: A guide to methods and applications. 18 (1), 315-322 (1990).
- Kepler, R. M., Humber, R. A., Bischoff, J. F., Rehner, S. A. Clarification of generic and species boundaries for Metarhizium and related fungi through multigene phylogenetics. Mycologia. 106 (4), 811-829 (2014).
- Kepler, R. M. A phylogenetically-based nomenclature for Cordycipitaceae (Hypocreales). IMA Fungus. 8 (2), 335-353 (2017).
- National Resource for Biomedical Supercomputing. GeneDoc: a tool for editing and annotating multiple sequence alignments. Pittsburgh Supercomputing Center's National Resource for Biomedical Supercomputing. , Available from: http://www.nrbsc.org/downloads (1997).
- Thompson, J. D., Gibson, T. J., Plewniak, F., Jeanmougin, F., Higgins, D. G. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research. 25 (24), 4876-4882 (1997).
- Kumar, S., Stecher, G., Tamura, K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets. Molecular Biology and Evolution. 33 (7), 1870-1874 (2016).
- Herlinda, S., Mulyati, S. I. Selection of isolates of entomopathogenic fungi and the bioefficacy of their liquid production against Leptocorisa oratorius nymphs. Microbiology Indonesia. 2 (3), 9(2008).
- Herlinda, S., Irsan, C., Mayasari, R., Septariani, S. Identification and selection of entomopathogenic fungi as biocontrol agents for Aphis gossypii from South Sumatra. Microbiology Indonesia. 4 (3), 137-142 (2010).
- Montes-Bazurto, L. G., Peteche-Yonda, Y., Medina-Cardenas, H. C., Bustillo-Pardey, A. E. Selection of entomopathogenic fungi for the biological control of Demotispa neivai (Coleoptera: Chrysomelidae) in oil palm plantations in Colombia. Journal of Entomological Science. 55 (3), 388-404 (2020).
- Shin, T. -Y., Choi, J. -B., Bae, S. -M., Koo, H. -N., Woo, S. -D. Study on selective media for isolation of entomopathogenic fungi. International Journal of Industrial Entomology. 20 (1), 7-12 (2010).
- Sharma, L., Oliveira, I., Torres, L., Marques, G. Entomopathogenic fungi in Portuguese vineyards soils: Suggesting a 'Galleria-Tenebrio-bait method'as bait-insects Galleria and Tenebrio significantly underestimate the respective recoveries of Metarhizium (robertsii) and Beauveria (bassiana). MycoKeys. (38), 1(2018).
- Rodríguez, M., Gerding, M., France, A. Selección de Hongos Entomopatógenos para el Control de Varroa destructor (Acari: Varroidae). Chilean journal of agricultural research. 69 (4), 534-540 (2009).
- Yang, H., et al. Persistence of Metarhizium (Hypocreales: Clavicipitaceae) and Beauveria bassiana (Hypocreales: Clavicipitaceae) in tobacco soils and potential as biocontrol agents of Spodoptera litura (Lepidoptera: Noctuidae). Environmental entomology. 48 (1), 147-155 (2019).
- Muñiz-Reyes, E., Guzmán-Franco, A. W., Sánchez-Escudero, J., Nieto-Angel, R. Occurrence of entomopathogenic fungi in tejocote (C rataegus mexicana) orchard soils and their pathogenicity against R hagoletis pomonella. Journal of Applied Microbiology. 117 (5), 1450-1462 (2014).
- Lacey, L. A., et al. Goettel Insect pathogens as biological control agents: Back to the future. Journal of Invertebrate Pathology. 132, 1-41 (2015).
- Humber, R. A. Manual of techniques in insect pathology. , Elsevier. 153-185 (1997).
- Rehner, S. A., Buckley, E. A Beauveria phylogeny inferred from nuclear ITS and EF1-α sequences: evidence for cryptic diversification and links to Cordyceps teleomorphs. Mycologia. 97 (1), 84-98 (2005).
- Quandt, C. A., et al. Phylogenetic-based nomenclatural proposals for Ophiocordycipitaceae (Hypocreales) with new combinations in Tolypocladium. IMA fungus. 5 (1), 121-134 (2014).
- Shah, F. A., Wang, C. S., Butt, T. M. Nutrition influences growth and virulence of the insect-pathogenic fungus Metarhizium anisopliae. FEMS Microbiology Letters. 251 (2), 259-266 (2005).
- Ignoffo, C. Environmental factors affecting persistence of entomopathogens. Florida Entomologist. , 516-525 (1992).
- Rodrigues, I. W., Forim, M., Da Silva, M., Fernandes, J., Batista Filho, A. Effect of ultraviolet radiation on fungi Beauveria bassiana and Metarhizium anisopliae, pure and encapsulated, and bio-insecticide action on Diatraea saccharalis. Advances in Entomology. 4 (3), 151-162 (2016).
- Paula, A. R., Ribeiro, A., Lemos, F. J. A., Silva, C. P., Samuels, R. I. Neem oil increases the persistence of the entomopathogenic fungus Metarhizium anisopliae for the control of Aedes aegypti (Diptera: Culicidae) larvae. Parasites and Vectors. 12 (1), 1-9 (2019).
- Morley-Davies, J., Moore, D., Prior, C. Screening of Metarhizium and Beauveria spp. conidia with exposure to simulated sunlight and a range of temperatures. Mycological Research. 100 (1), 31-38 (1996).
- Rangel, D. E., Braga, G. U., Flint, S. D., Anderson, A. J., Roberts, D. W. Variations in UV-B tolerance and germination speed of Metarhizium anisopliae conidia produced on insects and artificial substrates. Journal of Invertebrate Pathology. 87 (2-3), 77-83 (2004).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon