Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Eine Schritt-für-Schritt-Methode zum Nachweis neutralisierender Antikörper gegen AAV mit einem kolorimetrischen zellbasierten Assay
In diesem Artikel
Zusammenfassung
Ein umfassendes Laborprotokoll und ein Analyse-Workflow werden für einen schnellen, kostengünstigen und unkomplizierten kolorimetrischen zellbasierten Assay zur Erkennung neutralisierender Elemente gegen AAV6 beschrieben.
Zusammenfassung
Rekombinante Adeno-assoziierte Viren (rAAV) haben sich als sicherer und erfolgreicher Vektor für die Übertragung von genetischem Material zur Behandlung verschiedener Gesundheitszustände sowohl im Labor als auch in der Klinik erwiesen. Bereits vorhandene neutralisierende Antikörper (NAbs) gegen AAV-Kapside stellen jedoch eine ständige Herausforderung für die erfolgreiche Verabreichung von Gentherapien sowohl in experimentellen Großtiermodellen als auch in menschlichen Populationen dar. Ein vorläufiges Screening auf Wirtsimmunität gegen AAV ist notwendig, um die Wirksamkeit von AAV-basierten Gentherapien sowohl als Forschungsinstrument als auch als klinisch tragfähiges Therapeutikum sicherzustellen. Dieses Protokoll beschreibt einen kolorimetrischen In-vitro-Assay zum Nachweis neutralisierender Faktoren gegen AAV-Serotyp 6 (AAV6). Der Assay nutzt die Reaktion zwischen einem AAV, das für ein alkalisches Phosphatase (AP)-Reportergen kodiert, und seinem Substrat NBT/BCIP, das bei kombination eine unlösliche quantifizierbare violette Färbung erzeugt.
In diesem Protokoll werden Serumproben mit einem AAV-exprimierenden AP kombiniert und inkubiert, um eine mögliche neutralisierende Aktivität zu ermöglichen. Die Virusserummischung wird anschließend den Zellen zugesetzt, um die virale Transduktion von AAVs zu ermöglichen, die nicht neutralisiert wurden. Das NBT/BCIP-Substrat wird zugegeben und durchläuft eine chromogene Reaktion, die der viralen Transduktion und neutralisierenden Aktivität entspricht. Der Anteil der farbigen Fläche wird mit einem kostenlosen Software-Tool quantifiziert, um neutralisierende Titer zu generieren. Dieser Assay zeigt eine starke positive Korrelation zwischen Färbung und Viruskonzentration. Die Beurteilung von Serumproben von Schafen vor und nach verabreichung eines rekombinanten AAV6 führte zu einem dramatischen Anstieg der neutralisierenden Aktivität (125 bis >10.000-fache Zunahme). Der Assay zeigte eine ausreichende Sensitivität, um neutralisierende Aktivität in >1:32.000 Serumverdünnungen nachzuweisen. Dieser Assay bietet eine einfache, schnelle und kostengünstige Methode zum Nachweis von NAbs gegen AAVs.
Einleitung
Adeno-assoziierte Viren (AAV) werden zunehmend als Vektoren für die Verabreichung von Gentherapien zur Erprobung von Behandlungen für verschiedene Gesundheitszustände verwendet, die sich auf das Herz-Kreislauf-, Kreislauf-, Augen- und Zentralnervensystem auswirken1,2,3,4,5. Die Beliebtheit von AAV-Vektoren als führende Gentherapieplattform beruht auf ihrem positiven Sicherheitsprofil, ihrer langfristigen Transgenexpression und ihren weitreichenden gewebespezifischen Tropismen1,6. Erfolgreiche Ergebnisse in Tierversuchen haben den Weg für über fünfzig klinische AAV-Gentherapie-Studien geebnet, die ihre Wirksamkeitsendpunkte erfolgreich erreicht haben7, sowie für die Freigabe des ersten kommerziell erhältlichen AAV-Gentherapeutikums, das von der US-amerikanischen Food and Drug Administration zugelassen wurde8. Nach ersten Erfolgen hat AAV in der Grundlagen- und klinischen Forschung als bevorzugter Vektor weiter an Zugkraft gewonnen und ist derzeit die einzige in vivo Gentherapie, die in den USA und Europa für den klinischen Einsatz zugelassen ist9. Nichtsdestotrotz bleibt das Vorhandensein bereits vorhandener neutralisierender Antikörper (NAbs) gegen AAV-Vektorkaside ein Hindernis sowohl für die präklinische Forschung als auch für die Wirksamkeit klinischer Studien. NAbs sind sowohl in naiven menschlichen als auch in tierischen Populationen vorhanden und hemmen die Gentransduktion nach in vivo Verabreichung eines AAV-Vektors1. Die AAV-Seropositivität ist ein Ausschlusskriterium für die meisten gentherapeutischen Studien, und daher ist das vorläufige Screening auf Wirtsimmunität sowohl im Labor als auch in der Klinik von entscheidender Bedeutung. Die Etablierung eines Assays, der das Vorhandensein von NAbs gegen AAV nachweisen kann, ist ein wesentlicher Schritt in der Pipeline eines AAV-Gentherapie-basierten Forschungsprojekts. Dieser Bericht konzentriert sich auf AAV6, das für Forscher aufgrund seiner effizienten und selektiven Transduktion in quergestreiften Muskeln (Herz und Skelettmuskel) von Interesse war 1,10,11,12. Die Gentherapie gilt als vielversprechende Strategie für das Targeting des Herzens, da es schwierig ist, das Herz ohne invasive Verfahren am offenen Herzen gezielt anzusprechen.
Die neutralisierende Aktivität wird normalerweise entweder mit einem zellbasierten In-vitro- oder einem In-vivo-Transduktionshemmungsassay bestimmt. Im lebenden Organismus NAb-Assays beinhalten in der Regel die Verabreichung von Serum von einer Testperson (z. B. Mensch oder Großtier) an Mäuse, gefolgt von einem AAV mit einem Reportergen, gefolgt von einem Test auf die Expression des Reportergens oder des entsprechenden Antigens. In-vitro-Assays bestimmen NAb-Titer durch Inkubation von Serum oder Plasma von einem Menschen oder Großtier in seriellen Verdünnungen mit einem rekombinanten AAV (rAAV), das ein Reportergen exprimiert. Zellen werden mit dem Serum/Virus-Gemisch infiziert, und das Ausmaß, in dem die Reportergenexpression gehemmt wird, wird im Vergleich zu Kontrollen bewertet. In-vitro-Assays werden aufgrund ihrer vergleichsweise niedrigeren Kosten, ihrer Schnelligkeit bei Tests und ihrer größeren Kapazität für die Standardisierung und Validierung häufig für das NAb-Screening verwendet13,14 im Vergleich zu In-vivo-Assays. Es wird häufig berichtet, dass In-vivo-Assays eine höhere Sensitivität aufweisen15,16, aber die gleiche Behauptung wurde in Bezug auf In-vitro-Assays gemacht14,17.
Bisher haben In-vitro-NAb-Assays hauptsächlich Lumineszenz (Luciferase) als Reportergen verwendet, um die Neutralisation nachzuweisen. Obwohl eine lichtbasierte Methode in vielen Kontexten Von Vorteil ist, kann ein kolorimetrischer/chromogener NAb-Assay unter bestimmten Umständen vorteilhaft sein. Kolorimetrische Assays zur Beurteilung der Neutralisation wurden erfolgreich für andere Viren wie Influenza und Adenovirus eingesetzt18,19. Ihre Attraktivität ergibt sich aus ihrer Einfachheit, ihren niedrigeren Kosten und der Anforderung an alltägliche Laborgeräte und -werkzeuge20. NAb-Assays, die ein Lumineszenz-basiertes Reportergen verwenden, erfordern kostspielige Substratkits, ein Luminometer und entsprechende Software für die Analyse21. Dieser kolorimetrische Assay hat den Vorteil, dass nur ein Lichtmikroskop und ein sehr billiges Substrat benötigt werden. Die Berichterstattung über die Sensitivität von kolorimetrischen gegenüber lumineszierenden Assays hat zu widersprüchlichen Ergebnissen geführt. Eine Studie schlug vor, dass lumineszenzbasierte ELISA-Assays eine höhere Sensitivität und vergleichbare Reproduzierbarkeit wie kolorimetrische Assays aufweisen22, während eine andere studierte, dass kolorimetrische ELISA-Assays eine höhere Sensitivität verleihen23. Hier wird ein detailliertes Protokoll für einen In-vitro-NAb-Assay gegen AAV bereitgestellt, der die chromogene Reaktion zwischen einem AAV- kodierenden alkalischen Phosphatase (AP) -Reportergen und einem nitroblauen Tetrazolium / 5-Brom-4-Chlor-3-indolylphosphat (NBT / BCIP) -Substrat verwendet. Dieses Schritt-für-Schritt-Protokoll wurde auf der Grundlage eines früheren Berichts entwickelt, der ein hPLAP-Reportergen (Human Placental Alkaline Phosphatase) (AAV6-hPLAP) verwendete, um die neutralisierende Aktivität gegen AAV24 nachzuweisen. Dieser Assay ist kostengünstig, zeiteffizient, einfach einzurichten und erfordert minimale technische Fähigkeiten, Laborgeräte und Reagenzien. Darüber hinaus bietet die Einfachheit dieses Assays das Potenzial, für breite Anwendungen in verschiedenen Arten von Zellen, Geweben oder viralen Serotypen optimiert zu werden.
Protokoll
Alle Aspekte der Tierpflege und -experimente wurden gemäß den Richtlinien des Florey Institute of Neuroscience and Mental Health und dem Australian Code for the Care and Use of Animals for Scientific Purposes gemäß Reference25 durchgeführt. Für die Studie wurden 1,5-3-jährige Merinoschafe verwendet. Eine schematische Übersicht über das Assay-Protokoll finden Sie in Abbildung 1.
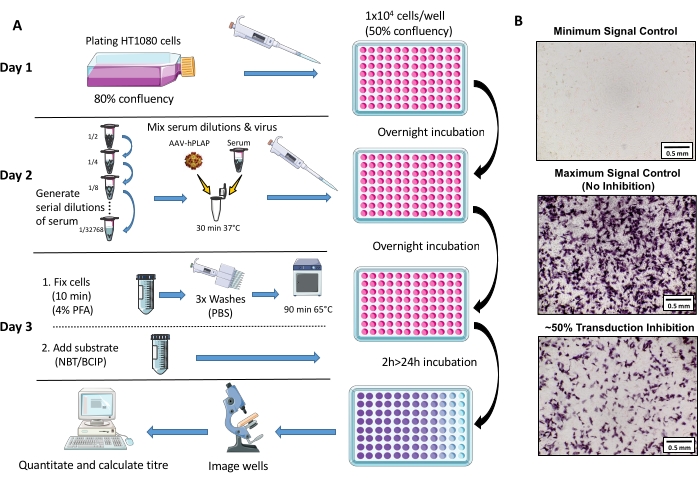
Abbildung 1: Schematische Darstellung des NAb-Assay-Protokolls. (A) Visuelle Darstellung des NAb-Assays zur Veranschaulichung der primären Schritte des Drei-Tage-Protokolls. Kurz gesagt, Zellen werden über Nacht gezüchtet und plattiert. Am folgenden Tag werden serielle Serumverdünnungen hergestellt, mit AAV inkubiert und dann über Nacht mit den Zellen inkubiert. Am nächsten Tag werden die Zellen fixiert, gewaschen, inkubiert, mit dem Substrat kombiniert und erneut inkubiert, gefolgt von Bildgebung und Quantifizierung. (B) Repräsentative Bilder einer minimalen Signalkontrolle (vollständige AAV-Hemmung), einer maximalen Signalkontrolle (keine Hemmung) und einer Schafsserumprobe mit ~50% Signalhemmung. Maßstabsleiste = 0,5 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
1. Erste Vorbereitung
- Zur Beurteilung bei Schafen: Blut in 8 ml Serumseparator-Gerinnselaktivierungsröhrchen (siehe Materialtabelle) entnehmen, die Blutprobe 20-30 min bei Raumtemperatur (RT) belassen und anschließend bei 2.100 x g für 15 min herunterdrehen. Der klare Überstand, der sich an der Oberseite der Röhrchen bildet, ist Serum. Aliquotieren Sie die klare wässrige Phase in Mikrozentrifugenröhrchen und lagern Sie sie bei -80 °C.
HINWEIS: Das Serum bei -80 °C bleibt ~5 Jahre haltbar. Blut wurde aus der Halsschlagader mit einer 16 G Nadel (Spitzenabschneidung) und einer Spritze von bewussten Tieren entnommen. - Hitzeinaktivieren Sie das fötale Rinderserum (FBS), indem Sie es 30 min lang in ein Wasserbad bei 56 °C geben und intermittierend schwenken. Legen Sie zur Präzision ein Thermometer in eine zweite Flasche, die ein äquivalentes Wasservolumen enthält, und geben Sie es gleichzeitig mit der FBS-Flasche in das Wärmebad. Beginnen Sie mit dem Timing, wenn das Thermometer 56 °C erreicht.
- Wenden Sie die richtige aseptische Technik und Zellkulturpraxis für alle nachfolgenden Schritte an, die in der Zellkulturhaube durchgeführt werden26,27. Sprühen Sie vor Gebrauch 70% Ethanol auf alle Gegenstände und die Haube und reinigen Sie es nach Fertigstellung mit 1% Natriumhypochlorit.
- Komplettes Modifiziertes Eagle Medium (DMEM) von Dulbecco durch Kombination von DMEM mit hohem Glucosegehalt (4,5 g/L), DMEM (89%) mit hitzeinaktiviertem FBS (10%) und Penicillin Streptomycin (1%). Kombinieren und filtern Sie mit einem sterilen Vakuumfiltrationssystem (0,22 μm Porengröße, Polyethersulfonmembran) (siehe Materialtabelle). Komplettes DMEM in Folie verpackt bei 4 °C lagern.
- HT1080-Zellen (siehe Materialtabelle) und Passage in einem 75 cm2 quadratischen Kolben wie in Referenz28 beschrieben einrichten. Erstellen Sie mehrere eingefrorene Zellbestände. Verwenden Sie keine Zellen nach 20 Passagen, da weiteres Passieren die Assay-Ergebnisse beeinflussen kann.
2. Tag 1 - Beschichtung der Zellen
- Passage HT1080-Zellen, wenn sie ~ 80% Konfluenz erreichen.
- Komplettes DMEM (hergestellt in Schritt 1.4), 0,05% Trypsin-EDTA und 1x phosphatgepufferte Kochsalzlösung (PBS) bis 37 °C in einem Wasserbad vorwärmen. Entfernen Sie das Wachstumsmedium aus den durchgelassenen Zellen mit einem Aspirationssystem.
HINWEIS: Alle Aspirationen in diesem Protokoll verwenden ein Vakuumsystem mit einem Schlauch, der an einer sterilen 5 ml serologischen Pipette befestigt ist. - Waschen Sie die Zellen in 10 ml vorgewärmtem (37 °C) 1x PBS und trypsinisieren Sie die Zellen für 3-4 min in 4 ml vorgewärmtem 0,05% Trypsin-EDTA, um die Zellen vom Kolben zu lösen.
- Inaktivieren Sie das Trypsin, indem Sie 6 ml vorgewärmtes komplettes DMEM hinzufügen und die Zellen in ein 50-ml-Röhrchen pipettieren. Berechnen Sie die Anzahl und Konzentration lebensfähiger Zellen mit einem Hämozytometer und der Trypanblau-Ausschlussmethode29.
- Verdünnen Sie die Zellen auf eine Konzentration von 1 x 105 Zellen/ml in vorgewärmtem vollständigem DMEM. Säen Sie 100 μL Zellen / Well in klare 96-Well-Flat-Bottomed-Platten (1 x 104 Zellen pro Well). Die Platte bei 37 °C, 5% Kohlendioxid (CO2) über Nacht für 16-22 h inkubieren.
3. Tag 2 - Infektion der Zellen
- Entfernen Sie die Platte(n) aus dem Inkubator und verwenden Sie ein Lichtmikroskop, um zu bestätigen, dass die Zellen gleichmäßig in den Vertiefungen verteilt sind und dass der Zusammenfluss ~ 50% beträgt. Wenn sich die Zellen nicht in einem Bereich von 45% -55% Konfluenz befinden, wiederholen Sie das Protokoll "Tag 1" und passen Sie die anfängliche Zellkonzentration entsprechend an.
- Erzeugen Sie serielle Verdünnungen der interessierenden Serumproben in 1,5 ml Mikrozentrifugenröhrchen unter Verwendung von vorgewärmtem vollständigem DMEM als Verdünnungsmittel. Tabelle 1 zeigt die Erzeugung einer Verdünnungskaskade für dreifache Proben.
- Um den Assay in dreifacher Ausfertigung durchzuführen, wird eine 7,5 x 106 Vektorgenomen (vg)/μL Arbeitslösung von AAV6-hPLAP (siehe Materialtabelle) durch Verdünnen einer Virusstammlösung in 1x PBS hergestellt.
- 66 μL der 7,5 x 106 vg/μL Virus-Arbeitslösung in jedes Röhrchen geben, das 264 μL Serum-/Medienverdünnung (330 μL Gesamtvolumen/Verdünnung, siehe Tabelle 1) enthält.
HINWEIS: Dies ist ein robuster Assay, der keine perfekten Kulturbedingungen erfordert. Um jedoch genau zu quantifizieren und sicherzustellen, dass jeder Assay-Lauf zuverlässig ist, ist es notwendig, Folgendes einzuschließen: (1) eine reine Virus- und Medienkontrolle, (2) eine reine Medienkontrolle und (3) eine NAb-Positivkontrollprobe auf allen Platten unter den gleichen experimentellen Bedingungen. Das beschriebene Volumen (330 μL) macht dreifache Proben +10% des Serum-Virus-Gemisches aus. Die Durchführung von Replikaten wird dringend empfohlen, um die neutralisierende Aktivität genau zu bestimmen.
- Mischen Sie die Virus-/Serumverdünnungen durch Pipettieren und geben Sie die Röhrchen, die die Virus/Serum-Gemische enthalten, in einen Inkubator bei 37 °C, 5% CO2 für 30 min, um eine mögliche Neutralisation zu ermöglichen.
- 100 μL der Virus/Serum-Mischung zu jeder Vertiefung auf die 96-Well-Platte mit 1 x 104 Zellen/Well für jede Verdünnung pipettieren.
HINWEIS: Dies erzeugt eine endgültige Viruskonzentration von 15k Viren / Zellmultiplikation der Infektion (MOI) in jedem Bohrloch. Tabelle 2 enthält ein Beispiel für ein 96-Well-Probenplattenlayout zur Beurteilung von Proben bis zu einer Verdünnung von 1/512. - Wickeln Sie die 96-Well-Platte mit Zellen, Serum und AAV-hPLAP in Folie und legen Sie sie in einen Inkubator bei 37 ° C, 5% CO2 über Nacht für 16-24 h, um AAV-Eintritt in die Zellen zu ermöglichen.
| Etikett der Verdünnungskaskade | Verdünnung | 3 x Probe (240 μL) + 10% Puffervolumen (24 μL) | Verhältnis von Serum zu Medien |
| Verdünnung 1 (D1) | 1/2 | 264 μL Serum 264 μL Medien | 50:50 |
| Verdünnung 2 (D2) | 1/4 | 264 μL D1 + 264 μL Medien | 25:75 |
| Verdünnung 3 (D3) | 1/8 | 264 μL D2 +264μL Medien | 12.5:87.5 |
| Verdünnung 4 (D4) | 1/16 | 264 μL D3 +264 μL Medien | 6.25:93.75 |
| Verdünnung 5 (D5) | 1/32 | 264 μL D4 +264 μL Medien | 3.13:96.87 |
| Verdünnung 6 (D6) | 1/64 | 264 μL D5 +264 μL Medien | 1.56:98.44 |
| Verdünnung 7 (D7) | 1/128 | 264 μL D5 +264 μL Medien | 0.78:99.22 |
| Verdünnung 8 (D8) | 1/256 | 264 μL D5 +264 μL Medien | 0.39:99.61 |
| Verdünnung 9 (D9) | 1/512 | 264 μL D7 + 264 μL Medien | 0.2:99.8 |
| Verdünnung 10 (D10) | 1/2048 | 132 μL D8 + 396 μL Medien | 0.05:99.95 |
| Verdünnung 11 (D11) | 1/8192 | 132 μL D9 + 396 μL Medien | 0.01:99.99 |
| Verdünnung 12 (D12) | 1/32768 | 132 μL D10 + 396 μL Medien | 0.003:99.997 |
Tabelle 1: Serum- und Verdünnungsmittelvolumina, die erforderlich sind, um serielle Serumverdünnungen in dreifacher Ausfertigung zu erzeugen.
| Serumprobe #1 | Serumprobe #2 | Serumprobe #3 | Mono AB (mAB), Steuerungen und zusätzliche Proben | |||||||||
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | |
| Ein | 1/2 | 1/2 | 1/2 | 1/2 | 1/2 | 1/2 | 1/2 | 1/2 | 1/2 | 50 ng MAb | 50 ng MAb | 50 ng MAb |
| B | 1/4 | 1/4 | 1/4 | 1/4 | 1/4 | 1/4 | 1/4 | 1/4 | 1/4 | 5 ng MAb | 5 ng MAb | 5 ng MAb |
| C | 1/8 | 1/8 | 1/8 | 1/8 | 1/8 | 1/8 | 1/8 | 1/8 | 1/8 | 0,5 ng MAb | 0,5 ng MAb | 0,5 ng MAb |
| D | 1/16 | 1/16 | 1/16 | 1/16 | 1/16 | 1/16 | 1/16 | 1/16 | 1/16 | MO (-C) | MO (-C) | MO (-C) |
| E | 1/32 | 1/32 | 1/32 | 1/32 | 1/32 | 1/32 | 1/32 | 1/32 | 1/32 | VO (+C) | VO (+C) | VO (+C) |
| F | 1/64 | 1/64 | 1/64 | 1/64 | 1/64 | 1/64 | 1/64 | 1/64 | 1/64 | Beispiel #1 1/512 | Beispiel #1 1/512 | Beispiel #1 1/512 |
| G | 1/256 | 1/256 | 1/256 | 1/256 | 1/256 | 1/256 | 1/256 | 1/256 | 1/256 | Beispiel #2 1/512 | Beispiel #2 1/512 | Beispiel #2 1/512 |
| H | 1/512 | 1/512 | 1/512 | 1/512 | 1/512 | 1/512 | 1/512 | 1/512 | 1/512 | Beispiel #3 1/512 | Beispiel #3 1/512 | Beispiel #3 1/512 |
Tabelle 2: Beispiel 96-Well-Plattenlayout zur Beurteilung naiver Serumproben in Verdünnungen von 1/2 bis 1/512. Höhere Verdünnungen werden in den Assay aufgenommen, wenn eine Probe beurteilt wird, von der bekannt ist, dass sie positiv auf AAV-NAbs ist (Proben nach der Verabreichung) oder wenn ein höherer Titer erforderlich ist. MO (-C): Nur-Medien-Steuerung. VO (+C): Nur Viren- und Medienkontrolle. mAb: Monoklonaler Antikörper gegen AAV (NAb-Positivkontrolle).
4. Tag 3 - Fixierung und Hinzufügen von Substrat zu den Zellen
- Vorwärmen Sie ein Aliquot von 1x PBS auf 37 °C (~25 ml/96-Well-Platte). Kühlen Sie separate Aliquots aus PBS (~ 25 ml / 96-Well-Platte) und doppelt destilliertem H2O (DDW, ~ 25 ml / 96-Well-Platte) auf 4 ° C. Lösen Sie ein Pellet aus BCIP/NBT (siehe Materialtabelle) in 10 mL DDW in einem 50 mL konischen Zentrifugenrohr durch Vortexieren (10 mL reicht für 2 x 96 Wellplatten).
- Aspirieren Sie das Medium aus den Vertiefungen der 96-Well-Platte mit einer serologischen Pipette oder ähnlichem, die an einem saugbasierten Aspirationssystem oder Rauchabzugssauger befestigt ist. Legen Sie die Spitze der serologischen Pipette vorsichtig in den Brunnen und entfernen Sie die Medien unter Vorsicht, um die anhaftenden Zellen nicht zu stören.
- Fügen Sie 50 μL RT 4% PFA zu jeder Vertiefung mit einer Pipette hinzu. Wickeln Sie die Platte in Folie und lassen Sie sie 10 Minuten bei RT, um die Zellen zu fixieren.
VORSICHT: Paraformaldehyd (PFA) ist ein wahrscheinliches Karzinogen und ist giftig durch Haut- oder Augenkontakt oder Inhalation. Griff in einem Abzug mit geeigneter persönlicher Schutzausrüstung sowie einer Gesichtsmaske. Machen Sie frische 4% PFA in PBS verdünnt (~ 7 ml erforderlich pro 96-Well-Platte).
- Fügen Sie 50 μL RT 4% PFA zu jeder Vertiefung mit einer Pipette hinzu. Wickeln Sie die Platte in Folie und lassen Sie sie 10 Minuten bei RT, um die Zellen zu fixieren.
- Waschen und aspirieren Sie die Zellen mit 200 μL RT 1x PBS. Wiederholen Sie diesen Schritt zweimal.
HINWEIS: Eine Mehrkanalpipette ist eine effiziente Option für die Pipettierschritte. - 200 μL vorgewärmtes PBS in jede Vertiefung pipettieren, die Platte in Folie wickeln und bei 65 °C für 90 min inkubieren, um die endogene alkalische Phosphataseaktivität zu denaturieren30.
- Saugen Sie Vertiefungen und Waschzellen mit 200 μL kaltem (4 °C) PBS. Erneut absaugen, 200 μl kaltes DDW einwaschen und erneut aspirieren.
- 50 μL des gelösten BCIP/NBT (hergestellt in Schritt 4.1) werden in jede Vertiefung pipettiert.
- Wickeln Sie den Teller in Folie und inkubieren Sie bei RT für 2-24 h.
HINWEIS: Seien Sie konsistent mit der Inkubationszeit zwischen den Läufen; Die zeitliche Flexibilität ermöglicht es den Benutzern, Brunnen entweder am 3. Tag oder am folgenden Tag zu fotografieren. - Nehmen Sie mit einer Lichtmikroskopkamera Fotos von jedem Bohrloch mit einem 4-fachen Objektiv auf, um sicherzustellen, dass die gleiche Belichtung, der gleiche Weißabgleich und die gleichen Lichteinstellungen für alle durchgeführten Assays konsistent verwendet werden.
- Positionieren Sie jede Vertiefung identisch und stellen Sie sicher, dass die Kanten der Vertiefung auf den Fotos nicht sichtbar sind. Speichern Sie Fotos im TIF-Format oder ähnlichem.
HINWEIS: Die spezifischen Einstellungen variieren zwischen den Mikroskopen, aber die Quantifizierung ist am effektivsten, wenn die Hintergrundbeleuchtung hoch und konsistent in den Vertiefungen ist (Abbildung 1B).
- Positionieren Sie jede Vertiefung identisch und stellen Sie sicher, dass die Kanten der Vertiefung auf den Fotos nicht sichtbar sind. Speichern Sie Fotos im TIF-Format oder ähnlichem.
5. Quantifizierung zur Bestimmung der neutralisierenden Aktivität mit ImageJ
- Laden Sie die frei verfügbare Software "ImageJ" herunter und installieren Sie sie (siehe Literaturverzeichnis).
- Öffnen Sie das zu analysierende Bild in BildJ, indem Sie Datei > Öffnen auswählen (Abbildung 2).
- Wenn Sie farbige Bilder verwenden, konvertieren Sie in Graustufen, indem Sie Bild > Typ > 8-Bit auswählen.
- Klicken Sie auf Bild > > Schwellenwert anpassen. Passen Sie den Schwellenwert an, bis alle farbigen Bereiche rot eingefärbt sind, der Hintergrund jedoch nicht. Nach Zugabe von NBT/BCIP lagert sich das farbige Produkt im Bereich um die Zellen ab, die hPLAP exprimieren.
HINWEIS: Es wird empfohlen, für alle Bilder, die auf derselben Platte aufgenommen wurden, dieselbe Schwellenwerteinstellung zu verwenden. - Klicken Sie auf Analysieren > Messungen festlegen , aktivieren Sie die Kontrollkästchen Fläche, Schwellenwert begrenzen, Flächenanteil und Anzeigebeschriftung und klicken Sie auf OK.
- Um den Signalwert (Prozentsatz der Färbung) einer bestimmten Vertiefung zu bestimmen, klicken Sie auf Analysieren > Messen. Die Spalte '% Area' des Popup-Fensters zeigt den Signalstand an.
- Führen Sie eine Quantifizierung für alle Stichprobenreplikate durch. Schließen Sie kontaminierte Vertiefungen, Vertiefungen mit ungleichmäßiger Zellverteilung oder Vertiefungen mit unterschiedlicher Zelldichte oder Beleuchtung aus.
HINWEIS: Beispiele für Bohrlöcher, die für den Ausschluss in Betracht gezogen werden sollten, finden Sie in der ergänzenden Abbildung 1 . Typischerweise können 3-4 Bohrlöcher den Ausschluss von einer 96-Well-Platte erfordern. Abbildung 2 zeigt eine visuelle Darstellung des Quantifizierungsprozesses mitHilfe von ImageJ.
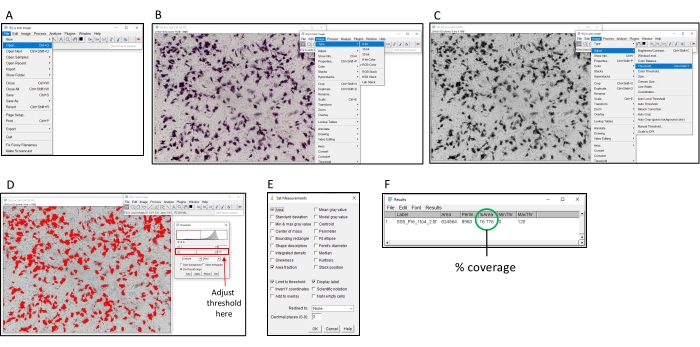
Abbildung 2: Schritte zum Bestimmen der prozentualen Farbgebung mit der ImageJ-Software. (A) Öffnen Sie das zu analysierende Bild mit der ImageJ-Software. (B) Konvertieren Sie das Bild in 8-Bit-Graustufen. (C) Öffnen Sie das Schwellenwertfenster. (D) Passen Sie den maximalen Schwellenwert so an, dass alle farbigen Bereiche abgedeckt sind, der Hintergrundbereich jedoch nicht (dieser Schwellenwert sollte auf einer gesamten Platte konsistent sein). (E) Wählen Sie die Dropbox "Analysieren", klicken Sie auf "Messungen festlegen" und aktivieren Sie "Fläche", "Flächenanteil", "Grenzschwellenwert" und "Anzeigebeschriftung" und klicken Sie auf "OK". (F) Klicken Sie auf "Messen", um die abgedeckte Fläche zu messen. Die Fläche in % gibt den Anteil des Bildes an, der farbig war. Dieser kann dann mit den Kontrollproben verwendet werden, um den TI50-Titer zu bestimmen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
6. Bestimmung des Titers der Transduktionshemmung (TI50)
- Bestimmen Sie die durchschnittliche Anzeige aus Replikaten (mithilfe der in Schritt 5 beschriebenen Schritte) für Folgendes: (1) Nur-Medien-Steuerung (Baseline-Signalmessung). (2) Nur Virus + Medienkontrolle (maximale Signalablesung). (3) Virus- und Serumproben von Interesse.
- Berechnen Sie den Prozentsatz der Hemmung mit der folgenden Formel:
100 - [(Testprobensignalauslesung (Virus + Serumprobe von Interesse) - Baseline-Signalanzeige (nur Medienkontrolle)) / (maximale Signalauslesung (nur Medien und Virus) - Baseline-Signalauslesung) x 100] = % Transduktionshemmung13. - Berechnen Sie die prozentuale Transduktionshemmung aus allen Replikaten jeder Verdünnung für alle Proben nach der Formel in 6.2. Bestimmen Sie die durchschnittliche Transduktionshemmung zwischen den technischen Replikaten für jede Verdünnung für alle Proben und Kontrollen.
- Berechnen Sie den 50%igen Transduktionshemmtiter (TI50-Titer ) einer Interessierenprobe, indem Sie die niedrigste Verdünnung der Probe bestimmen, die eine Transduktionshemmung der hPLAP-Aktivität von 50 % oder mehr ergibt. Wenn z. B. eine 1/8-Verdünnung einer Probe eine Transduktionshemmung von mehr als 50% aufweist, basierend auf der in 6.2 durchgeführten Berechnung (und eine 1/4-Verdünnung nicht), geben Sie den TI50-Titer als 1/8 an.
7. Bestimmung von neutralisierten AAV-Partikeln
- Berechnen Sie die Anzahl der neutralisierten AAV-Partikel pro μL Serum für eine bestimmte Probe unter Verwendung der folgenden Formel:
((MOI x Zellzahl/Well) / (Serumvolumen / Verdünnungsfaktor des TI50-Titers )) / 2 = neutralisierte AAV-Partikel / μL Serum9.
HINWEIS: Die Division durch 2 berücksichtigt den TI50, der 50% der neutralisierten Partikel misst. Für eine Probe, die einen TI50-Titer von 1/4 (25% Serum, 75% Verdünnungsmittel) ergibt, in dem der Assay 80 μL unverdünntes Serum und einen MOI von 15k auf 1 x 104 Zellen plattiert verwendet, wird die folgende Berechnung verwendet: ((15000 x 10000) / (80/4)) / 2 = 3,75x106 neutralisierte Partikel / μL Serum.
Ergebnisse
Transduktionsassay zur Bestimmung der optimalen Virusdosis für die Plattenabdeckung
HT1080-Zellen, eine etablierte Fibrosarkom-Zelllinie, wurden für diesen Assay ausgewählt. Eine Konzentration von 1 x 104 HT1080-Zellen/Well lieferte ~50% Zellkonfluenz in jeder Vertiefung einer 96-Well-Platte. Um die optimale Viruskonzentration für den Assay zu bestimmen, wurde ein rAAV, das für ein hPLAP(human placental alkaline phosphatase) Reportergen (AAV6-hPLAP)31 kodiert, i...
Diskussion
Dieser Bericht beschreibt einen kolorimetrischen Assay, der das Ausmaß der AAV-Neutralisation in einer bestimmten Serumprobe bewertet, indem er eine chromogene Reaktion bewertet, die dem Grad der in vitro viralen Transduktion entspricht. Die Entwicklung des Protokolls basierte auf der bekannten chromogenen Reaktion zwischen dem Enzym alkalische Phosphatase und NBT/BCIP, die weithin als Färbewerkzeug für den Nachweis von Proteintargets in Anwendungen wie der Immunhistochemie und als Reporterwerkzeug zur Bewert...
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Diese Studie wurde durch einen Projektzuschuss des National Health and Medical Research Council für JRM und CJT (ID 1163732) und teilweise durch das Operational Infrastructure Support Program der viktorianischen Regierung finanziert. SB wird durch ein gemeinsames Baker Heart and Diabetes Institute-La Trobe University Doctoral Scholarship unterstützt. KLW wird von der Shine On Foundation und einem Future Leader Fellowship der National Heart Foundation of Australia (ID 102539) unterstützt. JRM wird durch ein Senior Research Fellowship (ID 1078985) des National Health and Medical Research Council unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin/EDTA | Gibco | 25300-054 | |
| 50 mL conical centrifuge tube | Falcon | 14-432-22 | Or equivalent |
| 75 cm2 square flasks | Falcon | 353136 | Or equivalent |
| 96 well flat bottomed plate | Falcon | 353072 | |
| AAV6-CMV-hPLAP Vector | Muscle Research & Therapeutics Lab (University of Melbourne, Australia) AAV6-CMV-hPLAP can be provided upon request. | ||
| Aluminium foil | |||
| Anti-AAV6 (intact particle) mouse monoclonal antibody, (ADK6) | PROGEN | 610159 | Positive control monoclonal antibody |
| BCIP/NBT | SIGMAFAST | B5655 | |
| Cell and tissue culture safety cabinet | |||
| Electronic Pipette | 5 & 10 mL stripette inserts | ||
| Fetal Bovine Serum | Gibco | 10099-141 | |
| Haemocytometer | |||
| High glucose Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965118 | |
| HT1080 cells | ATCC | ||
| ImageJ Software | Freely available: https://imagej.nih.gov/ij/download.html | ||
| Incubator | 37 °C, 5% CO2 | ||
| Light microscope with camera | Capable of taking photos with a 4x objective lens | ||
| Oven | For a 65 °C incubation | ||
| Paraformaldehyde | MERCK | 30525-89-4 | |
| Penicillin Streptomycin | Gibco | 15140-122 | |
| Phosphate buffered saline | |||
| Pipettes and tips | 20 μL, 200 μL & 1 mL single pipettes and tips & 200 μL multichannel pipette | ||
| Stericup quick release filter | Millipore | S2GPU10RE | Used for combining media reagents |
| Trypan blue solution | Sigma-Aldrich | T8154 | |
| VACUETTE TUBE 8 ml CAT Serum Separator Clot Activator | Greiner BIO-ONE | 455071 | Used for serum collection & processing from sheep |
| Water bath |
Referenzen
- Bass-Stringer, S., et al. Adeno-associated virus gene therapy: Translational progress and future prospects in the treatment of heart failure. Heart, Lung and Circulation. 27 (11), 1285-1300 (2018).
- Casey, G. A., Papp, K. M., MacDonald, I. M. Ocular gene therapy with adeno-associated virus vectors: current outlook for patients and researchers. Journal of Ophthalmic and Vision Research. 15 (3), 396-399 (2020).
- Lykken, E. A., Shyng, C., Edwards, R. J., Rozenberg, A., Gray, S. J. Recent progress and considerations for AAV gene therapies targeting the central nervous system. Journal of Neurodevelopmental Disorders. 10 (1), 16 (2018).
- Guggino, W. B., Cebotaru, L. Adeno-Associated Virus (AAV) gene therapy for cystic fibrosis: Current barriers and recent developments. Expert Opinion on Biological Therapy. 17 (10), 1265-1273 (2017).
- Perrin, G. Q., Herzog, R. W., Markusic, D. M. Update on clinical gene therapy for hemophilia. Blood. 133 (5), 407-414 (2019).
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature Reviews Drug Discovery. 18 (5), 358-378 (2019).
- Kuzmin, D. A., et al. The clinical landscape for AAV gene therapies. Nature Reviews Drug Discovery. 20 (3), 173-174 (2021).
- Russell, S., et al. Efficacy and safety of voretigene neparvovec (AAV2-hRPE65v2) in patients with RPE65-mediated inherited retinal dystrophy: A randomised, controlled, open-label, phase 3 trial. Lancet. 390 (10097), 849-860 (2017).
- Weber, T. Anti-AAV Antibodies in AAV gene therapy: Current challenges and possible solutions. Frontiers in Immunology. 12, 658399 (2021).
- Weeks, K. L., et al. Phosphoinositide 3-kinase p110alpha is a master regulator of exercise-induced cardioprotection and PI3K gene therapy rescues cardiac dysfunction. Circulation: Heart Failure. 5 (4), 523-534 (2012).
- Gregorevic, P., et al. Systemic delivery of genes to striated muscles using adeno-associated viral vectors. Nature Medicine. 10 (8), 828-834 (2004).
- Bernardo, B. C., et al. Gene delivery of medium chain acyl-coenzyme A dehydrogenase induces physiological cardiac hypertrophy and protects against pathological remodelling. Clinical Science (London). 132 (3), 381-397 (2018).
- Meliani, A., et al. Determination of anti-adeno-associated virus vector neutralizing antibody titer with an in vitro reporter system. Human Gene Therapy Methods. 26 (2), 45-53 (2015).
- Falese, L., et al. Strategy to detect pre-existing immunity to AAV gene therapy. Gene Therapy. 24 (12), 768-778 (2017).
- Wang, D., et al. Adeno-Associated virus neutralizing antibodies in large animals and their impact on brain intraparenchymal gene transfer. Molecular Therapy - Methods & Clinical Development. 11, 65-72 (2018).
- Wang, M., et al. Prediction of adeno-associated virus neutralizing antibody activity for clinical application. Gene Therapy. 22 (12), 984-992 (2015).
- Kruzik, A., et al. Detection of biologically relevant low-titer neutralizing antibodies against adeno-associated virus require sensitive in vitro assays. Human Gene Therapy Methods. 30 (2), 35-43 (2019).
- Lehtoranta, L., Villberg, A., Santanen, R., Ziegler, T. A novel, colorimetric neutralization assay for measuring antibodies to influenza viruses. Journal of Virological Methods. 159 (2), 271-276 (2009).
- Johnston, P. B., Grayston, J. T., Loosli, C. G. Adenovirus neutralizing antibody determination by colorimetric assay. Proceedings of the Society for Experimental Biology and Medicine. 94 (2), 338-343 (1957).
- Xiaoli Zhu, T. G. . Nano-Inspired Biosensors for Protein Assay with Clinical Applications. , 237-264 (2019).
- Jungmann, A., Muller, O., Rapti, K. Cell-based measurement of neutralizing antibodies against adeno-associated virus (AAV). Methods in Molecular Biology. 1521, 109-126 (2017).
- Samineni, S., et al. Optimization, comparison, and application of colorimetric vs. chemiluminescence based indirect sandwich ELISA for measurement of human IL-23. Journal of Immunoassay and Immunochemistry. 27 (2), 183-193 (2006).
- Siddiqui, J., Remick, D. G. Improved sensitivity of colorimetric compared to chemiluminescence ELISAs for cytokine assays. Journal of Immunoassay and Immunochemistry. 24 (3), 273-283 (2003).
- Arnett, A. L., Garikipati, D., Wang, Z., Tapscott, S., Chamberlain, J. S. Immune responses to rAAV6: The Influence of canine parvovirus vaccination and neonatal administration of viral vector. Frontiers in Microbiology. 2, 220 (2011).
- Australian code for the care and use of animals for scientific purposes. National Health and Medical Research Council Available from: https://www.nhmrc.gov.au/about-us/publications/australian-code-care-and-use-animals-scientific-purposes (2013)
- Coecke, S., et al. Guidance on good cell culture practice. A report of the second ECVAM task force on good cell culture practice. Alternatives to Laboratory Animals. 33 (3), 261-287 (2005).
- Journal of Visualized Experiments. General Laboratory Techniques. Journal of Visualized Experiments Database. , (2018).
- AAV-HT1080 Cells. Stratagene Available from: https://www.chem-agilent.com/pdf/strata/240109.pdf (2003)
- Strober, W. Trypan blue exclusion test of cell viability. Current Protocols in Immunology. 111 (3), 1-3 (2015).
- Bieber, S., et al. Extracorporeal delivery of rAAV with metabolic exchange and oxygenation. Scientific Reports. 3, 1538 (2013).
- Winbanks, C. E., Beyer, C., Qian, H., Gregorevic, P. Transduction of skeletal muscles with common reporter genes can promote muscle fiber degeneration and inflammation. PLoS One. 7 (12), 51627 (2012).
- Thomas, C. J., et al. Evidence that the MEK/ERK but not the PI3K/Akt pathway is required for protection from myocardial ischemia-reperfusion injury by 3',4'-dihydroxyflavonol. European Journal of Pharmacology. 758, 53-59 (2015).
- Barger, A., et al. Use of alkaline phosphatase staining to differentiate canine osteosarcoma from other vimentin-positive tumors. Veterinary Pathology. 42 (2), 161-165 (2005).
- Gregorevic, P., et al. Evaluation of vascular delivery methodologies to enhance rAAV6-mediated gene transfer to canine striated musculature. Molecular Therapy. 17 (8), 1427-1433 (2009).
- Sharma, A., Ghosh, A., Hansen, E. T., Newman, J. M., Mohan, R. R. Transduction efficiency of AAV 2/6, 2/8 and 2/9 vectors for delivering genes in human corneal fibroblasts. Brain Research Bulletin. 81 (2-3), 273-278 (2010).
- Smejkal, G. B., Kaul, C. A. Stability of nitroblue tetrazolium-based alkaline phosphatase substrates. Journal of Histochemistry & Cytochemistry. 49 (9), 1189-1190 (2001).
- Falese, L., et al. Strategy to detect pre-existing immunity to AAV gene therapy. Gene Therapy. 24 (12), 768-778 (2017).
- Orlowski, A., et al. Successful transduction with AAV Vectors after selective depletion of anti-aav antibodies by immunoadsorption. Molecular Therapy - Methods & Clinical Development. 16, 192-203 (2020).
- Goossens, K., et al. Differential microRNA expression analysis in blastocysts by whole mount in situ hybridization and reverse transcription quantitative polymerase chain reaction on laser capture microdissection samples. Analytical Biochemistry. 423 (1), 93-101 (2012).
- Entrican, G., Wattegedera, S. R., Griffiths, D. J. Exploiting ovine immunology to improve the relevance of biomedical models. Molecular Immunology. 66 (1), 68-77 (2015).
- Walters, E. M., Prather, R. S. Advancing swine models for human health and diseases. Molecular Medicine. 110 (3), 212-215 (2013).
- Rapti, K., et al. Neutralizing antibodies against AAV serotypes 1, 2, 6, and 9 in sera of commonly used animal models. Molecular Therapy. 20 (1), 73-83 (2012).
- Tellez, J., et al. Characterization of naturally-occurring humoral immunity to AAV in sheep. PLoS One. 8 (9), 75142 (2013).
- Gupta, S., et al. Recommendations for the validation of cell-based assays used for the detection of neutralizing antibody immune responses elicited against biological therapeutics. Journal of Pharmaceutical and Biomedical Analysis. 55 (5), 878-888 (2011).
- Gupta, S., et al. Recommendations for the design, optimization, and qualification of cell-based assays used for the detection of neutralizing antibody responses elicited to biological therapeutics. Journal of Immunological Methods. 321 (1-2), 1-18 (2007).
- Shankar, G., et al. Recommendations for the validation of immunoassays used for detection of host antibodies against biotechnology products. Journal of Pharmaceutical and Biomedical Analysis. 48 (5), 1267-1281 (2008).
- U.S. Department of Health and Human Services Food and Drug Administration. Center for Drug Evaluation and Research (CDER). Immunogenicity Testing of Therapeutic Protein Products — Developing and Validating Assays for Anti-Drug Antibody Detection. U.S. Department of Health and Human Services Food and Drug Administration. , (2019).
- Baatartsogt, N., et al. A sensitive and reproducible cell-based assay via secNanoLuc to detect neutralizing antibody against adeno-associated virus vector capsid. Molecular Therapy - Methods & Clinical Development. 22, 162-171 (2021).
- Watano, R., Ohmori, T., Hishikawa, S., Sakata, A., Mizukami, H. Utility of micro mini pigs for evaluating liver-mediated gene expression in the presence of neutralizing antibody against vector capsid. Gene Therapy. 27 (9), 427-434 (2020).
- Majowicz, A., et al. Therapeutic hFIX activity achieved after single AAV5-hFIX treatment in Hemophilia B patients and NHPs with pre-existing anti-AAV5 NABs. Molecular Therapy - Methods & Clinical Development. 14, 27-36 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten