Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Une méthode étape par étape pour détecter les anticorps neutralisants contre l’AAV à l’aide d’un test colorimétrique à base de cellules
Dans cet article
Résumé
Un protocole de laboratoire complet et un flux de travail d’analyse sont décrits pour un test colorimétrique rapide, rentable et simple basé sur les cellules afin de détecter les éléments neutralisants contre AAV6.
Résumé
Les virus adéno-associés recombinants (rAAV) se sont avérés être un vecteur sûr et efficace pour le transfert de matériel génétique afin de traiter divers problèmes de santé en laboratoire et en clinique. Cependant, les anticorps neutralisants préexistants (NAbs) contre les capsides AAV posent un défi permanent pour l’administration réussie de thérapies géniques à la fois dans les grands modèles expérimentaux animaux et dans les populations humaines. Un dépistage préliminaire de l’immunité de l’hôte contre l’AAV est nécessaire pour assurer l’efficacité des thérapies géniques à base d’AAV en tant qu’outil de recherche et en tant qu’agent thérapeutique cliniquement viable. Ce protocole décrit un test colorimétrique in vitro pour détecter les facteurs neutralisants contre le sérotype 6 de l’AAV (AAV6). Le test utilise la réaction entre un AAV codant pour un gène rapporteur de phosphatase alcaline (AP) et son substrat NBT / BCIP, qui génère une tache violette quantifiable insoluble lors de la combinaison.
Dans ce protocole, les échantillons de sérum sont combinés avec un AAV exprimant l’AP et incubés pour permettre une activité neutralisante potentielle. Le mélange de sérum viral est ensuite ajouté aux cellules pour permettre la transduction virale de tous les AAV qui n’ont pas été neutralisés. Le substrat NBT/BCIP est ajouté et subit une réaction chromogène, correspondant à la transduction virale et à l’activité neutralisante. La proportion de surface colorée est quantifiée à l’aide d’un outil logiciel gratuit pour générer des titres neutralisants. Ce test montre une forte corrélation positive entre la coloration et la concentration virale. L’évaluation d’échantillons de sérum de moutons avant et après l’administration d’un AAV6 recombinant a entraîné une augmentation spectaculaire de l’activité neutralisante (augmentation de 125 à >10 000 fois). Le test a montré une sensibilité adéquate pour détecter l’activité neutralisante dans >1:32 000 dilutions sériques. Ce test fournit une méthode simple, rapide et rentable pour détecter les NAbs contre les AAV.
Introduction
Les virus adéno-associés (AAV) sont de plus en plus utilisés comme vecteurs pour l’administration de thérapies géniques à des traitements d’essai pour divers problèmes de santé qui ont un impact sur les systèmes cardiovasculaire, pulmonaire, circulatoire, oculaire et nerveux central1,2,3,4,5. La popularité des vecteurs AAV en tant que plate-forme de thérapie génique de premier plan découle de leur profil d’innocuité positif, de leur expression transgénique à long terme et de leurs tropismes spécifiques aux tissus1,6. Les résultats positifs des études animales ont ouvert la voie à plus de cinquante essais cliniques de thérapie génique AAV qui ont atteint avec succès leurs critères d’efficacité7, ainsi qu’à la sortie du premier médicament de thérapie génique AAV disponible dans le commerce approuvé par la Food and Drug Administration des États-Unis8. Après les premiers succès, l’AAV a continué de gagner du terrain dans les secteurs de la recherche fondamentale et clinique en tant que vecteur de choix et est actuellement la seule thérapie génique in vivo approuvée pour une utilisation clinique aux États-Unis et en Europe9. Néanmoins, la présence d’anticorps neutralisants préexistants (NAbs) contre les capsides vectorielles AAV reste un obstacle à la recherche préclinique et à l’efficacité des essais cliniques. Les NAbs sont présents dans les populations humaines et animales naïves et inhibent la transduction des gènes après l’administration in vivo d’un vecteur AAV1. La séropositivité à l’AAV est un critère d’exclusion pour la plupart des essais de thérapie génique et, par conséquent, le dépistage préliminaire de l’immunité de l’hôte est crucial en laboratoire et en clinique. L’établissement d’un test capable de détecter la présence de NAbs contre l’AAV est une étape essentielle dans le pipeline de tout projet de recherche basé sur la thérapie génique AAV. Ce rapport se concentre sur AAV6 qui a intéressé les chercheurs en raison de sa transduction efficace et sélective dans les muscles striés (cœur et muscle squelettique)1,10,11,12. La thérapie génique est considérée comme une stratégie prometteuse pour cibler le cœur, car il est difficile de cibler spécifiquement le cœur sans procédures invasives à cœur ouvert.
L’activité neutralisante est généralement déterminée à l’aide d’un test d’inhibition de la transduction in vitro ou in vivo à base de cellules. In vivo Les tests NAb impliquent généralement l’administration de sérum d’un sujet testé (par exemple, humain ou gros animal) à des souris, suivie d’un AAV avec un gène rapporteur, suivi d’un test d’expression du gène rapporteur ou de l’antigène correspondant. Les essais in vitro déterminent les titres de NAb en incubant du sérum ou du plasma d’un humain ou d’un gros animal dans des dilutions en série avec un AAV recombinant (rAAV) qui exprime un gène rapporteur. Les cellules sont infectées par le mélange sérum/virus, et la mesure dans laquelle l’expression du gène rapporteur est inhibée est évaluée par rapport aux témoins. Les tests in vitro sont largement utilisés pour le dépistage du NAb en raison de leur coût comparativement inférieur, de la rapidité des tests et de la plus grande capacité de normalisation et de validation13,14 par rapport aux essais in vivo. On rapporte souvent que les essais in vivo ont une plus grande sensibilité15,16, mais la même allégation a été faite concernant les essais in vitro14,17.
À ce jour, les tests NAb in vitro ont principalement utilisé la luminescence (luciférase) comme gène rapporteur pour détecter la neutralisation. Bien qu’une méthode basée sur la lumière ait du mérite dans de nombreux contextes, un test colorimétrique / chromogène NAb peut être avantageux dans certaines circonstances. Des tests colorimétriques pour évaluer la neutralisation ont été utilisés avec succès pour d’autres virus tels que la grippe et l’adénovirus18,19. Leur attrait provient de leur simplicité, de leur coût inférieur et de l’exigence de ne disposer que d’appareils et d’outils de laboratoire quotidiens20. Les tests NAb qui utilisent un gène rapporteur basé sur la luminescence nécessitent des kits de substrat coûteux, un luminomètre et un logiciel correspondant pour l’analyse21. Ce test colorimétrique a l’avantage de ne nécessiter qu’un microscope optique et un substrat très bon marché. La déclaration de la sensibilité des tests colorimétriques par rapport aux tests luminescents a donné des résultats contradictoires. Une étude a suggéré que les tests ELISA basés sur la luminescence présentent une plus grande sensibilité et une reproductibilité comparable aux tests colorimétriques22, tandis qu’une autre a révélé que les tests ELISA basés sur la colorimétrie conféraient une plus grande sensibilité23. Ici, un protocole détaillé pour un test NAb in vitro contre l’AAV qui utilise la réaction chromogène entre un AAV codant pour un gène rapporteur de phosphatase alcaline (AP) et un substrat de tétrazolium bleu nitro / 5-bromo-4-chloro-3-indolyl phosphate (NBT / BCIP) est fourni. Ce protocole étape par étape a été développé sur la base d’un rapport précédent qui utilisait un gène rapporteur hPLAP (phosphatase alcaline placentaire humaine) (AAV6-hPLAP) pour détecter l’activité neutralisante contre AAV24. Ce test est rentable, rapide, facile à configurer et nécessite un minimum de compétences techniques, d’équipement de laboratoire et de réactifs. De plus, la simplicité de ce test lui donne le potentiel d’être optimisé pour de larges applications sur différents types de cellules, de tissus ou de sérotypes viraux.
Protocole
Tous les aspects des soins et de l’expérimentation animale ont été menés conformément aux lignes directrices du Florey Institute of Neuroscience and Mental Health et au Code australien pour le soin et l’utilisation des animaux à des fins scientifiques suivant la référence25. Des brebis mérinos âgées de 1,5 à 3 ans ont été utilisées pour l’étude. Un aperçu schématique du protocole d’essai est fourni à la figure 1.
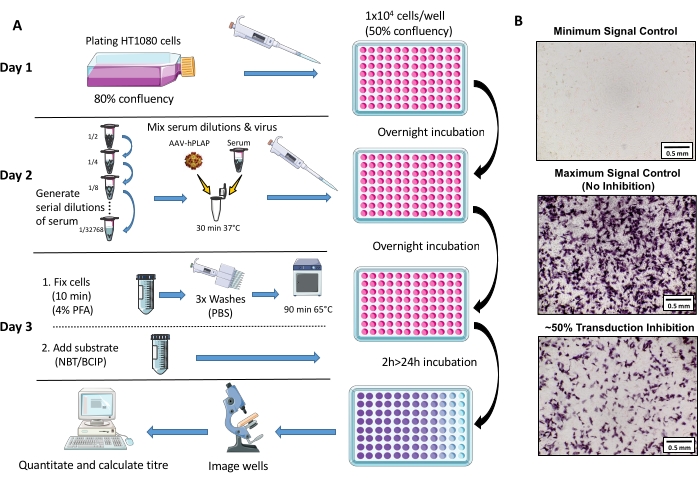
Figure 1 : Diagramme schématique du protocole de test NAb. (A) Représentation visuelle du test NAb illustrant les principales étapes impliquées dans le protocole de trois jours. En bref, les cellules sont cultivées et plaquées pendant la nuit. Le lendemain, des dilutions en série du sérum sont préparées, incubées avec de l’AAV, puis incubées avec les cellules pendant la nuit. Le lendemain, les cellules sont fixées, lavées, incubées, combinées avec le substrat et incubées à nouveau, suivies de l’imagerie et de la quantification. (B) Images représentatives d’un contrôle de signal minimum (inhibition complète de l’AAV), d’un contrôle de signal maximal (pas d’inhibition) et d’un échantillon de sérum ovin avec une inhibition du signal d’environ 50%. Barre d’échelle = 0,5 mm. Veuillez cliquer ici pour afficher une version agrandie de cette figure.
1. Préparation initiale
- Pour l’évaluation chez le mouton : prélever le sang dans des tubes d’activation de caillots séparateurs de sérum de 8 mL (voir tableau des matériaux), laisser l’échantillon de sang à température ambiante (RT) pendant 20 à 30 min, puis tourner à 2 100 x g pendant 15 min. Le surnageant clair qui se forme au sommet des tubes est le sérum. Aliquoter la phase aqueuse claire dans des tubes de microcentrifugation et stocker à -80 °C.
REMARQUE: Le sérum à -80 ° C reste stable pendant environ 5 ans. Le sang a été prélevé dans la veine carotide à l’aide d’une aiguille de 16 G (coupure de pointe) et d’une seringue d’animaux conscients. - Chauffer le sérum bovin fœtal (FBS) en le plaçant au bain-marie à 56 °C pendant 30 min et tourbillonner par intermittence. Pour plus de précision, placez un thermomètre dans une deuxième bouteille contenant un volume d’eau équivalent et ajoutez-le au bain de chaleur en même temps que la bouteille FBS. Commencez la synchronisation lorsque le thermomètre atteint 56 °C.
- Utiliser une technique aseptique appropriée et une pratique de culture cellulaire pour toutes les étapes ultérieures effectuées dans la culture cellulaire26,27. Vaporiser 70% d’éthanol sur tous les objets et la hotte avant utilisation et nettoyer avec 1% d’hypochlorite de sodium à la fin.
- Compléter le milieu Eagle modifié (DMEM) de Dulbecco en combinant un DMEM élevé en glucose (4,5 g / L) DMEM (89%) avec FBS inactivé par la chaleur (10%) et streptomycine à la pénicilline (1%). Combiner et filtrer à l’aide d’un système de filtration sous vide stérile (taille des pores de 0,22 μm, membrane de polyéthersulfone) (voir tableau des matériaux). Conserver le DMEM complet enveloppé dans du papier d’aluminium à 4 °C.
- Établir des cellules HT1080 (voir Tableau des matériaux) et passer dans une fiole carrée de 75 cm2 comme décrit à la référence 28. Créez plusieurs stocks congelés de cellules. N’utilisez pas de cellules après 20 passages, car un passage supplémentaire peut influencer les résultats du test.
2. Jour 1 - Placage des cellules
- Passage des cellules HT1080 lorsqu’elles atteignent ~ 80% de confluence.
- DMEM complet préchauffé (préparé à l’étape 1.4), 0,05 % de trypsine-EDTA et 1x solution saline tamponnée au phosphate (PBS) à 37 °C au bain-marie. Retirez le milieu de croissance des cellules traversées à l’aide d’un système d’aspiration.
REMARQUE: Toute aspiration dans ce protocole utilise un système de vide avec un tube attaché à une pipette sérologique stérile de 5 mL. - Laver les cellules dans 10 mL de PBS préchauffé (37 °C) et trypsiniser les cellules pendant 3 à 4 min dans 4 mL de trypsine-EDTA préchauffée à 0,05 % pour détacher les cellules de la fiole.
- Inactiver la trypsine en ajoutant 6 mL de DMEM complet préchauffé et pipeter les cellules dans un tube de 50 mL. Calculer le nombre et la concentration de cellules viables à l’aide d’un hémocytomètre et de la méthode d’exclusion du bleu de trypan29.
- Diluer les cellules à une concentration de 1 x 105 cellules/mL dans du DMEM complet préchauffé. Ensemencez 100 μL de cellules/puits dans des plaques claires à fond plat de 96 puits (1 x 104 cellules par puits). Incuber la plaque à 37 °C, 5% de dioxyde de carbone (CO2) pendant la nuit pendant 16-22 h.
3. Jour 2 - Infecter les cellules
- Retirez les plaques de l’incubateur et utilisez un microscope optique pour confirmer que les cellules sont dispersées uniformément dans les puits et que la confluence est d’environ 50%. Si les cellules ne se trouvent pas dans une plage de confluence de 45 % à 55 %, répétez le protocole du « Jour 1 » et ajustez la concentration cellulaire initiale en conséquence.
- Générer des dilutions en série des échantillons de sérum d’intérêt dans des tubes de microcentrifugation de 1,5 mL en utilisant du DMEM complet préchauffé comme diluant. Le tableau 1 illustre la génération d’une cascade de dilution pour les échantillons triples.
- Pour effectuer le dosage en triplicate, préparer un 7,5 x 106 génomes vectoriels (vg)/μL de solution de travail d’AAV6-hPLAP (voir Tableau des matériaux) en diluant une solution mère de virus dans 1x PBS.
- Ajouter 66 μL de la solution de travail du virus 7,5 x 106 vg/μL à chaque tube contenant 264 μL de dilution sérique/milieu (330 μL de volume total/dilution, voir tableau 1).
REMARQUE: Il s’agit d’un test robuste qui ne nécessite pas de conditions de culture parfaites. Cependant, pour quantifier avec précision et s’assurer que chaque essai est fiable, il est nécessaire d’inclure les éléments suivants: (1) un contrôle du virus et du milieu uniquement, (2) un contrôle du milieu uniquement et (3) un échantillon de contrôle positif NAb sur toutes les plaques dans les mêmes conditions expérimentales. Le volume décrit (330 μL) représente des échantillons triples +10% du mélange sérum et virus. L’exécution de répétitions est fortement recommandée pour la détermination précise de l’activité de neutralisation.
- Mélanger les dilutions virus/sérum par pipetage et placer les tubes contenant les mélanges virus/sérum dans un incubateur à 37 °C, 5 % de CO2 pendant 30 min pour permettre une neutralisation potentielle.
- Pipette 100 μL du mélange virus/sérum dans chaque puits sur la plaque de 96 puits contenant 1 x 104 cellules/puits pour chaque dilution.
REMARQUE: Cela générera une concentration virale finale de 15k virus / multiplicité cellulaire de l’infection (MOI) dans chaque puits. Le tableau 2 fournit un exemple de disposition de plaque d’échantillonnage de 96 puits pour évaluer les échantillons à une dilution de 1/512. - Envelopper la plaque de 96 puits contenant des cellules, du sérum et de l’AAV-hPLAP dans une feuille et placer dans un incubateur à 37 °C, 5% de CO2 pendant la nuit pendant 16-24 h pour permettre à l’AAV d’entrer dans les cellules.
| Étiquette de cascade de dilution | Dilution | 3 x échantillon (240 μL) + 10 % de volume tampon (24 μL) | Rapport sérum:média |
| Dilution 1 (D1) | 1/2 | 264 μL de sérum 264 μL de milieu | 50:50 |
| Dilution 2 (D2) | 1/4 | 264 μL D1 + 264 μL | 25:75 |
| Dilution 3 (D3) | 1/8 | 264 μL D2 + 264μL média | 12.5:87.5 |
| Dilution 4 (D4) | 1/16 | 264 μL D3 +264 μL média | 6.25:93.75 |
| Dilution 5 (D5) | 1/32 | 264 μL D4 +264 μL média | 3.13:96.87 |
| Dilution 6 (D6) | 1/64 | 264 μL D5 +264 μL média | 1.56:98.44 |
| Dilution 7 (D7) | 1/128 | 264 μL D5 +264 μL média | 0.78:99.22 |
| Dilution 8 (D8) | 1/256 | 264 μL D5 +264 μL média | 0.39:99.61 |
| Dilution 9 (D9) | 1/512 | 264 μL D7 + 264 μL de média | 0.2:99.8 |
| Dilution 10 (D10) | 1/2048 | 132 μL D8 + 396 μL de média | 0.05:99.95 |
| Dilution 11 (D11) | 1/8192 | 132 μL D9 + 396 μL de média | 0.01:99.99 |
| Dilution 12 (D12) | 1/32768 | 132 μL D10 + 396 μL de média | 0.003:99.997 |
Tableau 1 : Volumes de sérum et de diluant nécessaires pour générer des dilutions en série du sérum en triple.
| Échantillon de sérum #1 | Échantillon de sérum #2 | Échantillon de sérum #3 | Mono AB (mAB), commandes et échantillons supplémentaires | |||||||||
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | |
| Un | 1/2 | 1/2 | 1/2 | 1/2 | 1/2 | 1/2 | 1/2 | 1/2 | 1/2 | 50 ng MAb | 50 ng MAb | 50 ng MAb |
| B | 1/4 | 1/4 | 1/4 | 1/4 | 1/4 | 1/4 | 1/4 | 1/4 | 1/4 | 5 ng MAb | 5 ng MAb | 5 ng MAb |
| C | 1/8 | 1/8 | 1/8 | 1/8 | 1/8 | 1/8 | 1/8 | 1/8 | 1/8 | 0,5 ng MAb | 0,5 ng MAb | 0,5 ng MAb |
| D | 1/16 | 1/16 | 1/16 | 1/16 | 1/16 | 1/16 | 1/16 | 1/16 | 1/16 | MO (-C) | MO (-C) | MO (-C) |
| E | 1/32 | 1/32 | 1/32 | 1/32 | 1/32 | 1/32 | 1/32 | 1/32 | 1/32 | VO (+C) | VO (+C) | VO (+C) |
| F | 1/64 | 1/64 | 1/64 | 1/64 | 1/64 | 1/64 | 1/64 | 1/64 | 1/64 | Exemple #1 1/512 | Exemple #1 1/512 | Exemple #1 1/512 |
| G | 1/256 | 1/256 | 1/256 | 1/256 | 1/256 | 1/256 | 1/256 | 1/256 | 1/256 | Exemple #2 1/512 | Exemple #2 1/512 | Exemple #2 1/512 |
| H | 1/512 | 1/512 | 1/512 | 1/512 | 1/512 | 1/512 | 1/512 | 1/512 | 1/512 | Exemple #3 1/512 | Exemple #3 1/512 | Exemple #3 1/512 |
Tableau 2 : Exemple de disposition de plaques à 96 puits pour l’évaluation d’échantillons de sérum naïfs dans des dilutions allant de 1/2 à 1/512. Des dilutions plus élevées sont incorporées dans le test si l’on évalue un échantillon connu pour être positif pour les NAbs AAV (échantillons post-administration) ou si un titre plus élevé est nécessaire. MO (-C) : Contrôle des supports uniquement. VO (+C) : Contrôle des virus et des médias uniquement. mAb : Anticorps monoclonal contre l’AAV (NAb positif témoin).
4. Jour 3 - Fixation et ajout de substrat aux cellules
- Préchauffez une aliquote de 1x PBS à 37 °C (~25 mL/plaque de 96 puits). Refroidir les aliquotes séparées de PBS (~25 mL/plaque de 96 puits) et de H2O double distillé (DDW, ~25 mL/plaque de 96 puits) à 4 °C. Dissoudre une pastille de BCIP/NBT (voir Tableau des matériaux) dans 10 mL de DDW dans un tube de centrifugeuse conique de 50 mL par vortex (10 mL suffisent pour 2 x 96 plaques de puits).
- Aspirer le milieu des puits de la plaque de 96 puits à l’aide d’une pipette sérologique ou similaire fixée à un système d’aspiration à base d’aspiration ou à un aspirateur de hotte. Placez doucement l’extrémité de la pipette sérologique dans le puits et retirez le milieu en prenant soin de ne pas perturber les cellules adhérentes.
- Ajouter 50 μL de RT 4 % de PFA à chaque puits à l’aide d’une pipette. Enveloppez la plaque dans du papier d’aluminium et laissez-la à TA pendant 10 minutes pour fixer les cellules.
ATTENTION : Le paraformaldéhyde (PFA) est probablement cancérogène et toxique par contact cutané, visuel ou par inhalation. Manipuler dans une hotte avec un équipement de protection individuelle approprié ainsi qu’un masque facial. Faire du PFA frais à 4 % dilué dans du PBS (~ 7 mL requis par plaque de 96 puits).
- Ajouter 50 μL de RT 4 % de PFA à chaque puits à l’aide d’une pipette. Enveloppez la plaque dans du papier d’aluminium et laissez-la à TA pendant 10 minutes pour fixer les cellules.
- Lavez et aspirez les cellules avec 200 μL de RT 1x PBS. Répétez cette étape deux fois.
REMARQUE: Une pipette multicanal est une option efficace pour les étapes de pipetage. - Pipette 200 μL de PBS préchauffé dans chaque puits, envelopper la plaque dans du papier d’aluminium et incuber à 65 °C pendant 90 min pour dénaturer l’activité endogène de la phosphatase alcaline30.
- Aspirer les puits et laver les cellules avec 200 μL de PBS froid (4 °C). Aspirer à nouveau, laver dans 200 μl de DDW froid et aspirer à nouveau.
- Pipette 50 μL du BCIP/NBT dissous (préparé à l’étape 4.1) dans chaque puits.
- Envelopper la plaque dans du papier d’aluminium et incuber à RT pendant 2-24 h.
REMARQUE: Soyez cohérent avec le temps d’incubation entre les exécutions; la flexibilité temporelle permet aux utilisateurs de photographier les puits le jour 3 ou le lendemain. - À l’aide d’un appareil photo au microscope optique, prenez des photos de chaque puits à l’aide d’un objectif 4x, en veillant à ce que la même exposition, le même équilibrage des blancs et les mêmes réglages de lumière soient utilisés de manière cohérente pour tous les tests effectués.
- Positionnez chaque puits de manière identique et assurez-vous que les bords du puits ne sont pas visibles sur les photos. Enregistrez des photos au format TIF ou similaire.
REMARQUE : Les réglages spécifiques varient d’un microscope à l’autre, mais la quantification sera plus efficace si l’éclairage de fond est élevé et constant dans l’ensemble des puits (Figure 1B).
- Positionnez chaque puits de manière identique et assurez-vous que les bords du puits ne sont pas visibles sur les photos. Enregistrez des photos au format TIF ou similaire.
5. Quantification pour déterminer l’activité de neutralisation à l’aide d’ImageJ
- Téléchargez et installez le logiciel gratuit « ImageJ » (voir Tableau des matériaux).
- Ouvrez l’image à analyser dans ImageJ en sélectionnant Fichier > Ouvrir (Figure 2).
- Si vous utilisez des images colorées, convertissez-les en niveaux de gris en sélectionnant Image > Type > 8 bits.
- Cliquez sur Image > Ajuster > seuil. Ajustez le seuil jusqu’à ce que toutes les zones colorées soient colorées en rouge, mais pas l’arrière-plan. Lors de l’ajout de NBT / BCIP, le produit coloré se déposera dans la zone autour des cellules exprimant hPLAP.
REMARQUE: Il est recommandé d’utiliser le même paramètre de seuil pour toutes les images capturées sur la même plaque. - Cliquez sur Analyser > Définir les mesures et cochez les cases à cocher Zone, Limite au seuil, Fraction de surface et Afficher et cliquez sur OK.
- Pour déterminer la lecture du signal (pourcentage de coloration) d’un puits donné, cliquez sur Analyser > Mesurer. La colonne '% Area' de la fenêtre contextuelle affiche la lecture du signal.
- Effectuer la quantification pour tous les réplicats d’échantillons. Exclure les puits contaminés, les puits présentant une distribution cellulaire inégale ou les puits dont la densité cellulaire ou l’éclairage varie.
REMARQUE : Voir la figure supplémentaire 1 pour des exemples de puits dont l’exclusion devrait être envisagée. En règle générale, 3 à 4 puits peuvent nécessiter une exclusion d’une plaque de 96 puits. La figure 2 fournit une représentation visuelle du processus de quantification à l’aide d’ImageJ.
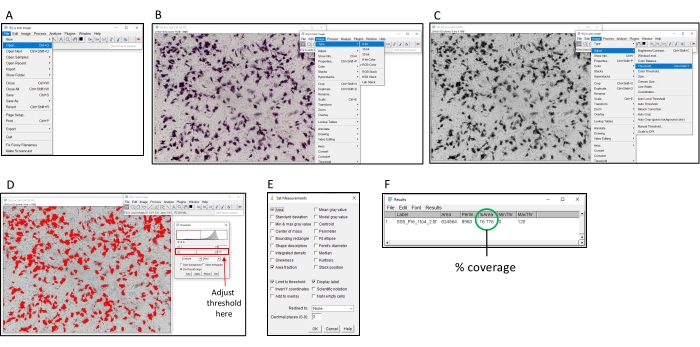
Figure 2 : Étapes permettant de déterminer le pourcentage de coloration à l’aide du logiciel ImageJ. (A) Ouvrez l’image à analyser avec le logiciel ImageJ. (B) Convertissez l’image en niveaux de gris 8 bits. (C) Ouvrez la fenêtre de seuil. (D) Ajustez le seuil maximal pour que toutes les zones colorées soient couvertes, mais pas la zone d’arrière-plan (ce seuil doit être cohérent sur une plaque entière). (E) Sélectionnez la dropbox 'Analyser', cliquez sur 'Définir les mesures' et cochez 'Zone', 'Fraction de surface', 'Seuil limite' et 'Afficher l’étiquette', puis cliquez sur 'OK'. (F) Cliquez sur « Mesurer » pour mesurer la zone couverte. La zone % indique la proportion de l’image qui a été colorée. Cela peut ensuite être utilisé avec les échantillons de contrôle pour déterminer le titre TI50. Veuillez cliquer ici pour voir une version agrandie de cette figure.
6. Détermination du titre d’inhibition de la transduction (TI50)
- Déterminez la lecture moyenne des réplications (à l’aide des étapes décrites à l’étape 5) pour les éléments suivants : (1) Contrôle du média uniquement (lecture du signal de référence). (2) Contrôle du virus + média uniquement (lecture maximale du signal). (3) Virus + échantillons de sérum d’intérêt.
- Calculez le pourcentage d’inhibition à l’aide de la formule suivante :
100 - [(Lecture du signal de l’échantillon d’essai (virus + échantillon sérique d’intérêt) - lecture du signal de base (contrôle du média uniquement)) / (lecture maximale du signal (média et virus uniquement) - lecture du signal de base) x 100] = % Inhibition de la transduction13. - Calculer le pourcentage d’inhibition de la transduction à partir de tous les réplicats de chaque dilution pour tous les échantillons en utilisant la formule indiquée au point 6.2. Déterminer l’inhibition moyenne de la transduction entre les répétitions techniques pour chaque dilution pour tous les échantillons et témoins.
- Calculer le titre d’inhibition de la transduction à 50 % (titre TI50 ) d’un échantillon d’intérêt en déterminant la dilution la plus faible de l’échantillon qui produit une inhibition de transduction de 50 % ou plus de l’activité hPLAP. Par exemple, si une dilution de 1/8 d’un échantillon présente une inhibition de transduction supérieure à 50 % d’après le calcul effectué au point 6.2 (et qu’une dilution de 1/4 ne le fait pas), indiquez que le titre TI50 est de 1/8.
7. Détermination des particules d’AAV neutralisées
- Calculer le nombre de particules DVA neutralisées par μL de sérum pour un échantillon donné en utilisant la formule suivante :
((MOI x nombre de cellules/puits) / (volume de sérum / facteur de dilution du titre TI50 )) / 2 = particules AAV neutralisées / μL de sérum9.
NOTE: La division par 2 représente le TI50 mesurant 50% des particules neutralisées. Pour un échantillon qui donne un titre TI50 de 1/4 (25% de sérum, 75% de diluant) dans lequel le dosage a utilisé 80 μL de sérum non dilué et un MOI de 15k plaqué sur 1 x 104 cellules, le calcul suivant serait utilisé: ((15000 x 10000) / (80/4)) / 2 = 3,75x106 particules neutralisées / μL de sérum.
Résultats
Test de transduction pour établir le dosage viral optimal pour la couverture des plaques
Les cellules HT1080, une lignée cellulaire de fibrosarcome bien établie, ont été sélectionnées pour ce test. Une concentration de 1 x 104 cellules HT1080 / puits a fourni ~ 50% de confluence cellulaire dans chaque puits d’une plaque de 96 puits. Pour déterminer la concentration virale optimale pour le dosage, un rAAV codant pour un gène rapporteur hPLAP (phosphatase alcaline placentaire humai...
Discussion
Ce rapport décrit un test colorimétrique qui évalue l’étendue de la neutralisation de l’AAV dans un échantillon de sérum donné en évaluant une réaction chromogène correspondant au degré de transduction virale in vitro. Le développement du protocole était basé sur la réaction chromogène connue entre l’enzyme phosphatase alcaline et nbT / BCIP, qui a été largement utilisée comme outil de coloration pour la détection de cibles protéiques dans des applications telles que l’immunohistochi...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Cette étude a été financée par une subvention de projet du Conseil national de la santé et de la recherche médicale à JRM et CJT (ID 1163732) et en partie par le programme de soutien à l’infrastructure opérationnelle du gouvernement victorien. SB est soutenu par une bourse de doctorat conjointe baker Heart and Diabetes Institute-La Trobe University. KLW est soutenu par la Shine On Foundation et une future bourse Future Leader de la National Heart Foundation of Australia (ID 102539). JRM est soutenu par une bourse de recherche principale du Conseil national de la santé et de la recherche médicale (ID 1078985).
matériels
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin/EDTA | Gibco | 25300-054 | |
| 50 mL conical centrifuge tube | Falcon | 14-432-22 | Or equivalent |
| 75 cm2 square flasks | Falcon | 353136 | Or equivalent |
| 96 well flat bottomed plate | Falcon | 353072 | |
| AAV6-CMV-hPLAP Vector | Muscle Research & Therapeutics Lab (University of Melbourne, Australia) AAV6-CMV-hPLAP can be provided upon request. | ||
| Aluminium foil | |||
| Anti-AAV6 (intact particle) mouse monoclonal antibody, (ADK6) | PROGEN | 610159 | Positive control monoclonal antibody |
| BCIP/NBT | SIGMAFAST | B5655 | |
| Cell and tissue culture safety cabinet | |||
| Electronic Pipette | 5 & 10 mL stripette inserts | ||
| Fetal Bovine Serum | Gibco | 10099-141 | |
| Haemocytometer | |||
| High glucose Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965118 | |
| HT1080 cells | ATCC | ||
| ImageJ Software | Freely available: https://imagej.nih.gov/ij/download.html | ||
| Incubator | 37 °C, 5% CO2 | ||
| Light microscope with camera | Capable of taking photos with a 4x objective lens | ||
| Oven | For a 65 °C incubation | ||
| Paraformaldehyde | MERCK | 30525-89-4 | |
| Penicillin Streptomycin | Gibco | 15140-122 | |
| Phosphate buffered saline | |||
| Pipettes and tips | 20 μL, 200 μL & 1 mL single pipettes and tips & 200 μL multichannel pipette | ||
| Stericup quick release filter | Millipore | S2GPU10RE | Used for combining media reagents |
| Trypan blue solution | Sigma-Aldrich | T8154 | |
| VACUETTE TUBE 8 ml CAT Serum Separator Clot Activator | Greiner BIO-ONE | 455071 | Used for serum collection & processing from sheep |
| Water bath |
Références
- Bass-Stringer, S., et al. Adeno-associated virus gene therapy: Translational progress and future prospects in the treatment of heart failure. Heart, Lung and Circulation. 27 (11), 1285-1300 (2018).
- Casey, G. A., Papp, K. M., MacDonald, I. M. Ocular gene therapy with adeno-associated virus vectors: current outlook for patients and researchers. Journal of Ophthalmic and Vision Research. 15 (3), 396-399 (2020).
- Lykken, E. A., Shyng, C., Edwards, R. J., Rozenberg, A., Gray, S. J. Recent progress and considerations for AAV gene therapies targeting the central nervous system. Journal of Neurodevelopmental Disorders. 10 (1), 16 (2018).
- Guggino, W. B., Cebotaru, L. Adeno-Associated Virus (AAV) gene therapy for cystic fibrosis: Current barriers and recent developments. Expert Opinion on Biological Therapy. 17 (10), 1265-1273 (2017).
- Perrin, G. Q., Herzog, R. W., Markusic, D. M. Update on clinical gene therapy for hemophilia. Blood. 133 (5), 407-414 (2019).
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature Reviews Drug Discovery. 18 (5), 358-378 (2019).
- Kuzmin, D. A., et al. The clinical landscape for AAV gene therapies. Nature Reviews Drug Discovery. 20 (3), 173-174 (2021).
- Russell, S., et al. Efficacy and safety of voretigene neparvovec (AAV2-hRPE65v2) in patients with RPE65-mediated inherited retinal dystrophy: A randomised, controlled, open-label, phase 3 trial. Lancet. 390 (10097), 849-860 (2017).
- Weber, T. Anti-AAV Antibodies in AAV gene therapy: Current challenges and possible solutions. Frontiers in Immunology. 12, 658399 (2021).
- Weeks, K. L., et al. Phosphoinositide 3-kinase p110alpha is a master regulator of exercise-induced cardioprotection and PI3K gene therapy rescues cardiac dysfunction. Circulation: Heart Failure. 5 (4), 523-534 (2012).
- Gregorevic, P., et al. Systemic delivery of genes to striated muscles using adeno-associated viral vectors. Nature Medicine. 10 (8), 828-834 (2004).
- Bernardo, B. C., et al. Gene delivery of medium chain acyl-coenzyme A dehydrogenase induces physiological cardiac hypertrophy and protects against pathological remodelling. Clinical Science (London). 132 (3), 381-397 (2018).
- Meliani, A., et al. Determination of anti-adeno-associated virus vector neutralizing antibody titer with an in vitro reporter system. Human Gene Therapy Methods. 26 (2), 45-53 (2015).
- Falese, L., et al. Strategy to detect pre-existing immunity to AAV gene therapy. Gene Therapy. 24 (12), 768-778 (2017).
- Wang, D., et al. Adeno-Associated virus neutralizing antibodies in large animals and their impact on brain intraparenchymal gene transfer. Molecular Therapy - Methods & Clinical Development. 11, 65-72 (2018).
- Wang, M., et al. Prediction of adeno-associated virus neutralizing antibody activity for clinical application. Gene Therapy. 22 (12), 984-992 (2015).
- Kruzik, A., et al. Detection of biologically relevant low-titer neutralizing antibodies against adeno-associated virus require sensitive in vitro assays. Human Gene Therapy Methods. 30 (2), 35-43 (2019).
- Lehtoranta, L., Villberg, A., Santanen, R., Ziegler, T. A novel, colorimetric neutralization assay for measuring antibodies to influenza viruses. Journal of Virological Methods. 159 (2), 271-276 (2009).
- Johnston, P. B., Grayston, J. T., Loosli, C. G. Adenovirus neutralizing antibody determination by colorimetric assay. Proceedings of the Society for Experimental Biology and Medicine. 94 (2), 338-343 (1957).
- Xiaoli Zhu, T. G. . Nano-Inspired Biosensors for Protein Assay with Clinical Applications. , 237-264 (2019).
- Jungmann, A., Muller, O., Rapti, K. Cell-based measurement of neutralizing antibodies against adeno-associated virus (AAV). Methods in Molecular Biology. 1521, 109-126 (2017).
- Samineni, S., et al. Optimization, comparison, and application of colorimetric vs. chemiluminescence based indirect sandwich ELISA for measurement of human IL-23. Journal of Immunoassay and Immunochemistry. 27 (2), 183-193 (2006).
- Siddiqui, J., Remick, D. G. Improved sensitivity of colorimetric compared to chemiluminescence ELISAs for cytokine assays. Journal of Immunoassay and Immunochemistry. 24 (3), 273-283 (2003).
- Arnett, A. L., Garikipati, D., Wang, Z., Tapscott, S., Chamberlain, J. S. Immune responses to rAAV6: The Influence of canine parvovirus vaccination and neonatal administration of viral vector. Frontiers in Microbiology. 2, 220 (2011).
- Australian code for the care and use of animals for scientific purposes. National Health and Medical Research Council Available from: https://www.nhmrc.gov.au/about-us/publications/australian-code-care-and-use-animals-scientific-purposes (2013)
- Coecke, S., et al. Guidance on good cell culture practice. A report of the second ECVAM task force on good cell culture practice. Alternatives to Laboratory Animals. 33 (3), 261-287 (2005).
- Journal of Visualized Experiments. General Laboratory Techniques. Journal of Visualized Experiments Database. , (2018).
- AAV-HT1080 Cells. Stratagene Available from: https://www.chem-agilent.com/pdf/strata/240109.pdf (2003)
- Strober, W. Trypan blue exclusion test of cell viability. Current Protocols in Immunology. 111 (3), 1-3 (2015).
- Bieber, S., et al. Extracorporeal delivery of rAAV with metabolic exchange and oxygenation. Scientific Reports. 3, 1538 (2013).
- Winbanks, C. E., Beyer, C., Qian, H., Gregorevic, P. Transduction of skeletal muscles with common reporter genes can promote muscle fiber degeneration and inflammation. PLoS One. 7 (12), 51627 (2012).
- Thomas, C. J., et al. Evidence that the MEK/ERK but not the PI3K/Akt pathway is required for protection from myocardial ischemia-reperfusion injury by 3',4'-dihydroxyflavonol. European Journal of Pharmacology. 758, 53-59 (2015).
- Barger, A., et al. Use of alkaline phosphatase staining to differentiate canine osteosarcoma from other vimentin-positive tumors. Veterinary Pathology. 42 (2), 161-165 (2005).
- Gregorevic, P., et al. Evaluation of vascular delivery methodologies to enhance rAAV6-mediated gene transfer to canine striated musculature. Molecular Therapy. 17 (8), 1427-1433 (2009).
- Sharma, A., Ghosh, A., Hansen, E. T., Newman, J. M., Mohan, R. R. Transduction efficiency of AAV 2/6, 2/8 and 2/9 vectors for delivering genes in human corneal fibroblasts. Brain Research Bulletin. 81 (2-3), 273-278 (2010).
- Smejkal, G. B., Kaul, C. A. Stability of nitroblue tetrazolium-based alkaline phosphatase substrates. Journal of Histochemistry & Cytochemistry. 49 (9), 1189-1190 (2001).
- Falese, L., et al. Strategy to detect pre-existing immunity to AAV gene therapy. Gene Therapy. 24 (12), 768-778 (2017).
- Orlowski, A., et al. Successful transduction with AAV Vectors after selective depletion of anti-aav antibodies by immunoadsorption. Molecular Therapy - Methods & Clinical Development. 16, 192-203 (2020).
- Goossens, K., et al. Differential microRNA expression analysis in blastocysts by whole mount in situ hybridization and reverse transcription quantitative polymerase chain reaction on laser capture microdissection samples. Analytical Biochemistry. 423 (1), 93-101 (2012).
- Entrican, G., Wattegedera, S. R., Griffiths, D. J. Exploiting ovine immunology to improve the relevance of biomedical models. Molecular Immunology. 66 (1), 68-77 (2015).
- Walters, E. M., Prather, R. S. Advancing swine models for human health and diseases. Molecular Medicine. 110 (3), 212-215 (2013).
- Rapti, K., et al. Neutralizing antibodies against AAV serotypes 1, 2, 6, and 9 in sera of commonly used animal models. Molecular Therapy. 20 (1), 73-83 (2012).
- Tellez, J., et al. Characterization of naturally-occurring humoral immunity to AAV in sheep. PLoS One. 8 (9), 75142 (2013).
- Gupta, S., et al. Recommendations for the validation of cell-based assays used for the detection of neutralizing antibody immune responses elicited against biological therapeutics. Journal of Pharmaceutical and Biomedical Analysis. 55 (5), 878-888 (2011).
- Gupta, S., et al. Recommendations for the design, optimization, and qualification of cell-based assays used for the detection of neutralizing antibody responses elicited to biological therapeutics. Journal of Immunological Methods. 321 (1-2), 1-18 (2007).
- Shankar, G., et al. Recommendations for the validation of immunoassays used for detection of host antibodies against biotechnology products. Journal of Pharmaceutical and Biomedical Analysis. 48 (5), 1267-1281 (2008).
- U.S. Department of Health and Human Services Food and Drug Administration. Center for Drug Evaluation and Research (CDER). Immunogenicity Testing of Therapeutic Protein Products — Developing and Validating Assays for Anti-Drug Antibody Detection. U.S. Department of Health and Human Services Food and Drug Administration. , (2019).
- Baatartsogt, N., et al. A sensitive and reproducible cell-based assay via secNanoLuc to detect neutralizing antibody against adeno-associated virus vector capsid. Molecular Therapy - Methods & Clinical Development. 22, 162-171 (2021).
- Watano, R., Ohmori, T., Hishikawa, S., Sakata, A., Mizukami, H. Utility of micro mini pigs for evaluating liver-mediated gene expression in the presence of neutralizing antibody against vector capsid. Gene Therapy. 27 (9), 427-434 (2020).
- Majowicz, A., et al. Therapeutic hFIX activity achieved after single AAV5-hFIX treatment in Hemophilia B patients and NHPs with pre-existing anti-AAV5 NABs. Molecular Therapy - Methods & Clinical Development. 14, 27-36 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon