Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein effizientes Clearing-Protokoll zur Untersuchung der Saatgutentwicklung bei Tomaten (Solanum lycopersicum L.)
In diesem Artikel
Zusammenfassung
Der Tomatensamen ist ein wichtiges Modell für das Studium der Genetik und Entwicklungsbiologie während der Pflanzenvermehrung. Dieses Protokoll ist nützlich, um Tomatensamen in verschiedenen Entwicklungsstadien zu entfernen, um die feinere embryonale Struktur zu beobachten.
Zusammenfassung
Tomate (Solanum lycopersicum L.) ist eine der wichtigsten Cash Crops weltweit. Der Tomatensamen ist ein wichtiges Modell für das Studium der Genetik und Entwicklungsbiologie während der Pflanzenvermehrung. Die Visualisierung einer feineren embryonalen Struktur innerhalb eines Tomatensamens wird oft durch Samenhüllenschleim, mehrzellige Schichtintegument und ein dickwandiges Endosperm behindert, das durch mühsames Einbetten aufgelöst werden muss. Eine einfachere Alternative ist der Einsatz von Gewebereinigungstechniken, die das Saatgut mit chemischen Mitteln nahezu transparent machen. Obwohl herkömmliche Clearingverfahren einen tiefen Einblick in kleinere Samen mit dünnerer Samenhülle ermöglichen, ist die Klärung von Tomatensamen vor allem in den späten Entwicklungsstadien eine technische Herausforderung.
Hier wird ein schnelles und arbeitssparendes Clearing-Protokoll vorgestellt, um die Entwicklung von Tomatensamen 3 bis 23 Tage nach der Blüte zu beobachten, wenn die embryonale Morphologie fast abgeschlossen ist. Diese Methode kombiniert eine in Arabidopsis weit verbreitete Chloralhydrat-basierte Reinigungslösung mit anderen Modifikationen, einschließlich des Wegfalls der Formalin-Aceto-Alkohol (FAA) -Fixierung, der Zugabe von Natriumhypochlorit-Behandlung von Samen, der Entfernung der erweichten Samenschichtschleimhaut sowie des Waschens und der Vakuumbehandlung. Diese Methode kann zur effizienten Reinigung von Tomatensamen in verschiedenen Entwicklungsstadien angewendet werden und ist nützlich bei der vollständigen Überwachung des Entwicklungsprozesses von mutierten Samen mit guter räumlicher Auflösung. Dieses Clearing-Protokoll kann auch auf die Tiefenbildgebung anderer kommerziell wichtiger Arten in den Solanaceae angewendet werden.
Einleitung
Tomate (S. lycopersicum L.) ist eine der wichtigsten Gemüsekulturen der Welt, mit einer Produktion von 186,8 Millionen Tonnen fleischigen Früchten von 5,1 Millionen Hektar im Jahr 20201. Es gehört zur großen Familie der Solanaceae mit etwa 2.716 Arten2, darunter viele kommerziell wichtige Kulturen wie Auberginen, Paprika, Kartoffeln und Tabak. Die Kulturtomate ist eine diploide Art (2n = 2x = 24) mit einer Genomgröße von ca. 900Mb3. Seit langem werden große Anstrengungen unternommen, um Tomatendomestizierung und -züchtung zu züchten, indem wünschenswerte Merkmale aus wilden Solanum spp. Es gibt über 5.000 Tomatenakzessionen, die im Tomato Genetics Resource Center aufgeführt sind, und mehr als 80.000 Keimplasma von Tomaten werden weltweit gelagert4. Die Tomatenpflanze ist mehrjährig im Gewächshaus und vermehrt sich durch Samen. Ein reifer Tomatensamen besteht aus drei Hauptkompartimenten: einem ausgewachsenen Embryo, einem zellulären Endosperm und einer harten Samenschale 5,6 (Abbildung 1A). Nach doppelter Befruchtung geht die Entwicklung von zellulärem Endosperm der Entwicklung von Zygoten voraus. Bei ~5-6 Tagen nach der Blüte (DAF) wird erstmals ein zweizelliger Proembryo beobachtet, wenn das Endosperm aus sechs bis acht Kernen besteht7. Bei Solanum pimpinellifolium nähert sich der Embryo nach 20 DAF seiner endgültigen Größe, und die Samen sind nach 32 DAF8 für die Keimung geeignet. Wenn sich der Embryo entwickelt, wird das Endosperm allmählich absorbiert und nur eine kleine Menge Endosperm verbleibt im Samen. Das restliche Endosperm besteht aus mikropylarem Endosperm, das die Radikelspitze umgibt, und seitlichem Endosperm im Rest des Samens 9,10. Die äußere Samenhülle wird aus verdickter und verholzter äußerer Epidermis des Integuments entwickelt, und mit den abgestorbenen Schichten von Integumentresten bilden sie eine harte Schale, um den Embryo und das Endosperm zu schützen5.
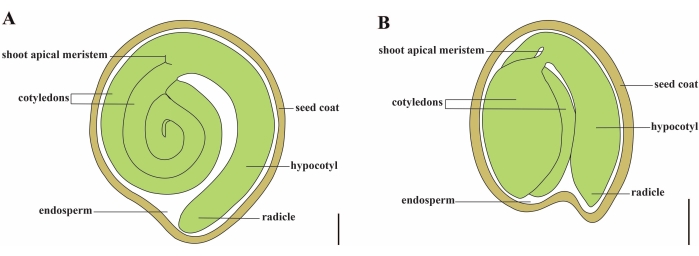
Abbildung 1: Schematische Darstellung eines reifen Samens in Solanum lycopersicum und Arabidopsis thaliana. (A) Längsanatomie eines reifen Tomatensamens. (B) Längsanatomie eines reifen Arabidopsis-Samens. Ein reifer Tomatensamen ist etwa 70-mal größer als ein Arabidopsis-Samen. Maßstabsbalken = (A) 400 μm, (B) 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Die Produktion hochwertiger Tomatensamen hängt von der Koordination zwischen dem Embryo, dem Endosperm und den mütterlichen Samenbestandteilen ab11. Die Analyse von Schlüsselgenen und Netzwerken in der Samenentwicklung erfordert eine tiefe und vollständige phänotypische Aufzeichnung von mutierten Samen. Konventionelle Einbettungsschnitttechniken, wie der Halbdünnschliff und der Paraffinschnitt, werden häufig auf Tomatensamen angewendet, um die lokalen und feineren Strukturen des Embryoszu beobachten 12,13,14,15. Die Analyse der Seed-Entwicklung aus dünnen Schnitten ist jedoch in der Regel mühsam und es fehlt die räumliche Auflösung der z-Achse. Im Vergleich dazu ist die Gewebereinigung eine schnelle und effiziente Methode, um das Entwicklungsstadium von embryonalen Defekten zu bestimmen, die am wahrscheinlichsten auftreten16. Das Clearing-Verfahren verringert die Undurchsichtigkeit des inneren Gewebes durch Homogenisierung des Brechungsindex mit einem oder mehreren biochemischen Mitteln16. Die gesamte Gewebereinigung ermöglicht die Beobachtung einer pflanzlichen Gewebestruktur, ohne ihre Integrität zu zerstören, und die Kombination von Clearing-Technologie und dreidimensionaler Bildgebung ist zu einer idealen Lösung geworden, um Informationen über die Morphologie und den Entwicklungszustand eines Pflanzenorgans zu erhalten17,18. Im Laufe der Jahre wurden Samenreinigungstechniken bei verschiedenen Pflanzenarten eingesetzt, darunter Arabidopsis thaliana, Hordeum vulgare und Beta vulgaris 19,20,21,22,23. Unter diesen war die Whole-mount-Eizellenreinigungstechnologie ein effizienter Ansatz zur Untersuchung der Samenentwicklung von Arabidopsis, aufgrund ihrer geringen Größe, 4-5 Schichten der Samenhüllenzelle und des nuklearen Endosperms24,25. Mit der kontinuierlichen Aktualisierung verschiedener Clearing-Mischungen, wie dem Auftauchen der Hoyer-Lösung26, wurden die inneren Strukturen der Gerstenzelle mit einem hohen Grad an Klarheit abgebildet, obwohl ihr Endosperm den Großteil der Samen ausmacht. Die Embryogenese von Zuckerrüben kann durch Reinigung in Kombination mit Vakuumbehandlung und Erweichung mit Salzsäurebeobachtet werden 19. Im Gegensatz zu den oben genannten Arten wurden jedoch keine embryologischen Beobachtungen durch Clearing-Protokolle in Tomatensamen berichtet. Dies verhindert eine detaillierte Untersuchung der Embryonal- und Samenentwicklung von Tomaten.
Chloralhydrat wird üblicherweise als Reinigungslösung verwendet, die es ermöglicht, die eingetauchten Gewebe und Zellen auf verschiedenen optischen Ebenen darzustellen und die Zellen oder Gewebekomponenten im Wesentlichen zu erhalten 27,28,29. Das auf Chloralhydrat basierende Clearing-Protokoll wurde erfolgreich für die gesamte Reinigung von Samen verwendet, um den Embryo und das Endosperm von Arabidopsis21,28 zu beobachten. Diese Clearing-Lösung ist jedoch nicht effizient bei der Reinigung von Tomatensamen, die undurchlässiger sind als Arabidopsis-Samen. Zu den physischen Barrieren gehören: (1) das Tomatenintegument hat fast 20 Zellschichten bei 3 bis 15 DAF 30,31, (2) das Tomatenendosperm ist zellulär, nicht vom Kerntyp 32, und (3) Tomatensamen sind etwa70-mal größer in der Größe33,34 und (4) produzieren große Mengen an Samenmantelschleimstoffen, die das Eindringen von Reinigungsreagenzien blockieren und die Visualisierung von Embryozellen beeinträchtigen.
Daher stellt dieser Bericht eine optimierte Chloralhydrat-basierte Clearing-Methode für die vollständige Reinigung von Tomatensamen in verschiedenen Stadien vor, die eine tiefe Bildgebung des Embryo-Entwicklungsprozesses ermöglicht (Abbildung 2).
Protokoll
1. Vorbereitung von Lösungen
- Bereiten Sie das FAA-Fixiermittel durch Zugabe von 2,5 ml 37% Formaldehyd, 2,5 ml Eisessig und 45 ml 70% Ethanol in einem 50-ml-Zentrifugenröhrchen vor. Vortex und lagern Sie es bei 4 °C. FAA Fixiermittel kurz vor Gebrauch frisch zubereiten.
ACHTUNG: 37% Formaldehyd ist ätzend und potenziell krebserregend, wenn es exponiert oder eingeatmet wird. Das Fixiermittel muss in einem Abzug unter Tragen geeigneter persönlicher Schutzausrüstung durchgeführt werden. - Bereiten Sie die Reinigungslösung vor, indem Sie 5 ml 100% Glycerin, 40 g Chloralhydrat und 10 ml destilliertes Wasser in einer 100-ml-Glasflasche hinzufügen, die mit Zinnfolie umwickelt ist. Mit einem Magnetrührer über Nacht bei Raumtemperatur auflösen. Die vorbereitete Lösung kann bis zu 6 Monate bei 4 °C gelagert werden.
ACHTUNG: Chloralhydrat ist krebserregend und riecht stechend. Führen Sie das Experiment in einem Abzug durch und tragen Sie geeignete persönliche Schutzausrüstung. Chloralhydrat lässt sich leicht in der Luft entwässern und sollte nicht in großen Mengen gelagert werden. Die Reinigungslösung kann sich bei Lichteinwirkung zersetzen und sollte von Licht ferngehalten werden. - Bereiten Sie die Desinfektionslösung vor, indem Sie 10 ml 6% Natriumhypochlorit, 40 ml destilliertes Wasser und 50 μL Tween-20 in einem 50-ml-Zentrifugenröhrchen hinzufügen. Vortex und lagern Sie es bei Raumtemperatur. Bereiten Sie die Desinfektionslösung kurz vor Gebrauch frisch zu.
HINWEIS: Der effektive Chlorgehalt von Natriumhypochlorit, der für eine lange Zeit platziert wurde, kann abnehmen, und der Gehalt an 6% Natriumhypochlorit kann entsprechend der tatsächlichen Situation erhöht werden.
2. Saatgutsammlung
- Tomatensamen (S. lycopersicum L. cv. Micro-Tom, siehe Materialtabelle) in 8 cm × 8 cm × 8 cm großen quadratischen Becken mit einer 1:1-Mischung aus Blütennährstofferde und Substrat (v/v) aussäen (siehe Materialtabelle) und wachsen in einem Wachstumsraum mit 16/8 h Hell-Dunkel-Perioden bei 24 ± 2 °C (Tag) und 18 ± 2 °C (Nacht), 60% -70% relative Luftfeuchtigkeit und eine Lichtintensität von 4.000 Lux. Etwa 6 Wochen später traten die Pflanzen in die Blütephase ein.
- Markieren Sie das Blütedatum unabhängiger Blüten an der Anthese (Öffnungswinkel der Blütenblätter beträgt 90°) und notieren Sie den Tag nach der Blüte (DAF).
- Ernten Sie die Früchte von 3 bis 23 DAF-Tomatenpflanzen und legen Sie sie sofort auf Eis. Teilen Sie Früchte (Samen oder Embryonen) von 3 bis 23 DAF in frühe (3-10 DAF), mittlere (11-16 DAF) und späte (17-23 DAF) Früchte (Samen oder Embryonen).
HINWEIS: Sammeln Sie nicht zu viele Früchte auf einmal und stellen Sie sicher, dass die Samen in jeder Frucht innerhalb von 1 Stunde zur weiteren Behandlung entfernt werden. - Für frühe Früchte brechen Sie die Früchte und legen Sie sie auf einen Objektträger und sammeln Sie frische Samen sorgfältig mit einer Präzisionszange unter dem Stereomikroskop (siehe Materialtabelle) bei 1x bis 4-facher Vergrößerung. Für mittlere und späte Früchte brechen Sie die Früchte und sammeln Sie frische Samen direkt mit einer Präzisionszange.
3. Chloralhydrat-basierte Reinigung von Samen
HINWEIS: Konventionelle35 und optimierte Protokolle wurden in dieser Studie hinsichtlich ihrer Saatgutreinigungseffizienz verglichen.
- Clearing mit einem herkömmlichen Protokoll
- Frisches Saatgut (erhalten in Schritt 2.4) sofort in ein 2-ml-Zentrifugenröhrchen mit 1,5 ml FAA-Fixiermittel geben und auf einem Orbitalschüttler (5 U/min, siehe Materialtabelle) 4 h bei Raumtemperatur inkubieren.
- Dehydrieren Sie diese Samen in einer Ethanolserie von 70%, 95% und 100% Ethanol (v / v) für jeweils 1 h auf einem Orbitalschüttler (5 U/min).
- Legen Sie die Samen in 3-5 Tropfen Reinigungslösung (~ 100 μL) auf den Objektträger und bedecken Sie die Probe vorsichtig mit einem Deckglas. Ersetzen Sie den Objektträger durch einen einzelnen konkaven Objektträger (siehe Materialtabelle) für mittlere und späte Samen.
- Bewahren Sie diese Objektträger oder einzelnen konkaven Objektträger bei Raumtemperatur für 30 min (3 DAF), 2 h (6 DAF), 1 Tag (9 DAF), 3 Tage (12 DAF) oder 7 Tage (14 bis 22 DAF) auf, abhängig von den Entwicklungsstadien des Saatguts (je jünger die Materialien, desto schneller die Clearing-Geschwindigkeit).
- Beobachten Sie die Proben mit einem DIC-Mikroskop (Differential Interference Contrast), das mit einer Digitalkamera (siehe Materialtabelle) bei 10-facher, 20-facher und 40-facher Vergrößerung ausgestattet ist. Passen Sie die Durchlichthelligkeit, den DIC-Schieberegler und die Kondensatorblende in Echtzeit für jede Probe an und optimieren Sie sie.
- Clearing mit dem optimierten Protokoll
- Frisches Saatgut (erhalten in Schritt 2.4) wird direkt in ein 2-ml-Zentrifugenröhrchen mit 1,5 ml Desinfektionslösung gegeben (Schritt 1.3).
HINWEIS: Die Anzahl der Samen, die in der 2-ml-Zentrifugenröhre gesammelt werden, variiert je nach Entwicklungsstadium der Samen. Einzelheiten sind in Tabelle 1 aufgeführt. - Inkubieren Sie die Proben auf einem Orbitalschüttler (30 U/min) bei Raumtemperatur für 3 bis 50 Minuten, bis der innerste Saatmantelumriss deutlich sichtbar ist. Entsorgen Sie die Desinfektionslösung.
HINWEIS: Die erforderliche Inkubationszeit hängt vom Saatgutentwicklungsstadium ab (je später die Samen, desto länger ist die Inkubationszeit). Einzelheiten sind in Tabelle 1 aufgeführt. - Bei mittleren und späten Samen die Samen auf einen Objektträger übertragen und den Samenschleim mit einer Pinzette und einer Seziernadel unter einem Stereomikroskop bei 1- bis 4-facher Vergrößerung entfernen. Die Samen werden mit einer Pinzette wieder in das ursprüngliche Zentrifugenröhrchen überführt.
HINWEIS: Dieser Schritt ist für frühe Samen nicht erforderlich. - Waschen Sie die Samen 5x mit 1,5 ml entionisiertem Wasser, jeweils 10 s. Entsorgen Sie das entionisierte Wasser.
- Eine Reinigungslösung von 2 × Volumen der Samen zugeben, gefolgt von einer Vakuumbehandlung für 0 bis 50 min (Tabelle 1) mit einer Vakuumpumpe (siehe Materialtabelle). Die intermittierende Vakuumbehandlung wird mit jeder Vakuumbehandlung für 10 Minuten im Abstand von 10 Minuten durchgeführt.
- Ersetzen Sie durch eine frische Reinigungslösung von 2 × Volumen der Samen. Bewahren Sie das 2-ml-Zentrifugenröhrchen mit den Samen 30 Minuten bis 7 Tage lang bei Raumtemperatur und vor Licht geschützt auf, um die Reinigung zu erleichtern (Tabelle 1). Während der Inkubation wird bei späten Embryonen die Clearing-Lösung täglich durch frische Lösung ersetzt und die Samen 10 min lang einer Vakuumbehandlung unterzogen.
- Bereiten Sie die geklärten Samen auf Objektträgern oder einfach konkaven Objektträgern vor und beobachten Sie sie mit dem DIC-Mikroskop, das mit einer Digitalkamera in 10-facher, 20-facher und 40-facher Vergrößerung ausgestattet ist. Passen Sie die Durchlichthelligkeit, den DIC-Schieberegler und die Kondensatorblende in Echtzeit für jede Probe an und optimieren Sie sie.
- Frisches Saatgut (erhalten in Schritt 2.4) wird direkt in ein 2-ml-Zentrifugenröhrchen mit 1,5 ml Desinfektionslösung gegeben (Schritt 1.3).
Ergebnisse
Wenn Tomatensamen mit einer herkömmlichen Methode wie bei Arabidopsis entfernt wurden, blockierten dichte Endospermzellen die Visualisierung früher Tomatenembryonen bei 3 DAF und 6 DAF (Abbildung 3A,B). Als das Gesamtvolumen des Embryos zunahm, war ein kugelförmiger Embryo bei 9 DAF kaum zu unterscheiden (Abbildung 3C). Als die Samengröße jedoch weiter zunahm, nahm ihre Permeabilität ab, was zu einem unscharfen Herzembryo bei 12 D...
Diskussion
Im Vergleich zum mechanischen Schneiden ist die Clearing-Technologie für die dreidimensionale Bildgebung vorteilhafter, da sie die Integrität von Pflanzengeweben oder Organen bewahrt16. Herkömmliche Clearing-Protokolle sind aufgrund des leichteren Eindringens chemischer Lösungen oft auf kleine Proben beschränkt. Tomatensamen sind eine problematische Probe für die Gewebereinigung, da sie etwa 70-mal größer sind als ein Arabidopsis-Samen und mehr Durchlässigkeitsbarrieren aufweisen...
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Die Autoren danken Dr. Jie Le und Dr. Xiufen Song für ihre hilfreichen Vorschläge zur differentiellen Interferenzkontrastmikroskopie bzw. zur konventionellen Clearing-Methode. Diese Forschung wurde von der National Natural Science Foundation of China (31870299) und der Youth Innovation Promotion Association der Chinesischen Akademie der Wissenschaften finanziert. Abbildung 2 wurde mit BioRender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 1,000 µL pipette | GILSON | FA10006M | |
| 1,000 µL pipette tips | Corning | T-1000-B | |
| 2 ml centrifuge tube | Axygen | MCT-200-C | |
| 37% formaldehyde | DAMAO | 685-2013 | |
| 5,000 µL pipette | Eppendorf | 3120000275 | |
| 5,000 µL pipette tips | biosharp | BS-5000-TL | |
| 50 ml centrifuge tube | Corning | 430829 | |
| Absolute Ethanol | BOYUAN | 678-2002 | |
| Bottle glass | Fisher | FB800-100 | |
| Chloral Hydrate | Meryer | M13315-100G | |
| Coverslip | Leica | 384200 | |
| DIC microscope | Zeiss | Axio Imager A1 | 10x, 20x and 40x magnification |
| Disinfectant | QIKELONGAN | 17-9185 | |
| Dissecting needle | Bioroyee | 17-9140 | |
| Flower nutrient soil | FANGJIE | ||
| Forceps | HAIOU | 4-94 | |
| Glacial Acetic Acid | BOYUAN | 676-2007 | |
| Glycerol | Solarbio | G8190 | |
| Magnetic stirrer | IKA | RET basic | |
| Micro-Tom | Tomato Genetics Resource Center | LA3911 | |
| Orbital shaker | QILINBEIER | QB-206 | |
| Seeding substrate | PINDSTRUP | LV713/018-LV252 | Screening:0-10 mm |
| Single concave slide | HUABODEYI | HBDY1895 | |
| Slide | Leica | 3800381 | |
| Stereomicroscope | Leica | S8 APO | 1x to 4x magnification |
| Tin foil | ZAOWUFANG | 613 | |
| Tween 20 | Sigma | P1379 | |
| Vacuum pump | SHIDING | SHB-III | |
| Vortex meter | Silogex | MX-S |
Referenzen
- . FAOSTAT Available from: https://www.fao.org/faostat/en/#data/QCL (2022)
- Olmstead, R. G., Bohs, L. A summary of molecular systematic research in Solanaceae: 1982-2006. Acta Horticulturae. 745, 255-268 (2007).
- Consortium, T. G. The tomato genome sequence provides insights into fleshy fruit evolution. Nature. 485 (7400), 635-641 (2012).
- Ebert, A. W., Chou, Y. Y. The tomato collection maintained by AVRDC - The World Vegetable Center: composition, germplasm dissemination and use in breeding. Acta Horticulturae. 1101, 169-176 (2015).
- Hilhorst, H., Groot, S., Bino, R. J. The tomato seed as a model system to study seed development and germination. Acta Botanica Neerlandica. 47, 169-183 (1998).
- Chaban, I. A., Gulevich, A. A., Kononenko, N. V., Khaliluev, M. R., Baranova, E. N. Morphological and structural details of tomato seed coat formation: A different functional role of the inner and outer epidermises in unitegmic ovule. Plants-Basel. 11 (9), 1101 (2022).
- Iwahori, S. High temperature injuries in tomato. V. Fertilization and development of embryo with special reference to the abnormalities caused by high temperature. Journal of The Japanese Society for Horticultural Science. 35 (4), 379-386 (1966).
- Xiao, H., et al. Integration of tomato reproductive developmental landmarks and expression profiles, and the effect of SUN. on fruit shape. BMC Plant Biology. 9 (1), 49 (2009).
- Karssen, C. M., Haigh, A. M., Toorn, P., Weges, R., Taylorson, R. B. Physiological mechanisms involved in seed priming. Recent advances in the development and germination of seeds. NATO ASI Series. 187, (1989).
- Nonogaki, H. Seed dormancy and germination-emerging mechanisms and new hypotheses. Frontiers in Plant Science. 5, 233 (2014).
- Doll, N. M., Ingram, G. C. Embryo-endosperm interactions. Annual Review of Plant Biology. 73, 293-321 (2022).
- Serrani, J. C., Fos, M., Atarés, A., García-Martínez, J. L. Effect of gibberellin and auxin on parthenocarpic fruit growth induction in the cv micro-tom of tomato. Journal of Plant Growth Regulation. 26 (3), 211-221 (2007).
- Yang, C., et al. A regulatory gene induces trichome formation and embryo lethality in tomato. Proceedings of the National Academy of Sciences USA. 108 (29), 11836-11841 (2011).
- Goetz, S., et al. Role of cis-12-oxo-phytodienoic acid in tomato embryo development. Plant Physiology. 158 (4), 1715-1727 (2012).
- Ko, H. Y., Ho, L. H., Neuhaus, H. E., Guo, W. J. Transporter SlSWEET15 unloads sucrose from phloem and seed coat for fruit and seed development in tomato. Plant Physiology. 187 (4), 2230-2245 (2021).
- Richardson, D. S., Lichtman, J. W. Clarifying tissue clearing. Cell. 162 (2), 246-257 (2015).
- Kurihara, D., Mizuta, Y., Sato, Y., Higashiyama, T. ClearSee: A rapid optical clearing reagent for whole-plant fluorescence imaging. Development. 142 (23), 4168-4179 (2015).
- Vieites-Prado, A., Renier, N. Tissue clearing and 3D imaging in developmental biology. Development. 148 (18), (2021).
- Kwiatkowska, M., Kadłuczka, D., Wędzony, M., Dedicova, B., Grzebelus, E. Refinement of a clearing protocol to study crassinucellate ovules of the sugar beet (Beta vulgaris L., Amaranthaceae). Plant Methods. 15, 71 (2019).
- Ponitka, A., Ślusarkiewicz-Jarzina, A. Cleared-ovule technique used for rapid access to early embryo development in Secale cereale × Zea mays crosses. Acta Biologica Cracoviensia. Series Botanica. 46, 133-137 (2014).
- Ceccato, L., et al. Maternal control of PIN1 is required for female gametophyte development in Arabidopsis. PLoS One. 8 (6), 66148 (2013).
- Wilkinson, L. G., Tucker, M. R. An optimised clearing protocol for the quantitative assessment of sub-epidermal ovule tissues within whole cereal pistils. Plant Methods. 13, 67 (2017).
- Hedhly, A., Vogler, H., Eichenberger, C., Grossniklaus, U. Whole-mount clearing and staining of Arabidopsis flower organs and Siliques. Journal of Visualized Experiments. (134), e56441 (2018).
- Creff, A., Brocard, L., Ingram, G. A mechanically sensitive cell layer regulates the physical properties of the Arabidopsis seed coat. Nature Communications. 6, 6382 (2015).
- Yang, T., et al. The B3 domain-containing transcription factor ZmABI19 coordinates expression of key factors required for maize seed development and grain filling. Plant Cell. 33 (1), 104-128 (2021).
- Anderson, L. E. Hoyer's solution as a rapid permanent mounting medium for bryophytes. Bryologist. 57 (3), 242-244 (1954).
- Herr, J. M. A new clearing-squash technique for the study of ovule development in angiosperms. American Journal of Botany. 58 (8), 785-790 (1971).
- Yadegari, R., et al. Cell differentiation and morphogenesis are uncoupled in Arabidopsis raspberry embryos. The Plant Cell. 6 (12), 1713-1729 (1995).
- Grini, P. E., Jurgens, G., Hulskamp, M. Embryo and endosperm development is disrupted in the female gametophytic capulet mutants of Arabidopsis. Genetics. 162 (4), 1911-1925 (2002).
- Kataoka, K., Uemachi, A., Yazawa, S. Fruit growth and pseudoembryo development affected by uniconazole, an inhibitor of gibberellin biosynthesis, in pat-2 and auxin-Induced parthenocarpic tomato fruits. Scientia Horticulturae. 98 (1), 9-16 (2003).
- de Jong, M., Wolters-Arts, M., Feron, R., Mariani, C., Vriezen, W. H. The Solanum lycopersicum auxin response factor 7 (SlARF7) regulates auxin signaling during tomato fruit set and development. Plant Journal. 57 (1), 160-170 (2009).
- Roth, M., Florez-Rueda, A. M., Paris, M., Stadler, T. Wild tomato endosperm transcriptomes reveal common roles of genomic imprinting in both nuclear and cellular endosperm. Plant Journal. 95 (6), 1084-1101 (2018).
- Orsi, C. H., Tanksley, S. D. Natural variation in an ABC transporter gene associated with seed size evolution in tomato species. PLoS Genetics. 5 (1), 1000347 (2009).
- Herridge, R. P., Day, R. C., Baldwin, S., Macknight, R. C. Rapid analysis of seed size in Arabidopsis for mutant and QTL discovery. Plant Methods. 7 (1), 3 (2011).
- Xu, T. T., Ren, S. C., Song, X. F., Liu, C. M. CLE19 expressed in the embryo regulates both cotyledon establishment and endosperm development in Arabidopsis. Journal of Experimental Botany. 66 (17), 5217-5227 (2015).
- Ghadiri Alamdari, N., Salmasi, S., Almasi, H. Tomato seed mucilage as a new source of biodegradable film-forming material: effect of glycerol and cellulose nanofibers on the characteristics of resultant films. Food and Bioprocess Technology. 14 (12), 2380-2400 (2021).
- Gardner, R. O. An overview of botanical clearing technique. Stain Technology. 50 (2), 99-105 (1975).
- Beresniewicz, M. M., Taylor, A. G., Goffinet, M. C., Terhune, B. T. Characterization and location of a semipermeable layer in seed coats of leek and onion (Liliaceae), tomato and pepper (Solanaceae). Seed Science and Technology. 23 (1), 123-134 (1995).
- Stebbins, G. L. A bleaching and clearing method for plant tissues. Science. 87 (2245), 21-22 (1938).
- Debenham, E. M. A modified technique for the microscopic examination of the xylem of whole plants or plant organs. Annals of Botany. 3 (2), 369-373 (1939).
- Morley, T. Accelerated clearing of plant leaves by NaOH in association with oxygen. Stain Technology. 43 (6), 315-319 (1968).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten