Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un protocole de défrichement efficace pour l’étude du développement des semences de tomates (Solanum lycopersicum L.)
Dans cet article
Résumé
La graine de tomate est un modèle important pour étudier la génétique et la biologie du développement pendant la reproduction des plantes. Ce protocole est utile pour éliminer les graines de tomates à différents stades de développement afin d’observer la structure embryonnaire plus fine.
Résumé
La tomate (Solanum lycopersicum L.) est l’une des principales cultures commerciales au monde. La graine de tomate est un modèle important pour étudier la génétique et la biologie du développement pendant la reproduction des plantes. La visualisation d’une structure embryonnaire plus fine dans une graine de tomate est souvent entravée par le mucilage du tégument, le tégument multicellulaire et un endosperme à paroi épaisse, qui doit être résolu par une encastre-section laborieuse. Une alternative plus simple consiste à utiliser des techniques de nettoyage des tissus qui rendent la graine presque transparente à l’aide d’agents chimiques. Bien que les procédures de défrichage conventionnelles permettent de mieux comprendre les petites graines avec un tégument plus mince, le défrichement des semences de tomates continue d’être techniquement difficile, en particulier dans les derniers stades de développement.
Un protocole de défrichement rapide et économique permet d’observer le développement des graines de tomates de 3 à 23 jours après la floraison, lorsque la morphologie embryonnaire est presque terminée. Cette méthode combine une solution de nettoyage à base d’hydrate de chloral largement utilisée chez Arabidopsis avec d’autres modifications, y compris l’omission de la fixation formol-acéto-alcool (FAA), l’ajout d’un traitement à l’hypochlorite de sodium des graines, l’élimination du mucilage ramolli du tégument et le lavage et le traitement sous vide. Cette méthode peut être appliquée pour un nettoyage efficace des graines de tomates à différents stades de développement et est utile pour le suivi complet du processus de développement des graines mutantes avec une bonne résolution spatiale. Ce protocole de défrichage peut également être appliqué à l’imagerie en profondeur d’autres espèces commercialement importantes dans les Solanaceae.
Introduction
La tomate (S. lycopersicum L.) est l’une des cultures légumières les plus importantes au monde, avec une production de 186,8 millions de tonnes de fruits charnus sur 5,1 millions d’hectares en 20201. Il appartient à la grande famille des solanacées avec environ 2 716 espèces2, y compris de nombreuses cultures commercialement importantes telles que l’aubergine, les poivrons, la pomme de terre et le tabac. La tomate cultivée est une espèce diploïde (2n = 2x = 24) avec une taille de génome d’environ 900 Mb3. Pendant longtemps, de grands efforts ont été faits pour la domestication et la reproduction de la tomate en sélectionnant des caractères souhaitables chez Solanum spp. Il y a plus de 5 000 accessions de tomates répertoriées dans le Centre de ressources génétiques sur la tomate et plus de 80 000 germoplasmes de tomates sont stockés dans le monde4. Le plant de tomate est vivace en serre et se propage par graines. Une graine de tomate mature se compose de trois compartiments principaux : un embryon adulte, un endosperme résiduel de type cellulaire et un tégument dur 5,6 (figure 1A). Après une double fécondation, le développement de l’endosperme de type cellulaire précède le développement des zygotes. À ~5-6 jours après la floraison (DAF), le proembryon bicellulaire est observé pour la première fois lorsque l’endosperme est constitué de six à huit noyaux7. Chez Solanum pimpinellifolium, l’embryon approche de sa taille finale après 20 DAF, et les graines sont viables pour la germination après 32 DAF8. Au fur et à mesure que l’embryon se développe, l’endosperme est progressivement absorbé et seule une petite quantité d’endosperme reste dans la graine. L’endosperme résiduel est constitué d’endosperme micropylaire entourant l’extrémité radiculaire et d’endosperme latéral dans le reste de la graine 9,10. Le tégument externe est développé à partir de l’épiderme externe épaissi et lignifié du tégument, et avec les couches mortes de restes de tégument, ils forment une coquille dure pour protéger l’embryon et l’endosperme5.
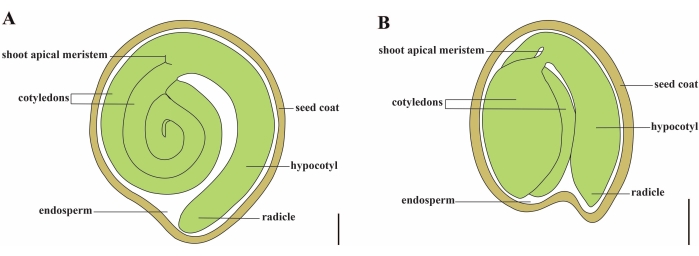
Figure 1 : Représentation schématique d’une graine mature chez Solanum lycopersicum et Arabidopsis thaliana. (A) Anatomie longitudinale d’une graine de tomate mature. (B) Anatomie longitudinale d’une graine d’Arabidopsis mature. Une graine de tomate mûre est environ 70 fois plus grande qu’une graine d’Arabidopsis. Barres d’échelle = (A) 400 μm, (B) 100 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La production de semences de tomates de haute qualité dépend de la coordination entre l’embryon, l’endosperme et les composants maternelsdes semences 11. La dissection de gènes et de réseaux clés dans le développement des semences nécessite un enregistrement phénotypique approfondi et complet des graines mutantes. Les techniques conventionnelles d’encastrement-section, telles que la section semi-mince et la section de paraffine, sont largement appliquées aux graines de tomates pour observer les structures locales et plus fines de l’embryon12,13,14,15. Cependant, l’analyse du développement des graines à partir de sections minces est généralement laborieuse et manque de résolution spatiale de l’axe z. En comparaison, la clairsemage tissulaire est une méthode rapide et efficace pour déterminer le stade de développement des anomalies embryonnaires les plus susceptibles de se produire16. La méthode de nettoyage réduit l’opacité des tissus internes en homogénéisant l’indice de réfraction avec un ou plusieurs agents biochimiques16. Le nettoyage des tissus entiers permet d’observer la structure d’un tissu végétal sans détruire son intégrité, et la combinaison de la technologie de nettoyage et de l’imagerie tridimensionnelle est devenue une solution idéale pour obtenir des informations sur la morphologie et l’état de développement d’un organe végétal17,18. Au fil des ans, des techniques de débroussaillage ont été utilisées chez diverses espèces végétales, notamment Arabidopsis thaliana, Hordeum vulgare et Beta vulgaris 19,20,21,22,23. Parmi celles-ci, la technologie de défrichage des ovules à montage entier a été une approche efficace pour étudier le développement des graines d’Arabidopsis, en raison de sa petite taille, de ses 4-5 couches de cellules de tégument et de l’endosperme de type nucléaire24,25. Avec la mise à jour continue de différents mélanges de nettoyage, tels que l’émergence de la solution26 de Hoyer, les structures internes de l’ovule d’orge ont été imagées avec un haut degré de clarté, bien que son endosperme constitue la majeure partie des graines. L’embryogenèse de la betterave sucrière peut être observée par débroussaillage combiné à un traitement sous vide et ramollissement à l’acide chlorhydrique19. Néanmoins, contrairement aux espèces mentionnées ci-dessus, les observations embryologiques par protocoles de nettoyage dans les graines de tomates n’ont pas été rapportées. Cela empêche une étude détaillée du développement embryonnaire et des semences de tomates.
L’hydrate de chloral est couramment utilisé comme solution de compensation qui permet aux tissus immergés et aux cellules d’être affichés sur différents plans optiques et préserve considérablement les cellules ou les composants tissulaires27,28,29. Le protocole de nettoyage à base d’hydrate de chloral a été utilisé avec succès pour le nettoyage complet des graines afin d’observer l’embryon et l’endosperme d’Arabidopsis21,28. Cependant, cette solution de nettoyage n’est pas efficace pour éliminer les graines de tomates, qui sont plus imperméables que les graines d’Arabidopsis. Les barrières physiques comprennent: (1) le tégument de la tomate a près de 20 couches cellulaires à 3 à 15 DAF 30,31, (2) l’endosperme de la tomate est de type cellulaire, pas de type nucléaire32, et (3) les graines de tomate sont environ 70 fois plus grandes en taille33,34 et (4) produisent de grandes quantités de mucilage de tégument, ce qui bloque la pénétration des réactifs d’élimination et affecte la visualisation des cellules embryonnaires.
Par conséquent, ce rapport présente une méthode optimisée de nettoyage à base d’hydrate de chloral pour l’élimination complète des graines de tomates à différents stades, ce qui permet une imagerie approfondie du processus de développement de l’embryon (Figure 2).
Protocole
1. Préparation des solutions
- Préparer le fixateur FAA en ajoutant 2,5 mL de formaldéhyde à 37 %, 2,5 mL d’acide acétique glacial et 45 mL d’éthanol à 70 % dans un tube à centrifuger de 50 mL. Vortex et conserver à 4 °C. Préparer fraîchement le fixateur FAA juste avant utilisation.
ATTENTION : 37 % de formaldéhyde est corrosif et potentiellement cancérogène en cas d’exposition ou d’inhalation. Le fixateur doit être effectué dans une hotte tout en portant un équipement de protection individuelle approprié. - Préparer la solution de nettoyage en ajoutant 5 mL de glycérol à 100 %, 40 g d’hydrate de chloral et 10 mL d’eau distillée dans une bouteille en verre de 100 ml enveloppée de papier d’aluminium. Dissoudre à l’aide d’un agitateur magnétique pendant une nuit à température ambiante. La solution préparée peut être conservée à 4 °C jusqu’à 6 mois.
ATTENTION : L’hydrate de chloral est cancérigène et dégage une odeur âcre. Effectuez l’expérience dans une hotte et portez un équipement de protection individuelle approprié. L’hydrate de chloral est facile à déliquescer dans l’air et ne doit pas être stocké en grande quantité. La solution de compensation peut se décomposer lorsqu’elle est exposée à la lumière et doit être tenue à l’écart de la lumière. - Préparer la solution désinfectante en ajoutant 10 mL d’hypochlorite de sodium à 6 %, 40 mL d’eau distillée et 50 μL de Tween-20 dans un tube à centrifuger de 50 mL. Vortex et stockez-le à température ambiante. Préparez fraîchement la solution désinfectante juste avant utilisation.
NOTE: La teneur en chlore efficace de l’hypochlorite de sodium qui a été placé pendant une longue période peut diminuer, et la teneur en hypochlorite de sodium à 6% peut être augmentée en fonction de la situation réelle.
2. Collecte des semences
- Semez des graines de tomates (S. lycopersicum L. cv. Micro-Tom, voir tableau des matières) dans des bassins carrés de 8 cm × 8 cm × 8 cm remplis d’un mélange 1:1 de sol nutritif de fleurs et de substrat (v/v) (voir tableau des matières), et poussez dans une salle de croissance avec 16/8 h de périodes lumière/obscurité à 24 ± 2 °C (jour) et 18 ± 2 °C (nuit), 60% à 70% d’humidité relative et une intensité lumineuse de 4 000 Lux. Environ 6 semaines plus tard, les plantes sont entrées dans la phase de floraison.
- Marquez la date de floraison des fleurs indépendantes à l’anthèse (l’angle d’ouverture des pétales est de 90°) et enregistrez le lendemain de la floraison (DAF).
- Récoltez les fruits de 3 à 23 plants de tomates DAF et mettez-les immédiatement sur la glace. Répartir les fruits (graines ou embryons) de 3 à 23 DAF en fruits précoces (3-10 DAF), moyens (11-16 DAF) et tardifs (17-23 DAF) (graines ou embryons).
REMARQUE: Ne récoltez pas trop de fruits à la fois et assurez-vous que les graines de chaque fruit sont dépouillées dans les 1 h pour un traitement ultérieur. - Pour les fruits primeurs, cassez le fruit et mettez-le sur une lame, et recueillez soigneusement les graines fraîches avec une pince de précision au stéréomicroscope (voir le tableau des matériaux) à un grossissement de 1x à 4x. Pour les fruits moyens et tardifs, cassez les fruits et collectez directement les graines fraîches à l’aide d’une pince de précision.
3. Élimination des semences à base d’hydrate de chloral
REMARQUE : Les protocoles conventionnels35 et optimisés ont été comparés dans cette étude pour leur efficacité de défrichement des semences.
- Compensation à l’aide d’un protocole conventionnel
- Placer immédiatement les graines fraîches (obtenues à l’étape 2.4) dans un tube à centrifuger de 2 mL contenant 1,5 mL de fixateur FAA et incuber sur un agitateur orbital (5 tr/min, voir le tableau des matières) pendant 4 h à température ambiante.
- Déshydrater ces graines dans une série d’éthanol de 70%, 95% et 100% éthanol (v/v) pendant 1 h chacune sur un agitateur orbital (5 rpm).
- Placer les graines dans 3 à 5 gouttes de solution de nettoyage (~100 μL) sur la lame et couvrir délicatement l’échantillon d’une lame. Remplacer la lame par une seule lame concave (voir le tableau des matières) pour les graines moyennes et tardives.
- Conservez ces lames ou lames concaves simples à température ambiante pendant 30 min (3 DAF), 2 h (6 DAF), 1 jour (9 DAF), 3 jours (12 DAF) ou 7 jours (14 à 22 DAF) selon les stades de développement du matériel semencier (plus les matériaux sont jeunes, plus la vitesse de dégagement est rapide).
- Observez les échantillons à l’aide d’un microscope à contraste interférentiel différentiel (CIV) équipé d’un appareil photo numérique (voir le tableau des matériaux) à un grossissement de 10x, 20x et 40x. Ajustez et optimisez la luminosité de la lumière transmise, le curseur DIC et l’ouverture du condensateur en temps réel pour chaque échantillon et capturez des images.
- Effacement à l’aide du protocole optimisé
- Placer les graines fraîches (obtenues à l’étape 2.4) directement dans un tube à centrifuger de 2 mL contenant 1,5 mL de solution désinfectante (étape 1.3).
REMARQUE : Le nombre de graines recueillies dans le tube à centrifuger de 2 ml varie selon le stade de développement des graines. Les détails sont présentés dans le tableau 1. - Incuber les échantillons sur un agitateur orbital (30 rpm) à température ambiante pendant 3 à 50 minutes jusqu’à ce que le contour le plus interne du tégument soit clairement visible. Jetez la solution désinfectante.
NOTE: Le temps d’incubation requis dépend du stade de développement de la graine (plus les graines sont tardives, plus le temps d’incubation sera long). Les détails sont présentés dans le tableau 1. - Pour les graines moyennes et tardives, transférer les graines sur une lame et retirer le mucilage de la graine avec une pince et une aiguille à dissection sous un stéréomicroscope à un grossissement de 1x à 4x. Transférez les graines dans le tube centrifuge d’origine à l’aide d’une pince.
REMARQUE : Cette étape n’est pas nécessaire pour les graines précoces. - Lavez les graines 5x avec 1,5 mL d’eau désionisée, 10 s à chaque fois. Jetez l’eau désionisée.
- Ajouter une solution de nettoyage de 2 × le volume des graines, suivie d’un traitement sous vide pendant 0 à 50 min (tableau 1) avec une pompe à vide (voir tableau des matériaux). Un traitement intermittent sous vide est effectué avec chaque traitement sous vide pendant 10 min espacés de 10 minutes.
- Remplacer par une solution de nettoyage fraîche de 2 × le volume des graines. Conserver le tube à centrifuger de 2 ml contenant les graines à température ambiante et à l’abri de la lumière pendant 30 minutes à 7 jours pour faciliter le nettoyage (tableau 1). Pendant l’incubation, pour les embryons tardifs, remplacer quotidiennement la solution de clairance par une solution fraîche et soumettre les graines à un traitement sous vide pendant 10 min.
- Préparez les graines dégagées sur des lames ou des lames concaves simples et observez avec le microscope DIC équipé d’un appareil photo numérique à un grossissement de 10x, 20x et 40x. Ajustez et optimisez la luminosité de la lumière transmise, le curseur DIC et l’ouverture du condensateur en temps réel pour chaque échantillon et capturez des images.
- Placer les graines fraîches (obtenues à l’étape 2.4) directement dans un tube à centrifuger de 2 mL contenant 1,5 mL de solution désinfectante (étape 1.3).
Résultats
Lorsque les graines de tomates ont été éliminées à l’aide d’une méthode conventionnelle comme chez Arabidopsis, des cellules endospermatiques denses ont bloqué la visualisation des embryons de tomates précoces à 3 DAF et 6 DAF (Figure 3A,B). Au fur et à mesure que le volume total de l’embryon augmentait, un embryon globulaire était à peine discernable à 9 DAF (Figure 3C). Cependant, à mesure que la taille de la grain...
Discussion
Par rapport à la section mécanique, la technologie de nettoyage est plus avantageuse pour l’imagerie tridimensionnelle car elle conserve l’intégrité des tissus ou organes végétaux16. Les protocoles de nettoyage conventionnels sont souvent limités à de petits échantillons en raison de la pénétration plus facile des solutions chimiques. La graine de tomate est un échantillon problématique pour le nettoyage des tissus car elle est environ 70 fois plus grande qu’une graine d’A...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Les auteurs remercient le Dr Jie Le et le Dr Xiufen Song pour leurs suggestions utiles sur la microscopie différentielle à contraste interférentiel et la méthode de compensation conventionnelle, respectivement. Cette recherche a été financée par la Fondation nationale des sciences naturelles de Chine (31870299) et l’Association de promotion de l’innovation des jeunes de l’Académie chinoise des sciences. La figure 2 a été créée avec BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| 1,000 µL pipette | GILSON | FA10006M | |
| 1,000 µL pipette tips | Corning | T-1000-B | |
| 2 ml centrifuge tube | Axygen | MCT-200-C | |
| 37% formaldehyde | DAMAO | 685-2013 | |
| 5,000 µL pipette | Eppendorf | 3120000275 | |
| 5,000 µL pipette tips | biosharp | BS-5000-TL | |
| 50 ml centrifuge tube | Corning | 430829 | |
| Absolute Ethanol | BOYUAN | 678-2002 | |
| Bottle glass | Fisher | FB800-100 | |
| Chloral Hydrate | Meryer | M13315-100G | |
| Coverslip | Leica | 384200 | |
| DIC microscope | Zeiss | Axio Imager A1 | 10x, 20x and 40x magnification |
| Disinfectant | QIKELONGAN | 17-9185 | |
| Dissecting needle | Bioroyee | 17-9140 | |
| Flower nutrient soil | FANGJIE | ||
| Forceps | HAIOU | 4-94 | |
| Glacial Acetic Acid | BOYUAN | 676-2007 | |
| Glycerol | Solarbio | G8190 | |
| Magnetic stirrer | IKA | RET basic | |
| Micro-Tom | Tomato Genetics Resource Center | LA3911 | |
| Orbital shaker | QILINBEIER | QB-206 | |
| Seeding substrate | PINDSTRUP | LV713/018-LV252 | Screening:0-10 mm |
| Single concave slide | HUABODEYI | HBDY1895 | |
| Slide | Leica | 3800381 | |
| Stereomicroscope | Leica | S8 APO | 1x to 4x magnification |
| Tin foil | ZAOWUFANG | 613 | |
| Tween 20 | Sigma | P1379 | |
| Vacuum pump | SHIDING | SHB-III | |
| Vortex meter | Silogex | MX-S |
Références
- . FAOSTAT Available from: https://www.fao.org/faostat/en/#data/QCL (2022)
- Olmstead, R. G., Bohs, L. A summary of molecular systematic research in Solanaceae: 1982-2006. Acta Horticulturae. 745, 255-268 (2007).
- Consortium, T. G. The tomato genome sequence provides insights into fleshy fruit evolution. Nature. 485 (7400), 635-641 (2012).
- Ebert, A. W., Chou, Y. Y. The tomato collection maintained by AVRDC - The World Vegetable Center: composition, germplasm dissemination and use in breeding. Acta Horticulturae. 1101, 169-176 (2015).
- Hilhorst, H., Groot, S., Bino, R. J. The tomato seed as a model system to study seed development and germination. Acta Botanica Neerlandica. 47, 169-183 (1998).
- Chaban, I. A., Gulevich, A. A., Kononenko, N. V., Khaliluev, M. R., Baranova, E. N. Morphological and structural details of tomato seed coat formation: A different functional role of the inner and outer epidermises in unitegmic ovule. Plants-Basel. 11 (9), 1101 (2022).
- Iwahori, S. High temperature injuries in tomato. V. Fertilization and development of embryo with special reference to the abnormalities caused by high temperature. Journal of The Japanese Society for Horticultural Science. 35 (4), 379-386 (1966).
- Xiao, H., et al. Integration of tomato reproductive developmental landmarks and expression profiles, and the effect of SUN. on fruit shape. BMC Plant Biology. 9 (1), 49 (2009).
- Karssen, C. M., Haigh, A. M., Toorn, P., Weges, R., Taylorson, R. B. Physiological mechanisms involved in seed priming. Recent advances in the development and germination of seeds. NATO ASI Series. 187, (1989).
- Nonogaki, H. Seed dormancy and germination-emerging mechanisms and new hypotheses. Frontiers in Plant Science. 5, 233 (2014).
- Doll, N. M., Ingram, G. C. Embryo-endosperm interactions. Annual Review of Plant Biology. 73, 293-321 (2022).
- Serrani, J. C., Fos, M., Atarés, A., García-Martínez, J. L. Effect of gibberellin and auxin on parthenocarpic fruit growth induction in the cv micro-tom of tomato. Journal of Plant Growth Regulation. 26 (3), 211-221 (2007).
- Yang, C., et al. A regulatory gene induces trichome formation and embryo lethality in tomato. Proceedings of the National Academy of Sciences USA. 108 (29), 11836-11841 (2011).
- Goetz, S., et al. Role of cis-12-oxo-phytodienoic acid in tomato embryo development. Plant Physiology. 158 (4), 1715-1727 (2012).
- Ko, H. Y., Ho, L. H., Neuhaus, H. E., Guo, W. J. Transporter SlSWEET15 unloads sucrose from phloem and seed coat for fruit and seed development in tomato. Plant Physiology. 187 (4), 2230-2245 (2021).
- Richardson, D. S., Lichtman, J. W. Clarifying tissue clearing. Cell. 162 (2), 246-257 (2015).
- Kurihara, D., Mizuta, Y., Sato, Y., Higashiyama, T. ClearSee: A rapid optical clearing reagent for whole-plant fluorescence imaging. Development. 142 (23), 4168-4179 (2015).
- Vieites-Prado, A., Renier, N. Tissue clearing and 3D imaging in developmental biology. Development. 148 (18), (2021).
- Kwiatkowska, M., Kadłuczka, D., Wędzony, M., Dedicova, B., Grzebelus, E. Refinement of a clearing protocol to study crassinucellate ovules of the sugar beet (Beta vulgaris L., Amaranthaceae). Plant Methods. 15, 71 (2019).
- Ponitka, A., Ślusarkiewicz-Jarzina, A. Cleared-ovule technique used for rapid access to early embryo development in Secale cereale × Zea mays crosses. Acta Biologica Cracoviensia. Series Botanica. 46, 133-137 (2014).
- Ceccato, L., et al. Maternal control of PIN1 is required for female gametophyte development in Arabidopsis. PLoS One. 8 (6), 66148 (2013).
- Wilkinson, L. G., Tucker, M. R. An optimised clearing protocol for the quantitative assessment of sub-epidermal ovule tissues within whole cereal pistils. Plant Methods. 13, 67 (2017).
- Hedhly, A., Vogler, H., Eichenberger, C., Grossniklaus, U. Whole-mount clearing and staining of Arabidopsis flower organs and Siliques. Journal of Visualized Experiments. (134), e56441 (2018).
- Creff, A., Brocard, L., Ingram, G. A mechanically sensitive cell layer regulates the physical properties of the Arabidopsis seed coat. Nature Communications. 6, 6382 (2015).
- Yang, T., et al. The B3 domain-containing transcription factor ZmABI19 coordinates expression of key factors required for maize seed development and grain filling. Plant Cell. 33 (1), 104-128 (2021).
- Anderson, L. E. Hoyer's solution as a rapid permanent mounting medium for bryophytes. Bryologist. 57 (3), 242-244 (1954).
- Herr, J. M. A new clearing-squash technique for the study of ovule development in angiosperms. American Journal of Botany. 58 (8), 785-790 (1971).
- Yadegari, R., et al. Cell differentiation and morphogenesis are uncoupled in Arabidopsis raspberry embryos. The Plant Cell. 6 (12), 1713-1729 (1995).
- Grini, P. E., Jurgens, G., Hulskamp, M. Embryo and endosperm development is disrupted in the female gametophytic capulet mutants of Arabidopsis. Genetics. 162 (4), 1911-1925 (2002).
- Kataoka, K., Uemachi, A., Yazawa, S. Fruit growth and pseudoembryo development affected by uniconazole, an inhibitor of gibberellin biosynthesis, in pat-2 and auxin-Induced parthenocarpic tomato fruits. Scientia Horticulturae. 98 (1), 9-16 (2003).
- de Jong, M., Wolters-Arts, M., Feron, R., Mariani, C., Vriezen, W. H. The Solanum lycopersicum auxin response factor 7 (SlARF7) regulates auxin signaling during tomato fruit set and development. Plant Journal. 57 (1), 160-170 (2009).
- Roth, M., Florez-Rueda, A. M., Paris, M., Stadler, T. Wild tomato endosperm transcriptomes reveal common roles of genomic imprinting in both nuclear and cellular endosperm. Plant Journal. 95 (6), 1084-1101 (2018).
- Orsi, C. H., Tanksley, S. D. Natural variation in an ABC transporter gene associated with seed size evolution in tomato species. PLoS Genetics. 5 (1), 1000347 (2009).
- Herridge, R. P., Day, R. C., Baldwin, S., Macknight, R. C. Rapid analysis of seed size in Arabidopsis for mutant and QTL discovery. Plant Methods. 7 (1), 3 (2011).
- Xu, T. T., Ren, S. C., Song, X. F., Liu, C. M. CLE19 expressed in the embryo regulates both cotyledon establishment and endosperm development in Arabidopsis. Journal of Experimental Botany. 66 (17), 5217-5227 (2015).
- Ghadiri Alamdari, N., Salmasi, S., Almasi, H. Tomato seed mucilage as a new source of biodegradable film-forming material: effect of glycerol and cellulose nanofibers on the characteristics of resultant films. Food and Bioprocess Technology. 14 (12), 2380-2400 (2021).
- Gardner, R. O. An overview of botanical clearing technique. Stain Technology. 50 (2), 99-105 (1975).
- Beresniewicz, M. M., Taylor, A. G., Goffinet, M. C., Terhune, B. T. Characterization and location of a semipermeable layer in seed coats of leek and onion (Liliaceae), tomato and pepper (Solanaceae). Seed Science and Technology. 23 (1), 123-134 (1995).
- Stebbins, G. L. A bleaching and clearing method for plant tissues. Science. 87 (2245), 21-22 (1938).
- Debenham, E. M. A modified technique for the microscopic examination of the xylem of whole plants or plant organs. Annals of Botany. 3 (2), 369-373 (1939).
- Morley, T. Accelerated clearing of plant leaves by NaOH in association with oxygen. Stain Technology. 43 (6), 315-319 (1968).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon