Method Article
Multiplex-Barcoding-Bildanalyse zur Immunprofilierung und räumlichen Kartierung bei der Einzelzellanalyse von Paraffingewebeproben
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Die Multiplex-Barcoding-Bildanalyse hat kürzlich die Charakterisierung der Tumormikroumgebung verbessert und ermöglicht umfassende Studien der Zellzusammensetzung, des funktionellen Zustands und der Zell-Zell-Interaktionen. In dieser Arbeit beschreiben wir ein Färbe- und Bildgebungsprotokoll unter Verwendung des Barcodings von Oligonukleotid-konjugierten Antikörpern und Zyklusbildgebung, das die Verwendung einer hochdimensionalen Bildanalysetechnik ermöglicht.
Zusammenfassung
Die Multiplex-Bildgebungstechnologie mit Antikörper-Barcoding mit Oligonukleotiden, die mehrere Epitope im selben Gewebeabschnitt nacheinander detektiert, ist eine effektive Methode zur Tumorbewertung, die das Verständnis der Tumormikroumgebung verbessert. Die Visualisierung der Proteinexpression in formalinfixierten, in Paraffin eingebetteten Geweben wird erreicht, wenn ein spezifischer Fluorophor über komplementäre Oligonukleotide an einen Antikörper-gebundenen Barcode getempert wird und dann eine Probenbildgebung durchgeführt wird. Tatsächlich ermöglicht diese Methode die Verwendung von anpassbaren Panels mit mehr als 40 Antikörpern in einer einzigen Gewebefärbereaktion. Diese Methode ist mit frisch gefrorenem Gewebe, formalinfixiertem, in Paraffin eingebettetem Gewebe, kultivierten Zellen und mononukleären Zellen des peripheren Blutes kompatibel, was bedeutet, dass Forscher diese Technologie verwenden können, um eine Vielzahl von Probentypen mit Einzelzellauflösung zu betrachten. Diese Methode beginnt mit einem manuellen Färbe- und Fixierungsprotokoll, und alle Antikörper-Barcodes werden mit einem Antikörpercocktail aufgebracht. Das Färbefluidik-Instrument ist vollständig automatisiert und führt iterative Zyklen der Markierung, Bildgebung und Entfernung spektral unterschiedlicher Fluorophore durch, bis alle Biomarker mit einem Standard-Fluoreszenzmikroskop abgebildet wurden. Die Bilder werden dann über alle Bildgebungszyklen hinweg gesammelt und kompiliert, um eine Einzelzellauflösung für alle Marker zu erreichen. Die einstufige Färbung und die schonende Entfernung von Fluorophoren ermöglichen nicht nur eine hochgradig gemultiplexte Biomarkeranalyse, sondern konservieren die Probe auch für zusätzliche nachgelagerte Analysen, falls gewünscht (z. B. Hämatoxylin- und Eosinfärbung). Darüber hinaus ermöglicht die Bildanalysesoftware die Bildverarbeitung – Driftkompensation, Hintergrundsubtraktion, Zellsegmentierung und Clustering – sowie die Visualisierung und Analyse der Bilder und Zellphänotypen für die Erstellung von räumlichen Netzwerkkarten. Zusammenfassend lässt sich sagen, dass diese Technologie ein computergestütztes Mikrofluidiksystem und ein Fluoreszenzmikroskop verwendet, um fluoreszenzmarkierte DNA-Sonden, die komplementär zu gewebegebundenen, Oligonukleotid-konjugierten Antikörpern sind, iterativ zu hybridisieren, abzubilden und zu streifen.
Einleitung
Die Tumormikroumgebung (TME) ist extrem heterogen und besteht aus Tumorzellen, Tumorstromazellen, Immunzellen, nichtzellulären Komponenten der extrazellulären Matrix und zahlreichen reichlich vorhandenen Molekülen, die von Tumor-, Stroma- und Immunzellen produziert und freigesetzt werden 1,2. Die sich häufenden Belege zeigen, dass das TME eine zentrale Rolle bei der Reprogrammierung der Tumordifferenzierung, des Wachstums, der Invasion, der Metastasierung und des Ansprechens auf Therapien spielt3.
Zu verstehen, wie verschiedene Zelltypen im TME interagieren und über Signalnetzwerke miteinander kommunizieren, ist für die Verbesserung der Krebsdiagnose, die Optimierung der Immuntherapie und die Entwicklung neuer Behandlungen von entscheidender Bedeutung4. Traditionelle Gewebemikroskopietechniken wie Immunhistochemie (IHC) und Immunfluoreszenz (IF) werden seit Jahrzehnten eingesetzt, um die Zelltypen, die Häufigkeit und die Kommunikation in Tumorproben zu untersuchen. Leider können diese Techniken in der Regel nur einen oder zwei Proteinmarker in einem Gewebeschnitt auswerten und können die komplexen räumlichen und strukturellen Beziehungen zwischen diesen Zellen nicht aufdecken 5,8,7.
In den letzten zwei Jahrzehnten haben sich mehrere Multiplex-Bildgebungstechnologien etabliert8. Diese Technologien bieten einen deutlich verbesserten Überblick über die Zusammensetzung, Funktion und Lage von Immunzellen innerhalb der TME, was zu schnellen Fortschritten in der Fähigkeit führt, komplexe TMEs auf Einzelzellebene zu identifizieren und räumlich zu profilieren 9,10. Die räumlichen und strukturellen Beziehungen verschiedener Tumor- und Immunzellen im TME stehen heute im Vordergrund biologischer und klinischer Studien mit diesen Multiplex-Bildgebungstechnologien11,12.
Die kürzlich entwickelte Multiplex-Bildgebungstechnologie mit Oligonukleotid-konjugierten Antikörper-Barcoding ist eine einflussreiche biologische Einzelzell-Forschungsplattform, die auf dem Nachweis von Oligonukleotid-konjugierten Antikörpern in formalinfixierten, paraffineingebetteten (FFPE) Proben basiert13,14. Gegenwärtig ermöglicht diese Multiplex-Bildgebungstechnologie die gleichzeitige Bildgebung von mehr als 100 Markern in einem einzigen Gewebeschnitt15, was die Anzahl der Zelltypen, die in situ unterscheidbar sind, erhöht hat. Dies ermöglicht eine räumliche Analyse von Tumor- und Immunzellen, die mit herkömmlichen Immunphänotypisierungsansätzen nicht möglich ist16.
In dieser Arbeit beschreiben wir ein optimiertes Protokoll für die Konjugation von gereinigten Antikörpern an Oligonukleotide und die Validierung dieser Konjugation unter Verwendung der Multiplex-Bildgebungsplattform und eines Multicycle-Bildgebungsverfahrens mit FFPE-Gewebe. Darüber hinaus beschreiben wir die grundlegenden Verfahren der Bildverarbeitung und Datenanalyse, die bei dieser Technologie zum Einsatz kommen.
Protokoll
Diese retrospektive Studie wurde vom Institutional Review Board des MD Anderson Cancer Center der University of Texas genehmigt. Die FFPE-Gewebeproben wurden von Patienten bei MD Anderson im Rahmen der routinemäßigen Standardversorgung entnommen. Es wurden keine diagnostischen oder therapeutischen Eingriffe durchgeführt. Für die Verwendung der gesammelten Proben für die Forschung und Veröffentlichung wurde eine Einverständniserklärung der Patienten eingeholt.
1. Antikörperquellen, die für das Design des Antikörperpanels verwendet werden
- Erstellen Sie ein Antikörperpanel für die Multiplex-Bildgebung, nachdem Sie die Qualität der Gewebe und Proteine sorgfältig geprüft haben. Für das Design des Antikörperpanels werden drei Antikörperquellen in Betracht gezogen: 1) vollständig kommerziell validierte Antikörper, 2) gescreente Antikörper mit Multiplex-Bildgebungstechnologie und 3) vom Endbenutzer gemeinsam genutzte Antikörper.
HINWEIS: Antikörper mit Multiplex-Bildgebungstechnologie haben sich als wirksam erwiesen und sind bei den Anbietern erhältlich. Diese gescreenten Antikörper können nach der Oligonukleotid-Konjugation durch den Anwender auf die Multiplex-Bildfärbung appliziert werden. - Wenn ein Protein von Interesse in den oben genannten Quellen nicht gefunden werden kann, verwenden Sie die Antikörperklone, von denen bekannt ist, dass sie mit IHC arbeiten. Darüber hinaus werden für diese Multiplex-Bildgebungstechnologie IgG-Isotypen anstelle von IgM-Klonen empfohlen, da die Ausfallrate bei IgM höher ist als bei IgG-Klonen.
2. Vor der Antikörperkonjugation
- Bei der Identifizierung von Antikörperklonen für die Konjugation mithilfe von Multiplex-Imaging-Barcodes sollten Sie den Kauf von trägerfreien Antikörpern in phosphatgepufferter Kochsalzlösung (PBS) oder einem ähnlichen Puffer in Betracht ziehen. Es ist bekannt, dass Trägerproteine, einschließlich BSA, Gluten, Glycerin und andere Proteinzusätze, die Konjugationskapazität reduzieren.
- Wählen Sie den am besten geeigneten Antikörperklon aus und optimieren Sie vor der Konjugation immer die Färbebedingungen (d. h. Antigengewinnung und Titration). Tun Sie dies, indem Sie einen unkonjugierten Antikörperklon auf positives und negatives Gewebe für diesen Antikörper mit Standard-IF oder IHC auftragen.
HINWEIS: Wenn ein gereinigter Antikörper nicht im Handel erhältlich ist, muss vor der Konjugation ein Antikörperreinigungsprozess durchgeführt werden. Das Aufreinigungsverfahren, das mit Antikörper-Aufreinigungskits verwendet wird, wird hier nicht erörtert.
3. Antikörper-Konjugation
- Besorge dir die Antikörper-Konjugationsreagenzien. Handelsübliche Konjugationskits enthalten eine Filterblockierlösung, eine Reduktionslösung 2, eine Konjugationslösung, eine Reinigungslösung, eine Antikörper-Aufbewahrungslösung (alle bei 4 °C gelagert) und eine Reduktionslösung 1 (bei −20 °C gelagert).
- Konjugation
HINWEIS: Ein gereinigter Antikörper wird mit einem Reduktionsmittel behandelt, so dass die reduzierten Einheiten des Antikörpers mit dem bei dieser Technologie verwendeten Multiplex-Imaging-Barcode reagieren und so eine kovalente Bindung eingehen können. Dieser Prozess dauert etwa 4,5 h und führt zu etwa 120 μl konjugiertem Antikörper, der 1 Jahr lang lebensfähig ist. Der gesamte Spin-down wird bei Raumtemperatur (RT) durchgeführt, und der Durchfluss wird verworfen, mit Ausnahme des allerletzten Schritts (3.2.9), in dem ein Sammelröhrchen den konjugierten Antikörper enthält.- Aspirieren und geben Sie 500 μl der Filterblockerlösung mit einer Pipette auf die 50-kDa-Molekulargewichts-Cutoff-Filtersäulen und schleudern Sie sie 2 Minuten lang bei 12.000 x g herunter, um die Bindung unspezifischer Antikörper zu blockieren.
Anmerkungen: Wenn Restlösung an der Oberseite der Säule erkennbar ist, drehen Sie den Filter im Auffangröhrchen um und schleudern Sie ihn 2 Minuten lang bei 3.000 x g nach unten. - 50 μg des Antikörpers in einer 100 μl großen Lösung in die Filtersäule pipettieren und 8 min lang bei 12.000 x g abschleudern.
Anmerkungen: Verwenden Sie ein Spektralphotometer, um die Konzentration des gereinigten Antikörpers zu messen und das Volumen der Lösung zu berechnen, das 50 μg des Antikörpers entspricht. Wenn das Volumen der Antikörperlösung weniger als 100 μl beträgt, stellen Sie das Volumen auf 100 μl ein, indem Sie 1x PBS hinzufügen. 1 μg unkonjugierter Antikörper zur Bestätigung der Konjugation aufbewahren (Schritt 4.4). - Pipettieren Sie 260 μl Reduktionsmastermix (20 μl Reduktionslösung 1 gemischt mit 825 μl Reduktionslösung 2, was für drei Antikörperkonjugationsreaktionen ausreicht) in jede Filtersäule. Ziehen Sie die Mastermischung 2-3 Sekunden lang vorsichtig vor oder pipettieren Sie auf und ab, um die Lösung mit dem Antikörper zu vermischen. Bei RT 30 Minuten inkubieren.
- Nach der Inkubation die Filtersäulen bei 12.000 x g für 8 min herunterschleudern. Geben Sie 450 μl Konjugationslösung in die Filtersäulen und schleudern Sie sie 8 Minuten lang bei 12.000 x g .
- Resuspendieren Sie den gewünschten Barcode in 10 μl nukleasefreiem Wasser und 210 μl Konjugationslösung.
Anmerkungen: Bereiten Sie unmittelbar vor der Verwendung eine Antikörpermarkierung für die Färbung vor. Antikörper-Barcode-Aliquots dürfen nicht wiederverwendet werden. - Nach Abschluss des Spin-downs in Schritt 3.2.4 wird der resuspendierte Antikörper-Tag (ca. 220 μl) in jede entsprechende Filtersäule gegeben. Pipettieren Sie die Mischung vorsichtig auf und ab, um die Reagenzien zu mischen. Schließen Sie die Deckel der Filtersäule und inkubieren Sie die Konjugationsreaktion bei RT für 2 h. Nach 2 h drehen Sie die Filtersäulen bei 12.000 x g für 8 min herunter.
HINWEIS: Für das Bestätigungsprotokoll wird empfohlen, 5 μl der konjugierten Lösung in einem Polymerase-Kettenreaktionsröhrchen beiseite zu legen und bei 4 °C zu lagern (siehe unten). - Pipettieren Sie 450 μl der Reinigungslösung in jede Filtersäule und schleudern Sie sie 8 Minuten lang bei 12.000 x g . Wiederholen Sie den Vorgang dreimal.
- Pipettieren Sie 100 μl der Aufbewahrungslösung in die Filtersäulen. Pipettieren Sie die Mischung mehr als 10 Mal vorsichtig auf und ab und waschen Sie die Seiten der Filter in der Säule sorgfältig.
Anmerkungen: Lösen Sie 50 μg Antikörper in 100 μl der Aufbewahrungslösung auf. Wenn die Konjugationsreaktion mit mehr als 50 μg Antikörper begonnen wird, fügen Sie in diesem Verhältnis mehr Speicherlösung hinzu. - Invertieren Sie die Filterspalten in einem Frischsammelröhrchen. Bei 3.000 x g für 2 min bei RT abschleudern. Bewahren Sie die gesammelte Lösung auf. Die konjugierte Antikörperlösung wird in sterile Schraubröhrchen pipettiert und bis zu 1 Jahr bei 4 °C gelagert.
HINWEIS: Ein konjugierter Antikörper sollte nach 2 Tagen mit der Multiplex-Bildgebungstechnologie getestet werden. Vorherige Tests können zu einer hohen nuklearen Hintergrundfärbung führen.
- Aspirieren und geben Sie 500 μl der Filterblockerlösung mit einer Pipette auf die 50-kDa-Molekulargewichts-Cutoff-Filtersäulen und schleudern Sie sie 2 Minuten lang bei 12.000 x g herunter, um die Bindung unspezifischer Antikörper zu blockieren.
4. Konjugationsbestätigung
HINWEIS: Vor der Durchführung von Färbeversuchen mit einem benutzerkonjugierten Antikörper unter Verwendung der Multiplex-Bildgebungstechnologie sollte die Konjugation durch Gelelektrophorese mit 5 μl des konjugierten Antikörpers (siehe Schritt 3.2.6) zusammen mit 1 μg eines nicht konjugierten Antikörpers (normalerweise in 2 μl der Mischung) als Kontrolle bestätigt werden. Eine erfolgreiche Antikörperkonjugation wird durch eine Erhöhung des Molekulargewichts des Antikörpers demonstriert. Dieses Bestätigungsprotokoll bewertet jedoch nur den Erfolg der chemischen Reaktion für die Konjugation und befasst sich nicht mit der Antikörpervalidierung, die für die Multiplex-Bildgebung verwendet wird.
- Pipettieren Sie 8 μl und 11 μl nukleasefreies Wasser in den reservierten konjugierten Antikörper bzw. den unkonjugierten Antikörper der Kontrolle, um ein Endvolumen von 13 μl zu erhalten.
- Pipettieren Sie 5 μl LDS-Probenpuffer (oder ein anderes Natriumdodecylsulfat-Polyacrylamid-Gelelektrophoresesystem) und 2 μl Probenreduktionsmittel in jede Probe des reservierten konjugierten Antikörpers und denaturieren Sie die Proben 10 Minuten lang in einem Trockenbad bei 95 °C.
- Während die Proben denaturiert werden, verdünnen Sie 40 ml MOPS SDS-Laufpuffer in 760 ml Reinstwasser, geben Sie ein Gel in den Tank eines Elektrophoresesystems und gießen Sie den verdünnten Laufpuffer über das Gel.
- Nach Ablauf der 10-minütigen Denaturierungsphase wird eine Vertiefung des Gels mit einem vorgefärbten Proteinstandard, eine mit dem unkonjugierten Antikörper (aus Schritt 3.2.2) und die restlichen Vertiefungen mit den konjugierten Antikörperproben beladen. Als nächstes wird das Gel mit 150 V betrieben, bis der Proteinstandard am Ende des Gels erscheint.
Anmerkungen: Das Gel haftet leicht an mikrowellengeeigneten Behältern und kann daher reißen, daher muss das Gel in den folgenden Schritten mit Vorsicht behandelt werden. - Übertragen Sie das Gel nach Abschluss des Laufs in einen mikrowellengeeigneten Behälter, der mit Reinstwasser vorgefüllt ist, und erhitzen Sie es in der Mikrowelle, bis die erste Blase im Wasser sichtbar ist.
Anmerkungen: Die Zeit, in der sich Blasen bilden, variiert stark je nach verwendeter Mikrowelle. - Lassen Sie das Wasser aus dem Behälter ab, gießen Sie etwa 250 ml Coomassie G-250 Beize über das Gel und erhitzen Sie das Gel in der Mikrowelle, bis die erste Blase sichtbar ist. Nehmen Sie anschließend den Behälter mit dem Gel und dem Coomassie G-250-Fleck aus der Mikrowelle und stellen Sie ihn für 10 Minuten auf einen Shaker.
- Lassen Sie den Fleck nach dem Schütteln vorsichtig abtropfen, ersetzen Sie ihn durch etwa 200 ml Reinstwasser und stellen Sie den Behälter dann auf den Shaker, um das Gel zu waschen.
- Lassen Sie das Reinstwasser ab und ersetzen Sie es fünfmal durch neues Reinstwasser oder bis die Reste des Flecks im Wasserbad nicht mehr sichtbar sind. Lassen Sie das Gel über Nacht auf dem Shaker waschen, bis bei Bedarf Bänder sichtbar sind, bevor Sie das Gel fotografieren (Abbildung 1).
HINWEIS: Die Antikörper, die in der Multiplex-Bildgebung verwendet werden, sollten Färbemuster aufweisen, die mit denen von farbstoffkonjugierten Antikörpern vergleichbar sind. Gewebeschnitte mit bekannten Antigene, die positiv für konjugierte Antikörper sind, können mit Oligonukleotid-konjugierten und farbstoffkonjugierten Antikörpern gefärbt werden. In dieser Arbeit waren jeweils die Gewebemorphologien für beide Antikörpertypen äquivalent und aufeinander abgestimmt, ebenso wie die erwartete Zellverteilung, basierend auf der Biologie der interessierenden Proteine und der Testgewebeproben. Dieses Ergebnis demonstriert die Wirksamkeit der Verwendung von Oligonukleotid-konjugierten Antikörpereinheiten für Gewebefärbungs-basierte Ansätze mit FFPE-Gewebe.
5. Färbung von Oligonukleotid-konjugierten Antikörpern
- Bereiten Sie Deckgläser für die Platzierung von Taschentüchern vor.
- Tränken Sie die Deckgläser 24 h lang in einer 0,1%igen Poly-L-Lysin-Lösung bei RT, um die Gewebehaftung zu verbessern.
- Lassen Sie nach dem Einweichen die Poly-L-Lysin-Lösung ab und waschen Sie die Deckgläser 30 s lang mit Reinstwasser. Wiederholen Sie das Waschen vier- bis sechsmal. Entfernen Sie die Deckgläser aus dem Reinstwasser, das zum Waschen verwendet wird, und legen Sie sie zum Trocknen über Nacht auf ein fusselfreies Handtuch.
HINWEIS: Ein Histologe muss das ausgewählte Gewebe in 5 μm dicke Abschnitte schneiden, sie auf die Mitte eines mit Poly-L-Lysin geladenen Deckglases legen und über Nacht trocknen lassen. Nach dem Trocknen dürfen die Deckgläser mit den Gewebeschnitten bei 4 °C nicht länger als 6 Monate gelagert werden. Das Färbepanel in diesem Protokoll enthielt 26 Marker. Bei dem in diesem Protokoll verwendeten Gewebe handelte es sich um normales menschliches Mandelgewebe. Die Einweichzeit des Deckglases sollte 1 Woche nicht überschreiten. Poly-L-Lysin-beschichtete Deckgläser können bei RT gelagert werden und müssen innerhalb von 2 Monaten nach der Herstellung verwendet werden.
- Legen Sie den Deckglashalter am Tag vor dem Färben über Nacht in einen 60 °C heißen Ofen.
- Legen Sie die Deckglasprobe am nächsten Tag in einen vorgeheizten Deckglashalter bei 60 °C. Prüfen Sie nach 30 Minuten Backzeit, ob das Paraffin vom Taschentuch abgeschmolzen ist.
- Legen Sie den Deckglashalter/die Probe schnell in die folgende Lösungsreihe: zwei Runden Entwachsungsmittel für jeweils 6 Minuten; zwei Runden 100% Ethanol für jeweils 5 Minuten; eine Runde 90%iges Ethanol für 5 Minuten; eine Runde 70%iges Ethanol für 5 Minuten; eine Runde 50%iges Ethanol für 5 Minuten; eine Runde 30%iges Ethanol für 5 Minuten; und zwei Runden mit Diethylpyrocarbonat (DEPC) behandeltem Reinstwasser für jeweils 5 Minuten.
Anmerkungen: Alle Verdünnungen von Ethanol werden mit DEPC-behandeltem Reinstwasser hergestellt. Wenn Xylol zum Entwachsen verwendet wird, sollte es in einer Haube verwendet werden. - Gehen Sie wie folgt vor, während Sie den Deckglashalter/die Probe der Lösungsserie aussetzen:
- Bereiten Sie eine Feuchtigkeitskammer vor, indem Sie eine leere Pipettenspitzenbox mit einem wassergetränkten Papiertuch auf den Boden legen.
- Füllen Sie einen Schnellkochtopf mit so viel Wasser, dass ein 50-ml-Becherglas zur Hälfte bedeckt ist.
- Geben Sie 5 ml Methanol pro Probe bei 4 °C in das Becherglas.
- AR9-Puffer mit DEPC-behandeltem Reinstwasser auf 1x verdünnen; pro Deckglashalter werden ca. 50 mL des verdünnten Puffers benötigt.
- Nachdem die Lösungsserie abgeschlossen ist, füllen Sie ein 50-ml-Glasbecherglas mit ca. 40 mL 1x AR9-Puffer, tauchen Sie den Deckglashalter/die Probe in das Becherglas und decken Sie den Becher-/Deckglashalter vollständig mit Aluminiumfolie ab.
- Stellen Sie den mit Aluminiumfolie überzogenen Becher-/Deckglashalter in den mit Wasser gefüllten Schnellkochtopf und kochen Sie ihn 20 Minuten lang unter hohem Druck (ca. 15 psi).
- Entfernen Sie nach dem Garen den Becher-/Deckglashalter, wickeln Sie die Alufolie vorsichtig aus und lassen Sie den Becher-/Deckglashalter etwa 10 Minuten bei RT abkühlen.
- Entfernen Sie den Deckglashalter/die Probe aus dem 1x AR9-Puffer und tauchen Sie ihn in zwei Runden DEPC-behandeltes Reinstwasser, wobei Sie die Proben in beiden Runden jeweils 2 Minuten lang inkubieren.
- Entnehmen Sie während der Inkubation den Hydratationspuffer, die blockierenden N-, die blockierende G-, die blockierende J- und die blockierende S-Lösung sowie das Antikörperverdünnungsmittel/den Antikörperblock aus dem Multiplex-Imaging-Färbekit. Beschriften Sie bei zwei Deckglasproben 6-Well-Platten für die Lösungen mit den in Abbildung 2 gezeigten Konfigurationen.
- Legen Sie die Deckglasprobe für jeweils 2 Minuten in zwei Runden à 5 ml des Hydratationspuffers.
- Nach beiden Platzierungsrunden in den Hydratationspuffer wird die Deckglasprobe in 5 ml des Multiplex-Antikörper-Verdünnungsmittels/-blocks gegeben und 20-30 Minuten bei RT inkubiert (nicht länger als 30 Minuten).
- Bereiten Sie während der Inkubation einen Antikörpercocktail zu, indem Sie eine 200-μl-Mastermischung aus Multiplex-Antikörperverdünnung/-block, N-Blocker, G-Blocker, J-Blocker und S-Blocker-Lösungen herstellen.
HINWEIS: Berechnen Sie die Gesamtantikörpermenge basierend auf der Anzahl der Marker und der validierten Titration jedes Markers und subtrahieren Sie die Gesamtantikörpermenge von der Mastermischung. Für sechs Marker mit einer Titration von jeweils 1:200 würde die Gleichung beispielsweise lauten: 200 μl Mastermix − 6 μl Antikörpercocktail = 194 μl Mastermix.
- Nach der Inkubation in dem Multiplex-Antikörperverdünnungsmittel/-block wird die Deckglasprobe in die in Schritt 5.5.1 vorbereitete Feuchtekammer gegeben, 190 μl des Antikörpercocktails auf die Deckglasprobe pipettiert und die Deckglasprobe 3 h lang bei RT inkubiert.
- Waschen Sie die Deckglasprobe nach der Inkubation in zwei Runden mit jeweils 2 min Verdünnungsmittel/Block des Multiplex-Imaging-Antikörpers.
- Um die gebundenen Antikörper am Gewebe auf dem Deckglas zu fixieren, führen Sie die Schritte 5.7.3-5.7.5 in der Feuchtekammer aus.
- Inkubieren Sie die Deckgläser 10 Minuten lang mit 16 % Formaldehyd, verdünnt auf 1,6 % mit Vorratslösung, und waschen Sie die Deckgläser dann dreimal mit 1x PBS.
- Inkubieren Sie die Deckgläser 5 min lang mit 4 °C Methanol und waschen Sie die Deckgläser anschließend dreimal mit 1x PBS.
- Inkubieren Sie die Deckgläser 20 Minuten lang mit 5 ml des mit 1x PBS verdünnten Fixierreagenzes und waschen Sie die Deckgläser dann dreimal mit 1x PBS.
- Lagern Sie die gefärbten Deckglasproben bis zu 2 Wochen im Lagerpuffer bei 4 °C
6. Multiplex-Imaging-Reporterplatte
HINWEIS: Eine 96-Well-Platte, die als Reporterplatte bezeichnet wird und Barcode-Fluorophore in einzelnen Wells enthält, wird nach speziell entwickelten Multiplex-Bildgebungsexperimenten hergestellt und korreliert mit jeder gefärbten Deckglasprobe. Die folgenden Schritte dienen der Vorbereitung der Reporterplatte.
- Bereiten Sie einen Reporter-Mastermix vor, indem Sie 4.880 μl nukleasefreies Wasser, 600 μl 10-fachen Multiplex-Bildgebungspuffer, 500 μl eines Assay-Reagenzes und 20 μl Kernfärbelösung kombinieren. Dies reicht für 20 Zyklen aller Bohrungen.
- Füllen Sie in jedem Zyklus des speziell entwickelten Multiplex-Bildgebungsexperiments eine Vertiefung mit 245 μl einer Lösung, die den Reporter-Mastermix und die spezifischen Barcode-Fluorophore für diesen Zyklus enthält.
HINWEIS: In Abbildung 3 sehen Sie die Konfiguration der Reporterplatte, die in diesem Protokoll für ein Karzinompanel verwendet wird. - Um die Fluorophore mit Barcode zu schützen, kleben Sie eine Folienplattenabdeckung über die Reporterplatte und legen Sie die Platte in das Multiplex-Bildgebungsgerät.
- Lagern Sie die Reporterplatte in einer dunklen Box bei 4 °C bis zu 2 Wochen.
7. Kalibrierung und Betrieb des Multiplex-Bildgebungsgeräts
HINWEIS: Das hochauflösende bildgebende Fluoreszenzmikroskop erfasst vier verschiedene Fluoreszenzkanäle in jedem Multiplex-Bildgebungszyklus bei 20-fachem, 100 % Anregungslicht und mit geringer Photobleichung.
- Kalibrieren Sie den Fokus der Bildgebung mithilfe des DAPI-Kanals, indem Sie ein Probendeckglas auf den Mikroskoptisch legen und 700 μl einer 1:1.500-Titration der Kernfärbelösung manuell auf das Gewebe pipettieren.
Anmerkungen: Das Deckglas wird während der Probenwäsche und Bildgebung auf dem Mikroskoptisch aufbewahrt. - Um das Multiplex-Bildgebungsgerät herzustellen, verdünnen Sie 10x Multiplex-Bildgebungspuffer auf 1x mit DEPC-behandeltem Reinstwasser und füllen Sie Reagenzflaschen mit geeigneten Lösungen/Lösungsmitteln, einschließlich des verdünnten 1x Multiplex-Bildgebungspuffers, DEPC-behandeltem Reinstwasser und Dimethylsulfoxid (DMSO).
- Sobald die Reagenzflaschen ordnungsgemäß befüllt sind, geben Sie das Versuchsdesign in die Multiplex-Software für bildgebende Instrumente ein. Bestimmen Sie den richtigen Zyklus, die Well-Nummern, die Z-Stapel-Stellen, den Markernamen, die Klasse und die Belichtungszeit für jeden Zyklus (Abbildung 4); Stellen Sie alle Parameter des Mikroskops ein. und wählen Sie die gewünschten Bereiche auf dem Beispieldeckglas aus, die abgebildet werden sollen.
- Klicken Sie in der Steuerungssoftware auf die Schaltfläche Experiment (Abbildung 4A). Klicken Sie im Fenster " Experiment einrichten und verwalten " auf die Schaltfläche " Neue Vorlage" (Abbildung 4B).
- Geben Sie den Projektnamen in das Feld neben der Schaltfläche Projekt ein (Abbildung 4C). Geben Sie die Gesamtzahl der Zyklen ein oder wählen Sie sie aus (Abbildung 4D).
- Klicken Sie auf die Schaltfläche Kanalzuweisung , geben Sie die Informationen für jeden Zyklus in die Spalten ein (Abbildung 4E) und klicken Sie auf die Schaltfläche Vorlage speichern . Starten Sie das Experiment, indem Sie auf die Schaltfläche Experiment starten klicken.
HINWEIS: Während eines Multiplex-Bildgebungsexperiments holt das Gerät die Barcode-Fluorophore aus einer Vertiefung der Reporterplatte (maximal vier Fluorophore pro Vertiefung, einschließlich DAPI), gibt sie direkt auf das Probendeckglas ab und bildet die interessierenden Bereiche für jeden Fluoreszenzkanal ab. Nach der gesamten Bildgebung für diesen Zyklus wäscht das Gerät die Barcode-Fluorophore ab und gibt den nächsten Zyklus von Reportern aus (von der nächsten Vertiefung auf der Reporterplatte). Die Bildgebung wird so lange fortgesetzt, bis alle Zyklen mit 26 Markern abgeschlossen sind.
8. Bildersammlung
HINWEIS: Multiplex-Bilder können mit jedem angepassten inversen Fluoreszenzmikroskop aufgenommen werden, das mit vier Fluoreszenzkanälen (DAPI, Cy3, Cy5 und Cy7) konfiguriert und mit einem Plan Fluor 20x-Objektiv ausgestattet ist. Die Bildgebung und das Waschen der Deckglasproben erfolgen iterativ automatisch mit einem speziell entwickelten Fluidik-Aufbau. Die Bilder werden mit der Prozessorsoftware (v1.8.0.7) im QPTIFF-Format aufgenommen.
- Klicken Sie im Prozessorfenster auf die Schaltfläche Eingabe und wählen Sie den Namen des Experiments aus.
- Wählen Sie im Abschnitt " Verarbeitungsoptionen " die Option " Hintergrundsubtraktion", " Entfaltung", " Erweiterte Schärfentiefe" und " Schattierungskorrektur" aus und aktivieren Sie sie. Klicken Sie auf die Schaltfläche Start .
9. Bildanalyse
HINWEIS: Die aufgenommenen Bilder können zur nachgelagerten Analyse in eine patentierte automatisierte Bildanalysesoftware oder ein Open-Source-Softwareprogramm (Abbildung 5) hochgeladen werden.
- Klicken Sie auf das QuPath-Symbol auf dem Computer und öffnen Sie die Software. Ziehen Sie die QPTIFF-Datei in das Viewer-Fenster .
- Klicken Sie auf die Schaltfläche Helligkeit & Kontrast, um das Fenster Helligkeit & Kontrast zu öffnen. Aktivieren oder deaktivieren Sie die Markierung in der Spalte Ausgewählt, um das Markierungssignal anzuzeigen oder zu schließen.
- Klicken Sie auf die Schaltfläche Zum Anpassen zoomen , um den Interessenbereich zu vergrößern oder zu verkleinern.
HINWEIS: Die Visualisierung von Multiplex-Bilddaten bietet dem Benutzer einen tiefen Einblick in die Mikroumgebung des Gewebes. Einzelne Marker in FFPE-Proben können im Fluoreszenzformat (Abbildung 6 und Abbildung 7) oder in der Pathologieansicht visualisiert werden. Mehrere Berechnungsplattformen können verwendet werden, um die zusammengesetzten Bilder zu verarbeiten und die gemultiplexten Gewebebilddaten zu analysieren. Mit dieser Software kann die räumliche Phänotypisierung sowie die Entdeckung seltener Zellen und die Berechnung der Zellnachbarschaft mit Ganzobjektträgerbildern von ultrahochgemultiplextem Gewebe durchgeführt werden, die mit der Multiplex-Bildgebungstechnologie erzeugt wurden. Siehe Abbildung 6 für die 26 Antikörper im Karzinompanel und in der Mandelprobe (ergänzende Abbildung 1).
Ergebnisse
Wir verwendeten FFPE-Mandelproben, um ein immunonkologisches Panel mit 26 Markern zu entwickeln, um den Immunstatus von FFPE-Gewebe mithilfe eines Barcode-Bildanalysesystems zu veranschaulichen. Insgesamt werden derzeit 19 Antikörper in anderen Multiplex-Bildgebungsstudien in unserem Labor verwendet. Alle Marker wurden mit FFPE-Gewebe mit chromogenem IHC getestet. Alle Antikörper wurden an einzigartige DNA-Oligonukleotide konjugiert. Beim Einrichten der Deckgläser mit dem webbasierten Gerätemanager (Abbildung 4) für diese Barcode-Bildanalysetechnologie ist zu beachten, dass der erste und letzte Zyklus immer "leer" sind (Abbildung 2 und Abbildung 4), was die Hintergrundfluoreszenzsignale liefert, die von den spezifischen Signalen der Antikörper subtrahiert werden sollen. Nachdem Bilder im QPTIFF-Format mit dem Fluoreszenzmikroskop aufgenommen wurden, können sie mit mehreren patentierten automatisierten Bildanalyseprogrammen oder Open-Source-Softwareprogrammen visualisiert werden. Zusammengesetzte Bilder können alle Marker oder ausgewählte Marker anzeigen, um eine bessere Ansicht der Signale zu ermöglichen (Abbildung 5). Darüber hinaus kann jeder Antikörper visuell auf nukleäre, zytoplasmatische oder membranöse Lokalisation untersucht werden. Immun-, Tumor- und Stromazellen können leicht identifiziert werden. Anschließend kann die Bildanalyse Aufschluss über die Signalintensität, den Dynamikumfang und die räumliche Verteilung aller Marker geben (Abbildung 6). Diese Technik ermöglichte es uns, alle 26 Marker auf subzellulärer Ebene in einem einzigen Gewebeschnitt zu analysieren (Abbildung 7). Durch die Analyse der Kolokalisation der Marker konnten wir die zellulären Phänotypen identifizieren, die räumliche Zellposition lokalisieren, den Abstand zwischen den Zellen berechnen und die Verteilung der Zellen ermitteln. Die entscheidende Wirkung dieser Technologie ist die Präsentation eines robusten Panels mit 26 Markern, das sich auf den Immunstatus der Gewebemikroumgebung konzentriert.
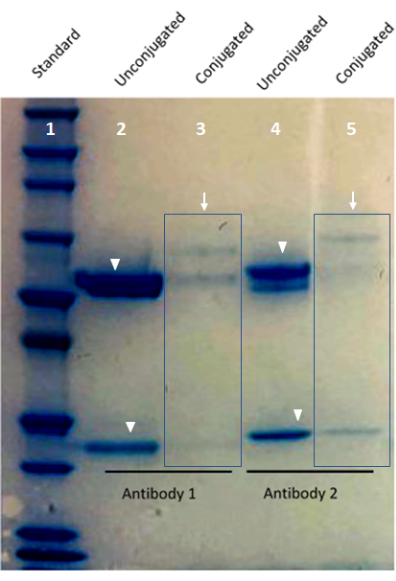
Abbildung 1: Bild der Validierung von kundenspezifisch konjugierten Antikörpern mit einem Bis-Tris-Proteingel. Bahn 1 des Gels zeigt den Proteinstandard. Bahn 2 und Bahn 4 zeigen Barcode-konjugierte Antikörper (Pfeile). Bahn 3 und Bahn 5 zeigen die schweren und leichten Kettenbänder eines unkonjugierten Antikörpers (Pfeilspitzen). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
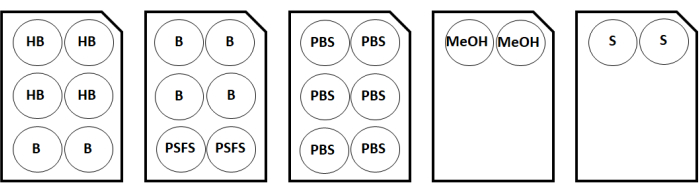
Abbildung 2: Konfigurationskarte der Färbeplatte für zwei Deckglasproben. Abkürzungen: HB = Hydratationspuffer; B = Antikörper-Verdünnungsmittel/-Block; PSFS = Fixierlösung nach der Färbung; PBS = phosphatgepufferte Kochsalzlösung; MeOH = Methanol; S = Speicherlösung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 3: Konfiguration der Reporterplatte. Jeder konjugierte Antikörper hat einen Barcode, der für einen bestimmten Reporter komplementär ist. Um die Reporterplatte einzurichten, sollte jeder konjugierte Antikörper und der dazugehörige Reporter aufgelistet werden. Als nächstes wird jedem Antikörper eine Zyklusnummer zugewiesen. Die Leistung von zwei Blindzyklen (C1 und C18) wird verwendet, um den Grad der Autofluoreszenz in den drei Fluoreszenzkanälen zu bewerten und um die Hintergrundsubtraktion nach der Bildgebung mit der Bilderfassungssteuerungssoftware zu untersuchen. Ein Software-Assistent überprüft das Gerät in dieser Phase, um sicherzustellen, dass alle Einstellungen korrekt sind (Abbildung 4). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
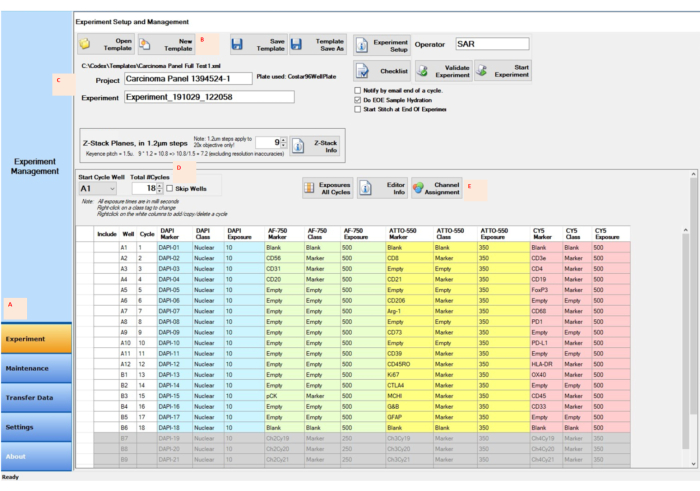
Abbildung 4: Bildaufnahme mit der Steuerungssoftware für den Versuchsaufbau. (A) Wählen Sie die Registerkarte Experiment in der linken unteren Ecke der Steuerungssoftware, um die Einrichtung vorzubereiten und zu starten. (B,C) Wählen Sie Neue Vorlage aus, um die experimentellen Einstellungen mit einem neuen Projekt- und Experimentnamen einzugeben. (D) Ändern Sie das Startzyklusfeld und die Anzahl der Zyklen, um die Position des Reporters in der 96-Well-Reporterplatte widerzuspiegeln. (E) Ordnen Sie den vier für den Versuchslauf vorgesehenen Kanälen die richtigen Fluoreszenzkanäle zu. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 5: Bildvisualisierung mit webbasierter Software (QuPath). Das Betrachterfenster zeigt 26 Marker im FFPE-Schnitt des gefärbten Mandelgewebes. Im Fenster Helligkeit & Kontrast werden die Markierungen mit den Häkchen angezeigt. Schließlich wird im Viewer-Fenster das FFPE-Sample mit den ausgewählten Markern angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
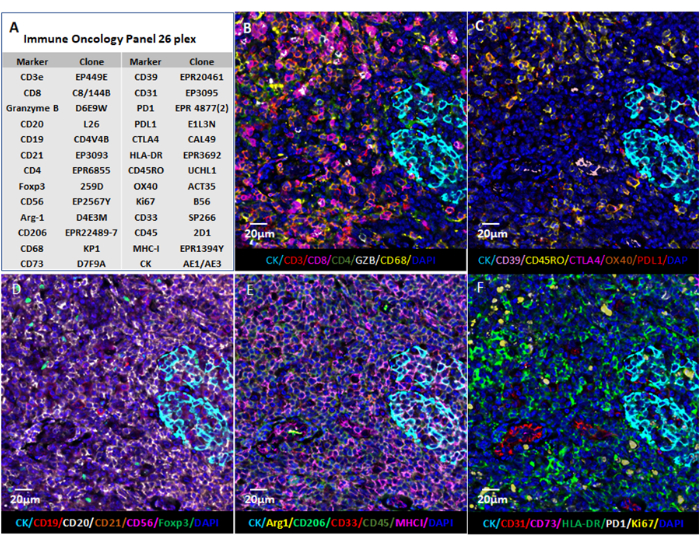
Abbildung 6: Bildvisualisierung mit webbasierter Software. (A) Für die 26 Marker wurden Mandelgewebeschnitte gefärbt, und die Bilder der Schnitte im QPTIFF-Format wurden mit kommerzieller digitaler Diabetrachter-Software oder Open-Source-Software (QuPath) zur Kommentierung und Überprüfung visualisiert. (B-F) Sechs Markierungen wurden in derselben Anmerkung angezeigt, um eine bessere Sicht auf die Signale zu ermöglichen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
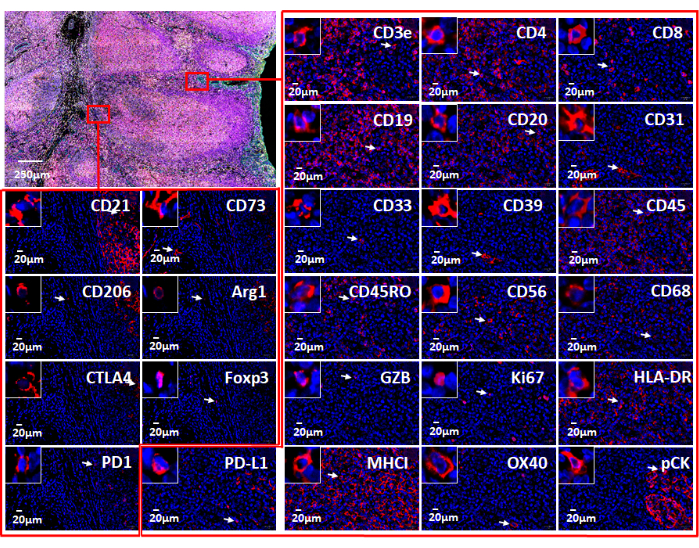
Abbildung 7: Ansichten der 26 einzelnen Marker, die mit webbasierter Software verwendet werden. Die Markerexpression im Mandelgewebe wird mittels Immunfluoreszenzfärbung mit einem immunonkologischen Panel (oben links) dargestellt. Einzelne Markierungen in zwei kleinen Bereichen (rote Rechtecke) werden angezeigt. Die vergrößerte Einfügung zeigt Zellen an, die für diese Markierungen positiv sind (weiße Pfeile). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
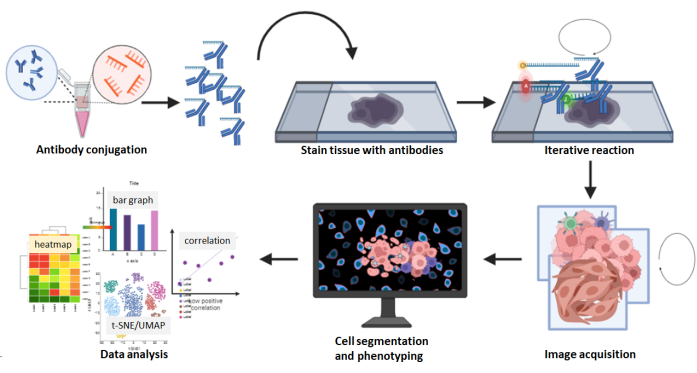
Abbildung 8: Zusammenfassung des Arbeitsablaufs für die Multiplex-Bildaufnahme. FFPE-Gewebeschnitte wurden mit dem 26-Antikörper-Panel angefärbt, gefolgt von einer Mehrzyklusreaktion. Rohbilder der gefärbten Schnitte wurden rechnerisch verarbeitet, und eine Zelldichte- und räumliche Analyse wurde mit den zusammengesetzten Bildern durchgeführt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ergänzende Abbildung 1: IHC-Validierung im Mandelgewebe. FFPE-Gewebeschnitte wurden mit einem individuellen Antikörper angefärbt. Die Markerausprägung im Mandelgewebe wird bei geringer Vergrößerung dargestellt, und die vergrößerte Einlage zeigt Zellen, die positiv für den Marker sind (rote Rechtecke). Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Das TME spielt eine wesentliche Rolle bei der Entwicklung, dem Fortschreiten und dem Ansprechen auf die Behandlung von Krebs. Darüber hinaus kann die Dichte spezifischer tumorinfiltrierender Lymphozyten-Subpopulationen im TME als prognostischer Biomarker für bestimmte Krebsarten dienen. Bemerkenswert ist, dass neben der zellulären Zusammensetzung des TME auch die räumlichen Eigenschaften eines Tumors einen Überblick über das Verständnis der Tumorbiologie und die Identifizierung potenzieller prognostischer Biomarker geben können12,17.
Da zahlreiche Immunzellpopulationen an der Reaktion auf Krebs oder Krebs beteiligt sind, wird ein besseres Verständnis dieser Zellen und ihrer räumlichen Beziehungen zueinander und zu Krebszellen dazu beitragen, neue immuntherapeutische Strategien zu identifizieren. Frühere Studien haben die Lage und räumliche Verteilung von TME-Zellen auf der Grundlage der Gewebestruktur in den intratumoralen und peritumoralen Bereichen und den invasiven Rändern der Tumorzellen stratifiziert18,19. In den letzten 15 Jahren haben technologische Fortschritte die phänotypische Analyse einzelner Zellen auf der Grundlage ihrer räumlichen Ausbreitung zu einem neuartigen, einflussreichen Werkzeug für die Untersuchung der TME und die Kategorisierung potenzieller Biomarker für die Tumorimmuntherapie gemacht. Die Multiplex-IF-Histochemie kann mehrere biologische Marker gleichzeitig schätzen20.
Ähnlich wie bei der Oligonukleotid-konjugierten Antikörperstrategie werden vier Arten von proteinbasierten Multiplex-Plattformen verwendet, um das TME zu untersuchen: Chromogen-, Fluoreszenz-, DNA-Barcode- und Metallisotopen-markierte Antikörper-Detektionssysteme. Die kostengünstigen chromogenen IHC-Plattformen ermöglichen die Visualisierung ganzer Objektträger und die pathologische Beurteilung mittels herkömmlicher Hellfeldmikroskopie. Bei Multiplex-IF und IHC werden Antikörper verwendet, die mit Fluorophoren konjugiert sind. Die Multiplex-IF/IHC-Plattform detektiert Antikörper mit hoher Spezifität und kann zielgerichtete Antikörper auch auf subzellulärer Ebene quantifizieren 6,21. Darüber hinaus kann aufgrund der Beschaffenheit von Chromogenen und Fluorophoren die Verwendung eines Antikörperpanels die Expression von bis zu 10 Biomarkern auf einem einzigen Objektträger erfassen. Auf Metallisotopen-basierten Plattformen werden metallmarkierte Antikörper verwendet, um eine Multiplex-Bildgebung mit Einzelzell- und räumlicher Auflösung und hoher Empfindlichkeit für einzelne Gewebeabschnittedurchzuführen 22. Theoretisch ermöglichen diese metallkonjugierten Antikörperansätze den gleichzeitigen Nachweis von mehr als 100 Biomarkern auf einem einzigen Gewebeschnitt. Eine Herausforderung bei der Isotopenmarkierungstechnik ist die isobare Interferenz, die verhindert, dass eine 100%ige Reinheit der Anreicherung erreicht wird23. Darüber hinaus nimmt die Interferenz mit zunehmender Anzahl der Marker zu. DNA-konjugierte Antikörper-Nachweisplattformen erkennen Antikörper, die mit eindeutigen DNA-Barcodes markiert sind. Auf diesen Plattformen können mehr als 40 Biomarker gleichzeitig mit hoher Spezifität erfasst werden6.
Die Multiplex-Bildgebung ist eine kommerziell erhältliche DNA-Barcode-markierte Antikörper-Nachweisplattform, mit der DNA-konjugierte Antikörper in einem Schritt auf einen einzelnen Gewebeobjektträger aufgebracht werden können (Abbildung 8). Im Gegensatz zur Multiplex-Ionenstrahl-Bildgebungsplattform, die die Verwendung von goldbeschichteten Objektträgern erfordert, die von den Herstellern bezogen werden, benötigt die Multiplex-Bildgebungsplattform für die Gewebevorbereitung nur normale Deckgläser oder Objektträger, die mit 0,1 % Poly-L-Lysin beschichtet sind, um dem Gewebe zu helfen, daran zu haften und das Gewebe während des Färbe- und Bildgebungsprozesses intakt zu halten. Die Verwendung von Gewebeschnitten auf Deckgläsern innerhalb von 4 Wochen nach dem Schnitt wird empfohlen, da die längere Lagerung von ungefärbten Objektträgern zu einer Verringerung der Antigenität führt. Eine gefärbte Deckglasprobe kann bis zu 2 Wochen in einem Lagerpuffer bei 4 °C aufbewahrt werden, ohne dass das Färbesignal verloren geht. Für die Lagerung der Deckglasproben ist keine spezielle Ausrüstung erforderlich. Das Multiplex-Bildgebungssystem wurde aufgerüstet, um normale Objektträger anstelle von Deckgläsern zu verwenden, was die Färbung größerer Gewebe und eine einfache Handhabung ermöglicht. Bei Verwendung einer Reduktionslösung für die Antikörperkonjugation (Schritt 3.2.3) sollte die Reaktion auf nicht mehr als 30 min begrenzt werden, um eine Schädigung der Antikörper zu vermeiden. Die Blockierungspuffer in Schritt 5.6.6 sollten frisch vorbereitet werden, und die Blockierungspuffer dürfen nicht wiederverwendet werden.
Im Vergleich zu chromogen-, fluoreszenz- und metallisotopenmarkierten Multiplex-Antikörper-Detektionsplattformen hat die Multiplex-Bildgebungstechnologie einige Vorteile. So sind beispielsweise mehr als 60 vorgefertigte Antikörper-Panels für die Multiplex-Bildgebung im Handel erhältlich, was dazu beiträgt, Zeit und Kosten bei der Antikörperkonjugation und -validierung zu sparen, und die Zahl der vorgefertigten Antikörper-Panels wächst. Diese Antikörper, zu denen der Karzinommarker Pan-Cytokeratin, der Melanommarker SOX10, der Gefäßmarker CD31, der Stromamarker SMA und zahlreiche Immunzellmarker gehören, sind validiert und experimentierreif. Für Antikörper, die nicht vorgefertigt sind, ist das kommerziell erhältliche Konjugationskit, das für die Verwendung mit Multiplex-Bildgebung entwickelt wurde, unkompliziert und benutzerfreundlich. Kundenkonjugierte Antikörper sind bei Lagerung bei 4 °C 1 Jahr haltbar. Darüber hinaus ist für die Aufnahme der Bilder kein Aufwärmen der Maschine erforderlich. Bei dieser Multiplex-Bildgebungstechnologie führen die iterativen Wasch-, Hybridisierungs- und Stripping-Schritte in der Bildaufnahme selten zu einer verringerten Markerintensität oder einer verschlechterten Gewebemorphologie 5,24,25. Darüber hinaus werden zusammengesetzte Bilder im QPTIFF-Format mit einem einfachen Dreifarben-Fluoreszenzmikroskop aufgenommen und können mit digitaler Analysesoftware von Drittanbietern hochgeladen und analysiert werden. Die Färbemarker können mit Einzelzellauflösung visualisiert werden, und Zellphänotypen können durch die Kolokalisation der Marker charakterisiert werden (Abbildung 6 und Abbildung 7). Die umfassende Analyse eines Multiplexbildes zeigt außerdem die Gewebekompartimente, die Quantifizierung von Einzelzellmarkern sowie die Daten zum nächsten Nachbarn und zur Nähe (Abbildung 8).
Eine Herausforderung bei der Multiplex-Bildanalyse ist die Zelltypidentifikation. Wenn mehr Einzelobjekt-Klassifikatoren auf ein Bild angewendet werden, werden in der Regel ungewöhnlichere Phänotypen annotiert. Daher wird empfohlen, bekannte Marker zu verwenden, die nicht im selben Klassifikator co-exprimiert werden, und nur den phänotypbezogenen Klassifikator auf die Annotation einzelner Zellen anzuwenden. Variationen in der Zelltyp-Annotation führen zu wesentlich unterschiedlichen räumlichen Ergebnissen, wie z. B. Unterschiede in der räumlichen Verteilung der Zellen und der zellulären Nachbarschaftsanalyse26,27.
Die Multiplex-Bildanalyse hat sich bei der Färbung und Bildgebung vieler Probentypen als erfolgreich erwiesen, darunter FFPE-Gewebe, frisch gefrorenes Gewebe, archivierte ganze Objektträger und Gewebe-Microarrays. Multiplexbilder von Brust-, Gehirn-, Lungen-, Milz-, Nieren-, Lymphknoten- und Hautgewebeschnitten können mit tiefen einzelligen räumlichen Phänotypisierungsdaten aufgenommen werden 5,16,25,28.
In Zukunft werden weitere vorgefertigte Antikörper für die Multiplex-Bildgebung erwartet. Darüber hinaus ist die Entwicklung spezifischer Software für die Multiplex-Bildanalyse dringend erforderlich. Derzeit gibt es viele kommerziell erhältliche und quelloffene Softwareprogramme für die Hi-Plex-Bildanalyse29, aber die Wissenschaftler benötigen immer noch Hilfe bei der Erstellung eines Standard-Workflows für diese Analysen30,31. Obwohl die mit diesem Protokoll aufgenommenen zusammengesetzten Bilder mit Software von Drittanbietern kompatibel sind, kann dies zu zusätzlichen Kosten für den Benutzer führen. Ein weiterer Nachteil der Multiplex-Bildgebungstechnologie ist die Signalreduktion bei der Detektion von Kernproteinen nach iterativem Waschen, Hybridisieren und mit großen Antikörperplatten. Glücklicherweise kann dies minimiert werden, indem die Barcode-Fluorophore bei frühen Zyklen beim Design der Reporterplatten abgerufen werden. Vor kurzem wurde diese Plattform mit einem neuen Hochgeschwindigkeits-Scansystem aufgerüstet, das die Zeit bis zur Erstellung von zusammengesetzten Bildern drastisch verkürzt hat32. Darüber hinaus wurde über eine neue Strategie mit Tyramid-konjugierten Barcodes berichtet, um die auf Oligonukleotiden konjugierte Antikörper-Barcoding-basierte Bildgebung zu verbessern. Diese Technologie zielt darauf ab, Färbesignale zu verstärken, für die Barcode-konjugierte Antikörper schwer zu erhalten sind33.
Offenlegungen
Die Autoren haben keine Konflikte offenzulegen.
Danksagungen
Die Autoren danken Donald R. Norwood von Editing Services, Research Medical Library bei MD Anderson für die Bearbeitung dieses Artikels und dem Multiplex-IF- und Bildanalyselabor in der Abteilung für Translationale Molekularpathologie bei MD Anderson. Dieses Projekt wurde zum Teil von der Moonshots-Plattform des Translational Molecular Pathology-Immunoprofiling Laboratory (TMP-IL) am Department of Translational Molecular Pathology, dem MD Anderson Cancer Center der University of Texas und dem NCI Cooperative Agreement U24CA224285 (an das MD Anderson Cancer Center CIMAC) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 10x AR9 Buffer | Akoya Biosciences | AR900250ML | |
| 10x Buffer | Akoya Biosciences | 7000001 | |
| 16% paraformaldehyde | Thermo Fisher Scientific | 28906 | |
| 1X Antibody Diluent/Block | Akoya Biosciences | ARD1001EA | |
| Antibody Conjugation Kit | Akoya Biosciences | 7000009 | Contains Filter Blocking Solution, Antibody Reduction Solution 1, Antibody Reduction Solution 2, Conjugation Solution, Purification Solution, Antibody Storage Solution |
| Assay Reagent | Akoya Biosciences | 7000002 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| Ethanol, 200 proof | |||
| G Blocker V2 | Akoya Biosciences | 240199 | |
| Histoclear | Thermo Fisher Scientific | 50-329-50 | |
| Methanol | Sigma-Aldrich | 34860-1L-R | |
| Milli-Q Integral 10 | Millipore | ZRXQ010WW | |
| Niknon Fluorescence microscope | Keyence Corp. of America | BZ-X810 | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| Nuclease-free water | Thermo Fisher Scientific | AM9938 | |
| NuPAGE | Thermo Fisher Scientific | NP0008 | |
| PBS | Thermo Fisher Scientific | 14190136 | |
| PhenoCycler Barcodes/Reporters Combination | Akoya Biosciences | 5450004 (BX025/RX025) | |
| 5450003 (BX022/RX022) | |||
| 5450023 (BX002/RX002) | |||
| 5250002 (BX020/RX020) | |||
| 2520003 (BX023/RX023) | |||
| 5250005 (BX029/RX029) | |||
| 5250007 (BX035/RX035) | |||
| 5250012 (BX052/RX052) | |||
| 5550012 (BX030/RX030) | |||
| 5550015 (BX042/RX042) | |||
| 5550014 (BX036/RX036) | |||
| QuPath | Open-Source | https://qupath.github.io/ | |
| SimplyBlue SafeStain | Thermo Fisher Scientific | LC6065 | |
| Staining Kit | (Akoya Biosciences | 7000008 | Contains Hydration Buffer, N Blocker, J Blocker, S Blocker, Fixative Reagent, Storage Buffer |
Referenzen
- Hanahan, D., Coussens, L. M. Accessories to the crime: Functions of cells recruited to the tumor microenvironment. Cancer Cell. 21 (3), 309-322 (2012).
- Labani-Motlagh, A., Ashja-Mahdavi, M., Loskog, A. The tumor microenvironment: A milieu hindering and obstructing antitumor immune responses. Frontiers in Immunology. 11, 940 (2020).
- Jin, M. Z., Jin, W. L. The updated landscape of tumor microenvironment and drug repurposing. Signal Transduction and Targeted Therapy. 5 (1), 166 (2020).
- Waldman, A. D., Fritz, J. M., Lenardo, M. J. A guide to cancer immunotherapy: From T cell basic science to clinical practice. Nature Reviews Immunology. 20 (11), 651-668 (2020).
- Black, S., et al. CODEX multiplexed tissue imaging with DNA-conjugated antibodies. Nature Protocols. 16 (8), 3802-3835 (2021).
- Tan, W. C. C., et al. Overview of multiplex immunohistochemistry/immunofluorescence techniques in the era of cancer immunotherapy. Cancer Communications. 40 (4), 135-153 (2020).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 117 (52), 33455-33465 (2020).
- Allam, M., Cai, S., Coskun, A. F. Multiplex bioimaging of single-cell spatial profiles for precision cancer diagnostics and therapeutics. NPJ Precision Oncology. 4, 11 (2020).
- Eng, J., et al. A framework for multiplex imaging optimization and reproducible analysis. Communications Biology. 5 (1), 438 (2022).
- Taube, J. M., et al. Multi-institutional TSA-amplified multiplexed immunofluorescence reproducibility evaluation (MITRE) study. Journal for Immunotherapy of Cancer. 9 (7), e002197 (2021).
- Binnewies, M., et al. Understanding the tumor immune microenvironment (TIME) for effective therapy. Nature Medicine. 24 (5), 541-550 (2018).
- Heindl, A., Nawaz, S., Yuan, Y. Mapping spatial heterogeneity in the tumor microenvironment: A new era for digital pathology. Laboratory Investigation. 95 (4), 377-384 (2015).
- Kuswanto, W., Nolan, G., Lu, G. Highly multiplexed spatial profiling with CODEX: bioinformatic analysis and application in human disease. Seminars in Immunopathology. 45 (1), 145-157 (2022).
- Phillips, D., et al. Immune cell topography predicts response to PD-1 blockade in cutaneous T cell lymphoma. Nature Communications. 12 (1), 6726 (2021).
- Jhaveri, N., et al. Deep ultrahigh-plex spatial phenotyping of human cancer tissues. Cancer Research. 82, 3877 (2022).
- Goltsev, Y., et al. Deep profiling of mouse splenic architecture with CODEX multiplexed imaging. Cell. 174 (4), 968-981 (2018).
- Yuan, Y. Spatial heterogeneity in the tumor microenvironment. Cold Spring Harbor Perspectives in Medicine. 6 (8), a026583 (2016).
- Bruck, O., et al. Spatial immunoprofiling of the intratumoral and peritumoral tissue of renal cell carcinoma patients. Modern Pathology. 34 (12), 2229-2241 (2021).
- Tsujikawa, T., et al. Prognostic significance of spatial immune profiles in human solid cancers. Cancer Science. 111 (10), 3426-3434 (2020).
- Hoyt, C. C. Multiplex immunofluorescence and multispectral imaging: forming the basis of a clinical test platform for immuno-oncology. Frontiers in Molecular Biosciences. 8, 674747 (2021).
- Parra, E. R., et al. Identification of distinct immune landscapes using an automated nine-color multiplex immunofluorescence staining panel and image analysis in paraffin tumor tissues. Scientific Reports. 11 (1), 4530 (2021).
- Elaldi, R., et al. High dimensional imaging mass cytometry panel to visualize the tumor immune microenvironment contexture. Frontiers in Immunology. 12, 666233 (2021).
- Campos Clemente, L., Shi, O., Rojas, F., Parra, E. R. Expanding the comprehension of the tumor microenvironment using mass spectrometry imaging of formalin-fixed and paraffin-embedded tissue samples. Journal of Visualized Experiments. (184), e64015 (2022).
- Schurch, C. M., et al. Coordinated cellular neighborhoods orchestrate antitumoral immunity at the colorectal cancer invasive front. Cell. 182 (5), 1341-1359 (2020).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by CODEX tissue imaging. Frontiers in Immunology. 12, 687673 (2021).
- Hickey, J. W., Tan, Y., Nolan, G. P., Goltsev, Y. Strategies for accurate cell type identification in CODEX multiplexed imaging data. Frontiers in Immunology. 12, 727626 (2021).
- Parra, E. R. Methods to determine and analyze the cellular spatial distribution extracted from multiplex immunofluorescence data to understand the tumor microenvironment. Frontiers in Molecular Biosciences. 8, 668340 (2021).
- Tzoras, E., et al. Dissecting tumor-immune microenvironment in breast cancer at a spatial and multiplex resolution. Cancers. 14 (8), 1999 (2022).
- Francisco-Cruz, A., Parra, E. R., Tetzlaff, M. T. Wistuba, II. Multiplex immunofluorescence assays. Methods in Molecular Biology. 2055, 467-495 (2020).
- Shakya, R., Nguyen, T. H., Waterhouse, N., Khanna, R. Immune contexture analysis in immuno-oncology: applications and challenges of multiplex fluorescent immunohistochemistry. Clinical and Translational Immunology. 9 (10), e1183 (2020).
- Wilson, C. M., et al. Challenges and opportunities in the statistical analysis of multiplex immunofluorescence data. Cancers. 13 (12), 3031 (2021).
- DeRosa, J. Setting a new standard for spatial omics: An integrated multiomics approach: every single cell, two different analytes, one unbiased picture. Genetic Engineering & Biotechnology News. 42, 26-28 (2022).
- Simonson, P. D., Valencia, I., Patel, S. S. Tyramide-conjugated DNA barcodes enable signal amplification for multiparametric CODEX imaging. Communications Biology. 5 (1), 627 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten