Method Article
Análise de Imagem de Código de Barras Multiplexado para Caracterização de Imunoprofiling e Mapeamento Espacial na Análise Unicelular de Amostras de Parafina
Neste Artigo
Resumo
A análise de imagens multiplexadas com código de barras tem melhorado recentemente a caracterização do microambiente tumoral, permitindo estudos abrangentes da composição celular, estado funcional e interações célula-célula. Neste artigo, descrevemos um protocolo de coloração e imagem usando o código de barras de anticorpos conjugados a oligonucleotídeos e imagens cíclicas, que permite o uso de uma técnica de análise de imagens de alta dimensão.
Resumo
A tecnologia de imagem multiplexada usando código de barras de anticorpos com oligonucleotídeos, que detecta sequencialmente múltiplos epítopos no mesmo corte de tecido, é uma metodologia eficaz para avaliação tumoral que melhora a compreensão do microambiente tumoral. A visualização da expressão de proteínas em tecidos fixados em formalina e emblocados em parafina é obtida quando um fluoróforo específico é recozido em um código de barras ligado a anticorpos via oligonucleotídeos complementares e, em seguida, imagens de amostras são realizadas; De fato, esse método permite o uso de painéis personalizáveis de mais de 40 anticorpos em uma única reação de coloração de tecido. Este método é compatível com tecido fresco congelado, tecido fixado em formalina, tecido embebido em parafina, células cultivadas e células mononucleares do sangue periférico, o que significa que os pesquisadores podem usar essa tecnologia para visualizar uma variedade de tipos de amostra em resolução de célula única. Este método começa com um protocolo manual de coloração e fixação, e todos os códigos de barras de anticorpos são aplicados usando um coquetel de anticorpos. O instrumento de coloração fluídica é totalmente automatizado e realiza ciclos iterativos de marcação, geração de imagens e remoção de fluoróforos espectralmente distintos até que todos os biomarcadores tenham sido imageados usando um microscópio de fluorescência padrão. As imagens são então coletadas e compiladas em todos os ciclos de imagem para alcançar resolução de célula única para todos os marcadores. A coloração em etapa única e a remoção suave do fluoróforo não apenas permitem a análise de biomarcadores altamente multiplexados, mas também preservam a amostra para análise adicional a jusante, se desejado (por exemplo, coloração de hematoxilina e eosina). Além disso, o software de análise de imagens permite o processamento de imagens - compensação de deriva, subtração de fundo, segmentação celular e agrupamento - bem como a visualização e análise das imagens e fenótipos celulares para a geração de mapas de redes espaciais. Em resumo, essa tecnologia emprega um sistema microfluídico computadorizado e um microscópio de fluorescência para hibridizar, obter imagens e retirar sondas de DNA marcadas fluorescentemente que são complementares aos anticorpos conjugados a oligonucleotídeos ligados ao tecido.
Introdução
O microambiente tumoral (TME) é extremamente heterogêneo, constituído por células tumorais, células estromais tumorais, células imunes, componentes não celulares da matriz extracelular e numerosas moléculas abundantes produzidas e liberadas por células tumorais, estromais eimunes1,2. Evidências acumuladas demonstram que a ETM tem um papel fundamental na reprogramação da diferenciação, crescimento, invasão, metástase e resposta a terapiastumorais3.
Compreender como os diferentes tipos celulares da EMT interagem e se comunicam entre si por meio de redes de sinalização é essencial para melhorar o diagnóstico do câncer, otimizar a imunoterapia e desenvolver novos tratamentos4. Técnicas tradicionais de microscopia tecidual, incluindo imunohistoquímica (IHQ) e imunofluorescência (IF), têm sido usadas há décadas para estudar os tipos celulares, abundância e comunicações em amostras tumorais. Infelizmente, essas técnicas tipicamente podem avaliar apenas um ou dois marcadores proteicos em um corte tecidual e não podem revelar as complexas relações espaciais e estruturais entre essas células 5,8,7.
Nas últimas duas décadas, várias tecnologias de imagem multiplexada foram estabelecidas8. Essas tecnologias fornecem visões muito melhores da composição, função e localização das células imunes dentro da TME, levando a rápidos avanços na capacidade de identificar e perfilar espacialmente TMEs complexas no nível de célula única 9,10. As relações espaciais e estruturais de várias células tumorais e imunes na EMT estão atualmente na vanguarda dos estudos biológicos e clínicos que utilizam essas tecnologias de imagem multiplexada11,12.
A tecnologia de imagem multiplexada recentemente desenvolvida usando código de barras de anticorpos conjugados com oligonucleotídeos é uma plataforma de pesquisa biológica unicelular influente baseada na detecção de anticorpos conjugados a oligonucleotídeos em amostras fixadas em formalina e emblocadas em parafina (FFPE)13,14. Atualmente, essa tecnologia de imagem multiplexada permite a obtenção simultânea de imagens de mais de 100 marcadores em um único corte de tecido15, o que aumentou o número de tipos celulares que são distinguíveis in situ. Isso permite um nível de análise espacial de células tumorais e imunes que não é possível usando as abordagens tradicionais deimunofenotipagem16.
Neste artigo, descrevemos um protocolo otimizado para conjugar anticorpos purificados contra oligonucleotídeos e validar essa conjugação usando a plataforma de imagem multiplexada e um procedimento de imagem multiciclo com tecido FFPE. Além disso, descrevemos os procedimentos básicos de processamento e análise de dados utilizados com essa tecnologia.
Protocolo
Este estudo retrospectivo foi aprovado pelo Comitê de Ética em Pesquisa do MD Anderson Cancer Center da Universidade do Texas. As amostras de tecido FFPE foram coletadas de pacientes no MD Anderson como parte do tratamento padrão de rotina. Não foram realizadas intervenções diagnósticas ou terapêuticas. Consentimento informado foi obtido dos pacientes para utilização das amostras coletadas para pesquisa e publicação.
1. Fontes de anticorpos usadas para o design do painel de anticorpos
- Crie um painel de anticorpos para imagens multiplexadas depois de considerar cuidadosamente a qualidade dos tecidos e proteínas de interesse. Três fontes de anticorpos são consideradas para o design do painel de anticorpos: 1) anticorpos totalmente validados comercialmente, 2) anticorpos multiplexados rastreados por tecnologia de imagem e 3) anticorpos compartilhados pelo usuário final.
NOTA: Os anticorpos rastreados por tecnologia de imagem multiplexada mostraram funcionar e estão disponíveis nos fornecedores. Esses anticorpos rastreados podem ser aplicados na coloração de imagens multiplexadas após conjugação de oligonucleotídeos pelo usuário. - Se uma proteína de interesse não puder ser encontrada nas fontes mencionadas acima, empregue os clones de anticorpos que são conhecidos por trabalhar com IHQ. Além disso, isotipos de IgG em vez de clones de IgM são recomendados para essa tecnologia de imagem multiplexada devido à maior taxa de falha com clones de IgM do que com clones de IgG.
2. Antes da conjugação de anticorpos
- Ao identificar clones de anticorpos para conjugação usando códigos de barras de imagem multiplexados, considere a compra de anticorpos livres de transportador em solução salina tamponada com fosfato (PBS) ou um tampão semelhante. Proteínas carreadoras, incluindo BSA, glúten, glicerol e outros aditivos proteicos, são conhecidas por reduzir a capacidade de conjugação.
- Selecione o clone de anticorpos mais adequado e sempre otimize as condições de coloração (isto é, recuperação e titulação do antígeno) antes da conjugação. Faça isso aplicando um clone de anticorpo não conjugado ao tecido positivo e negativo para esse anticorpo usando IF ou IHQ padrão.
NOTA: Se um anticorpo purificado não estiver comercialmente disponível, um processo de purificação de anticorpos deve ser realizado antes da conjugação. O procedimento de purificação usado com kits de purificação de anticorpos não é discutido aqui.
3. Conjugação de anticorpos
- Obter os reagentes de conjugação de anticorpos. Os kits de conjugação comercialmente disponíveis contêm uma solução de bloqueio de filtro, solução de redução 2, solução de conjugação, solução de purificação, solução de armazenamento de anticorpos (todos armazenados a 4 °C) e solução de redução 1 (armazenados a -20 °C).
- Conjugação
NOTA: Um anticorpo purificado é tratado com um agente redutor, permitindo que as metades reduzidas do anticorpo reajam com o código de barras de imagem multiplexado usado com esta tecnologia e, assim, formem uma ligação covalente. Esse processo leva cerca de 4,5 h e resulta em aproximadamente 120 μL de anticorpo conjugado, o que é viável por 1 ano. Todo o spin-down é conduzido à temperatura ambiente (TR), e o flow-through é descartado, exceto na última etapa (3.2.9), na qual um tubo de coleta contém o anticorpo conjugado.- Aspirar e aplicar 500 μL da solução de bloqueio do filtro usando uma pipeta nas colunas do filtro de corte de peso molecular de 50 kDa e girar para baixo a 12.000 x g por 2 min para bloquear a ligação de anticorpos inespecíficos.
NOTA: Se a solução residual na parte superior da coluna for perceptível, inverta o filtro no tubo de coleta e gire para baixo a 3.000 x g por 2 min. - Pipetar 50 μg do anticorpo em um volume de 100 μL de solução para a coluna filtrante e girar para baixo a 12.000 x g por 8 min.
NOTA: Utilizar um espectrofotómetro para medir a concentração do anticorpo purificado e calcular o volume de solução correspondente a 50 μg do anticorpo. Se o volume da solução de anticorpos for inferior a 100 μL, ajuste o volume para 100 μL adicionando 1x PBS. Reter 1 μg de anticorpo não conjugado para confirmação da conjugação (passo 4.4). - Pipetar 260 μL de mistura mestre de redução (20 μL de solução de redução 1 misturada com 825 μL de solução de redução 2, suficiente para três reações de conjugação de anticorpos) para cada coluna filtrante. Vórtice suavemente a mistura mestra por 2-3 s, ou pipetar para cima e para baixo para misturar a solução com o anticorpo. Incubar em TR por 30 min.
- Após a incubação, gire as colunas do filtro a 12.000 x g por 8 min. Adicionar 450 μL de solução de conjugação às colunas filtrantes e girar para baixo a 12.000 x g durante 8 min.
- Ressuspender o código de barras desejado em 10 μL de água livre de nuclease e 210 μL de solução de conjugação.
NOTA: Prepare uma etiqueta de anticorpos imediatamente antes de usar para a coloração. Não reutilize alíquotas de código de barras de anticorpos. - Após a conclusão da rotação descendente na etapa 3.2.4, adicionar a etiqueta de anticorpos ressuspensa (aproximadamente 220 μL) em cada coluna de filtro correspondente. Pipetar a mistura para cima e para baixo suavemente para misturar os reagentes. Feche as tampas da coluna filtrante e incube a reação de conjugação em RT por 2 h. Após 2 h, gire as colunas do filtro a 12.000 x g por 8 min.
NOTA: Para o protocolo de confirmação recomenda-se reservar 5 μL da solução conjugada num tubo de reacção em cadeia da polimerase e armazená-la a 4 °C (ver abaixo). - Pipetar 450 μL da solução de purificação para cada coluna filtrante e girar para baixo a 12.000 x g durante 8 minutos. Repita três vezes.
- Pipetar 100 μL da solução de armazenamento para as colunas filtrantes. Pipetar suavemente a mistura para cima e para baixo mais de 10 vezes e lavar cuidadosamente as laterais dos filtros na coluna.
NOTA: Dissolver 50 μg de anticorpo em 100 μL da solução de armazenamento. Se a reação de conjugação for iniciada com mais de 50 μg de anticorpo, adicione mais solução de armazenamento nessa proporção. - Inverta as colunas de filtro em um tubo de coleta novo. Gire para baixo a 3.000 x g por 2 min em RT. Mantenha a solução coletada. Pipetar a solução de anticorpos conjugados em tubos estéreis com tampa de rosca e armazenar por até 1 ano a 4 °C.
NOTA: Um anticorpo conjugado deve ser testado com a tecnologia de imagem multiplexada após 2 dias. Testes anteriores a isso podem resultar em alta coloração nuclear de fundo.
- Aspirar e aplicar 500 μL da solução de bloqueio do filtro usando uma pipeta nas colunas do filtro de corte de peso molecular de 50 kDa e girar para baixo a 12.000 x g por 2 min para bloquear a ligação de anticorpos inespecíficos.
4. Confirmação da conjugação
NOTA: Antes de realizar experiências de coloração com um anticorpo conjugado pelo utilizador utilizando a tecnologia de imagem multiplexada, a conjugação deve ser confirmada utilizando electroforese em gel com 5 μL do anticorpo conjugado (ver passo 3.2.6) juntamente com 1 μg de um anticorpo não conjugado (geralmente em 2 μL da mistura) como controlo. Uma conjugação de anticorpos bem sucedida será demonstrada por um aumento no peso molecular do anticorpo. No entanto, este protocolo de confirmação avalia apenas o sucesso da reação química para conjugação e não aborda a validação de anticorpos usada para imagens multiplexadas.
- Pipetar 8 μL e 11 μL de água livre de nucleases para o anticorpo conjugado reservado e controlar o anticorpo não conjugado, respectivamente, para obter um volume final de 13 μL.
- Pipetar 5 μL de tampão de amostra LDS (ou outro sistema de electroforese em gel de dodecil sulfato de sódio-poliacrilamida) e 2 μL de agente redutor de amostra em cada amostra do anticorpo conjugado reservado e desnaturar as amostras num banho seco a 95 °C durante 10 minutos.
- Enquanto as amostras estão desnaturando, diluir 40 mL de tampão de corrida MOPS SDS em 760 mL de água ultrapura, colocar um gel no tanque de um sistema de eletroforese e despejar o tampão de corrida diluído sobre o gel.
- Uma vez completado o período de desnaturação de 10 min, carregar um poço do gel com um padrão proteico pré-corado, um com o anticorpo não conjugado (a partir do passo 3.2.2) e os poços restantes com as amostras de anticorpos conjugados. Em seguida, passe o gel a 150 V até que o padrão proteico apareça no final do gel.
NOTA: O gel adere facilmente a recipientes seguros para micro-ondas e, portanto, pode rasgar, por isso o gel deve ser manuseado com cautela nas etapas seguintes. - Após a conclusão da corrida, transfira o gel para um recipiente seguro para micro-ondas pré-preenchido com água ultrapura e aqueça-o em um micro-ondas até que a primeira bolha na água seja visualizada.
NOTA: O tempo para a formação de bolhas varia muito dependendo do micro-ondas utilizado. - Escorra a água do recipiente, despeje cerca de 250 mL de coloração Coomassie G-250 sobre o gel e aqueça o gel em um micro-ondas até que a primeira bolha seja visualizada. Depois, retire o recipiente com o gel e a mancha Coomassie G-250 do micro-ondas e coloque-o em um agitador por 10 min.
- Depois de agitar, escorra cuidadosamente a mancha, substitua-a por cerca de 200 mL de água ultrapura e, em seguida, coloque o recipiente no agitador para lavar o gel.
- Escorra a água ultrapura e substitua-a por água ultrapura nova cinco vezes ou até que os restos da mancha não sejam aparentes no banho-maria. Deixe o gel lavar durante a noite no agitador até que as bandas fiquem visíveis, se necessário, antes de fotografar o gel (Figura 1).
NOTA: Os anticorpos usados em imagens multiplexadas devem ter padrões de coloração comparáveis aos dos anticorpos conjugados com corantes. Cortes teciduais com antígenos conhecidos positivos para anticorpos conjugados podem ser corados com anticorpos conjugados a oligonucleotídeos e corantes. Neste trabalho, em cada caso, as morfologias teciduais para ambos os tipos de anticorpos foram equivalentes e harmonizadas entre si, bem como a distribuição celular prevista, com base na biologia das proteínas de interesse e das amostras de tecido teste. Este resultado demonstra a eficácia do uso de metades de anticorpos conjugados com oligonucleotídeos para abordagens baseadas em coloração tecidual usando tecido FFPE.
5. Coloração de anticorpos conjugados a oligonucleotídeos
- Prepare lamínulas para colocação de tecidos.
- Mergulhe as lamínulas em uma solução de poli-L-lisina a 0,1% por 24 h na RT para melhorar a aderência tecidual.
- Após a imersão, escorra a solução de poli-L-lisina e lave as lamínulas com água ultrapura por 30 s. Repita a lavagem de quatro a seis vezes. Retire as lamínulas da água ultrapura usada para lavar e coloque-as em uma toalha sem fiapos para secar durante a noite.
NOTA: Um histologista deve cortar o tecido selecionado em cortes com 5 μm de espessura, colocá-los no centro de uma lamínula carregada de poli-L-lisina e deixá-los secar durante a noite. Após a secagem, as lamínulas com os cortes de tecido devem ser armazenadas a 4 °C por um período máximo de 6 meses. O painel de coloração deste protocolo continha 26 marcadores. O tecido utilizado neste protocolo foi tecido tonsilar humano normal. O tempo de imersão não deve exceder 1 semana. As lamínulas revestidas de poli-L-lisina podem ser armazenadas em RT e devem ser utilizadas no prazo de 2 meses após a preparação.
- No dia anterior à mancha, coloque o suporte de tampa em um forno a 60 °C durante a noite.
- No dia seguinte, coloque a amostra de lamínula num suporte de lamínula pré-aquecido num forno a 60 °C. Depois de assar por 30 min, verifique se a parafina derreteu longe do tecido.
- Coloque rapidamente o suporte/amostra da tampa na seguinte série de soluções: duas rodadas de agente desparafinador por 6 min cada; duas rodadas de etanol 100% por 5 min cada; uma rodada de etanol 90% por 5 min; uma rodada de etanol 70% por 5 min; uma rodada de etanol 50% por 5 min; uma rodada de etanol 30% por 5 min; e duas rodadas de água ultrapura tratada com dietilpirocarbonato (DEPC) por 5 min cada.
NOTA: Todas as diluições de etanol são preparadas com água ultrapura tratada com DEPC. Se o xileno for usado para desparafinação, ele deve ser usado em uma coifa. - Ao submeter o suporte/amostra da tampa à série de soluções, faça o seguinte:
- Prepare uma câmara de umidade colocando uma caixa de ponta de pipeta vazia com uma toalha de papel embebida em água na parte inferior.
- Encha uma panela de pressão com água suficiente para cobrir metade um copo de 50 mL.
- Colocar 5 ml de metanol por amostra no copo a 4 °C.
- Diluir o tampão AR9 com água ultrapura tratada com DEPC a 1x; são necessários cerca de 50 ml do tampão diluído por suporte de lamínula.
- Depois que a série de soluções estiver completa, encha um copo de vidro de 50 mL com cerca de 40 mL de tampão 1x AR9, submerja o suporte/amostra de tampa no copo e cubra completamente o copo/suporte de deslizamento com folha de alumínio.
- Coloque o copo coberto com papel alumínio na panela de pressão cheia de água e cozinhe em alta pressão (cerca de 15 psi) por 20 min.
- Após o cozimento, remova o copo/suporte de tampa, desembrulhe cuidadosamente o papel alumínio e deixe o copo/suporte de tampa esfriar em RT por cerca de 10 minutos.
- Remova o suporte/amostra da tampa do tampão AR9 1x e submerja-o em duas rodadas de água ultrapura tratada com DEPC, realizando uma incubação das amostras em ambas as rodadas por 2 min cada.
- Durante a incubação, recupere o tampão de hidratação, o bloqueio N, o bloqueio G, o bloqueio J e o bloqueio S e o diluente/bloqueio de anticorpos do kit de coloração de imagem multiplexada. Para duas amostras de lamínula, rotule as placas de 6 poços para as soluções usando as configurações mostradas na Figura 2.
- Colocar a amostra em duas rodadas de 5 mL do tampão de hidratação por 2 min cada.
- Após ambas as rodadas de colocação no tampão de hidratação, colocar a amostra de lamínula em 5 mL do diluente/bloco de anticorpos de imagem multiplexado e incubar por 20-30 min em RT (não exceder 30 min).
- Durante a incubação, prepare um coquetel de anticorpos fazendo uma mistura mestra de 200 μL de diluente/bloqueio de anticorpos de imagem multiplexada, bloqueador N, bloqueador G, bloqueador J e soluções de bloqueador S.
NOTA: Calcule a quantidade total de anticorpos com base no número de marcadores e na titulação validada de cada marcador e subtraia a quantidade total de anticorpos da mistura mestre. Por exemplo, para seis marcadores, cada um com uma titulação de 1:200, a equação seria 200 μL de mistura mestre − 6 μL de coquetel de anticorpos = 194 μL de mistura mestre.
- Após a incubação no diluente/bloco de anticorpos de imagem multiplexada, colocar a amostra de lamínula na câmara de humidade preparada na etapa 5.5.1, pipetar 190 μL do cocktail de anticorpos na amostra de lamínula e incubar a amostra de lamínula em RT durante 3 h.
- Após a incubação, lavar a amostra em dois rounds com 5 mL do diluente/bloco de anticorpos multiplexados por imagem por 2 min cada.
- Para fixar os anticorpos ligados ao tecido na lamínula, execute as etapas 5.7.3-5.7.5 na câmara de umidade.
- Incubar as lamínulas por 10 min com formaldeído a 16% diluído a 1,6% com solução de armazenamento e, em seguida, lavar as lamínulas três vezes com 1x PBS.
- Incubar as lamínulas durante 5 minutos com metanol a 4 °C e, em seguida, lavar as lamínulas três vezes com 1x PBS.
- Incubar as lamínulas por 20 min com 5 mL do reagente fixador diluído com 1x PBS e, em seguida, lavar as lamínulas três vezes com 1x PBS.
- Conservar as amostras coradas no tampão de armazenamento a 4 °C durante um período máximo de 2 semanas
6. Placa de repórter de imagem multiplexada
NOTA: Uma placa de 96 poços, referida como placa de relatório, contendo fluoróforos com código de barras em poços individuais é preparada de acordo com experimentos de imagem multiplexados projetados sob medida e correlaciona-se com cada amostra de lamínula corada. Os passos a seguir são para a preparação da chapa do repórter.
- Prepare uma mistura mestre de repórter combinando 4.880 μL de água livre de nuclease, 600 μL de 10x tampão de imagem multiplexado, 500 μL de um reagente de ensaio e 20 μL de solução de coloração nuclear. Isso será suficiente para 20 ciclos de todos os poços.
- Em cada ciclo do experimento de imagem multiplexada personalizado, preencha um poço com 245 μL de uma solução contendo a mistura mestre do repórter e os fluoróforos com código de barras específicos para esse ciclo.
NOTA: Consulte a Figura 3 para a configuração da placa repórter usada neste protocolo para um painel de carcinoma. - Para proteger os fluoróforos com código de barras, cole uma tampa de placa de folha sobre a placa repórter e coloque a placa dentro do instrumento de imagem multiplexado.
- Conservar a placa do repórter numa caixa escura a 4 °C até 2 semanas.
7. Calibrando e executando a máquina de imagem multiplexada
NOTA: O microscópio de fluorescência de imagem de alta resolução captura quatro canais de fluorescência diferentes em cada ciclo de imagem multiplexado a 20x, 100% de luz de excitação e com baixo fotoclareamento.
- Calibrar o foco da imagem usando o canal DAPI colocando uma lamínula de amostra no estágio do microscópio, pipetando manualmente 700 μL de uma titulação 1:1.500 de solução corante nuclear no tecido.
NOTA: A lamínula é retida no estágio do microscópio durante a lavagem da amostra e a obtenção de imagens. - Para preparar o instrumento de imagem multiplexado, dilua 10x o buffer de imagem multiplexado para 1x usando água ultrapura tratada com DEPC e encha garrafas de reagente com soluções/solventes apropriados, incluindo o tampão de imagem multiplexado diluído 1x, água ultrapura tratada com DEPC e dimetilsulfóxido (DMSO).
- Uma vez que os frascos de reagente estejam enchidos adequadamente, insira o desenho experimental no software gerenciador de instrumentos de imagem multiplexado; designar o ciclo correto, os números dos poços, os locais da pilha z, o nome do marcador, a classe e o tempo de exposição para cada ciclo (Figura 4); definir todos os parâmetros do microscópio; e selecione as regiões de interesse na folha de capa da amostra a ser fotografada.
- Clique no botão Experimento no software de controle (Figura 4A). Na janela Experiment Setup and Management , clique no botão New Template (Figura 4B).
- Digite o nome do projeto no espaço ao lado do botão Project (Figura 4C). Digite ou selecione o número total de ciclos (Figura 4D).
- Clique no botão Channel Assignment , digite as informações de cada ciclo nas colunas (Figura 4E) e clique no botão Save Template . Inicie o experimento clicando no botão Iniciar experimento .
NOTA: Durante um experimento de imagem multiplexada, o instrumento recupera os fluoróforos com código de barras de um poço da placa repórter (máximo de quatro fluoróforos por poço, incluindo DAPI), os dispensa diretamente na folha de cobertura da amostra e obtém imagens das regiões de interesse para cada canal de fluorescência. Seguindo todas as imagens desse ciclo, o instrumento lava os fluoróforos com código de barras e dispensa o próximo ciclo de repórteres (do próximo poço na placa do repórter). As imagens continuam até que todos os ciclos usando 26 marcadores tenham sido concluídos.
8. Coleta de imagens
NOTA: As imagens multiplexadas podem ser coletadas usando qualquer microscópio de fluorescência invertido adaptado configurado com quatro canais de fluorescência (DAPI, Cy3, Cy5 e Cy7) e equipado com uma lente Plan Fluor 20x. A obtenção de imagens e a lavagem das amostras de lamínula são realizadas de forma iterativa automaticamente usando uma configuração fluídica especialmente desenvolvida. As imagens são adquiridas utilizando o software Processor (v1.8.0.7) no formato QPTIFF.
- Na janela do processador, clique no botão Entrada e selecione o nome do experimento.
- Na seção Opções de Processamento , selecione e marque Subtração em Segundo Plano, Deconvolução, Profundidade de Campo Estendida e Correção de Sombreamento. Clique no botão Iniciar .
9. Análise das imagens
NOTA: As imagens adquiridas podem ser carregadas em um software patenteado de análise automatizada de imagens ou em um programa de software de código aberto (Figura 5) para análise a jusante.
- Clique no ícone QuPath no computador e abra o software. Arraste o arquivo QPTIFF para a janela do Visualizador .
- Clique no botão Brilho e Contraste, que abrirá a janela Brilho e Contraste. Marque ou desmarque o marcador na coluna Selecionado para mostrar ou fechar o sinal do marcador.
- Clique no botão Zoom para ajustar para ampliar ou reduzir a área de interesse.
NOTA: A visualização de dados de imagem multiplexada fornece ao usuário uma visão profunda do microambiente do tecido. Marcadores individuais em amostras FFPE podem ser visualizados em formato de fluorescência (Figura 6 e Figura 7) ou em visão anatomopatológica. Várias plataformas computacionais podem ser usadas para processar as imagens compostas e analisar os dados de imagens de tecido multiplexadas. Usando este software, a fenotipagem espacial, bem como a descoberta de células raras e computação de vizinhança celular, pode ser realizada com imagens de lâminas inteiras de tecido multiplexado ultra-alto gerado através da tecnologia de imagem multiplexada. Ver Figura 6 para os 26 anticorpos no painel de carcinoma e amostra de amígdalas (Figura suplementar 1).
Resultados
Empregamos amostras de tonsilas FFPE para desenvolver um painel imuno-oncológico de 26 marcadores para ilustrar o estado imunológico do tecido FFPE usando um sistema de análise de imagem com código de barras. No total, 19 anticorpos são atualmente usados em outros estudos de imagem multiplexados em nosso laboratório. Todos os marcadores foram testados usando tecido FFPE com IHQ cromogênica. Todos os anticorpos foram conjugados a oligonucleotídeos únicos de DNA. Ao configurar as lamínulas usando o gerenciador de instrumentos baseado na web (Figura 4) para essa tecnologia de análise de imagens de código de barras, deve-se notar que o primeiro e o último ciclos são sempre "em branco" (Figura 2 e Figura 4), o que fornece os sinais de fluorescência de fundo a serem subtraídos dos sinais específicos dos anticorpos. Após a coleta de imagens no formato QPTIFF com o microscópio de fluorescência, elas podem ser visualizadas usando vários softwares patenteados de análise automatizada de imagens ou programas de software de código aberto. Imagens compostas podem mostrar todos os marcadores ou marcadores selecionados para uma melhor visualização dos sinais (Figura 5). Além disso, cada anticorpo pode ser avaliado visualmente quanto à localização nuclear, citoplasmática ou membranosa. Células imunológicas, tumorais e estromais podem ser facilmente identificadas. Posteriormente, a análise das imagens pode fornecer informações sobre a intensidade do sinal, a amplitude dinâmica e a distribuição espacial de todos os marcadores (Figura 6). Essa técnica permitiu analisar todos os 26 marcadores em nível subcelular em um único corte tecidual (Figura 7). Analisando a co-localização dos marcadores, foi possível identificar os fenótipos celulares, localizar a posição espacial celular, calcular a distância entre as células e encontrar a distribuição das células. O impacto crucial dessa tecnologia é a apresentação de um robusto painel de 26 marcadores focado no estado imunológico do microambiente tecidual.
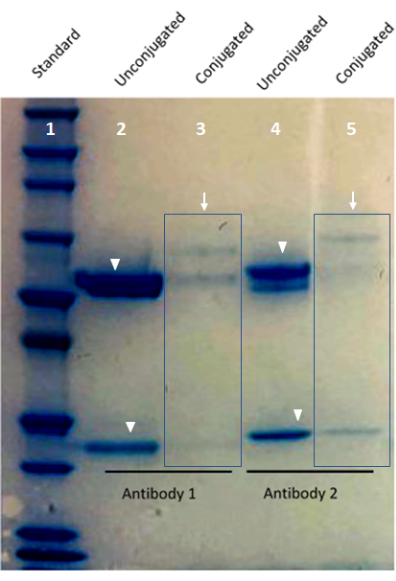
Figura 1: Imagem de validação de anticorpos conjugados personalizados usando um gel de proteína Bis-Tris. A faixa 1 do gel mostra o padrão proteico. As faixas 2 e 4 apresentam anticorpos conjugados ao código de barras (setas). A faixa 3 e a faixa 5 mostram as bandas de cadeia pesada e leve de um anticorpo não conjugado (pontas de setas). Clique aqui para ver uma versão maior desta figura.
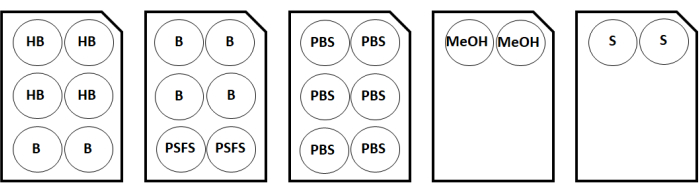
Figura 2: Mapa de configuração da placa de coloração para duas amostras de lamínula. Abreviações: HB = tampão de hidratação; B = diluente/bloqueio de anticorpos; PSFS = solução fixadora pós-coloração; PBS = solução salina tamponada com fosfato; MeOH = metanol; S = solução de armazenamento. Clique aqui para ver uma versão maior desta figura.

Figura 3: Configuração da placa do repórter. Cada anticorpo conjugado tem um código de barras que é complementar a um repórter específico. Para montar a placa de repórter, cada anticorpo conjugado e seu correspondente repórter devem ser listados. Em seguida, a cada anticorpo é atribuído um número de ciclo. O desempenho de dois ciclos em branco (C1 e C18) é utilizado para avaliar o nível de autofluorescência nos três canais de fluorescência e para a subtração de fundo pós-imagem usando o software de controle de aquisição de imagens. Um assistente de software verificará o instrumento neste estágio para garantir que todas as configurações estejam corretas (Figura 4). Clique aqui para ver uma versão maior desta figura.
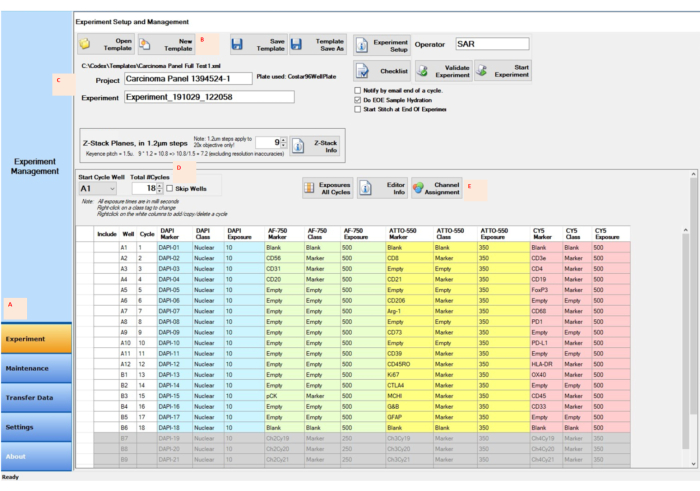
Figura 4: Aquisição das imagens utilizando o software de controle para a montagem do experimento. (A) Selecione a guia Experiência no canto inferior esquerdo do software de controle para preparar e iniciar a configuração. (B,C) Selecione Novo modelo para inserir as configurações experimentais com um novo nome de projeto e experimento. (D) Altere o poço do ciclo inicial e o número de ciclos para refletir a localização do repórter na placa do repórter de 96 poços. (E) Atribuir os canais de fluorescência adequados aos quatro canais designados para a corrida experimental. Clique aqui para ver uma versão maior desta figura.

Figura 5: Visualização de imagens utilizando software baseado na web (QuPath). A janela do visualizador mostra 26 marcadores no corte FFPE do tecido amigdaliano corado. A janela Brilho e Contraste mostra os marcadores com as marcas de seleção. Finalmente, a janela do visualizador mostra a amostra FFPE com os marcadores selecionados. Clique aqui para ver uma versão maior desta figura.
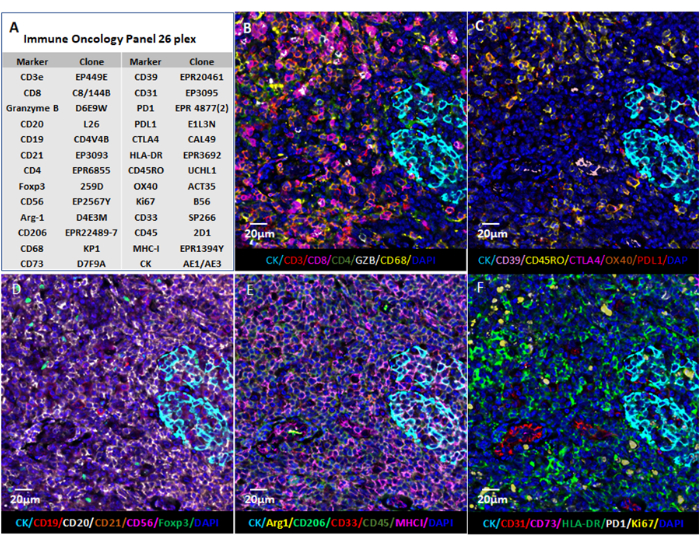
Figura 6: Visualização das imagens utilizando software baseado na web. (A) Cortes de tecido tonsilar foram corados para os 26 marcadores, e as imagens dos cortes em formato QPTIFF foram visualizadas usando software visualizador digital comercial de lâminas ou software livre (QuPath) para anotação e revisão. (B-F) Seis marcadores foram exibidos na mesma anotação para melhor visualização dos sinais. Clique aqui para ver uma versão maior desta figura.
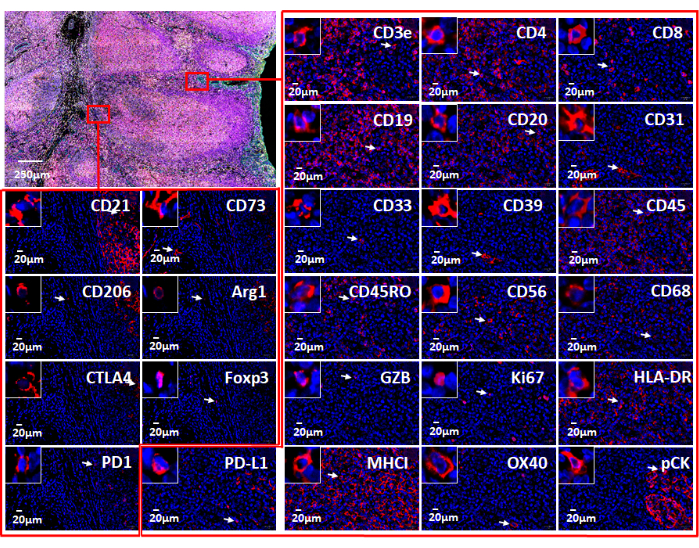
Figura 7: Visualizações dos 26 marcadores individuais usados com software baseado na web. A expressão do marcador no tecido tonsilar é demonstrada através da coloração por imunofluorescência com um painel de imuno-oncologia (canto superior esquerdo). Marcadores individuais em duas pequenas áreas (retângulos vermelhos) são mostrados. A inserção ampliada mostra células positivas para esses marcadores (setas brancas). Clique aqui para ver uma versão maior desta figura.
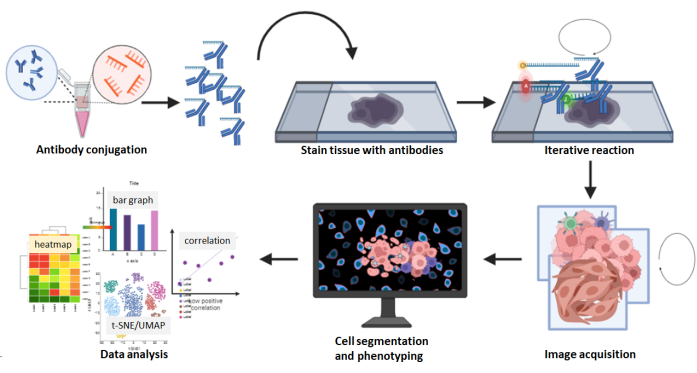
Figura 8: Resumo do fluxo de trabalho de aquisição de imagens multiplexadas. Cortes teciduais FFPE foram corados usando o painel de 26 anticorpos, seguido por uma reação multiciclo. Imagens brutas dos cortes corados foram processadas computacionalmente, e uma análise espacial e de densidade celular foi realizada usando as imagens compostas. Clique aqui para ver uma versão maior desta figura.
Figura Suplementar 1: Validação da IHQ em tecido tonsilar. Cortes teciduais FFPE foram corados com um anticorpo individual. A expressão do marcador no tecido tonsilar é mostrada em uma ampliação baixa, e a inserção ampliada mostra células positivas para o marcador (retângulos vermelhos). Clique aqui para baixar este arquivo.
Discussão
A EMT desempenha um papel essencial no desenvolvimento, progressão e resposta ao tratamento do câncer. Além disso, a densidade de subtipos específicos de linfócitos infiltrantes de tumor na ETM pode servir como um biomarcador prognóstico para certos tipos de câncer. Notavelmente, além da composição celular da ETM, as características espaciais de um tumor podem fornecer um esboço para o entendimento da biologia do tumor e identificação de potenciais biomarcadoresprognósticos12,17.
Como numerosas populações de células imunes estão envolvidas em respostas pró-câncer ou anticâncer, uma melhor compreensão dessas células e suas relações espaciais entre si e com as células cancerosas ajudará a orientar a identificação de novas estratégias imunoterápicas. Estudos prévios estratificaram a localização e a distribuição espacial das células TME com base na estrutura tecidual nas áreas intratumorais e peritumorais e nas margens invasivas das célulastumorais18,19. Nos últimos 15 anos, os avanços tecnológicos tornaram a análise fenotípica de células individuais com base em sua dispersão espacial uma ferramenta nova e influente para estudar a ETM e categorizar potenciais biomarcadores para imunoterapia tumoral. A histoquímica do FI multiplex pode estimar simultaneamente múltiplos marcadores biológicos20.
Semelhante à estratégia de anticorpos conjugados com oligonucleotídeo, quatro tipos de plataformas multiplex baseadas em proteínas são usados para estudar a TME: sistemas de detecção de anticorpos marcados com cromogênio, fluorescência, DNA barcode e isótopos metálicos. As plataformas de IHQ cromogênicas de baixo custo permitem a visualização de lâminas inteiras e a avaliação patológica usando microscopia convencional de campo claro. No FI multiplexado e na IHQ, anticorpos conjugados com fluoróforos são usados. A plataforma multiplex IF/IHQ detecta anticorpos com alta especificidade e pode quantificar os anticorpos direcionados mesmo em nível subcelular 6,21. Além disso, devido à natureza dos cromógenos e fluoróforos, o uso de um painel de anticorpos pode capturar a expressão de até 10 biomarcadores em uma única lâmina. Em plataformas baseadas em isótopos metálicos, anticorpos marcados com metal são usados para realizar imagens multiplexadas com resolução espacial e de célula única, e alta sensibilidade para cortes individuais de tecido22. Teoricamente, essas abordagens de anticorpos conjugados a metais permitem a detecção simultânea de mais de 100 biomarcadores em uma única seção de tecido. Um desafio da técnica de marcação isotópica é a interferência isobárica, que impede que se atinja 100% de pureza do enriquecimento23. Além disso, a interferência aumenta à medida que o número de marcadores aumenta. As plataformas de detecção de anticorpos conjugados com DNA reconhecem anticorpos marcados com códigos de barras de DNA exclusivos. Mais de 40 biomarcadores podem ser capturados simultaneamente com alta especificidade nessasplataformas6.
A imagem multiplexada é uma plataforma de detecção de anticorpos marcados com código de barras de DNA comercialmente disponível para a aplicação de anticorpos conjugados ao DNA em uma única lâmina de tecido em uma única etapa (Figura 8). Para a etapa de preparação do tecido, ao contrário da plataforma de imagem por feixe de íons multiplexado, que requer o uso de lâminas revestidas com ouro obtidas dos fabricantes, a plataforma de imagem multiplexada requer apenas lamínulas regulares ou lâminas revestidas com poli-L-lisina a 0,1% para ajudar o tecido a aderir a ele e manter o tecido intacto durante o processo de coloração e imagem. Recomenda-se o uso de cortes de tecido em lamínulas dentro de 4 semanas após a secção, pois o armazenamento prolongado de lâminas não coradas resulta em redução da antigenicidade. Uma amostra de lamínula corada pode ser mantida em tampão de armazenamento a 4 °C por até 2 semanas sem perder seu sinal de coloração. Não é necessário nenhum equipamento especial para o armazenamento das amostras de lamínula. O sistema de imagem multiplexado foi atualizado para usar lâminas regulares em vez de lamínulas, o que permite a coloração de tecidos maiores e fácil manuseio. Ao utilizar uma solução de redução para conjugação de anticorpos (passo 3.2.3), a reação deve ser limitada a um máximo de 30 minutos para evitar danos aos anticorpos. Os buffers de bloqueio da etapa 5.6.6 devem ser preparados recentemente e os buffers de bloqueio não devem ser reutilizados.
Em comparação com plataformas de detecção de anticorpos multiplex marcadas com cromógenos, fluorescência e isótopos metálicos, a tecnologia de imagem multiplexada tem certas vantagens. Por exemplo, mais de 60 painéis de anticorpos pré-projetados para imagens multiplexadas estão comercialmente disponíveis, o que ajuda a economizar tempo e custos na conjugação e validação de anticorpos, e o número de painéis de anticorpos pré-projetados está crescendo. Esses anticorpos, que incluem o marcador de carcinoma pancitoqueratina, o marcador de melanoma SOX10, o marcador vascular CD31, o marcador estromal SMA e vários marcadores de células imunes, são validados e prontos para experimentação. Para anticorpos que não são pré-projetados, o kit de conjugação disponível comercialmente projetado para uso com imagens multiplexadas é simples e fácil de usar. Os anticorpos conjugados pelo cliente são bons por 1 ano quando armazenados a 4 °C. Além disso, o aquecimento da máquina não é necessário para capturar as imagens. Nessa tecnologia de imagem multiplexada, as etapas iterativas de lavagem, hibridização e remoção na aquisição das imagens raramente resultam em diminuição da intensidade dos marcadores ou morfologia tecidual degradada 5,24,25. Além disso, as imagens compostas são capturadas no formato QPTIFF com um microscópio de fluorescência tricolor simples e podem ser carregadas e analisadas usando software de análise digital de terceiros. Os marcadores de coloração podem ser visualizados em resolução unicelular, e os fenótipos celulares podem ser caracterizados através da co-localização dos marcadores (Figura 6 e Figura 7). A análise abrangente de uma imagem multiplexada revela ainda os compartimentos teciduais, a quantificação do marcador unicelular e os dados de vizinhança e proximidade mais próximos (Figura 8).
Um desafio na análise de imagens multiplexadas é a identificação do tipo celular. Normalmente, quando mais classificadores de objeto único são aplicados a uma imagem, fenótipos mais incomuns serão anotados. Portanto, recomenda-se o uso de marcadores conhecidos que não são co-expressos no mesmo classificador e a aplicação apenas do classificador relacionado ao fenótipo à anotação de células únicas. Variações na anotação do tipo celular resultarão em resultados espaciais substancialmente diferentes, tais como diferenças na distribuição espacial celular e análise de vizinhança celular26,27.
A análise de imagens multiplexadas provou ser bem-sucedida na coloração e geração de imagens de muitos tipos de amostras, incluindo tecido FFPE, tecido fresco congelado, lâminas inteiras arquivadas e microarrays de tecido. Imagens multiplexadas de cortes de mama, cérebro, pulmão, baço, rim, linfonodo e tecido cutâneo podem ser adquiridas com dados profundos de fenotipagem espacial unicelular 5,16,25,28.
No futuro, mais anticorpos pré-projetados para imagens multiplexadas são esperados. Além disso, o desenvolvimento de softwares específicos para análise de imagens multiplexadas é extremamente necessário. Atualmente, existem muitos softwares disponíveis comercialmente e de código aberto para análise de imagens Hi-Plex29, mas os cientistas ainda precisam de ajuda na criação de um fluxo de trabalho padrão para essas análises30,31. Embora as imagens compostas capturadas usando este protocolo sejam compatíveis com software de terceiros, isso pode resultar em custos extras para o usuário. Outra desvantagem da tecnologia de imagem multiplexada é a redução do sinal na detecção de proteínas nucleares após lavagem iterativa, hibridização e remoção com grandes painéis de anticorpos. Felizmente, isso pode ser minimizado recuperando os fluoróforos com código de barras nos ciclos iniciais ao projetar as placas do repórter. Recentemente, essa plataforma foi atualizada com um novo sistema de varredura de alta velocidade, que reduziu drasticamente o tempo de obtenção de imagens compostas32. Além disso, uma nova estratégia usando códigos de barras conjugados com tiramida foi relatada para melhorar a imagem baseada em código de barras de anticorpos conjugados a oligonucleotídeo. Essa tecnologia visa amplificar sinais de coloração para os quais anticorpos conjugados com código de barras são difíceis de obter33.
Divulgações
Os autores não têm conflitos a revelar.
Agradecimentos
Os autores agradecem a Donald R. Norwood da Editing Services, Research Medical Library at MD Anderson pela edição deste artigo e ao IF multiplex e laboratório de análise de imagens do Departamento de Patologia Molecular Translacional do MD Anderson. Este projeto foi apoiado em parte pelo laboratório Translational Molecular Pathology-Immunoprofiling (TMP-IL) Moonshots Platform no Departamento de Patologia Molecular Translacional, The University of Texas MD Anderson Cancer Center e NCI Cooperative Agreement U24CA224285 (para o MD Anderson Cancer Center CIMAC).
Materiais
| Name | Company | Catalog Number | Comments |
| 10x AR9 Buffer | Akoya Biosciences | AR900250ML | |
| 10x Buffer | Akoya Biosciences | 7000001 | |
| 16% paraformaldehyde | Thermo Fisher Scientific | 28906 | |
| 1X Antibody Diluent/Block | Akoya Biosciences | ARD1001EA | |
| Antibody Conjugation Kit | Akoya Biosciences | 7000009 | Contains Filter Blocking Solution, Antibody Reduction Solution 1, Antibody Reduction Solution 2, Conjugation Solution, Purification Solution, Antibody Storage Solution |
| Assay Reagent | Akoya Biosciences | 7000002 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| Ethanol, 200 proof | |||
| G Blocker V2 | Akoya Biosciences | 240199 | |
| Histoclear | Thermo Fisher Scientific | 50-329-50 | |
| Methanol | Sigma-Aldrich | 34860-1L-R | |
| Milli-Q Integral 10 | Millipore | ZRXQ010WW | |
| Niknon Fluorescence microscope | Keyence Corp. of America | BZ-X810 | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| Nuclease-free water | Thermo Fisher Scientific | AM9938 | |
| NuPAGE | Thermo Fisher Scientific | NP0008 | |
| PBS | Thermo Fisher Scientific | 14190136 | |
| PhenoCycler Barcodes/Reporters Combination | Akoya Biosciences | 5450004 (BX025/RX025) | |
| 5450003 (BX022/RX022) | |||
| 5450023 (BX002/RX002) | |||
| 5250002 (BX020/RX020) | |||
| 2520003 (BX023/RX023) | |||
| 5250005 (BX029/RX029) | |||
| 5250007 (BX035/RX035) | |||
| 5250012 (BX052/RX052) | |||
| 5550012 (BX030/RX030) | |||
| 5550015 (BX042/RX042) | |||
| 5550014 (BX036/RX036) | |||
| QuPath | Open-Source | https://qupath.github.io/ | |
| SimplyBlue SafeStain | Thermo Fisher Scientific | LC6065 | |
| Staining Kit | (Akoya Biosciences | 7000008 | Contains Hydration Buffer, N Blocker, J Blocker, S Blocker, Fixative Reagent, Storage Buffer |
Referências
- Hanahan, D., Coussens, L. M. Accessories to the crime: Functions of cells recruited to the tumor microenvironment. Cancer Cell. 21 (3), 309-322 (2012).
- Labani-Motlagh, A., Ashja-Mahdavi, M., Loskog, A. The tumor microenvironment: A milieu hindering and obstructing antitumor immune responses. Frontiers in Immunology. 11, 940 (2020).
- Jin, M. Z., Jin, W. L. The updated landscape of tumor microenvironment and drug repurposing. Signal Transduction and Targeted Therapy. 5 (1), 166 (2020).
- Waldman, A. D., Fritz, J. M., Lenardo, M. J. A guide to cancer immunotherapy: From T cell basic science to clinical practice. Nature Reviews Immunology. 20 (11), 651-668 (2020).
- Black, S., et al. CODEX multiplexed tissue imaging with DNA-conjugated antibodies. Nature Protocols. 16 (8), 3802-3835 (2021).
- Tan, W. C. C., et al. Overview of multiplex immunohistochemistry/immunofluorescence techniques in the era of cancer immunotherapy. Cancer Communications. 40 (4), 135-153 (2020).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 117 (52), 33455-33465 (2020).
- Allam, M., Cai, S., Coskun, A. F. Multiplex bioimaging of single-cell spatial profiles for precision cancer diagnostics and therapeutics. NPJ Precision Oncology. 4, 11 (2020).
- Eng, J., et al. A framework for multiplex imaging optimization and reproducible analysis. Communications Biology. 5 (1), 438 (2022).
- Taube, J. M., et al. Multi-institutional TSA-amplified multiplexed immunofluorescence reproducibility evaluation (MITRE) study. Journal for Immunotherapy of Cancer. 9 (7), e002197 (2021).
- Binnewies, M., et al. Understanding the tumor immune microenvironment (TIME) for effective therapy. Nature Medicine. 24 (5), 541-550 (2018).
- Heindl, A., Nawaz, S., Yuan, Y. Mapping spatial heterogeneity in the tumor microenvironment: A new era for digital pathology. Laboratory Investigation. 95 (4), 377-384 (2015).
- Kuswanto, W., Nolan, G., Lu, G. Highly multiplexed spatial profiling with CODEX: bioinformatic analysis and application in human disease. Seminars in Immunopathology. 45 (1), 145-157 (2022).
- Phillips, D., et al. Immune cell topography predicts response to PD-1 blockade in cutaneous T cell lymphoma. Nature Communications. 12 (1), 6726 (2021).
- Jhaveri, N., et al. Deep ultrahigh-plex spatial phenotyping of human cancer tissues. Cancer Research. 82, 3877 (2022).
- Goltsev, Y., et al. Deep profiling of mouse splenic architecture with CODEX multiplexed imaging. Cell. 174 (4), 968-981 (2018).
- Yuan, Y. Spatial heterogeneity in the tumor microenvironment. Cold Spring Harbor Perspectives in Medicine. 6 (8), a026583 (2016).
- Bruck, O., et al. Spatial immunoprofiling of the intratumoral and peritumoral tissue of renal cell carcinoma patients. Modern Pathology. 34 (12), 2229-2241 (2021).
- Tsujikawa, T., et al. Prognostic significance of spatial immune profiles in human solid cancers. Cancer Science. 111 (10), 3426-3434 (2020).
- Hoyt, C. C. Multiplex immunofluorescence and multispectral imaging: forming the basis of a clinical test platform for immuno-oncology. Frontiers in Molecular Biosciences. 8, 674747 (2021).
- Parra, E. R., et al. Identification of distinct immune landscapes using an automated nine-color multiplex immunofluorescence staining panel and image analysis in paraffin tumor tissues. Scientific Reports. 11 (1), 4530 (2021).
- Elaldi, R., et al. High dimensional imaging mass cytometry panel to visualize the tumor immune microenvironment contexture. Frontiers in Immunology. 12, 666233 (2021).
- Campos Clemente, L., Shi, O., Rojas, F., Parra, E. R. Expanding the comprehension of the tumor microenvironment using mass spectrometry imaging of formalin-fixed and paraffin-embedded tissue samples. Journal of Visualized Experiments. (184), e64015 (2022).
- Schurch, C. M., et al. Coordinated cellular neighborhoods orchestrate antitumoral immunity at the colorectal cancer invasive front. Cell. 182 (5), 1341-1359 (2020).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by CODEX tissue imaging. Frontiers in Immunology. 12, 687673 (2021).
- Hickey, J. W., Tan, Y., Nolan, G. P., Goltsev, Y. Strategies for accurate cell type identification in CODEX multiplexed imaging data. Frontiers in Immunology. 12, 727626 (2021).
- Parra, E. R. Methods to determine and analyze the cellular spatial distribution extracted from multiplex immunofluorescence data to understand the tumor microenvironment. Frontiers in Molecular Biosciences. 8, 668340 (2021).
- Tzoras, E., et al. Dissecting tumor-immune microenvironment in breast cancer at a spatial and multiplex resolution. Cancers. 14 (8), 1999 (2022).
- Francisco-Cruz, A., Parra, E. R., Tetzlaff, M. T. Wistuba, II. Multiplex immunofluorescence assays. Methods in Molecular Biology. 2055, 467-495 (2020).
- Shakya, R., Nguyen, T. H., Waterhouse, N., Khanna, R. Immune contexture analysis in immuno-oncology: applications and challenges of multiplex fluorescent immunohistochemistry. Clinical and Translational Immunology. 9 (10), e1183 (2020).
- Wilson, C. M., et al. Challenges and opportunities in the statistical analysis of multiplex immunofluorescence data. Cancers. 13 (12), 3031 (2021).
- DeRosa, J. Setting a new standard for spatial omics: An integrated multiomics approach: every single cell, two different analytes, one unbiased picture. Genetic Engineering & Biotechnology News. 42, 26-28 (2022).
- Simonson, P. D., Valencia, I., Patel, S. S. Tyramide-conjugated DNA barcodes enable signal amplification for multiparametric CODEX imaging. Communications Biology. 5 (1), 627 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados