Method Article
Analisi di immagini di codici a barre multiplexed per l'immunoprofilazione e la caratterizzazione della mappatura spaziale nell'analisi a singola cellula di campioni di tessuto di paraffina
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
L'analisi delle immagini con codici a barre multiplexed ha recentemente migliorato la caratterizzazione del microambiente tumorale, consentendo studi completi sulla composizione cellulare, sullo stato funzionale e sulle interazioni cellula-cellula. Qui, descriviamo un protocollo di colorazione e imaging che utilizza il codice a barre di anticorpi coniugati con oligonucleotidi e l'imaging ciclico, che consente l'uso di una tecnica di analisi delle immagini ad alta dimensionalità.
Abstract
La tecnologia di imaging multiplexed che utilizza il codice a barre anticorpale con oligonucleotidi, che rileva sequenzialmente più epitopi nella stessa sezione di tessuto, è una metodologia efficace per la valutazione del tumore che migliora la comprensione del microambiente tumorale. La visualizzazione dell'espressione proteica in tessuti fissati in formalina e incorporati in paraffina si ottiene quando uno specifico fluoroforo viene ricotto a un codice a barre legato agli anticorpi tramite oligonucleotidi complementari e quindi viene eseguita l'imaging del campione; Infatti, questo metodo consente l'uso di pannelli personalizzabili di oltre 40 anticorpi in una singola reazione di colorazione tissutale. Questo metodo è compatibile con tessuto fresco congelato, tessuto fissato in formalina, tessuto incorporato in paraffina, cellule in coltura e cellule mononucleate del sangue periferico, il che significa che i ricercatori possono utilizzare questa tecnologia per visualizzare una varietà di tipi di campioni a risoluzione singola cellulare. Questo metodo inizia con un protocollo di colorazione e fissaggio manuale e tutti i codici a barre degli anticorpi vengono applicati utilizzando un cocktail di anticorpi. Lo strumento di fluidica di colorazione è completamente automatizzato ed esegue cicli iterativi di etichettatura, imaging e rimozione di fluorofori spettralmente distinti fino a quando tutti i biomarcatori non sono stati ripresi utilizzando un microscopio a fluorescenza standard. Le immagini vengono quindi raccolte e compilate in tutti i cicli di imaging per ottenere una risoluzione a cella singola per tutti i marcatori. La colorazione in un'unica fase e la delicata rimozione dei fluorofori non solo consentono un'analisi dei biomarcatori altamente multiplexata, ma conservano anche il campione per ulteriori analisi a valle, se lo si desidera (ad esempio, colorazione con ematossilina ed eosina). Inoltre, il software di analisi delle immagini consente l'elaborazione delle immagini - compensazione della deriva, sottrazione dello sfondo, segmentazione cellulare e clustering - nonché la visualizzazione e l'analisi delle immagini e dei fenotipi cellulari per la generazione di mappe di rete spaziali. In sintesi, questa tecnologia impiega un sistema di microfluidica computerizzato e un microscopio a fluorescenza per ibridare iterativamente, visualizzare e strisciare sonde di DNA marcate con fluorescenza che sono complementari agli anticorpi coniugati con oligonucleotidi legati ai tessuti.
Introduzione
Il microambiente tumorale (TME) è estremamente eterogeneo, costituito da cellule tumorali, cellule stromali tumorali, cellule immunitarie, componenti non cellulari della matrice extracellulare e numerose molecole abbondanti prodotte e rilasciate da cellule tumorali, stromale e immunitarie 1,2. Prove crescenti dimostrano che la TME ha un ruolo fondamentale nella riprogrammazione della differenziazione tumorale, della crescita, dell'invasione, delle metastasi e della risposta alle terapie3.
Comprendere come i diversi tipi di cellule nella TME interagiscono e comunicano tra loro attraverso reti di segnalazione è essenziale per migliorare la diagnosi del cancro, ottimizzare l'immunoterapia e sviluppare nuovi trattamenti4. Le tecniche tradizionali di microscopia tissutale, tra cui l'immunoistochimica (IHC) e l'immunofluorescenza (IF), sono state utilizzate per decenni per studiare i tipi di cellule, l'abbondanza e le comunicazioni nei campioni tumorali. Sfortunatamente, queste tecniche in genere possono valutare solo uno o due marcatori proteici in una sezione tissutale e non possono rivelare le complesse relazioni spaziali e strutturali tra queste cellule 5,8,7.
Negli ultimi due decenni, sono state stabilite diverse tecnologie di imaging multiplexed8. Queste tecnologie forniscono una visione molto migliore della composizione, della funzione e della posizione delle cellule immunitarie all'interno della TME, portando a rapidi progressi nella capacità di identificare e profilare spazialmente TME complesse a livello di singola cellula 9,10. Le relazioni spaziali e strutturali di varie cellule tumorali e immunitarie nella TME sono ora all'avanguardia negli studi biologici e clinici che utilizzano queste tecnologie di imaging multiplexate11,12.
La tecnologia di imaging multiplexata recentemente sviluppata che utilizza codici a barre di anticorpi coniugati con oligonucleotidi è un'influente piattaforma di ricerca biologica a singola cellula basata sulla rilevazione di anticorpi coniugati con oligonucleotidi in campioni fissati in formalina e incorporati in paraffina (FFPE)13,14. Attualmente, questa tecnologia di imaging multiplex consente l'imaging simultaneo di oltre 100 marcatori in una singola sezione tissutale15, che ha aumentato il numero di tipi di cellule distinguibili in situ. Ciò consente un livello di analisi spaziale delle cellule tumorali e immunitarie che non è possibile utilizzando approcci di immunofenotipizzazione tradizionali16.
Qui, descriviamo un protocollo ottimizzato per coniugare anticorpi purificati agli oligonucleotidi e convalidare questa coniugazione utilizzando la piattaforma di imaging multiplexata e una procedura di imaging multiciclo con tessuto FFPE. Inoltre, descriviamo le procedure di base di elaborazione delle immagini e analisi dei dati utilizzate con questa tecnologia.
Protocollo
Questo studio retrospettivo è stato approvato dall'Institutional Review Board dell'MD Anderson Cancer Center dell'Università del Texas. I campioni di tessuto FFPE sono stati raccolti dai pazienti di MD Anderson come parte delle cure standard di routine. Non sono stati eseguiti interventi diagnostici o terapeutici. Il consenso informato è stato ottenuto dai pazienti per l'utilizzo dei campioni raccolti per la ricerca e la pubblicazione.
1. Fonti di anticorpi utilizzate per la progettazione del pannello anticorpale
- Creare un pannello anticorpale per l'imaging multiplex dopo aver considerato attentamente la qualità dei tessuti e delle proteine di interesse. Tre fonti di anticorpi sono considerate per la progettazione del pannello anticorpale: 1) anticorpi completamente convalidati commercialmente, 2) anticorpi sottoposti a screening con tecnologia di imaging multiplex e 3) anticorpi condivisi dall'utente finale.
NOTA: gli anticorpi sottoposti a screening con tecnologia di imaging multiplexed hanno dimostrato di funzionare e sono disponibili presso i fornitori. Questi anticorpi schermati possono essere applicati alla colorazione multiplex dell'immagine dopo la coniugazione oligonucleotidica da parte dell'utente. - Se una proteina di interesse non può essere trovata nelle fonti sopra menzionate, impiegare i cloni di anticorpi che sono noti per funzionare con IHC. Inoltre, gli isotipi IgG piuttosto che i cloni IgM sono raccomandati per questa tecnologia di imaging multiplexata a causa del più alto tasso di fallimento con IgM rispetto ai cloni IgG.
2. Prima della coniugazione anticorpale
- Quando si identificano cloni di anticorpi per la coniugazione utilizzando codici a barre di imaging multiplexati, prendere in considerazione l'acquisto di anticorpi senza vettore in soluzione salina tamponata con fosfato (PBS) o un tampone simile. Le proteine di trasporto, tra cui BSA, glutine, glicerolo e altri additivi proteici, sono noti per ridurre la capacità di coniugazione.
- Selezionare il clone anticorpale più adatto e ottimizzare sempre le condizioni di colorazione (cioè il recupero e la titolazione dell'antigene) prima della coniugazione. Fate questo applicando un clone anticorpale non coniugato al tessuto positivo e negativo per questo anticorpo usando IF o IHC standard.
NOTA: Se un anticorpo purificato non è disponibile in commercio, è necessario eseguire un processo di purificazione degli anticorpi prima della coniugazione. La procedura di purificazione utilizzata con i kit di purificazione degli anticorpi non è discussa nel presente documento.
3. Coniugazione anticorpale
- Ottenere i reagenti di coniugazione anticorpale. I kit di coniugazione disponibili in commercio contengono una soluzione che blocca il filtro, una soluzione di riduzione 2, una soluzione di coniugazione, una soluzione di purificazione, una soluzione di stoccaggio di anticorpi (tutti conservati a 4 °C) e una soluzione di riduzione 1 (conservata a -20 °C).
- Coniugazione
NOTA: Un anticorpo purificato viene trattato con un agente riducente, consentendo alle frazioni ridotte dell'anticorpo di reagire con il codice a barre di imaging multiplex utilizzato con questa tecnologia e, quindi, formare un legame covalente. Questo processo richiede circa 4,5 ore e si traduce in circa 120 μL di anticorpo coniugato, che è vitale per 1 anno. Tutta la rotazione viene condotta a temperatura ambiente (RT) e il flusso continuo viene scartato tranne nell'ultima fase (3.2.9), in cui una provetta di raccolta contiene l'anticorpo coniugato.- Aspirare e applicare 500 μL della soluzione che blocca il filtro utilizzando una pipetta sulle colonne del filtro di taglio del peso molecolare da 50 kDa e ruotare verso il basso a 12.000 x g per 2 minuti per bloccare il legame anticorpale non specifico.
NOTA: Se la soluzione residua nella parte superiore della colonna è evidente, capovolgere il filtro nel tubo di raccolta e ruotare verso il basso a 3.000 x g per 2 minuti. - Pipettare 50 μg di anticorpo in un volume di 100 μL di soluzione alla colonna del filtro e centrifugare a 12.000 x g per 8 minuti.
NOTA: Utilizzare uno spettrofotometro per misurare la concentrazione dell'anticorpo purificato e calcolare il volume di soluzione corrispondente a 50 μg dell'anticorpo. Se il volume della soluzione anticorpale è inferiore a 100 μL, regolare il volume a 100 μL aggiungendo 1x PBS. Conservare 1 μg di anticorpo non coniugato per la conferma della coniugazione (fase 4.4). - Pipettare 260 μL di miscela master di riduzione (20 μL di soluzione di riduzione 1 miscelata con 825 μL di soluzione di riduzione 2, sufficiente per tre reazioni di coniugazione anticorpale) su ciascuna colonna filtrante. Vortice delicatamente la miscela principale per 2-3 s, o pipettare su e giù per mescolare la soluzione con l'anticorpo. Incubare a RT per 30 min.
- Dopo l'incubazione, ruotare le colonne del filtro a 12.000 x g per 8 minuti. Aggiungere 450 μL di soluzione di coniugazione alle colonne del filtro e centrifugare a 12.000 x g per 8 minuti.
- Risospendere il codice a barre desiderato in 10 μL di acqua priva di nucleasi e 210 μL di soluzione di coniugazione.
NOTA: Preparare un tag anticorpale immediatamente prima dell'uso per la colorazione. Non riutilizzare le aliquote dei codici a barre anticorpali. - Dopo aver completato lo spin-down al punto 3.2.4, aggiungere il tag anticorpale risospeso (circa 220 μL) in ciascuna colonna del filtro corrispondente. Pipettare delicatamente la miscela su e giù per miscelare i reagenti. Chiudere i coperchi delle colonne filtranti e incubare la reazione di coniugazione a RT per 2 ore. Dopo 2 ore, ruotare le colonne del filtro a 12.000 x g per 8 minuti.
NOTA: Per il protocollo di conferma si raccomanda di mettere da parte 5 μL della soluzione coniugata in un tubo di reazione a catena della polimerasi e conservarla a 4 °C (vedere sotto). - Pipettare 450 μL della soluzione di purificazione in ciascuna colonna filtrante e centrifugare a 12.000 x g per 8 minuti. Ripeti tre volte.
- Pipettare 100 μL della soluzione di stoccaggio nelle colonne del filtro. Pipettare delicatamente la miscela su e giù più di 10 volte e lavare accuratamente i lati dei filtri nella colonna.
NOTA: Sciogliere 50 μg di anticorpi in 100 μL della soluzione di conservazione. Se la reazione di coniugazione viene avviata con più di 50 μg di anticorpo, aggiungere altra soluzione di conservazione a questo rapporto. - Capovolgere le colonne del filtro in un tubo di raccolta fresco. Girare a 3.000 x g per 2 minuti a RT. Conservare la soluzione raccolta. Pipettare la soluzione anticorpale coniugata in tubi sterili a vite e conservare fino a 1 anno a 4 °C.
NOTA: Un anticorpo coniugato deve essere testato con la tecnologia di imaging multiplex dopo 2 giorni. I test precedenti a questo possono causare un'elevata colorazione nucleare di fondo.
- Aspirare e applicare 500 μL della soluzione che blocca il filtro utilizzando una pipetta sulle colonne del filtro di taglio del peso molecolare da 50 kDa e ruotare verso il basso a 12.000 x g per 2 minuti per bloccare il legame anticorpale non specifico.
4. Conferma della coniugazione
NOTA: Prima di eseguire esperimenti di colorazione con un anticorpo coniugato dall'utente utilizzando la tecnologia di imaging multiplexato, la coniugazione deve essere confermata utilizzando l'elettroforesi su gel con 5 μL di anticorpo coniugato (vedere punto 3.2.6) insieme a 1 μg di un anticorpo non coniugato (di solito in 2 μL della miscela) come controllo. Una coniugazione anticorpale di successo sarà dimostrata da un aumento del peso molecolare dell'anticorpo. Tuttavia, questo protocollo di conferma valuta solo il successo della reazione chimica per la coniugazione e non affronta la convalida degli anticorpi utilizzata per l'imaging multiplexato.
- Pipettare 8 μL e 11 μL di acqua priva di nucleasi rispettivamente nell'anticorpo coniugato riservato e nell'anticorpo non coniugato di controllo per ottenere un volume finale di 13 μL.
- Pipettare 5 μL di tampone per campioni LDS (o altro sistema di elettroforesi su gel di sodio dodecilsolfato-poliacrilammide) e 2 μL di agente riducente del campione in ciascun campione dell'anticorpo coniugato riservato e denaturare i campioni in un bagno asciutto a 95 °C per 10 minuti.
- Mentre i campioni sono denaturati, diluire 40 ml di tampone MOPS SDS in 760 ml di acqua ultrapura, posizionare un gel nel serbatoio di un sistema di elettroforesi e versare il tampone diluito sul gel.
- Una volta completato il periodo di denaturazione di 10 minuti, caricare un pozzetto del gel con uno standard proteico pre-colorato, uno con l'anticorpo non coniugato (dal punto 3.2.2) e i pozzetti rimanenti con i campioni di anticorpi coniugati. Quindi, eseguire il gel a 150 V fino a quando lo standard proteico appare alla fine del gel.
NOTA: Il gel aderisce facilmente ai contenitori sicuri per microonde e, quindi, può strapparsi, quindi il gel deve essere maneggiato con cautela nei seguenti passaggi. - Al termine della corsa, trasferire il gel in un contenitore sicuro per microonde preriempito con acqua ultrapura e riscaldarlo in un forno a microonde fino a visualizzare la prima bolla nell'acqua.
NOTA: Il tempo di formazione delle bolle varia notevolmente a seconda del microonde utilizzato. - Scolare l'acqua dal contenitore, versare circa 250 ml di colorazione Coomassie G-250 sul gel e riscaldare il gel in un forno a microonde fino a visualizzare la prima bolla. Successivamente, rimuovere il contenitore con il gel e la macchia Coomassie G-250 dal microonde e posizionarlo su uno shaker per 10 minuti.
- Dopo aver agitato, drenare accuratamente la macchia, sostituirla con circa 200 ml di acqua ultrapura, quindi posizionare il contenitore sullo shaker per lavare il gel.
- Drenare l'acqua ultrapura e sostituirla con nuova acqua ultrapura cinque volte o fino a quando i resti della macchia non sono evidenti nel bagnomaria. Lasciare lavare il gel per una notte sullo shaker fino a quando le bande sono visibili, se necessario, prima di fotografare il gel (Figura 1).
NOTA: Gli anticorpi utilizzati nell'imaging multiplex devono avere modelli di colorazione paragonabili a quelli degli anticorpi coniugati con colorante. Le sezioni di tessuto con antigeni noti positivi per anticorpi coniugati possono essere colorate con anticorpi coniugati con oligonucleotidi e coniugati con coloranti. In questo lavoro, in ciascun caso, le morfologie tissutali per entrambi i tipi di anticorpi erano equivalenti e armonizzate tra loro, così come la distribuzione cellulare prevista, basata sulla biologia delle proteine di interesse e sui campioni di tessuto di prova. Questo risultato dimostra l'efficacia dell'utilizzo di frazioni anticorpali coniugate con oligonucleotidi per approcci basati sulla colorazione dei tessuti utilizzando il tessuto FFPE.
5. Colorazione anticorpale coniugata con oligonucleotidi
- Preparare i vetrini di copertura per il posizionamento dei tessuti.
- Immergere i vetrini in una soluzione di poli-L-lisina allo 0,1% per 24 ore a RT per migliorare l'aderenza del tessuto.
- Dopo l'immersione, scolare la soluzione di poli-L-lisina e lavare i vetrini con acqua ultrapura per 30 s. Ripetere il lavaggio da quattro a sei volte. Rimuovere i coprislip dall'acqua ultrapura utilizzata per il lavaggio e metterli su un asciugamano privo di lanugine per asciugarli durante la notte.
NOTA: Un istologo deve tagliare il tessuto selezionato in sezioni spesse 5 μm, posizionarle al centro di un coprislip caricato con poli-L-lisina e lasciarle asciugare durante la notte. Dopo l'asciugatura, i vetrini con le sezioni di tessuto devono essere conservati a 4 °C per non più di 6 mesi. Il pannello di colorazione in questo protocollo conteneva 26 marcatori. Il tessuto utilizzato in questo protocollo era normale tessuto tonsilla umano. Il tempo di ammollo della copertina non deve superare 1 settimana. I coprivetrini rivestiti di poli-L-lisina possono essere conservati presso RT e devono essere utilizzati entro 2 mesi dalla preparazione.
- Il giorno prima della colorazione, posizionare il portaslip in un forno a 60 °C per tutta la notte.
- Il giorno seguente, collocare il campione di coprislip in un portavetrino preriscaldato in un forno a 60 °C. Dopo aver cotto per 30 minuti, controllare che la paraffina si sia sciolta dal tessuto.
- Posizionare rapidamente il portafoglio/campione nella seguente serie di soluzioni: due cicli di agente decerante per 6 minuti ciascuno; due cicli di etanolo al 100% per 5 minuti ciascuno; un giro di etanolo al 90% per 5 min; un giro di etanolo al 70% per 5 min; un giro di etanolo al 50% per 5 min; un giro di etanolo al 30% per 5 minuti; e due cicli di acqua ultrapura trattata con dietil pirocarbonato (DEPC) per 5 minuti ciascuno.
NOTA: Tutte le diluizioni di etanolo sono preparate con acqua ultrapura trattata con DEPC. Se lo xilene viene utilizzato per la deceratura, dovrebbe essere usato in un cappuccio. - Mentre sottoponete il portafoglio/campione alla serie di soluzioni, effettuate le seguenti operazioni:
- Preparare una camera di umidità posizionando una scatola di punta per pipette vuota con un tovagliolo di carta imbevuto d'acqua sul fondo.
- Riempire una pentola a pressione con acqua sufficiente a coprire a metà un becher da 50 ml.
- Introdurre 5 mL di metanolo per campione nel becher a 4 °C.
- Diluire il tampone AR9 con acqua ultrapura trattata con DEPC a 1x; sono necessari circa 50 ml del tampone diluito per portafoglio.
- Al termine della serie di soluzioni, riempire un becher di vetro da 50 mL con circa 40 mL di 1x tampone AR9, immergere il portafoglio/campione nel becher e coprire completamente il portavetrina/coprivetrino con un foglio di alluminio.
- Posizionare il becher rivestito di alluminio nella pentola a pressione riempita d'acqua e cuocere ad alta pressione (circa 15 psi) per 20 minuti.
- Dopo la cottura, rimuovere il portabicchiere/coprifoglio, scartare con cura il foglio di alluminio e lasciare raffreddare il porta-becher/coprislip a RT per circa 10 minuti.
- Rimuovere il portafoglio/campione dal tampone 1x AR9 e immergerlo in due cicli di acqua ultrapura trattata con DEPC, eseguendo un'incubazione dei campioni in entrambi i cicli per 2 minuti ciascuno.
- Durante l'incubazione, recuperare il tampone di idratazione, le soluzioni di blocco N, G, J bloccante e S bloccante e il diluente/blocco anticorpale dal kit di colorazione per imaging multiplexato. Per due campioni di vetrino, etichettare le piastre a 6 pozzetti per le soluzioni utilizzando le configurazioni mostrate nella Figura 2.
- Introdurre il campione di coprifoglietto in due cicli da 5 ml di tampone di idratazione per 2 minuti ciascuno.
- Dopo entrambi i cicli di posizionamento nel tampone di idratazione, posizionare il campione di vetrino in 5 ml del diluente/blocco dell'anticorpo multiplex e incubare per 20-30 minuti a RT (non superare i 30 minuti).
- Durante l'incubazione, preparare un cocktail di anticorpi realizzando una miscela master da 200 μL di diluente/blocco di anticorpi multiplexati di imaging, bloccanti N, bloccanti G, bloccanti J e soluzioni bloccanti S.
NOTA: Calcolare la quantità totale di anticorpi in base al numero di marcatori e alla titolazione convalidata di ciascun marcatore e sottrarre la quantità totale di anticorpi dalla miscela principale. Ad esempio, per sei marcatori, ciascuno con una titolazione 1:200, l'equazione sarebbe 200 μL di master mix − 6 μL di cocktail anticorpale = 194 μL di master mix.
- Dopo l'incubazione nel diluente/blocco dell'anticorpo multiplexato, introdurre il campione di vetrino nella camera di umidità preparata al punto 5.5.1, pipettare 190 μL del cocktail di anticorpi sul campione di vetrino e incubare il campione di vetrino a RT per 3 ore.
- Dopo l'incubazione, lavare il campione di vetrino in due cicli con 5 ml del diluente/blocco dell'anticorpo di imaging multiplex per 2 minuti ciascuno.
- Per fissare gli anticorpi legati al tessuto sul vetrino, eseguire i passaggi 5.7.3-5.7.5 nella camera di umidità.
- Incubare i coprivetrini per 10 minuti con formaldeide al 16% diluita all'1,6% con soluzione di conservazione, quindi lavare i coprivetrini tre volte con 1x PBS.
- Incubare i coprivetrini per 5 minuti con metanolo a 4 °C, quindi lavare i coprivetrini tre volte con 1x PBS.
- Incubare i coprivetrini per 20 minuti con 5 ml del reagente fissativo diluito con 1x PBS, quindi lavare i vetrini tre volte con 1x PBS.
- Conservare i campioni di copertina macchiati nel tampone di conservazione a 4 °C per un massimo di 2 settimane
6. Piastra reporter di imaging multiplexed
NOTA: Una piastra da 96 pozzetti, denominata targa reporter, contenente fluorofori con codice a barre in singoli pozzetti viene preparata in base a esperimenti di imaging multiplexati progettati su misura e correlata a ciascun campione di copertina colorato. I seguenti passaggi sono per la preparazione della targa del reporter.
- Preparare un master mix reporter combinando 4.880 μL di acqua priva di nucleasi, 600 μL di tampone di imaging multiplexato 10x, 500 μL di un reagente di analisi e 20 μL di soluzione di colorazione nucleare. Questo sarà sufficiente per 20 cicli di tutti i pozzi.
- In ogni ciclo dell'esperimento di imaging multiplexato progettato su misura, riempire un pozzetto con 245 μL di una soluzione contenente la miscela master reporter e i fluorofori specifici con codice a barre per quel ciclo.
NOTA: vedere la Figura 3 per la configurazione della piastra reporter utilizzata in questo protocollo per un pannello di carcinoma. - Per proteggere i fluorofori con codice a barre, far aderire un coperchio della piastra di alluminio sopra la piastra reporter e posizionare la piastra all'interno dello strumento di imaging multiplexato.
- Conservare la lastra reporter in una scatola scura a 4 °C per un massimo di 2 settimane.
7. Calibrazione e funzionamento della macchina di imaging multiplexata
NOTA: Il microscopio a fluorescenza ad alta risoluzione cattura quattro diversi canali di fluorescenza in ciascun ciclo di imaging multiplex a 20x, luce di eccitazione al 100% e con basso photobleaching.
- Calibrare la messa a fuoco dell'imaging utilizzando il canale DAPI posizionando un vetrino di copertura del campione sullo stadio del microscopio, pipettando manualmente 700 μL di una titolazione 1:1.500 della soluzione di colorazione nucleare sul tessuto.
NOTA: Il coprislip viene trattenuto sul palco del microscopio durante il lavaggio del campione e l'imaging. - Per preparare lo strumento di imaging multiplexato, diluire il tampone di imaging multiplexato 10x a 1x utilizzando acqua ultrapura trattata con DEPC e riempire i flaconi di reagenti con soluzioni/solventi appropriati, tra cui il tampone di imaging multiplexato 1x diluito, l'acqua ultrapura trattata con DEPC e il dimetilsolfossido (DMSO).
- Una volta riempiti i flaconi del reagente in modo appropriato, inserire il progetto sperimentale nel software di gestione degli strumenti di imaging multiplexato; designare il ciclo corretto, i numeri dei pozzi, i posti z-stack, il nome del marcatore, la classe e il tempo di esposizione per ciascun ciclo (Figura 4); impostare tutti i parametri del microscopio; e selezionare le regioni di interesse sul foglio di copertina del campione da fotografare.
- Fare clic sul pulsante Esperimento nel software di controllo (Figura 4A). Nella finestra Impostazione e gestione dell'esperimento , fare clic sul pulsante Nuovo modello (Figura 4B).
- Digitare il nome del progetto nello spazio accanto al pulsante Progetto (Figura 4C). Digitare o selezionare il numero totale di cicli (Figura 4D).
- Fare clic sul pulsante Assegnazione canale , digitare le informazioni per ciascun ciclo nelle colonne (Figura 4E) e fare clic sul pulsante Salva modello . Avvia l'esperimento facendo clic sul pulsante Avvia esperimento .
NOTA: Durante un esperimento di imaging multiplexato, lo strumento recupera i fluorofori con codice a barre da un pozzetto della piastra reporter (massimo quattro fluorofori per pozzetto, incluso DAPI), li eroga direttamente sul vetrino di copertura del campione e visualizza le regioni di interesse per ciascun canale di fluorescenza. Dopo tutte le immagini per quel ciclo, lo strumento lava via i fluorofori con codice a barre e dispensa il ciclo successivo di reporter (dal pozzetto successivo sulla piastra del reporter). L'imaging continua fino al completamento di tutti i cicli utilizzando 26 marcatori.
8. Raccolta di immagini
NOTA: Le immagini multiplexate possono essere raccolte utilizzando qualsiasi microscopio a fluorescenza invertita adattato configurato con quattro canali di fluorescenza (DAPI, Cy3, Cy5 e Cy7) e dotato di una lente Plan Fluor 20x. L'imaging e il lavaggio dei campioni di copertina vengono eseguiti automaticamente in modo iterativo utilizzando una configurazione fluidica appositamente sviluppata. Le immagini vengono acquisite utilizzando il software Processor (v1.8.0.7) in formato QPTIFF.
- Nella finestra del processore, fai clic sul pulsante Input e seleziona il nome dell'esperimento.
- Nella sezione Opzioni di elaborazione , selezionate e selezionate Sottrazione sfondo, Deconvoluzione, Profondità di campo estesa e Correzione ombreggiatura. Fare clic sul pulsante Start .
9. Analisi delle immagini
NOTA: Le immagini acquisite possono essere caricate su un software di analisi automatica delle immagini brevettato o su un programma software open source (Figura 5) per l'analisi a valle.
- Fare clic sull'icona QuPath sul computer e aprire il software. Trascinare il file QPTIFF nella finestra Visualizzatore .
- Fare clic sul pulsante Luminosità e contrasto, che aprirà la finestra Luminosità e contrasto. Selezionare o deselezionare l'indicatore nella colonna Selezionato per mostrare o chiudere il segnale del marcatore.
- Fare clic sul pulsante Zoom per adattare per ingrandire o ridurre l'area di interesse.
NOTA: la visualizzazione dei dati delle immagini multiplexed fornisce all'utente uno sguardo approfondito nel microambiente tissutale. I singoli marcatori nei campioni FFPE possono essere visualizzati in formato di fluorescenza (Figura 6 e Figura 7) o in vista patologica. Diverse piattaforme computazionali possono essere utilizzate per elaborare le immagini composite e analizzare i dati delle immagini tissutali multiplexate. Utilizzando questo software, la fenotipizzazione spaziale, così come la scoperta di cellule rare e il calcolo del vicinato cellulare, possono essere realizzati con immagini di interi vetrini di tessuto multiplexato ultra-alto generato tramite la tecnologia di imaging multiplexato. Vedere la Figura 6 per i 26 anticorpi nel pannello del carcinoma e nel campione di tonsille (Figura supplementare 1).
Risultati
Abbiamo utilizzato campioni di tonsille FFPE per sviluppare un pannello di immunooncologia a 26 marcatori per illustrare lo stato immunitario del tessuto FFPE utilizzando un sistema di analisi delle immagini con codice a barre. Complessivamente, 19 anticorpi sono attualmente utilizzati in altri studi di imaging multiplex nel nostro laboratorio. Tutti i marcatori sono stati testati utilizzando tessuto FFPE con IHC cromogenico. Tutti gli anticorpi sono stati coniugati a oligonucleotidi del DNA unici. Quando si impostano i coprivetrini utilizzando il gestore di strumenti basato sul web (Figura 4) per questa tecnologia di analisi delle immagini con codice a barre, va notato che il primo e l'ultimo ciclo sono sempre "vuoti" (Figura 2 e Figura 4), che fornisce i segnali di fluorescenza di fondo da sottrarre dai segnali specifici degli anticorpi. Dopo che le immagini in formato QPTIFF sono state raccolte con il microscopio a fluorescenza, possono essere visualizzate utilizzando diversi software di analisi delle immagini automatizzati brevettati o programmi software open source. Le immagini composite possono mostrare tutti i marcatori o i marcatori selezionati per una migliore visualizzazione dei segnali (Figura 5). Inoltre, ogni anticorpo può essere valutato visivamente per la localizzazione nucleare, citoplasmatica o membranosa. Le cellule immunitarie, tumorali e stromali possono essere facilmente identificate. Successivamente, l'analisi delle immagini può fornire informazioni sull'intensità del segnale, sulla gamma dinamica e sulla distribuzione spaziale di tutti i marcatori (Figura 6). Questa tecnica ci ha permesso di analizzare tutti i 26 marcatori a livello subcellulare in una singola sezione di tessuto (Figura 7). Analizzando la co-localizzazione dei marcatori, abbiamo potuto identificare i fenotipi cellulari, localizzare la posizione spaziale delle cellule, calcolare la distanza tra le cellule e trovare la distribuzione delle cellule. L'impatto cruciale di questa tecnologia è la presentazione di un robusto pannello di 26 marcatori focalizzato sullo stato immunitario del microambiente tissutale.
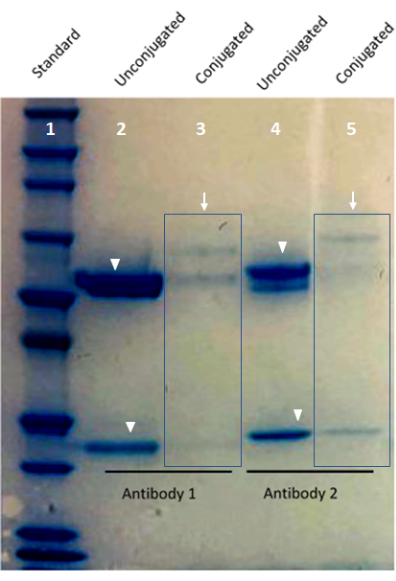
Figura 1: Immagine della convalida degli anticorpi coniugati su misura utilizzando un gel proteico Bis-Tris. La corsia 1 del gel mostra lo standard proteico. La corsia 2 e la corsia 4 mostrano anticorpi coniugati con codice a barre (frecce). La corsia 3 e la corsia 5 mostrano le bande di catena pesanti e leggere di un anticorpo non coniugato (punte di freccia). Fare clic qui per visualizzare una versione ingrandita di questa figura.
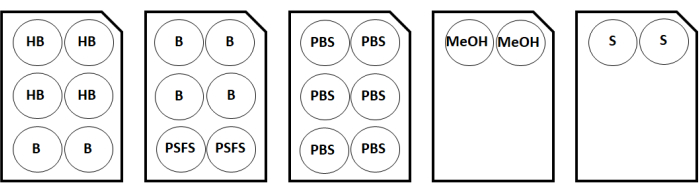
Figura 2: Mappa di configurazione della piastra di colorazione per due campioni di copertina. Abbreviazioni: HB = tampone di idratazione; B = diluente/blocco anticorpale; PSFS = soluzione fissativa post-colorazione; PBS = soluzione salina tamponata fosfato; MeOH = metanolo; S = soluzione di archiviazione. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 3: Configurazione della piastra Reporter. Ogni anticorpo coniugato ha un codice a barre che è complementare a un reporter specifico. Per impostare la targa del reporter, devono essere elencati tutti gli anticorpi coniugati e il relativo reporter. Successivamente, a ciascun anticorpo viene assegnato un numero di ciclo. L'esecuzione di due cicli vuoti (C1 e C18) viene utilizzata per valutare il livello di autofluorescenza nei tre canali di fluorescenza e per la sottrazione dello sfondo post-imaging utilizzando il software di controllo dell'acquisizione delle immagini. Una procedura guidata del software controllerà lo strumento in questa fase per assicurarsi che tutte le impostazioni siano corrette (Figura 4). Fare clic qui per visualizzare una versione ingrandita di questa figura.
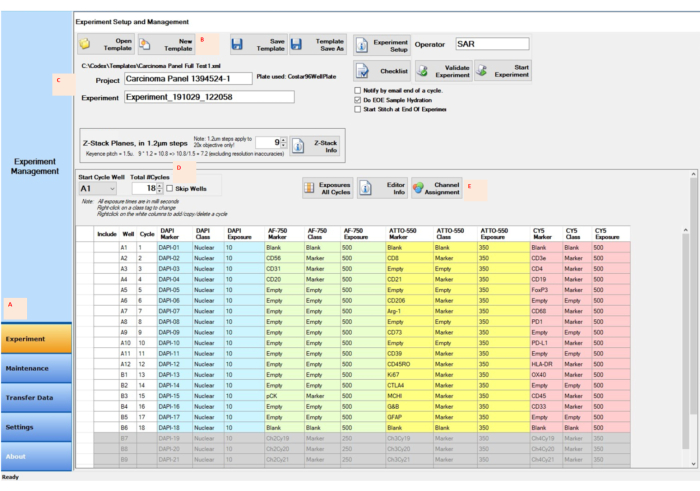
Figura 4: Acquisizione dell'immagine utilizzando il software di controllo per l'impostazione dell'esperimento. (A) Selezionare la scheda Esperimento nell'angolo in basso a sinistra del software di controllo per preparare e avviare l'installazione. (B,C) Selezionare Nuovo modello per inserire le impostazioni sperimentali con un nuovo nome di progetto e sperimentale. (D) Modificare il ciclo iniziale e il numero di cicli in modo che riflettano la posizione del reporter nella piastra del reporter a 96 pozzetti. (E) Assegnare i canali di fluorescenza appropriati ai quattro canali designati per l'esecuzione sperimentale. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 5: Visualizzazione delle immagini mediante software basato sul Web (QuPath). La finestra del visualizzatore mostra 26 marcatori nella sezione FFPE del tessuto tonsillare colorato. La finestra Luminosità e contrasto mostra i marcatori con i segni di spunta. Infine, la finestra del visualizzatore mostra l'esempio FFPE con i marcatori selezionati. Fare clic qui per visualizzare una versione ingrandita di questa figura.
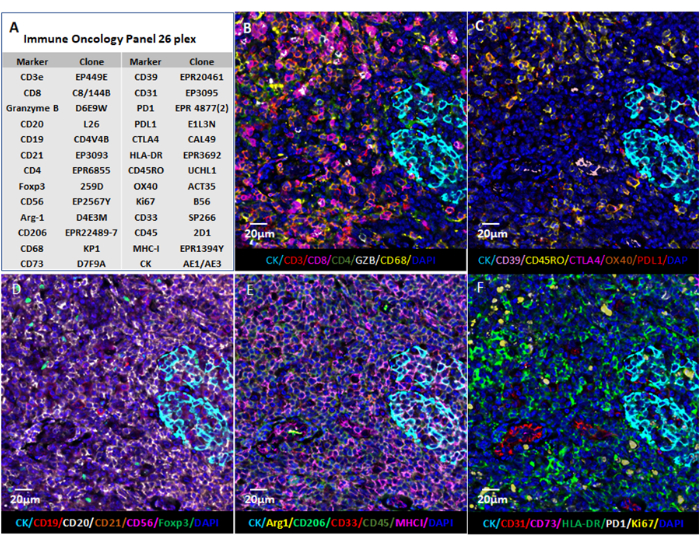
Figura 6: Visualizzazione delle immagini mediante software basato sul web. (A) Le sezioni di tessuto tonsillare sono state colorate per i 26 marcatori e le immagini delle sezioni in formato QPTIFF sono state visualizzate utilizzando un software di visualizzazione di diapositive digitali commerciali o software open source (QuPath) per l'annotazione e la revisione. (B-F) Sei marcatori sono stati visualizzati nella stessa annotazione per una migliore visione dei segnali. Fare clic qui per visualizzare una versione ingrandita di questa figura.
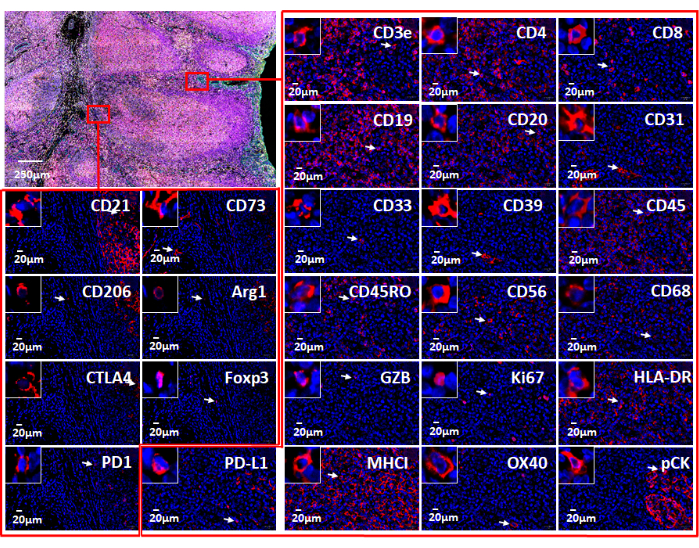
Figura 7: Viste dei 26 singoli marcatori utilizzati con il software basato sul Web. L'espressione del marcatore nel tessuto tonsillare è mostrata tramite colorazione a immunofluorescenza con un pannello di immunooncologia (in alto a sinistra). Vengono mostrati i singoli marcatori in due piccole aree (rettangoli rossi). L'inserto ingrandito mostra le celle positive per questi marcatori (frecce bianche). Fare clic qui per visualizzare una versione ingrandita di questa figura.
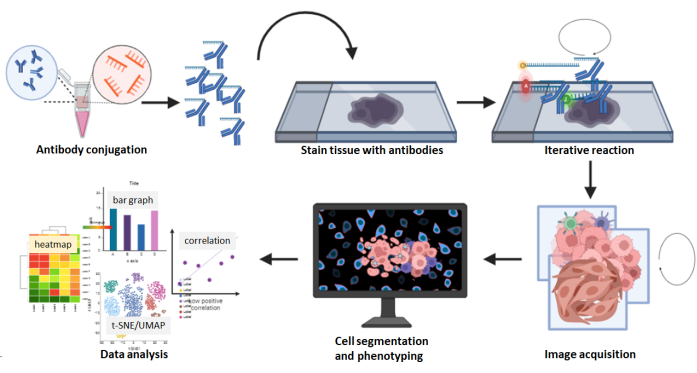
Figura 8: Riepilogo del flusso di lavoro di acquisizione di immagini multiplexate. Le sezioni di tessuto FFPE sono state colorate utilizzando il pannello anticorpale 26 seguito da una reazione multiciclo. Le immagini grezze delle sezioni colorate sono state elaborate computazionalmente e una densità cellulare e un'analisi spaziale sono state eseguite utilizzando le immagini composite. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Figura supplementare 1: Convalida IHC nel tessuto tonsillare. Le sezioni di tessuto FFPE sono state colorate utilizzando un singolo anticorpo. L'espressione del marcatore nel tessuto tonsillare viene mostrata a basso ingrandimento e l'inserto ingrandito mostra le cellule positive per il marcatore (rettangoli rossi). Clicca qui per scaricare questo file.
Discussione
La TME svolge un ruolo essenziale nello sviluppo, nella progressione e nelle risposte terapeutiche del cancro. Inoltre, la densità di specifici sottogruppi di linfociti infiltranti il tumore nella TME può servire come biomarcatore prognostico per alcuni tipi di cancro. Sorprendentemente, oltre alla composizione cellulare della TME, le caratteristiche spaziali di un tumore possono fornire un quadro per comprendere la biologia del tumore e identificare potenziali biomarcatori prognostici12,17.
Poiché numerose popolazioni di cellule immunitarie sono coinvolte nelle risposte protumorali o antitumorali, una migliore comprensione di queste cellule e delle loro relazioni spaziali tra loro e con le cellule tumorali aiuterà a guidare l'identificazione di nuove strategie immunoterapeutiche. Studi precedenti hanno stratificato la posizione e la distribuzione spaziale delle cellule TME in base alla struttura tissutale nelle aree intratumorali e peritumorali e ai margini invasivi delle cellule tumorali18,19. Negli ultimi 15 anni, i progressi tecnologici hanno reso l'analisi fenotipica delle singole cellule basata sulla loro dispersione spaziale uno strumento nuovo e influente per studiare la TME e classificare potenziali biomarcatori per l'immunoterapia tumorale. L'istochimica IF multiplex può stimare contemporaneamente più marcatori biologici20.
Analogamente alla strategia anticorpale coniugata con oligonucleotidi, per studiare la TME vengono utilizzati quattro tipi di piattaforme multiplex basate su proteine: sistemi di rilevamento di anticorpi marcati con cromogeno, fluorescenza, codice a barre del DNA e isotopi metallici. Le convenienti piattaforme cromogeniche IHC consentono la visualizzazione dell'intero vetrino e la valutazione patologica utilizzando la microscopia convenzionale a campo chiaro. Nell'IF multiplexed e nell'IHC vengono utilizzati anticorpi coniugati con fluorofori. La piattaforma multiplex IF/IHC rileva anticorpi con elevata specificità e può quantificare anticorpi mirati anche a livello subcellulare 6,21. Inoltre, a causa della natura dei cromogeni e dei fluorofori, l'uso di un pannello anticorpale può catturare l'espressione di un massimo di 10 biomarcatori su un singolo vetrino. Su piattaforme basate su isotopi metallici, gli anticorpi marcati con metallo vengono utilizzati per eseguire imaging multiplex con risoluzione spaziale e a singola cellula e alta sensibilità per singole sezioni di tessuto22. Teoricamente, questi approcci anticorpali coniugati con metalli consentono il rilevamento simultaneo di oltre 100 biomarcatori su una singola sezione di tessuto. Una sfida della tecnica di etichettatura degli isotopi è l'interferenza isobarica, che impedisce di raggiungere il 100% di purezza dell'arricchimento23. Inoltre, l'interferenza aumenta all'aumentare del numero di marcatori. Le piattaforme di rilevamento degli anticorpi coniugati con DNA riconoscono gli anticorpi marcati con codici a barre DNA univoci. Più di 40 biomarcatori possono essere catturati simultaneamente con elevata specificità su queste piattaforme6.
L'imaging multiplexed è una piattaforma di rilevamento di anticorpi marcata con codice a barre DNA disponibile in commercio per l'applicazione di anticorpi coniugati con DNA a un singolo vetrino tissutale in un unico passaggio (Figura 8). Per la fase di preparazione del tessuto, a differenza della piattaforma di imaging a fascio ionico multiplexato, che richiede l'uso di vetrini rivestiti in oro ottenuti dai produttori, la piattaforma di imaging multiplexata richiede solo vetrini o vetrini regolari rivestiti con poli-L-lisina allo 0,1% per aiutare il tessuto ad aderire ad esso e mantenere intatto il tessuto durante il processo di colorazione e imaging. Si raccomanda l'uso di sezioni di tessuto sui vetrini entro 4 settimane dal sezionamento, poiché la conservazione prolungata di vetrini non colorati comporta una riduzione dell'antigenicità. Un campione di coprivetrino colorato può essere conservato in un tampone di stoccaggio a 4 °C per un massimo di 2 settimane senza perdere il segnale di colorazione. Non sono necessarie attrezzature speciali per la conservazione dei campioni di copertina. Il sistema di imaging multiplexato è stato aggiornato per utilizzare vetrini regolari anziché copertine, che consentono la colorazione di tessuti più grandi e una facile manipolazione. Quando si utilizza una soluzione di riduzione per la coniugazione degli anticorpi (fase 3.2.3), la reazione deve essere limitata a non più di 30 minuti per evitare danni agli anticorpi. I buffer di blocco di cui al punto 5.6.6 devono essere preparati al momento e i buffer di blocco non devono essere riutilizzati.
Rispetto alle piattaforme di rilevamento di anticorpi multiplex marcati con cromogeno, fluorescenza e isotopi metallici, la tecnologia di imaging multiplex presenta alcuni vantaggi. Ad esempio, sono disponibili in commercio più di 60 pannelli anticorpali predefiniti per l'imaging multiplexato, il che aiuta a risparmiare tempo e costi nella coniugazione e nella convalida degli anticorpi e il numero di pannelli anticorpali predefiniti è in crescita. Questi anticorpi, che includono il marcatore di carcinoma pan-citocheratina, il marcatore di melanoma SOX10, il marcatore vascolare CD31, il marcatore stromale SMA e numerosi marcatori di cellule immunitarie, sono validati e pronti per la sperimentazione. Per gli anticorpi che non sono preprogettati, il kit di coniugazione disponibile in commercio progettato per l'uso con imaging multiplex è semplice e facile da usare. Gli anticorpi coniugati con il cliente sono validi per 1 anno se conservati a 4 °C. Inoltre, il riscaldamento della macchina non è necessario per acquisire le immagini. In questa tecnologia di imaging multiplexato, le fasi iterative di lavaggio, ibridazione e stripping nell'acquisizione dell'immagine raramente comportano una diminuzione dell'intensità del marcatore o una degradazione della morfologia del tessuto 5,24,25. Inoltre, le immagini composite vengono acquisite in formato QPTIFF con un semplice microscopio a fluorescenza a tre colori e possono essere caricate e analizzate utilizzando software di analisi digitale di terze parti. I marcatori di colorazione possono essere visualizzati alla risoluzione di una singola cellula e i fenotipi cellulari possono essere caratterizzati attraverso la co-localizzazione dei marcatori (Figura 6 e Figura 7). L'analisi completa di un'immagine multiplex rivela ulteriormente i compartimenti tissutali, la quantificazione dei marcatori a singola cellula e i dati sul vicino più vicino e sulla prossimità (Figura 8).
Una sfida nell'analisi delle immagini multiplex è l'identificazione del tipo di cella. Di solito, quando più classificatori di oggetti singoli vengono applicati a un'immagine, verranno annotati fenotipi più non comuni. Pertanto, si raccomanda di utilizzare marcatori noti che non sono co-espressi nello stesso classificatore e applicare solo il classificatore correlato al fenotipo all'annotazione di singole cellule. Le variazioni nell'annotazione del tipo di cellula daranno luogo a risultati spaziali sostanzialmente diversi, come le differenze nella distribuzione spaziale delle cellule e l'analisi del vicinato cellulare26,27.
L'analisi delle immagini multiplexed ha dimostrato di avere successo nella colorazione e nell'imaging di molti tipi di campioni, tra cui tessuto FFPE, tessuto fresco congelato, vetrini interi archiviati e microarray tissutali. Le immagini multiplexate di sezioni di seno, cervello, polmone, milza, rene, linfonodi e tessuto cutaneo possono essere acquisite con dati di fenotipizzazione spaziale profonda a singola cellula 5,16,25,28.
In futuro sono attesi altri anticorpi predefiniti per l'imaging multiplexato. Inoltre, è estremamente necessario sviluppare un software specifico per l'analisi delle immagini multiplexate. Attualmente, esistono molti programmi software disponibili in commercio e open source per l'analisi delle immagini Hi-Plex29, ma gli scienziati hanno ancora bisogno di aiuto per creare un flusso di lavoro standard per queste analisi30,31. Sebbene le immagini composite acquisite utilizzando questo protocollo siano compatibili con software di terze parti, ciò potrebbe comportare costi aggiuntivi per l'utente. Un altro svantaggio della tecnologia di imaging multiplexata è la riduzione del segnale nel rilevamento delle proteine nucleari dopo il lavaggio iterativo, l'ibridazione e lo stripping con grandi pannelli di anticorpi. Fortunatamente, questo può essere ridotto al minimo recuperando i fluorofori con codice a barre nei primi cicli durante la progettazione delle lastre reporter. Recentemente, questa piattaforma è stata aggiornata con un nuovo sistema di scansione ad alta velocità, che ha ridotto drasticamente il tempo per ottenere immagini composite32. Inoltre, è stata riportata una nuova strategia che utilizza codici a barre coniugati con tiramide per migliorare l'imaging basato su codici a barre anticorpali coniugati con oligonucleotidi. Questa tecnologia mira ad amplificare i segnali di colorazione per i quali è difficile ottenere anticorpi coniugati con codice a barre33.
Divulgazioni
Gli autori non hanno conflitti da rivelare.
Riconoscimenti
Gli autori ringraziano Donald R. Norwood di Editing Services, Research Medical Library di MD Anderson per aver curato questo articolo e il laboratorio di analisi multiplex IF e immagini del Dipartimento di Patologia Molecolare Traslazionale di MD Anderson. Questo progetto è stato sostenuto in parte dalla piattaforma Moonshots del laboratorio di patologia molecolare traslazionale-immunoprofilazione (TMP-IL) presso il Dipartimento di Patologia molecolare traslazionale, dall'MD Anderson Cancer Center dell'Università del Texas e dal NCI Cooperative Agreement U24CA224285 (al MD Anderson Cancer Center CIMAC).
Materiali
| Name | Company | Catalog Number | Comments |
| 10x AR9 Buffer | Akoya Biosciences | AR900250ML | |
| 10x Buffer | Akoya Biosciences | 7000001 | |
| 16% paraformaldehyde | Thermo Fisher Scientific | 28906 | |
| 1X Antibody Diluent/Block | Akoya Biosciences | ARD1001EA | |
| Antibody Conjugation Kit | Akoya Biosciences | 7000009 | Contains Filter Blocking Solution, Antibody Reduction Solution 1, Antibody Reduction Solution 2, Conjugation Solution, Purification Solution, Antibody Storage Solution |
| Assay Reagent | Akoya Biosciences | 7000002 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| Ethanol, 200 proof | |||
| G Blocker V2 | Akoya Biosciences | 240199 | |
| Histoclear | Thermo Fisher Scientific | 50-329-50 | |
| Methanol | Sigma-Aldrich | 34860-1L-R | |
| Milli-Q Integral 10 | Millipore | ZRXQ010WW | |
| Niknon Fluorescence microscope | Keyence Corp. of America | BZ-X810 | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| Nuclease-free water | Thermo Fisher Scientific | AM9938 | |
| NuPAGE | Thermo Fisher Scientific | NP0008 | |
| PBS | Thermo Fisher Scientific | 14190136 | |
| PhenoCycler Barcodes/Reporters Combination | Akoya Biosciences | 5450004 (BX025/RX025) | |
| 5450003 (BX022/RX022) | |||
| 5450023 (BX002/RX002) | |||
| 5250002 (BX020/RX020) | |||
| 2520003 (BX023/RX023) | |||
| 5250005 (BX029/RX029) | |||
| 5250007 (BX035/RX035) | |||
| 5250012 (BX052/RX052) | |||
| 5550012 (BX030/RX030) | |||
| 5550015 (BX042/RX042) | |||
| 5550014 (BX036/RX036) | |||
| QuPath | Open-Source | https://qupath.github.io/ | |
| SimplyBlue SafeStain | Thermo Fisher Scientific | LC6065 | |
| Staining Kit | (Akoya Biosciences | 7000008 | Contains Hydration Buffer, N Blocker, J Blocker, S Blocker, Fixative Reagent, Storage Buffer |
Riferimenti
- Hanahan, D., Coussens, L. M. Accessories to the crime: Functions of cells recruited to the tumor microenvironment. Cancer Cell. 21 (3), 309-322 (2012).
- Labani-Motlagh, A., Ashja-Mahdavi, M., Loskog, A. The tumor microenvironment: A milieu hindering and obstructing antitumor immune responses. Frontiers in Immunology. 11, 940 (2020).
- Jin, M. Z., Jin, W. L. The updated landscape of tumor microenvironment and drug repurposing. Signal Transduction and Targeted Therapy. 5 (1), 166 (2020).
- Waldman, A. D., Fritz, J. M., Lenardo, M. J. A guide to cancer immunotherapy: From T cell basic science to clinical practice. Nature Reviews Immunology. 20 (11), 651-668 (2020).
- Black, S., et al. CODEX multiplexed tissue imaging with DNA-conjugated antibodies. Nature Protocols. 16 (8), 3802-3835 (2021).
- Tan, W. C. C., et al. Overview of multiplex immunohistochemistry/immunofluorescence techniques in the era of cancer immunotherapy. Cancer Communications. 40 (4), 135-153 (2020).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 117 (52), 33455-33465 (2020).
- Allam, M., Cai, S., Coskun, A. F. Multiplex bioimaging of single-cell spatial profiles for precision cancer diagnostics and therapeutics. NPJ Precision Oncology. 4, 11 (2020).
- Eng, J., et al. A framework for multiplex imaging optimization and reproducible analysis. Communications Biology. 5 (1), 438 (2022).
- Taube, J. M., et al. Multi-institutional TSA-amplified multiplexed immunofluorescence reproducibility evaluation (MITRE) study. Journal for Immunotherapy of Cancer. 9 (7), e002197 (2021).
- Binnewies, M., et al. Understanding the tumor immune microenvironment (TIME) for effective therapy. Nature Medicine. 24 (5), 541-550 (2018).
- Heindl, A., Nawaz, S., Yuan, Y. Mapping spatial heterogeneity in the tumor microenvironment: A new era for digital pathology. Laboratory Investigation. 95 (4), 377-384 (2015).
- Kuswanto, W., Nolan, G., Lu, G. Highly multiplexed spatial profiling with CODEX: bioinformatic analysis and application in human disease. Seminars in Immunopathology. 45 (1), 145-157 (2022).
- Phillips, D., et al. Immune cell topography predicts response to PD-1 blockade in cutaneous T cell lymphoma. Nature Communications. 12 (1), 6726 (2021).
- Jhaveri, N., et al. Deep ultrahigh-plex spatial phenotyping of human cancer tissues. Cancer Research. 82, 3877 (2022).
- Goltsev, Y., et al. Deep profiling of mouse splenic architecture with CODEX multiplexed imaging. Cell. 174 (4), 968-981 (2018).
- Yuan, Y. Spatial heterogeneity in the tumor microenvironment. Cold Spring Harbor Perspectives in Medicine. 6 (8), a026583 (2016).
- Bruck, O., et al. Spatial immunoprofiling of the intratumoral and peritumoral tissue of renal cell carcinoma patients. Modern Pathology. 34 (12), 2229-2241 (2021).
- Tsujikawa, T., et al. Prognostic significance of spatial immune profiles in human solid cancers. Cancer Science. 111 (10), 3426-3434 (2020).
- Hoyt, C. C. Multiplex immunofluorescence and multispectral imaging: forming the basis of a clinical test platform for immuno-oncology. Frontiers in Molecular Biosciences. 8, 674747 (2021).
- Parra, E. R., et al. Identification of distinct immune landscapes using an automated nine-color multiplex immunofluorescence staining panel and image analysis in paraffin tumor tissues. Scientific Reports. 11 (1), 4530 (2021).
- Elaldi, R., et al. High dimensional imaging mass cytometry panel to visualize the tumor immune microenvironment contexture. Frontiers in Immunology. 12, 666233 (2021).
- Campos Clemente, L., Shi, O., Rojas, F., Parra, E. R. Expanding the comprehension of the tumor microenvironment using mass spectrometry imaging of formalin-fixed and paraffin-embedded tissue samples. Journal of Visualized Experiments. (184), e64015 (2022).
- Schurch, C. M., et al. Coordinated cellular neighborhoods orchestrate antitumoral immunity at the colorectal cancer invasive front. Cell. 182 (5), 1341-1359 (2020).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by CODEX tissue imaging. Frontiers in Immunology. 12, 687673 (2021).
- Hickey, J. W., Tan, Y., Nolan, G. P., Goltsev, Y. Strategies for accurate cell type identification in CODEX multiplexed imaging data. Frontiers in Immunology. 12, 727626 (2021).
- Parra, E. R. Methods to determine and analyze the cellular spatial distribution extracted from multiplex immunofluorescence data to understand the tumor microenvironment. Frontiers in Molecular Biosciences. 8, 668340 (2021).
- Tzoras, E., et al. Dissecting tumor-immune microenvironment in breast cancer at a spatial and multiplex resolution. Cancers. 14 (8), 1999 (2022).
- Francisco-Cruz, A., Parra, E. R., Tetzlaff, M. T. Wistuba, II. Multiplex immunofluorescence assays. Methods in Molecular Biology. 2055, 467-495 (2020).
- Shakya, R., Nguyen, T. H., Waterhouse, N., Khanna, R. Immune contexture analysis in immuno-oncology: applications and challenges of multiplex fluorescent immunohistochemistry. Clinical and Translational Immunology. 9 (10), e1183 (2020).
- Wilson, C. M., et al. Challenges and opportunities in the statistical analysis of multiplex immunofluorescence data. Cancers. 13 (12), 3031 (2021).
- DeRosa, J. Setting a new standard for spatial omics: An integrated multiomics approach: every single cell, two different analytes, one unbiased picture. Genetic Engineering & Biotechnology News. 42, 26-28 (2022).
- Simonson, P. D., Valencia, I., Patel, S. S. Tyramide-conjugated DNA barcodes enable signal amplification for multiparametric CODEX imaging. Communications Biology. 5 (1), 627 (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon