Method Article
Analyse d’images de codes à barres multiplexés pour l’immunoprofilage et la caractérisation de la cartographie spatiale dans l’analyse unicellulaire d’échantillons de tissus de paraffine
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
L’analyse d’images de codes à barres multiplexés a récemment amélioré la caractérisation du microenvironnement tumoral, permettant des études approfondies de la composition cellulaire, de l’état fonctionnel et des interactions cellule-cellule. Nous décrivons ici un protocole de coloration et d’imagerie utilisant le codage à barres d’anticorps conjugués oligonucléotidiques et l’imagerie cyclique, qui permet l’utilisation d’une technique d’analyse d’images de haute dimension.
Résumé
La technologie d’imagerie multiplexée utilisant le codage à barres d’anticorps avec des oligonucléotides, qui détecte séquentiellement plusieurs épitopes dans la même section de tissu, est une méthodologie efficace pour l’évaluation de la tumeur qui améliore la compréhension du microenvironnement tumoral. La visualisation de l’expression des protéines dans les tissus fixés au formol et incorporés dans la paraffine est réalisée lorsqu’un fluorophore spécifique est recuit à un code-barres lié à un anticorps via des oligonucléotides complémentaires, puis que l’imagerie de l’échantillon est effectuée; En effet, cette méthode permet l’utilisation de panels personnalisables de plus de 40 anticorps dans une seule réaction de coloration tissulaire. Cette méthode est compatible avec les tissus frais congelés, les tissus fixés au formol, incorporés dans la paraffine, les cellules en culture et les cellules mononucléaires du sang périphérique, ce qui signifie que les chercheurs peuvent utiliser cette technologie pour visualiser une variété de types d’échantillons à une résolution unicellulaire. Cette méthode commence par un protocole de coloration et de fixation manuelle, et tous les codes-barres d’anticorps sont appliqués à l’aide d’un cocktail d’anticorps. L’instrument de fluidique de coloration est entièrement automatisé et effectue des cycles itératifs de marquage, d’imagerie et d’élimination des fluorophores spectralement distincts jusqu’à ce que tous les biomarqueurs aient été imagés à l’aide d’un microscope à fluorescence standard. Les images sont ensuite collectées et compilées à travers tous les cycles d’imagerie pour obtenir une résolution unicellulaire pour tous les marqueurs. La coloration en une seule étape et l’élimination douce des fluorophores permettent non seulement une analyse hautement multiplexée des biomarqueurs, mais préservent également l’échantillon pour une analyse supplémentaire en aval si désiré (p. ex. coloration à l’hématoxyline et à l’éosine). En outre, le logiciel d’analyse d’images permet le traitement d’images - compensation de dérive, soustraction d’arrière-plan, segmentation cellulaire et regroupement - ainsi que la visualisation et l’analyse des images et des phénotypes cellulaires pour la génération de cartes de réseaux spatiaux. En résumé, cette technologie utilise un système microfluidique informatisé et un microscope à fluorescence pour hybrider, imager et dépouiller itérativement des sondes d’ADN marquées par fluorescence qui sont complémentaires aux anticorps conjugués oligonucléotidiques liés aux tissus.
Introduction
Le microenvironnement tumoral (TME) est extrêmement hétérogène, composé de cellules tumorales, de cellules stromales tumorales, de cellules immunitaires, de composants non cellulaires de la matrice extracellulaire et de nombreuses molécules abondantes produites et libérées par les cellules tumorales, stromales et immunitaires 1,2. L’accumulation de preuves démontre que le TME joue un rôle central dans la reprogrammation de la différenciation, de la croissance, de l’invasion, des métastases et de la réponse aux thérapies3.
Comprendre comment les différents types de cellules dans le TME interagissent et communiquent entre eux par le biais de réseaux de signalisation est essentiel pour améliorer le diagnostic du cancer, optimiser l’immunothérapie et développer de nouveaux traitements4. Les techniques traditionnelles de microscopie tissulaire, y compris l’immunohistochimie (IHC) et l’immunofluorescence (IF), sont utilisées depuis des décennies pour étudier les types cellulaires, l’abondance et les communications dans les échantillons tumoraux. Malheureusement, ces techniques ne peuvent généralement évaluer qu’un ou deux marqueurs protéiques dans une coupe de tissu et ne peuvent pas révéler les relations spatiales et structurelles complexes entre ces cellules 5,8,7.
Au cours des deux dernières décennies, plusieurs technologies d’imagerie multiplexées ont été mises en place8. Ces technologies fournissent des vues nettement améliorées de la composition, de la fonction et de l’emplacement des cellules immunitaires au sein du TME, ce qui conduit à des progrès rapides dans la capacité d’identifier et de profiler spatialement les TME complexes au niveau unicellulaire 9,10. Les relations spatiales et structurelles de diverses cellules tumorales et immunitaires dans le TME sont maintenant à l’avant-garde des études biologiques et cliniques utilisant ces technologies d’imagerie multiplexées11,12.
La technologie d’imagerie multiplexée récemment développée utilisant le codage à barres d’anticorps conjugués aux oligonucléotides est une plateforme de recherche biologique unicellulaire influente basée sur la détection d’anticorps conjugués à oligonucléotides dans des échantillons fixés au formol et incorporés à la paraffine (FFPE)13,14. Actuellement, cette technologie d’imagerie multiplexée permet l’imagerie simultanée de plus de 100 marqueurs dans une seule sectiontissulaire 15, ce qui a augmenté le nombre de types de cellules qui se distinguent in situ. Cela permet un niveau d’analyse spatiale des cellules tumorales et immunitaires qui n’est pas possible avec les approches traditionnelles d’immunophénotypage16.
Nous décrivons ici un protocole optimisé pour conjuguer des anticorps purifiés aux oligonucléotides et valider cette conjugaison à l’aide de la plateforme d’imagerie multiplexée et d’une procédure d’imagerie multicycle avec tissu FFPE. De plus, nous décrivons les procédures de base de traitement d’images et d’analyse de données utilisées avec cette technologie.
Protocole
Cette étude rétrospective a été approuvée par le comité d’examen institutionnel du MD Anderson Cancer Center de l’Université du Texas. Les échantillons de tissus FFPE ont été prélevés chez des patients de MD Anderson dans le cadre des soins standard de routine. Aucune intervention diagnostique ou thérapeutique n’a été effectuée. Le consentement éclairé des patients a été obtenu pour l’utilisation des échantillons prélevés à des fins de recherche et de publication.
1. Sources d’anticorps utilisées pour la conception du panel d’anticorps
- Créez un panel d’anticorps pour l’imagerie multiplexée après avoir soigneusement examiné la qualité des tissus et des protéines d’intérêt. Trois sources d’anticorps sont prises en compte pour la conception du panel d’anticorps : 1) les anticorps entièrement validés commercialement, 2) les anticorps multiplexés criblés par la technologie d’imagerie et 3) les anticorps partagés par l’utilisateur final.
REMARQUE : Les anticorps dépistés par la technologie d’imagerie multiplexée ont fait leurs preuves et sont disponibles auprès des fournisseurs. Ces anticorps criblés peuvent être appliqués à la coloration d’images multiplexées après conjugaison d’oligonucléotides par l’utilisateur. - Si une protéine d’intérêt ne peut pas être trouvée dans les sources mentionnées ci-dessus, utilisez les clones d’anticorps connus pour fonctionner avec IHC. De plus, les isotypes IgG plutôt que les clones IgM sont recommandés pour cette technologie d’imagerie multiplexée en raison du taux d’échec plus élevé avec les clones IgM qu’avec les clones IgG.
2. Avant la conjugaison des anticorps
- Lors de l’identification de clones d’anticorps pour la conjugaison à l’aide de codes-barres d’imagerie multiplexés, envisagez d’acheter des anticorps sans support dans une solution saline tamponnée au phosphate (PBS) ou un tampon similaire. Les protéines porteuses, y compris le BSA, le gluten, le glycérol et d’autres additifs protéiques, sont connues pour réduire la capacité de conjugaison.
- Sélectionnez le clone d’anticorps le plus approprié et optimisez toujours les conditions de coloration (c.-à-d. récupération et titrage de l’antigène) avant la conjugaison. Pour ce faire, appliquez un clone d’anticorps non conjugué sur les tissus positifs et négatifs pour cet anticorps en utilisant l’IF ou l’IHC standard.
REMARQUE: Si un anticorps purifié n’est pas disponible dans le commerce, un processus de purification des anticorps doit être effectué avant la conjugaison. La procédure de purification utilisée avec les kits de purification d’anticorps n’est pas abordée ici.
3. Conjugaison des anticorps
- Obtenir les réactifs de conjugaison des anticorps. Les trousses de conjugaison disponibles dans le commerce contiennent une solution de blocage du filtre, une solution de réduction 2, une solution de conjugaison, une solution de purification, une solution de stockage d’anticorps (toutes stockées à 4 °C) et une solution de réduction 1 (stockées à −20 °C).
- Conjugaison
REMARQUE: Un anticorps purifié est traité avec un agent réducteur, ce qui permet aux fractions réduites de l’anticorps de réagir avec le code-barres d’imagerie multiplexé utilisé avec cette technologie et, ainsi, de former une liaison covalente. Ce processus prend environ 4,5 heures et donne environ 120 μL d’anticorps conjugués, ce qui est viable pendant 1 an. Tout le spin-down est effectué à température ambiante (RT) et l’écoulement continu est éliminé, sauf à la toute dernière étape (3.2.9), dans laquelle un tube de collecte contient l’anticorps conjugué.- Aspirer et appliquer 500 μL de la solution de blocage du filtre à l’aide d’une pipette sur les colonnes filtrantes de coupure de poids moléculaire de 50 kDa, puis tourner vers le bas à 12 000 x g pendant 2 minutes pour bloquer la liaison d’anticorps non spécifiques.
REMARQUE: Si une solution résiduelle en haut de la colonne est visible, inverser le filtre dans le tube de collecte et tourner vers le bas à 3 000 x g pendant 2 min. - Introduire à la pipette 50 μg de l’anticorps dans un volume de solution de 100 μL sur la colonne filtrante et faire tourner vers le bas à 12 000 x g pendant 8 min.
NOTE: Utilisez un spectrophotomètre pour mesurer la concentration de l’anticorps purifié et pour calculer le volume de solution correspondant à 50 μg de l’anticorps. Si le volume de la solution d’anticorps est inférieur à 100 μL, ajuster le volume à 100 μL en ajoutant 1x PBS. Conserver 1 μg d’anticorps non conjugué pour la confirmation de la conjugaison (étape 4.4). - Pipeter 260 μL de mélange maître de réduction (20 μL de solution de réduction 1 mélangée à 825 μL de solution de réduction 2, ce qui est suffisant pour trois réactions de conjugaison d’anticorps) à chaque colonne filtrante. Tourbillonner doucement le mélange principal pendant 2-3 s, ou pipeter de haut en bas pour mélanger la solution avec l’anticorps. Incuber à TA pendant 30 min.
- Après l’incubation, faire tourner les colonnes filtrantes à 12 000 x g pendant 8 min. Ajouter 450 μL de solution de conjugaison aux colonnes filtrantes et faire tourner vers le bas à 12 000 x g pendant 8 min.
- Remettez en suspension le code-barres souhaité dans 10 μL d’eau sans nucléase et 210 μL de solution de conjugaison.
REMARQUE : Préparez une étiquette d’anticorps immédiatement avant l’utilisation pour la coloration. Ne réutilisez pas les aliquotes de code-barres d’anticorps. - Une fois l’essorage terminé à l’étape 3.2.4, ajouter l’étiquette d’anticorps remis en suspension (environ 220 μL) dans chaque colonne de filtre correspondante. Pipeter doucement le mélange de haut en bas pour mélanger les réactifs. Fermer les couvercles de la colonne filtrante et incuber la réaction de conjugaison à TA pendant 2 h. Après 2 h, faites tourner les colonnes filtrantes à 12 000 x g pendant 8 min.
NOTE: Il est recommandé de mettre de côté 5 μL de la solution conjuguée dans un tube de réaction en chaîne par polymérase et de la stocker à 4 °C pour le protocole de confirmation (voir ci-dessous). - Pipeter 450 μL de la solution de purification dans chaque colonne filtrante et faire tourner vers le bas à 12 000 x g pendant 8 min. Répétez trois fois.
- Pipeter 100 μL de la solution de stockage dans les colonnes filtrantes. Pipeter doucement le mélange de haut en bas plus de 10 fois et laver soigneusement les côtés des filtres dans la colonne.
REMARQUE : Dissoudre 50 μg d’anticorps dans 100 μL de la solution de stockage. Si la réaction de conjugaison est commencée avec plus de 50 μg d’anticorps, ajouter plus de solution de stockage à ce rapport. - Inversez les colonnes filtrantes dans un nouveau tube de collecte. Faire tourner vers le bas à 3 000 x g pendant 2 min à TA. Conserver la solution recueillie. Introduire à la pipette la solution d’anticorps conjugués dans des tubes à vis stériles et conserver jusqu’à 1 an à 4 °C.
REMARQUE: Un anticorps conjugué doit être testé avec la technologie d’imagerie multiplexée après 2 jours. Les essais préalables peuvent entraîner une coloration nucléaire de fond élevée.
- Aspirer et appliquer 500 μL de la solution de blocage du filtre à l’aide d’une pipette sur les colonnes filtrantes de coupure de poids moléculaire de 50 kDa, puis tourner vers le bas à 12 000 x g pendant 2 minutes pour bloquer la liaison d’anticorps non spécifiques.
4. Confirmation de la conjugaison
REMARQUE : Avant d’effectuer des expériences de coloration avec un anticorps conjugué par l’utilisateur à l’aide de la technologie d’imagerie multiplexée, la conjugaison doit être confirmée en utilisant l’électrophorèse sur gel avec 5 μL de l’anticorps conjugué (voir étape 3.2.6) avec 1 μg d’anticorps non conjugué (habituellement dans 2 μL du mélange) comme témoin. Une conjugaison réussie de l’anticorps sera démontrée par une augmentation du poids moléculaire de l’anticorps. Cependant, ce protocole de confirmation évalue uniquement le succès de la réaction chimique pour la conjugaison et ne traite pas de la validation des anticorps utilisée pour l’imagerie multiplexée.
- Pipeter 8 μL et 11 μL d’eau exempte de nucléase dans l’anticorps conjugué réservé et contrôler l’anticorps non conjugué, respectivement, pour obtenir un volume final de 13 μL.
- Introduire à la pipette 5 μL de LDS (ou un autre système d’électrophorèse sur gel dodécylsulfate-polyacrylamide de sodium) et 2 μL d’agent réducteur d’échantillon dans chaque échantillon de l’anticorps conjugué réservé, et dénaturer les échantillons dans un bain sec à 95 °C pendant 10 min.
- Pendant la dénaturation des échantillons, diluer 40 mL de tampon MOPS SDS dans 760 mL d’eau ultrapure, placer un gel dans le réservoir d’un système d’électrophorèse et verser le tampon courant dilué sur le gel.
- Une fois la période de dénaturation de 10 minutes terminée, charger un puits du gel avec un étalon protéique pré-coloré, un avec l’anticorps non conjugué (à partir de l’étape 3.2.2) et les puits restants avec les échantillons d’anticorps conjugués. Ensuite, exécutez le gel à 150 V jusqu’à ce que l’étalon protéique apparaisse à la fin du gel.
REMARQUE: Le gel adhère facilement aux récipients allant au micro-ondes et, par conséquent, peut se déchirer, de sorte que le gel doit être manipulé avec prudence dans les étapes suivantes. - Une fois l’exécution terminée, transférez le gel dans un récipient allant au micro-ondes prérempli d’eau ultrapure et chauffez-le au micro-ondes jusqu’à ce que la première bulle dans l’eau soit visualisée.
REMARQUE: Le temps de formation des bulles varie considérablement en fonction du micro-ondes utilisé. - Vidangez l’eau du contenant, versez environ 250 ml de colorant Coomassie G-250 sur le gel et chauffez le gel au micro-ondes jusqu’à ce que la première bulle soit visualisée. Ensuite, retirez le récipient avec le gel et la coloration Coomassie G-250 du micro-ondes et placez-le sur un shaker pendant 10 min.
- Après avoir agité, égoutter soigneusement la tache, la remplacer par environ 200 ml d’eau ultrapure, puis placer le récipient sur le shaker pour laver le gel.
- Vidangez l’eau ultrapure et remplacez-la par de l’eau ultrapure neuve cinq fois ou jusqu’à ce que les restes de la tache ne soient pas apparents dans le bain-marie. Laissez le gel laver toute la nuit sur le shaker jusqu’à ce que des bandes soient visibles, si nécessaire, avant de photographier le gel (Figure 1).
REMARQUE : Les anticorps utilisés en imagerie multiplexée doivent présenter des profils de coloration comparables à ceux des anticorps conjugués à un colorant. Les coupes de tissus avec des antigènes connus positifs pour les anticorps conjugués peuvent être colorées avec des anticorps conjugués à des oligonucléotides et des anticorps conjugués à un colorant. Dans ce travail, dans chaque cas, les morphologies tissulaires des deux types d’anticorps étaient équivalentes et harmonisées l’une avec l’autre, ainsi que la distribution cellulaire anticipée, basée sur la biologie des protéines d’intérêt et des échantillons de tissus testés. Ce résultat démontre l’efficacité de l’utilisation de groupements d’anticorps conjugués aux oligonucléotides pour les approches basées sur la coloration tissulaire utilisant du tissu FFPE.
5. Coloration par anticorps conjugués aux oligonucléotides
- Préparez les lames de couverture pour le placement des tissus.
- Faire tremper les lames de couverture dans une solution de poly-L-lysine à 0,1% pendant 24 h à TA pour améliorer l’adhérence des tissus.
- Après trempage, égoutter la solution de poly-L-lysine et laver les lamelles de couverture à l’eau ultrapure pendant 30 s. Répétez le lavage quatre à six fois. Retirez les lamelles de couverture de l’eau ultrapure utilisée pour le lavage et placez-les sur une serviette non pelucheuse pour sécher pendant la nuit.
REMARQUE: Un histologiste doit couper le tissu sélectionné en sections de 5 μm d’épaisseur, les placer au centre d’une lame de couverture chargée en poly-L-lysine et les laisser sécher pendant la nuit. Après séchage, les lamelles de couverture avec les sections de tissu doivent être conservées à 4 °C pendant 6 mois au maximum. Le panneau de coloration de ce protocole contenait 26 marqueurs. Le tissu utilisé dans ce protocole était du tissu amygdale humain normal. Le temps de trempage de la housse ne doit pas dépasser 1 semaine. Les lamelles de couverture revêtues de poly-L-lysine peuvent être stockées chez RT et doivent être utilisées dans les 2 mois suivant leur préparation.
- La veille de la coloration, placez le porte-couvercle dans un four à 60 °C pendant la nuit.
- Le lendemain, placez l’échantillon de bordereau dans un porte-lame-couvercle préchauffé dans un four à 60 °C. Après la cuisson pendant 30 minutes, vérifiez que la paraffine a fondu loin du tissu.
- Placer rapidement le porte-lame/échantillon dans la série de solutions suivante : deux cycles d’agent déparaffinant pendant 6 min chacun; deux cycles d’éthanol à 100 % pendant 5 min chacun; un tour d’éthanol à 90% pendant 5 min; un tour d’éthanol à 70% pendant 5 min; un tour d’éthanol à 50% pendant 5 min; un tour d’éthanol à 30% pendant 5 min; et deux cycles d’eau ultrapure traitée au pyrocarbonate de diéthyle (DEPC) pendant 5 minutes chacun.
NOTE: Toutes les dilutions d’éthanol sont préparées avec de l’eau ultrapure traitée au DEPC. Si le xylène est utilisé pour le déparaffinage, il doit être utilisé dans une hotte. - Lorsque vous soumettez le porte-feuillet/échantillon à la série de solutions, procédez comme suit :
- Préparez une chambre d’humidité en plaçant une boîte vide d’embout de pipette avec une serviette en papier imbibée d’eau au fond.
- Remplissez un autocuiseur avec suffisamment d’eau pour couvrir à moitié un bécher de 50 mL.
- Placer 5 mL de méthanol par échantillon dans le bécher à 4 °C.
- Diluer le tampon AR9 avec de l’eau ultrapure traitée DEPC à 1x; environ 50 mL du tampon dilué sont nécessaires par porte-lame.
- Une fois la série de solutions terminée, remplissez un bécher en verre de 50 ml avec environ 40 ml de 1x tampon AR9, immergez le porte-couvercle/échantillon dans le bécher et recouvrez complètement le porte-lame/couvercle avec du papier d’aluminium.
- Placer le porte-bécher/couvercle recouvert de papier d’aluminium dans l’autocuiseur rempli d’eau et cuire à haute pression (environ 15 psi) pendant 20 min.
- Après la cuisson, retirez le porte-bécher/couvercle, déballez soigneusement le papier d’aluminium et laissez le porte-bécher/couvercle refroidir à TA pendant environ 10 minutes.
- Retirez le porte-lame/échantillon du tampon AR9 1x et immergez-le dans deux cycles d’eau ultrapure traitée par DEPC, en effectuant une incubation des échantillons dans les deux tours pendant 2 minutes chacun.
- Pendant l’incubation, récupérez le tampon d’hydratation, le bloc N, le blocage G, le blocage J et le blocage des solutions S, ainsi que le diluant/bloc d’anticorps du kit de coloration d’imagerie multiplexé. Pour deux échantillons de lamelles de couverture, étiqueter les plaques à 6 puits pour les solutions en utilisant les configurations illustrées à la figure 2.
- Placer l’échantillon de la lamelle de couverture en deux cycles de 5 mL du tampon d’hydratation pendant 2 minutes chacun.
- Après les deux cycles de placement dans le tampon d’hydratation, placer l’échantillon de capuchon dans 5 ml du diluant/bloc d’anticorps d’imagerie multiplexé et incuber pendant 20 à 30 minutes à TA (ne pas dépasser 30 min).
- Pendant l’incubation, préparez un cocktail d’anticorps en préparant un mélange maître de 200 μL de diluant/bloc d’anticorps d’imagerie multiplexé, de bloqueur N, de bloqueur G, de bloqueur J et de solutions de bloqueur S.
REMARQUE : Calculez la quantité totale d’anticorps en fonction du nombre de marqueurs et du titrage validé de chaque marqueur, et soustrayez la quantité totale d’anticorps du mélange maître. Par exemple, pour six marqueurs, chacun avec un titrage de 1:200, l’équation serait 200 μL de mélange maître − 6 μL de cocktail d’anticorps = 194 μL de mélange maître.
- Après incubation dans le diluant/bloc d’anticorps d’imagerie multiplexé, placer l’échantillon de lamelle de couverture dans la chambre d’humidité préparée à l’étape 5.5.1, pipeter 190 μL du cocktail d’anticorps sur l’échantillon de lamelle de couverture et incuber l’échantillon de lamelle de couverture à TA pendant 3 h.
- Après l’incubation, laver l’échantillon de couvercle en deux cycles avec 5 mL du diluant/bloc d’anticorps imageur multiplexé pendant 2 minutes chacun.
- Pour fixer les anticorps liés au tissu sur la lame, effectuez les étapes 5.7.3-5.7.5 dans la chambre d’humidité.
- Incuber les lames de couverture pendant 10 min avec 16% de formaldéhyde dilué à 1,6% avec une solution de stockage, puis laver les lamelles trois fois avec 1x PBS.
- Incuber les lames de couverture pendant 5 min avec du méthanol à 4 °C, puis laver les lamelles trois fois avec 1x PBS.
- Incuber les lames de couverture pendant 20 min avec 5 mL du réactif fixateur dilué avec 1x PBS, puis laver les lamelles trois fois avec 1x PBS.
- Conservez les échantillons de lamelles tachées dans le tampon de stockage à 4 °C pendant 2 semaines maximum
6. Plaque rapporteur d’imagerie multiplexée
REMARQUE : Une plaque de 96 puits, appelée plaque rapporteure, contenant des fluorophores à code à barres dans des puits individuels est préparée selon des expériences d’imagerie multiplexées conçues sur mesure et est corrélée avec chaque échantillon de feuillet de couverture coloré. Les étapes suivantes concernent la préparation de la plaque rapporteure.
- Préparer un mélange-maître rapporteur en combinant 4 880 μL d’eau exempte de nucléases, 600 μL de tampon d’imagerie multiplexé 10x, 500 μL d’un réactif de dosage et 20 μL de solution de coloration nucléaire. Ce sera suffisant pour 20 cycles de tous les puits.
- À chaque cycle de l’expérience d’imagerie multiplexée conçue sur mesure, remplissez un puits avec 245 μL d’une solution contenant le mélange principal rapporteur et les fluorophores à code-barres spécifiques pour ce cycle.
REMARQUE : Voir la figure 3 pour la configuration de la plaque de rapport utilisée dans ce protocole pour un panneau de carcinome. - Pour protéger les fluorophores à code-barres, collez un couvercle de plaque en aluminium sur la plaque rapporteur et placez la plaque dans l’instrument d’imagerie multiplexé.
- Conservez la plaque rapporteur dans une boîte sombre à 4 °C pendant 2 semaines maximum.
7. Étalonnage et fonctionnement de l’appareil d’imagerie multiplexé
REMARQUE: Le microscope à fluorescence d’imagerie haute résolution capture quatre canaux de fluorescence différents dans chaque cycle d’imagerie multiplexé à 20x, 100% de lumière d’excitation et avec un faible photoblanchiment.
- Calibrez la focalisation de l’imagerie à l’aide du canal DAPI en plaçant une lame de couverture d’échantillon sur la platine du microscope, en pipetant manuellement 700 μL d’un titrage 1:1 500 de solution de coloration nucléaire sur le tissu.
REMARQUE: La lamelle de couverture est conservée sur l’étage du microscope pendant le lavage et l’imagerie de l’échantillon. - Pour préparer l’instrument d’imagerie multiplexé, diluer 10x tampon d’imagerie multiplexé à 1x avec de l’eau ultrapure traitée DEPC et remplir les flacons de réactifs avec des solutions/solvants appropriés, y compris le tampon d’imagerie multiplexé 1x dilué, de l’eau ultrapure traitée par DEPC et du diméthylsulfoxyde (DMSO).
- Une fois les flacons de réactifs remplis correctement, entrez le plan expérimental dans le logiciel de gestion d’instruments d’imagerie multiplexé; désigner le cycle, les numéros de puits, les emplacements de la pile Z, le nom du marqueur, la classe et le temps d’exposition appropriés pour chaque cycle (figure 4); régler tous les paramètres du microscope; et sélectionnez les régions d’intérêt sur l’exemple de bordereau de couverture à imager.
- Cliquez sur le bouton Expérimenter dans le logiciel de contrôle (Figure 4A). Dans la fenêtre Configuration et gestion des expériences , cliquez sur le bouton Nouveau modèle (Figure 4B).
- Tapez le nom du projet dans l’espace situé à côté du bouton Projet (Figure 4C). Tapez ou sélectionnez le nombre total de cycles (Figure 4D).
- Cliquez sur le bouton Channel Assignment (Attribution de canal ), saisissez les informations pour chaque cycle dans les colonnes (Figure 4E), puis cliquez sur le bouton Save Template (Enregistrer le modèle ). Démarrez l’expérience en cliquant sur le bouton Démarrer l’expérience .
REMARQUE: Au cours d’une expérience d’imagerie multiplexée, l’instrument récupère les fluorophores à code-barres d’un puits de la plaque rapporteur (maximum de quatre fluorophores par puits, y compris DAPI), les distribue directement sur la feuille de couverture de l’échantillon et imagera les régions d’intérêt pour chaque canal de fluorescence. Après toute l’imagerie de ce cycle, l’instrument lave les fluorophores à code-barres et distribue le prochain cycle de rapporteurs (à partir du puits suivant sur la plaque du rapporteur). L’imagerie se poursuit jusqu’à ce que tous les cycles utilisant 26 marqueurs soient terminés.
8. Collection d’images
REMARQUE: Les images multiplexées peuvent être collectées à l’aide de n’importe quel microscope à fluorescence inversée adapté configuré avec quatre canaux de fluorescence (DAPI, Cy3, Cy5 et Cy7) et équipé d’une lentille Plan Fluor 20x. L’imagerie et le lavage des échantillons de feuillet de couverture sont effectués automatiquement de manière itérative à l’aide d’une configuration fluidique spécialement développée. Les images sont acquises à l’aide du logiciel Processor (v1.8.0.7) au format QPTIFF.
- Dans la fenêtre du processeur, cliquez sur le bouton Entrée et sélectionnez le nom de l’expérience.
- Dans la section Options de traitement, sélectionnez et cochez Soustraction d’arrière-plan, Déconvolution, Profondeur de champ étendue et Correction de l’ombrage. Cliquez sur le bouton Démarrer.
9. Analyse d’images
REMARQUE : Les images acquises peuvent être téléchargées vers un logiciel breveté d’analyse d’images automatisée ou un logiciel open source (Figure 5) pour une analyse en aval.
- Cliquez sur l’icône QuPath sur l’ordinateur et ouvrez le logiciel. Faites glisser le fichier QPTIFF vers la fenêtre Viewer .
- Cliquez sur le bouton Luminosité et contraste, qui ouvrira la fenêtre Luminosité et contraste. Cochez ou décochez le marqueur dans la colonne Sélectionné pour afficher ou fermer le signal du marqueur.
- Cliquez sur le bouton Zoom pour ajuster pour effectuer un zoom avant ou arrière sur la zone d’intérêt.
REMARQUE: La visualisation de données d’image multiplexées fournit à l’utilisateur un aperçu approfondi du microenvironnement tissulaire. Les marqueurs individuels dans les échantillons FFPE peuvent être visualisés en fluorescence (Figure 6 et Figure 7) ou en vue pathologique. Plusieurs plates-formes de calcul peuvent être utilisées pour traiter les images composites et analyser les données d’images tissulaires multiplexées. À l’aide de ce logiciel, le phénotypage spatial, ainsi que la découverte de cellules rares et le calcul du voisinage cellulaire, peuvent être réalisés avec des images entières de tissus multiplexés ultra-élevés générées via la technologie d’imagerie multiplexée. Voir la figure 6 pour les 26 anticorps dans le panel de carcinome et l’échantillon d’amygdale (Figure supplémentaire 1).
Résultats
Nous avons utilisé des échantillons d’amygdales FFPE pour développer un panel d’immuno-oncologie à 26 marqueurs afin d’illustrer le statut immunitaire du tissu FFPE à l’aide d’un système d’analyse d’images à code-barres. Dans l’ensemble, 19 anticorps sont actuellement utilisés dans d’autres études d’imagerie multiplexée dans notre laboratoire. Tous les marqueurs ont été testés en utilisant du tissu FFPE avec IHC chromogénique. Tous les anticorps ont été conjugués à des oligonucléotides d’ADN uniques. Lors de la configuration des bordereaux à l’aide du gestionnaire d’instruments basé sur le Web (Figure 4) pour cette technologie d’analyse d’images à code-barres, il convient de noter que le premier et le dernier cycle sont toujours « blancs » (Figure 2 et Figure 4), ce qui fournit les signaux de fluorescence de fond à soustraire des signaux spécifiques des anticorps. Une fois que les images au format QPTIFF ont été collectées avec le microscope à fluorescence, elles peuvent être visualisées à l’aide de plusieurs logiciels d’analyse d’images automatisés brevetés ou de logiciels open source. Les images composites peuvent montrer tous les marqueurs ou les marqueurs sélectionnés pour une meilleure vue des signaux (Figure 5). De plus, chaque anticorps peut être évalué visuellement pour la localisation nucléaire, cytoplasmique ou membraneuse. Les cellules immunitaires, tumorales et stromales peuvent être facilement identifiées. Par la suite, l’analyse d’images peut fournir des informations sur l’intensité du signal, la plage dynamique et la distribution spatiale de tous les marqueurs (Figure 6). Cette technique nous a permis d’analyser les 26 marqueurs au niveau subcellulaire dans une seule coupe de tissu (Figure 7). En analysant la co-localisation des marqueurs, nous avons pu identifier les phénotypes cellulaires, localiser la position spatiale des cellules, calculer la distance entre les cellules et trouver la distribution des cellules. L’impact crucial de cette technologie est la présentation d’un panel robuste de 26 marqueurs axé sur le statut immunitaire du microenvironnement tissulaire.
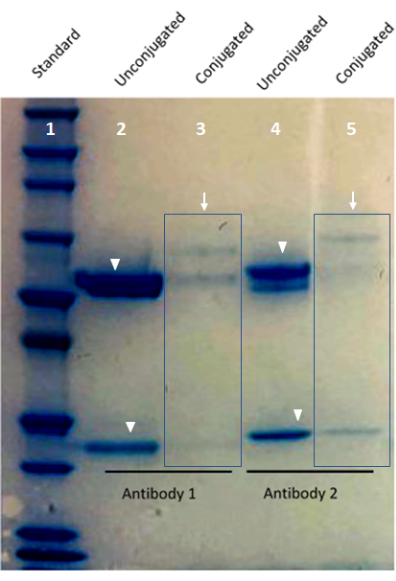
Figure 1 : Image de validation d’anticorps conjugués sur mesure à l’aide d’un gel de protéine Bis-Tris. La voie 1 du gel montre la norme protéique. La voie 2 et la voie 4 montrent des anticorps conjugués par code-barres (flèches). Les voies 3 et 5 montrent les bandes de chaîne lourdes et légères d’un anticorps non conjugué (pointes de flèches). Veuillez cliquer ici pour voir une version agrandie de cette figure.
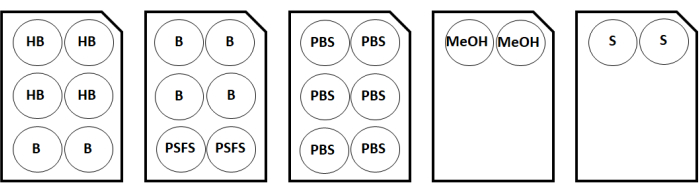
Figure 2 : Carte de configuration des plaques colorantes pour deux échantillons de lames de couverture. Abréviations : HB = tampon d’hydratation; B = diluant/bloc d’anticorps; PSFS = solution fixatrice post-coloration; PBS = solution saline tamponnée au phosphate; MeOH = méthanol; S = solution de stockage. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Configuration de la plaque rapporteure. Chaque anticorps conjugué a un code-barres complémentaire à un rapporteur spécifique. Pour configurer la plaque rapporteure, chaque anticorps conjugué et son rapporteur correspondant doivent être répertoriés. Ensuite, un numéro de cycle est attribué à chaque anticorps. La performance de deux cycles blancs (C1 et C18) est utilisée pour évaluer le niveau d’autofluorescence dans les trois canaux de fluorescence et pour la soustraction de fond post-imagerie à l’aide du logiciel de contrôle d’acquisition d’image. Un assistant logiciel vérifiera l’instrument à ce stade pour s’assurer que tous les réglages sont corrects (Figure 4). Veuillez cliquer ici pour voir une version agrandie de cette figure.
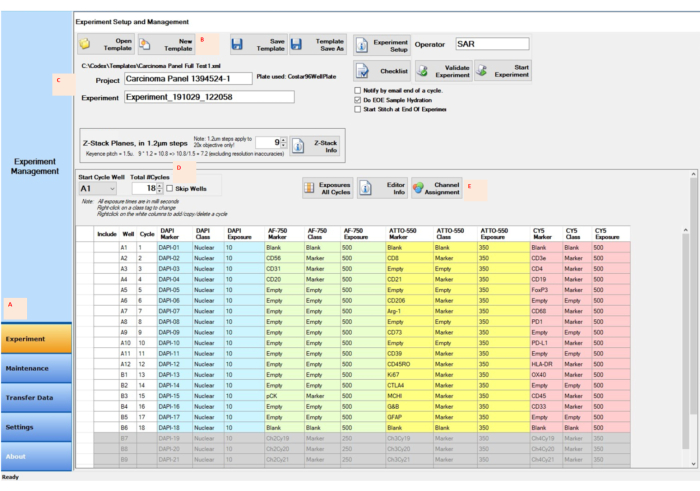
Figure 4 : Acquisition d’images à l’aide du logiciel de contrôle pour la configuration de l’expérience. (A) Sélectionnez l’onglet Expérience dans le coin inférieur gauche du logiciel de contrôle pour préparer et démarrer la configuration. (B,C) Sélectionnez Nouveau modèle pour entrer les paramètres expérimentaux avec un nouveau nom de projet et d’expérience. (D) Modifier le puits de cycle de démarrage et le nombre de cycles pour refléter l’emplacement du rapporteur dans la plaque de rapport de 96 puits. (E) Attribuer les canaux de fluorescence appropriés aux quatre canaux désignés pour l’essai. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Visualisation d’images à l’aide d’un logiciel Web (QuPath). La fenêtre du visualiseur montre 26 marqueurs dans la section FFPE du tissu des amygdales colorées. La fenêtre Luminosité et contraste affiche les marqueurs avec les coches. Enfin, la fenêtre de visualisation affiche l’échantillon FFPE avec les marqueurs sélectionnés. Veuillez cliquer ici pour voir une version agrandie de cette figure.
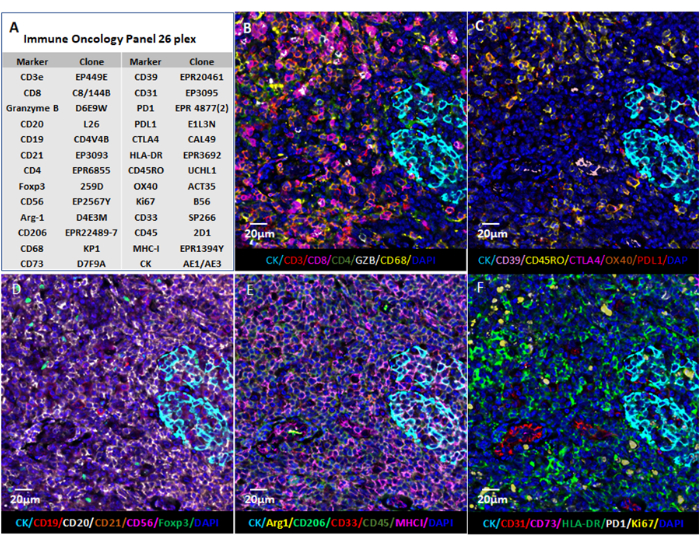
Figure 6 : Visualisation d’images à l’aide d’un logiciel Web. (A) Les coupes de tissu des amygdales ont été colorées pour les 26 marqueurs, et les images des sections en format QPTIFF ont été visualisées à l’aide d’un logiciel commercial de visualisation de diapositives numériques ou d’un logiciel open source (QuPath) pour annotation et examen. (B-F) Six marqueurs ont été affichés dans la même annotation pour une meilleure vue des signaux. Veuillez cliquer ici pour voir une version agrandie de cette figure.
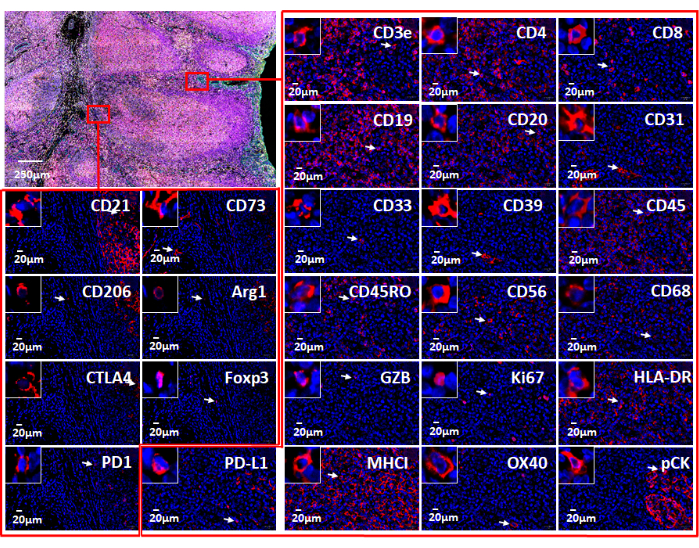
Figure 7 : Vues des 26 marqueurs individuels utilisés avec les logiciels Web. L’expression du marqueur dans le tissu des amygdales est montrée par coloration par immunofluorescence avec un panel d’immuno-oncologie (en haut à gauche). Des marqueurs individuels dans deux petites zones (rectangles rouges) sont montrés. L’encart agrandi affiche des cellules positives pour ces marqueurs (flèches blanches). Veuillez cliquer ici pour voir une version agrandie de cette figure.
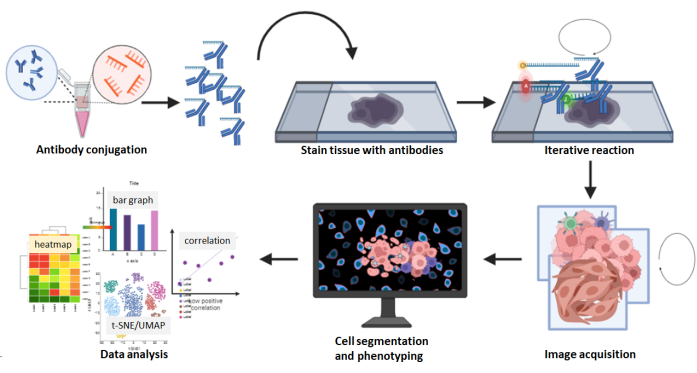
Figure 8 : Résumé du flux de travail d’acquisition d’images multiplexées. Les coupes de tissu FFPE ont été colorées à l’aide du panel de 26 anticorps suivis d’une réaction multicycle. Les images brutes des sections colorées ont été traitées informatiquement, et une analyse de la densité cellulaire et de l’espace a été effectuée à l’aide des images composites. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire 1 : Validation de l’IHC dans le tissu des amygdales. Les coupes de tissu FFPE ont été colorées à l’aide d’un anticorps individuel. L’expression du marqueur dans le tissu des amygdales est affichée à un faible grossissement, et l’insert zoomé montre des cellules positives pour le marqueur (rectangles rouges). Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Le CCM joue un rôle essentiel dans le développement, la progression et les réponses au traitement du cancer. De plus, la densité de sous-ensembles spécifiques de lymphocytes infiltrant les tumeurs dans le TME peut servir de biomarqueur pronostique pour certains types de cancer. Remarquablement, en plus de la composition cellulaire du TME, les caractéristiques spatiales d’une tumeur peuvent fournir un aperçu pour comprendre la biologie de la tumeur et identifier les biomarqueurs pronostiques potentiels12,17.
Comme de nombreuses populations de cellules immunitaires sont impliquées dans les réponses procancéreuses ou anticancéreuses, une meilleure compréhension de ces cellules et de leurs relations spatiales entre elles et avec les cellules cancéreuses aidera à orienter l’identification de nouvelles stratégies immunothérapeutiques. Des études antérieures ont stratifié l’emplacement et la distribution spatiale des cellules TME en fonction de la structure tissulaire dans les zones intratumorales et péritumorales et des marges invasives des cellules tumorales18,19. Au cours des 15 dernières années, les progrès technologiques ont fait de l’analyse phénotypique des cellules individuelles en fonction de leur dispersion spatiale un nouvel outil influent pour étudier l’ETM et catégoriser les biomarqueurs potentiels de l’immunothérapie tumorale. L’histochimie IF multiplex permet d’estimer simultanément plusieurs marqueurs biologiques20.
À l’instar de la stratégie des anticorps conjugués aux oligonucléotides, quatre types de plateformes multiplex à base de protéines sont utilisés pour étudier l’EMT : les systèmes de détection d’anticorps marqués par chromogènes, fluorescences, codes à barres ADN et isotopes métalliques. Les plates-formes IHC chromogènes rentables permettent la visualisation de lames entières et l’évaluation pathologique en utilisant la microscopie à fond clair conventionnelle. Dans les IF et IHC multiplexés, des anticorps conjugués avec des fluorophores sont utilisés. La plateforme multiplex IF/IHC détecte les anticorps à haute spécificité et peut quantifier les anticorps ciblés même au niveau subcellulaire 6,21. De plus, en raison de la nature des chromogènes et des fluorophores, l’utilisation d’un panel d’anticorps peut capturer l’expression de jusqu’à 10 biomarqueurs sur une seule lame. Sur les plates-formes à base d’isotopes métalliques, les anticorps marqués au métal sont utilisés pour effectuer une imagerie multiplexée avec une résolution unicellulaire et spatiale, et une sensibilité élevée pour des sections de tissus individuelles22. Théoriquement, ces approches d’anticorps conjugués au métal permettent la détection simultanée de plus de 100 biomarqueurs sur une seule coupe de tissu. L’un des défis de la technique de marquage isotopique est l’interférence isobare, qui empêche la pureté de 100% de l’enrichissement d’être atteinte23. De plus, les interférences augmentent à mesure que le nombre de marqueurs augmente. Les plateformes de détection d’anticorps conjugués à l’ADN reconnaissent les anticorps marqués avec des codes-barres ADN uniques. Plus de 40 biomarqueurs peuvent être capturés simultanément avec une grande spécificité sur ces plateformes6.
L’imagerie multiplexée est une plateforme de détection d’anticorps marqués par code-barres ADN disponible dans le commerce permettant d’appliquer des anticorps conjugués à l’ADN à une seule lame tissulaire en une seule étape (Figure 8). Pour l’étape de préparation des tissus, contrairement à la plate-forme d’imagerie par faisceau d’ions multiplexé, qui nécessite l’utilisation de lames recouvertes d’or obtenues auprès des fabricants, la plate-forme d’imagerie multiplexée ne nécessite que des lames de recouvrement régulières ou des lames recouvertes de poly-L-lysine à 0,1% pour aider le tissu à y adhérer et à garder le tissu intact pendant le processus de coloration et d’imagerie. L’utilisation de coupes de tissu sur les lames de couverture dans les 4 semaines suivant la sectionnement est recommandée, car le stockage prolongé des lames non tachées entraîne une réduction de l’antigénicité. Un échantillon de feuillet de couverture coloré peut être conservé dans un tampon de stockage à 4 °C jusqu’à 2 semaines sans perdre son signal de coloration. Aucun équipement spécial n’est requis pour le stockage des échantillons de lames de couverture. Le système d’imagerie multiplexé a été mis à niveau pour utiliser des lames régulières au lieu de lames de couverture, ce qui permet la coloration de tissus plus gros et une manipulation facile. Lors de l’utilisation d’une solution de réduction pour la conjugaison des anticorps (étape 3.2.3), la réaction doit être limitée à 30 minutes au maximum pour éviter d’endommager les anticorps. Les tampons de blocage de l’étape 5.6.6 doivent être fraîchement préparés et les tampons de blocage ne doivent pas être réutilisés.
Par rapport aux plates-formes de détection d’anticorps multiplex marqués par chromogènes, fluorescence et isotopes métalliques, la technologie d’imagerie multiplexée présente certains avantages. Par exemple, plus de 60 panels d’anticorps préconçus pour l’imagerie multiplexée sont disponibles dans le commerce, ce qui permet d’économiser du temps et de l’argent dans la conjugaison et la validation des anticorps, et le nombre de panels d’anticorps préconçus augmente. Ces anticorps, qui comprennent le marqueur du carcinome pancytokératine, le marqueur du mélanome SOX10, le marqueur vasculaire CD31, le marqueur stromal SMA et de nombreux marqueurs de cellules immunitaires, sont validés et prêts à être expérimentés. Pour les anticorps qui ne sont pas préconçus, le kit de conjugaison disponible dans le commerce conçu pour être utilisé avec l’imagerie multiplexée est simple et convivial. Les anticorps conjugués par le client sont bons pendant 1 an lorsqu’ils sont conservés à 4 °C. De plus, le préchauffage de la machine n’est pas nécessaire pour capturer les images. Dans cette technologie d’imagerie multiplexée, les étapes itératives de lavage, d’hybridation et de décapage dans l’acquisition de l’image entraînent rarement une diminution de l’intensité du marqueur ou une dégradation de la morphologie tissulaire 5,24,25. De plus, les images composites sont capturées au format QPTIFF avec un simple microscope à fluorescence tricolore et peuvent être téléchargées et analysées à l’aide d’un logiciel d’analyse numérique tiers. Les marqueurs de coloration peuvent être visualisés à une résolution unicellulaire, et les phénotypes cellulaires peuvent être caractérisés via la co-localisation des marqueurs (Figure 6 et Figure 7). L’analyse complète d’une image multiplexée révèle en outre les compartiments tissulaires, la quantification des marqueurs unicellulaires et les données de voisinage et de proximité les plus proches (Figure 8).
Un défi dans l’analyse d’images multiplexées est l’identification du type de cellule. Habituellement, lorsque plusieurs classificateurs à objet unique sont appliqués à une image, des phénotypes plus rares seront annotés. Par conséquent, il est recommandé d’utiliser des marqueurs connus qui ne sont pas co-exprimés dans le même classificateur et d’appliquer uniquement le classificateur lié au phénotype à l’annotation de cellules individuelles. Les variations dans l’annotation du type de cellule entraîneront des résultats spatiaux sensiblement différents, tels que des différences dans la distribution spatiale cellulaire et l’analyse du voisinage cellulaire26,27.
L’analyse d’images multiplexées s’est avérée efficace pour colorer et imager de nombreux types d’échantillons, y compris les tissus FFPE, les tissus frais congelés, les lames entières archivées et les puces à tissus. Des images multiplexées de sections de seins, de cerveaux, de poumons, de rate, de reins, de ganglions lymphatiques et de tissus cutanés peuvent être acquises avec des données de phénotypage spatial unicellulaire profondes 5,16,25,28.
À l’avenir, davantage d’anticorps préconçus pour l’imagerie multiplexée sont attendus. En outre, le développement d’un logiciel spécifique pour l’analyse d’images multiplexées est grandement nécessaire. Actuellement, de nombreux logiciels disponibles dans le commerce et open source pour l’analyse d’images Hi-Plex existent29, mais les scientifiques ont encore besoin d’aide pour créer un flux de travail standard pour ces analyses30,31. Bien que les images composites capturées à l’aide de ce protocole soient compatibles avec des logiciels tiers, cela peut entraîner des coûts supplémentaires pour l’utilisateur. Un autre inconvénient de la technologie d’imagerie multiplexée est la réduction du signal dans la détection des protéines nucléaires après le lavage itératif, l’hybridation et le stripping avec de grands panels d’anticorps. Heureusement, cela peut être minimisé en récupérant les fluorophores à code-barres dès les premiers cycles lors de la conception des plaques rapporteures. Récemment, cette plate-forme a été mise à niveau avec un nouveau système de numérisation à grande vitesse, ce qui a considérablement réduit le temps d’obtention d’images composites32. De plus, une nouvelle stratégie utilisant des codes-barres conjugués au tyramide a été signalée pour améliorer l’imagerie basée sur le codage à barres des anticorps conjugués aux oligonucléotides. Cette technologie vise à amplifier les signaux de coloration pour lesquels les anticorps conjugués code-barres sont difficiles à obtenir33.
Déclarations de divulgation
Les auteurs n’ont aucun conflit à révéler.
Remerciements
Les auteurs remercient Donald R. Norwood des services de révision, Research Medical Library de MD Anderson pour la rédaction de cet article et le laboratoire multiplex IF et d’analyse d’images du département de pathologie moléculaire translationnelle de MD Anderson. Ce projet a été soutenu en partie par la plateforme Moonshots du laboratoire de pathologie moléculaire translationnelle et d’immunoprofilage (TMP-IL) du département de pathologie moléculaire translationnelle, le MD Anderson Cancer Center de l’Université du Texas et le NCI Cooperative Agreement U24CA224285 (au MD Anderson Cancer Center CIMAC).
matériels
| Name | Company | Catalog Number | Comments |
| 10x AR9 Buffer | Akoya Biosciences | AR900250ML | |
| 10x Buffer | Akoya Biosciences | 7000001 | |
| 16% paraformaldehyde | Thermo Fisher Scientific | 28906 | |
| 1X Antibody Diluent/Block | Akoya Biosciences | ARD1001EA | |
| Antibody Conjugation Kit | Akoya Biosciences | 7000009 | Contains Filter Blocking Solution, Antibody Reduction Solution 1, Antibody Reduction Solution 2, Conjugation Solution, Purification Solution, Antibody Storage Solution |
| Assay Reagent | Akoya Biosciences | 7000002 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| Dimethyl sulfoxide | Avantor/VWR | BDH1115-4LP | |
| Ethanol, 200 proof | |||
| G Blocker V2 | Akoya Biosciences | 240199 | |
| Histoclear | Thermo Fisher Scientific | 50-329-50 | |
| Methanol | Sigma-Aldrich | 34860-1L-R | |
| Milli-Q Integral 10 | Millipore | ZRXQ010WW | |
| Niknon Fluorescence microscope | Keyence Corp. of America | BZ-X810 | |
| Nuclear Stain | Akoya Biosciences | 7000003 | |
| Nuclease-free water | Thermo Fisher Scientific | AM9938 | |
| NuPAGE | Thermo Fisher Scientific | NP0008 | |
| PBS | Thermo Fisher Scientific | 14190136 | |
| PhenoCycler Barcodes/Reporters Combination | Akoya Biosciences | 5450004 (BX025/RX025) | |
| 5450003 (BX022/RX022) | |||
| 5450023 (BX002/RX002) | |||
| 5250002 (BX020/RX020) | |||
| 2520003 (BX023/RX023) | |||
| 5250005 (BX029/RX029) | |||
| 5250007 (BX035/RX035) | |||
| 5250012 (BX052/RX052) | |||
| 5550012 (BX030/RX030) | |||
| 5550015 (BX042/RX042) | |||
| 5550014 (BX036/RX036) | |||
| QuPath | Open-Source | https://qupath.github.io/ | |
| SimplyBlue SafeStain | Thermo Fisher Scientific | LC6065 | |
| Staining Kit | (Akoya Biosciences | 7000008 | Contains Hydration Buffer, N Blocker, J Blocker, S Blocker, Fixative Reagent, Storage Buffer |
Références
- Hanahan, D., Coussens, L. M. Accessories to the crime: Functions of cells recruited to the tumor microenvironment. Cancer Cell. 21 (3), 309-322 (2012).
- Labani-Motlagh, A., Ashja-Mahdavi, M., Loskog, A. The tumor microenvironment: A milieu hindering and obstructing antitumor immune responses. Frontiers in Immunology. 11, 940 (2020).
- Jin, M. Z., Jin, W. L. The updated landscape of tumor microenvironment and drug repurposing. Signal Transduction and Targeted Therapy. 5 (1), 166 (2020).
- Waldman, A. D., Fritz, J. M., Lenardo, M. J. A guide to cancer immunotherapy: From T cell basic science to clinical practice. Nature Reviews Immunology. 20 (11), 651-668 (2020).
- Black, S., et al. CODEX multiplexed tissue imaging with DNA-conjugated antibodies. Nature Protocols. 16 (8), 3802-3835 (2021).
- Tan, W. C. C., et al. Overview of multiplex immunohistochemistry/immunofluorescence techniques in the era of cancer immunotherapy. Cancer Communications. 40 (4), 135-153 (2020).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 117 (52), 33455-33465 (2020).
- Allam, M., Cai, S., Coskun, A. F. Multiplex bioimaging of single-cell spatial profiles for precision cancer diagnostics and therapeutics. NPJ Precision Oncology. 4, 11 (2020).
- Eng, J., et al. A framework for multiplex imaging optimization and reproducible analysis. Communications Biology. 5 (1), 438 (2022).
- Taube, J. M., et al. Multi-institutional TSA-amplified multiplexed immunofluorescence reproducibility evaluation (MITRE) study. Journal for Immunotherapy of Cancer. 9 (7), e002197 (2021).
- Binnewies, M., et al. Understanding the tumor immune microenvironment (TIME) for effective therapy. Nature Medicine. 24 (5), 541-550 (2018).
- Heindl, A., Nawaz, S., Yuan, Y. Mapping spatial heterogeneity in the tumor microenvironment: A new era for digital pathology. Laboratory Investigation. 95 (4), 377-384 (2015).
- Kuswanto, W., Nolan, G., Lu, G. Highly multiplexed spatial profiling with CODEX: bioinformatic analysis and application in human disease. Seminars in Immunopathology. 45 (1), 145-157 (2022).
- Phillips, D., et al. Immune cell topography predicts response to PD-1 blockade in cutaneous T cell lymphoma. Nature Communications. 12 (1), 6726 (2021).
- Jhaveri, N., et al. Deep ultrahigh-plex spatial phenotyping of human cancer tissues. Cancer Research. 82, 3877 (2022).
- Goltsev, Y., et al. Deep profiling of mouse splenic architecture with CODEX multiplexed imaging. Cell. 174 (4), 968-981 (2018).
- Yuan, Y. Spatial heterogeneity in the tumor microenvironment. Cold Spring Harbor Perspectives in Medicine. 6 (8), a026583 (2016).
- Bruck, O., et al. Spatial immunoprofiling of the intratumoral and peritumoral tissue of renal cell carcinoma patients. Modern Pathology. 34 (12), 2229-2241 (2021).
- Tsujikawa, T., et al. Prognostic significance of spatial immune profiles in human solid cancers. Cancer Science. 111 (10), 3426-3434 (2020).
- Hoyt, C. C. Multiplex immunofluorescence and multispectral imaging: forming the basis of a clinical test platform for immuno-oncology. Frontiers in Molecular Biosciences. 8, 674747 (2021).
- Parra, E. R., et al. Identification of distinct immune landscapes using an automated nine-color multiplex immunofluorescence staining panel and image analysis in paraffin tumor tissues. Scientific Reports. 11 (1), 4530 (2021).
- Elaldi, R., et al. High dimensional imaging mass cytometry panel to visualize the tumor immune microenvironment contexture. Frontiers in Immunology. 12, 666233 (2021).
- Campos Clemente, L., Shi, O., Rojas, F., Parra, E. R. Expanding the comprehension of the tumor microenvironment using mass spectrometry imaging of formalin-fixed and paraffin-embedded tissue samples. Journal of Visualized Experiments. (184), e64015 (2022).
- Schurch, C. M., et al. Coordinated cellular neighborhoods orchestrate antitumoral immunity at the colorectal cancer invasive front. Cell. 182 (5), 1341-1359 (2020).
- Phillips, D., et al. Highly multiplexed phenotyping of immunoregulatory proteins in the tumor microenvironment by CODEX tissue imaging. Frontiers in Immunology. 12, 687673 (2021).
- Hickey, J. W., Tan, Y., Nolan, G. P., Goltsev, Y. Strategies for accurate cell type identification in CODEX multiplexed imaging data. Frontiers in Immunology. 12, 727626 (2021).
- Parra, E. R. Methods to determine and analyze the cellular spatial distribution extracted from multiplex immunofluorescence data to understand the tumor microenvironment. Frontiers in Molecular Biosciences. 8, 668340 (2021).
- Tzoras, E., et al. Dissecting tumor-immune microenvironment in breast cancer at a spatial and multiplex resolution. Cancers. 14 (8), 1999 (2022).
- Francisco-Cruz, A., Parra, E. R., Tetzlaff, M. T. Wistuba, II. Multiplex immunofluorescence assays. Methods in Molecular Biology. 2055, 467-495 (2020).
- Shakya, R., Nguyen, T. H., Waterhouse, N., Khanna, R. Immune contexture analysis in immuno-oncology: applications and challenges of multiplex fluorescent immunohistochemistry. Clinical and Translational Immunology. 9 (10), e1183 (2020).
- Wilson, C. M., et al. Challenges and opportunities in the statistical analysis of multiplex immunofluorescence data. Cancers. 13 (12), 3031 (2021).
- DeRosa, J. Setting a new standard for spatial omics: An integrated multiomics approach: every single cell, two different analytes, one unbiased picture. Genetic Engineering & Biotechnology News. 42, 26-28 (2022).
- Simonson, P. D., Valencia, I., Patel, S. S. Tyramide-conjugated DNA barcodes enable signal amplification for multiparametric CODEX imaging. Communications Biology. 5 (1), 627 (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon