Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Immunfärbungsbasierter Nachweis dynamischer Veränderungen in Erythrozytenproteinen
In diesem Artikel
Zusammenfassung
Die Erfassung dynamischer Veränderungen in der Proteinaktivierung entkernter roter Blutkörperchen stellt methodische Herausforderungen dar, wie z.B. die Konservierung dynamischer Veränderungen akuter Stimuli für eine spätere Bewertung. Das vorgestellte Protokoll beschreibt Probenvorbereitungs- und Färbetechniken, die die Konservierung und Analyse relevanter Proteinveränderungen und den anschließenden Nachweis ermöglichen.
Zusammenfassung
Die Antikörpermarkierung von Erythrozytenproteinen (RBC) ist eine häufig verwendete, semiquantitative Methode, um Veränderungen des Gesamtproteingehalts oder akute Veränderungen der Proteinaktivierungszustände zu erkennen. Es erleichtert die Beurteilung von Erythrozytenbehandlungen, die Charakterisierung von Unterschieden in bestimmten Krankheitszuständen und die Beschreibung zellulärer Kohärenzen. Der Nachweis einer akut veränderten Proteinaktivierung (z.B. durch Mechanotransduktion) erfordert eine adäquate Probenvorbereitung, um ansonsten temporäre Proteinmodifikationen zu erhalten. Das Grundprinzip beinhaltet die Immobilisierung der Zielbindungsstellen der gewünschten Erythrozytenproteine, um die initiale Bindung spezifischer Primärantikörper zu ermöglichen. Die Probe wird weiterverarbeitet, um optimale Bedingungen für die Bindung des Sekundärantikörpers an den entsprechenden Primärantikörper zu gewährleisten. Die Auswahl von nicht-fluoreszierenden Sekundärantikörpern erfordert eine zusätzliche Behandlung, einschließlich der Biotin-Avidin-Kopplung und der Anwendung von 3,3-Diaminobenzidin-Tetrahydrochlorid (DAB), um die Färbung zu entwickeln, die in Echtzeit unter dem Mikroskop kontrolliert werden muss, um die Oxidation und damit die Färbeintensität rechtzeitig zu stoppen. Für die Detektion der Färbeintensität werden Bilder mit einem Standard-Lichtmikroskop aufgenommen. In einer Modifikation dieses Protokolls kann stattdessen ein Fluorescein-konjugierter Sekundärantikörper eingesetzt werden, was den Vorteil hat, dass kein weiterer Entwicklungsschritt notwendig ist. Dieses Verfahren erfordert jedoch ein Fluoreszenzobjektiv, das an einem Mikroskop befestigt ist, um die Färbung zu detektieren. Angesichts des semi-quantitativen Charakters dieser Methoden ist es unerlässlich, mehrere Kontrollfärbungen bereitzustellen, um unspezifische Antikörperreaktionen und Hintergrundsignale zu berücksichtigen. Hier stellen wir sowohl Färbeprotokolle als auch die entsprechenden analytischen Verfahren vor, um die jeweiligen Ergebnisse und Vorteile der verschiedenen Färbetechniken zu vergleichen und zu diskutieren.
Einleitung
Rote Blutkörperchen (RBCs) durchqueren das Herz-Kreislauf-System 70 bis 140 Tage lang, mit einem mittleren Erythrozytenalter von etwa 115 Tagen 1,2. Seneszente oder beschädigte Erythrozyten werden durch Erythrophagozytose, einen effizienten Clearing-Prozess, der von Makrophagen angetrieben wird, aus dem Kreislauf entfernt3. Die vorgegebene Lebensdauer dieser Zellen ist eine Folge der Abgabe der Zellorganellen, einschließlich des Zellkerns, der Mitochondrien und der Ribosomen, während der Differenzierung und Reifung4. Daher sind zirkulierende Erythrozyten frei von einer Translationsmaschinerie, was die Synthese neuer Proteine ausschließt3. Daraus folgt, dass dynamische, posttranslationale Modifikationen an bestehenden Proteinen den einzigen praktikablen Mechanismus der akuten, biochemischen Regulation als Reaktion auf extrazelluläre und intrazelluläre Stressoren darstellen, die auf Erythrozyten einwirken5.
Mechanische Kräfte scheinen die wichtigsten extrazellulären Signale zu sein, die die Aktivierung oder Modulation biochemischer Signalwege innerhalb von Erythrozyten verursachen. Die Entdeckung des mechanosensitiven Proteins Piezo1 in Erythrozytenmembranen6 inspirierte mehrere Forschungslinien, die die mechanisch aktivierte Signalübertragung in diesen Zellen untersuchten7. Jüngste Fortschritte haben beispielsweise gezeigt, dass die physikalischen Eigenschaften von Erythrozyten aktiv durch akute und dynamische Veränderungen von Proteinenreguliert werden 8, einschließlich der posttranslationalen Phosphorylierung und Ubiquitinierung9. Da sich diese normalen Modifikationen bei bestimmten Erkrankungen unterscheiden 9,10,11, scheint es von wissenschaftlichem und klinischem Interesse zu sein, den Aktivierungszustand von Erythrozytenproteinen zu bestimmen, insbesondere in Bezug auf mechanobiologische Prozesse.
Die Bestimmung akuter Veränderungen in den Aktivierungszuständen von Erythrozytenproteinen bringt einige methodische Herausforderungen mit sich. Zum Beispiel erfordert die Lagerung von Erythrozytenproben für die spätere Analyse die Konservierung der modifizierten Erythrozytenproteine, da posttranslationale Modifikationen nicht dauerhaft sind. Darüber hinaus sind klassische Proteinnachweismethoden (z. B. Western Blotting) in Erythrozyten aufgrund der geringen Häufigkeit von Proteinen im Vergleich zum Hämoglobin, das ~98% des Proteingehalts in diesen Zellen ausmacht, notorisch schwer zu standardisieren12. Daher ist die Antikörper-basierte Färbung chemisch konservierter Erythrozyten die Methode der Wahl bei der Untersuchung akuter Modifikationen wichtiger Erythrozytenproteine, wie z. B. der RBC-spezifischen Isoform der Stickstoffmonoxid-Synthase (RBC-NOS)13,14. Es wurde gezeigt, dass Erythrozyten-NOS enzymatisch Stickstoffmonoxid (NO) produziert, das für wesentliche Erythrozyteneigenschaften, einschließlich der Erythrozytenverformbarkeit, unverzichtbar zu seinscheint 15,16,17. Posttranslationale Modifikationen von RBC-NOS regulieren die katalytische Enzymaktivität, wobei die Phosphorylierung des Serin-1177-Rests beschrieben wird, um die Enzymaktivität zu erhöhen, während die Phosphorylierung der Reste Serin 114 oder Threonin 495 mit einer verminderten RBC-NOS-Aktivität in Verbindung gebracht wurde18,19.
Insgesamt tragen temporäre Modifikationen von Erythrozytenproteinen zu einer wichtigen zellulären Funktion bei, und standardisierte Protokolle, die den Nachweis dieser modifizierten Proteine ermöglichen, sind von großem Wert. In dieser Arbeit stellen wir zwei unterschiedliche Protokolle vor, die spezifische Antikörper nutzen, um den Nachweis der Aktivierung des RBC-NOS-Proteins zu erleichtern, und diskutieren Empfehlungen für die Datenanalyse und -interpretation.
Die Leistung der beschriebenen Protokolle wurde durch Messung des gut berichteten Anstiegs der Phosphorylierung von RBC-NOS am Serin-1177-Rest als Reaktion auf mechanische Kräfte bewertet, die die im menschlichen Gefäßsystem auftretenden Kräfte widerspiegeln (5 Pa).
Protokoll
Die hier beschriebenen Protokolle stehen im Einklang mit der Deklaration von Helsinki und wurden von den Ethikkommissionen der Deutschen Sporthochschule Köln (16.9.2013) und der Griffith University (2019/808) genehmigt. Die Probanden wurden untersucht, um sicherzustellen, dass keine relevanten Pathologien vorhanden waren, und erhielten eine schriftliche Einverständniserklärung.
1. Färbung von Erythrozytenproteinen mit immunhistochemischen Protokollen
HINWEIS: Eine detaillierte Liste der erforderlichen Chemikalien und Materialien finden Sie in der Materialtabelle. In den folgenden Abschnitten wird die Herstellung der erforderlichen Lösungen beschrieben, gefolgt von einer detaillierten Beschreibung des immunhistochemischen Protokolls (Abbildung 1).
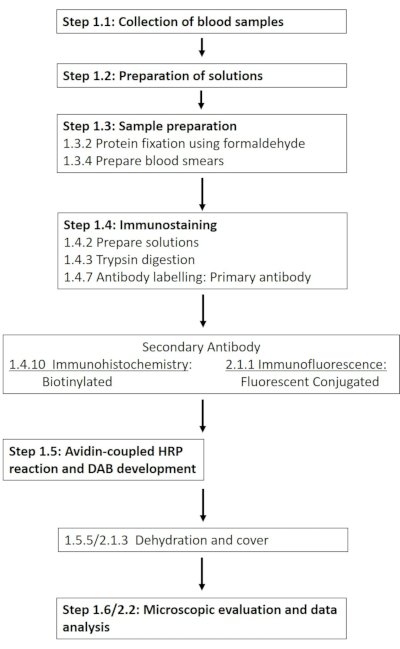
Abbildung 1: Schematische Darstellung der einzelnen Schritte, die für die immunhistochemische und Immunfluoreszenzfärbung von Erythrozyten-NOS an der Phosphorylierungsstelle 1177 erforderlich sind. Es wird ein typischer Arbeitsablauf der vorgestellten Protokolle vorgestellt, der von der Lösungsvorbereitung über die Blutentnahme bis hin zur Antikörper-basierten Detektion und Visualisierung reicht. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Entnahme der Blutprobe
- Entnehmen Sie Blutproben (10 ml Vollblut) von sieben gesunden Männern. Bei den Freiwilligen handelte es sich um gesunde Personen, die Berichten zufolge frei von kardiovaskulären, hämatologischen, neurologischen, endokrinen oder metabolischen Erkrankungen waren. Die Freiwilligen waren auch Nichtraucher.
- Das Blut wird mit einer sterilen Nadel und Spritze aus einer prominenten Vene im antecubitalen Bereich des Unterarms entnommen und sofort in Röhrchen umgefüllt, die mit einem der folgenden Antikoagulanzien beschichtet sind: Natriumheparin (für die Immunhistochemie) oder Ethylendiamintetraessigsäure (EDTA; für die Fluoreszenzmarkierung).
- Die antikoagulierten Blutproben werden mit einem Couette-Schersystem,wie oben 7,20 beschrieben, genau kontrollierten mechanischen Kräften ausgesetzt.
- Die Schervorrichtung besteht aus einem drehbaren Becher und einem stationären Bob, die durch einen Spalt von 300 μm voneinander getrennt sind. Die Blutprobe wird in den Spalt überführt, wodurch die präzise steuerbare Rotationsgeschwindigkeit des Bechers gut kontrollierte mechanische Kräfte auf die Probe ausübt. Die hier präsentierten repräsentativen Daten wurden durch Anwendung mechanischer Kräfte erzeugt, die die im menschlichen Gefäßsystem auftretenden Kräfte (5 Pa) über einen längeren Zeitraum (300 s) widerspiegeln.
- Herstellung von Lösungen, die für die Immunfärbung erforderlich sind
- Bereiten Sie die Lösungen wie in Tabelle 1 vor. Die Lösungen können vor der Durchführung des Immunfärbeverfahrens hergestellt und bei einer bestimmten Temperatur gelagert werden, bis sie benötigt werden.
HINWEIS: Die Lagerdauer kann je nach Chemikalien variieren. Prüfen Sie das Sicherheitsdatenblatt.
- Bereiten Sie die Lösungen wie in Tabelle 1 vor. Die Lösungen können vor der Durchführung des Immunfärbeverfahrens hergestellt und bei einer bestimmten Temperatur gelagert werden, bis sie benötigt werden.
- Probenvorbereitung
- Verarbeiten Sie Vollblut unmittelbar nach der Entnahme oder experimentellen Behandlung (falls zutreffend; z. B. mechanischer Reiz in unseren Proben; siehe Schritt 1.1.3), um kurzlebige Effekte erkennen zu können, z. B. nach Scherbelastung, Belastung, kurzzeitiger Hypoxie-Exposition usw.
- Führen Sie die Erythrozytenproteinfixierung mit Formaldehyd wie folgt durch: Vollblut im Verhältnis 1:2 in 4%iger Paraformaldehydlösung 20 Minuten lang bei Raumtemperatur (RT) verdünnen. Die Probe wird bei 132 x g für 3 min bei RT zentrifugiert und der Überstand vorsichtig durch Pipettieren entfernt.
- Das Erythrozytenpellet wird in zwei Volumina von 0,1 mol/l phosphatgepufferter Kochsalzlösung (PBS) (1:3-Verdünnung) resuspendiert und 5 min bei RT inkubiert. Wiederholen Sie die Zentrifugation wie oben beschrieben und entfernen Sie den Überstand, der klar sein sollte, durch Pipettieren. Resuspendieren Sie das Erythrozytenpellet in einem Volumen von 0,1 mol/L PBS (1:2-Verdünnung).
- Bereiten Sie Blutausstriche wie folgt vor: Beschriften Sie einen Objektträger mit der Proben-ID, dem applizierten Antikörper usw. mit einem alkoholbeständigen Stift (z. B. Bleistift). Geben Sie dann 10 μl der vorbereiteten Erythrozytenlösung direkt über das Etikettenfeld.
- Legen Sie einen zweiten Objektträger in einem Winkel von ca. 45° auf die Probe und verteilen Sie die Probe gleichmäßig entlang des Objektträgers. Fixieren Sie den Objektträger über einem Bunsenbrenner, indem Sie die Probe mit konstanter Bewegung 5-7 s lang über den Brenner schweben lassen.
HINWEIS: Es wird empfohlen, die Proben vor der Hitzefixierung an der Luft zu trocknen. Die Proben können bis zur Färbung bei RT gelagert werden (Abbildung 2).
- Immunhistochemische Färbung
- Markieren Sie auf jedem Objektträger zwei Bereiche: einen Testbereich, in dem die Erythrozyten mit dem jeweiligen Primär- und Sekundärantikörper inkubiert werden, und einen Kontrollbereich, in dem der Primärantikörper durch eine Kontrolllösung ersetzt wird. Dieser Bereich dient als Kontrolle innerhalb des Assays, um das Hintergrundsignal der Erythrozyten zu bestimmen.
- Bereiten Sie die Lösungen vor oder während des Eingriffs gemäß Tabelle 2 vor. Es wird ein Volumen von 300 μl pro Testbereich und 200 μl pro Kontrollbereich benötigt.
HINWEIS: Es ist wichtig zu beachten, dass diese antikörperbasierten Färbeverfahren eher semiquantitative als quantitative Daten liefern. Um sicherzustellen, dass aus den gewonnenen Daten aussagekräftige Schlussfolgerungen gezogen werden können, sind daher geeignete Kontrollproben (d.h. als Referenzpunkt für die bewerteten relativen Veränderungen der Proteinaktivierung) unbedingt erforderlich. - Für den Trypsinaufschluss werden 0,1 % Trypsin aufgetaut und auf RT äquilibriert. Markieren Sie mit einem Fettstift zwei Bereiche auf jedem Objektträger: einen Testbereich (2/3 des Objektträgers) und einen Kontrollbereich (1/3 des Objektträgers). Tragen Sie Tris-gepufferte Kochsalzlösung (TBS) vorsichtig mit Transferpipetten auf, um die Probenbereiche zu waschen. Lassen Sie die TBS 30 s ruhen und gießen Sie sie dann ab. Wiederholen Sie den Waschschritt.
Hinweis: Fügen Sie die folgenden Lösungen sowohl dem Steuerelement als auch dem Testbereich hinzu, sofern nicht anders beschrieben. Die Bereiche müssen mit der jeweiligen Lösung ausreichend abgedeckt sein. Tragen Sie die Lösungen mit Einweg-Transferpipetten auf und wechseln Sie die Pipetten nach jeder Lösung. - Fügen Sie 0,1 % Trypsin hinzu, decken Sie die Objektträger mit einer speziell angefertigten Inkubationskammer mit einem Deckel oder Aluminiumfolie ab und inkubieren Sie 30 Minuten lang bei 37 °C in einem Inkubator. Stoppen Sie nach der Inkubation die Enzymreaktion durch Zugabe von Leitungswasser. Gießen Sie die Lösung ab. Waschen Sie beide Bereiche 3x mit TBS, wie oben beschrieben.
- Um die Peroxidaseaktivierung zu blockieren, fügen Sie Methanollösung hinzu und inkubieren Sie 30 Minuten lang bei RT. Decken Sie die Objektträger während dieser Zeit ab. Gießen Sie die Lösung ab.
ANMERKUNG: Für den Immunfluoreszenznachweis (Abschnitt 2) kann dieser Schritt entfallen, da die Blockierung endogener Peroxidasen dazu dient, ein künstliches Signal zu verhindern, das durch den Nachweis mit Meerrettichperoxidase (HRP) und 3,3′-Diaminobenzidinhydrat (DAB) verursacht wird. - Waschen Sie beide Bereiche 3x mit TBS, wie in Schritt 1.4.3 beschrieben. Fügen Sie 3%ige Magermilchlösung hinzu und inkubieren Sie 30 Minuten lang bei RT, um zu blockieren. Gießen Sie die Lösung ab. Danach nicht mehr waschen.
- Primärantikörper (AB) nur in den Testbereich geben. Geben Sie die AB-Steuerungslösung (siehe Tabelle 2) in den Kontrollbereich ein. Die Proben werden über Nacht bei 4 °C inkubiert. Decken Sie die Objektträger während dieser Zeit ab, um ein Austrocknen zu verhindern.
HINWEIS: Vermeiden Sie es, die Lösungen aus dem Test- in den Kontrollbereich zu übertragen, da dies die Kontrollwerte beeinflusst. - Gießen Sie die Antikörperlösung ab. Waschen Sie beide Bereiche 3x mit TBS, wie in Schritt 1.4.3 beschrieben.
- Führen Sie einen Blockierungsschritt aus, um eine unspezifische Bindung zu verhindern. Fügen Sie 3% normales Ziegenserum hinzu und inkubieren Sie 30 Minuten lang bei RT. Gießen Sie die Lösung ab.
- Fügen Sie die sekundäre Antikörperlösung hinzu und inkubieren Sie 30 Minuten lang bei RT. Gießen Sie die Antikörperlösung ab. Waschen Sie die Stellen 3x mit TBS, wie in Schritt 1.4.3 beschrieben.
- Entwicklung von Färbung und Abdeckung
- Die Avidin-gekoppelte HRP-Reaktion (siehe Verdünnung in Tabelle 2) wird durch Zugabe von verdünnter Avidin-Peroxidase-Lösung durchgeführt und 30 Minuten lang bei RT inkubiert.
- Bereiten Sie die DAB-Mischung vor, um die Immunfärbung vor der Verwendung gemäß Tabelle 3 zu entwickeln.
VORSICHT: DAB ist gefährlich. Beachten Sie die Gefahrenhinweise H341 (im Verdacht, genetische Defekte zu verursachen) und H350 (kann Krebs verursachen). Beachten Sie die folgenden Sicherheitshinweise: P201, P202, P280, P308+P313, P405 und P501. - Führen Sie die DAB-Kontrollfärbung wie folgt durch: Die HRP-Lösung aus Schritt 1.5.1 wird von einem Objektträger entnommen und vor dem Färben mit einem kleinen Volumen (z. B. 1 ml) der vorbereiteten DAB-Lösung in einem separaten Zentrifugenröhrchen gemischt. Die Mischung sollte eine braun/graue Farbe entwickeln.
- Legen Sie die Objektträger unter ein Mikroskop (mindestens 200-fache Vergrößerung) und geben Sie DAB-Lösung in beide Bereiche. Überwachen Sie die Färbung der Erythrozyten kontinuierlich und stoppen Sie die Färbung, indem Sie die DAB-Lösung mit einer Einwegpipette entfernen, bevor der Hintergrund zu färben beginnt. Für die RBC-NOS-Serin-1177-Färbung beträgt die DAB-Inkubationszeit etwa 17 Minuten.
- Führen Sie eine Probentrocknung durch. Legen Sie die Objektträger in ein Glasgestell und tauchen Sie sie jeweils 5 s lang in Ethanollösungen verschiedener Verdünnungen, beginnend mit 70 %, gefolgt von 96 %, dann 100 % und schließlich in Xylol. Nehmen Sie die Objektträger aus dem Gestell und legen Sie sie auf ein Taschentuch mit Erythrozyten an der Oberseite, um überschüssige Flüssigkeit aufzunehmen.
- Geben Sie zwei oder drei Tropfen Eindeckmedium auf den Objektträger. Decken Sie die Folie mit einem Deckglas ab. Vermeiden Sie den Einschluss von Luftblasen, da dies die mikroskopische Auswertung behindert. Trocknen Sie die Proben mindestens über Nacht in einem Abzug.
- Durchführung mikroskopischer Auswertungen
- Zur Visualisierung und Bildgebung werden Objektträger in ein Durchlichtmikroskop mit mindestens 200-facher Vergrößerung gelegt. Stellen Sie sicher, dass das Mikroskop mit einer Kamera gekoppelt ist, um Bilder der gefärbten Erythrozyten aufzunehmen.
- Schalten Sie die Lichtquelle des Mikroskops ein. Schalten Sie die am Mikroskop angeschlossene Kamera ein und starten Sie die Mikroskopsteuerungssoftware. Verwenden Sie die Hellfeldmikroskopie, um den geeigneten Fokus zu bestimmen, indem Sie die Grob- und Feinfokuseinstellknöpfe drehen und die Fokusebene finden, auf der Erythrozyten sichtbar sind.
- Stellen Sie die Hintergrundwerte der Bilder auf 220 ± 5 Grauwerte ein, gemessen an drei zellenfreien Bereichen der Folie. Öffnen Sie dazu das erste Bild mit der frei verfügbaren Software ImageJ. Wählen Sie die Option Mittlere Grauwerte über das Panel Messungen festlegen. Verwenden Sie das ovale Symbol, um den Grauwert mit dem Befehl Messen zu messen. Liegen die Messwerte außerhalb des zulässigen Bereichs, passen Sie die Hintergrundbeleuchtung am Mikroskop entsprechend an. Wiederholen Sie diese Schritte, bis die Hintergrundwerte korrekt sind.
HINWEIS: Diese Hintergrundwerte sollten für alle aufgenommenen Bilder innerhalb des angegebenen Bereichs liegen. Andernfalls sind die Daten nicht vergleichbar. - Um den Grauwert der Erythrozyten zu analysieren, markieren Sie die Kante jedes Erythrozyten mit dem Auswahlwerkzeug Oval in der ImageJ-Software. Ermitteln Sie die Grauwerte einzelner RBCs mit dem Befehl Messen . Messen Sie die Grauwerte von mindestens 50 Erythrozyten aus dem Test und von mindestens 10 Erythrozyten aus dem Kontrollbereich.
HINWEIS: Die Gesamtzahl der analysierten Erythrozyten kann individuell angepasst werden. Je mehr Erythrozyten analysiert werden, desto aussagekräftiger ist das Ergebnis. Die Gesamtzahl der analysierten Erythrozyten sollte jedoch für jede Erkrankung, jeden Probanden usw. innerhalb eines bestimmten Experiments vergleichbar sein.- Vermeiden Sie die Analyse von Erythrozyten in der Nähe des Fettstifts, da die Verfärbung in diesem Bereich unvollständig sein kann. Vermeiden Sie die Analyse von überlappenden Erythrozyten, da dies die Färbeintensität beeinflusst. Verwenden Sie nur Erythrozyten, die von anderen Erythrozyten getrennt sind. Analysieren Sie mindestens fünf Bilder aus dem Test und mindestens zwei Bilder aus dem Kontrollbereich für die Bewertung von 50/10 Erythrozyten. Beziehen Sie Erythrozyten aus jedem Bild in die endgültige Analyse ein.
- Um die Färbeintensität zu berechnen, berechnen Sie die Endsignale wie folgt:
(Einzelkontrollbereich RBC - mittlerer Hintergrund des Testbereichs) - (Einzelkontrollbereich RBC - mittlerer Kontrollbereichshintergrund)

Abbildung 2: Darstellung des Fixierprozesses und der Blutausstrichentstehung. (Schema, das mit BioRender.com erstellt wurde.) Verdünnte Blutproben werden chemisch in Paraformaldehyd fixiert, dann zentrifugiert und mit phosphatgepufferter Kochsalzlösung gewaschen. Zum Schluss wird resuspendiertes Blut auf einen Objektträger geschmiert und durch Schweben über einer Bunsenbrennerflamme thermisch fixiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Zubereitungs- und Lagerbedingungen für Lösungen, die für die immunhistochemische Färbung erforderlich sind. Die Lösung kann vor dem Protokoll vorbereitet werden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Beschreibung von Antikörperlösungen zur sofortigen Anwendung. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 3: Komponenten und Protokoll zur Vorbereitung der DAB-Lösung für den sofortigen Einsatz. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
2. Fluoreszenzmarkierung von Erythrozytenproteinen
ANMERKUNG: Der folgende Abschnitt skizziert eine Anpassung des immunhistochemischen Protokolls, das mit dem Ziel entwickelt wurde, die Verwendung von Antikörpern mit fluoreszierenden Konjugaten zu ermöglichen (Abbildung 1).
Die Vorbereitung der Blutprobe für das Immunfluoreszenzprotokoll ist identisch mit der in Abschnitt 1 beschriebenen, so dass der folgende Abschnitt mit der Färbung der Proben beginnt.
- Immunfluoreszenz-Färbung
- Mit dem sekundären, fluoreszierenden konjugierten Antikörper (für das Volumen siehe Schritt 1.4.2) für 30 Minuten bei lichtgeschützter RT inkubieren, entweder mit einer speziell angefertigten Inkubationskammer oder Aluminiumfolie.
- Gießen Sie die sekundäre Antikörperlösung ab und waschen Sie die Proben 3x mit TBS. Lassen Sie die letzte Wäsche auf den Proben, um ein Austrocknen zu verhindern.
- Gießen Sie das TBS ab und nehmen Sie die Proben nacheinander aus dem Probenrack, um eine längere Lichteinwirkung zu vermeiden. Um die Proben zu dehydrieren, werden die Objektträger jeweils ~5 s lang verschiedenen Ethanollösungen ausgesetzt, beginnend mit 70 %, gefolgt von 90 % und schließlich 100 %. Belichten Sie die Objektträger 5 s lang mit Xylol/Xylol-Lösung.
- Bereiten Sie ein Deckglas mit zwei oder drei Tropfen Eindeckmedium vor und montieren Sie dann den Objektträger mit dem Deckglas. Sorgen Sie für eine gleichmäßige Verteilung des Eindeckmediums, indem Sie leichten Druck mit einer Pinzette oder einem ähnlichen, sterilen Metallinstrument ausüben. Beseitigen Sie Luftblasen, die sonst die Bildgebung stören könnten, mit demselben Instrument. Lassen Sie die Proben über Nacht an einem dunklen und trockenen Ort trocknen.
- Mikroskopische Auswertung
- Zur Visualisierung und Bildgebung legen Sie den Objektträger mit einer Gesamtvergrößerung von mindestens 400x auf den Mikroskoptisch. Schalten Sie die Mikroskoplichtquelle und die Leuchtstofflampe ein und stellen Sie sicher, dass die Leuchtstofflichtquelle auf maximale Intensität eingestellt ist.
- Schalten Sie die am Mikroskop angeschlossene Kamera ein und starten Sie die Mikroskopsteuerungssoftware. Verwenden Sie die Hellfeldmikroskopie, um den geeigneten Fokus zu bestimmen, indem Sie die Knöpfe für die Grob- und Feinfokuseinstellung drehen und die Fokusebene finden, auf der Erythrozyten sichtbar sind.
HINWEIS: Übermäßige Lichteinwirkung kann zu einem Photobleichen der fluoreszierenden Konjugate führen. Zu diesem Zweck kann ein Objektträger aus einem früheren Experiment verwendet werden, um die Lichteinwirkung der noch zu analysierenden Objektträger zu minimieren. - Bestimmen Sie die optimale Laserintensität, indem Sie Erythrozyten im Kontrollbereich inspizieren. Stellen Sie sicher, dass die Intensität hoch genug ist, dass die Erythrozyten sichtbar sind, ohne ein großes Hintergrundsignal zu erzeugen. Halten Sie die Intensität und Belichtungszeit zwischen Bereichen und Proben konsistent, um die Vergleichbarkeit zu gewährleisten.
- Erfassen Sie Hellfeld- und Fluoreszenzbilder in mindestens drei verschiedenen Bereichen des Testbereichs des Objektträgers, die nach dem Zufallsprinzip ausgewählt werden. Um einen Bereich auszuwählen, schwenken Sie mit den Bedienelementen des Mikroskoptisches von den Rändern des Objektträgers weg, die durch den Lipidstift markiert sind. Wählen Sie einen Bereich aus, der eine gleichmäßige Verteilung einer einzelnen RBC-Schicht aufweist.
- Stellen Sie die Belichtungszeit für fluoreszierende Bilder auf 1 s ein und nehmen Sie ein Bild mit den Softwaresteuerungen der Mikroskopsteuerungssoftware auf. Wechseln Sie in den Hellfeld-Modus, stellen Sie die Belichtungszeit auf "Auto" und nehmen Sie das entsprechende Hellfeldbild auf.
- Erfassen Sie Hellfeld- und Fluoreszenzbilder in mindestens zwei verschiedenen, zufällig ausgewählten Bereichen des Kontrollbereichs des Objektträgers, indem Sie Schritt 2.2.3 wiederholen.
- Speichern Sie die Bilder im .tif-Format, um die ursprünglichen Grauwerte der Pixel beizubehalten, Komprimierung zu verhindern und Metadaten der Erfassung zu speichern.
- Messen Sie die Grauwerte der erfassten Erythrozyten. Öffnen Sie ImageJ (oder FIJI21; siehe Zusatzdatei 1). Ermitteln Sie die Grauwerte der einzelnen Erythrozyten. Markieren Sie dazu jede einzelne Zelle mit dem Auswahlwerkzeug Oval und analysieren Sie sie mit dem Befehl Messen .
HINWEIS: Erythrozyten sollten nicht analysiert werden, wenn sie sich mit anderen Zellen überlappen, da dies das resultierende Signal verstärken kann. - Markieren Sie zwischen drei und fünf Bereiche, die frei von Erythrozyten sind, und bestimmen Sie die Grauwerte, um ein Maß für das Hintergrundsignal zu erhalten. Analysieren Sie mindestens 150 Erythrozyten aus mindestens drei unterschiedlichen Bildern für jeden Testbereich und mindestens 50 Erythrozyten aus mindestens zwei unterschiedlichen Bildern für jeden Kontrollbereich.
HINWEIS: Die Analyse weiterer Zellen/Bereiche kann empfohlen werden, um die Variabilität zu minimieren. Da die Erythrozytenpopulation von Natur aus heterogen ist, kann die Zell-Zell-Variabilität des Signals je nach interessiertem Protein signifikant sein. - Führen Sie eine alternative Datenanalyse der aufgenommenen Bilder mit einem Makrobefehl durch. Erstellen/installieren Sie einen Makrobefehl über die FIJI-Version von ImageJ für die automatische Auswahl, Hintergrundkorrektur und Grauwertanalyse eines bestimmten Bildes.
HINWEIS: Diese Routine verwendet automatische Schwellenwerte, um die in einem bestimmten Bild vorhandenen Zellen anhand einer Kopie des Originalbildes zu erkennen, wodurch eine Überlagerung erzeugt wird, die dem Originalbild auferlegt wird, um Grauwerte von fluoreszierenden Erythrozyten zu extrahieren. Das Makro wird als .ijm-Datei als Supplementary Coding File 1 abgelegt. - Öffnen Sie die Bilddatei im .tif-Format, bereit für die Analyse in FIJI. Öffnen Sie das Makro RBC fluorescence.ijm (Supplementary Coding File 1) und klicken Sie auf Ausführen.
HINWEIS: Das Makro ist für Bilder eingerichtet, die mit 600-facher Vergrößerung und einem großen Signal-Rausch-Verhältnis aufgenommen wurden. Die automatische Auswahl von Zellen sollte vom Prüfarzt überprüft werden.
- Berechnen Sie die Endsignale wie folgt:
(Prüfbereich RBC - Prüfbereichshintergrund) - (Regelbereich RBC - Kontrollbereich Hintergrund) für die manuelle Analyse;
(Testbereich - Kontrollbereich) für die automatisierte Analyse.
Ergebnisse
Das vorgestellte Protokoll, das Methoden beschreibt, die den Nachweis akuter Veränderungen in Erythrozytenproteinen erleichtern, wurde an einer bekannten mechanisch sensitiven Proteinveränderung getestet: der Phosphorylierung von Erythrozyten-NOS am Serin-1177-Rest. Vollblut wurde von gesunden Probanden gewonnen und anschließend in zwei separate Aliquots aufgeteilt. Eine gegebene Blutprobe wurde 300 s lang mechanischer Scherbelastung physiologischer Größenordnung (5 Pa) ausgesetzt, von der zuvor gezeigt wurde, dass ...
Diskussion
Neuere Literatur deutet stark darauf hin, dass das Erythrozyten-NOS-Protein von entscheidender Bedeutung für die Regulation der Verformbarkeit von Erythrozytenist 15,22,23, was wiederum ihre Passage durch enge Kapillaren erleichtert 24. Die Proteinaktivität hängt in hohem Maße von posttranslationalen Proteinmodifikationen ab, insbesondere von der Phosphorylierung bestimmter Reste18
Offenlegungen
Alle Autoren haben offengelegt, dass es keine Interessenkonflikte gibt.
Danksagungen
LK bedankt sich für die Unterstützung durch ein Stipendium des australischen Forschungsausbildungsprogramms.
Materialien
| Name | Company | Catalog Number | Comments |
| 3,3′-Diaminobenzidin -tetrahydrochloride Hydrate | Sigma/Merck | D5637 | DAB |
| Ammoniumchloride | Merck /Millipore | 101145 | NH4Cl |
| Centrifuge 5427 R | Eppendorf | 5409000010 | |
| Coverslips | VWR | 631-0147 | |
| di-sodium Hydrogen Phosphate Dihydrate | Merck /Millipore | 106580 | Na2HPO4. 2 H2O |
| Disposable transfer pipettes | VWR | 612-6803 | |
| Entellan | Merck /Millipore | 107961 | rapid mounting medium for microscopy |
| Ethanol denaturated using 1 % methyl ethyl ketone (MEK) | Hofmann | 642 | |
| Glucose-Oxidase | Sigma/Merck | G2133 | |
| Grease pencil | Dako | S 2002 | |
| Horse-radish peroxidase/ExtrAvidin−Peroxidase | Sigma/Merck | E-2886 | HRP |
| Hydrochloric acid | Merck /Millipore | 109057 | HCl |
| Hydrogen peroxide, 30% | Merck /Millipore | 107203 | H2O2 |
| ImageJ Software | Freeware | ||
| Laser-assisted optical rotational cell analyser (LORCA) | RR Mechatronics | Ektacytometer instrument used for shearing | |
| Methanol | Merck /Millipore | 106009 | |
| Microscope slides | VWR | 630-1985 | |
| Nickel(II)-sulfate Hexahydrate | Sigma/Merck | N4882 | NiSO4.6H2O |
| Normal Goat serum | Agilent/DAKO | X0907 | NGS |
| Paraformaldehyde | Merck /Millipore | 818715 | PFA |
| Pipettes Eppendorf Reference 2 | VWR | 613-5836/ 613-5839 | |
| Rabbit Anti-phospho eNOS Antibody (Ser1177) | Merck/Millipore | 07-428-I | Primary Antibody |
| Reaction tubes, 2ml | Eppendorf | 30120094 | |
| Secondary Antibody goat anti rabbit | Agilent/DAKO | E0432 | Secondary Antibody |
| Skim milk powder | Bio-Rad | 170-6404 | |
| Sodium chloride | Merck /Millipore | 106404 | NaCl |
| Sodium Dihydrogen Phosphate Monohydrate | Merck /Millipore | 106346 | NaH2PO4.H2O |
| Sodium hydroxide, 1 M | Merck /Millipore | 150706 | NaOH |
| Tris(hydroxymethyl)-aminomethane | Merck /Millipore | 108382 | Tris |
| Trypsin | Sigma/Merck | T7409 | |
| Tween20 | Merck /Millipore | 822184 | |
| Whatman Glas microfiber filter, quality GF/F | Merck /Millipore | WHA1825047 | |
| Xylol | VWR Chemicals | 2,89,73,465 | |
| ß-D-Glucose monohydrate | Merck /Millipore | 14431-43-7 |
Referenzen
- Cohen, R. M., et al. Red cell life span heterogeneity in hematologically normal people is sufficient to alter HbA1c. Blood. 112 (10), 4284-4291 (2008).
- Mock, D. M., et al. Red blood cell (RBC) survival determined in humans using RBCs labeled at multiple biotin densities. Transfusion. 51 (5), 1047-1057 (2011).
- Thiagarajan, P., Parker, C. J., Prchal, J. T. How do red blood cells die?. Frontiers in Physiology. 12, 655393 (2021).
- Moras, M., Lefevre, S. D., Ostuni, M. A. From erythroblasts to mature red blood cells: organelle clearance in mammals. Frontiers in Physiology. 8, 1076 (2017).
- Pretini, V., et al. Red blood cells: chasing interactions. Frontiers in Physiology. 10, 945 (2019).
- Cahalan, S. M., et al. Piezo1 links mechanical forces to red blood cell volume. eLife. 4, e07370 (2015).
- Kuck, L., Peart, J. N., Simmonds, M. J. Piezo1 regulates shear-dependent nitric oxide production in human erythrocytes. American Journal of Physiology. Heart and Circulatory Physiology. 323 (1), H24-H37 (2022).
- Kuck, L., Peart, J. N., Simmonds, M. J. Active modulation of human erythrocyte mechanics. American Journal of Physiology. Cell Physiology. 319 (2), C250-C257 (2020).
- Strader, M. B., et al. Post-translational modification as a response to cellular stress induced by hemoglobin oxidation in sickle cell disease. Scientific Reports. 10 (1), 14218 (2020).
- Pecankova, K., Majek, P., Cermak, J., Dyr, J. E. Posttranslational modifications of red blood cell ghost proteins as "signatures" for distinguishing between low- and high-risk myelodysplastic syndrome patients. Turkish Journal of Haematology. 34 (1), 111-113 (2017).
- Grau, M., et al. High red blood cell nitric oxide synthase activation is not associated with improved vascular function and red blood cell deformability in sickle cell anaemia. British Journal of Haematology. 168 (5), 728-736 (2015).
- Sae-Lee, W., et al. The protein organization of a red blood cell. Cell Reports. 40 (3), 111103 (2022).
- Suhr, F., et al. Moderate exercise promotes human RBC-NOS activity, NO production and deformability through Akt kinase pathway. PLoS One. 7 (9), e45982 (2012).
- Kuck, L., Grau, M., Bloch, W., Simmonds, M. J. Shear stress ameliorates superoxide impairment to erythrocyte deformability with concurrent nitric oxide synthase activation. Frontiers in Physiology. 10, 36 (2019).
- Grau, M., et al. RBC-NOS-dependent S-nitrosylation of cytoskeletal proteins improves RBC deformability. PLoS One. 8 (2), e56759 (2013).
- Simmonds, M. J., Detterich, J. A., Connes, P. Nitric oxide, vasodilation and the red blood cell. Biorheology. 51 (2-3), 121-134 (2014).
- Bor-Kucukatay, M., Wenby, R. B., Meiselman, H. J., Baskurt, O. K. Effects of nitric oxide on red blood cell deformability. American Journal of Physiology. Heart and Circulatory Physiology. 284 (5), H1577-H1584 (2003).
- Suhr, F., et al. Intensive exercise induces changes of endothelial nitric oxide synthase pattern in human erythrocytes. Nitric Oxide: Biology and Chemistry. 20 (2), 95-103 (2009).
- Grau, M., et al. Regulation of red blood cell deformability is independent of red blood cell-nitric oxide synthase under hypoxia. Clinical Hemorheology and Microcirculation. 63 (3), 199-215 (2016).
- Grau, M., Kuck, L., Dietz, T., Bloch, W., Simmonds, M. J. Sub-fractions of red blood cells respond differently to shear exposure following superoxide treatment. Biology. 10 (1), 47 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Ozüyaman, B., Grau, M., Kelm, M., Merx, M. W., Kleinbongard, P. RBC NOS: regulatory mechanisms and therapeutic aspects. Trends in Molecular Medicine. 14 (7), 314-322 (2008).
- Kleinbongard, P., et al. Red blood cells express a functional endothelial nitric oxide synthase. Blood. 107 (7), 2943-2951 (2006).
- McMahon, T. J. Red blood cell deformability, vasoactive mediators, and adhesion. Frontiers in Physiology. 10, 1417 (2019).
- Bizjak, D. A., Brinkmann, C., Bloch, W., Grau, M. Increase in red blood cell-nitric oxide synthase dependent nitric oxide production during red blood cell aging in health and disease: a study on age dependent changes of rheologic and enzymatic properties in red blood cells. PLoS One. 10 (4), 0125206 (2015).
- Di Pietro, N., et al. Nitric oxide synthetic pathway and cGMP levels are altered in red blood cells from end-stage renal disease patients. Molecular and Cellular Biochemistry. 417 (1-2), 155-167 (2016).
- Grau, M., et al. Even patients with mild COVID-19 symptoms after SARS-CoV-2 infection show prolonged altered red blood cell morphology and rheological parameters. Journal of Cellular and Molecular Medicine. 26 (10), 3022-3030 (2022).
- Mozar, A., et al. Red blood cell nitric oxide synthase modulates red blood cell deformability in sickle cell anemia. Clinical Hemorheology and Microcirculation. 64 (1), 47-53 (2016).
- Ulker, P., Gunduz, F., Meiselman, H. J., Baskurt, O. K. Nitric oxide generated by red blood cells following exposure to shear stress dilates isolated small mesenteric arteries under hypoxic conditions. Clinical Hemorheology and Microcirculation. 54 (4), 357-369 (2013).
- Nader, E., et al. Hydroxyurea therapy modulates sickle cell anemia red blood cell physiology: Impact on RBC deformability, oxidative stress, nitrite levels and nitric oxide synthase signalling pathway. Nitric Oxide: Biology and Chemistry. 81, 28-35 (2018).
- Fischer, U. M., Schindler, R., Brixius, K., Mehlhorn, U., Bloch, W. Extracorporeal circulation activates endothelial nitric oxide synthase in erythrocytes. The Annals of Thoracic Surgery. 84 (6), 2000-2003 (2007).
- Horobin, J. T., Sabapathy, S., Kuck, L., Simmonds, M. J. Shear stress and RBC-NOS Serine1177 Phosphorylation in humans: a dose response. Life. 11 (1), 36 (2021).
- Kuck, L., Grau, M., Simmonds, M. J. Recovery time course of erythrocyte deformability following exposure to shear is dependent upon conditioning shear stress. Biorheology. 54 (5-6), 141-152 (2018).
- Grau, M., et al. Effect of acute exercise on RBC deformability and RBC nitric oxide synthase signalling pathway in young sickle cell anaemia patients. Scientific Reports. 9 (1), 11813 (2019).
- Feelisch, M. . Methods in Nitric Oxide Research. , (1998).
- Cortese-Krott, M. M., et al. Human red blood cells at work: identification and visualization of erythrocytic eNOS activity in health and disease. Blood>. 120 (20), 4229-4237 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten