Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Détection basée sur l’immunomarquage d’altérations dynamiques dans les protéines des globules rouges
Dans cet article
Résumé
La capture des changements dynamiques dans l’activation protéique des globules rouges énucléés pose des défis méthodologiques, comme la préservation des changements dynamiques des stimuli aigus pour une évaluation ultérieure. Le protocole présenté décrit les techniques de préparation et de coloration des échantillons qui permettent la préservation et l’analyse des changements protéiques pertinents et la détection ultérieure.
Résumé
Le marquage des anticorps des protéines des globules rouges (GR) est une méthode semi-quantitative couramment utilisée pour détecter les changements dans la teneur globale en protéines ou les altérations aiguës des états d’activation des protéines. Il facilite l’évaluation des traitements des globules rouges, la caractérisation des différences dans certains états pathologiques et la description des cohérences cellulaires. La détection d’une altération aiguë de l’activation protéique (p. ex. par mécanotransduction) nécessite une préparation adéquate de l’échantillon pour préserver les modifications protéiques autrement temporaires. Le principe de base comprend l’immobilisation des sites de liaison cibles des protéines rouges souhaitées pour permettre la liaison initiale d’anticorps primaires spécifiques. L’échantillon est ensuite traité pour garantir des conditions optimales pour la liaison de l’anticorps secondaire à l’anticorps primaire correspondant. La sélection d’anticorps secondaires non fluorescents nécessite un traitement supplémentaire, y compris le couplage biotine-avidine et l’application de 3,3-diaminobenzidine-tétrachlorhydrate (DAB) pour développer la coloration, qui doit être contrôlée en temps réel au microscope afin d’arrêter l’oxydation, et donc l’intensité de la coloration, à temps. Pour la détection de l’intensité de la coloration, les images sont prises à l’aide d’un microscope optique standard. Dans une modification de ce protocole, un anticorps secondaire conjugué à la fluorescéine peut être appliqué à la place, ce qui présente l’avantage qu’aucune autre étape de développement n’est nécessaire. Cette procédure, cependant, nécessite un objectif de fluorescence attaché à un microscope pour la détection des colorations. Compte tenu de la nature semi-quantitative de ces méthodes, il est impératif de fournir plusieurs colorants de contrôle pour tenir compte des réactions d’anticorps non spécifiques et des signaux de fond. Ici, nous présentons à la fois les protocoles de coloration et les processus analytiques correspondants pour comparer et discuter des résultats et avantages respectifs des différentes techniques de coloration.
Introduction
Les globules rouges (GR) traversent le système cardiovasculaire pendant 70 à 140 jours, avec un âge moyen des globules rouges d’environ 115 jours 1,2. Les globules rouges sénescents ou endommagés sont éliminés de la circulation par érythrophagocytose, un processus d’élimination efficace entraîné par les macrophages3. La durée de vie prédéterminée de ces cellules est une conséquence de l’abandon des organites cellulaires, y compris le noyau, les mitochondries et les ribosomes, au cours de la différenciation et de la maturation4. Ainsi, les globules rouges circulants sont dépourvus de machinerie translationnelle, empêchant la synthèse de nouvelles protéines3. Il s’ensuit que les modifications dynamiques post-traductionnelles des protéines existantes représentent le seul mécanisme viable de régulation biochimique aiguë en réponse aux facteurs de stress extracellulaires et intracellulaires agissant sur les globules rouges5.
Les forces mécaniques semblent être les principaux indices extracellulaires qui provoquent l’activation ou la modulation des voies biochimiques dans les globules rouges. La découverte de la protéine mécanosensible, Piezo1, dans les membranes des globules rouges6 a inspiré plusieurs lignes de recherche sur la signalisation activée mécaniquement dans ces cellules7. Par exemple, des progrès récents ont montré que les propriétés physiques des globules rouges sont activement régulées par des changements aigus et dynamiques des protéines8, ce qui comprend la phosphorylation post-traductionnelle et l’ubiquitination9. Étant donné que ces modifications normales diffèrent dans certaines maladies 9,10,11, il semble être d’intérêt scientifique et clinique de déterminer l’état d’activation des protéines rouges, en particulier en relation avec les processus mécanobiologiques.
La détermination des changements aigus dans les états d’activation des protéines rouges pose certains défis méthodologiques. Par exemple, le stockage d’échantillons de globules rouges en vue d’une analyse ultérieure nécessite la préservation des protéines de globules rouges modifiées, car les modifications post-traductionnelles ne sont pas durables. De plus, les méthodes classiques de détection des protéines (par exemple, le transfert Western) sont notoirement difficiles à normaliser dans les globules rouges en raison de la faible abondance des protéines par rapport à l’hémoglobine, qui représente ~98% de la teneur en protéines de ces cellules12. Ainsi, la coloration par anticorps des globules rouges conservés chimiquement a été la méthode de choix pour étudier les modifications aiguës d’importantes protéines rouges, telles que l’isoforme spécifique des globules rouges de l’oxyde nitrique synthase (RBC-NOS)13,14. Il a été démontré que RBC-NOS produit de l’oxyde nitrique (NO) par voie enzymatique, ce qui semble indispensable pour les propriétés essentielles des globules rouges, y compris la déformabilité des globules rouges15,16,17. Les modifications post-traductionnelles de RBC-NOS régulent l’activité enzymatique catalytique, la phosphorylation du résidu de sérine 1177 étant décrite pour augmenter l’activité enzymatique, tandis que la phosphorylation des résidus sérine 114 ou thréonine 495 a été associée à une diminution de l’activité RBC-NOS18,19.
Collectivement, les modifications temporaires des protéines des globules rouges contribuent à une fonction cellulaire importante, et les protocoles normalisés qui permettent la détection de ces protéines modifiées sont d’une grande valeur. Ici, nous présentons deux protocoles distincts qui exploitent des anticorps spécifiques pour faciliter la détection de l’activation de la protéine RBC-NOS, et discutons des recommandations pour l’analyse et l’interprétation des données.
La performance des protocoles décrits a été évaluée en mesurant l’augmentation bien rapportée de la phosphorylation des globules rouges NOS au niveau du résidu de sérine 1177 en réponse à des forces mécaniques reflétant celles qui se produisent dans le système vasculaire humain (5 Pa).
Protocole
Les protocoles décrits ici sont conformes à la Déclaration d’Helsinki et ont été approuvés par les comités d’éthique de l’Université allemande des sports de Cologne (16/09/2013) et de l’Université Griffith (2019/808). Les volontaires ont été sélectionnés pour s’assurer de l’absence de pathologies pertinentes et ont fourni un consentement éclairé écrit.
1. Coloration des protéines des globules rouges à l’aide de protocoles d’immunohistochimie
REMARQUE : Une liste détaillée des produits chimiques et des matériaux requis est fournie dans le tableau des matières. Les sections suivantes décrivent la préparation des solutions requises, suivies d’une description détaillée du protocole d’immunohistochimie (Figure 1).
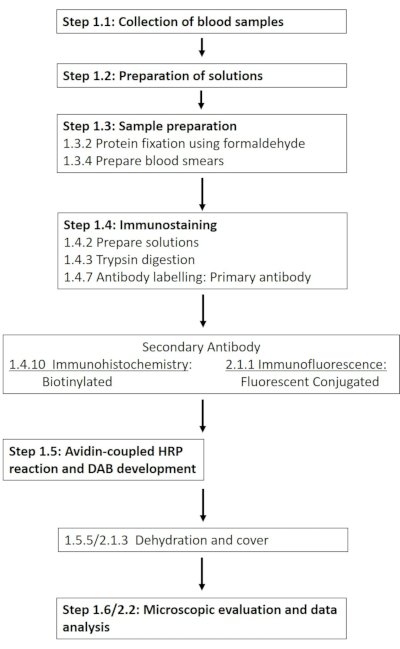
Figure 1 : Schéma des différentes étapes requises pour la coloration immunohistochimique et immunofluorescence des globules rouges au site de phosphorylation 1177. Un flux de travail typique des protocoles présentés allant de la préparation de la solution et du prélèvement sanguin à la détection et à la visualisation basées sur les anticorps est présenté. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Prélèvement d’un échantillon de sang
- Obtenir des échantillons de sang (10 ml de sang total) de sept hommes en bonne santé. Les volontaires étaient des individus en bonne santé apparemment exempts de maladies cardiovasculaires, hématologiques, neurologiques, endocriniennes ou métaboliques. Les volontaires étaient également non-fumeurs.
- Prélever du sang à l’aide d’une aiguille et d’une seringue stériles dans une veine proéminente de la région antécubitale de l’avant-bras et transférer immédiatement dans des tubes recouverts de l’un des anticoagulants suivants: héparine de sodium (pour l’immunohistochimie) ou acide éthylènediaminetétraacétique (EDTA; pour le marquage fluorescent).
- Exposer les échantillons de sang anticoagulé à des forces mécaniques contrôlées avec précision à l’aide d’un système de cisaillement de type Couette, tel que décrit précédemmentà la section 7,20.
- L’appareil de cisaillement comprend une coupelle rotative et un bob fixe, séparés par un espace de 300 μm. Transférez l’échantillon de sang dans l’espace, ce qui provoque la vitesse de rotation contrôlable avec précision de la coupelle pour exercer des forces mécaniques bien contrôlées sur l’échantillon. Les données représentatives présentées ici ont été produites en appliquant des forces mécaniques reflétant celles qui se produisent dans le système vasculaire humain (5 Pa) pendant des durées prolongées (300 s).
- Préparation des solutions nécessaires à l’immunomarquage
- Préparez les solutions comme indiqué dans le tableau 1. Les solutions peuvent être préparées avant d’effectuer la procédure d’immunomarquage et stockées à une température donnée jusqu’à ce qu’elles soient nécessaires.
REMARQUE: La durée de stockage peut varier entre les produits chimiques. Vérifiez la fiche signalétique.
- Préparez les solutions comme indiqué dans le tableau 1. Les solutions peuvent être préparées avant d’effectuer la procédure d’immunomarquage et stockées à une température donnée jusqu’à ce qu’elles soient nécessaires.
- Préparation des échantillons
- Traiter le sang total immédiatement après le retrait ou le traitement expérimental (le cas échéant; par exemple, stimulus mécanique dans nos échantillons; voir étape 1.1.3) pour pouvoir détecter les effets de courte durée, par exemple après l’application de contraintes de cisaillement, l’exercice, l’exposition à l’hypoxie de courte durée, etc.
- Effectuer la fixation des protéines rouges à l’aide de formaldéhyde comme suit : diluer le sang total dans un rapport de 1:2 dans une solution de paraformaldéhyde à 4 % pendant 20 minutes à température ambiante (RT). Centrifuger l’échantillon à 132 x g pendant 3 min à TA et retirer délicatement le surnageant par pipetage.
- Resuspendre la pastille de globule rouge dans deux volumes de solution saline tamponnée au phosphate (PBS) de 0,1 mol/L (dilution 1:3) et incuber pendant 5 min à TA. Répéter la centrifugation comme décrit ci-dessus et retirer le surnageant, qui doit être clair, par pipetage. Resuspendre la pastille de globule de globule de RBC dans un volume de 0,1 mol/L de PBS (dilution 1:2).
- Préparez les frottis sanguins comme suit : étiquetez une lame de microscope avec l’ID de l’échantillon, l’anticorps appliqué, etc. à l’aide d’un stylo résistant à l’alcool (p. ex. crayon). Ensuite, ajoutez 10 μL de la solution de globule rouge préparée juste au-dessus du champ de l’étiquette.
- Placer une deuxième lame sur l’échantillon à un angle d’environ 45° et disperser l’échantillon uniformément le long de la lame. Fixez la lame à chaud sur un brûleur Bunsen en faisant passer l’échantillon au-dessus du brûleur avec un mouvement constant pendant 5 à 7 s.
REMARQUE: Il est recommandé de sécher les échantillons à l’air avant la fixation thermique. Les échantillons peuvent être conservés à RT jusqu’à coloration (Figure 2).
- Coloration immunohistochimique
- Marquez deux zones sur chaque lame: une zone d’essai dans laquelle les globules rouges sont incubés avec les anticorps primaire et secondaire respectifs, et une zone de contrôle dans laquelle l’anticorps primaire est remplacé par une solution témoin. Cette zone sert de contrôle à l’intérieur du test pour déterminer le signal de fond des globules rouges.
- Préparer des solutions avant ou pendant la procédure, conformément au tableau 2. Un volume de 300 μL est nécessaire par zone d’essai et de 200 μL par zone témoin.
REMARQUE: Il est important de noter que ces procédures de coloration basées sur les anticorps produisent des données semi-quantitatives plutôt que quantitatives. Par conséquent, pour s’assurer que des conclusions significatives peuvent être tirées des données produites, des échantillons témoins appropriés (c.-à-d. pour fournir un point de référence pour les changements relatifs évalués dans l’activation des protéines) sont absolument nécessaires. - Pour la digestion de la trypsine, décongeler 0,1% de trypsine et équilibrer à TA. Marquez deux zones sur chaque lame à l’aide d’un crayon à graisse: une zone d’essai (2/3 de la lame) et une zone de contrôle (1/3 de la lame). Appliquez soigneusement une solution saline tamponnée au tris (TBS) à l’aide de pipettes de transfert pour laver les zones d’échantillonnage. Laissez le TBS reposer pendant 30 s, puis versez-le. Répétez l’étape de lavage.
REMARQUE : ajoutez les solutions suivantes à la zone de contrôle et à la zone de test, sauf indication contraire. Les zones doivent être suffisamment couvertes avec la solution respective. Appliquez les solutions à l’aide de pipettes de transfert jetables et changez de pipette après chaque solution. - Ajouter 0,1% de trypsine, couvrir les lames à l’aide d’une chambre d’incubation spécialement conçue avec un couvercle ou une feuille d’aluminium et incuber pendant 30 minutes à 37 °C dans un incubateur. Après l’incubation, arrêter la réaction enzymatique en ajoutant de l’eau du robinet. Versez la solution. Lavez les deux zones 3x avec TBS, comme décrit ci-dessus.
- Pour bloquer l’activation de la peroxydase, ajouter la solution de méthanol et incuber pendant 30 minutes à TA. Couvrez les lames pendant ce temps. Versez la solution.
NOTE: Pour la détection par immunofluorescence (section 2), cette étape peut être omise, étant donné que le blocage des peroxydases endogènes sert à prévenir le signal artificiel causé par la détection avec la peroxydase de raifort (HRP) et l’hydrate de 3,3′-diaminobenzidine (DAB). - Lavez les deux zones 3x avec TBS, comme décrit à l’étape 1.4.3. Ajouter la solution de lait écrémé à 3% et incuber pendant 30 min à TA pour bloquer. Versez la solution. Ne vous lavez pas après.
- Ajouter l’anticorps primaire (AB) uniquement dans la zone d’essai. Ajouter une solution de contrôle AB (voir Tableau 2) à la zone de contrôle. Incuber les échantillons à 4 °C pendant la nuit. Couvrez les lames pendant ce temps pour éviter le séchage.
REMARQUE : Évitez de transférer les solutions de la zone de test à la zone de contrôle, car cela influencerait les valeurs de contrôle. - Versez la solution d’anticorps. Lavez les deux zones 3x avec TBS, comme décrit à l’étape 1.4.3.
- Effectuez une étape de blocage pour empêcher la liaison non spécifique. Ajouter 3% de sérum de chèvre normal et incuber pendant 30 min à TA. Verser la solution.
- Ajouter la solution d’anticorps secondaires et incuber pendant 30 min à TA. Verser la solution d’anticorps. Lavez les zones 3x avec TBS, comme décrit à l’étape 1.4.3.
- Développement de la coloration et de la couverture
- Effectuer la réaction HRP couplée à l’avidine (voir dilution dans le tableau 2) en ajoutant une solution diluée d’avidine-peroxydase et en incubant pendant 30 min à TA. Verser la solution.
- Préparer le mélange DAB pour développer l’immunomarquage avant utilisation, conformément au tableau 3.
ATTENTION : Le DAB est dangereux. Considérez les mentions de danger H341 (soupçonnée de causer des anomalies génétiques) et H350 (peut causer le cancer). Tenez compte des conseils de prudence suivants : P201, P202, P280, P308+P313, P405 et P501. - Effectuer la coloration de contrôle DAB comme suit: recueillir la solution HRP de l’étape 1.5.1 à partir d’une lame et mélanger avec un petit volume (par exemple, 1 mL) de solution DAB préparée dans un tube à centrifuger séparé avant la coloration. Le mélange devrait développer une couleur brune / grise.
- Placez les lames sous un microscope (grossissement d’au moins 200x) et ajoutez une solution DAB aux deux zones. Surveillez la coloration des globules rouges en continu et arrêtez la coloration en retirant la solution DAB avec une pipette jetable avant que l’arrière-plan ne commence à se colorer. Pour la coloration à la sérine 1177 RBC-NOS, le temps d’incubation du DAB est d’environ 17 min.
- Effectuer la déshydratation de l’échantillon. Placer les lames dans une grille en verre et les immerger pendant 5 s chacune dans des solutions d’éthanol de différentes dilutions, en commençant par 70%, puis 96%, puis 100%, et enfin dans du xylol. Retirez les lames de la grille et placez-les sur un mouchoir, avec des globules rouges en haut pour absorber l’excès de liquide.
- Ajouter deux ou trois gouttes de support de montage tout au long de la lame. Couvrez la diapositive à l’aide d’un bordereau de couverture. Évitez l’inclusion de bulles d’air, car cela entrave l’évaluation microscopique. Sécher les échantillons au moins toute la nuit dans une hotte.
- Réalisation d’une évaluation microscopique
- Pour la visualisation et l’imagerie, placez les lames dans un microscope à lumière transmise avec un grossissement d’au moins 200x. Assurez-vous que le microscope est couplé à une caméra pour prendre des photos des globules rouges colorés.
- Allumez la source lumineuse du microscope. Allumez la caméra fixée au microscope et démarrez le logiciel de contrôle du microscope. Utilisez la microscopie à fond clair pour déterminer la mise au point appropriée en tournant les boutons de réglage grossier et fin de la mise au point et en trouvant le niveau de mise au point où les globules rouges sont visibles.
- Définissez les valeurs d’arrière-plan des images sur 220 ± 5 valeurs de gris, mesurées sur trois zones sans cellules de la diapositive. Pour ce faire, ouvrez la première image à l’aide du logiciel gratuit ImageJ. Sélectionnez l’option Valeurs moyennes de gris à l’aide du panneau Définir les mesures. Utilisez l’icône ovale pour mesurer la valeur de gris à l’aide de la commande Mesurer . Si les valeurs mesurées sont hors de portée, ajustez la lumière de fond au microscope en conséquence. Répétez ces étapes jusqu’à ce que les valeurs d’arrière-plan soient correctes.
REMARQUE: Ces valeurs d’arrière-plan doivent être dans la plage donnée pour toutes les images prises. Sinon, les données ne sont pas comparables. - Pour l’analyse de la valeur de gris RBC, marquez le bord de chaque RBC à l’aide de l’outil de sélection Ovale du logiciel ImageJ. Déterminez les valeurs de gris des RBC individuels à l’aide de la commande Mesurer . Mesurez les valeurs de gris d’un minimum de 50 globules rouges à partir du test et d’un minimum de 10 globules rouges de la zone de contrôle.
REMARQUE : Le nombre total de globules rouges analysés peut être ajusté individuellement. Plus il y a de globules rouges analysés, plus le résultat est significatif. Cependant, le nombre total de globules rouges analysés devrait être comparable pour chaque condition, sujet, etc. au sein d’une expérience donnée.- Évitez l’analyse des globules rouges près du crayon à graisse, car la coloration pourrait être incomplète dans cette zone. Évitez l’analyse des globules rouges qui se chevauchent, car cela affecte l’intensité de la coloration. N’utilisez que des globules rouges séparés des autres globules rouges. Analyser au moins cinq images du test et au moins deux images de la zone de contrôle pour l’évaluation des globules rouges 50/10. Inclure les globules rouges de chaque image dans l’analyse finale.
- Pour calculer l’intensité de coloration, calculez les signaux finaux comme suit :
(zone d’essai individuelle RBC - fond de la zone d’essai moyenne) - (zone de contrôle individuelle RBC - zone de contrôle moyenne arrière-plan)

Figure 2 : Représentation du processus de fixation et de la génération de frottis sanguins. (Schéma créé avec BioRender.com.) Les échantillons de sang dilués sont fixés chimiquement dans du paraformaldéhyde, puis centrifugés et lavés avec une solution saline tamponnée au phosphate. Enfin, le sang remis en suspension est étalé sur une lame de verre et fixé thermiquement en planant au-dessus d’une flamme de brûleur Bunsen. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Conditions de préparation et de stockage des solutions nécessaires à la coloration immunohistochimique. La solution peut être préparée avant le protocole. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 2 : Description des solutions d’anticorps pour utilisation immédiate. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 3 : Composants et protocole de préparation de la solution DAB pour une utilisation immédiate. Veuillez cliquer ici pour télécharger ce tableau.
2. Marquage fluorescent des protéines des globules rouges
REMARQUE : La section suivante décrit une adaptation du protocole immunohistochimique, développé dans le but de permettre l’utilisation d’anticorps avec des conjugués fluorescents (Figure 1).
La préparation des échantillons de sang pour le protocole d’immunofluorescence est identique à celle décrite à la section 1, de sorte que la section suivante commence par la coloration des échantillons.
- Coloration par immunofluorescence
- Incuber avec l’anticorps conjugué fluorescent secondaire (pour le volume, voir étape 1.4.2) pendant 30 min à TA à l’abri de la lumière, à l’aide d’une chambre d’incubation spécialement conçue ou d’une feuille d’aluminium.
- Versez la solution d’anticorps secondaire et lavez les échantillons 3x avec TBS. Laissez le lavage final sur les échantillons pour éviter qu’ils ne sèchent.
- Versez le TBS et retirez les échantillons du support à échantillons un à la fois pour éviter une exposition prolongée à la lumière. Pour déshydrater les échantillons, exposez les lames à différentes solutions d’éthanol pendant ~5 s chacune, en commençant par 70%, puis 90% et enfin 100%. Exposer les lames à une solution de xylol/xylène pendant 5 s.
- Préparez un bordereau de couverture avec deux ou trois gouttes de support de montage, puis montez la glissière avec le bordereau. Assurez une répartition uniforme du support de montage en appliquant une légère pression avec une pince à épiler ou un instrument métallique stérile similaire. Éliminez les bulles d’air, qui pourraient autrement interférer avec l’imagerie, avec le même instrument. Laissez les échantillons sécher toute la nuit dans un espace sombre et sec.
- Évaluation microscopique
- Pour la visualisation et l’imagerie, placez la lame sur la platine du microscope avec un grossissement total d’au moins 400x. Allumez la source lumineuse du microscope et la source de lumière fluorescente, en veillant à ce que la source de lumière fluorescente soit ajustée à une intensité maximale.
- Allumez la caméra fixée au microscope et démarrez le logiciel de contrôle du microscope. Utilisez la microscopie à fond clair pour déterminer la mise au point appropriée en tournant les boutons de réglage de la mise au point grossière et fine et en trouvant le niveau de mise au point où les globules rouges sont visibles.
REMARQUE: Une exposition excessive à la lumière peut provoquer un photoblanchiment des conjugués fluorescents. On peut utiliser une diapositive d’une expérience précédente à cette fin, pour minimiser l’exposition à la lumière des lames qui doivent encore être analysées. - Déterminez l’intensité laser optimale en inspectant les globules rouges dans la zone de contrôle. Assurez-vous que l’intensité est suffisamment élevée pour que les globules rouges soient visibles, tout en ne produisant pas un signal de fond important. Maintenir l’intensité et le temps d’exposition cohérents entre les zones et les échantillons pour assurer la comparabilité.
- Capturez des images en fond clair et fluorescentes dans au moins trois zones distinctes de la zone de test de la diapositive, sélectionnées au hasard. Pour sélectionner une zone, éloignez-vous des bords de la lame marqués par le stylo lipidique, à l’aide des commandes de l’étage du microscope. Sélectionnez une zone qui présente une distribution uniforme d’une couche de globules rouges singulière.
- Réglez le temps d’exposition sur 1 s pour les images fluorescentes et capturez une image à l’aide des commandes logicielles du logiciel de contrôle du microscope. Passez en mode fond clair, réglez le temps d’exposition sur 'Auto' et capturez l’image en fond clair correspondante.
- Capturez des images en fond clair et fluorescentes dans au moins deux zones distinctes sélectionnées au hasard de la zone de contrôle de la diapositive en répétant l’étape 2.2.3.
- Enregistrez les images dans .tif format pour préserver les valeurs de gris d’origine des pixels, éviter la compression et transporter les métadonnées de l’acquisition.
- Mesurez les valeurs de gris des globules rouges capturés. Ouvrez ImageJ (ou FIJI21 ; voir Fichier supplémentaire 1). Déterminez les valeurs de gris de chaque GRE. À cette fin, marquez chaque cellule individuelle avec l’outil de sélection ovale et analysez à l’aide de la commande Mesurer .
REMARQUE: Les globules rouges ne doivent pas être analysés s’ils se chevauchent avec d’autres cellules, car cela peut augmenter le signal résultant. - Mettez en surbrillance entre trois et cinq zones exemptes de globules rouges et déterminez les valeurs de gris pour fournir une mesure du signal de fond. Analyser au moins 150 globules rouges provenant d’au moins trois images distinctes pour chaque zone d’essai, et au moins 50 globules rouges provenant d’au moins deux images distinctes pour chaque zone de contrôle.
REMARQUE : L’analyse d’un plus grand nombre de cellules/zones peut être conseillée pour minimiser la variabilité. Étant donné que la population de globules rouges est intrinsèquement hétérogène, la variabilité cellule-cellule du signal peut être importante, selon la protéine d’intérêt. - Effectuez une analyse alternative des données des images capturées à l’aide de la commande macro. Créez/installez une commande macro via la version FIJI d’ImageJ pour la sélection automatisée, la correction de l’arrière-plan et l’analyse des valeurs de gris d’une image donnée.
REMARQUE: Cette routine utilise un seuillage automatisé pour détecter les cellules présentes dans une image donnée à l’aide d’une copie de l’image originale, produisant une superposition qui est imposée à l’image originale pour extraire les valeurs de gris des GR fluorescents. La macro est déposée sous la forme d’un fichier .ijm, en tant que fichier de codage supplémentaire 1. - Ouvrez le fichier image dans .tif format prêt pour l’analyse dans FIJI. Ouvrez la macro RBC fluorescence.ijm (Supplementary Coding File 1) et cliquez sur Exécuter.
REMARQUE: La macro est configurée pour les images obtenues avec un grossissement de 600x et un rapport signal sur bruit élevé. La sélection automatique des cellules doit être examinée par l’investigateur.
- Calculez les signaux finaux comme suit :
(zone d’essai RBC - contexte de la zone d’essai) - (zone de contrôle RBC - contexte de la zone de contrôle) pour l’analyse manuelle;
(zone d’essai - zone de contrôle) pour l’analyse automatisée.
Résultats
Le protocole présenté, décrivant les méthodes qui facilitent la détection des altérations aiguës des protéines rouges, a été testé sur une altération protéique bien connue et sensible mécaniquement : la phosphorylation des globules rouges-NOS au niveau du résidu de sérine 1177. Le sang total a été obtenu à partir de volontaires sains et ensuite divisé en deux aliquotes distinctes. Un échantillon de sang donné a été exposé à une contrainte de cisaillement mécanique d’une ampleur physiologique ...
Discussion
La littérature récente suggère fortement que la protéine RBC-NOS est d’une importance cruciale pour la régulation de la déformabilité des globules rouges 15,22,23, ce qui facilite leur passage à travers les capillaires étroits 24. L’activité protéique dépend fortement des modifications protéiques post-traductionnelles, en particulier de la phosphorylation de certains résidus
Déclarations de divulgation
Tous les auteurs ont révélé qu’il n’y avait pas de conflits d’intérêts.
Remerciements
LK reconnaît le soutien d’une bourse du programme de formation à la recherche du gouvernement australien.
matériels
| Name | Company | Catalog Number | Comments |
| 3,3′-Diaminobenzidin -tetrahydrochloride Hydrate | Sigma/Merck | D5637 | DAB |
| Ammoniumchloride | Merck /Millipore | 101145 | NH4Cl |
| Centrifuge 5427 R | Eppendorf | 5409000010 | |
| Coverslips | VWR | 631-0147 | |
| di-sodium Hydrogen Phosphate Dihydrate | Merck /Millipore | 106580 | Na2HPO4. 2 H2O |
| Disposable transfer pipettes | VWR | 612-6803 | |
| Entellan | Merck /Millipore | 107961 | rapid mounting medium for microscopy |
| Ethanol denaturated using 1 % methyl ethyl ketone (MEK) | Hofmann | 642 | |
| Glucose-Oxidase | Sigma/Merck | G2133 | |
| Grease pencil | Dako | S 2002 | |
| Horse-radish peroxidase/ExtrAvidin−Peroxidase | Sigma/Merck | E-2886 | HRP |
| Hydrochloric acid | Merck /Millipore | 109057 | HCl |
| Hydrogen peroxide, 30% | Merck /Millipore | 107203 | H2O2 |
| ImageJ Software | Freeware | ||
| Laser-assisted optical rotational cell analyser (LORCA) | RR Mechatronics | Ektacytometer instrument used for shearing | |
| Methanol | Merck /Millipore | 106009 | |
| Microscope slides | VWR | 630-1985 | |
| Nickel(II)-sulfate Hexahydrate | Sigma/Merck | N4882 | NiSO4.6H2O |
| Normal Goat serum | Agilent/DAKO | X0907 | NGS |
| Paraformaldehyde | Merck /Millipore | 818715 | PFA |
| Pipettes Eppendorf Reference 2 | VWR | 613-5836/ 613-5839 | |
| Rabbit Anti-phospho eNOS Antibody (Ser1177) | Merck/Millipore | 07-428-I | Primary Antibody |
| Reaction tubes, 2ml | Eppendorf | 30120094 | |
| Secondary Antibody goat anti rabbit | Agilent/DAKO | E0432 | Secondary Antibody |
| Skim milk powder | Bio-Rad | 170-6404 | |
| Sodium chloride | Merck /Millipore | 106404 | NaCl |
| Sodium Dihydrogen Phosphate Monohydrate | Merck /Millipore | 106346 | NaH2PO4.H2O |
| Sodium hydroxide, 1 M | Merck /Millipore | 150706 | NaOH |
| Tris(hydroxymethyl)-aminomethane | Merck /Millipore | 108382 | Tris |
| Trypsin | Sigma/Merck | T7409 | |
| Tween20 | Merck /Millipore | 822184 | |
| Whatman Glas microfiber filter, quality GF/F | Merck /Millipore | WHA1825047 | |
| Xylol | VWR Chemicals | 2,89,73,465 | |
| ß-D-Glucose monohydrate | Merck /Millipore | 14431-43-7 |
Références
- Cohen, R. M., et al. Red cell life span heterogeneity in hematologically normal people is sufficient to alter HbA1c. Blood. 112 (10), 4284-4291 (2008).
- Mock, D. M., et al. Red blood cell (RBC) survival determined in humans using RBCs labeled at multiple biotin densities. Transfusion. 51 (5), 1047-1057 (2011).
- Thiagarajan, P., Parker, C. J., Prchal, J. T. How do red blood cells die?. Frontiers in Physiology. 12, 655393 (2021).
- Moras, M., Lefevre, S. D., Ostuni, M. A. From erythroblasts to mature red blood cells: organelle clearance in mammals. Frontiers in Physiology. 8, 1076 (2017).
- Pretini, V., et al. Red blood cells: chasing interactions. Frontiers in Physiology. 10, 945 (2019).
- Cahalan, S. M., et al. Piezo1 links mechanical forces to red blood cell volume. eLife. 4, e07370 (2015).
- Kuck, L., Peart, J. N., Simmonds, M. J. Piezo1 regulates shear-dependent nitric oxide production in human erythrocytes. American Journal of Physiology. Heart and Circulatory Physiology. 323 (1), H24-H37 (2022).
- Kuck, L., Peart, J. N., Simmonds, M. J. Active modulation of human erythrocyte mechanics. American Journal of Physiology. Cell Physiology. 319 (2), C250-C257 (2020).
- Strader, M. B., et al. Post-translational modification as a response to cellular stress induced by hemoglobin oxidation in sickle cell disease. Scientific Reports. 10 (1), 14218 (2020).
- Pecankova, K., Majek, P., Cermak, J., Dyr, J. E. Posttranslational modifications of red blood cell ghost proteins as "signatures" for distinguishing between low- and high-risk myelodysplastic syndrome patients. Turkish Journal of Haematology. 34 (1), 111-113 (2017).
- Grau, M., et al. High red blood cell nitric oxide synthase activation is not associated with improved vascular function and red blood cell deformability in sickle cell anaemia. British Journal of Haematology. 168 (5), 728-736 (2015).
- Sae-Lee, W., et al. The protein organization of a red blood cell. Cell Reports. 40 (3), 111103 (2022).
- Suhr, F., et al. Moderate exercise promotes human RBC-NOS activity, NO production and deformability through Akt kinase pathway. PLoS One. 7 (9), e45982 (2012).
- Kuck, L., Grau, M., Bloch, W., Simmonds, M. J. Shear stress ameliorates superoxide impairment to erythrocyte deformability with concurrent nitric oxide synthase activation. Frontiers in Physiology. 10, 36 (2019).
- Grau, M., et al. RBC-NOS-dependent S-nitrosylation of cytoskeletal proteins improves RBC deformability. PLoS One. 8 (2), e56759 (2013).
- Simmonds, M. J., Detterich, J. A., Connes, P. Nitric oxide, vasodilation and the red blood cell. Biorheology. 51 (2-3), 121-134 (2014).
- Bor-Kucukatay, M., Wenby, R. B., Meiselman, H. J., Baskurt, O. K. Effects of nitric oxide on red blood cell deformability. American Journal of Physiology. Heart and Circulatory Physiology. 284 (5), H1577-H1584 (2003).
- Suhr, F., et al. Intensive exercise induces changes of endothelial nitric oxide synthase pattern in human erythrocytes. Nitric Oxide: Biology and Chemistry. 20 (2), 95-103 (2009).
- Grau, M., et al. Regulation of red blood cell deformability is independent of red blood cell-nitric oxide synthase under hypoxia. Clinical Hemorheology and Microcirculation. 63 (3), 199-215 (2016).
- Grau, M., Kuck, L., Dietz, T., Bloch, W., Simmonds, M. J. Sub-fractions of red blood cells respond differently to shear exposure following superoxide treatment. Biology. 10 (1), 47 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Ozüyaman, B., Grau, M., Kelm, M., Merx, M. W., Kleinbongard, P. RBC NOS: regulatory mechanisms and therapeutic aspects. Trends in Molecular Medicine. 14 (7), 314-322 (2008).
- Kleinbongard, P., et al. Red blood cells express a functional endothelial nitric oxide synthase. Blood. 107 (7), 2943-2951 (2006).
- McMahon, T. J. Red blood cell deformability, vasoactive mediators, and adhesion. Frontiers in Physiology. 10, 1417 (2019).
- Bizjak, D. A., Brinkmann, C., Bloch, W., Grau, M. Increase in red blood cell-nitric oxide synthase dependent nitric oxide production during red blood cell aging in health and disease: a study on age dependent changes of rheologic and enzymatic properties in red blood cells. PLoS One. 10 (4), 0125206 (2015).
- Di Pietro, N., et al. Nitric oxide synthetic pathway and cGMP levels are altered in red blood cells from end-stage renal disease patients. Molecular and Cellular Biochemistry. 417 (1-2), 155-167 (2016).
- Grau, M., et al. Even patients with mild COVID-19 symptoms after SARS-CoV-2 infection show prolonged altered red blood cell morphology and rheological parameters. Journal of Cellular and Molecular Medicine. 26 (10), 3022-3030 (2022).
- Mozar, A., et al. Red blood cell nitric oxide synthase modulates red blood cell deformability in sickle cell anemia. Clinical Hemorheology and Microcirculation. 64 (1), 47-53 (2016).
- Ulker, P., Gunduz, F., Meiselman, H. J., Baskurt, O. K. Nitric oxide generated by red blood cells following exposure to shear stress dilates isolated small mesenteric arteries under hypoxic conditions. Clinical Hemorheology and Microcirculation. 54 (4), 357-369 (2013).
- Nader, E., et al. Hydroxyurea therapy modulates sickle cell anemia red blood cell physiology: Impact on RBC deformability, oxidative stress, nitrite levels and nitric oxide synthase signalling pathway. Nitric Oxide: Biology and Chemistry. 81, 28-35 (2018).
- Fischer, U. M., Schindler, R., Brixius, K., Mehlhorn, U., Bloch, W. Extracorporeal circulation activates endothelial nitric oxide synthase in erythrocytes. The Annals of Thoracic Surgery. 84 (6), 2000-2003 (2007).
- Horobin, J. T., Sabapathy, S., Kuck, L., Simmonds, M. J. Shear stress and RBC-NOS Serine1177 Phosphorylation in humans: a dose response. Life. 11 (1), 36 (2021).
- Kuck, L., Grau, M., Simmonds, M. J. Recovery time course of erythrocyte deformability following exposure to shear is dependent upon conditioning shear stress. Biorheology. 54 (5-6), 141-152 (2018).
- Grau, M., et al. Effect of acute exercise on RBC deformability and RBC nitric oxide synthase signalling pathway in young sickle cell anaemia patients. Scientific Reports. 9 (1), 11813 (2019).
- Feelisch, M. . Methods in Nitric Oxide Research. , (1998).
- Cortese-Krott, M. M., et al. Human red blood cells at work: identification and visualization of erythrocytic eNOS activity in health and disease. Blood>. 120 (20), 4229-4237 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon