È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Rilevazione basata sull'immunocolorazione di alterazioni dinamiche nelle proteine dei globuli rossi
In questo articolo
Riepilogo
Catturare i cambiamenti dinamici nell'attivazione proteica dei globuli rossi enucleati pone sfide metodologiche, come la conservazione dei cambiamenti dinamici agli stimoli acuti per una valutazione successiva. Il protocollo presentato descrive la preparazione del campione e le tecniche di colorazione che consentono la conservazione e l'analisi dei cambiamenti proteici rilevanti e la successiva rilevazione.
Abstract
La marcatura anticorpale delle proteine dei globuli rossi (RBC) è un metodo semi-quantitativo comunemente usato per rilevare cambiamenti nel contenuto proteico complessivo o alterazioni acute negli stati di attivazione delle proteine. Facilita la valutazione dei trattamenti con globuli rossi, la caratterizzazione delle differenze in alcuni stati patologici e la descrizione delle coerenze cellulari. Il rilevamento di un'attivazione proteica acutamente alterata (ad esempio, attraverso la meccanotrasduzione) richiede un'adeguata preparazione del campione per preservare modificazioni proteiche altrimenti temporanee. Il principio di base include l'immobilizzazione dei siti di legame bersaglio delle proteine RBC desiderate per consentire il legame iniziale di specifici anticorpi primari. Il campione viene ulteriormente elaborato per garantire condizioni ottimali per il legame dell'anticorpo secondario all'anticorpo primario corrispondente. La selezione di anticorpi secondari non fluorescenti richiede un trattamento aggiuntivo, compreso l'accoppiamento biotina-avidina e l'applicazione di 3,3-diaminobenzidina-tetracloridrato (DAB) per sviluppare la colorazione, che deve essere controllata in tempo reale al microscopio per fermare l'ossidazione, e quindi l'intensità della colorazione, in tempo. Per il rilevamento dell'intensità della colorazione, le immagini vengono scattate utilizzando un microscopio ottico standard. In una modifica di questo protocollo, può essere applicato invece un anticorpo secondario coniugato con fluoresceina, che ha il vantaggio che non è necessaria alcuna ulteriore fase di sviluppo. Questa procedura, tuttavia, richiede un obiettivo di fluorescenza collegato a un microscopio per il rilevamento della colorazione. Data la natura semi-quantitativa di questi metodi, è imperativo fornire diverse colorazioni di controllo per tenere conto delle reazioni anticorpali non specifiche e dei segnali di fondo. Qui, presentiamo sia i protocolli di colorazione che i corrispondenti processi analitici per confrontare e discutere i rispettivi risultati e vantaggi delle diverse tecniche di colorazione.
Introduzione
I globuli rossi (RBC) attraversano il sistema cardiovascolare per 70-140 giorni, con un'età media dei globuli rossi di circa 115 giorni 1,2. I globuli rossi senescenti o danneggiati vengono rimossi dalla circolazione mediante eritrofagocitosi, un efficiente processo di compensazione guidato dai macrofagi3. La durata della vita predeterminata di queste cellule è una conseguenza della resa degli organelli cellulari, incluso il nucleo, i mitocondri e i ribosomi, durante la differenziazione e la maturazione4. Pertanto, i globuli rossi circolanti sono privi di un meccanismo traslazionale, precludendo la sintesi di nuove proteine3. Ne consegue che le modificazioni dinamiche post-traduzionali delle proteine esistenti rappresentano l'unico meccanismo praticabile di regolazione biochimica acuta in risposta a fattori di stress extracellulari e intracellulari che agiscono sui globuli rossi5.
Le forze meccaniche sembrano essere i principali segnali extracellulari che causano l'attivazione o la modulazione dei percorsi biochimici all'interno dei globuli rossi. La scoperta della proteina meccanosensibile, Piezo1, nelle membrane RBC6 ha ispirato diverse linee di ricerca che studiano la segnalazione attivata meccanicamente in queste cellule7. Ad esempio, recenti progressi hanno dimostrato che le proprietà fisiche dei globuli rossi sono attivamente regolate da cambiamenti acuti e dinamici delle proteine8, che includono la fosforilazione post-traduzionale e l'ubiquitinazione9. Poiché queste normali modificazioni differiscono in alcune malattie 9,10,11, sembra essere di interesse scientifico e clinico determinare lo stato di attivazione delle proteine RBC, in particolare in relazione ai processi meccanobiologici.
La determinazione di cambiamenti acuti negli stati di attivazione della proteina RBC pone alcune sfide metodologiche. Ad esempio, la conservazione dei campioni di RBC per analisi successive richiede la conservazione delle proteine RBC modificate, poiché le modifiche post-traduzionali non sono durevoli. Inoltre, i metodi classici di rilevamento delle proteine (ad esempio, western blotting) sono notoriamente difficili da standardizzare nei globuli rossi a causa della bassa abbondanza di proteine rispetto all'emoglobina, che rappresenta ~ 98% del contenuto proteico in queste cellule12. Pertanto, la colorazione basata su anticorpi di globuli rossi conservati chimicamente è stato il metodo di scelta quando si studiano le modificazioni acute di importanti proteine RBC, come l'isoforma specifica dei globuli rossi dell'ossido nitrico sintasi (RBC-NOS)13,14. È stato dimostrato che RBC-NOS produce enzimaticamente ossido nitrico (NO), che sembra indispensabile per le proprietà essenziali dei globuli rossi, inclusa la deformabilità dei globuli rossi15,16,17. Le modificazioni post-traduzionali di RBC-NOS regolano l'attività enzimatica catalitica, con la fosforilazione del residuo di serina 1177 descritta per aumentare l'attività enzimatica, mentre la fosforilazione dei residui serina 114 o treonina 495 è stata collegata a una diminuzione dell'attività RBC-NOS18,19.
Collettivamente, le modifiche temporanee delle proteine RBC contribuiscono a importanti funzioni cellulari e i protocolli standardizzati che consentono il rilevamento di queste proteine modificate sono di alto valore. Qui, presentiamo due protocolli distinti che sfruttano anticorpi specifici per facilitare la rilevazione dell'attivazione della proteina RBC-NOS e discutiamo le raccomandazioni per l'analisi e l'interpretazione dei dati.
Le prestazioni dei protocolli descritti sono state valutate misurando l'aumento ben riportato della fosforilazione di RBC-NOS al residuo di serina 1177 in risposta a forze meccaniche riflettenti quelle che si verificano all'interno del sistema vascolaristico umano (5 Pa).
Protocollo
I protocolli qui descritti sono in linea con la Dichiarazione di Helsinki e sono stati approvati dai comitati etici dell'Università tedesca dello sport di Colonia (16/09/2013) e dell'Università Griffith (2019/808). I volontari sono stati sottoposti a screening per garantire l'assenza di patologie rilevanti e hanno fornito il consenso informato scritto.
1. Colorazione delle proteine RBC utilizzando protocolli immunoistochimici
NOTA: un elenco dettagliato delle sostanze chimiche e dei materiali richiesti è fornito nella tabella dei materiali. Le sezioni seguenti descrivono la preparazione delle soluzioni richieste, seguite da una descrizione dettagliata del protocollo immunoistochimico (Figura 1).
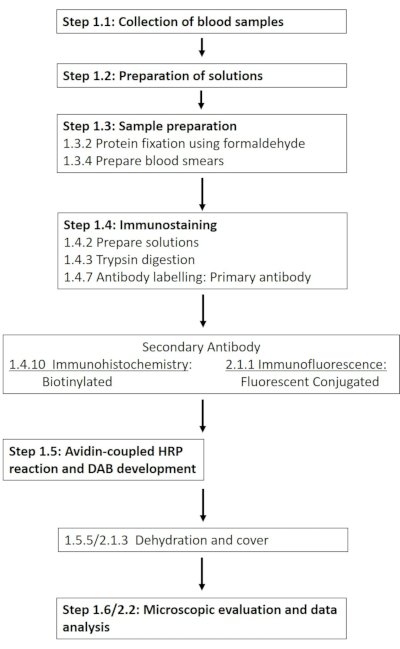
Figura 1: Schema delle singole fasi necessarie per la colorazione immunoistochimica e immunofluorescenza di RBC-NOS nel sito di fosforilazione 1177. Viene presentato un flusso di lavoro tipico dei protocolli presentati che si estende dalla preparazione della soluzione e dal prelievo di sangue al rilevamento e alla visualizzazione basati sugli anticorpi. Fare clic qui per visualizzare una versione ingrandita di questa figura.
- Raccolta del campione di sangue
- Ottenere campioni di sangue (10 ml di sangue intero) da sette maschi sani. I volontari erano individui sani privi di malattie cardiovascolari, ematologiche, neurologiche, endocrine o metaboliche. I volontari erano anche non fumatori.
- Raccogliere il sangue usando un ago sterile e una siringa da una vena prominente nella regione antecubitale dell'avambraccio e trasferirlo immediatamente in tubi rivestiti con uno dei seguenti anticoagulanti: eparina di sodio (per immunoistochimica) o acido etilendiamminotetraacetico (EDTA; per l'etichettatura fluorescente).
- Esporre i campioni di sangue anticoagulati a forze meccaniche controllate con precisione utilizzando un sistema di cesoiatura tipo Couette, comedescritto prima 7,20.
- L'apparato di taglio comprende una coppa girevole e un bob stazionario, separati da uno spazio di 300 μm. Trasferire il campione di sangue nello spazio, il che fa sì che la velocità di rotazione controllabile con precisione della tazza eserciti forze meccaniche ben controllate sul campione. I dati rappresentativi qui presentati sono stati prodotti applicando forze meccaniche che riflettono quelle che si verificano all'interno del sistema vascolare umano (5 Pa) per durate prolungate (300 s).
- Preparazione delle soluzioni necessarie per l'immunocolorazione
- Preparare le soluzioni come nella Tabella 1. Le soluzioni possono essere preparate prima di eseguire la procedura di immunocolorazione e conservate a una determinata temperatura fino al momento del bisogno.
NOTA: la durata di conservazione può variare tra le sostanze chimiche. Controllare la scheda di sicurezza dei materiali.
- Preparare le soluzioni come nella Tabella 1. Le soluzioni possono essere preparate prima di eseguire la procedura di immunocolorazione e conservate a una determinata temperatura fino al momento del bisogno.
- Preparazione del campione
- Processare il sangue intero immediatamente dopo la sospensione o il trattamento sperimentale (ove applicabile; ad esempio, stimolo meccanico nei nostri campioni; vedere il punto 1.1.3) per essere in grado di rilevare effetti di breve durata, ad esempio dopo l'applicazione di stress da taglio, l'esercizio fisico, l'esposizione all'ipossia di breve durata, ecc.
- Eseguire la fissazione della proteina RBC usando formaldeide come segue: diluire il sangue intero in un rapporto di 1: 2 in soluzione di paraformaldeide al 4% per 20 minuti a temperatura ambiente (RT). Centrifugare il campione a 132 x g per 3 minuti a RT e rimuovere con attenzione il surnatante mediante pipettaggio.
- Risospendere il pellet RBC in due volumi di soluzione salina tamponata fosfato 0,1 mol/L (diluizione 1:3) e incubare per 5 minuti a RT. Ripetere la centrifugazione come descritto sopra e rimuovere il surnatante, che dovrebbe essere chiaro, mediante pipettaggio. Risospendere il pellet RBC in un volume di 0,1 mol/L PBS (diluizione 1:2).
- Preparare strisci di sangue come segue: etichettare un vetrino da microscopio con l'ID del campione, l'anticorpo applicato, ecc. utilizzando una penna resistente all'alcool (ad esempio, matita). Quindi, aggiungere 10 μL della soluzione RBC preparata proprio sopra il campo dell'etichetta.
- Posizionare un secondo vetrino sul campione con un angolo di circa 45° e disperdere uniformemente il campione lungo il vetrino. Fissare a caldo la slitta su un bruciatore Bunsen facendo passare il campione sopra il bruciatore con movimento costante per 5-7 s.
NOTA: Si consiglia di asciugare all'aria i campioni prima del fissaggio a caldo. I campioni possono essere conservati in RT fino alla colorazione (Figura 2).
- Colorazione immunoistochimica
- Contrassegnare due aree su ciascun vetrino: un'area di test in cui i globuli rossi sono incubati con il rispettivo anticorpo primario e secondario e un'area di controllo in cui l'anticorpo primario viene sostituito da una soluzione di controllo. Quest'area funge da controllo interno del saggio per determinare il segnale di fondo dei globuli rossi.
- Preparare le soluzioni prima o durante la procedura, come da tabella 2. È necessario un volume di 300 μL per area di prova e 200 μL per area di controllo.
NOTA: È importante notare che queste procedure di colorazione basate su anticorpi producono dati semi-quantitativi, piuttosto che quantitativi. Pertanto, per garantire che si possano trarre conclusioni significative dai dati prodotti, sono assolutamente necessari campioni di controllo appropriati (cioè per fornire un punto di riferimento per i cambiamenti relativi valutati nell'attivazione delle proteine). - Per la digestione della tripsina, scongelare lo 0,1% di tripsina ed equilibrare a RT. Segnare due aree su ciascun vetrino usando una matita grassa: un'area di prova (2/3 del vetrino) e un'area di controllo (1/3 del vetrino). Applicare con cautela la soluzione salina tris-buffered (TBS) utilizzando pipette transfer per lavare le aree del campione. Lasciare riposare il TBS per 30 secondi e poi versarlo. Ripetere la fase di lavaggio.
NOTA: aggiungere le seguenti soluzioni sia al controllo che all'area di test, se non diversamente descritto. Le aree devono essere sufficientemente coperte con la rispettiva soluzione. Applicare le soluzioni utilizzando pipette transfer monouso e cambiare pipette dopo ogni soluzione. - Aggiungere tripsina allo 0,1%, coprire i vetrini utilizzando una camera di incubazione appositamente costruita con coperchio o foglio di alluminio e incubare per 30 minuti a 37 °C in un'incubatrice. Dopo l'incubazione, interrompere la reazione enzimatica aggiungendo acqua di rubinetto. Versare la soluzione. Lavare entrambe le aree 3 volte con TBS, come descritto sopra.
- Per bloccare l'attivazione della perossidasi, aggiungere una soluzione di metanolo e incubare per 30 minuti su RT. Coprire le diapositive durante quel periodo. Versare la soluzione.
NOTA: Per la rilevazione con immunofluorescenza (paragrafo 2), questo passaggio può essere omesso, dato che il blocco delle perossidasi endogene serve a prevenire il segnale artefattuale causato dalla rilevazione con perossidasi di rafano (HRP) e 3,3′-Diaminobenzidina idrato (DAB). - Lavare entrambe le aree 3 volte con TBS, come descritto al punto 1.4.3. Aggiungere una soluzione di latte scremato al 3% e incubare per 30 minuti a RT per bloccare. Versare la soluzione. Non lavare dopo.
- Aggiungere l'anticorpo primario (AB) solo all'area di test. Aggiungere la soluzione di controllo AB (vedere Tabella 2) all'area di controllo. Incubare i campioni a 4 °C durante la notte. Coprire le diapositive durante quel periodo per evitare l'asciugatura.
NOTA: evitare di trasferire le soluzioni dal test all'area di controllo, poiché ciò influenzerebbe i valori di controllo. - Versare la soluzione anticorpale. Lavare entrambe le aree 3 volte con TBS, come descritto al punto 1.4.3.
- Eseguire un passaggio di blocco per impedire l'associazione non specifica. Aggiungere il 3% di siero di capra normale e incubare per 30 minuti a RT. Versare la soluzione.
- Aggiungere la soluzione anticorpale secondaria e incubare per 30 minuti a RT. Versare la soluzione anticorpale. Lavare le aree 3 volte con TBS, come descritto al punto 1.4.3.
- Sviluppo di colorazione e copertura
- Effettuare la reazione HRP accoppiata ad avidina (vedere diluizione nella Tabella 2) aggiungendo una soluzione diluita di avidina-perossidasi e incubando per 30 minuti a RT. Versare la soluzione.
- Preparare la miscela DAB per sviluppare l'immunocolorazione prima dell'uso, come da Tabella 3.
ATTENZIONE: DAB è pericoloso. Considerare le indicazioni di pericolo H341 (sospettato di causare difetti genetici) e H350 (può causare il cancro). Considerare i seguenti consigli di prudenza: P201, P202, P280, P308+P313, P405 e P501. - Eseguire la colorazione di controllo DAB come segue: raccogliere la soluzione HRP dal punto 1.5.1 da un vetrino e miscelare con un piccolo volume (ad esempio, 1 ml) di soluzione DAB preparata in una provetta da centrifuga separata prima della colorazione. La miscela dovrebbe sviluppare un colore marrone / grigio.
- Posizionare i vetrini al microscopio (ingrandimento di almeno 200x) e aggiungere la soluzione DAB a entrambe le aree. Monitorare continuamente la colorazione dei globuli rossi e interrompere la colorazione rimuovendo la soluzione DAB con una pipetta usa e getta prima che lo sfondo inizi a colorarsi. Per la colorazione con serina 1177 RBC-NOS, il tempo di incubazione DAB è di circa 17 minuti.
- Eseguire la disidratazione del campione. Posizionare i vetrini in una griglia di vetro e immergere per 5 s ciascuno in soluzioni di etanolo di varie diluizioni, iniziando con il 70%, seguito dal 96% e poi dal 100% e infine in xilolo. Rimuovere i vetrini dal rack e posizionarli su un fazzoletto, con globuli rossi nella parte superiore per assorbire il liquido in eccesso.
- Aggiungere due o tre gocce di supporto di montaggio in tutta la diapositiva. Coprire la diapositiva con un coprivetrino. Evitare l'inclusione di bolle d'aria, in quanto ciò ostacola la valutazione microscopica. Asciugare i campioni almeno per una notte in una cappa aspirante.
- Esecuzione della valutazione microscopica
- Per la visualizzazione e l'imaging, posizionare i vetrini in un microscopio a luce trasmessa con un ingrandimento di almeno 200x. Assicurarsi che il microscopio sia accoppiato a una fotocamera per scattare foto dei globuli rossi macchiati.
- Accendere la sorgente luminosa del microscopio. Accendere la fotocamera collegata al microscopio e avviare il software di controllo del microscopio. Utilizzare la microscopia a campo chiaro per determinare la messa a fuoco appropriata ruotando le manopole di regolazione della messa a fuoco grossolane e fini e trovando il livello di messa a fuoco in cui sono visibili i globuli rossi.
- Impostare i valori di sfondo delle immagini su 220 ± 5 valori di grigio, misurati su tre aree prive di celle della diapositiva. Per fare ciò, aprire la prima immagine utilizzando il software gratuito ImageJ. Selezionare l'opzione Valori medi di grigio utilizzando il pannello Imposta misure. Utilizzare l'icona ovale per misurare il valore grigio utilizzando il comando Misura . Se i valori misurati sono fuori portata, regolare di conseguenza la luce di fondo al microscopio. Ripetere questi passaggi finché i valori di sfondo non sono corretti.
NOTA: questi valori di sfondo devono rientrare nell'intervallo specificato per tutte le immagini scattate. In caso contrario, i dati non sono comparabili. - Per l'analisi del valore di grigio RBC, contrassegnare il bordo di ciascun RBC utilizzando lo strumento di selezione Oval all'interno del software ImageJ. Determinare i valori di grigio dei singoli RBC utilizzando il comando Misura . Misurare i valori di grigio di un minimo di 50 RBC dal test e un minimo di 10 RBC dall'area di controllo.
NOTA: Il numero totale di globuli rossi analizzati può essere regolato individualmente. Più RBC analizzati, più significativo è il risultato. Tuttavia, il numero totale di globuli rossi analizzati dovrebbe essere confrontabile per ogni condizione, soggetto, ecc. all'interno di un determinato esperimento.- Evitare l'analisi dei globuli rossi vicino alla matita grassa, poiché la colorazione potrebbe essere incompleta in quest'area. Evitare l'analisi dei globuli rossi sovrapposti, poiché ciò influisce sull'intensità della colorazione. Utilizzare solo RBC separati dagli altri RBC. Analizzare almeno cinque immagini dal test e almeno due immagini dall'area di controllo per la valutazione di 50/10 RBC. Includere RBC da ogni immagine nell'analisi finale.
- Per calcolare l'intensità della colorazione, calcola i segnali finali come:
(RBC per area di prova individuale - sfondo medio dell'area di prova) - (RBC per area di controllo individuale - sfondo medio dell'area di controllo)

Figura 2: Rappresentazione del processo fissativo e della generazione di striscio di sangue. (Schema creato con BioRender.com.) I campioni di sangue diluiti vengono fissati chimicamente in paraformaldeide, quindi centrifugati e lavati con soluzione salina tamponata con fosfato. Infine, il sangue risospeso viene spalmato su un vetrino e fissato termicamente passando sopra una fiamma del bruciatore Bunsen. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Tabella 1: Condizioni di preparazione e conservazione delle soluzioni necessarie per la colorazione immunoistochimica. La soluzione può essere preparata prima del protocollo. Clicca qui per scaricare questa tabella.
Tabella 2: Descrizione delle soluzioni anticorpali per uso immediato. Clicca qui per scaricare questa tabella.
Tabella 3: Componenti e protocollo per preparare la soluzione DAB per l'uso immediato. Clicca qui per scaricare questa tabella.
2. Marcatura fluorescente delle proteine RBC
NOTA: La sezione seguente delinea un adattamento del protocollo immunoistochimico, sviluppato con l'obiettivo di consentire l'uso di anticorpi con coniugati fluorescenti (Figura 1).
La preparazione del campione di sangue per il protocollo di immunofluorescenza è identica a quella descritta nel paragrafo 1, quindi la sezione seguente inizia dalla colorazione dei campioni.
- Colorazione con immunofluorescenza
- Incubare con l'anticorpo secondario coniugato fluorescente (per il volume, vedere punto 1.4.2) per 30 minuti a RT al riparo dalla luce, utilizzando una camera di incubazione appositamente costruita o un foglio di alluminio.
- Versare la soluzione anticorpale secondaria e lavare i campioni 3 volte con TBS. Lasciare il lavaggio finale sui campioni per evitare che si secchino.
- Versare il TBS e rimuovere i campioni dal rack dei campioni uno alla volta per evitare un'esposizione prolungata alla luce. Per disidratare i campioni, esporre i vetrini a diverse soluzioni di etanolo per ~ 5 s ciascuna, iniziando con il 70%, seguito dal 90% e infine dal 100%. Esporre i vetrini alla soluzione di xilo/xilene per 5 s.
- Preparare un coprivetrino con due o tre gocce di supporto di montaggio e quindi montare il vetrino con il coprivetrino. Garantire una distribuzione uniforme del mezzo di montaggio applicando una leggera pressione con una pinzetta o uno strumento metallico sterile simile. Elimina le bolle d'aria, che altrimenti potrebbero interferire con l'imaging, con lo stesso strumento. Lasciare asciugare i campioni per una notte in uno spazio buio e asciutto.
- Valutazione microscopica
- Per la visualizzazione e l'imaging, posizionare il vetrino sul palco del microscopio con un ingrandimento totale di almeno 400x. Accendere la sorgente luminosa del microscopio e la sorgente luminosa fluorescente, assicurandosi che la sorgente luminosa fluorescente sia regolata alla massima intensità.
- Accendere la fotocamera collegata al microscopio e avviare il software di controllo del microscopio. Utilizzare la microscopia a campo chiaro per determinare la messa a fuoco appropriata ruotando le manopole di regolazione della messa a fuoco grossolana e fine e trovando il livello di messa a fuoco in cui sono visibili i globuli rossi.
NOTA: Un'eccessiva esposizione alla luce può causare il fotosbiancamento dei coniugati fluorescenti. Si può usare una diapositiva di un precedente esperimento per questo scopo, per ridurre al minimo l'esposizione alla luce dei vetrini che devono ancora essere analizzati. - Determinare l'intensità laser ottimale ispezionando i globuli rossi nell'area di controllo. Assicurati che l'intensità sia abbastanza alta da rendere visibili i globuli rossi, senza produrre un segnale di fondo di grandi dimensioni. Mantenere l'intensità e il tempo di esposizione coerenti tra aree e campioni per garantire la comparabilità.
- Acquisire immagini in campo chiaro e fluorescenti in almeno tre aree distinte dell'area di test del vetrino, selezionate a caso. Per selezionare un'area, allontanatevi dai bordi della diapositiva contrassegnati dalla penna lipidica, utilizzando i controlli dello stage del microscopio. Selezionate un'area che mostri una distribuzione uniforme di un singolo livello RBC.
- Impostare il tempo di esposizione su 1 s per le immagini fluorescenti e acquisire un'immagine utilizzando i controlli software del software di controllo del microscopio. Passare alla modalità brightfield, impostare il tempo di esposizione su "Auto" e acquisire l'immagine in campo chiaro corrispondente.
- Acquisire immagini in campo chiaro e fluorescenti in almeno due aree distinte selezionate casualmente dell'area di controllo della diapositiva ripetendo il passaggio 2.2.3.
- Salvare le immagini in .tif formato per preservare i valori di grigio originali dei pixel, impedire la compressione e trasportare i metadati dell'acquisizione.
- Misurare i valori di grigio dei globuli rossi acquisiti. Aprire ImageJ (o FIJI21; vedere File supplementare 1). Determinare i valori di grigio dei singoli globuli rossi. A tal fine, contrassegnare ogni singola cella con lo strumento di selezione ovale e analizzare utilizzando il comando Misura .
NOTA: i globuli rossi non devono essere analizzati se si sovrappongono ad altre celle, poiché ciò potrebbe aumentare il segnale risultante. - Evidenziare da tre a cinque aree prive di globuli rossi e determinare i valori di grigio per fornire una misura del segnale di fondo. Analizzare almeno 150 RBC da almeno tre immagini distinte per ogni area di test e almeno 50 RBC da almeno due immagini distinte per ogni area di controllo.
NOTA: L'analisi di più celle / aree può essere consigliata per ridurre al minimo la variabilità. Dato che la popolazione di globuli rossi è intrinsecamente eterogenea, la variabilità cellula-cellula nel segnale può essere significativa, a seconda della proteina di interesse. - Eseguire analisi alternative dei dati delle immagini acquisite utilizzando il comando macro. Creare/installare un comando macro tramite la versione FIJI di ImageJ per la selezione automatica, la correzione dello sfondo e l'analisi dei valori di grigio di una determinata immagine.
NOTA: questa routine utilizza la soglia automatica per rilevare le celle presenti in una determinata immagine utilizzando una copia dell'immagine originale, producendo una sovrapposizione che viene imposta sull'immagine originale per estrarre i valori di grigio dei globuli rossi fluorescenti. La macro viene depositata come file ijm, come file di codifica supplementare 1. - Aprire il file immagine in .tif formato pronto per l'analisi nelle FIGI. Aprire la macro RBC fluorescence.ijm (Supplementary Coding File 1) e fare clic su Esegui.
NOTA: la macro è impostata per immagini ottenute con ingrandimento 600x e un ampio rapporto segnale-rumore. La selezione automatica delle cellule deve essere esaminata dallo sperimentatore.
- Calcola i segnali finali come:
(area di prova RBC - sfondo area test) - (area di controllo RBC - sfondo area di controllo) per l'analisi manuale;
(area di test - area di controllo) per analisi automatizzate.
Risultati
Il protocollo presentato, che descrive metodi che facilitano la rilevazione di alterazioni acute nelle proteine RBC, è stato testato su una ben nota alterazione proteica meccanicamente sensibile: la fosforilazione di RBC-NOS sul residuo di serina 1177. Il sangue intero è stato ottenuto da volontari sani e successivamente diviso in due aliquote separate. Un dato campione di sangue è stato esposto a sollecitazioni meccaniche di taglio di entità fisiologica (5 Pa) per 300 s, che in precedenza ha dimostrato di provocare ...
Discussione
La letteratura recente suggerisce altamente che la proteina RBC-NOS è di cruciale importanza per la regolazione della deformabilità dei globuli rossi 15,22,23, che a sua volta facilita il loro passaggio attraverso capillari stretti 24. L'attività proteica dipende fortemente dalle modificazioni proteiche post-traduzionali, in particolare dalla fosforilazione di alcuni residui18....
Divulgazioni
Tutti gli autori hanno rivelato che non ci sono conflitti di interesse.
Riconoscimenti
LK riconosce il sostegno di una borsa di studio del programma di formazione alla ricerca del governo australiano.
Materiali
| Name | Company | Catalog Number | Comments |
| 3,3′-Diaminobenzidin -tetrahydrochloride Hydrate | Sigma/Merck | D5637 | DAB |
| Ammoniumchloride | Merck /Millipore | 101145 | NH4Cl |
| Centrifuge 5427 R | Eppendorf | 5409000010 | |
| Coverslips | VWR | 631-0147 | |
| di-sodium Hydrogen Phosphate Dihydrate | Merck /Millipore | 106580 | Na2HPO4. 2 H2O |
| Disposable transfer pipettes | VWR | 612-6803 | |
| Entellan | Merck /Millipore | 107961 | rapid mounting medium for microscopy |
| Ethanol denaturated using 1 % methyl ethyl ketone (MEK) | Hofmann | 642 | |
| Glucose-Oxidase | Sigma/Merck | G2133 | |
| Grease pencil | Dako | S 2002 | |
| Horse-radish peroxidase/ExtrAvidin−Peroxidase | Sigma/Merck | E-2886 | HRP |
| Hydrochloric acid | Merck /Millipore | 109057 | HCl |
| Hydrogen peroxide, 30% | Merck /Millipore | 107203 | H2O2 |
| ImageJ Software | Freeware | ||
| Laser-assisted optical rotational cell analyser (LORCA) | RR Mechatronics | Ektacytometer instrument used for shearing | |
| Methanol | Merck /Millipore | 106009 | |
| Microscope slides | VWR | 630-1985 | |
| Nickel(II)-sulfate Hexahydrate | Sigma/Merck | N4882 | NiSO4.6H2O |
| Normal Goat serum | Agilent/DAKO | X0907 | NGS |
| Paraformaldehyde | Merck /Millipore | 818715 | PFA |
| Pipettes Eppendorf Reference 2 | VWR | 613-5836/ 613-5839 | |
| Rabbit Anti-phospho eNOS Antibody (Ser1177) | Merck/Millipore | 07-428-I | Primary Antibody |
| Reaction tubes, 2ml | Eppendorf | 30120094 | |
| Secondary Antibody goat anti rabbit | Agilent/DAKO | E0432 | Secondary Antibody |
| Skim milk powder | Bio-Rad | 170-6404 | |
| Sodium chloride | Merck /Millipore | 106404 | NaCl |
| Sodium Dihydrogen Phosphate Monohydrate | Merck /Millipore | 106346 | NaH2PO4.H2O |
| Sodium hydroxide, 1 M | Merck /Millipore | 150706 | NaOH |
| Tris(hydroxymethyl)-aminomethane | Merck /Millipore | 108382 | Tris |
| Trypsin | Sigma/Merck | T7409 | |
| Tween20 | Merck /Millipore | 822184 | |
| Whatman Glas microfiber filter, quality GF/F | Merck /Millipore | WHA1825047 | |
| Xylol | VWR Chemicals | 2,89,73,465 | |
| ß-D-Glucose monohydrate | Merck /Millipore | 14431-43-7 |
Riferimenti
- Cohen, R. M., et al. Red cell life span heterogeneity in hematologically normal people is sufficient to alter HbA1c. Blood. 112 (10), 4284-4291 (2008).
- Mock, D. M., et al. Red blood cell (RBC) survival determined in humans using RBCs labeled at multiple biotin densities. Transfusion. 51 (5), 1047-1057 (2011).
- Thiagarajan, P., Parker, C. J., Prchal, J. T. How do red blood cells die?. Frontiers in Physiology. 12, 655393 (2021).
- Moras, M., Lefevre, S. D., Ostuni, M. A. From erythroblasts to mature red blood cells: organelle clearance in mammals. Frontiers in Physiology. 8, 1076 (2017).
- Pretini, V., et al. Red blood cells: chasing interactions. Frontiers in Physiology. 10, 945 (2019).
- Cahalan, S. M., et al. Piezo1 links mechanical forces to red blood cell volume. eLife. 4, e07370 (2015).
- Kuck, L., Peart, J. N., Simmonds, M. J. Piezo1 regulates shear-dependent nitric oxide production in human erythrocytes. American Journal of Physiology. Heart and Circulatory Physiology. 323 (1), H24-H37 (2022).
- Kuck, L., Peart, J. N., Simmonds, M. J. Active modulation of human erythrocyte mechanics. American Journal of Physiology. Cell Physiology. 319 (2), C250-C257 (2020).
- Strader, M. B., et al. Post-translational modification as a response to cellular stress induced by hemoglobin oxidation in sickle cell disease. Scientific Reports. 10 (1), 14218 (2020).
- Pecankova, K., Majek, P., Cermak, J., Dyr, J. E. Posttranslational modifications of red blood cell ghost proteins as "signatures" for distinguishing between low- and high-risk myelodysplastic syndrome patients. Turkish Journal of Haematology. 34 (1), 111-113 (2017).
- Grau, M., et al. High red blood cell nitric oxide synthase activation is not associated with improved vascular function and red blood cell deformability in sickle cell anaemia. British Journal of Haematology. 168 (5), 728-736 (2015).
- Sae-Lee, W., et al. The protein organization of a red blood cell. Cell Reports. 40 (3), 111103 (2022).
- Suhr, F., et al. Moderate exercise promotes human RBC-NOS activity, NO production and deformability through Akt kinase pathway. PLoS One. 7 (9), e45982 (2012).
- Kuck, L., Grau, M., Bloch, W., Simmonds, M. J. Shear stress ameliorates superoxide impairment to erythrocyte deformability with concurrent nitric oxide synthase activation. Frontiers in Physiology. 10, 36 (2019).
- Grau, M., et al. RBC-NOS-dependent S-nitrosylation of cytoskeletal proteins improves RBC deformability. PLoS One. 8 (2), e56759 (2013).
- Simmonds, M. J., Detterich, J. A., Connes, P. Nitric oxide, vasodilation and the red blood cell. Biorheology. 51 (2-3), 121-134 (2014).
- Bor-Kucukatay, M., Wenby, R. B., Meiselman, H. J., Baskurt, O. K. Effects of nitric oxide on red blood cell deformability. American Journal of Physiology. Heart and Circulatory Physiology. 284 (5), H1577-H1584 (2003).
- Suhr, F., et al. Intensive exercise induces changes of endothelial nitric oxide synthase pattern in human erythrocytes. Nitric Oxide: Biology and Chemistry. 20 (2), 95-103 (2009).
- Grau, M., et al. Regulation of red blood cell deformability is independent of red blood cell-nitric oxide synthase under hypoxia. Clinical Hemorheology and Microcirculation. 63 (3), 199-215 (2016).
- Grau, M., Kuck, L., Dietz, T., Bloch, W., Simmonds, M. J. Sub-fractions of red blood cells respond differently to shear exposure following superoxide treatment. Biology. 10 (1), 47 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Ozüyaman, B., Grau, M., Kelm, M., Merx, M. W., Kleinbongard, P. RBC NOS: regulatory mechanisms and therapeutic aspects. Trends in Molecular Medicine. 14 (7), 314-322 (2008).
- Kleinbongard, P., et al. Red blood cells express a functional endothelial nitric oxide synthase. Blood. 107 (7), 2943-2951 (2006).
- McMahon, T. J. Red blood cell deformability, vasoactive mediators, and adhesion. Frontiers in Physiology. 10, 1417 (2019).
- Bizjak, D. A., Brinkmann, C., Bloch, W., Grau, M. Increase in red blood cell-nitric oxide synthase dependent nitric oxide production during red blood cell aging in health and disease: a study on age dependent changes of rheologic and enzymatic properties in red blood cells. PLoS One. 10 (4), 0125206 (2015).
- Di Pietro, N., et al. Nitric oxide synthetic pathway and cGMP levels are altered in red blood cells from end-stage renal disease patients. Molecular and Cellular Biochemistry. 417 (1-2), 155-167 (2016).
- Grau, M., et al. Even patients with mild COVID-19 symptoms after SARS-CoV-2 infection show prolonged altered red blood cell morphology and rheological parameters. Journal of Cellular and Molecular Medicine. 26 (10), 3022-3030 (2022).
- Mozar, A., et al. Red blood cell nitric oxide synthase modulates red blood cell deformability in sickle cell anemia. Clinical Hemorheology and Microcirculation. 64 (1), 47-53 (2016).
- Ulker, P., Gunduz, F., Meiselman, H. J., Baskurt, O. K. Nitric oxide generated by red blood cells following exposure to shear stress dilates isolated small mesenteric arteries under hypoxic conditions. Clinical Hemorheology and Microcirculation. 54 (4), 357-369 (2013).
- Nader, E., et al. Hydroxyurea therapy modulates sickle cell anemia red blood cell physiology: Impact on RBC deformability, oxidative stress, nitrite levels and nitric oxide synthase signalling pathway. Nitric Oxide: Biology and Chemistry. 81, 28-35 (2018).
- Fischer, U. M., Schindler, R., Brixius, K., Mehlhorn, U., Bloch, W. Extracorporeal circulation activates endothelial nitric oxide synthase in erythrocytes. The Annals of Thoracic Surgery. 84 (6), 2000-2003 (2007).
- Horobin, J. T., Sabapathy, S., Kuck, L., Simmonds, M. J. Shear stress and RBC-NOS Serine1177 Phosphorylation in humans: a dose response. Life. 11 (1), 36 (2021).
- Kuck, L., Grau, M., Simmonds, M. J. Recovery time course of erythrocyte deformability following exposure to shear is dependent upon conditioning shear stress. Biorheology. 54 (5-6), 141-152 (2018).
- Grau, M., et al. Effect of acute exercise on RBC deformability and RBC nitric oxide synthase signalling pathway in young sickle cell anaemia patients. Scientific Reports. 9 (1), 11813 (2019).
- Feelisch, M. . Methods in Nitric Oxide Research. , (1998).
- Cortese-Krott, M. M., et al. Human red blood cells at work: identification and visualization of erythrocytic eNOS activity in health and disease. Blood>. 120 (20), 4229-4237 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon