É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Detecção de Alterações Dinâmicas em Proteínas Eritrocitárias Baseadas em Imunomarcação
Neste Artigo
Resumo
A captação de mudanças dinâmicas na ativação proteica de hemácias enucleadas apresenta desafios metodológicos, como a preservação de alterações dinâmicas em estímulos agudos para posterior avaliação. O protocolo apresentado descreve técnicas de preparo e coloração de amostras que permitem a preservação e análise de alterações proteicas relevantes e posterior detecção.
Resumo
A marcação de anticorpos de proteínas eritrocitárias é um método semiquantitativo comumente usado para detectar mudanças no conteúdo global de proteínas ou alterações agudas em estados de ativação proteica. Facilita a avaliação de tratamentos eritrocitários, a caracterização de diferenças em determinados estados patológicos e a descrição de coerências celulares. A detecção de ativação proteica agudamente alterada (por exemplo, através de mecanotransdução) requer preparação adequada da amostra para preservar modificações proteicas temporárias. O princípio básico inclui a imobilização dos sítios de ligação alvo das proteínas eritrocitárias desejadas para permitir a ligação inicial de anticorpos primários específicos. A amostra é posteriormente processada para garantir condições ideais para a ligação do anticorpo secundário ao anticorpo primário correspondente. A seleção de anticorpos secundários não fluorescentes requer tratamento adicional, incluindo o acoplamento biotina-avidina e a aplicação de 3,3-diaminobenzidina-tetracloridrato (DAB) para desenvolver a coloração, que precisa ser controlada em tempo real sob um microscópio para interromper a oxidação e, assim, colorir a intensidade a tempo. Para a detecção da intensidade da coloração, as imagens são obtidas usando um microscópio de luz padrão. Em uma modificação deste protocolo, um anticorpo secundário conjugado à fluoresceína pode ser aplicado em seu lugar, o que tem a vantagem de que nenhuma etapa adicional de desenvolvimento é necessária. Este procedimento, no entanto, requer uma objetiva de fluorescência acoplada a um microscópio para detecção da coloração. Dada a natureza semiquantitativa desses métodos, é imperativo fornecer várias colorações de controle para explicar reações de anticorpos inespecíficos e sinais de fundo. Aqui, apresentamos os protocolos de coloração e os processos analíticos correspondentes para comparar e discutir os respectivos resultados e vantagens das diferentes técnicas de coloração.
Introdução
As hemácias (hemácias) atravessam o sistema cardiovascular por 70 a 140 dias, com idade média das hemácias de aproximadamente 115 dias 1,2. As hemácias senescentes ou lesadas são removidas da circulação por eritrofagocitose, um eficiente processo de clareamento conduzido por macrófagos3. O tempo de vida predeterminado dessas células é uma consequência da entrega das organelas celulares, incluindo o núcleo, as mitocôndrias e os ribossomos, durante a diferenciação e maturação4. Assim, as hemácias circulantes são desprovidas de maquinaria translacional, impossibilitando a síntese de novas proteínas3. Conclui-se que modificações dinâmicas e pós-traducionais em proteínas existentes representam o único mecanismo viável de regulação bioquímica aguda em resposta a estressores extracelulares e intracelulares que atuam sobre as hemácias5.
As forças mecânicas parecem ser as principais pistas extracelulares que causam a ativação ou modulação de vias bioquímicas dentro das hemácias. A descoberta da proteína mecanossensível, Piezo1, em membranas eritrocitárias6 inspirou várias linhas de pesquisa investigando a sinalização ativada mecanicamente nessas células7. Por exemplo, avanços recentes têm mostrado que as propriedades físicas das hemácias são ativamente reguladas por mudanças agudas e dinâmicas das proteínas8, o que inclui fosforilação pós-traducional e ubiquitinação9. Como essas modificações normais diferem em certas doenças 9,10,11, parece ser de interesse científico e clínico determinar o estado de ativação das proteínas eritrocitárias, especificamente em relação aos processos mecanobiológicos.
A determinação de alterações agudas nos estados de ativação de proteínas eritrocitárias apresenta alguns desafios metodológicos. Por exemplo, o armazenamento de amostras de hemácias para análise posterior requer a preservação das proteínas eritrocitárias modificadas, uma vez que as modificações pós-traducionais não são duráveis. Além disso, os métodos clássicos de detecção de proteínas (por exemplo, western blotting) são notoriamente difíceis de padronizar em hemácias devido à baixa abundância de proteínas em relação à hemoglobina, que responde por ~98% do conteúdo proteico nessas células12. Assim, a coloração de hemácias quimicamente preservadas com base em anticorpos tem sido o método de escolha na investigação de modificações agudas de importantes proteínas eritrocitárias, como a isoforma eritrocitária específica da óxido nítrico sintase (RBC-NOS)13,14. Demonstrou-se que as hemácias-NOS produzem enzimaticamente óxido nítrico (NO), o que parece indispensável para propriedades essenciais das hemácias, incluindo a deformabilidade das hemácias15,16,17. Modificações pós-traducionais de RBC-NOS regulam a atividade enzimática catalítica, com a fosforilação do resíduo de serina 1177 sendo descrita para aumentar a atividade enzimática, enquanto a fosforilação dos resíduos serina 114 ou treonina 495 tem sido associada à diminuição da atividade de RBC-NOS18,19.
Coletivamente, modificações temporárias de proteínas eritrocitárias contribuem para importante função celular, e protocolos padronizados que permitem a detecção dessas proteínas modificadas são de alto valor. Aqui, apresentamos dois protocolos distintos que exploram anticorpos específicos para facilitar a detecção da ativação da proteína eritrocitária-NOS, e discutimos recomendações para análise e interpretação dos dados.
O desempenho dos protocolos descritos foi avaliado medindo-se o aumento bem relatado na fosforilação de eritrócitos-NOS no resíduo de serina 1177 em resposta a forças mecânicas reflexivas daquelas que ocorrem dentro da vasculatura humana (5 Pa).
Protocolo
Os protocolos aqui descritos estão alinhados com a Declaração de Helsinque e foram aprovados pelos Comitês de Ética da Universidade Alemã de Esportes de Colônia (16/9/2013) e da Universidade Griffith (2019/808). Os voluntários foram triados para garantir a ausência de patologias relevantes e assinaram o termo de consentimento livre e esclarecido.
1. Coloração de proteínas eritrocitárias utilizando protocolos de imunohistoquímica
NOTA: Uma lista detalhada dos produtos químicos e materiais necessários é fornecida na Tabela de Materiais. As seções a seguir descrevem o preparo das soluções necessárias, seguidas de uma descrição detalhada do protocolo de imuno-histoquímica (Figura 1).
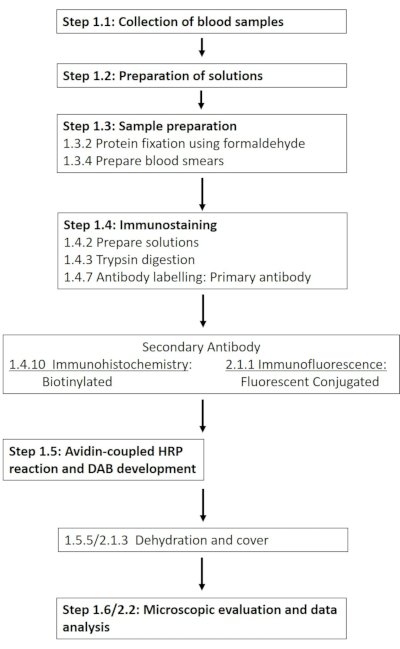
Figura 1: Esquema dos passos individuais necessários para a coloração imunoistoquímica e imunofluorescência das hemácias-NOS no sítio de fosforilação 1177. Um fluxo de trabalho típico dos protocolos apresentados, desde o preparo da solução e coleta de sangue até a detecção e visualização baseada em anticorpos, é apresentado. Clique aqui para ver uma versão maior desta figura.
- Coleta de amostra de sangue
- Obter amostras de sangue (10 mL de sangue total) de sete homens saudáveis. Os voluntários eram indivíduos saudáveis e livres de doenças cardiovasculares, hematológicas, neurológicas, endócrinas ou metabólicas. Os voluntários também não eram tabagistas.
- Coletar sangue usando uma agulha estéril e seringa de uma veia proeminente na região antecubital do antebraço e transferir imediatamente para tubos revestidos com um dos seguintes anticoagulantes: heparina sódica (para imunohistoquímica) ou ácido etilenodiaminotetracético (EDTA; para marcação fluorescente).
- Expor as amostras de sangue anticoagulado a forças mecânicas controladas com precisão usando um sistema de cisalhamento tipo Couette, como descrito anteriormente 7,20.
- O aparelho de cisalhamento é composto por um copo giratório e um bob estacionário, separados por um gap de 300 μm. Transfira a amostra de sangue para a lacuna, o que faz com que a velocidade de rotação precisamente controlável do copo exerça forças mecânicas bem controladas sobre a amostra. Os dados representativos aqui apresentados foram produzidos pela aplicação de forças mecânicas reflexivas daquelas que ocorrem dentro da vasculatura humana (5 Pa) por longas durações (300 s).
- Preparação de soluções necessárias para imunomarcação
- Prepare as soluções conforme a Tabela 1. As soluções podem ser preparadas antes da realização do procedimento de imunomarcação e armazenadas em determinada temperatura até que seja necessário.
NOTA: A duração do armazenamento pode variar entre os produtos químicos. Verifique a ficha de dados de segurança do material.
- Prepare as soluções conforme a Tabela 1. As soluções podem ser preparadas antes da realização do procedimento de imunomarcação e armazenadas em determinada temperatura até que seja necessário.
- Preparo da amostra
- Processar o sangue total imediatamente após a retirada ou tratamento experimental (quando aplicável; por exemplo, estímulo mecânico em nossas amostras; ver etapa 1.1.3) para ser capaz de detectar efeitos de curta duração, por exemplo, após a aplicação de tensão de cisalhamento, exercício, exposição à hipóxia de curta duração, etc.
- Realizar a fixação da proteína eritrocitária com formaldeído da seguinte forma: diluir o sangue total na proporção de 1:2 em solução de paraformaldeído a 4% por 20 min à temperatura ambiente (TR). Centrifugar a amostra a 132 x g durante 3 min em RT e remover cuidadosamente o sobrenadante por pipetagem.
- Ressuspender o pellet de hemácias em dois volumes de solução salina tamponada com fosfato (PBS) 0,1 mol/L (diluição 1:3) e incubar por 5 min na RT. Repita a centrifugação conforme descrito acima e remova o sobrenadante, que deve estar claro, por pipetagem. Ressuspender a pastilha de hemácias em um volume de PBS 0,1 mol/L (diluição 1:2).
- Prepare esfregaços de sangue da seguinte forma: rotule uma lâmina de microscópio com o ID da amostra, anticorpo aplicado, etc. usando uma caneta resistente ao álcool (por exemplo, lápis). Em seguida, adicione 10 μL da solução de hemácia preparada logo acima do campo do rótulo.
- Coloque uma segunda lâmina sobre a amostra em um ângulo de aproximadamente 45° e disperse a amostra uniformemente ao longo da lâmina. Fixe o calor da lâmina sobre um queimador de Bunsen passando a amostra sobre o queimador com movimento constante por 5-7 s.
NOTA: Recomenda-se secar ao ar as amostras antes da fixação térmica. As amostras podem ser armazenadas em RT até a coloração (Figura 2).
- Coloração imunoistoquímica
- Marque duas áreas em cada lâmina: uma área de teste na qual as hemácias são incubadas com o respectivo anticorpo primário e secundário e uma área de controle na qual o anticorpo primário é substituído por uma solução de controle. Esta área serve como um controle interno do ensaio para determinar o sinal de fundo das hemácias.
- Preparar soluções antes ou durante o procedimento, conforme Tabela 2. É necessário um volume de 300 μL por área de teste e 200 μL por área de controle.
NOTA: É importante notar que esses procedimentos de coloração baseados em anticorpos produzem dados semiquantitativos, em vez de quantitativos. Assim, para garantir que conclusões significativas possam ser tiradas dos dados produzidos, amostras de controle apropriadas (ou seja, para fornecer um ponto de referência para as alterações relativas avaliadas na ativação de proteínas) são absolutamente necessárias. - Para a digestão de tripsina, descongele a tripsina a 0,1% e equilibre para RT. Marque duas áreas em cada lâmina usando um lápis de gordura: uma área de teste (2/3 da lâmina) e uma área de controle (1/3 da lâmina). Aplique cuidadosamente solução salina tamponada com tris (TBS) usando pipetas de transferência para lavar as áreas da amostra. Deixe o TBS descansar por 30 s e depois despeje. Repita a etapa de lavagem.
Observação : adicione as seguintes soluções para o controle e a área de teste, a menos que descrito de outra forma. As áreas precisam ser suficientemente cobertas com a respectiva solução. Aplicar as soluções utilizando pipetas de transferência descartáveis e alternar pipetas após cada solução. - Adicione 0,1% de tripsina, cubra as lâminas usando uma câmara de incubação construída especificamente com uma tampa ou papel alumínio e incube por 30 min a 37 °C em uma incubadora. Após a incubação, pare a reação enzimática adicionando água da torneira. Despeje a solução. Lave ambas as áreas 3x com TBS, conforme descrito acima.
- Para bloquear a ativação da peroxidase, adicione solução de metanol e incube por 30 min em RT. Cubra as lâminas durante esse tempo. Despeje a solução.
NOTA: Para a detecção de imunofluorescência (secção 2), este passo pode ser omitido, uma vez que o bloqueio das peroxidases endógenas serve para prevenir o sinal artifactual causado pela detecção com peroxidase de raiz forte (HRP) e hidrato de 3,3′-diaminobenzidina (DAB). - Lavar ambas as áreas 3x com TBS, conforme descrito no passo 1.4.3. Adicionar 3% de solução de leite desnatado e incubar por 30 min em RT para bloquear. Despeje a solução. Não lave depois.
- Adicionar anticorpo primário (AB) apenas à área de teste. Adicione a solução de controle AB (consulte a Tabela 2) à área de controle. Incubar as amostras a 4 °C durante a noite. Cubra as lâminas durante esse tempo para evitar a secagem.
NOTA: Evite transferir as soluções do teste para a área de controle, pois isso influenciará os valores de controle. - Despeje a solução de anticorpos. Lavar ambas as áreas 3x com TBS, conforme descrito no passo 1.4.3.
- Execute uma etapa de bloqueio para evitar vinculação inespecífica. Adicionar 3% de soro normal de cabra e incubar por 30 min em RT.
- Adicione a solução de anticorpos secundários e incube por 30 min na RT. Despeje a solução de anticorpos. Lavar as áreas 3x com TBS, conforme descrito no passo 1.4.3.
- Desenvolvimento de coloração e cobertura
- Efectuar a reacção HRP associada à avidina (ver diluição no quadro 2) adicionando solução diluída de avidina-peroxidase e incubando durante 30 minutos em RT.
- Preparar a mistura DAB para desenvolver a imunomarcação antes do uso, conforme Tabela 3.
CUIDADO: DAB é perigoso. Considere as advertências de perigo H341 (suspeita de causar defeitos genéticos) e H350 (pode causar câncer). Considere as seguintes declarações de precaução: P201, P202, P280, P308+P313, P405 e P501. - Realizar a coloração de controle DAB da seguinte forma: coletar a solução de HRP da etapa 1.5.1 de uma lâmina e misturar com um pequeno volume (por exemplo, 1 mL) de solução DAB preparada em um tubo de centrífuga separado antes da coloração. A mistura deve desenvolver uma cor marrom/cinza.
- Coloque as lâminas sob um microscópio (aumento de pelo menos 200x) e adicione solução DAB a ambas as áreas. Monitore a coloração das hemácias continuamente e pare a coloração removendo a solução DAB com uma pipeta descartável antes que o fundo comece a colorir. Para a coloração de serina 1177 RBC-NOS, o tempo de incubação do DAB é de cerca de 17 min.
- Realizar a desidratação da amostra. Coloque as lâminas em uma cremalheira de vidro e mergulhe por 5 s cada em soluções de etanol de várias diluições, começando com 70%, seguido por 96%, depois 100% e, finalmente, em xilol. Retire as lâminas do rack e coloque em um tecido, com hemácias na parte superior para absorver o excesso de líquido.
- Adicione duas ou três gotas de meio de montagem em todo o slide. Cubra o slide usando uma lamínula. Evitar a inclusão de bolhas de ar, pois isso dificulta a avaliação microscópica. Seque as amostras pelo menos durante a noite em um exaustor.
- Realização de avaliação microscópica
- Para visualização e obtenção de imagens, coloque as lâminas em um microscópio de luz transmitida com aumento de pelo menos 200x. Certifique-se de que o microscópio esteja acoplado a uma câmera para tirar fotos das hemácias coradas.
- Ligue a fonte de luz do microscópio. Ligue a câmera acoplada ao microscópio e inicie o software de controle do microscópio. Use a microscopia de campo brilhante para determinar o foco apropriado, girando os botões de ajuste de foco grosso e fino e encontrando o nível de foco onde as hemácias são visíveis.
- Defina os valores de plano de fundo das imagens como 220 ± 5 valores de cinza, medidos em três áreas livres de células do slide. Para fazer isso, abra a primeira imagem usando o software disponível gratuitamente ImageJ. Selecione a opção Valores médios de cinza usando o painel Definir medidas. Use o ícone oval para medir o valor cinza usando o comando Medir . Se os valores medidos estiverem fora do intervalo, ajuste a luz de fundo no microscópio de acordo. Repita essas etapas até que os valores de plano de fundo estejam corretos.
Observação : esses valores de plano de fundo devem estar dentro do intervalo fornecido para todas as imagens tiradas. Caso contrário, os dados não serão comparáveis. - Para análise do valor de cinza RBC, marque a borda de cada RBC usando a ferramenta de seleção Oval dentro do software ImageJ. Determine os valores de cinza de hemácias individuais usando o comando Measure . Medir os valores de cinza de um mínimo de 50 hemácias do teste e um mínimo de 10 hemácias da área de controle.
NOTA: O número total de hemácias analisadas pode ser ajustado individualmente. Quanto mais hemácias analisadas, mais significativo é o resultado. No entanto, o número total de hemácias analisadas deve ser comparável para cada condição, sujeito, etc. dentro de um determinado experimento.- Evite a análise de hemácias perto do lápis de gordura, pois a coloração pode estar incompleta nessa área. Evite a análise de hemácias sobrepostas, pois isso afeta a intensidade da coloração. Utilizar somente hemácias separadas de outras hemácias. Analisar pelo menos cinco imagens do teste e pelo menos duas imagens da área de controle para a avaliação de 50/10 hemácias. Incluir hemácias de cada foto na análise final.
- Para calcular a intensidade de coloração, calcule os sinais finais como:
(RBC da área de teste individual - fundo médio da área de teste) - (RBC da área de controle individual - fundo da área de controle médio)

Figura 2: Representação do processo fixador e geração de esfregaço sanguíneo. (Esquema criado com BioRender.com.) As amostras de sangue diluídas são fixadas quimicamente em paraformaldeído, depois centrifugadas e lavadas com solução salina tamponada com fosfato. Finalmente, o sangue ressuspendido é manchado em uma lâmina de vidro e fixado termicamente através do pairar sobre uma chama de queimador de Bunsen. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Condições de preparo e armazenamento das soluções necessárias para a coloração imunoistoquímica. A solução pode ser preparada antes do protocolo. Clique aqui para baixar esta tabela.
Tabela 2: Descrição das soluções de anticorpos para uso imediato. Clique aqui para baixar esta tabela.
Tabela 3: Componentes e protocolo para preparar a solução DAB para uso imediato. Clique aqui para baixar esta tabela.
2. Marcação fluorescente de proteínas eritrocitárias
NOTA: A seção a seguir descreve uma adaptação do protocolo de imunohistoquímica, desenvolvido com o objetivo de possibilitar o uso de anticorpos com conjugados fluorescentes (Figura 1).
A preparação da amostra de sangue para o protocolo de imunofluorescência é idêntica à descrita na secção 1, pelo que a secção seguinte começa a partir da coloração das amostras.
- Coloração por imunofluorescência
- Incubar com o anticorpo conjugado fluorescente secundário (para o volume, ver passo 1.4.2) durante 30 minutos em RT protegido da luz, utilizando uma câmara de incubação construída especificamente ou folha de alumínio.
- Deite a solução de anticorpos secundários e lave as amostras 3x com TBS. Deixe a lavagem final nas amostras para evitar que sequem.
- Despeje o TBS e remova as amostras do rack de amostras, uma de cada vez, para evitar a exposição prolongada à luz. Para desidratar as amostras, exponha as lâminas a diferentes soluções de etanol por ~5 s cada, começando com 70%, seguido por 90% e, finalmente, 100%. Expor as lâminas à solução de xilol/xileno por 5 s.
- Prepare uma lamínula com duas ou três gotas de meio de montagem e, em seguida, monte a corrediça com a lamínula. Garanta a distribuição uniforme do meio de montagem aplicando uma leve pressão com uma pinça ou um instrumento de metal estéril similar. Elimine bolhas de ar, que podem interferir com a imagem, com o mesmo instrumento. Deixe as amostras secar durante a noite em um espaço escuro e seco.
- Avaliação microscópica
- Para visualização e aquisição de imagens, colocar a lâmina no palco do microscópio com um aumento total de pelo menos 400x. Ligue a fonte de luz do microscópio e a fonte de luz fluorescente, garantindo que a fonte de luz fluorescente seja ajustada para a intensidade máxima.
- Ligue a câmera acoplada ao microscópio e inicie o software de controle do microscópio. Use a microscopia de campo brilhante para determinar o foco apropriado, girando os botões de ajuste de foco grosso e fino e encontrando o nível de foco onde as hemácias são visíveis.
NOTA: A exposição excessiva à luz pode causar fotobranqueamento dos conjugados fluorescentes. Pode-se utilizar uma lâmina de um experimento anterior para esse fim, para minimizar a exposição à luz das lâminas que ainda serão analisadas. - Determine a intensidade ideal do laser inspecionando as hemácias na área de controle. Certifique-se de que a intensidade seja alta o suficiente para que as hemácias estejam visíveis, enquanto não produzem um grande sinal de fundo. Mantenha a intensidade e o tempo de exposição consistentes entre áreas e amostras para garantir a comparabilidade.
- Capture imagens de campo claro e fluorescentes em pelo menos três áreas distintas da área de teste da lâmina, selecionadas aleatoriamente. Para selecionar uma área, afaste-se das bordas da lâmina marcadas pela caneta lipídica, usando os controles de estágio do microscópio. Selecione uma área que exiba distribuição uniforme de uma camada singular de eritrócitos.
- Defina o tempo de exposição para 1 s para imagens fluorescentes e capture uma imagem usando os controles de software do software de controle do microscópio. Alterne para o modo de campo brilhante, defina o tempo de exposição como 'Automático' e capture a imagem de campo brilhante correspondente.
- Capture imagens de campo brilhante e fluorescentes em pelo menos duas áreas distintas selecionadas aleatoriamente da área de controle da lâmina repetindo a etapa 2.2.3.
- Salve as imagens em .tif formato para preservar os valores de cinza originais dos pixels, impedir a compactação e transportar metadados da aquisição.
- Meça os valores de cinza das hemácias capturadas. Abra o ImageJ (ou FIJI21; consulte Arquivo Suplementar 1). Determine os valores de cinza de hemácias individuais. Para isso, marque cada célula individual com a ferramenta de seleção Oval e analise usando o comando Medir .
NOTA: As hemácias não devem ser analisadas se se sobreporem a outras células, pois isso pode aumentar o sinal resultante. - Realce entre três e cinco áreas livres de hemácias e determine os valores de cinza para fornecer uma medida do sinal de fundo. Analisar pelo menos 150 hemácias de pelo menos três imagens distintas para cada área de teste e pelo menos 50 hemácias de pelo menos duas imagens distintas para cada área de controle.
NOTA: A análise de mais células/áreas pode ser aconselhada para minimizar a variabilidade. Dado que a população de eritrócitos é inerentemente heterogênea, a variabilidade célula-célula no sinal pode ser significativa, dependendo da proteína de interesse. - Execute a análise de dados alternativos das imagens capturadas usando o comando macro. Crie/instale um comando de macro por meio da versão FIJI do ImageJ para seleção automatizada, correção de plano de fundo e análise de valor cinza de uma determinada imagem.
Observação : essa rotina usa o limiar automatizado para detectar as células presentes em uma determinada imagem usando uma cópia da imagem original, produzindo uma sobreposição que é imposta à imagem original para extrair valores de cinza de hemácias fluorescentes. A macro é depositada como um arquivo .ijm, como arquivo de codificação suplementar 1. - Abra o arquivo de imagem em .tif formato pronto para análise em FIJI. Abra a macro fluorescence.ijm (Supplementary Coding File 1) do RBC e clique em Executar.
NOTA: A macro está configurada para imagens obtidas com ampliação de 600x e uma grande relação sinal-ruído. A seleção automática de células deve ser revista pelo investigador.
- Calcule os sinais finais como:
(área de teste RBC - fundo da área de teste) - (área de controle RBC - fundo da área de controle) para análise manual;
(área de teste - área de controle) para análise automatizada.
Resultados
O protocolo apresentado, descrevendo métodos que facilitam a detecção de alterações agudas em proteínas eritrocitárias, foi testado em uma conhecida alteração proteica mecanicamente sensível: a fosforilação de hemácias-NOS no resíduo de serina 1177. O sangue total foi obtido de voluntários saudáveis e posteriormente dividido em duas alíquotas separadas. Uma determinada amostra de sangue foi exposta a tensão de cisalhamento mecânica de magnitude fisiológica (5 Pa) por 300 s, que foi previamente demonst...
Discussão
A literatura recente sugere que a proteína RBC-NOS é de fundamental importância para a regulação da deformabilidade das hemácias 15,22,23, o que, por sua vez, facilita sua passagem por capilares estreitos 24. A atividade proteica depende fortemente de modificações proteicas pós-traducionais, particularmente da fosforilação de certos resíduos18. O foco de interesse est?...
Divulgações
Todos os autores declararam não haver conflitos de interesse.
Agradecimentos
A LK reconhece o apoio de uma bolsa do Programa de Treinamento em Pesquisa do Governo Australiano.
Materiais
| Name | Company | Catalog Number | Comments |
| 3,3′-Diaminobenzidin -tetrahydrochloride Hydrate | Sigma/Merck | D5637 | DAB |
| Ammoniumchloride | Merck /Millipore | 101145 | NH4Cl |
| Centrifuge 5427 R | Eppendorf | 5409000010 | |
| Coverslips | VWR | 631-0147 | |
| di-sodium Hydrogen Phosphate Dihydrate | Merck /Millipore | 106580 | Na2HPO4. 2 H2O |
| Disposable transfer pipettes | VWR | 612-6803 | |
| Entellan | Merck /Millipore | 107961 | rapid mounting medium for microscopy |
| Ethanol denaturated using 1 % methyl ethyl ketone (MEK) | Hofmann | 642 | |
| Glucose-Oxidase | Sigma/Merck | G2133 | |
| Grease pencil | Dako | S 2002 | |
| Horse-radish peroxidase/ExtrAvidin−Peroxidase | Sigma/Merck | E-2886 | HRP |
| Hydrochloric acid | Merck /Millipore | 109057 | HCl |
| Hydrogen peroxide, 30% | Merck /Millipore | 107203 | H2O2 |
| ImageJ Software | Freeware | ||
| Laser-assisted optical rotational cell analyser (LORCA) | RR Mechatronics | Ektacytometer instrument used for shearing | |
| Methanol | Merck /Millipore | 106009 | |
| Microscope slides | VWR | 630-1985 | |
| Nickel(II)-sulfate Hexahydrate | Sigma/Merck | N4882 | NiSO4.6H2O |
| Normal Goat serum | Agilent/DAKO | X0907 | NGS |
| Paraformaldehyde | Merck /Millipore | 818715 | PFA |
| Pipettes Eppendorf Reference 2 | VWR | 613-5836/ 613-5839 | |
| Rabbit Anti-phospho eNOS Antibody (Ser1177) | Merck/Millipore | 07-428-I | Primary Antibody |
| Reaction tubes, 2ml | Eppendorf | 30120094 | |
| Secondary Antibody goat anti rabbit | Agilent/DAKO | E0432 | Secondary Antibody |
| Skim milk powder | Bio-Rad | 170-6404 | |
| Sodium chloride | Merck /Millipore | 106404 | NaCl |
| Sodium Dihydrogen Phosphate Monohydrate | Merck /Millipore | 106346 | NaH2PO4.H2O |
| Sodium hydroxide, 1 M | Merck /Millipore | 150706 | NaOH |
| Tris(hydroxymethyl)-aminomethane | Merck /Millipore | 108382 | Tris |
| Trypsin | Sigma/Merck | T7409 | |
| Tween20 | Merck /Millipore | 822184 | |
| Whatman Glas microfiber filter, quality GF/F | Merck /Millipore | WHA1825047 | |
| Xylol | VWR Chemicals | 2,89,73,465 | |
| ß-D-Glucose monohydrate | Merck /Millipore | 14431-43-7 |
Referências
- Cohen, R. M., et al. Red cell life span heterogeneity in hematologically normal people is sufficient to alter HbA1c. Blood. 112 (10), 4284-4291 (2008).
- Mock, D. M., et al. Red blood cell (RBC) survival determined in humans using RBCs labeled at multiple biotin densities. Transfusion. 51 (5), 1047-1057 (2011).
- Thiagarajan, P., Parker, C. J., Prchal, J. T. How do red blood cells die?. Frontiers in Physiology. 12, 655393 (2021).
- Moras, M., Lefevre, S. D., Ostuni, M. A. From erythroblasts to mature red blood cells: organelle clearance in mammals. Frontiers in Physiology. 8, 1076 (2017).
- Pretini, V., et al. Red blood cells: chasing interactions. Frontiers in Physiology. 10, 945 (2019).
- Cahalan, S. M., et al. Piezo1 links mechanical forces to red blood cell volume. eLife. 4, e07370 (2015).
- Kuck, L., Peart, J. N., Simmonds, M. J. Piezo1 regulates shear-dependent nitric oxide production in human erythrocytes. American Journal of Physiology. Heart and Circulatory Physiology. 323 (1), H24-H37 (2022).
- Kuck, L., Peart, J. N., Simmonds, M. J. Active modulation of human erythrocyte mechanics. American Journal of Physiology. Cell Physiology. 319 (2), C250-C257 (2020).
- Strader, M. B., et al. Post-translational modification as a response to cellular stress induced by hemoglobin oxidation in sickle cell disease. Scientific Reports. 10 (1), 14218 (2020).
- Pecankova, K., Majek, P., Cermak, J., Dyr, J. E. Posttranslational modifications of red blood cell ghost proteins as "signatures" for distinguishing between low- and high-risk myelodysplastic syndrome patients. Turkish Journal of Haematology. 34 (1), 111-113 (2017).
- Grau, M., et al. High red blood cell nitric oxide synthase activation is not associated with improved vascular function and red blood cell deformability in sickle cell anaemia. British Journal of Haematology. 168 (5), 728-736 (2015).
- Sae-Lee, W., et al. The protein organization of a red blood cell. Cell Reports. 40 (3), 111103 (2022).
- Suhr, F., et al. Moderate exercise promotes human RBC-NOS activity, NO production and deformability through Akt kinase pathway. PLoS One. 7 (9), e45982 (2012).
- Kuck, L., Grau, M., Bloch, W., Simmonds, M. J. Shear stress ameliorates superoxide impairment to erythrocyte deformability with concurrent nitric oxide synthase activation. Frontiers in Physiology. 10, 36 (2019).
- Grau, M., et al. RBC-NOS-dependent S-nitrosylation of cytoskeletal proteins improves RBC deformability. PLoS One. 8 (2), e56759 (2013).
- Simmonds, M. J., Detterich, J. A., Connes, P. Nitric oxide, vasodilation and the red blood cell. Biorheology. 51 (2-3), 121-134 (2014).
- Bor-Kucukatay, M., Wenby, R. B., Meiselman, H. J., Baskurt, O. K. Effects of nitric oxide on red blood cell deformability. American Journal of Physiology. Heart and Circulatory Physiology. 284 (5), H1577-H1584 (2003).
- Suhr, F., et al. Intensive exercise induces changes of endothelial nitric oxide synthase pattern in human erythrocytes. Nitric Oxide: Biology and Chemistry. 20 (2), 95-103 (2009).
- Grau, M., et al. Regulation of red blood cell deformability is independent of red blood cell-nitric oxide synthase under hypoxia. Clinical Hemorheology and Microcirculation. 63 (3), 199-215 (2016).
- Grau, M., Kuck, L., Dietz, T., Bloch, W., Simmonds, M. J. Sub-fractions of red blood cells respond differently to shear exposure following superoxide treatment. Biology. 10 (1), 47 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Ozüyaman, B., Grau, M., Kelm, M., Merx, M. W., Kleinbongard, P. RBC NOS: regulatory mechanisms and therapeutic aspects. Trends in Molecular Medicine. 14 (7), 314-322 (2008).
- Kleinbongard, P., et al. Red blood cells express a functional endothelial nitric oxide synthase. Blood. 107 (7), 2943-2951 (2006).
- McMahon, T. J. Red blood cell deformability, vasoactive mediators, and adhesion. Frontiers in Physiology. 10, 1417 (2019).
- Bizjak, D. A., Brinkmann, C., Bloch, W., Grau, M. Increase in red blood cell-nitric oxide synthase dependent nitric oxide production during red blood cell aging in health and disease: a study on age dependent changes of rheologic and enzymatic properties in red blood cells. PLoS One. 10 (4), 0125206 (2015).
- Di Pietro, N., et al. Nitric oxide synthetic pathway and cGMP levels are altered in red blood cells from end-stage renal disease patients. Molecular and Cellular Biochemistry. 417 (1-2), 155-167 (2016).
- Grau, M., et al. Even patients with mild COVID-19 symptoms after SARS-CoV-2 infection show prolonged altered red blood cell morphology and rheological parameters. Journal of Cellular and Molecular Medicine. 26 (10), 3022-3030 (2022).
- Mozar, A., et al. Red blood cell nitric oxide synthase modulates red blood cell deformability in sickle cell anemia. Clinical Hemorheology and Microcirculation. 64 (1), 47-53 (2016).
- Ulker, P., Gunduz, F., Meiselman, H. J., Baskurt, O. K. Nitric oxide generated by red blood cells following exposure to shear stress dilates isolated small mesenteric arteries under hypoxic conditions. Clinical Hemorheology and Microcirculation. 54 (4), 357-369 (2013).
- Nader, E., et al. Hydroxyurea therapy modulates sickle cell anemia red blood cell physiology: Impact on RBC deformability, oxidative stress, nitrite levels and nitric oxide synthase signalling pathway. Nitric Oxide: Biology and Chemistry. 81, 28-35 (2018).
- Fischer, U. M., Schindler, R., Brixius, K., Mehlhorn, U., Bloch, W. Extracorporeal circulation activates endothelial nitric oxide synthase in erythrocytes. The Annals of Thoracic Surgery. 84 (6), 2000-2003 (2007).
- Horobin, J. T., Sabapathy, S., Kuck, L., Simmonds, M. J. Shear stress and RBC-NOS Serine1177 Phosphorylation in humans: a dose response. Life. 11 (1), 36 (2021).
- Kuck, L., Grau, M., Simmonds, M. J. Recovery time course of erythrocyte deformability following exposure to shear is dependent upon conditioning shear stress. Biorheology. 54 (5-6), 141-152 (2018).
- Grau, M., et al. Effect of acute exercise on RBC deformability and RBC nitric oxide synthase signalling pathway in young sickle cell anaemia patients. Scientific Reports. 9 (1), 11813 (2019).
- Feelisch, M. . Methods in Nitric Oxide Research. , (1998).
- Cortese-Krott, M. M., et al. Human red blood cells at work: identification and visualization of erythrocytic eNOS activity in health and disease. Blood>. 120 (20), 4229-4237 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados