Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
CRISPR-Cas9 Genom-Editierung von Rattenembryonen mit Hilfe des Adeno-assoziierten Virus (AAV) und der Elektroporation von 2-Zell-Embryonen
In diesem Artikel
Zusammenfassung
Dieses Protokoll stellt einen optimierten Ansatz zur Herstellung genetisch veränderter Rattenmodelle dar. Das Adeno-assoziierte Virus (AAV) wird verwendet, um eine DNA-Reparaturvorlage zu liefern, und die Elektroporation wird verwendet, um CRISPR-Cas9-Reagenzien zu verabreichen, um den Genom-Editierungsprozess im 2-Zell-Embryo abzuschließen.
Zusammenfassung
Die Genom-Editing-Technologie wird häufig eingesetzt, um genetisch veränderte Tiere, einschließlich Ratten, zu züchten. Die zytoplasmatische oder pronukleäre Injektion von DNA-Reparaturschablonen und CRISPR-Cas-Reagenzien ist die häufigste Verabreichungsmethode in Embryonen. Diese Art der Mikromanipulation erfordert jedoch den Zugang zu spezialisierten Geräten, ist mühsam und erfordert ein gewisses Maß an technischem Geschick. Darüber hinaus führen Mikroinjektionstechniken aufgrund der mechanischen Belastung des Embryos oft zu einer geringeren Überlebensrate des Embryos. In diesem Protokoll haben wir eine optimierte Methode entwickelt, um große DNA-Reparaturschablonen zu liefern, die in Verbindung mit der CRISPR-Cas9-Genom-Editierung arbeiten, ohne dass eine Mikroinjektion erforderlich ist. Dieses Protokoll kombiniert die AAV-vermittelte DNA-Verabreichung von einzelsträngigen DNA-Spendervorlagen mit der Verabreichung von CRISPR-Cas9-Ribonukleoprotein (RNP) durch Elektroporation, um 2-Zell-Embryonen zu modifizieren. Mit dieser neuartigen Strategie haben wir erfolgreich gezielte Knock-in-Rattenmodelle hergestellt, die DNA-Sequenzen mit einer Größe von 1,2 bis 3,0 kb mit Wirkungsgraden zwischen 42 % und 90 % einfügen.
Einleitung
Die Entwicklung von CRISPR-basierten Genom-Editierungswerkzeugen hat unsere Fähigkeit beschleunigt, neue und anspruchsvollere gentechnisch veränderte Rattenmodelle effizient zu erstellen. Single-guide-RNA wird zusammen mit Cas9-Nuklease kombiniert, um Ribonukleoprotein-Komplexe (RNP) zu bilden, die auf DNA-Sequenzen von Interesse innerhalb des Genoms abzielen und zu doppelsträngigen DNA-Brüchen führen. Da zelluläre DNA-Reparaturmechanismen fehleranfällig sind, werden während des Reparaturprozesses Insertionen und Deletionen (INDELs) eingeführt, die die Funktion eines Zielgens stören können. Wenn eine gewünschte modifizierte DNA-Sequenz (Reparatur-Matrize) zusammen mit Genom-Editing-Reagenzien verabreicht wird, erfolgt das Einfügen der Reparatur-Matrize in die Region, die den doppelsträngigen DNA-Bruch enthält, durch einen Prozess, der als homologiegesteuerte Reparatur (HDR) bezeichnet wird. Dies ist eine effektive Strategie zur Generierung von Tiermodellen mit gezielten DNA-Insertionen/-Substitutionen (Knock-Ins). Eine Einschränkung besteht darin, dass Knock-in-Sequenzen oft sehr groß sind, was nachweislich die Effizienz der Geneditierung verringert und somit die Generierung des gewünschten Modells erschwert1. Zu den Strategien zur Steigerung der Knock-in-Effizienz gehörten die Linearisierung von Reparaturschablonen für doppelsträngige DNA (dsDNA) und einzelsträngige DNA (ssDNA) sowie die chemische Modifikation von DNA-Reparaturschablonen 2,3,4. Darüber hinaus wurde die pronukleäre Mikroinjektion zusammen mit HDR-stimulierenden Verbindungen, die Anwendung von elektrischen Impulsen in Verbindung mit der Mikroinjektion und die zeitgesteuerte Mikroinjektion in 2-Zell-Embryonen versucht 5,6,7. Trotz des Erfolgs einiger dieser Ansätze bleibt der Einbau von DNA-Sequenzen, die größer als 1,0 kb sind, technisch anspruchsvoll.
Die Elektroporation, eine gängige Methode zum Einbringen von Reagenzien in kultivierte Zelllinien, bietet eine Alternative zur Mikroinjektion zur Verabreichung von CRISPR-Cas9-Komponenten in Embryonen. Die Elektroporation von Embryonen, die erstmals in Rattenembryonen8 nachgewiesen wurde, wurde seitdem erfolgreich als Verabreichungsmethode bei Mäusen 9,10,11,12,13, Schweinen 14,15 und anderen Tiermodellorganismen eingesetzt 16,17,18. Die Embryonen, die in dem Medium mit den CRISPR-Cas9-Reagenzien suspendiert sind, werden in eine Küvette oder auf einen Objektträger zwischen zwei Elektroden gegeben und direkten elektrischen Stromimpulsen ausgesetzt. Dadurch entstehen vorübergehende Öffnungen in der Zona pellucida und der Plasmamembran des Embryos, durch die die CRISPR-Cas9-Komponenten in die Embryonen gelangen. Typischerweise werden elektrische "Poring"-Impulse auf mittlerer Ebene verwendet, um die temporären Öffnungen zu erzeugen, gefolgt von elektrischen "Transferimpulsen" auf niedrigerer Ebene, die die Bewegung der negativ geladenen Genom-Editing-Komponenten erleichtern. Die Elektroporation von Embryonen ist effizient, hat einen hohen Durchsatz und ist einfach durchzuführen. Während sich die Elektroporation von Embryonen bei der Einführung kleiner (<200 bp) ssDNA-Reparaturschablonen als sehr erfolgreich erwiesen hat, gibt es nur wenige Berichte über eine erfolgreiche Elektroporation größerer (>1,0 kb) Reparaturschablonen13,19. Diese Größenbeschränkung stellt eine wesentliche Einschränkung der Embryo-Elektroporation für die Erzeugung von Knock-in-Tiermodellen dar, die große Insertionen erfordern.
Im Rahmen der Gentherapie werden Adeno-assoziierte Viren (AAVs) aufgrund ihrer effizienten In-vivo-Infektiosität sowohl von sich teilenden als auch von sich nicht teilenden Zellen, ihrer fehlenden Pathogenität und ihrer seltenen genomischen Integration seit langem als Vehikel für die Verabreichung von genetischem Material verwendet20,21. In jüngster Zeit wurden in weiteren Studien AAVs mit der CRISPR-Cas9-Technologie kombiniert, um DNA-Reparaturschablonen und CRISPR-Reagenzien einzuführen 22,23,24. Dieser Ansatz ermöglicht die Verabreichung größerer DNA-Reparaturschablonen, ohne dass Mikroinjektionstechniken erforderlich sind.
Der HDR-Signalweg ist in den späten S- und G2-Phasen des Zellzyklus aktiver25,26. In in vitro durchgeführten Studien wurden signifikante Steigerungen der Knock-in-Effizienz durch die Verabreichung von CRISPR-Cas9-RNPs und DNA-Reparatur-Templates in G2-synchronisierte Zellen oder durch die Beschränkung des Vorhandenseins des Cas9-Proteins auf späte S- und G2-Phasen unter Verwendung eines Cas9-Geminin-Fusionsproteins2 erreicht. Darüber hinaus gibt es ein wichtiges zygotisches Genomaktivierungsereignis (ZGA), das während der verlängerten G2-Phase des Embryos im 2-Zell-Stadium auftritt und mit einem offenen Chromatinzustand verbunden ist. Es wird spekuliert, dass dies den CRISPR-Cas9-RNPs und Reparaturvorlagen einen besseren Zugang zur genomischen DNA verschafft.
Unser Ziel war es, auf all diesen Beobachtungen aufzubauen, indem wir den AAV-Ansatz mit der Elektroporation von Embryonen kombinierten, um CRISPR-Cas9-RNPs im 2-Zell-Stadium der Embryonalentwicklung einzuführen. Diese Strategie nutzt die größere DNA-Reparatur-Template-Lieferkapazität von AAV, die technische Leichtigkeit der Elektroporation und den optimaleren 2-Zell-Zeitpunkt für die genomische Zugänglichkeit während der Embryonalentwicklung, um eine effiziente Methode für die gezielte Genmanipulation von DNA-Insertionen zu schaffen. Wie in diesem Protokoll hervorgehoben, ermöglicht unsere optimierte Methode die Herstellung von gezielten Knock-in-Rattenmodellen mit Insertionen von DNA-Sequenzen mit einer Größe von 1,2 bis 3,0 kb, ohne dass Mikroinjektionstechniken erforderlich sind.
Protokoll
Alle Versuchsverfahren wurden vom Institutional Animal Care and Use Committee der University of Missouri (ACUC-Protokoll #25580) genehmigt und gemäß den Richtlinien des Leitfadens für die Verwendung und Pflege von Labortieren durchgeführt.
1. AAV-vermittelte Lieferung von DNA-Reparaturschablonen
- Entwerfen Sie das DNA-Konstrukt so, dass es die gewünschte Knock-in-Sequenz zwischen zwei Homologiearmen enthält. Typische Homologie-Armlängen sind 300-500 bp lang. Stellen Sie sicher, dass das gesamte DNA-Reparatur-Template (Homologiearme + gewünschte Knock-in-Sequenz) von invertierten terminalen Wiederholungen (ITRs) flankiert wird, indem Sie in ein geeignetes Plasmid-Rückgrat klonieren und in rekombinantes ssAAV1 oder ssAAV6 (Serotypen 1 oder 6) verpacken.
HINWEIS: Wenn die sgRNA-Zielsequenz in der DNA-Reparaturvorlage vorhanden ist, ist es wichtig, stille Mutationen zu entwickeln, um diese Sequenz zu stören und so eine CRISPR-Cas9-vermittelte Editierung des korrekt anvisierten Allels zu verhindern.
VORSICHT: Rekombinante virale AAV-Vektoren sind nicht integrierend und als BSL-1 klassifiziert. Für das Pipettieren und die Handhabung von Embryonen, die dem Virus ausgesetzt waren, werden aseptische Standardtechniken verwendet. - Künstlich hergestelltes ssAAV1 oder ssAAV6 (verpackt mit DNA-Reparatur-Template) zu 30 μl KSOM-R-Medium27,28 bis zu einer Endkonzentration von 3 × 107 Genomkopien (GC)/μl unter Mineralöl in 35 mm Petrischale geben.
- Platzieren Sie Zygoten mit sichtbaren Vorkernen in das KSOM-R-Medium, das technisch hergestelltes ssAAV enthält.
HINWEIS: Bei Verwendung dieses Protokolls ist es nicht erforderlich, die Zona pellucida zu verdünnen, da die AAV-Serotypen 1 und 6 in der Lage sind, die DNA-Reparaturschablonen für eine effiziente Verabreichung von DNA-Reparaturschablonen leicht zu passieren. - Bei 37 °C mit 5 % CO2 und maximaler Luftfeuchtigkeit für 18-20 h inkubieren, um eine ausreichende Virustransduktion zu ermöglichen.
2. Vorbereitung der Elektroporation
- Am nächsten Tag wird eine Elektroporationsmischung mit 100 ng/μl sgRNA und 100 ng/μl Cas9-Protein, verdünnt in Opti-MEM-Medien in einem sterilen RNase-freien Röhrchen hergestellt. Vorsichtig mischen.
- 10 Minuten bei Raumtemperatur inkubieren, um die RNP-Bildung zu ermöglichen.
- Schalten Sie während der Inkubationszeit den Elektroporator ein und schließen Sie die Kabel an eine Glasschieberelektrode mit einem Abstand von 1,5 mm an.
- Stellen Sie die Elektroporationsparameter wie in Tabelle 1 beschrieben ein.
3. Elektroporation von 2-Zell-Embryonen
- Pipettieren Sie vorsichtig 5 μl der CRISPR-Elektroporationsmischung zwischen die beiden Elektroden auf dem Objektträger, wobei Sie darauf achten müssen, dass keine Luftblasen in die Lösung gelangen.
- Entfernen Sie den Embryo im 2-Zell-Stadium aus der AAV-Lösung. Bringen Sie Embryonen im 2-Zell-Stadium in die Mischung, ohne dass sie die Seiten der Elektroden berühren. Dies geschieht, um eine Lyse zu vermeiden.
- Drücken Sie die Ohm-Taste , um die Impedanz zu messen. Stellen Sie sicher, dass die Impedanz zwischen 0,100 und 0,300 kΩ liegt. Das Reaktionsvolumen ändert die Impedanz (Volumen hinzufügen, um die Impedanz zu verringern, und Volumen subtrahieren, um die Impedanz zu erhöhen).
- Drücken Sie auf Start. Der Vorgang dauert nur wenige Sekunden.
- Die Embryonen werden unmittelbar nach der Elektroporation entnommen und 3 Mal in frischen 30-μl-Tropfen KSOM-R-Medien gewaschen.
- Die Embryonen werden wieder in einen 37 °C warmen Inkubator mit 5 %CO2 und maximaler Luftfeuchtigkeit für die In-vitro-Kultur in 500 μl Tropfen KSOM-R-Medien unter Mineralöl gelegt. Alternativ können Embryonen chirurgisch auf eine 1,5 Tage postkoitum (dpc) pseudoschwangere weibliche Ratte übertragen werden, um lebende Nachkommen zu zeugen.
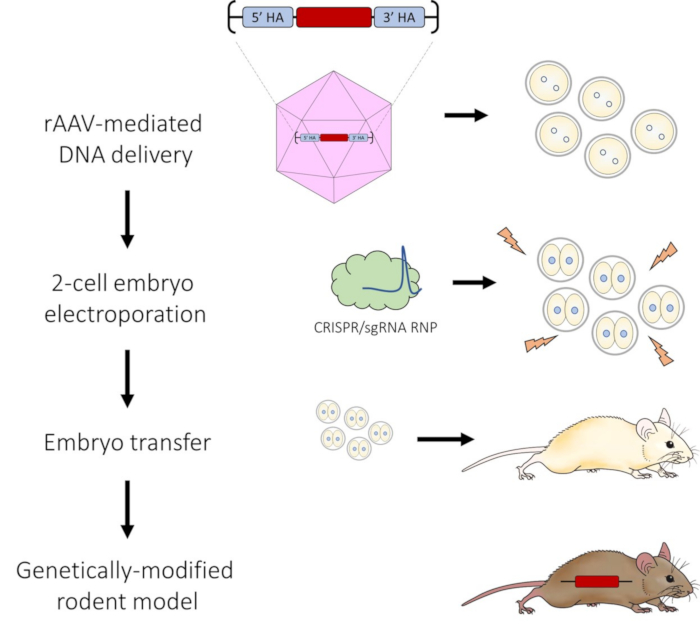
Abbildung 1: Schematische Darstellung der AAV-vermittelten DNA-Verabreichung und der 2-Zell-Embryo-Elektroporations-Pipeline für die CRISPR-Cas9-Genom-Editierung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Gemäß dem Protokoll gibt es eine effektive AAV-vermittelte Verabreichung der DNA-Reparaturvorlage, die eine hocheffiziente HDR ermöglicht, nachdem 2-Zell-Embryonen mit CRISPR-Cas9-RNPs elektroporiert wurden. Wie im Video gezeigt, führt ein erfolgreicher Elektroporationsprozess dazu, dass sich auf jeder Elektrode Blasen bilden (Abbildung 2C) und die Impedanz im Bereich von 0,100 und 0,300 kΩ bleibt. Nach der Elektroporation ist es nicht ungewöhnlich, das...
Diskussion
Das Genom-Editing-System CRISPR-Cas hat die Gentechnik revolutioniert, indem es die effiziente Herstellung sowohl einfacher als auch komplexer, maßgeschneiderter genetischer Veränderungen in einer Vielzahl von Tierarten ermöglicht. Häufige Verfeinerungen und Verbesserungen der Techniken im Zusammenhang mit der Genom-Editierung erhöhen seine Vielseitigkeit. Hier haben wir einen neuen Ansatz beschrieben, der die ssAAV-vermittelte DNA-Verabreichung zusammen mit der Elektroporation von 2-Zell-Embryonen von CRISPR-Cas9-R...
Offenlegungen
Die Autoren haben keine Interessenkonflikte zu deklarieren.
Danksagungen
Die Autoren bedanken sich bei Nolan Davis für die Unterstützung bei der Videografie und Videobearbeitung.
Materialien
| Name | Company | Catalog Number | Comments |
| Leads with alligator clips for electrodes | NepaGene | C117 | |
| Mineral oil | MilliporeSigma | M5310 | |
| NepaGene21 Super Electroporator | NepaGene | NEPA21 | |
| Platinum plate electrodes on slide glass | NepaGene | CUY501P1-1.5 | |
| PURedit Cas9 Protein | MilliporeSigma | PECAS9 | |
| sgRNA (chemically-modified) | Synthego | N/A | |
| ssAAV1 or ssAAV6 packaged DNA repair template | Vectorbuilder | N/A |
Referenzen
- Lau, C. H., Tin, C., Suh, Y. CRISPR-based strategies for targeted transgene knock-in and gene correction. Fac Rev. 9, 20 (2020).
- Gutschner, T., Haemmerle, M., Genovese, G., Draetta, G. F., Chin, L. Post-translational Regulation of Cas9 during G1 Enhances Homology-Directed Repair. Cell Rep. 14 (6), 1555-1566 (2016).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biol. 18 (1), 35 (2017).
- Miura, H., Quadros, R. M., Gurumurthy, C. B., Ohtsuka, M. Easi-CRISPR for creating knock-in and conditional knockout mouse models using long ssDNA donors. Nat Protoc. 13 (1), 195-215 (2018).
- Gu, B., Posfai, E., Gertsenstein, M., Rossant, J. Efficient generation of large-fragment knock-in mouse models using 2-cell (2C)-homologous recombination (HR)-CRISPR. Curr Protoc Mouse Biol. 10 (1), e67 (2020).
- Gu, B., Posfai, E., Rossant, J. Efficient generation of targeted large insertions by microinjection into two-cell-stage mouse embryos. Nat Biotechnol. 36 (7), 632-637 (2018).
- Kurihara, T., et al. DNA repair protein RAD51 enhances the CRISPR/Cas9-mediated knock-in efficiency in brain neurons. Biochem Biophys Res Commun. 524 (3), 621-628 (2020).
- Kaneko, T., Sakuma, T., Yamamoto, T., Mashimo, T. Simple knockout by electroporation of engineered endonucleases into intact rat embryos. Sci Rep. 4, 6382 (2014).
- Chen, S., Lee, B., Lee, A. Y., Modzelewski, A. J., He, L. Highly efficient mouse genome editing by CRISPR ribonucleoprotein electroporation of zygotes. J Biol Chem. 291 (28), 14457-14467 (2016).
- Hashimoto, M., Yamashita, Y., Takemoto, T. Electroporation of Cas9 protein/sgRNA into early pronuclear zygotes generates non-mosaic mutants in the mouse. Dev Biol. 418 (1), 1-9 (2016).
- Wang, W., et al. Delivery of Cas9 protein into mouse zygotes through a series of electroporation dramatically increases the efficiency of model creation. J Genet Genomics. 43 (5), 319-327 (2016).
- Troder, S. E., et al. An optimized electroporation approach for efficient CRISPR/Cas9 genome editing in murine zygotes. PLoS One. 13 (5), e0196891 (2018).
- Teixeira, M., et al. Electroporation of mice zygotes with dual guide RNA/Cas9 complexes for simple and efficient cloning-free genome editing. Sci Rep. 8 (1), 474 (2018).
- Tanihara, F., et al. Efficient generation of GGTA1-deficient pigs by electroporation of the CRISPR/Cas9 system into in vitro-fertilized zygotes. BMC Biotechnol. 20 (1), 40 (2020).
- Tanihara, F., et al. Generation of CD163-edited pig via electroporation of the CRISPR/Cas9 system into porcine in vitro-fertilized zygotes. Anim Biotechnol. 32 (2), 147-154 (2021).
- Camargo, L. S. A., Owen, J. R., Van Eenennaam, A. L., Ross, P. J. Efficient one-step knockout by electroporation of ribonucleoproteins into zona-intact bovine embryos. Front Genet. 11, 570069 (2020).
- Lin, J. C., Van Eenennaam, A. L. Electroporation-mediated genome editing of livestock zygotes. Front Genet. 12, 648482 (2021).
- Betters, E., Charney, R. M., Garcia-Castro, M. I. Electroporation and in vitro culture of early rabbit embryos. Data Brief. 21, 316-320 (2018).
- Miyasaka, Y., et al. CLICK: one-step generation of conditional knockout mice. BMC Genomics. 19 (1), 318 (2018).
- Epstein, B. E., Schaffer, D. V. Combining engineered nucleases with adeno-associated viral vectors for therapeutic gene editing. Adv Exp Med Biol. 1016, 29-42 (2017).
- Gaj, T., Epstein, B. E., Schaffer, D. V. Genome engineering using adeno-associated virus: basic and clinical research applications. Mol Ther. 24 (3), 458-464 (2016).
- Chen, S., et al. CRISPR-READI: Efficient generation of knockin mice by CRISPR RNP electroporation and AAV donor infection. Cell Rep. 27 (13), 3780-3789 (2019).
- Mizuno, N., et al. Intra-embryo gene cassette knockin by CRISPR/CAS9-mediated genome editing with adeno-associated viral vector. iScience. 9, 286-297 (2018).
- Davis, D. J., et al. Efficient DNA knock-in using AAV-mediated delivery with 2-cell embryo CRISPR-Cas9 electroporation. Front Genome Ed. 5, 1256451 (2023).
- Smirnikhina, S. A., Zaynitdinova, M. I., Sergeeva, V. A., Lavrov, A. V. Improving homology-directed repair in genome editing experiments by influencing the cell cycle. Int J Mol Sci. 23 (11), (2022).
- Takata, M., et al. Homologous recombination and non-homologous end-joining pathways of DNA double-strand break repair have overlapping roles in the maintenance of chromosomal integrity in vertebrate cells. EMBO J. 17 (18), 5497-5508 (1998).
- Men, H., Stone, B. J., Bryda, E. C. Media optimization to promote rat embryonic development to the blastocyst stage in vitro. Theriogenology. 151, 81-85 (2020).
- Men, H., Amos-Landgraf, J. M., Bryda, E. C., Franklin, C. L. KSOM-R supports both mouse and rat preimplantation embryo development in vitro. Theriogenology. 198, 69-74 (2023).
- Romeo, C., et al. AAV diffuses across zona pellucida for effortless gene delivery to fertilized eggs. Biochem Biophys Res Commun. 526 (1), 85-90 (2020).
- Fulka, J., Moor, R. M., Fulka, J. Electrofusion of mammalian oocytes and embryonic cells. Methods Mol Biol. 48, 309-316 (1995).
- Rems, L., et al. Cell electrofusion using nanosecond electric pulses. Sci Rep. 3, 3382 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten