É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
CRISPR-Cas9 Edição Genômica de Embriões de Rato usando Vírus Adenoassociado (AAV) e Eletroporação de Embriões de 2 Células
Neste Artigo
Resumo
Este protocolo apresenta uma abordagem otimizada para a produção de modelos de ratos geneticamente modificados. O vírus adenoassociado (AAV) é usado para entregar um modelo de reparo de DNA, e a eletroporação é usada para entregar reagentes CRISPR-Cas9 para completar o processo de edição do genoma em embrião de 2 células.
Resumo
A tecnologia de edição do genoma é amplamente utilizada para produzir animais geneticamente modificados, incluindo ratos. A injeção citoplasmática ou pronuclear de moldes de reparo de DNA e reagentes CRISPR-Cas é o método de liberação mais comum em embriões. No entanto, esse tipo de micromanipulação requer acesso a equipamentos especializados, é trabalhoso e requer um certo nível de habilidade técnica. Além disso, as técnicas de microinjeção frequentemente resultam em menor sobrevivência do embrião devido ao estresse mecânico sobre o embrião. Neste protocolo, desenvolvemos um método otimizado para entregar grandes modelos de reparo de DNA para trabalhar em conjunto com a edição do genoma CRISPR-Cas9 sem a necessidade de microinjeção. Este protocolo combina a entrega de DNA mediada por AAV de modelos de doadores de DNA de fita simples juntamente com a entrega de ribonucleoproteína CRISPR-Cas9 (RNP) por eletroporação para modificar embriões de 2 células. Usando esta nova estratégia, produzimos com sucesso modelos de ratos knock-in direcionados carregando a inserção de sequências de DNA de 1,2 a 3,0 kb de tamanho com eficiências entre 42% e 90%.
Introdução
O desenvolvimento de ferramentas de edição do genoma baseadas em CRISPR acelerou nossa capacidade de gerar eficientemente novos e mais sofisticados modelos de ratos geneticamente modificados. O RNA guia-único, juntamente com a nuclease Cas9, é combinado para formar complexos de ribonucleoproteína (RNP) que têm como alvo sequências de DNA de interesse dentro do genoma e resultam em quebras de DNA de fita dupla. Como os mecanismos de reparo do DNA celular são propensos a erros, inserções e deleções (INDELs) são introduzidas durante o processo de reparo que podem interromper a função de um gene alvo. Quando há co-entrega de uma sequência de DNA projetada desejada (modelo de reparo) juntamente com reagentes de edição do genoma, a inserção do molde de reparo na região que contém a quebra de DNA de fita dupla ocorre por meio de um processo chamado reparo dirigido por homologia (HDR). Esta é uma estratégia eficaz para gerar modelos animais com inserções/substituições de DNA direcionadas (knock-ins). Uma limitação é que as sequências knock-in são frequentemente grandes em tamanho, o que demonstrou reduzir a eficiência da edição gênica, tornando a geração do modelo desejado mais difícil1. Estratégias para aumentar a eficiência do knock-in incluíram a linearização de modelos de reparo de DNA de fita dupla (dsDNA) e DNA de fita simples (ssDNA) e modificação química de modelos de reparo de DNA 2,3,4. Além disso, a microinjeção pronuclear juntamente com compostos estimuladores de HDR, a aplicação de pulsos elétricos em conjunto com a microinjeção e a microinjeção cronometrada em embriões de 2 células têm sido tentadas 5,6,7. Apesar do sucesso de algumas dessas abordagens, a incorporação de sequências de DNA maiores que 1,0 kb permanece tecnicamente desafiadora.
A eletroporação, que é um método comum para introduzir reagentes em linhagens celulares cultivadas, oferece uma alternativa à microinjeção para a entrega de componentes CRISPR-Cas9 em embriões. A eletroporação embrionária, demonstrada pela primeira vez em embriões de ratos8, tem sido utilizada com sucesso como método de liberação em camundongos 9,10,11,12,13, suínos 14,15 e outros organismos animaismodelo 16,17,18 . Os embriões, suspensos no meio contendo reagentes CRISPR-Cas9, são colocados em uma cubeta ou em uma lâmina de vidro entre dois eletrodos e submetidos a pulsos diretos de correntes elétricas. Isso cria aberturas transitórias na zona pelúcida e na membrana plasmática do embrião através das quais os componentes CRISPR-Cas9 entram nos embriões. Normalmente, pulsos elétricos de "poring" de nível médio são usados para criar as aberturas temporárias seguidas por "pulsos de transferência" elétricos de nível inferior que facilitam o movimento dos componentes de edição do genoma carregados negativamente. A eletroporação embrionária é eficiente, tem alto rendimento e é fácil de executar. No entanto, embora a eletroporação embrionária tenha se mostrado altamente bem-sucedida para a introdução de pequenos (<200 pb) modelos de reparo de ssDNA, há poucos relatos de eletroporação bem-sucedida de modelos de reparo maiores (>1,0 kb)13,19. Essa restrição de tamanho representa uma grande limitação da eletroporação embrionária por gerar modelos animais knock-in que requerem grandes inserções.
No contexto da terapia gênica, os vírus adenoassociados (AAVs) têm sido usados há muito tempo como veículos para liberar material genético devido à sua eficiente infectividade in vivo de células em divisão e não em divisão, ausência de patogenicidade e rara integração genômica20,21. Recentemente, mais estudos combinaram AAVs com a tecnologia CRISPR-Cas9 para introduzir modelos de reparo de DNA e reagentes CRISPR 22,23,24. Essa abordagem permite a entrega de modelos maiores de reparo de DNA sem a necessidade de técnicas de microinjeção.
A via HDR é mais ativa nas fases S tardia e G2 do ciclocelular25,26. Em estudos realizados in vitro, aumentos significativos na eficiência de knock-in foram alcançados pela entrega de RNPs CRISPR-Cas9 e modelos de reparo de DNA em células sincronizadas com G2 ou pela restrição da presença da proteína Cas9 às fases S e G2 tardias usando uma proteína de fusão Cas9-Geminin2. Além disso, há um grande evento de ativação do genoma zigótico (ZGA) que ocorre durante a fase G2 estendida do embrião em estágio de 2 células, e isso está associado a um estado de cromatina aberta. Especula-se que isso proporcione aos RNPs CRISPR-Cas9 e modelos de reparo maior acessibilidade ao DNA genômico.
Nosso objetivo foi desenvolver todas essas observações, combinando a abordagem AAV com eletroporação embrionária para introduzir RNPs CRISPR-Cas9 no estágio de 2 células do desenvolvimento embrionário. Essa estratégia aproveita a maior capacidade de entrega do molde de reparo de DNA do AAV, a facilidade técnica de eletroporação e o ponto de tempo de 2 células mais ideal para acessibilidade genômica durante o desenvolvimento do embrião para criar um método eficiente para engenharia genética direcionada de inserções de DNA. Como destacado neste protocolo, nosso método otimizado permite a produção de modelos de ratos knock-in direcionados carregando inserções de sequências de DNA de 1,2 a 3,0 kb de tamanho sem a necessidade de técnicas de microinjeção.
Protocolo
Todos os procedimentos experimentais foram aprovados pelo Comitê Institucional de Cuidados e Uso de Animais da Universidade de Missouri (protocolo ACUC #25580) e foram realizados de acordo com as diretrizes estabelecidas no Guia para o Uso e Cuidado de Animais de Laboratório.
1. Entrega do modelo de reparo de DNA mediado por AAV
- Projete a construção de DNA para conter a sequência de knock-in desejada entre dois braços de homologia. Os comprimentos típicos dos braços de homologia são de 300-500 pb de comprimento. Certifique-se de que todo o molde de reparo de DNA (braços de homologia + sequência de knock-in desejada) seja flanqueado por repetições terminais invertidas (ITRs) por clonagem em um backbone plasmidial apropriado e empacotamento em ssAAV1 ou ssAAV6 recombinantes (sorotipos 1 ou 6).
NOTA: Se a sequência alvo do sgRNA estiver presente no modelo de reparo de DNA, é importante projetar mutações silenciosas para interromper essa sequência, impedindo assim a edição mediada por CRISPR-Cas9 do alelo corretamente visado.
CUIDADO: Os vetores virais AAV recombinantes não são integradores e classificados como BSL-1. Técnicas assépticas padrão são usadas para pipetar e manipular embriões que foram expostos a vírus. - Adicionar ssAAV1 ou ssAAV6 projetados (embalados com molde de reparo de DNA) a 30 μL de meio KSOM-R27,28 para uma concentração final de 3 × 107 cópias do genoma (GC)/μL sob óleo mineral em placa de Petri de 35 mm.
- Coloque zigotos com pró-núcleos visíveis no meio KSOM-R contendo ssAAV projetado.
NOTA: Usando este protocolo, não há necessidade de afinar a zona pelúcida, pois os sorotipos 1 e 6 do AAV são capazes de passar prontamente para a entrega eficiente do molde de reparo de DNA. - Incubar a 37 °C com 5% de CO2 e humidade máxima durante 18-20 h para permitir uma transdução viral suficiente.
2. Preparação da eletroporação
- No dia seguinte, fazer uma mistura de eletroporação contendo 100 ng/μL de sgRNA e 100 ng/μL de proteína Cas9 diluída em meio Opti-MEM em um tubo estéril livre de RNase. Misture delicadamente.
- Incubar à temperatura ambiente por 10 min para permitir a formação da RNP.
- Durante o tempo de incubação, ligue o eletroporator e conecte os cabos a um eletrodo deslizante de vidro com folga de 1,5 mm.
- Defina os parâmetros de eletroporação conforme descrito na Tabela 1.
3. Eletroporação embrionária de 2 células
- Pipetar suavemente 5 μL da mistura de eletroporação CRISPR entre os dois eletrodos na lâmina de vidro, tomando cuidado para não introduzir bolhas de ar na solução.
- Remova o embrião em estágio de 2 células da solução de AAV. Alinhe embriões em estágio de 2 células na mistura sem permitir que eles toquem nas laterais dos eletrodos. Isso é feito para evitar a lise.
- Pressione o botão Ohm para medir a impedância. Verifique se a impedância está entre 0,100 e 0,300 kΩ. O volume da reação irá alterar a impedância (adicionar volume para diminuir a impedância e subtrair volume para aumentar a impedância).
- Pressione Iniciar. O processo leva alguns segundos para ser concluído.
- Remover os embriões imediatamente após a eletroporação e lavar 3 vezes em gotas frescas de 30 μL do meio KSOM-R.
- Colocar os embriões novamente em estufa a 37 °C com 5% de CO2 e umidade máxima para cultivo in vitro em gotas de 500 μL do meio KSOM-R sob óleo mineral. Alternativamente, transfira cirurgicamente embriões para uma rata pseudogestante pós-coito (dpc) de 1,5 dia para produzir descendentes vivos.
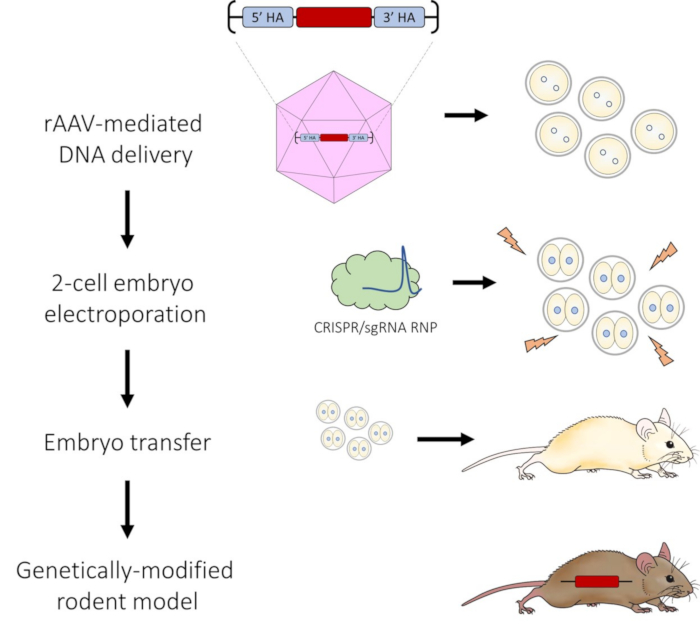
Figura 1: Esquema da entrega de DNA mediada por AAV e pipeline de eletroporação embrionária de 2 células para edição do genoma CRISPR-Cas9. Clique aqui para ver uma versão ampliada desta figura.
Resultados
Seguindo o protocolo, há uma entrega efetiva mediada por AAV do molde de reparo de DNA, permitindo HDR altamente eficiente após embriões de 2 células serem eletroporados com RNPs CRISPR-Cas9. Como mostrado no vídeo, um processo de eletroporação bem-sucedido resulta na formação de bolhas em cada eletrodo (Figura 2C) e a impedância permanece dentro da faixa de 0,100 e 0,300 kΩ. Após a eletroporação, não é incomum que os embriões inchem preenche...
Discussão
O sistema de edição do genoma CRISPR-Cas revolucionou o campo da engenharia genética ao permitir a produção eficiente de modificações genéticas simples e complexas e personalizadas em uma variedade de espécies animais. Refinamentos frequentes e melhorias nas técnicas associadas à edição do genoma aumentam sua versatilidade. Aqui descrevemos uma nova abordagem usando a entrega de DNA mediada por ssAAV juntamente com a eletroporação embrionária de 2 células de reagentes CRISPR-Cas9 em embriões de roedores...
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
Os autores gostariam de agradecer a Nolan Davis pela assistência com videografia e edição de vídeo.
Materiais
| Name | Company | Catalog Number | Comments |
| Leads with alligator clips for electrodes | NepaGene | C117 | |
| Mineral oil | MilliporeSigma | M5310 | |
| NepaGene21 Super Electroporator | NepaGene | NEPA21 | |
| Platinum plate electrodes on slide glass | NepaGene | CUY501P1-1.5 | |
| PURedit Cas9 Protein | MilliporeSigma | PECAS9 | |
| sgRNA (chemically-modified) | Synthego | N/A | |
| ssAAV1 or ssAAV6 packaged DNA repair template | Vectorbuilder | N/A |
Referências
- Lau, C. H., Tin, C., Suh, Y. CRISPR-based strategies for targeted transgene knock-in and gene correction. Fac Rev. 9, 20 (2020).
- Gutschner, T., Haemmerle, M., Genovese, G., Draetta, G. F., Chin, L. Post-translational Regulation of Cas9 during G1 Enhances Homology-Directed Repair. Cell Rep. 14 (6), 1555-1566 (2016).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biol. 18 (1), 35 (2017).
- Miura, H., Quadros, R. M., Gurumurthy, C. B., Ohtsuka, M. Easi-CRISPR for creating knock-in and conditional knockout mouse models using long ssDNA donors. Nat Protoc. 13 (1), 195-215 (2018).
- Gu, B., Posfai, E., Gertsenstein, M., Rossant, J. Efficient generation of large-fragment knock-in mouse models using 2-cell (2C)-homologous recombination (HR)-CRISPR. Curr Protoc Mouse Biol. 10 (1), e67 (2020).
- Gu, B., Posfai, E., Rossant, J. Efficient generation of targeted large insertions by microinjection into two-cell-stage mouse embryos. Nat Biotechnol. 36 (7), 632-637 (2018).
- Kurihara, T., et al. DNA repair protein RAD51 enhances the CRISPR/Cas9-mediated knock-in efficiency in brain neurons. Biochem Biophys Res Commun. 524 (3), 621-628 (2020).
- Kaneko, T., Sakuma, T., Yamamoto, T., Mashimo, T. Simple knockout by electroporation of engineered endonucleases into intact rat embryos. Sci Rep. 4, 6382 (2014).
- Chen, S., Lee, B., Lee, A. Y., Modzelewski, A. J., He, L. Highly efficient mouse genome editing by CRISPR ribonucleoprotein electroporation of zygotes. J Biol Chem. 291 (28), 14457-14467 (2016).
- Hashimoto, M., Yamashita, Y., Takemoto, T. Electroporation of Cas9 protein/sgRNA into early pronuclear zygotes generates non-mosaic mutants in the mouse. Dev Biol. 418 (1), 1-9 (2016).
- Wang, W., et al. Delivery of Cas9 protein into mouse zygotes through a series of electroporation dramatically increases the efficiency of model creation. J Genet Genomics. 43 (5), 319-327 (2016).
- Troder, S. E., et al. An optimized electroporation approach for efficient CRISPR/Cas9 genome editing in murine zygotes. PLoS One. 13 (5), e0196891 (2018).
- Teixeira, M., et al. Electroporation of mice zygotes with dual guide RNA/Cas9 complexes for simple and efficient cloning-free genome editing. Sci Rep. 8 (1), 474 (2018).
- Tanihara, F., et al. Efficient generation of GGTA1-deficient pigs by electroporation of the CRISPR/Cas9 system into in vitro-fertilized zygotes. BMC Biotechnol. 20 (1), 40 (2020).
- Tanihara, F., et al. Generation of CD163-edited pig via electroporation of the CRISPR/Cas9 system into porcine in vitro-fertilized zygotes. Anim Biotechnol. 32 (2), 147-154 (2021).
- Camargo, L. S. A., Owen, J. R., Van Eenennaam, A. L., Ross, P. J. Efficient one-step knockout by electroporation of ribonucleoproteins into zona-intact bovine embryos. Front Genet. 11, 570069 (2020).
- Lin, J. C., Van Eenennaam, A. L. Electroporation-mediated genome editing of livestock zygotes. Front Genet. 12, 648482 (2021).
- Betters, E., Charney, R. M., Garcia-Castro, M. I. Electroporation and in vitro culture of early rabbit embryos. Data Brief. 21, 316-320 (2018).
- Miyasaka, Y., et al. CLICK: one-step generation of conditional knockout mice. BMC Genomics. 19 (1), 318 (2018).
- Epstein, B. E., Schaffer, D. V. Combining engineered nucleases with adeno-associated viral vectors for therapeutic gene editing. Adv Exp Med Biol. 1016, 29-42 (2017).
- Gaj, T., Epstein, B. E., Schaffer, D. V. Genome engineering using adeno-associated virus: basic and clinical research applications. Mol Ther. 24 (3), 458-464 (2016).
- Chen, S., et al. CRISPR-READI: Efficient generation of knockin mice by CRISPR RNP electroporation and AAV donor infection. Cell Rep. 27 (13), 3780-3789 (2019).
- Mizuno, N., et al. Intra-embryo gene cassette knockin by CRISPR/CAS9-mediated genome editing with adeno-associated viral vector. iScience. 9, 286-297 (2018).
- Davis, D. J., et al. Efficient DNA knock-in using AAV-mediated delivery with 2-cell embryo CRISPR-Cas9 electroporation. Front Genome Ed. 5, 1256451 (2023).
- Smirnikhina, S. A., Zaynitdinova, M. I., Sergeeva, V. A., Lavrov, A. V. Improving homology-directed repair in genome editing experiments by influencing the cell cycle. Int J Mol Sci. 23 (11), (2022).
- Takata, M., et al. Homologous recombination and non-homologous end-joining pathways of DNA double-strand break repair have overlapping roles in the maintenance of chromosomal integrity in vertebrate cells. EMBO J. 17 (18), 5497-5508 (1998).
- Men, H., Stone, B. J., Bryda, E. C. Media optimization to promote rat embryonic development to the blastocyst stage in vitro. Theriogenology. 151, 81-85 (2020).
- Men, H., Amos-Landgraf, J. M., Bryda, E. C., Franklin, C. L. KSOM-R supports both mouse and rat preimplantation embryo development in vitro. Theriogenology. 198, 69-74 (2023).
- Romeo, C., et al. AAV diffuses across zona pellucida for effortless gene delivery to fertilized eggs. Biochem Biophys Res Commun. 526 (1), 85-90 (2020).
- Fulka, J., Moor, R. M., Fulka, J. Electrofusion of mammalian oocytes and embryonic cells. Methods Mol Biol. 48, 309-316 (1995).
- Rems, L., et al. Cell electrofusion using nanosecond electric pulses. Sci Rep. 3, 3382 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados