Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Édition du génome CRISPR-Cas9 d’embryons de rats à l’aide d’un virus adéno-associé (AAV) et d’une électroporation d’embryons à 2 cellules
Dans cet article
Résumé
Ce protocole présente une approche optimisée pour la production de modèles de rats génétiquement modifiés. Le virus adéno-associé (AAV) est utilisé pour fournir une matrice de réparation de l’ADN, et l’électroporation est utilisée pour délivrer des réactifs CRISPR-Cas9 afin de compléter le processus d’édition du génome dans un embryon à 2 cellules.
Résumé
La technologie d’édition du génome est largement utilisée pour produire des animaux génétiquement modifiés, y compris des rats. L’injection cytoplasmique ou pronucléaire de matrices de réparation de l’ADN et de réactifs CRISPR-Cas est la méthode d’administration la plus courante dans les embryons. Cependant, ce type de micromanipulation nécessite l’accès à du matériel spécialisé, est laborieux et nécessite un certain niveau de compétence technique. De plus, les techniques de micro-injection entraînent souvent une survie embryonnaire plus faible en raison du stress mécanique subi par l’embryon. Dans ce protocole, nous avons développé une méthode optimisée pour fournir de grands modèles de réparation de l’ADN qui fonctionnent en conjonction avec l’édition du génome CRISPR-Cas9 sans avoir besoin de micro-injection. Ce protocole combine l’administration d’ADN médiée par AAV de modèles de donneurs d’ADN simple brin avec l’administration de la ribonucléoprotéine (RNP) CRISPR-Cas9 par électroporation pour modifier les embryons à 2 cellules. À l’aide de cette nouvelle stratégie, nous avons réussi à produire des modèles ciblés de rats knock-in portant l’insertion de séquences d’ADN d’une taille de 1,2 à 3,0 kb avec des efficacités comprises entre 42 % et 90 %.
Introduction
Le développement d’outils d’édition du génome basés sur CRISPR a accéléré notre capacité à générer efficacement de nouveaux modèles de rats génétiquement modifiés plus sophistiqués. L’ARN guide unique, ainsi que la nucléase Cas9, sont combinés pour former des complexes ribonucléoprotéiques (RNP) qui ciblent les séquences d’ADN d’intérêt dans le génome et entraînent des cassures d’ADN double brin. Étant donné que les mécanismes de réparation de l’ADN cellulaire sont sujets aux erreurs, des insertions et des délétions (INDEL) sont introduites au cours du processus de réparation, ce qui peut perturber la fonction d’un gène cible. Lorsqu’il y a co-livraison d’une séquence d’ADN modifiée souhaitée (matrice de réparation) avec des réactifs d’édition du génome, l’insertion de la matrice de réparation dans la région contenant la cassure de l’ADN double brin se produit par un processus appelé réparation dirigée par homologie (HDR). Il s’agit d’une stratégie efficace pour générer des modèles animaux avec des insertions/substitutions d’ADN ciblées (knock-ins). L’une des limites est que les séquences knock-in sont souvent de grande taille, ce qui réduit l’efficacité de l’édition de gènes, ce qui rend la génération du modèle souhaité plus difficile1. Les stratégies visant à accroître l’efficacité de la frappe ont inclus la linéarisation des modèles de réparation de l’ADN double brin (ADNdb) et de l’ADN simple brin (ADNss) et la modification chimique des modèles de réparation de l’ADN 2,3,4. De plus, la micro-injection pronucléaire avec des composés stimulant le HDR, l’application d’impulsions électriques en conjonction avec la micro-injection et la micro-injection chronométrée dans des embryons à 2 cellules ont toutes été tentées 5,6,7. Malgré le succès de certaines de ces approches, l’incorporation de séquences d’ADN de plus de 1,0 kb reste techniquement difficile.
L’électroporation, qui est une méthode courante d’introduction de réactifs dans les lignées cellulaires en culture, offre une alternative à la micro-injection pour délivrer des composants CRISPR-Cas9 dans les embryons. L’électroporation embryonnaire, démontrée pour la première fois chez des embryons de rat8, a depuis été utilisée avec succès comme méthode d’administration chez les souris 9,10,11,12,13, les porcs 14,15 et d’autres organismes modèles animaux 16,17,18. Les embryons, en suspension dans le milieu contenant les réactifs CRISPR-Cas9, sont placés dans une cuvette ou sur une lame de verre entre deux électrodes et soumis à des impulsions directes de courants électriques. Cela crée des ouvertures transitoires dans la zone pellucide et la membrane plasmique de l’embryon à travers lesquelles les composants CRISPR-Cas9 pénètrent dans les embryons. En règle générale, des impulsions électriques de niveau intermédiaire sont utilisées pour créer les ouvertures temporaires, suivies d’impulsions de transfert électriques de niveau inférieur qui facilitent le mouvement des composants d’édition du génome chargés négativement. L’électroporation des embryons est efficace, a un débit élevé et est facile à réaliser. Cependant, alors que l’électroporation embryonnaire s’est avérée très efficace pour l’introduction de modèles de réparation d’ADNss de petite taille (<200 pb), il existe peu de rapports d’électroporation réussie de modèles de réparation plus grands (>1,0 kb)13,19. Cette restriction de taille représente une limitation majeure de l’électroporation embryonnaire pour la génération de modèles animaux knock-in nécessitant de grandes insertions.
Dans le contexte de la thérapie génique, les virus adéno-associés (AAV) ont longtemps été utilisés comme véhicules pour délivrer du matériel génétique en raison de leur infectiosité in vivo efficace des cellules en division et non en division, de leur manque de pathogénicité et de leur intégration génomique rare20,21. Récemment, d’autres études ont combiné les AAV avec la technologie CRISPR-Cas9 pour introduire des modèles de réparation de l’ADN et des réactifs CRISPR 22,23,24. Cette approche permet de fournir des modèles de réparation de l’ADN plus grands sans avoir besoin de techniques de micro-injection.
La voie HDR est plus active dans les phases tardives S et G2 du cycle cellulaire25,26. Dans les études réalisées in vitro, des augmentations significatives de l’efficacité de knock-in ont été obtenues par l’administration de RNP CRISPR-Cas9 et de matrices de réparation de l’ADN dans des cellules synchronisées G2 ou par la restriction de la présence de la protéine Cas9 aux phases tardives S et G2 à l’aide d’une protéine de fusion Cas9-Géminine2. De plus, il y a un événement majeur d’activation du génome zygotique (ZGA) qui se produit pendant la phase G2 prolongée de l’embryon au stade 2 cellules, et cela est associé à un état de chromatine ouvert. On suppose que cela donne aux RNP CRISPR-Cas9 et aux modèles de réparation une plus grande accessibilité à l’ADN génomique.
Notre objectif était de nous appuyer sur toutes ces observations, en combinant l’approche AAV avec l’électroporation de l’embryon pour introduire les RNP CRISPR-Cas9 au stade 2 cellules du développement embryonnaire. Cette stratégie tire parti de la plus grande capacité d’administration de la matrice de réparation de l’ADN de l’AAV, de la facilité technique de l’électroporation et du point de temps plus optimal à 2 cellules pour l’accessibilité génomique pendant le développement de l’embryon afin de créer une méthode efficace pour le génie génétique ciblé des insertions d’ADN. Comme le souligne ce protocole, notre méthode optimisée permet de produire des modèles ciblés de rats knock-in portant des insertions de séquences d’ADN d’une taille de 1,2 à 3,0 kb sans avoir besoin de techniques de micro-injection.
Access restricted. Please log in or start a trial to view this content.
Protocole
Toutes les procédures expérimentales ont été approuvées par le Comité institutionnel de protection et d’utilisation des animaux de l’Université du Missouri (protocole ACUC #25580) et ont été effectuées conformément aux directives énoncées dans le Guide pour l’utilisation et le soin des animaux de laboratoire.
1. Livraison d’un modèle de réparation de l’ADN médié par l’AAV
- Concevez la construction de l’ADN pour qu’elle contienne la séquence de knock-in souhaitée entre deux bras d’homologie. Les longueurs de bras d’homologie typiques sont de 300 à 500 pb de longueur. S’assurer que l’ensemble de la matrice de réparation de l’ADN (bras d’homologie + séquence de knock-in souhaitée) est flanqué de répétitions terminales inversées (ITR) en clonant dans un squelette plasmidique approprié et en empaquetant dans des ssAAV1 ou ssAAV6 recombinants (sérotypes 1 ou 6).
REMARQUE : Si la séquence cible de l’ARNsg est présente dans la matrice de réparation de l’ADN, il est important de concevoir des mutations silencieuses pour perturber cette séquence, empêchant ainsi l’édition médiée par CRISPR-Cas9 de l’allèle correctement ciblé.
ATTENTION : Les vecteurs viraux AAV recombinants ne sont pas intégrateurs et sont classés comme BSL-1. Les techniques aseptiques standard sont utilisées pour le pipetage et la manipulation des embryons qui ont été exposés au virus. - Ajouter du ssAAV1 ou du ssAAV6 (emballé avec un modèle de réparation de l’ADN) à 30 μL de milieu KSOM-R27,28 jusqu’à une concentration finale de 3 × 107 copies du génome (GC)/μL sous de l’huile minérale dans une boîte de Pétri de 35 mm.
- Placez les zygotes avec des pronoyaux visibles dans le milieu KSOM-R contenant du ssAAV modifié.
REMARQUE : À l’aide de ce protocole, il n’est pas nécessaire d’éclaircir la zone pellucide, car les sérotypes AAV 1 et 6 peuvent facilement passer à travers pour une livraison efficace de la matrice de réparation de l’ADN. - Incuber à 37 °C avec 5 % de CO2 et une humidité maximale pendant 18 à 20 h pour permettre une transduction virale suffisante.
2. Préparation de l’électroporation
- Le lendemain, préparez un mélange d’électroporation contenant 100 ng/μL d’ARNsg et 100 ng/μL de protéine Cas9 dilué dans un milieu Opti-MEM dans un tube stérile sans RNase. Mélangez délicatement.
- Incuber à température ambiante pendant 10 min pour permettre la formation de RNP.
- Pendant le temps d’incubation, allumez l’électroporateur et connectez les fils à une électrode coulissante en verre de 1,5 mm.
- Définissez les paramètres d’électroporation comme décrit dans le tableau 1.
3. Électroporation embryonnaire à 2 cellules
- Pipeter doucement 5 μL du mélange d’électroporation CRISPR entre les deux électrodes sur la lame de verre en prenant soin de ne pas introduire de bulles d’air dans la solution.
- Retirez l’embryon au stade 2 cellules de la solution AAV. Alignez les embryons au stade de 2 cellules dans le mélange sans leur permettre de toucher les côtés des électrodes. Ceci est fait pour éviter la lyse.
- Appuyez sur le bouton Ohm pour mesurer l’impédance. Assurez-vous que l’impédance est comprise entre 0,100 et 0,300 kΩ. Le volume de réaction modifiera l’impédance (ajouter du volume pour diminuer l’impédance et soustraire du volume pour augmenter l’impédance).
- Appuyez sur Démarrer. Le processus ne prend que quelques secondes.
- Prélever les embryons immédiatement après l’électroporation et les laver 3 fois dans des gouttes fraîches de 30 μL de milieu KSOM-R.
- Replacer les embryons dans un incubateur à 37 °C avec 5 % de CO2 et une humidité maximale pour une culture in vitro dans 500 μL de milieux KSOM-R sous huile minérale. Alternativement, transférer chirurgicalement les embryons à un rat femelle pseudo-coït post-coït (dpc) de 1,5 jour pour produire une progéniture vivante.
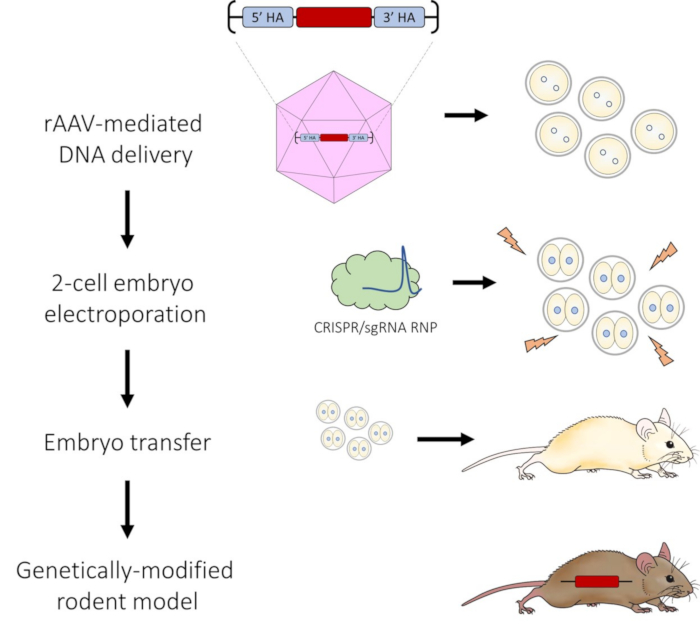
Figure 1 : Schéma de l’administration d’ADN médiée par l’AAV et du pipeline d’électroporation d’embryons à 2 cellules pour l’édition du génome CRISPR-Cas9. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Résultats
En suivant le protocole, il y a une administration efficace médiée par AAV du modèle de réparation de l’ADN, ce qui permet un HDR très efficace après que les embryons à 2 cellules aient été électroporés avec des RNP CRISPR-Cas9. Comme le montre la vidéo, un processus d’électroporation réussi entraîne la formation de bulles sur chaque électrode (Figure 2C) et l’impédance reste dans la plage de 0,100 à 0,300 kΩ. Après l’électroporat...
Access restricted. Please log in or start a trial to view this content.
Discussion
Le système d’édition du génome CRISPR-Cas a révolutionné le domaine du génie génétique en permettant la production efficace de modifications génétiques personnalisées simples et complexes chez une variété d’espèces animales. Des raffinements et des améliorations fréquents dans les techniques associées à l’édition du génome renforcent sa polyvalence. Nous avons décrit ici une nouvelle approche utilisant l’administration d’ADN médiée par ssAAV ainsi que l’électroporation embryonnaire à ...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Les auteurs tiennent à remercier Nolan Davis pour son aide dans la vidéographie et le montage vidéo.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Leads with alligator clips for electrodes | NepaGene | C117 | |
| Mineral oil | MilliporeSigma | M5310 | |
| NepaGene21 Super Electroporator | NepaGene | NEPA21 | |
| Platinum plate electrodes on slide glass | NepaGene | CUY501P1-1.5 | |
| PURedit Cas9 Protein | MilliporeSigma | PECAS9 | |
| sgRNA (chemically-modified) | Synthego | N/A | |
| ssAAV1 or ssAAV6 packaged DNA repair template | Vectorbuilder | N/A |
Références
- Lau, C. H., Tin, C., Suh, Y. CRISPR-based strategies for targeted transgene knock-in and gene correction. Fac Rev. 9, 20(2020).
- Gutschner, T., Haemmerle, M., Genovese, G., Draetta, G. F., Chin, L. Post-translational Regulation of Cas9 during G1 Enhances Homology-Directed Repair. Cell Rep. 14 (6), 1555-1566 (2016).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biol. 18 (1), 35(2017).
- Miura, H., Quadros, R. M., Gurumurthy, C. B., Ohtsuka, M. Easi-CRISPR for creating knock-in and conditional knockout mouse models using long ssDNA donors. Nat Protoc. 13 (1), 195-215 (2018).
- Gu, B., Posfai, E., Gertsenstein, M., Rossant, J. Efficient generation of large-fragment knock-in mouse models using 2-cell (2C)-homologous recombination (HR)-CRISPR. Curr Protoc Mouse Biol. 10 (1), e67(2020).
- Gu, B., Posfai, E., Rossant, J. Efficient generation of targeted large insertions by microinjection into two-cell-stage mouse embryos. Nat Biotechnol. 36 (7), 632-637 (2018).
- Kurihara, T., et al. DNA repair protein RAD51 enhances the CRISPR/Cas9-mediated knock-in efficiency in brain neurons. Biochem Biophys Res Commun. 524 (3), 621-628 (2020).
- Kaneko, T., Sakuma, T., Yamamoto, T., Mashimo, T. Simple knockout by electroporation of engineered endonucleases into intact rat embryos. Sci Rep. 4, 6382(2014).
- Chen, S., Lee, B., Lee, A. Y., Modzelewski, A. J., He, L. Highly efficient mouse genome editing by CRISPR ribonucleoprotein electroporation of zygotes. J Biol Chem. 291 (28), 14457-14467 (2016).
- Hashimoto, M., Yamashita, Y., Takemoto, T. Electroporation of Cas9 protein/sgRNA into early pronuclear zygotes generates non-mosaic mutants in the mouse. Dev Biol. 418 (1), 1-9 (2016).
- Wang, W., et al. Delivery of Cas9 protein into mouse zygotes through a series of electroporation dramatically increases the efficiency of model creation. J Genet Genomics. 43 (5), 319-327 (2016).
- Troder, S. E., et al. An optimized electroporation approach for efficient CRISPR/Cas9 genome editing in murine zygotes. PLoS One. 13 (5), e0196891(2018).
- Teixeira, M., et al. Electroporation of mice zygotes with dual guide RNA/Cas9 complexes for simple and efficient cloning-free genome editing. Sci Rep. 8 (1), 474(2018).
- Tanihara, F., et al. Efficient generation of GGTA1-deficient pigs by electroporation of the CRISPR/Cas9 system into in vitro-fertilized zygotes. BMC Biotechnol. 20 (1), 40(2020).
- Tanihara, F., et al. Generation of CD163-edited pig via electroporation of the CRISPR/Cas9 system into porcine in vitro-fertilized zygotes. Anim Biotechnol. 32 (2), 147-154 (2021).
- Camargo, L. S. A., Owen, J. R., Van Eenennaam, A. L., Ross, P. J. Efficient one-step knockout by electroporation of ribonucleoproteins into zona-intact bovine embryos. Front Genet. 11, 570069(2020).
- Lin, J. C., Van Eenennaam, A. L. Electroporation-mediated genome editing of livestock zygotes. Front Genet. 12, 648482(2021).
- Betters, E., Charney, R. M., Garcia-Castro, M. I. Electroporation and in vitro culture of early rabbit embryos. Data Brief. 21, 316-320 (2018).
- Miyasaka, Y., et al. CLICK: one-step generation of conditional knockout mice. BMC Genomics. 19 (1), 318(2018).
- Epstein, B. E., Schaffer, D. V. Combining engineered nucleases with adeno-associated viral vectors for therapeutic gene editing. Adv Exp Med Biol. 1016, 29-42 (2017).
- Gaj, T., Epstein, B. E., Schaffer, D. V. Genome engineering using adeno-associated virus: basic and clinical research applications. Mol Ther. 24 (3), 458-464 (2016).
- Chen, S., et al. CRISPR-READI: Efficient generation of knockin mice by CRISPR RNP electroporation and AAV donor infection. Cell Rep. 27 (13), 3780-3789 (2019).
- Mizuno, N., et al. Intra-embryo gene cassette knockin by CRISPR/CAS9-mediated genome editing with adeno-associated viral vector. iScience. 9, 286-297 (2018).
- Davis, D. J., et al. Efficient DNA knock-in using AAV-mediated delivery with 2-cell embryo CRISPR-Cas9 electroporation. Front Genome Ed. 5, 1256451(2023).
- Smirnikhina, S. A., Zaynitdinova, M. I., Sergeeva, V. A., Lavrov, A. V. Improving homology-directed repair in genome editing experiments by influencing the cell cycle. Int J Mol Sci. 23 (11), (2022).
- Takata, M., et al. Homologous recombination and non-homologous end-joining pathways of DNA double-strand break repair have overlapping roles in the maintenance of chromosomal integrity in vertebrate cells. EMBO J. 17 (18), 5497-5508 (1998).
- Men, H., Stone, B. J., Bryda, E. C. Media optimization to promote rat embryonic development to the blastocyst stage in vitro. Theriogenology. 151, 81-85 (2020).
- Men, H., Amos-Landgraf, J. M., Bryda, E. C., Franklin, C. L. KSOM-R supports both mouse and rat preimplantation embryo development in vitro. Theriogenology. 198, 69-74 (2023).
- Romeo, C., et al. AAV diffuses across zona pellucida for effortless gene delivery to fertilized eggs. Biochem Biophys Res Commun. 526 (1), 85-90 (2020).
- Fulka, J., Moor, R. M., Fulka, J. Electrofusion of mammalian oocytes and embryonic cells. Methods Mol Biol. 48, 309-316 (1995).
- Rems, L., et al. Cell electrofusion using nanosecond electric pulses. Sci Rep. 3, 3382(2013).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon