Method Article
3D-Druck von Bakterien zur Untersuchung von Motilität und Wachstum in komplexen porösen 3D-Medien
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt ein Verfahren für den dreidimensionalen (3D) Druck von Bakterienkolonien, um ihre Motilität und ihr Wachstum in komplexen porösen 3D-Hydrogelmatrizen zu untersuchen, die ihren natürlichen Lebensräumen ähnlicher sind als herkömmliche Flüssigkulturen oder Petrischale.
Zusammenfassung
Bakterien sind in komplexen dreidimensionalen (3D) porösen Umgebungen wie biologischen Geweben und Gelen sowie unterirdischen Böden und Sedimenten allgegenwärtig. Die meisten bisherigen Arbeiten konzentrierten sich jedoch auf Untersuchungen von Zellen in flüssigen Massen oder an flachen Oberflächen, die die Komplexität vieler natürlicher bakterieller Lebensräume nicht vollständig rekapitulieren. Hier wird diese Wissenslücke geschlossen, indem die Entwicklung einer Methode beschrieben wird, um dichte Bakterienkolonien in gestaute körnige Hydrogelmatrizen in 3D zu drucken. Diese Matrizen haben einstellbare Porengrößen und mechanische Eigenschaften; sie schließen die Zellen physisch ein und stützen sie so in 3D. Sie sind optisch transparent und ermöglichen eine direkte Visualisierung der Ausbreitung von Bakterien in ihrer Umgebung mithilfe von Bildgebung. Als Beweis für dieses Prinzip wird hier die Leistungsfähigkeit dieses Protokolls durch 3D-Druck und Abbildung von unbeweglichen und beweglichen Vibro cholerae sowie nicht-beweglichen Escherichia coli in gestauten granulären Hydrogelmatrizen mit unterschiedlichen interstitiellen Porengrößen demonstriert.
Einleitung
Bakterien bewohnen oft verschiedene, komplexe poröse 3D-Umgebungen, die von Schleimhautgelen im Darm und in der Lunge bis hin zum Boden im Bodenreichen 1,2,3,4,5,6,7,8,9,10,11,12,13,14,15, 16,17,18,19,20,21,
22,23,24,25. In diesen Umgebungen kann die bakterielle Bewegung durch Motilität oder Wachstum durch umgebende Hindernisse wie Polymernetzwerke oder Packungen fester Mineralkörner behindert werden, die die Fähigkeit der Zellen beeinflussen, sich in ihrer Umgebung auszubreiten26, Zugang zu Nährstoffquellen zu erhalten, neues Terrain zu besiedeln und schützende Biofilmgemeinschaften zu bilden27. Traditionelle Laborstudien verwenden jedoch in der Regel stark vereinfachte Geometrien, die sich auf Zellen in Flüssigkulturen oder auf flache Oberflächen konzentrieren. Diese Ansätze liefern zwar wichtige Einblicke in die Mikrobiologie, rekapitulieren jedoch nicht vollständig die Komplexität natürlicher Lebensräume, was zu dramatischen Unterschieden in den Wachstumsraten und dem Motilitätsverhalten im Vergleich zu Messungen in realen Umgebungen führt. Daher ist eine Methode zur Definition von Bakterienkolonien und zur Untersuchung ihrer Motilität und ihres Wachstums in porösen 3D-Umgebungen, die vielen ihrer natürlichen Lebensräume ähnlicher sind, dringend erforderlich.
Die Inokulation von Zellen in ein Agar-Gel und die anschließende Visualisierung ihrer makroskopischen Ausbreitung mit dem Auge oder mit einer Kamera bietet eine einfache Möglichkeit, dies zu erreichen, wie sie erstmals 1936 von Tittsler und Sandholzer vorgeschlagen wurde28. Dieser Ansatz weist jedoch eine Reihe wichtiger technischer Herausforderungen auf: (1) Während die Porengrößen im Prinzip durch Variation der Agarosekonzentration variiert werden können, ist die Porenstruktur solcher Gele schlecht definiert; (2) Lichtstreuung führt dazu, dass diese Gele trüb sind, was es schwierig macht, Zellen auf individueller Ebene mit hoher Auflösung und Genauigkeit zu visualisieren, insbesondere in großen Proben; (3) Wenn die Agarkonzentration zu groß ist, ist die Zellmigration auf die obere flache Oberfläche des Gels beschränkt; (4) Die komplexe Rheologie solcher Gele macht es schwierig, Inokula mit genau definierten Geometrien einzuführen.
Um diese Einschränkungen zu beheben, entwickelte Dattas Labor in früheren Arbeiten einen alternativen Ansatz, bei dem körnige Hydrogelmatrizen - bestehend aus gestauten, biokompatiblen Hydrogelpartikeln, die in flüssiger Bakterienkultur aufgequollen sind - als "poröse Petrischale" verwendet wurden, um Zellen in 3D einzuschließen. Diese Matrizen sind weiche, selbstheilende Fließspannungsfeststoffe; daher kann sich eine Injektionsmikrodüse im Gegensatz zu vernetzten Gelen, die in anderen Bioprinting-Verfahren verwendet werden, frei innerhalb der Matrix entlang eines beliebigen vorgeschriebenen 3D-Pfads bewegen, indem sie die Hydrogelpartikellokal neu anordnet 29. Diese Partikel verdichten sich dann schnell wieder und heilen sich selbst um injizierte Bakterien herum, wodurch die Zellen ohne zusätzliche schädliche Verarbeitung an Ort und Stelle bleiben. Bei diesem Verfahren handelt es sich also um eine Form des 3D-Drucks, die es ermöglicht, Bakterienzellen in einer gewünschten 3D-Struktur mit definierter Gemeinschaftszusammensetzung innerhalb einer porösen Matrix mit einstellbaren physikalisch-chemischen Eigenschaften anzuordnen. Darüber hinaus sind die Hydrogelmatrizen vollständig transparent, so dass die Zellen direkt bildgebend sichtbar gemacht werden können.
Der Nutzen dieses Ansatzes wurde bereits auf zwei Arten demonstriert. In einer Reihe von Studien wurden verdünnte Zellen in der gesamten Hydrogelmatrix verteilt, was Untersuchungen der Motilität einzelner Bakterien ermöglichte30,31. In einer anderen Reihe von Studien wurden mehrzellige Gemeinschaften mit einer Injektionsdüse, die auf einem programmierbaren Mikroskoptisch montiert war, in Gelen im Zentimetermaßstab 3D-gedruckt, was Studien über die Ausbreitung bakterieller Kollektive in ihrer Umgebung ermöglichte32,33. In beiden Fällen zeigten diese Studien bisher unbekannte Unterschiede in den Ausbreitungseigenschaften von Bakterien, die poröse Umgebungen bewohnen, im Vergleich zu denen in Flüssigkulturen/auf ebenen Oberflächen. Da sie jedoch auf einem Mikroskoptisch montiert waren, waren diese früheren Studien auf kleine Probenvolumina (~1 ml) und daher auf kurze experimentelle Zeitskalen beschränkt. Sie waren auch in ihrer Fähigkeit eingeschränkt, Inokula-Geometrien mit hoher räumlicher Auflösung zu definieren.
Hier wird die nächste Generation dieser experimentellen Plattform beschrieben, die beide Einschränkungen berücksichtigt. Insbesondere werden Protokolle bereitgestellt, mit denen man einen modifizierten 3D-Drucker mit einem angeschlossenen Spritzenextruder verwenden kann, um Bakterienkolonien in großem Maßstab in 3D zu drucken und abzubilden. Darüber hinaus zeigen repräsentative Daten, wie dieser Ansatz für die Untersuchung der Motilität und des Wachstums von Bakterien nützlich sein kann, am Beispiel des Biofilmbildners Vibrio cholerae und der planktonischen Escherichia coli . Dieser Ansatz ermöglicht es, Bakterienkolonien über lange Zeiträume zu erhalten und mit verschiedenen bildgebenden Verfahren sichtbar zu machen. Daher hat die Fähigkeit dieses Ansatzes, Bakteriengemeinschaften in porösen 3D-Lebensräumen zu untersuchen, ein enormes Forschungs- und Anwendungspotenzial, das sich auf die Behandlung und Untersuchung von Mikroben im Darm, in der Haut, in der Lunge und im Boden auswirkt. Darüber hinaus könnte dieser Ansatz in Zukunft für den 3D-Druck von bakterienbasierten künstlichen lebenden Materialien in komplexere freistehende Formen verwendet werden.
Protokoll
Dieser Ansatz besteht darin, einen kommerziellen 3D-Drucker für die Modellierung von Schmelzablagerungen in einen 3D-Biodrucker umzuwandeln, wobei ein zuvor von Tashman et al. festgelegtes Protokoll verwendet wird.34. Kurz gesagt, Tashman et al. ersetzten einen kommerziellen Extruderkopf durch einen maßgeschneiderten Spritzenpumpenextruder. Dieser Extruder ermöglicht den Druck von hochkonzentrierten flüssigen Suspensionen von Bakterienzellen in 3D, wobei sein extrudiertes Volumen und seine 3D-Position durch die Programmiersprache G-Code gesteuert werden. Das extrudierte Volumen wird in der Software durch den Extruderschritt (E-Schritt) vorgegeben und zusätzlich wie weiter unten beschrieben kalibriert. Diese Bakteriensuspensionen werden dabei direkt in eine körnige Hydrogelmatrix gedruckt, die als 3D-Träger für die Zellen dient. Im Folgenden beschreibt das Protokoll auch, wie Matrizen mit unterschiedlichen Polymerkonzentrationen hergestellt, die daraus resultierenden Änderungen der Porengröße und der rheologischen Eigenschaften charakterisiert und die anschließende bakterielle Motilität und das Wachstum durch direkte Bildgebung charakterisiert werden.
1. Umbau eines kommerziellen 3D-Druckers in einen 3D-Biodrucker
- Entfernen Sie den Extruder und die Heizung aus einem handelsüblichen 3D-Drucker (siehe Materialtabelle).
- Befolgen Sie die vorherigen Protokolle zur Herstellung des Spritzenpumpenextruders34, mit einer zusätzlichen Modifikation, um eine Einweg-Luer-Lock-Spritze aufzunehmen. Montieren Sie den Spritzenpumpenextruder am Drucker.
HINWEIS: Die CAD-Dateien, die für die Modifikation des Spritzenpumpenextruders für Kunststoffspritzen erforderlich sind, finden Sie in den Zusatzdateien 1-3. - Installieren und öffnen Sie die 3D-Druckersoftware (siehe Materialtabelle) auf einem Computer. Schließen Sie den 3D-Drucker an den Computer an.
- Laden Sie eine 1-ml-Einwegspritze mit einer Nadel geeigneter Größe in die 3D-gedruckten Klemmen, indem Sie die obere und untere Hälfte des Mechanismus ausrichten (Abbildung 1). Befestigen Sie die Klemmen mit drei M8-Innensechskantschrauben und dünnen Sechskantmuttern aus Stahl um die Spritze (siehe Materialtabelle). Der Spritzenkolben wird mit der Leitspindel im Spritzenpumpenextruder verbunden. Heben Sie den Kolben manuell an, indem Sie die Leitspindel drehen, um einen Luftspalt von 0,5 ml in der Spritze zu erzeugen.
- Wenn die Kontamination für das Experiment ein Problem darstellt, transportieren Sie den Spritzen-Klemm-Komplex in eine Biohaube und sterilisieren Sie ihn beim Eintritt mit 70%igem Ethanolspray, bevor Sie die folgenden Schritte ausführen.
2. Herstellung der Bakteriensuspension
- Für V. cholerae und E. coli über Nacht auf einer 2%igen Lennox LB (Luria Broth, siehe Materialtabelle) Agarplatte bei 37 °C wachsen.
- Bei V. cholerae inokulieren Sie die Zellen in 3 ml flüssiges LB mit zehn sterilen Glaskügelchen. Züchten Sie die Zellen in einem Schüttelinkubator bei 37 °C für 5-6 h bis zur mittleren exponentiellen Phase auf eine optische Dichte (OD) 600 von ~0,9.
- Bei E. coli inokulieren Sie die Zellen in 3 ml flüssiges LB. Züchten Sie die Zellen über Nacht in einem Schüttelinkubator bei 37 °C. Innokulieren Sie 200 μl der Übernachtkultur 3 h lang in frischem LB, bis der OD 0,6 erreicht.
- Übertragen Sie die Kultur in ein 10-ml-Zentrifugenröhrchen. Zentrifugieren Sie die Kultur 5 Minuten lang bei 5.000 x g bei Raumtemperatur, um ein Pellet zu bilden. Entfernen Sie den Überstand. Resuspendieren Sie mit ~10 μl flüssigem LB, um eine Zelldichte von ~9 x 1010 Zellen pro ml zu erreichen.
3. Laden der Bakteriensuspension in die Spritze
HINWEIS: Es gibt zwei Methoden zum Laden der Bakterien in die Spritze (Schritt 3.1 und Schritt 3.2). Schritt 3.1 funktioniert zum Laden kleiner Mengen von Bakteriensuspensionen, <200 μl, und Schritt 3.2 funktioniert zum Laden größerer Mengen von Bakteriensuspensionen, >200 μl. Schritt 3.1 wurde für die hier gezeigten repräsentativen Ergebnisse verwendet.
- Legen Sie eine leere 1-ml-Kunststoff-Luer-Lock-Spritze in den 3D-Biodrucker. Verbinden Sie den Spritzenkolben mit der Leitspindel. Ziehen Sie die Spritze manuell zurück, um 0,2 ml des Luftspalts hinzuzufügen, um Platz für die Bewegung des Kolbens in der Spritze zu schaffen, da für jede Versuchscharge ein kleines Volumen von Zellen ~20-50 μl verwendet wird.
- Befestigen Sie eine stumpfe Nadel mit der Nadelstärke, die für die erforderliche Größe der Druckmerkmale erforderlich ist, an der Spritzenspitze. Hier wird eine 2-Zoll-20-G-Nadel verwendet.
- Laden Sie die Bakteriensuspension in die Spritze, indem Sie ein 10-ml-Zentrifugenröhrchen mit dem Bakterieninokulum unter die Nadel legen. Drehen Sie die Schraube manuell, um den Spritzenkolben zurückzuziehen und die Zellen in die Spritze zu laden. Die Bakterienzellvolumina sind so klein, dass die Zellen oft nur in die Nadel geladen werden.
- Entfernen Sie den Kolben aus dem Spritzen-Klemm-Komplex und verwenden Sie eine andere Spritze und Nadel, um den Spritzen-Klemm-Komplex vorsichtig mit den gewünschten Bakteriensuspensionen zu befüllen, wobei darauf zu achten ist, dass keine Luftblasen eingeschlossen werden. Der Spritzen-Klemm-Komplex sollte leicht über den Rand mit der gewünschten Bakteriensuspension gefüllt und dann in den Bioprinter überführt werden.
- Führen Sie den Spritzen-Klemm-Komplex ohne Kolben vorsichtig in die entsprechende Buchse am Hauptkern des Biodrucker-Extruders ein.
- Stellen Sie sicher, dass sich der Druckerschlitten ungefähr auf halber Höhe der Leitspindel befindet und sich unter der geladenen Spritze eine Auffangschale befindet. Führen Sie dann den Kolben vorsichtig sowohl durch den Schlitten als auch durch den Spritzen-Klemm-Komplex ein, bis er am Schlitten einrastet. Drücken Sie den Kolben langsam in die Bakteriensuspension, um Luftblasen in der Spritze nicht einzuschließen.
- Schieben Sie die Adapterklemme über die Rückseite des Kolbens auf den Schlitten, um sie sowohl für Extrusions- als auch für Rückzugsmanöver zu sichern.
4. Kalibrierung des Extruderschritts auf das abgeschiedene Volumen
- Um den Extruderschritt (E-Schritt) auf das abgeschiedene Volumen zu kalibrieren, richten Sie zunächst den Biodrucker mit der genauen Spritze, der Spritzennadel und der abscheidenden Bakteriensuspension ein, die im Experiment verwendet werden. Hier wird eine 1 mL Luer-Lock-Spritze verwendet.
- Bestimmen Sie einen geschätzten E-Schrittbereich, über den kalibriert werden soll, indem Sie eine beliebige E-Schrittzahl (~200) extrudieren, und notieren Sie die Änderung des Kolbenvolumens mit Spritzenvolumenmarkierungen.
- Verwenden Sie dieses grobe Verhältnis von E-Schritt zu Volumen, um die E-Schritt-Einstellungen für die Durchführung des Kalibrierungs-Sweeps zu bestimmen. Wenn beispielsweise ein E-Schritt von 200 durch Sichtprüfung ca. 20 μl extrudiert und man 10-200 μl abscheiden möchte, testen Sie E-Schritte zwischen 100-2000.
- Um den linearen Kalibrierungssweep durchzuführen, markieren und messen Sie zunächst die Trockenmasse von zwanzig 1,5-ml-Probenahmeröhrchen auf einer Analysenwaage mit einer Empfindlichkeit von 0,1 mg.
- Extrudieren Sie die Bakteriensuspension in die vorgemessenen 1,5-ml-Röhrchen. Führen Sie für jeden E-Step mindestens 2 Replikationen durch. Wiederholen Sie dies für alle E-Schritte über den linearen Bereich und ersetzen Sie bei Bedarf die Bakteriensuspension. Wenn eine Kontamination für das Experiment ein Problem darstellt, wischen Sie die Außenseite der Spritzennadel nach jeder Probe mit einem fusselfreien Tuch ab, das mit 70% Ethanol getränkt ist.
- Messen Sie die Masse aller 1,5-ml-Röhrchen mit derselben Analysenwaage. Subtrahieren Sie den ersten Massenwert vom zweiten, um eine Nettomasse der abgeschiedenen Bakteriensuspension zu erhalten.
- Wandeln Sie die Masse der Bakteriensuspension in ein Volumen mit der Materialdichte um. Für viele Bakteriensuspensionen, die hauptsächlich aus Wasser bestehen, ist 1 g/ml eine geeignete Dichtenäherung.
- Führen Sie eine lineare Anpassung zwischen dem E-Schritt und dem extrudierten Volumen durch, um den Kalibrierungsprozess abzuschließen.
5. Vorbereitung der körnigen Hydrogelmatrix
- In einer Biosicherheitswerkbank trockenes Granulat aus vernetzten Acrylsäure/Alkylacrylat-Copolymeren (siehe Materialtabelle) zu 400 ml 2 % Lennox Luria-Bertani (LB) geben, um die Matrix steril zu halten; Es können jedoch auch andere flüssige Zellkulturmedien verwendet werden, um die Hydrogelmatrix aufzuquellen.
HINWEIS: Der Gewichtsprozentsatz des Granulats, das dem LB zugesetzt wird, hängt von der angestrebten Porengröße ab. In der vorliegenden Studie werden für eine 0,9%ige körnige Hydrogelmatrix 3,6 g Trockengranulat in die LB und für eine 1,2%ige körnige Hydrogelmatrix 4,8 g Trockengranulat in die LB gegeben. Das Hydrogel-Granulat wird homogen dispergiert, indem es 2 Minuten lang in einem Standmixer gemischt wird. - Stellen Sie nach dem Mischen den pH-Wert auf 7,4 ein, indem Sie 20 bis 500 μl-Schritte von 10 M Natriumhydroxid (NaOH) hinzufügen, um die Lebensfähigkeit der Zellen sicherzustellen. Messen Sie nach jeder Zugabe von NaOH den pH-Wert, indem Sie eine Pipettenspitze in die Mischung tauchen und dann die Hydrogelmatrix auf ein pH-Testpapier wischen.
HINWEIS: Wenn das NaOH hinzugefügt wird, erhöht sich die Viskosität der Mischung, wenn das Hydrogelgranulat zu quellen beginnt. Das gequollene Hydrogelgranulat hat einen Durchmesser von ~5 μm bis 10 μm und ist in einer Hydrogelmatrix zusammengeklemmt. Die innere Maschenweite des Granulats beträgt ~40 nm bis 100 nm, wie zuvor festgestellt32. Die Maschenweite ist groß genug, damit kleine Moleküle (z. B. Sauerstoff und Nährstoffe) frei diffundieren können, aber klein genug, damit Bakterien zwischen den interstitiellen Poren eingeschlossen werden können. - Übertragen Sie anschließend die körnige Hydrogelmatrix mit einer sterilen 50-ml-Kunststoffspritze in ein 50-ml-Zentrifugenröhrchen. Zentrifugieren Sie die Hydrogelmatrix bei 161 x g für 1 Minute bei Raumtemperatur, um die während des Mischvorgangs gebildeten Blasen zu entfernen.
- Lassen Sie die Hydrogelmatrix mindestens 2 Tage bei Raumtemperatur einwirken, um sicherzustellen, dass keine Kontamination aufgetreten ist. Die Kontamination erscheint als Mikrokolonien, die in der Hydrogelmatrix suspendiert sind. Zentrifugieren Sie die Hydrogelmatrix nach zwei Tagen bei 161 x g für 1 Minute, um alle zusätzlichen Blasen zu entfernen, die sich gebildet haben.
HINWEIS: Das Protokoll kann hier pausiert werden, indem die Hydrogelmatrix bis zu einer Woche bei Raumtemperatur gelagert wird. - Übertragen Sie in der Biosicherheitswerkbank mit einer sterilen 30-ml-Kunststoffspritze die gewünschte Menge an Hydrogel-Matrix in den Behälter, in dem der Druck erfolgt (hier wurden ~20 ml für einen 20-ml-Gewebekulturkolben oder 1 ml für 1-ml-Kunststoff-Mikroküvetten verwendet).
6. Charakterisierung der rheologischen Eigenschaften der granulären Hydrogelmatrix
- Laden Sie ~3 ml der Hydrogelmatrix in ein Scherrheometer (siehe Materialtabelle) mit einem Abstand von 1 mm zwischen aufgerauten parallelen Platten mit 50 mm Durchmesser, um die rheologischen Eigenschaften zu messen.
- Quantifizieren Sie das Fließverhalten mithilfe unidirektionaler Schermessungen am Scherrheometer, indem Sie die Scherspannung als Funktion eines logarithmischen Sweeps der Scherrate von 10-4 s-1 bis 102 s-1 messen (z. B. Abbildung 2A).
HINWEIS: Bei niedrigen Scherraten hat die Hydrogelmatrix eine konstante Scherspannung (die Fließspannung), die unabhängig von der Schergeschwindigkeit ist. Bei hohen Scherraten steigt die Scherspannung mit einer Potenzgesetzabhängigkeit von der Schergeschwindigkeit, was auf die Fluidisierung der Hydrogelmatrix hinweist. Dieses Ertrags-Stress-Verhalten ermöglicht es, Bakterien innerhalb der körnigen Hydrogelmatrix29 in 3D zu drucken. - Messen Sie die Speicher- und Verlustmodule G' bzw. G'' als Funktion der Frequenz unter Verwendung einer oszillatorischen Rheologie mit kleiner Amplitude mit einer Dehnungsamplitude von 1 % und Frequenzen zwischen 0,1 und 1 Hz (z. B. Abbildung 2B).
HINWEIS: Die ideale körnige Hydrogelmatrix für den 3D-Druck sollte einen Speichermodul haben, der größer als der Verlustmodul ist, was darauf hinweist, dass das Medium als gestauter elastischer Feststoff29 wirkt.
7. Charakterisierung der interstitiellen Porengröße der granulären Hydrogelmatrix
- 100 nm carboxylierte fluoreszierende Polystyrol-Nanopartikel (~3,6 x 1013 Partikel/ml, siehe Materialtabelle) in ihrer Verpackung 15 Minuten lang beschallen, um sie zu resuspendieren, um Aggregationen/Cluster von Partikeln aufzubrechen. Übertragen Sie 50 μl Nanopartikel in ein 1,5-ml-Mikrozentrifugenröhrchen.
- 10 min bei 9.500 x g bei Raumtemperatur zentrifugieren, bis sich das Pellet bildet und der Überstand klar ist. Entfernen Sie den Überstand und resuspendieren Sie das Pellet in 1 ml des flüssigen Wachstumsmediums (hier LB), das zur Herstellung der körnigen Hydrogelmatrix verwendet wird.
HINWEIS: Das Protokoll kann hier pausiert werden, indem die resuspendierten Nanopartikel bis zu 3 Monate bei 4 °C gelagert werden.
- 10 min bei 9.500 x g bei Raumtemperatur zentrifugieren, bis sich das Pellet bildet und der Überstand klar ist. Entfernen Sie den Überstand und resuspendieren Sie das Pellet in 1 ml des flüssigen Wachstumsmediums (hier LB), das zur Herstellung der körnigen Hydrogelmatrix verwendet wird.
- Beschallen Sie die resuspendierten Nanopartikel 30 Minuten lang. Übertragen Sie 1 ml körnige Hydrogelmatrix in ein 1,5-ml-Mikrozentrifugenröhrchen. Geben Sie 1 μl der resuspendierten Nanopartikel in die körnige Hydrogelmatrix und mischen Sie sie mit einer Pipettenspitze. Nach dem Mischen 30 s bei 161 x g bei Raumtemperatur zentrifugieren.
- Übertragen Sie die Hydrogelmatrix und die Nanopartikelmischung in eine Petrischale mit 35 mm Durchmesser und einem 0,1 mm dicken Glasboden. Der Brunnen hat einen Durchmesser von 20 mm und eine Tiefe von 1 mm. Legen Sie ein Glasdeckglas darauf und drücken Sie es nach unten, um den Fluss und die Verdunstung während der Bildgebung zu deaktivieren. Eine Alternative zum Glasdeckglas besteht darin, 1 ml Paraffinöl auf die Oberseite zu geben.
- Bilden Sie die Nanopartikel mit einem konfokalen Mikroskop mit einem 40-fachen Ölobjektiv mit 8-fachem zusätzlichem Zoom in der Bildgebungssoftware ab (siehe Materialtabelle).
HINWEIS: Anstelle eines zusätzlichen Digitalzooms in der Software kann ein Objektiv mit höherer Vergrößerung verwendet werden.- Stellen Sie eine Zeitschleife ohne Verzögerung (idealerweise ~19 Bilder/s) in einer einzigen Z-Ebene für 2 Minuten mit mindestens vier Nanopartikeln im Sichtfeld dar. Wiederholen Sie dies 15 bis 20 Mal an verschiedenen Orten, um genügend Daten für die Statistik zu sammeln (100 bis 200 Nanopartikel).
- Verwenden Sie eine Partikelverfolgungssoftware, um die Verschiebung der Partikel zu analysieren. Hier wird ein speziell geschriebenes Skript verwendet, das auf dem klassischen Crocker-Grier-Algorithmus basiert, um den Schwerpunkt des Nanopartikels35 zu verfolgen (siehe Zusatzdatei 7).
- Berechnen Sie aus der Partikelverfolgung die mittlere quadratische Verschiebung (MSD). Die MSD wird bei kurzen Längen und Zeitskalen eine freie Diffusion im Porenraum aufweisen und aufgrund des Einschlusses35 bei großen Längen und Zeitskalen zu subdiffusiver Skalierung übergehen.
- Identifizieren Sie die Längenskala, auf der der Übergang zur subdiffusiven Skalierung erfolgt, um die lokale Porengröße abzuschätzen. Berechnen Sie die Porengröße, indem Sie diese Längenskala zum Nanopartikeldurchmesser addieren. Wiederholen Sie die Porengrößenanalyse für jedes gemessene Nanopartikel. Dies ergibt eine Porengrößenverteilung, aus der eine mittlere Porengröße berechnet werden kann (z. B. Abbildung 3).
8. 3D Druckverfahren
- 3D-Druck von maßgefertigten Haltern für die Musterbehälter (siehe Ergänzungsdateien 4-6 für die CAD-Dateien ). Hier kommen Halter für die Gewebekulturkolben und Mikroküvetten zum Einsatz. Mit den Haltern kann der Drucker so programmiert werden, dass er mehrere Muster in einer einzigen Drucksitzung druckt. Legen Sie die Probenbehälter mit Hydrogelmedien in die Halter auf der Bauplattform.
- Öffnen Sie die 3D-Drucksoftware. Laden Sie einen vorprogrammierten G-Code in die Software. Für die repräsentativen Ergebnisse wird in Schritt 3.1 die Bakteriensuspension in den 3D-Drucker geladen.
HINWEIS: Ein Beispiel für einen G-Code zum Drucken linearer vertikaler Geometrien ist in Tabelle 1 angegeben. - Verschieben Sie mit der 3D-Drucksoftware die x-y-z-Ebenen , um den Druckkopf auf der x-y-Ebene zu zentrieren, um der erste Behälter zu sein, und positionieren Sie dann die z-Achse. Die Referenz-Z-Achse hebt den Druckkopf an. Drehen Sie die Schraube manuell langsam, um den Spritzenkolben zu drücken, bis eine kleine Menge der Bakteriensuspension an der Spitze der Nadel zu sehen ist.
- Wischen Sie die überschüssige Bakteriensuspension leicht mit einem sterilen Einwegtuch ab. Basierend auf der Höhe des Probenhalters, der Nadel und der Spritze senken Sie den Druckkopf mit der 3D-Drucksoftware in einem festen Abstand in das Hydrogelmedium im Probenbehälter Ihrer Wahl ab. Starten Sie den Druckvorgang mit einem Klick auf Drucken.
- Sobald der Druck abgeschlossen ist, schließen Sie die Probenbehälter. Wischen Sie den Drucker mit 70% Ethanol ab. Entsorgen Sie die Spritze und die Nadel ordnungsgemäß.
9. Anbau und Bildgebung der V. cholerae
- Verwenden Sie für die Bildgebung mit großem Sichtfeld eine Kamera mit einem Zoomobjektivaufsatz, um das Wachstum der Bildzellen mit einem Leuchtkasten zu fördern. Bilden Sie die Proben direkt nach dem Druck bei Raumtemperatur ab und übertragen Sie sie dann in einen Inkubator. Halten Sie die Proben während des Experiments zwischen den Bildgebungssitzungen bei 37 °C in einem stationären Inkubator.
- Nehmen Sie Bilder über einen gewünschten Zeitraum auf, um das Wachstumsverhalten über lange Zeiträume in der granularen Hydrogelmatrix zu beobachten.
Ergebnisse
Die Verwendung eines 3D-Biodruckers mit der körnigen Hydrogelmatrix erweitert die Möglichkeiten von Bioprintern, Bakterienkolonien in Formen zu drucken, die beim Drucken auf einem flachen Substrat aufgrund der niedrigen Viskosität der Bakteriensuspension zusammenbrechen würden. Die Auflösung des hier vorgestellten Ansatzes hängt von der Extrusionsgeschwindigkeit, der Größe der Nadel, der Geschwindigkeit des Druckkopfes, der Luft in der Nadel und der Viskosität der Bakteriensuspension ab. Aufgrund des geringen Volumens der Bakteriensuspension können beim Laden der Bakteriensuspension in Spritze und Nadel versehentlich Luftblasen eingebracht werden. Dies kann dazu führen, dass sich eine Luftblase in der endgültigen gedruckten Struktur ablagert (Abbildung 4). Eine andere Möglichkeit, Luftblasen in den Druck einzubringen, besteht darin, den Kolben nicht zu drücken, um vor dem Drucken einen kleinen Tropfen bakterieller Suspension an der Nadelspitze zu bilden, und an der Nadelspitze ein Luftspalt vorhanden ist. Wenn Sie den Spritzenkolben vor dem Drucken nicht drücken, kann dies auch dazu führen, dass unterschiedliche Zellvolumina in derselben Charge gedruckt werden. Im Laufe der Zeit löst sich die Luftblase jedoch im umgebenden Medium auf, wie in Abbildung 4 dargestellt.
Für die Kalibrierung des Extrusionsschritts hängt das abgeschiedene Volumen davon ab, wie der Linearantrieb des Druckkopfes den Spritzenkolben übersetzt, der Innendurchmesser der Spritze wirkt sich direkt auf das Volumen aus. Darüber hinaus beeinflussen die rheologischen Eigenschaften der Bakteriensuspension, wie leicht sie durch die Nadelkontraktion scheren, um reibungslos zu drucken. Daher sollte dieses Kalibrierungsverfahren für jedes Spritzen-/Nadel-/Bakteriensuspensions-Setup wiederholt werden. Prinzipiell könnte die Spritzenkalibrierung automatisiert werden. In der Praxis wäre es jedoch eine Herausforderung, einen solchen Code zu schreiben, der für viele Anwendungsfälle gilt. Beispielsweise müsste ein Benutzer, der die Extrusion von ca. 300 μl aus einer 1-ml-Spritze kalibrieren möchte, die Spritze viel häufiger nachfüllen als ein Benutzer, der um ein Zielvolumen von 30 μl kalibriert. Da die Konstante für die E-Step-zu-Volumen-Kalibrierung zu Beginn des Kalibrierungsprozesses unbekannt ist, kann der Benutzer möglicherweise nicht genau vorhersagen, wie oft ein erneutes Laden tatsächlich erforderlich ist. Um den Kalibrierungsprozess zu automatisieren, müssten außerdem die genauen Positionen jedes vorgewogenen 1,5-ml-Röhrchens angegeben werden. Um sicherzustellen, dass die gesamte Biotinte von der Nadel für eine genaue Kalibrierung in das Röhrchen abgeschieden wird, muss ein präziser Kontakt zwischen der Nadel und dem Röhrchenboden/der Röhrchenwand hergestellt werden. Ohne guten Kontakt können kleine Tröpfchen benetzt und an der Nadel haften bleiben. Daher kann jede Positionsänderung zwischen den Röhrchen erheblich zu Fehlern im Kalibrierungsprozess beitragen. Aus diesen Gründen empfehlen die Autoren, dass jeder Benutzer ein Kalibrierungsprogramm erstellt, das seinen individuellen Anforderungen entspricht.
Ein wesentliches Merkmal des hier vorgestellten Ansatzes ist die Möglichkeit, die Ausbreitung von Bakterien in ihrer Umgebung direkt über Motilität und Wachstum mittels Bildgebung zu visualisieren. In einer früheren Version des Protokolls für die Bildgebung wurden 6-Well-Platten mit 19 ml einer körnigen Hydrogelmatrix gefüllt. Aber selbst bei einem erfolgreichen 3D-Druck einer horizontalen Zelllinie, die auf einem inversen Mikroskop beobachtet werden könnte, würde auch auf der Oberseite des Mediums eine dichte Kolonie wachsen, was die Visualisierung mit der Hellfeldmikroskopie einschränkt (Abbildung 5). Die Kolonie auf der Oberseite resultierte wahrscheinlich aus einer Kontamination, als die Spritzennadel während des Drucks in das Medium eingeführt oder daraus entfernt wurde. Um dieses Problem zu umgehen, werden vertikale Zelllinien in Szintillationsfläschchen gedruckt (Abbildung 6A). Die Krümmung der zylindrischen Fläschchen führte jedoch zu Verzerrungen während der Bildgebung. Dies führte zur Auswahl von flachwandigen Küvetten und Gewebekulturflaschen als Probenbehälter für den Druck, die eine unverzerrte Bildgebung ermöglichten (Abbildung 6B-D). Eine Einschränkung von Gewebekulturflaschen ist der kleine geneigte Hals, der die Geometrien, die gedruckt werden können, begrenzt.
Zusammengenommen ermöglicht dieses Protokoll die Beobachtung der bakteriellen Motilität und des Wachstums in komplexen porösen Umgebungen über lange Zeiträume. Einige Beispiele sind in Abbildung 6B-G für Biofilm-bildende V. cholerae mittels Hellfeldmikroskopie sowie für Planktonzellen von E. coli mittels Laser-Scanning-Fluoreszenz-Konfokalmikroskopie dargestellt - was die Vielseitigkeit dieses Ansatzes demonstriert. In der Tat ist ein potenzielles Problem von Hydrogelmatrizen ihre mögliche Autofluoreszenz bei der Abbildung mit Fluoreszenzmikroskopie; die in Abbildung 6F,G gezeigten Bilder zeigen jedoch, dass eine solche Autofluoreszenz in der hier vorgestellten experimentellen Plattform minimal ist. Eine weitere Einschränkung solcher optischer Mikroskopieansätze ist ihre räumliche Auflösung, die durch die Beugungsgrenze bei ~100 s Nanometern festgelegt wird; diese Längenskala ist jedoch viel kleiner als die Größe einer einzelnen Bakterienzelle, und daher bieten optische Techniken die Möglichkeit, Bakterienzellen von der Skala einzelner Zellen (Abbildung 6G) bis zur Skala größerer, mehrzelliger Kolonienabzubilden 30,31,32,33. Weitere Beispiele werden im Folgenden beschrieben.
Wie oben erwähnt, kann der hier vorgestellte Ansatz verwendet werden, um Bakterienkolonien in kleinen (1 ml) und großen (20 ml) Matrixvolumina in 3D zu drucken und abzubilden. Daher werden die Unterschiede in den Ergebnissen, die mit verschiedenen Volumina erzielt wurden, im Folgenden anhand von 3D-gedruckten Kolonien von beweglichen und nicht-beweglichen V. cholerae als repräsentative Beispiele beschrieben. Die räumliche Auflösung (Breite der Linie) des Drucks wird durch den Innendurchmesser der Düse bestimmt. In den unten beschriebenen Beispielen ergibt eine Nadel mit einem Innendurchmesser von 0,6 mm eine anfängliche zylindrische Kolonie von 0,6 mm. Frühere Arbeiten haben gezeigt, dass die Druckauflösung durch die Verwendung einer gezogenen Glaskapillare mit einem Innendurchmesser von ~100-200 μm33 noch weiter reduziert werden kann.
Für die kleinen Volumina werden zwei verschiedene körnige Hydrogelmatrizen mit zwei unterschiedlichen Porengrößenverteilungen verwendet: eine mit einer mittleren Porengröße, die größer ist als der durchschnittliche Durchmesser einer V. cholerae-Zelle , 0,2-0,4μm 36, und die andere mit einer mittleren Porengröße, die kleiner als dieser Durchmesser ist. Die Proben über zwölf Tage werden abgebildet und durch Bildanalyse der räumlichen Ausdehnung der Kolonien im Laufe der Zeit gemessen (Abbildung 7 und Abbildung 8). Bei unbeweglichen Zellen, die sich nur durch Zellwachstum in ihrer Umgebung ausbreiten können, war die Geschwindigkeit der Flächenausdehnung zwischen den verschiedenen untersuchten Matrizen ähnlich (Abbildung 7), was darauf hindeutet, dass Unterschiede in der Matrixporengröße die zelluläre Ausbreitung durch Wachstum nicht beeinflussen - wie erwartet. Im Gegensatz dazu war für die beweglichen Zellen, die sich durch aktive Motilität in ihrer Umgebung ausbreiten, die Rate der räumlichen Ausdehnung von V. cholerae für die Hydrogelmatrix mit den größeren Poren höher - für die der Einschluss durch die Hydrogelkörner die Zellmotilität weniger behindert. Diese Unterschiede in der Bakterienausbreitung zeigten sich auch in den Koloniemorphologien (Abbildung 8). Die Kolonie von V. cholerae in Hydrogelmatrizen mit größeren Poren breitete sich durch glatte, diffuse Wolken aus (Abbildung 8A), was die Ausbreitung durch aktive Motilität widerspiegelt, wie zuvor beobachtet32. In Übereinstimmung mit dieser Interpretation werden diese diffusen Wolken bei nicht-beweglichen Zellen nicht beobachtet (Abbildung 8C). Außerdem sind die Poren so groß, dass die Zellen die Kügelchen nicht drücken, wenn sie durch die Poren schwimmen. Darüber hinaus beträgt die viskose Spannung, die beim Schwimmen ausgeübt wird, weniger als 1 Pa, was nicht ausreicht, um die umgebende Hydrogelmatrix nennenswert zu verformen. Im Gegensatz dazu breitet sich die Kolonie in Hydrogelmatrizen mit kleineren Poren nur durch raue, fraktalartige Wolken sowohl für bewegliche als auch für nicht-bewegliche Zellen aus (Abbildung 8B, D), was die Ausbreitung ausschließlich durch Zellwachstum widerspiegelt, während die Zellen wachsen, sie sich vorübergehend verformen und die umgebende Matrix abgeben. Da die Fließspannung der Hydrogelmatrizen viel kleiner ist als der Turgordruck der Zellen, bietet die Matrix in dieser Grenze der kleinen Porengröße nur einen schwachen Widerstand gegen Zellwachstum und scheint die 3D-gedruckte Struktur nicht stark zu beeinflussen, wie auch in unserer früheren Arbeit37 nachgewiesen wurde.
Ähnliche Ergebnisse werden für Experimente beobachtet, die in großvolumigen Proben durchgeführt wurden; Angesichts der größeren Fülle an Nährstoffen in diesen Proben könnten die Experimente das Zellwachstum jedoch über längere Zeiträume aufrechterhalten. Als Beispiel werden Ergebnisse mit den granulären Hydrogelmatrizen gezeigt, bei denen die durchschnittliche Porengröße kleiner war als die Zellgröße - und somit die zelluläre Ausbreitung hauptsächlich auf das Wachstum zurückzuführen war. Die Proben werden ~30 Tage lang in den körnigen Hydrogelmatrizen abgebildet und beobachteten in den ersten 150 Stunden eine ähnliche Flächenausdehnung sowohl für nicht-bewegliche als auch für bewegliche Zellen; bei noch längeren Zeiten wird jedoch eine starke Variabilität zwischen den Proben beobachtet, wobei einige Proben schnellere Ausbreitungsraten aufweisen (Abbildung 9 und Abbildung 10). Interessanterweise wird bei der erneuten Beprobung der Hydrogelmatrix nach dem Experiment - ein Vorteil des großen Volumens der Hydrogelmatrix - eine Abnahme der Fließspannung, der Speichermodule und der Verlustmodule gemessen (Abbildung 11), was darauf hindeutet, dass die Matrizen weicher wurden. Diese Veränderung könnte darauf zurückzuführen sein, dass die V. cholerae ein Molekül produzieren, das die Eigenschaften der Hydrogelmatrix verändert und die zelluläre Ausbreitung über lange Zeiträume fördert, was in zukünftigen Forschungen interessant sein wird. Beispiele für die groben, fraktalartigen Koloniemorphologien, die zu diesen Experimenten führen, sind in Abbildung 10 dargestellt.

Abbildung 1: Bilder des speziell angefertigten 3D-Bioprinters. (A) Biodrucker mit modifiziertem Spritzenpumpenextruderkopf mit Einweg-Luer-Lock-Spritze und -Nadel. Der Bioprinter ist ~46 cm breit. (B) Bild des 3D-Drucks von Zellen in Kolben, die mit gestauter Hydrogelmatrix gefüllt sind. Die Breite des Bildes beträgt 87 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
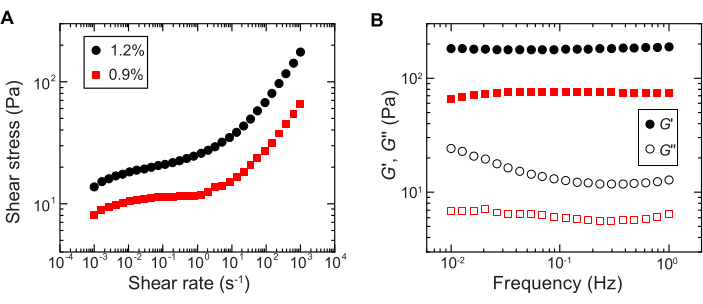
Abbildung 2: Charakterisierung der rheologischen Eigenschaften der granulären Hydrogelmatrizen. (A) Scherspannung in Abhängigkeit von der angelegten Schergeschwindigkeit. (B) Speicher- und Verlustmodul, G' bzw. G'', in Abhängigkeit von der Schwingungsfrequenz. Die Legende gibt den Hydrogel-Massenanteil an, der zur Herstellung jeder Hydrogelmatrix verwendet wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
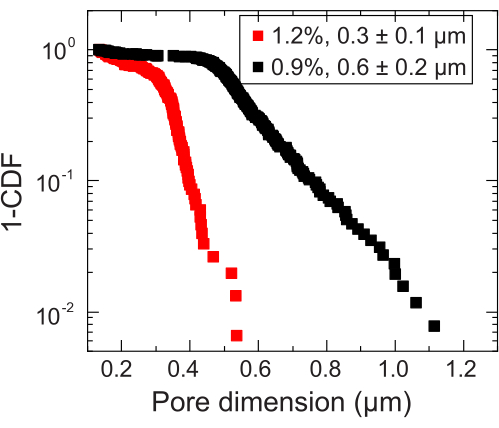
Abbildung 3: Porengrößenmessungen von zwei repräsentativen granularen Hydrogelmatrizen. Durch Tracking-Tracer wird die Verteilung der charakteristischen Porendimensionen für jede Hydrogelmatrix bestimmt. Die Daten werden durch 1-CDF dargestellt, wobei CDF die kumulative Verteilungsfunktion ist. Die Legende gibt den Hydrogel-Massenanteil an, der zur Herstellung jeder Matrix verwendet wurde. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
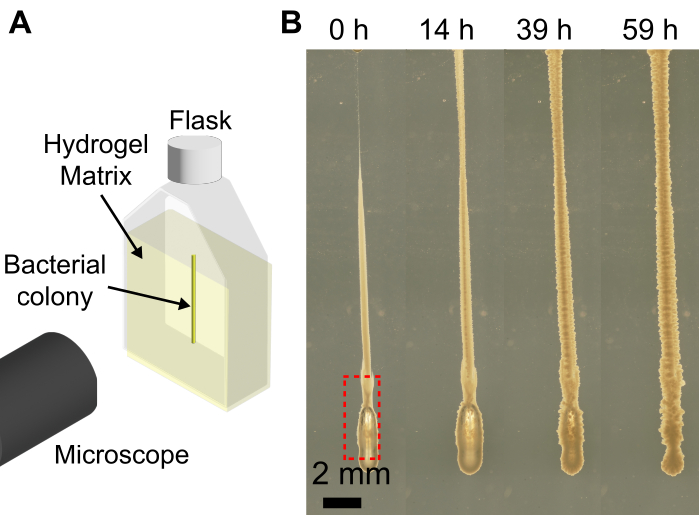
Abbildung 4: Beispiele für Blasen, die beim 3D-Druck von Bakterien gebildet wurden. (A) Schematische Darstellung des Bildgebungsaufbaus. (B) Schnappschüsse des Wachstums von V. cholerae mit einer Blase am unteren Rand des Abdrucks (roter Kasten) in 1,2% Hydrogelmatrix, die in LB geschwollen ist. Nach 59 Stunden Wachstum bei 37 °C ist die Luftblase vollständig aufgelöst und das Volk kollabiert aufgrund der Elastizität der Hydrogelmatrix zurück. Maßstabsleiste = 2 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Bilder der horizontalen Linie von beweglichen V. cholerae, gedruckt in einer Sechs-Well-Platte, die mit einer 1,2%igen Hydrogelmatrix gefüllt war, die in LB aufgequollen war, und dem Biofilm, der sich nach 48 Stunden Inkubation bei 37 °C auf der Oberseite bildete. (A) Schematische Darstellung des Bildgebungsaufbaus. (B) Die Biofilmbildung auf der Oberseite aufgrund von Verunreinigungen während des 3D-Drucks verringert die Opazität und ermöglicht keine klare Abbildung der horizontalen Linie. Die Oberseite bildet Falten, vermutlich aufgrund von unterschiedlichem Wachstum. Maßstabsleiste = 5 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
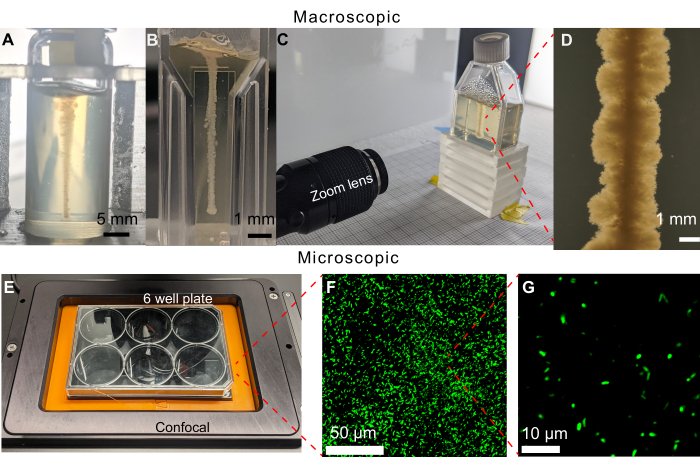
Abbildung 6: Beispiele für verschiedene Probenbehälter, die in dieser Methode verwendet werden können. (A) V. cholerae gedruckt in einem Glasfläschchen, das mit einer 1,2%igen granulären Hydrogelmatrix gefüllt war, nach 470 h. Die Krümmung des Fläschchens erschwert die Darstellung des Wachstums. Maßstabsbalken = 5 mm. (B) V. cholerae gedruckt in einer Mikroküvette, die mit 1,2 % körniger Hydrogelmatrix gefüllt ist. Die flachen Seiten ermöglichen eine klare Bildgebung, die kleinen Volumina begrenzen jedoch die Dauer der Experimente, bevor den Zellen die Wachstumssubstrate ausgehen. (C) Bildgebungsaufbau mit einem Zoomobjektiv zum Bild von V. cholerae , gedruckt in einem Gewebekulturkolben, der mit 1,2 % körniger Hydrogelmatrix gefüllt ist. Ähnlich wie beim Mikroküvettengehäuse ermöglichen die flachen Seiten eine klare Bildgebung. Die Gewebekulturflaschen können mit größeren Volumina körniger Hydrogelmatrix gefüllt werden, wodurch die Versuchszeit verlängert wird. (D) Bild des Zoomobjektivs der V. cholerae , gedruckt in einer 1,2%igen körnigen Hydrogelmatrix nach 100 Stunden Inkubation bei 37 °C. Maßstabsbalken = 1 mm. (E) Bildgebungsaufbau mit einem konfokalen Mikroskop zur Abbildung fluoreszierender Zellen. (F) 3D-Projektion von konfokalen Mikroskopaufnahmen von fluoreszierenden E. coli in der 1,2%igen granulären Hydrogelmatrix nach 10-tägiger Inkubation bei 37 °C. Maßstabsbalken = 50 μm. (G) Konfokalmikroskopische Aufnahme von fluoreszierenden E. coli in der Hydrogelmatrix mit Einzelzellauflösung. Maßstabsbalken = 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
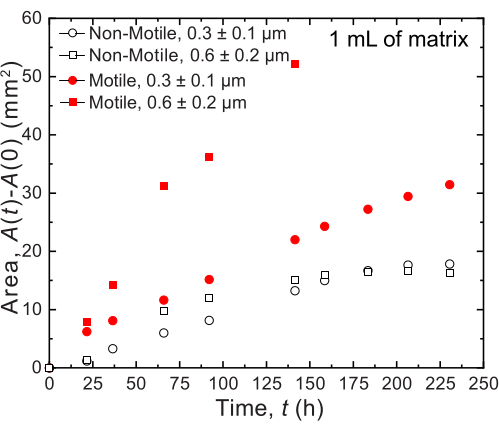
Abbildung 7: Flächenausdehnung in Abhängigkeit von der Zeit von Kolonien von V. cholerae, gedruckt in 1 ml granulärer Hydrogel-Stützmatrizen. Die Daten auf dem Diagramm stammen aus der Bildanalyse von Abbildung 8. Es wurde beobachtet, dass sich bewegliche Zellen (geschlossener roter Kreis und Quadrat) schneller ausbreiten als die nicht-beweglichen Zellen, was die Ausbreitung sowohl durch Wachstum als auch durch Motilität widerspiegelt. Darüber hinaus breiten sich die beweglichen Zellen in der Hydrogelmatrix mit einer großen Porengröße (rote Quadrate) schneller aus als die Zellen in der Hydrogelmatrix mit 0,3 μm Poren (rote Kreise). Nicht-bewegliche Zellen zeigen keinen Unterschied in der Flächenexpansionsrate zwischen den beiden Porengrößen, da sie nur wachsen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 8: Kolonien von V. cholerae , gedruckt in 1 ml granulärer Hydrogel-Stützmatrizen. (A) Schnappschüsse des Wachstums und der Motilität von beweglichen V. cholerae in einer 0,9%igen granulären Hydrogelmatrix, bei der die Porengröße größer als der durchschnittliche Zelldurchmesser ist. Pfeile zeigen eine diffuse Wolke an, die sich bildet, wenn sich Zellen durch die Hydrogelmatrix bewegen. (B) Momentaufnahmen des Wachstums und der Motilität von beweglichen V. cholerae in einer 1,2%igen körnigen Hydrogelmatrix, bei der die Porengröße kleiner als der durchschnittliche Zelldurchmesser ist. Pfeile zeigen eine raue, fraktalartige Wolke an, die sich durch Zellwachstum bildet. (C) Momentaufnahmen der zeitlichen Entwicklung von nicht-beweglichen V. cholerae in 0,9% körniger Hydrogelmatrix, bei der die Porengröße größer als der durchschnittliche Zelldurchmesser ist. In diesem Fall sind diffuse Wolken, die die Motilität widerspiegeln, nicht beobachtbar. (D) Momentaufnahmen des Wachstums von unbeweglichen V. cholerae in einer 1,2%igen körnigen Hydrogel-Trägermatrix, bei der die Porengröße kleiner als der durchschnittliche Zelldurchmesser ist. In diesem Fall sind wieder raue, fraktalartige Wolken zu beobachten, die das Wachstum widerspiegeln. Maßstabsleisten = 1 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
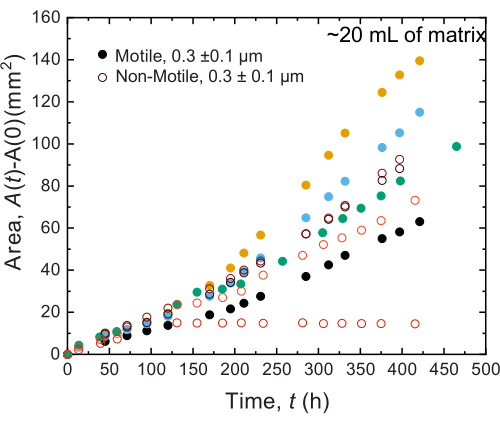
Abbildung 9: Flächenausdehnung in Abhängigkeit von der Zeit von Kolonien von V. cholerae, gedruckt in 20 ml 1,2% körniger Hydrogel-Stützmatrizen. Es wird beobachtet, dass sich unbewegliche (offene Kreise) und bewegliche Zellen (geschlossene Kreise) in den ersten 100 Stunden mit ähnlichen Geschwindigkeiten ausbreiten. Nach 100 Stunden werden Unterschiede in den Expansionsraten der Fläche beobachtet, möglicherweise aufgrund der Entwicklung der Variabilität über lange Zeiträume. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 10: Kolonien von V. cholerae, gedruckt in 22 ml 1,2 % körniger Hydrogelmatrizen, die bei 37 °C gezüchtet wurden. (Oben) Momentaufnahmen des Wachstums von beweglichen V. cholerae. (Unten) Momentaufnahmen des Wachstums von nicht-beweglichen V. cholerae. In beiden Fällen sind raue, fraktalartige Wolken zu beobachten, die das Wachstum widerspiegeln. Maßstabsleisten = 2 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
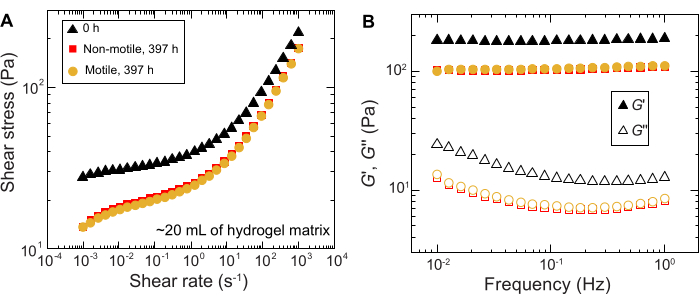
Abbildung 11: Charakterisierung der rheologischen Eigenschaften der 1,2%igen granulären Hydrogelmatrix vor (0 h) und nach dem Experiment (397 h). (A) Scherspannung in Abhängigkeit von der angelegten Schergeschwindigkeit. (B) Speicher- und Verlustmodul, G' bzw. G'', in Abhängigkeit von der angelegten Schwingungsfrequenz. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Gcode-Befehle | Aufgaben | |||
| M82 | Absoluter Extrusionsmodus | |||
| M302 S0 | Kaltfließpressen ermöglichen | |||
| M92 E14575 | Legen Sie die Extrusionsschritte pro mm fest | |||
| G92 X35.61 Y81 Z0 E0.0 | Stellen Sie die z-Achsen- und Extrusionsposition auf Null und die x-,y-Position auf 35,71 mm und y 88 mm ein, wo sich der Druckkopf befindet, wenn der G-Code beginnt | |||
| M221 S100 T0 | setzt die Durchflussmenge auf 100% | |||
| M107 | Schalten Sie den Lüfter aus | |||
| G1 F1 X35.61 Y81 E0.1 | Extrudieren Sie die 20 μl Biotinte mit einer Vorschubgeschwindigkeit von 50 μl/min in die Matrix, wo die aktuelle Position eingestellt wurde | |||
| G0 F200 Z60 | Nadel mit einer Geschwindigkeit von 200 mm/min aus der Matrix und dem Probenbehälter ziehen | |||
| G0 F500 X65.81 Y81.0 | Bewegen Sie die Nadel mit einer Geschwindigkeit von 500 mm/min zum nächsten Probenkontiiner | |||
| G0 F100 Z0 | Senken Sie die Nadel in den nächsten Probenbehälter auf die gleiche z-Position, an der der Druck mit einer Geschwindigkeit von 100 mm/min begonnen hat | |||
| G1 F1 X65.81 Y81.0 E0.2 | Extrudieren Sie die 20 μl Biotinte mit einer Vorschubgeschwindigkeit von 50 μl/min in die Matrix, wo die aktuelle Position eingestellt wurde | |||
| G0 F200 Z60 | Nadel mit einer Geschwindigkeit von 200 mm/min aus der Matrix und dem Probenbehälter ziehen | |||
| G0 F500 X96.01 Y81.0 | Bewegen Sie die Nadel mit einer Geschwindigkeit von 500 mm/min zum nächsten Probenkontiiner | |||
| G0 F100 Z0 | Senken Sie die Nadel in den Probenbehälter auf die gleiche z-Position, an der der Druck mit einer Geschwindigkeit von 100 mm/min begonnen hat | |||
| G1 F1 X96.01 Y81.0 E0.3 | Extrudieren Sie die 20 μl Biotinte mit einer Vorschubgeschwindigkeit von 50 μl/min in die Matrix, wo die aktuelle Position eingestellt wurde | |||
| G0 F200 Z60 | Nadel mit einer Geschwindigkeit von 200 mm/min aus der Matrix und dem Probenbehälter ziehen | |||
| G0 F500 X126.21 Y81.0 | Bewegen Sie die Nadel mit einer Geschwindigkeit von 500 mm/min zum nächsten Probenkontiiner | |||
| G0 F100 Z0 | Senken Sie die Nadel in den nächsten Probenbehälter auf die gleiche z-Position, an der der Druck mit einer Geschwindigkeit von 100 mm/min begonnen hat | |||
| G1 F1 X126.21 Y81.0 E0.4 | Extrudieren Sie die 20 μl Biotinte mit einer Vorschubgeschwindigkeit von 50 μl/min in die Matrix, wo die aktuelle Position eingestellt wurde | |||
| G0 F200 Z80 | Nadel mit einer Geschwindigkeit von 200 mm/min aus der Matrix und dem Probenbehälter ziehen | |||
Tabelle 1: G-Code-Programmierung zum Drucken vertikaler Linien der Bakteriensuspension.
Ergänzende Datei 1: STL-Datei für die untere Klemme 1 ml Einweg-Luer-Lock-Spritzen für den Spritzenextruder. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: STL-Datei für die obere Klemme für 1-ml-Einweg-Luer-Lock-Spritzen für den Spritzenextruder. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 3: STL-Datei für den Spritzenadapter für 1-ml-Einweg-Luer-Lock-Spritzen für den Spritzenextruder. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 4: STL-Datei für den Küvetten-Probenhalter. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 5: STL-Datei für den Probenhalter für Gewebekolben. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 6: STL-Datei für die 6-Well-Platte und das 35-mm-Petrischalen-Druckbett. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 7: Benutzerdefiniertes Skript für die Partikelverfolgung. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Kritische Schritte im Protokoll
Es ist wichtig sicherzustellen, dass bei der Herstellung jeder Hydrogelmatrix die Matrix in einer sterilen Umgebung hergestellt wird. Andernfalls kann es zu einer Kontamination kommen, die sich z.B. nach mehreren Tagen als Mikrokolonien (kleine Sphäroide) in der Matrix manifestiert. Während des Mischvorgangs ist es wichtig, dass alle trockenen körnigen Hydrogelpartikel aufgelöst werden. Darüber hinaus beginnt das Granulat beim Einstellen des pH-Werts jeder Hydrogelmatrix mit dem NaOH zu quellen, was die Viskosität der Hydrogelmatrix erhöht, was das Mischen erschwert. Die Verwendung des Standmixers trägt dazu bei, dass das NaOH gut in die Hydrogelmatrix gemischt wird. Während der Beladung jeder Bakteriensuspension können sich Lufteinschlüsse in der Nadel bilden. Um dieses Problem zu vermeiden, stellen Sie sicher, dass die Nadelspitze immer in der Bakteriensuspension im Zentrifugenröhrchen sitzt und nicht am Boden des Röhrchens oder in der Nähe der Oberseite. Eine andere Möglichkeit, dieses Problem zu lösen, besteht darin, große Zellvolumina zu züchten und somit größere Mengen der Bakteriensuspension zum Drucken zu haben.
Begrenzungen
Derzeit schränkt die niedrige Viskosität der Bakteriensuspension während des Drucks die Geometrien ein, die gedruckt werden können, und führt oft zu einer Biofilmbildung und einem Wachstum auf der Oberfläche der Hydrogel-Matrix aufgrund von Spurenzellen. Es gibt einige mögliche Methoden, um diese Einschränkung zu überwinden, darunter die Erhöhung der Viskosität der Bakteriensuspension oder die weitere Optimierung der 3D-Druckereinstellungen. Um die Viskosität der Bakteriensuspension zu erhöhen, könnte man die Bakteriensuspension mit einem anderen Polymer mischen - zum Beispiel Alginat, das zuvor für den 3D-Druck von Bakterien auf ebenen Oberflächen verwendet wurde38. Die Druckereinstellungen können weiter optimiert werden, um das Zurückziehen des Spritzenkolbens während des Herausziehens der Nadel aus der körnigen Hydrogelmatrix zu ermöglichen, was das Potenzial hätte, die Ablagerung von Zellen während des Entfernens der Nadel aus der Hydrogelmatrix zu verhindern.
Die Bedeutung der Methode im Vergleich zu bestehenden/alternativen Methoden
Das hier beschriebene Verfahren ermöglicht das Drucken von Bakterienkolonien in körnige Hydrogelmatrizen. Die körnigen Hydrogelmatrizen ermöglichen die Untersuchung des Einflusses externer Umweltfaktoren (z. B. Porengröße, Verformbarkeit der Matrix) auf die Motilität und das Wachstum von Bakterien. Während in dieser Arbeit LB als flüssiges Wachstumsmedium zum Quellen der Hydrogelmatrix verwendet wird, kann die Hydrogelmatrix mit anderen flüssigen Wachstumsmedien, einschließlich Medien mit Antibiotika, aufgequollen werden. Bisherige Methoden zur Untersuchung von Bakterien in geschlossenen Umgebungen waren durch die Länge der experimentellen Zeit, die Polymermaschenweite und die Steifigkeit der umgebenden Hydrogelmatrix begrenzt37,38. Es gibt bereits Protokolle zur Herstellung von granularen Hydrogelmatrizen aus verschiedenen Polymeren, so dass das Potenzial für die Untersuchung der Auswirkungen unterschiedlicher Umweltbedingungen auf die Beweglichkeit und das Wachstum von Bakterien enorm ist. Diese Methode ermöglicht die Untersuchung von Bakterien in Kontrollumgebungen, die die Umgebungen, die Bakterien in der realen Welt bewohnen, wie z. B. Wirtsschleim oder Boden, leichter rekapitulieren. Eine weitere Einschränkung vieler anderer Methoden ist die Deckkraft der umgebenden Matrix; Dieser Ansatz mit optisch transparenten Materialien bietet jedoch die Möglichkeit, z. B. die optogenetische Kontrolle und Musterung von Bakterien in 3D zu untersuchen.
Über die Untersuchung von Motilität und Wachstum hinaus überwindet das hier beschriebene 3D-Druckverfahren die Einschränkungen vieler anderer Biodruckverfahren, die die Abscheidung einer Biotinte auf einem Substrat erfordern und daher in der Höhe des von ihnen hergestellten künstlichen lebenden Materials begrenzt sind. In Zukunft kann dieses Bioprinting-Protokoll weiter ausgebaut werden, um biohybride Materialien herzustellen, indem Polymere mit biofilmbildenden Zellen gemischt werden. Die körnigen Hydrogel-Matrizen unterstützen den 3D-Druck von dickeren, größeren technischen lebenden Materialien und komplexeren Geometrien als viele andere aktuelle Bioprinting-Methoden für Bakterien. Während in dieser Arbeit nur V. cholerae und E. coli verwendet wurden, wurden auch andere Arten wie Pseudomonas aeruginosa erfolgreich in 3D gedruckt37. Über das Drucken hinaus kann der Drucker so angepasst werden, dass er nach dem Wachstum eine kontrollierte Probenahme von Bakterien durchführt, um beispielsweise festzustellen, ob genetische Veränderungen aufgetreten sind.
Offenlegungen
Die experimentelle Plattform, die in dieser Veröffentlichung zum 3D-Druck und zur Abbildung von Bakteriengemeinschaften verwendet wird, ist Gegenstand einer Patentanmeldung, die von der Princeton University im Namen von Tapomoy Bhattacharjee und S.S.D. eingereicht wurde (PCT-Anmeldenummer PCT/US/2020/030213).
Danksagungen
R.K.B. bedankt sich für die Unterstützung durch das Presidential Postdoctoral Research Fellows Program. Dieses Material basiert auch auf Arbeiten, die durch den NSF Graduate Research Fellowship Program Grant DGE-2039656 (an A.M.H.) unterstützt wurden. A.S.D.-M. und H.N.L. bedankt sich für die Unterstützung durch den Lidow Independent Work/Senior Thesis Fund an der Princeton University. Wir danken auch dem Labor von Bonnie Bassler für die Bereitstellung von Stämmen von V. cholerae. S.S.D. dankt der Unterstützung durch die NSF-Zuschüsse CBET-1941716, DMR-2011750 und EF-2124863 sowie durch den Eric and Wendy Schmidt Transformative Technology Fund, die New Jersey Health Foundation, das Pew Biomedical Scholars Program und das Camille Dreyfus Teacher-Scholar Program.
Materialien
| Name | Company | Catalog Number | Comments |
| 1 mL cuvettes | VWR | 97000-586 | |
| 1 mL Luer lock syringe | BH Supplies | BH1LL | |
| 10 M NaOH | Sigma-Aldrich | 72068 | |
| 100 nm carboxylated fluorescent polystyrene nanoparticles (FluoSpheres) | Invitrogen, (ThermoFischer Scientific) | F8803 | |
| 15 mL centrifuge tubes | ThermoFischer Scientific | 14-955-237 | |
| 20 G blunt needle | McMaster Carr | 75165A252 | |
| 25 mL tissue culture flasks | VWR | 10861-566 | |
| 3D printer | Lulzbot | LulzBot Mini 2 | |
| 3D printing software | Cura | Cura-Lulzbot | |
| 50 mL centrifuge tubes | ThermoFischer Scientific | 14-955-239 | |
| Agar | Sigma-Aldrich | A1296 | |
| Carbomer Granular Hydrogel Particles | Lubrizol | Carbopol 980NF | dry granules of crosslinked acrylic acid/alkyl acrylate copolymers |
| Centrifuge (2 mL tube capacity) | VWR | 2405-37 | |
| Centrifuge (50 mL tube capacity) | ThermoFischer Scientific | 75007200 | Sorvall (brand) ST 8 (model) |
| Confocal Microscope | Nikon | A1R+ inverted laserscanning confocal microscope | |
| Glass bottom petri dish | Cellvis | D35-10-1-N | |
| Lennox LB (Lubria Broth) | Sigma-Aldrich | L3022 | |
| M8 × 1.25 mm, 150 mm long, Fully Threaded Socket Cap | McMaster Carr | 91290A478 | |
| M8 × 1.25 mm, Brass Thin Hex Nut | McMaster Carr | 93187A300 | |
| Open-source syringe pump | Custom-made | Replistruder 4 | https://www.sciencedirect.com/science/article/pii/S2468067220300791 |
| Petri dish (60 mm round) | ThermoFischer Scientific | FB0875713A | |
| Shear Rheometer | Anton Paar | MCR 501 | |
| Ultrasonic cleaner | VWR | 97043-992 |
Referenzen
- Persat, A., et al. The mechanical world of bacteria. Cell. 161 (5), 988-997 (2015).
- Stoodley, P., Dodds, I., Beer, D. D., Scott, H. L., Boyle, J. D. Flowing biofilms as a transport mechanism for biomass through porous media under laminar and turbulent conditions in a laboratory reactor system. Biofouling. 21 (3-4), 161-168 (2005).
- Ludemann, H., Arth, I., Liesack, W. Spatial changes in the bacterial community structure along a vertical oxygen gradient in flooded paddy soil cores. Applied and Environmental Microbiology. 66 (2), 754-762 (2000).
- Sicard, J. F., Bihan, G. L., Vogeleer, P., Jacques, M., Harel, J. Interactions of intestinal bacteria with components of the intestinal mucus. Frontiers in Cellular and Infection Microbiology. 7, 387 (2017).
- Grice, E. A., Segre, J. A. The skin microbiome. Nature Reviews Microbiology. 9 (4), 244-253 (2011).
- Balzan, S., Quadros, C. D. A., Cleva, R. D., Zilberstein, B., Cecconello, I. Bacterial translocation: Overview of mechanisms and clinical impact. Journal of Gastroenterology and Hepatology. 22 (4), 464-471 (2007).
- Chaban, B., Hughes, H. V., Beeby, M. The flagellum in bacterial pathogens: For motility and a whole lot more. Seminars in Cell & Developmental Biology. 46, 91-103 (2015).
- Datta, S. S., Steinberg, A. P., Ismagilov, R. F. Polymers in the gut compress the colonic mucus hydrogel. Proceedings of the National Academy of Sciences. 113 (26), 7041-7046 (2016).
- Harman, M. W., et al. The heterogeneous motility of the Lyme disease spirochete in gelatin mimics dissemination through tissue. Proceedings of the National Academy of Sciences. 109 (8), 3059-3064 (2012).
- Ribet, D., Cossart, P. How bacterial pathogens colonize their hosts and invade deeper tissues. Microbes and Infection. 17 (3), 173-183 (2015).
- Siitonen, A., Nurminen, M. Bacterial motility is a colonization factor in experimental urinary tract infection. Infection and Immunity. 60 (9), 3918-3920 (1992).
- Lux, R., Miller, J. N., Park, N. H., Shi, W. Motility and chemotaxis in tissue penetration of oral epithelial cell layers by Treponema denticola. Infection and Immunity. 69 (10), 6276-6283 (2001).
- O’Neil, H. S., Marquis, H. Listeria monocytogenes flagella are used for motility, not as adhesins, to increase host cell invasion. Infection and Immunity. 74 (12), 6675-6681 (2006).
- Gill, C. O., Penney, N. Penetration of bacteria into meat. Applied and Environmental Microbiology. 33 (6), 1284-1286 (1977).
- Shirai, H., Datta, A. K., Oshita, S. Penetration of aerobic bacteria into meat: A mechanistic understanding. Journal of Food Engineering. 196, 193-207 (2017).
- Thornlow, D. N., Brackett, E. L., Gigas, J. M., Dessel, N. V., Forbes, N. S. Persistent enhancement of bacterial motility increases tumor penetration: Motility enhances bacterial tumor penetration. Biotechnology and Bioengineering. 112 (11), 2397-2405 (2015).
- Toley, B. J., Forbes, N. S. Motility is critical for effective distribution and accumulation of bacteria in tumor tissue. Integrative Biology. 4 (2), 165-176 (2011).
- Dechesne, A., Wang, G., Gülez, G., Or, D., Smets, B. F. Hydration-controlled bacterial motility and dispersal on surfaces. Proceedings of the National Academy of Sciences. 107 (32), 14369-14372 (2010).
- de Souza, R., Ambrosini, A., Passaglia, L. M. P. Plant growth-promoting bacteria as inoculants in agricultural soils. Genetics and Molecular Biology. 38 (4), 401-419 (2015).
- Turnbull, G. A., Morgan, J. A. W., Whipps, J. M., Saunders, J. R. The role of bacterial motility in the survival and spread of Pseudomonas fluorescens in soil and in the attachment and colonisation of wheat roots. FEMS Microbiology Ecology. 36 (1), 21-31 (2001).
- Watt, M., Kirkegaard, J. A., Passioura, J. B. Rhizosphere biology and crop productivity—a review. Soil Research. 44 (4), 299-317 (2006).
- Adadevoh, J. S. T., Ramsburg, C. A., Ford, R. M. Chemotaxis Increases the Retention of Bacteria in Porous Media with Residual NAPL Entrapment. Environmental Science & Technology. 52 (13), 7289-7295 (2018).
- Adadevoh, J. S. T., Triolo, S., Ramsburg, C. A., Ford, R. M. Chemotaxis Increases the Residence Time of Bacteria in Granular Media Containing Distributed Contaminant Sources. Environmental Science & Technology. 50 (1), 181-187 (2016).
- Ford, R. M., Harvey, R. W. Role of chemotaxis in the transport of bacteria through saturated porous media. Advances in Water Resources. 30 (6-7), 1608-1617 (2007).
- Wang, M., Ford, R. M., Harvey, R. W. Coupled effect of chemotaxis and growth on microbial distributions in organic-amended aquifer sediments: Observations from laboratory and field studies. Environmental Science & Technology. 42 (10), 3556-3562 (2008).
- Amchin, D. B., Ott, J. A., Bhattacharjee, T., Datta, S. S. Influence of confinement on the spreading of bacterial populations. PLoS Computational Biology. 18 (5), e1010063 (2022).
- Moore-Ott, J. A., Chiu, S., Amchin, D. B., Bhattacharjee, T., Datta, S. S. A biophysical threshold for biofilm formation. eLife. 11, e76380 (2022).
- Tittsler, R. P., Sandholzer, L. A. The use of semi-solid agar for the detection of bacterial motility. Journal of Bacteriology. 31 (6), 575-580 (1936).
- Bhattacharjee, T., et al. Polyelectrolyte scaling laws for microgel yielding near jamming. Soft Matter. 14 (9), 1559-1570 (2018).
- Bhattacharjee, T., Datta, S. S. Confinement and activity regulate bacterial motion in porous media. Soft Matter. 15 (48), 9920-9930 (2019).
- Bhattacharjee, T., Datta, S. S. Bacterial hopping and trapping in porous media. Nature Communications. 10 (1), 2075 (2019).
- Bhattacharjee, T., Amchin, D. B., Ott, J. A., Kratz, F., Datta, S. S. Chemotactic migration of bacteria in porous media. Biophysical Journal. 120 (16), 3483-3497 (2021).
- Bhattacharjee, T., Amchin, D. B., Alert, R., Ott, J. A., Datta, S. S. Chemotactic smoothing of collective migration. eLife. 11, e71226 (2022).
- Tashman, J. W., Shiwarski, D. J., Feinberg, A. W. A high performance open-source syringe extruder optimized for extrusion and retraction during FRESH 3D bioprinting. HardwareX. 9, e00170 (2021).
- Crocker, J. C., Grier, D. G. Methods of digital video microscopy for colloidal studies. Journal of Colloid and Interface Science. 179 (1), 298-310 (1996).
- Chatterjee, T., Chatterjee, B. K., Chakrabarti, P. Modelling of growth kinetics of Vibrio cholerae in presence of gold nanoparticles: Effect of size and morphology. Scientific Reports. 7 (1), 9671 (2017).
- Martínez-Calvo, A., et al. Morphological instability and roughening of growing 3D bacterial colonies. Proceedings of the National Academy of Sciences. 119 (43), e2208019119 (2022).
- Lehner, B. A. E., Schmieden, D. T., Meyer, A. S. A Straightforward approach for 3D bacterial printing. ACS Synthetic Biology. 6 (7), 1124-1130 (2017).
- Zhang, Q., et al. Morphogenesis and cell ordering in confined bacterial biofilms. Proceedings of the National Academy of Sciences. 118 (31), e2107107118 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten