Method Article
Impression 3D de bactéries pour étudier la motilité et la croissance dans des milieux poreux 3D complexes
Dans cet article
Résumé
Ce protocole décrit une procédure d’impression tridimensionnelle (3D) de colonies bactériennes pour étudier leur motilité et leur croissance dans des matrices d’hydrogel poreuses 3D complexes qui s’apparentent davantage à leurs habitats naturels que les cultures liquides conventionnelles ou les boîtes de Pétri.
Résumé
Les bactéries sont omniprésentes dans les environnements poreux tridimensionnels complexes (3D), tels que les tissus et les gels biologiques, ainsi que les sols et les sédiments souterrains. Cependant, la majorité des travaux antérieurs se sont concentrés sur l’étude de cellules dans des liquides en vrac ou sur des surfaces planes, qui ne récapitulent pas entièrement la complexité de nombreux habitats bactériens naturels. Ici, cette lacune dans les connaissances est comblée en décrivant le développement d’une méthode pour imprimer en 3D des colonies denses de bactéries dans des matrices d’hydrogel granulaires coincées. Ces matrices ont des tailles de pores et des propriétés mécaniques réglables ; ils confinent physiquement les cellules, les soutenant ainsi en 3D. Ils sont optiquement transparents, ce qui permet de visualiser directement la propagation bactérienne dans leur environnement à l’aide de l’imagerie. Comme preuve de ce principe, ici, la capacité de ce protocole est démontrée par l’impression 3D et l’imagerie de Vibro cholerae non mobile et mobile, ainsi que d’Escherichia coli non mobile, dans des matrices d’hydrogel granulaires coincées avec des pores interstitiels de différentes tailles.
Introduction
Les bactéries habitent souvent des environnements poreux 3D divers et complexes allant des gels muqueux dans l’intestin et les poumons au sol dans le sol 1,2,3,4,5,6,7,8,9,10,11,12,13,14,15, 16,17,18,19,20,21,
22,23,24,25. Dans ces contextes, le mouvement bactérien par motilité ou croissance peut être entravé par des obstacles environnants, tels que des réseaux de polymères ou des empilements de grains minéraux solides, influençant la capacité des cellules à se propager dans leur environnement26, à accéder aux sources de nutriments, à coloniser de nouveaux terrains et à former des communautés de biofilms protectrices27. Cependant, les études de laboratoire traditionnelles utilisent généralement des géométries très simplifiées, en se concentrant sur des cellules dans des cultures liquides ou sur des surfaces planes. Bien que ces approches fournissent des informations clés sur la microbiologie, elles ne récapitulent pas entièrement la complexité des habitats naturels, ce qui entraîne des différences spectaculaires dans les taux de croissance et le comportement de motilité par rapport aux mesures effectuées dans des contextes réels. Par conséquent, une méthode pour définir les colonies bactériennes et étudier leur motilité et leur croissance dans des environnements poreux 3D plus proches de bon nombre de leurs habitats naturels est absolument nécessaire.
L’inoculation de cellules dans un gel de gélose puis la visualisation de leur propagation macroscopique à l’œil nu ou à l’aide d’une caméra fournit un moyen simple d’y parvenir, comme l’ont proposé Tittsler et Sandholzer en 193628. Cependant, cette approche souffre d’un certain nombre de défis techniques clés : (1) alors que la taille des pores peut, en principe, être modifiée en faisant varier la concentration d’agarose, la structure des pores de ces gels est mal définie ; (2) La diffusion de la lumière rend ces gels troubles, ce qui rend difficile la visualisation des cellules à l’échelle individuelle avec une résolution et une fidélité élevées, en particulier dans les grands échantillons ; (3) Lorsque la concentration de gélose est trop importante, la migration cellulaire est limitée à la surface plane supérieure du gel ; (4) La rhéologie complexe de ces gels rend difficile l’introduction d’inoculums avec des géométries bien définies.
Pour remédier à ces limites, le laboratoire de Datta a développé une approche alternative utilisant des matrices d’hydrogel granulaires - composées de particules d’hydrogel biocompatibles coincées gonflées dans une culture bactérienne liquide - comme « boîtes de Pétri poreuses » pour confiner les cellules en 3D. Ces matrices sont des solides mous, auto-cicatrisants, à limite d’élasticité ; ainsi, contrairement aux gels réticulés utilisés dans d’autres procédés de bio-impression, une microbuse d’injection peut se déplacer librement à l’intérieur de la matrice le long de n’importe quel chemin 3D prescrit en réorganisant localement les particules d’hydrogel29. Ces particules se redensifient ensuite rapidement et s’auto-réparent autour des bactéries injectées, soutenant les cellules en place sans aucun traitement nocif supplémentaire. Ce procédé est donc une forme d’impression 3D qui permet d’organiser des cellules bactériennes - dans une structure 3D souhaitée, avec une composition de communauté définie - au sein d’une matrice poreuse aux propriétés physico-chimiques réglables. De plus, les matrices d’hydrogel sont complètement transparentes, ce qui permet de visualiser directement les cellules à l’aide de l’imagerie.
L’utilité de cette approche a déjà été démontrée de deux manières. Dans une série d’études, des cellules diluées ont été dispersées dans la matrice de l’hydrogel, ce qui a permis d’étudier la motilité des bactéries individuelles30,31. Dans une autre série d’études, les communautés multicellulaires ont été imprimées en 3D dans des gels à l’échelle centimétrique à l’aide d’une buse d’injection montée sur une platine de microscope programmable, ce qui a permis d’étudier la propagation des collectifs bactériens dans leur environnement32,33. Dans les deux cas, ces études ont révélé des différences jusqu’alors inconnues dans les caractéristiques de propagation des bactéries habitant des environnements poreux par rapport à celles de la culture liquide/sur des surfaces planes. Cependant, étant donné qu’elles étaient montées sur une platine de microscope, ces études antérieures étaient limitées à de petits volumes d’échantillons (~1 ml) et, par conséquent, à des échelles de temps expérimentales courtes. Ils étaient également limités dans leur capacité à définir des géométries d’inocula avec une haute résolution spatiale.
Ici, la prochaine génération de cette plate-forme expérimentale qui répond à ces deux limites est décrite. Plus précisément, des protocoles sont fournis par lesquels on peut utiliser une imprimante 3D modifiée avec un extrudeur de seringue attaché pour imprimer en 3D et imager des colonies bactériennes à grande échelle. De plus, des données représentatives indiquent comment cette approche peut être utile pour étudier la motilité et la croissance des bactéries, en utilisant les exemples de Vibrio cholerae et d’Escherichia coli planctonique. Cette approche permet de maintenir les colonies bactériennes sur de longues périodes et de les visualiser à l’aide de diverses techniques d’imagerie. Par conséquent, la capacité de cette approche à étudier les communautés bactériennes dans les habitats poreux 3D a un énorme potentiel de recherche et d’application, ayant un impact sur le traitement et l’étude des microbes dans l’intestin, la peau, les poumons et le sol. De plus, cette approche pourrait être utilisée à l’avenir pour imprimer en 3D des matériaux vivants à base de bactéries dans des formes autoportantes plus complexes.
Protocole
Cette approche consiste à convertir une imprimante 3D commerciale de modélisation par dépôt de fil fondu en une bio-imprimante 3D en utilisant un protocole précédemment établi par Tashman et al.34. En bref, Tashman et al. ont remplacé une tête d’extrudeuse commerciale par une extrudeuse à seringue sur mesure. Cette extrudeuse permet d’imprimer en 3D des suspensions liquides hautement concentrées de cellules bactériennes, avec son volume extrudé et sa position 3D contrôlés par le langage de programmation G-code. Le volume extrudé est spécifié dans le logiciel par l’étape de l’extrudeuse (E-step) et est en outre calibré comme décrit ci-dessous. Ces suspensions bactériennes sont ainsi imprimées directement dans une matrice d’hydrogel granulaire, qui agit comme un support 3D pour les cellules. Ci-dessous, le protocole décrit également comment préparer des matrices avec différentes concentrations de polymères, caractériser les changements résultants de la taille des pores et des propriétés rhéologiques, et caractériser la motilité et la croissance bactériennes ultérieures à l’aide de l’imagerie directe.
1. Conversion d’une imprimante 3D commerciale en bio-imprimante 3D
- Retirez l’extrudeuse et le chauffage d’une imprimante 3D commerciale (voir Tableau des matériaux).
- Suivez les protocoles précédents pour fabriquer l’extrudeuse de pousse-seringue34, avec une modification supplémentaire pour accueillir une seringue Luer lock jetable. Montez l’extrudeuse de pousse-seringue sur l’imprimante.
REMARQUE : Les fichiers CAO requis pour modifier l’extrudeuse de pousse-seringue pour les seringues en plastique sont fournis dans les fichiers supplémentaires 1 à 3. - Installez et ouvrez le logiciel de l’imprimante 3D (voir la table des matériaux) sur un ordinateur. Connectez l’imprimante 3D à l’ordinateur.
- Chargez une seringue jetable de 1 ml avec une aiguille de taille appropriée dans les pinces imprimées en 3D en alignant les moitiés supérieure et inférieure du mécanisme (Figure 1). Fixez les pinces autour de la seringue à l’aide de trois boulons à douille M8 et d’écrous hexagonaux en acier minces (voir tableau des matériaux). Le piston de la seringue se connecte à la vis mère de l’extrudeuse de la pompe à seringue. Soulevez manuellement le piston en tournant la vis-mère pour créer un espace d’air de 0,5 mL dans la seringue.
- Si la contamination est un problème pour l’expérience, transporter le complexe seringue-pince dans un biocapot et stériliser avec un spray d’éthanol à 70 % à l’entrée avant de terminer les étapes suivantes.
2. Préparation de la suspension bactérienne
- Pour V. cholerae et E. coli, cultiver pendant la nuit sur une gélose Lennox LB (bouillon de Luria, voir tableau des matières) à 2 % à 37 °C.
- Pour V. cholerae, inoculer les cellules dans 3 mL de LB liquide avec dix billes de verre stériles. Cultiver les cellules dans un incubateur à agitation à 37 °C pendant 5 à 6 h jusqu’à la phase exponentielle moyenne jusqu’à une densité optique (OD) 600 de ~0,9.
- Dans le cas d’E. coli, inoculer les cellules dans 3 mL de LB liquide. Cultiver les cellules dans un incubateur à agitation à 37 °C pendant la nuit. Innoculer 200 μL de la culture de nuit dans des LB frais pendant 3 h jusqu’à ce que la DO atteigne 0,6.
- Transférer la culture dans un tube à centrifuger de 10 ml. Centrifuger la culture pendant 5 min à 5 000 x g à température ambiante pour former une pastille. Retirez le surnageant. Remettre en suspension avec ~10 μL de LB liquide pour obtenir une densité cellulaire de ~9 x10 10 cellules par mL.
3. Chargement de la suspension bactérienne dans la seringue
REMARQUE : Deux méthodes sont fournies pour charger les bactéries dans la seringue (étape 3.1 et étape 3.2). L’étape 3.1 fonctionne pour charger de petits volumes de suspensions bactériennes, <200 μL, et l’étape 3.2 fonctionne pour charger de plus grands volumes de suspensions bactériennes, >200 μL. L’étape 3.1 a été utilisée pour les résultats représentatifs présentés ici.
- Chargez une seringue Luer lock en plastique vide de 1 ml dans la bio-imprimante 3D. Connectez le piston de la seringue avec la vis-mère. Rétractez manuellement la seringue pour ajouter 0,2 mL de l’entrefer afin de laisser de l’espace pour que le piston se déplace dans la seringue, car un petit volume de cellules ~20-50 μL est utilisé pour chaque lot d’expériences.
- Fixez une aiguille émoussée à l’extrémité de la seringue avec la taille d’aiguille nécessaire pour la taille des caractéristiques d’impression requises. Ici, une aiguille de 2 pouces 20 G est utilisée.
- Chargez la suspension bactérienne dans la seringue en plaçant un tube à centrifuger de 10 mL avec l’inoculum bactérien sous l’aiguille. Tournez manuellement la vis pour rétracter le piston de la seringue et chargez les cellules dans la seringue. Les volumes de cellules bactériennes sont si petits que souvent, les cellules ne sont chargées que dans l’aiguille.
- Retirez le piston du complexe seringue-pince et utilisez une autre seringue et une aiguille pour charger soigneusement le complexe seringue-pince avec les suspensions bactériennes souhaitées, en prenant soin d’éviter de piéger les bulles d’air. Le complexe seringue-pince doit être rempli légèrement sur le bord avec la suspension bactérienne souhaitée, puis transféré dans la bio-imprimante.
- Insérez soigneusement le complexe seringue-pince sans le piston dans la douille correspondante sur le noyau principal de l’extrudeuse bio-imprimante.
- Assurez-vous que le chariot de l’imprimante est à peu près à mi-hauteur de la vis-mère et qu’un plat de collecte est présent sous la seringue chargée. Ensuite, insérez soigneusement le piston à travers le chariot et le complexe seringue-pince jusqu’à ce qu’il s’accroche au chariot. Enfoncez lentement le piston dans la suspension bactérienne pour éviter de piéger des bulles d’air dans la seringue.
- Faites glisser la pince d’adaptation sur le chariot à l’arrière du piston pour le fixer en place pour les manœuvres d’extrusion et de rétraction.
4. Étalonnage de l’étape de l’extrudeuse au volume déposé
- Pour calibrer l’étape de l’extrudeuse (E-step) au volume déposé, configurez d’abord la bio-imprimante avec la seringue, l’aiguille de seringue et la suspension bactérienne de dépôt exactes qui seront utilisées dans l’expérience. Ici, une seringue Luer lock de 1 ml est utilisée.
- Déterminez une plage de pas E estimée à étalonner en extrudant un nombre arbitraire de pas E (~200) et notez le changement de volume du piston avec des marqueurs de volume de seringue.
- Utilisez ce rapport E-step/volume grossier pour déterminer les paramètres E-step pour effectuer le balayage d’étalonnage. Par exemple, si un E-step de 200 extrude environ 20 μL par inspection visuelle et que l’on veut déposer 10-200 μL, testez E-steps entre 100-2000.
- Pour effectuer le balayage d’étalonnage linéaire, étiquetez et mesurez d’abord la masse sèche de vingt tubes d’échantillonnage de 1,5 mL sur une balance analytique avec une sensibilité de 0,1 mg.
- Extruder la suspension bactérienne dans les tubes pré-mesurés de 1,5 ml. Pour chaque étape E, effectuez au moins 2 répétitions. Répétez l’opération pour tous les E-steps sur la plage linéaire, en remplaçant la suspension bactérienne si nécessaire. Si la contamination est un problème pour l’expérience, essuyez l’extérieur de l’aiguille de la seringue avec une lingette non pelucheuse saturée d’éthanol à 70 % après chaque échantillon.
- Mesurez la masse de tous les tubes de 1,5 mL avec la même balance analytique. Soustrayez la première valeur de masse de la seconde pour obtenir une masse nette de suspension bactérienne déposée.
- Convertir la masse de suspension bactérienne en un volume avec la densité du matériau. Pour de nombreuses suspensions bactériennes composées principalement d’eau, 1 g/mL est une approximation de densité appropriée.
- Effectuez un ajustement linéaire entre le pas en E et le volume extrudé pour terminer le processus d’étalonnage.
5. Préparation de la matrice d’hydrogel granulaire
- Dans une enceinte de sécurité biologique, ajouter des granules secs de copolymères d’acide acrylique réticulé et d’acrylate d’alkyle (voir le tableau des matériaux) à 400 mL de Lennox Luria-Bertani (LB) à 2 % pour maintenir la matrice stérile ; Cependant, d’autres milieux de culture cellulaire liquides peuvent également être utilisés pour gonfler la matrice d’hydrogel.
REMARQUE : Le pourcentage en poids des granulés ajoutés au LB dépend de la taille des pores que l’on vise. Dans la présente étude, pour une matrice d’hydrogel granulaire à 0,9 %, 3,6 g de granulés secs sont ajoutés à la LB et pour une matrice d’hydrogel granulaire à 1,2 %, 4,8 g de granulés secs sont ajoutés à la LB. Les granulés d’hydrogel sont dispersés de manière homogène en les mélangeant dans un batteur sur socle pendant 2 min. - Une fois mélangé, ajuster le pH à 7,4 en ajoutant des incréments de 20 à 500 μL d’hydroxyde de sodium (NaOH) de 10 M pour assurer la viabilité cellulaire. Après chaque ajout de NaOH, mesurez le pH en trempant une pointe de pipette dans le mélange, puis en essuyant la matrice d’hydrogel sur un papier réactif de pH.
REMARQUE : Au fur et à mesure que le NaOH est ajouté, la viscosité du mélange augmentera à mesure que les granulés d’hydrogel commenceront à gonfler. Les granules d’hydrogel gonflés ont un diamètre de ~5 μm à 10 μm et sont coincés ensemble dans une matrice d’hydrogel. La taille des mailles internes des granulés est de ~40 nm à 100 nm, comme précédemment établi32. La taille des mailles est suffisamment grande pour que les petites molécules (par exemple, l’oxygène et les nutriments) diffusent librement, mais suffisamment petites pour que les bactéries soient confinées entre les pores interstitiels. - Ensuite, transférez la matrice d’hydrogel granulaire dans un tube à centrifuger de 50 ml à l’aide d’une seringue en plastique stérile de 50 ml. Centrifuger la matrice d’hydrogel à 161 x g pendant 1 min à température ambiante pour éliminer les bulles formées pendant le processus de mélange.
- Laissez la matrice d’hydrogel reposer pendant au moins 2 jours à température ambiante pour vous assurer qu’aucune contamination ne s’est produite. La contamination se présente sous forme de microcolonies en suspension dans la matrice d’hydrogel. Après deux jours, centrifugez la matrice d’hydrogel à 161 x g pendant 1 min pour éliminer les bulles supplémentaires qui se sont formées.
REMARQUE : Le protocole peut être interrompu ici en stockant la matrice d’hydrogel à température ambiante jusqu’à une semaine. - Dans l’enceinte de sécurité biologique, à l’aide d’une seringue en plastique stérile de 30 ml, transférez la quantité désirée de matrice d’hydrogel dans le récipient où l’impression aura lieu (ici ~20 ml pour un flacon de culture tissulaire de 20 ml ou 1 ml pour des micro-cuvettes en plastique de 1 ml ont été utilisés).
6. Caractérisation des propriétés rhéologiques de la matrice granulaire de l’hydrogel
- Chargez ~3 mL de la matrice d’hydrogel dans un rhéomètre de cisaillement (voir Tableau des matériaux) avec un espace de 1 mm entre des plaques parallèles rugueuses de 50 mm de diamètre pour mesurer les propriétés rhéologiques.
- Quantifier le comportement d’élasticité à l’aide de mesures de cisaillement unidirectionnelles sur le rhéomètre de cisaillement en mesurant la contrainte de cisaillement en fonction d’un balayage logarithmique du taux de cisaillement de 10-4 s-1 à 102 s-1 (par exemple, Figure 2A).
REMARQUE : À de faibles taux de cisaillement, la matrice d’hydrogel aura une contrainte de cisaillement constante (la limite d’élasticité) qui est indépendante de la vitesse de cisaillement. À des taux de cisaillement élevés, la contrainte de cisaillement augmentera avec une dépendance de la loi de puissance sur le taux de cisaillement, indiquant la fluidisation de la matrice d’hydrogel. Ce comportement d’élasticité-stress permet d’imprimer en 3D des bactéries dans la matrice granulaired’hydrogel 29. - Mesurer les modules de stockage et de perte, G' et G'' respectivement, en fonction de la fréquence en utilisant une rhéologie oscillatoire de faible amplitude avec une amplitude de déformation de 1% et des fréquences comprises entre 0,1 et 1 Hz (par exemple, Figure 2B).
REMARQUE : La matrice d’hydrogel granulaire idéale pour l’impression 3D doit avoir un module de stockage supérieur au module de perte, ce qui indique que le support agit comme un solide élastiquecoincé 29.
7. Caractérisation de la taille des pores interstitiels de la matrice d’hydrogel granulaire
- Sonicate 100 nm de nanoparticules de polystyrène fluorescent carboxylé (~3,6 x10 13 particules/mL, voir tableau des matériaux) dans leur emballage pendant 15 min pour les remettre en suspension afin de briser les agrégats/amas de particules. Transférer 50 μL de nanoparticules dans un tube de microcentrifugation de 1,5 mL.
- Centrifuger pendant 10 min à 9 500 x g à température ambiante jusqu’à ce que la pastille se forme et que le surnageant soit clair. Retirer le surnageant et remettre la pastille en suspension dans 1 mL du milieu de croissance liquide (ici LB) utilisé pour préparer la matrice d’hydrogel granulaire.
REMARQUE : Le protocole peut être interrompu ici en stockant les nanoparticules remises en suspension à 4 °C pendant 3 mois maximum.
- Centrifuger pendant 10 min à 9 500 x g à température ambiante jusqu’à ce que la pastille se forme et que le surnageant soit clair. Retirer le surnageant et remettre la pastille en suspension dans 1 mL du milieu de croissance liquide (ici LB) utilisé pour préparer la matrice d’hydrogel granulaire.
- Soniquer les nanoparticules remises en suspension pendant 30 min. Transférer 1 mL de matrice d’hydrogel granulaire dans un tube de microcentrifugation de 1,5 mL. Ajoutez 1 μL de nanoparticules remises en suspension dans la matrice d’hydrogel granulaire et mélangez-les avec une pointe de pipette. Après mélange, centrifuger pendant 30 s à 161 x g à température ambiante.
- Transférez la matrice d’hydrogel et le mélange de nanoparticules dans une boîte de Pétri de 35 mm de diamètre avec un puits à fond en verre de 0,1 mm d’épaisseur. Le puits mesure 20 mm de diamètre et 1 mm de profondeur. Placez une lamelle en verre sur le dessus et appuyez pour désactiver l’écoulement et l’évaporation pendant l’imagerie. Une alternative à la lamelle en verre consiste à ajouter 1 ml d’huile de paraffine sur le dessus.
- Imagez les nanoparticules à l’aide d’un microscope confocal avec un objectif à huile 40x avec un zoom supplémentaire 8x dans le logiciel d’imagerie (voir Tableau des matériaux).
REMARQUE : Un objectif à grossissement plus élevé peut être utilisé au lieu d’utiliser un zoom numérique supplémentaire dans le logiciel.- Image d’une boucle temporelle sans délai (idéalement ~19 images/s) dans un seul plan z pendant 2 min avec au moins quatre nanoparticules dans le champ de vision. Répétez 15 à 20 fois à différents endroits pour collecter suffisamment de données pour les statistiques (100 à 200 nanoparticules).
- Utilisez un logiciel de suivi des particules pour analyser le déplacement des particules. Ici, un script personnalisé est utilisé sur la base de l’algorithme classique de Crocker-Grier pour suivre le centre de masse de la nanoparticule35 (voir le fichier supplémentaire 7).
- À partir du suivi des particules, calculez le déplacement quadratique moyen (MSD). Le MSD présentera une diffusion libre dans l’espace interstitiel à de courtes longueurs et échelles de temps et passera à une mise à l’échelle sous-diffusive à de grandes longueurs et échelles de temps en raison du confinement35.
- Identifier l’échelle de longueur où se produit la transition vers la mise à l’échelle sous-diffusive pour estimer la taille locale des pores. Calculez la taille des pores en ajoutant cette échelle de longueur au diamètre de la nanoparticule. Répétez l’analyse de la taille des pores pour chaque nanoparticule mesurée. Cela donnera une distribution de la taille des pores à partir de laquelle une taille moyenne des pores peut être calculée (par exemple, Figure 3).
8. 3D processus d’impression
- Imprimez en 3D des supports sur mesure pour les conteneurs d’échantillons (voir les fichiers supplémentaires 4-6 pour les fichiers CAO). Ici, des supports pour les flacons de culture tissulaire et les microcuvettes sont utilisés. Les supports permettent de programmer l’imprimante pour imprimer plusieurs échantillons en une seule session d’impression. Placez les récipients d’échantillons avec des supports d’hydrogel dans les supports de la plate-forme de construction.
- Ouvrez le logiciel d’impression 3D. Chargez un code g préprogrammé dans le logiciel. Pour obtenir des résultats représentatifs, l’étape 3.1 est utilisée pour charger la suspension bactérienne dans l’imprimante 3D.
REMARQUE : Un exemple de code g pour l’impression de géométries verticales linéaires est donné dans le Tableau 1. - Grâce au logiciel d’impression 3D, déplacez les plans x-y-z pour centrer la tête d’impression sur le plan x-y pour être le premier conteneur, puis accueillez l’axe z. L’axe z de guidage soulèvera la tête d’impression. Tournez manuellement la vis lentement pour enfoncer le piston de la seringue jusqu’à ce qu’une petite quantité de suspension bactérienne soit visible à l’extrémité de l’aiguille.
- Essuyez légèrement l’excès de suspension bactérienne avec une lingette stérile jetable. En fonction de la hauteur du porte-échantillon, de l’aiguille et de la seringue, à l’aide du logiciel d’impression 3D, abaissez la tête d’impression à une distance fixe dans le support d’hydrogel dans le récipient d’échantillon de votre choix. Lancez le processus d’impression en cliquant sur Imprimer.
- Une fois l’impression terminée, fermez les récipients d’échantillons. Essuyez l’imprimante avec de l’éthanol à 70 %. Jetez correctement la seringue et l’aiguille.
9. Culture et imagerie de V . cholerae
- Pour l’imagerie à grand champ de vision, utilisez une caméra avec un zoom pour imager la croissance cellulaire avec une lightbox. Imagez les échantillons juste après l’impression à température ambiante, puis transférez-les dans un incubateur. Maintenir les échantillons à 37 °C dans un incubateur fixe entre les séances d’imagerie pendant l’expérience.
- Capturez des images sur une période de temps souhaitée pour observer le comportement de croissance à de longues périodes dans la matrice d’hydrogel granulaire.
Résultats
L’utilisation d’une bio-imprimante 3D avec la matrice d’hydrogel granulaire élargit les capacités des bio-imprimantes à imprimer des colonies bactériennes dans des formes qui, lorsqu’elles sont imprimées sur un substrat plat, s’affaiseraient en raison de la faible viscosité de la suspension bactérienne. La résolution de l’approche présentée ici dépend de la vitesse d’extrusion, de la taille de l’aiguille, de la vitesse de la tête d’impression, de l’air dans l’aiguille et de la viscosité de la suspension bactérienne. En raison du faible volume de suspension bactérienne, des bulles d’air peuvent être introduites par inadvertance lors du chargement de la suspension bactérienne dans la seringue et l’aiguille. Cela peut entraîner le dépôt d’une bulle d’air dans la structure imprimée finale (Figure 4). Une autre façon d’introduire des bulles d’air dans l’impression est de ne pas appuyer sur le piston pour former une petite goutte de suspension bactérienne à la pointe de l’aiguille avant l’impression et un espace d’air existe à la pointe de l’aiguille. Ne pas enfoncer le piston de la seringue avant l’impression peut également entraîner l’impression de différents volumes de cellules dans le même lot. Cependant, au fil du temps, la bulle d’air se dissout dans le milieu environnant, comme le montre la figure 4.
Pour calibrer l’étape d’extrusion, le volume déposé dépend de la façon dont l’actionneur linéaire de la tête d’impression traduit le piston de la seringue, le diamètre intérieur de la seringue aura un impact direct sur le volume. De plus, les propriétés rhéologiques de la suspension bactérienne auront un impact sur la facilité avec laquelle elles cisaillent la contraction de l’aiguille pour imprimer en douceur. Ainsi, cette procédure d’étalonnage doit être refaite pour chaque configuration de seringue/aiguille/suspension bactérienne. En principe, l’étalonnage des seringues pourrait être automatisé. Cependant, dans la pratique, il serait difficile d’écrire un tel code qui s’applique largement à de nombreux cas d’utilisation. Par exemple, un utilisateur souhaitant calibrer l’extrusion d’environ 300 μL à partir d’une seringue de 1 mL devrait recharger la seringue beaucoup plus fréquemment qu’un utilisateur étalonnant autour d’un volume cible de 30 μL. Étant donné que la constante d’étalonnage E-step to volume est inconnue au début du processus d’étalonnage, l’utilisateur peut ne pas être en mesure de prédire exactement à quelle fréquence le rechargement est réellement nécessaire. De plus, pour automatiser le processus d’étalonnage, les positions exactes de chaque tube de 1,5 mL prépesé devraient être spécifiées. Pour s’assurer que toute la bio-encre est déposée de l’aiguille dans le tube pour un étalonnage précis, un contact précis doit être établi entre l’aiguille et le fond/la paroi du tube. Sans un bon contact, de petites gouttelettes peuvent rester mouillées et attachées à l’aiguille. Ainsi, toute variation de position entre les tubes peut grandement contribuer aux erreurs dans le processus d’étalonnage. Pour ces raisons, les auteurs recommandent à chaque utilisateur de créer un programme d’étalonnage qui répond à ses besoins uniques.
Une caractéristique clé de l’approche présentée ici est la capacité de visualiser les bactéries se propageant directement dans leur environnement via la motilité et la croissance à l’aide de l’imagerie. Dans une version antérieure du protocole d’imagerie, des plaques à 6 puits étaient remplies de 19 mL d’une matrice d’hydrogel granulaire. Cependant, même avec une impression 3D réussie d’une ligne horizontale de cellules pouvant être observée sur un microscope inversé, une colonie dense se développerait également sur la surface supérieure du milieu, limitant la visualisation avec la microscopie à fond clair (Figure 5). La colonie sur la surface supérieure résulte probablement d’une contamination lorsque l’aiguille de la seringue a été placée dans le support ou retirée du support pendant l’impression. Pour contourner ce problème, des lignes verticales de cellules sont imprimées dans des flacons à scintillation (Figure 6A). Cependant, la courbure des flacons cylindriques provoquait une distorsion lors de l’imagerie. Cela a conduit à la sélection de cuvettes à paroi plate et de flacons de culture tissulaire comme récipients d’échantillons pour l’impression, permettant une imagerie non déformée (Figure 6B-D). L’une des limites des flacons de culture tissulaire est le petit col incliné qui limite les géométries pouvant être imprimées.
L’ensemble de ces protocoles permet d’observer la motilité et la croissance bactériennes dans des environnements poreux complexes sur de longues échelles de temps. Quelques exemples sont présentés à la figure 6B-G pour V. cholerae formant un biofilm à l’aide de la microscopie à fond clair ainsi que pour les cellules planctoniques d’E. coli à l’aide de la microscopie confocale à fluorescence à balayage laser, démontrant la polyvalence de cette approche. En effet, un problème potentiel des matrices d’hydrogel est leur possible autofluorescence lorsqu’elles sont imagées à l’aide de la microscopie à fluorescence ; cependant, les images présentées dans la figure 6F,G démontrent qu’une telle autofluorescence est minime dans la plateforme expérimentale présentée ici. Une autre limite de ces approches de microscopie optique est leur résolution spatiale, qui est fixée par la limite de diffraction à ~100 s nanomètres ; cependant, cette échelle de longueur est beaucoup plus petite que la taille d’une cellule bactérienne individuelle et, par conséquent, les techniques optiques permettent d’imager les cellules bactériennes de l’échelle des cellules individuelles (Figure 6G) à l’échelle de colonies multicellulaires plus grandes 30,31,32,33. D’autres exemples sont décrits ci-dessous.
Comme indiqué ci-dessus, l’approche présentée ici peut être utilisée pour imprimer en 3D et imager des colonies bactériennes dans de petits (1 ml) et de grands volumes matriciels (20 ml). Par conséquent, les différences dans les résultats obtenus en utilisant différents volumes sont décrites ci-dessous, en utilisant des colonies imprimées en 3D de V. cholerae mobiles et non mobiles comme exemples représentatifs. La résolution spatiale (largeur de la ligne) de l’impression est définie par le diamètre intérieur de la buse. Dans les exemples décrits ci-dessous, une aiguille d’un diamètre intérieur de 0,6 mm donne une colonie cylindrique initiale de 0,6 mm. Des travaux antérieurs ont démontré que la résolution d’impression peut être encore réduite en utilisant un capillaire en verre tiré d’un diamètre intérieur de ~100-200 μm33.
Pour les petits volumes, deux matrices d’hydrogel granulaires différentes avec deux distributions de taille de pores différentes sont utilisées : l’une avec une taille moyenne de pores supérieure au diamètre moyen d’une cellule de V. cholerae, 0,2-0,4 μm36, et l’autre avec une taille moyenne de pores inférieure à ce diamètre. Les échantillons sur douze jours sont imagés et mesurés par l’analyse d’images de l’expansion surfacique des colonies au fil du temps (figure 7 et figure 8). Pour les cellules non mobiles, qui ne peuvent se propager dans leur environnement que par croissance cellulaire, le taux d’expansion surfacique était similaire entre les différentes matrices étudiées (Figure 7), indiquant que les différences de taille des pores de la matrice n’influencent pas la propagation cellulaire par la croissance - comme prévu. En revanche, pour les cellules mobiles qui se propagent dans leur environnement par motilité active, le taux d’expansion surfacique de V. cholerae était plus élevé pour la matrice d’hydrogel avec les pores plus grands, pour laquelle le confinement par les grains d’hydrogel entrave moins la motilité cellulaire. Ces différences dans la propagation bactérienne étaient également apparentes dans les morphologies des colonies (figure 8). La colonie de V. cholerae dans des matrices d’hydrogel à pores plus grands s’est propagée à travers des panaches lisses et diffus (Figure 8A), reflétant la propagation par motilité active comme observé précédemment32. En effet, conformément à cette interprétation, ces panaches diffus ne sont pas observés dans le cas des cellules non mobiles (Figure 8C). De plus, les pores sont suffisamment grands pour que les cellules ne poussent pas les perles lorsqu’elles nagent à travers les pores. De plus, la contrainte visqueuse appliquée par la nage est inférieure à 1 Pa, ce qui est insuffisant pour déformer sensiblement la matrice d’hydrogel environnante. En revanche, la colonie dans les matrices d’hydrogel avec des pores plus petits ne se propage que par des panaches rugueux de type fractal pour les cellules mobiles et non mobiles (Figure 8B,D), reflétant la propagation uniquement par la croissance cellulaire, tandis que les cellules se développent, elles se déforment transitoirement et produisent la matrice environnante. En fait, étant donné que la limite d’élasticité des matrices d’hydrogel est beaucoup plus faible que la pression de turgescence des cellules, dans cette limite de petite taille de pores, la matrice n’offre qu’une faible résistance à la croissance cellulaire et ne semble pas affecter fortement la structure imprimée en 3D, également comme vérifié dans nos travaux précédents37.
Des résultats similaires sont observés pour les expériences réalisées sur des échantillons de grand volume ; cependant, étant donné la plus grande abondance de nutriments dans ces échantillons, les expériences pourraient soutenir la croissance cellulaire sur des échelles de temps plus longues. À titre d’exemple, des résultats utilisant les matrices d’hydrogel granulaires où la taille moyenne des pores était inférieure à la taille de la cellule sont montrés - et donc, la propagation cellulaire était principalement due à la croissance. Les échantillons sont imagés pendant ~30 jours dans les matrices d’hydrogel granulaires et ont observé une expansion surfacique similaire pour les cellules non mobiles et mobiles pendant les 150 premières heures ; cependant, à des périodes encore plus longues, une forte variabilité est observée entre les échantillons, certains échantillons présentant des taux d’étalement plus rapides (figure 9 et figure 10). Il est intéressant de noter que lors du rééchantillonnage de la matrice d’hydrogel après l’expérience - un avantage du grand volume de la matrice d’hydrogel - une diminution de la limite d’élasticité, des modules de stockage et des modules de perte est mesurée (Figure 11), indiquant que les matrices sont devenues plus molles. Ce changement pourrait être dû au fait que V. cholerae produit une molécule qui modifie les propriétés de la matrice hydrogel et favorise la propagation cellulaire à long terme, ce qui sera intéressant à tester dans de futures recherches. Des exemples de morphologies de colonies rugueuses et fractales qui résultent de ces expériences sont présentés à la figure 10.

Figure 1 : Images de la bio-imprimante 3D sur mesure. (A) Bio-imprimante avec une tête d’extrusion de pousse-seringue modifiée avec une seringue et une aiguille Luer lock jetables. La bio-imprimante mesure ~46 cm de large. (B) Image de l’impression 3D de cellules dans des flacons remplis de matrice d’hydrogel coincée. La largeur de l’image est de 87 mm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
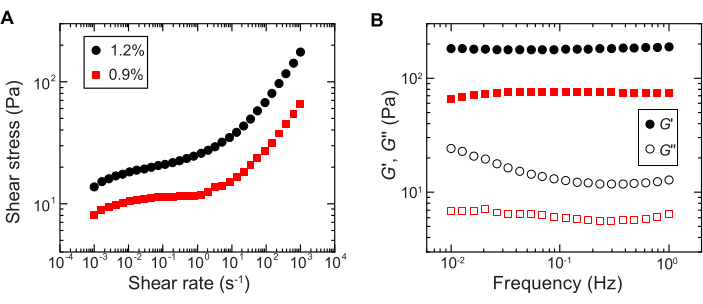
Figure 2 : Caractérisation des propriétés rhéologiques des matrices d’hydrogel granulaires. (A) Contrainte de cisaillement en fonction de la vitesse de cisaillement appliquée. (B) Modules de stockage et de perte, G' et G'' respectivement, en fonction de la fréquence d’oscillation. La légende indique la fraction massique d’hydrogel utilisée pour préparer chaque matrice d’hydrogel. Veuillez cliquer ici pour voir une version agrandie de cette figure.
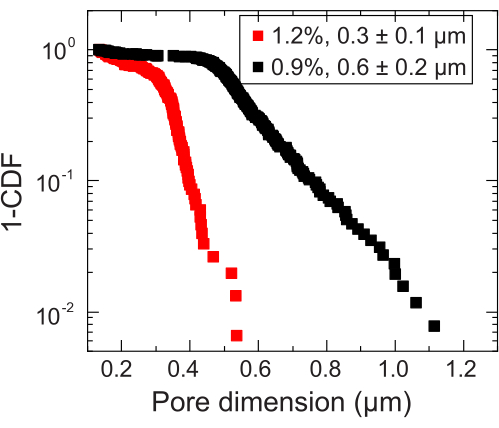
Figure 3 : Mesures de la taille des pores de deux matrices d’hydrogel granulaires représentatives. En suivant les traceurs, la distribution des dimensions caractéristiques des pores est déterminée pour chaque matrice d’hydrogel. Les données sont représentées par 1-CDF, où CDF est la fonction de distribution cumulative. La légende indique la fraction massique d’hydrogel utilisée pour préparer chaque matrice. Veuillez cliquer ici pour voir une version agrandie de cette figure.
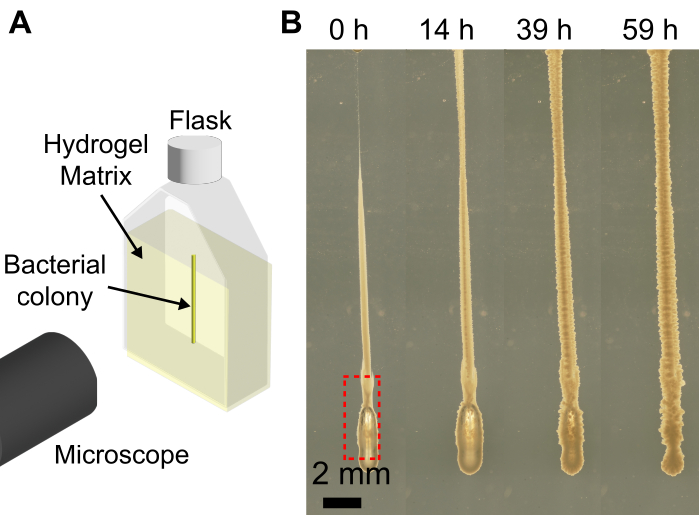
Figure 4 : Exemples de bulles formées lors de l’impression 3D de bactéries. (A) Schéma de l’installation d’imagerie. (B) Instantanés de la croissance de V. cholerae avec une bulle au bas de l’empreinte (encadré rouge) dans une matrice d’hydrogel à 1,2 % gonflée en LB. Après 59 h de croissance à 37 °C, la bulle d’air est complètement dissoute et la colonie s’effondre à nouveau en raison de l’élasticité de la matrice d’hydrogel. Barre d’échelle = 2 mm. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Images de la ligne horizontale de V. cholerae mobile imprimées dans une plaque à six puits remplie de matrice d’hydrogel à 1,2 % gonflée en LB et du biofilm qui s’est formé sur la surface supérieure après 48 h d’incubation à 37 °C. (A) Schéma de l’installation d’imagerie. (B) La formation de biofilm sur la surface supérieure due à la contamination lors de l’impression 3D diminue l’opacité et ne permet pas une imagerie claire de la ligne horizontale. La surface supérieure forme des plis, probablement dus à une croissance différentielle. Barre d’échelle = 5 mm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
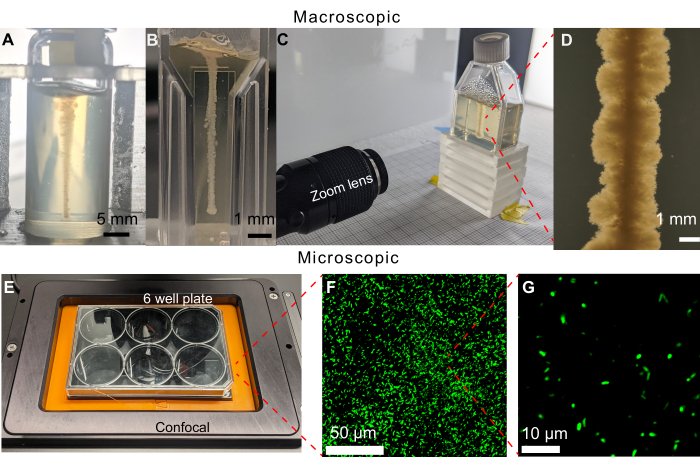
Figure 6 : Exemples de différents récipients d’échantillons pouvant être utilisés dans cette méthode. (A) V. cholerae imprimé dans un flacon en verre rempli d’une matrice d’hydrogel granulaire à 1,2 % après 470 h. La courbure du flacon rend difficile l’imagerie de la croissance. Barres d’échelle = 5 mm. (B) V. cholerae imprimé dans une micro-cuvette remplie d’une matrice d’hydrogel granulaire à 1,2 %. Les côtés plats permettent une imagerie claire, mais les petits volumes limitent la durée des expériences avant que les cellules ne manquent de substrats de croissance. (C) Installation d’imagerie avec un zoom pour imager V. cholerae imprimée dans un flacon de culture tissulaire rempli d’une matrice d’hydrogel granulaire à 1,2 %. Semblable au boîtier de la micro-cuvette, les côtés plats permettent une imagerie claire. Les flacons de culture tissulaire peuvent être remplis de plus grands volumes de matrice d’hydrogel granulaire, ce qui allonge la durée de l’expérience. (D) Image de la lentille zoom de V. cholerae imprimée à l’intérieur d’une matrice d’hydrogel granulaire à 1,2 % après 100 h d’incubation à 37 °C. Barre d’échelle = 1 mm. (E) Installation d’imagerie avec un microscope confocal pour imager les cellules fluorescentes. (F) Projection 3D de micrographies confocales d’E. coli fluorescent à l’intérieur de la matrice d’hydrogel granulaire à 1,2 % après 10 jours d’incubation à 37 °C. Barre d’échelle = 50 μm. (G) Micrographie confocale à résolution unicellulaire d’E. coli fluorescent à l’intérieur de la matrice d’hydrogel. Barre d’échelle = 10 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
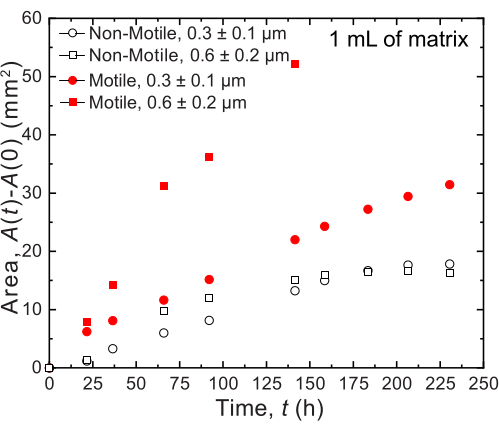
Figure 7 : Expansion de la surface en fonction du temps des colonies de V. cholerae imprimées dans 1 mL de matrices de support d’hydrogel granulaire. Les données sur le graphique proviennent de l’analyse d’image de la figure 8. Il a été observé que les cellules mobiles (cercle rouge fermé et carré) se propagent à un rythme plus rapide que les cellules non mobiles, reflétant la propagation à la fois par croissance et motilité. De plus, les cellules mobiles de la matrice d’hydrogel avec une grande taille de pores (carrés rouges) se propagent à un rythme plus rapide que les cellules de la matrice d’hydrogel avec des pores de 0,3 μm (cercles rouges). Les cellules non mobiles ne montrent aucune différence dans le taux d’expansion surfacique entre les deux tailles de pores, car elles ne font que croître. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 8 : Colonies de V. cholerae imprimées dans 1 mL de matrices de support d’hydrogel granulaire. (A) Instantanés de la croissance et de la motilité de V. cholerae mobile dans une matrice d’hydrogel granulaire à 0,9 % où la taille des pores est plus grande que le diamètre moyen des cellules. Les flèches indiquent un panache diffus qui se forme en raison du déplacement des cellules à travers la matrice d’hydrogel. (B) Instantanés de la croissance et de la motilité de V. cholerae mobile dans une matrice d’hydrogel granulaire à 1,2 % où la taille des pores est inférieure au diamètre moyen des cellules. Les flèches indiquent un panache rugueux, semblable à une fractale, qui se forme en raison de la croissance cellulaire. (C) Instantanés de l’évolution temporelle de V. cholerae non mobile dans une matrice d’hydrogel granulaire à 0,9 % où la taille des pores est plus grande que le diamètre moyen des cellules. Dans ce cas, les panaches diffus reflétant la motilité ne sont pas observables. (D) Instantanés de la croissance de V. cholerae non mobile dans une matrice de support d’hydrogel granulaire à 1,2 % où la taille des pores est inférieure au diamètre moyen des cellules. Dans ce cas, des panaches rugueux de type fractal reflétant la croissance sont à nouveau observables. Barres d’échelle = 1 mm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
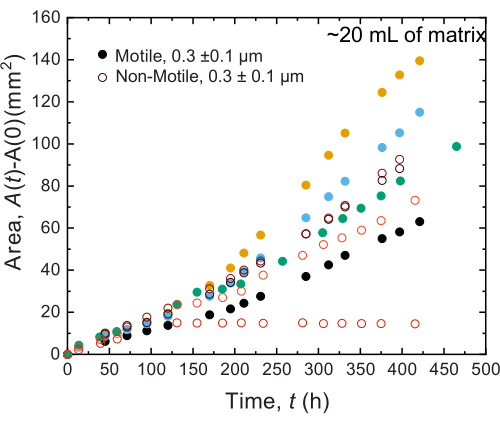
Figure 9 : Expansion de la surface en fonction du temps des colonies de V. cholerae imprimées dans 20 mL de matrices de support d’hydrogel granulaire à 1,2 %. On observe que les cellules non mobiles (cercles ouverts) et mobiles (cercles fermés) se propagent à des vitesses similaires pendant les 100 premières heures. Après 100 h, des différences dans les taux d’expansion surfacique sont observées, potentiellement dues à l’évolution de la variabilité à des moments longs. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 10 : Colonies de V. cholerae imprimées dans 22 mL de matrices d’hydrogel granulaires à 1,2 % cultivées à 37 °C. (En haut) Instantanés de la croissance de V. cholerae mobile. (En bas) Instantanés de la croissance de V. cholerae non mobile. Dans les deux cas, des panaches rugueux de type fractal reflétant la croissance sont observables. Barres d’échelle = 2 mm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
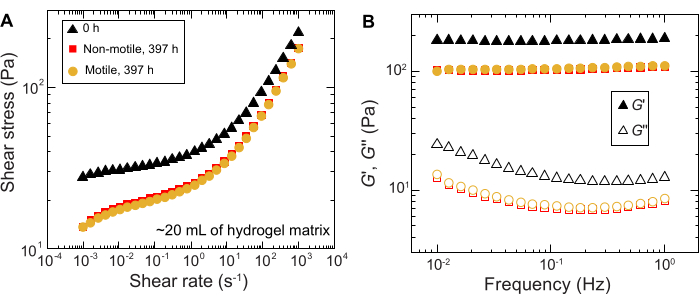
Figure 11 : Caractérisation des propriétés rhéologiques de la matrice d’hydrogel granulaire à 1,2 % avant (0 h) et après l’expérience (397 h). (A) Contrainte de cisaillement en fonction du taux de cisaillement appliqué. (B) Modules de stockage et de perte, G' et G'' respectivement, en fonction de la fréquence d’oscillation appliquée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Commandes Gcode | Tâches | |||
| M82 | Mode d’extrusion absolu | |||
| M302 S0 | Activer l’extrusion à froid | |||
| M92 E14575 | Définir les étapes d’extrusion par mm | |||
| G92 X35.61 Y81 Z0 E0.0 | Réglez l’axe z et la position d’extrusion à zéro, et la position x-,y- à 35,71 mm et y 88 mm où se trouve la tête d’impression au démarrage du g-code | |||
| M221 S100 T0 | règle le débit à 100 % | |||
| M107 | Éteignez le ventilateur | |||
| G1 F1 X35.61 Y81 E0.1 | Extruder les 20 μL de bio-encre dans la matrice à une vitesse d’alimentation de 50 μL/min où la position actuelle a été définie | |||
| G0 F200 Z60 | Retirez l’aiguille de la matrice et du récipient d’échantillon à une vitesse de 200 mm/min | |||
| G0 F500 X65.81 Y81.0 | Déplacer l’aiguille vers le conteneur d’échantillon suivant à une vitesse de 500 mm/min | |||
| G0 F100 Z0 | Abaissez l’aiguille dans le prochain récipient d’échantillon jusqu’à la même position z où l’impression a commencé à une vitesse de 100 mm/min | |||
| G1 F1 X65.81 Y81.0 E0.2 | Extruder les 20 μL de bio-encre dans la matrice à une vitesse d’alimentation de 50 μL/min où la position actuelle a été définie | |||
| G0 F200 Z60 | Retirez l’aiguille de la matrice et du récipient d’échantillon à une vitesse de 200 mm/min | |||
| G0 F500 X96.01 Y81.0 | Déplacer l’aiguille vers le conteneur d’échantillon suivant à une vitesse de 500 mm/min | |||
| G0 F100 Z0 | Abaissez l’aiguille dans le récipient d’échantillon à la même position z où l’impression a commencé à une vitesse de 100 mm/min | |||
| G1 F1 X96.01 Y81.0 E0.3 | Extruder les 20 μL de bio-encre dans la matrice à une vitesse d’alimentation de 50 μL/min où la position actuelle a été définie | |||
| G0 F200 Z60 | Retirez l’aiguille de la matrice et du récipient d’échantillon à une vitesse de 200 mm/min | |||
| G0 F500 X126.21 Y81.0 | Déplacer l’aiguille vers le conteneur d’échantillon suivant à une vitesse de 500 mm/min | |||
| G0 F100 Z0 | Abaissez l’aiguille dans le prochain récipient d’échantillon jusqu’à la même position z où l’impression a commencé à une vitesse de 100 mm/min | |||
| G1 F1 X126.21 Y81.0 E0.4 | Extruder les 20 μL de bio-encre dans la matrice à une vitesse d’alimentation de 50 μL/min où la position actuelle a été définie | |||
| G0 F200 Z80 | Retirez l’aiguille de la matrice et du récipient d’échantillon à une vitesse de 200 mm/min | |||
Tableau 1 : Programmation G-code pour l’impression de lignes verticales de la suspension bactérienne.
Fichier supplémentaire 1 : Fichier STL pour la pince inférieure Seringues Luer lock jetables de 1 mL pour l’extrudeuse de seringues. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 2 : Fichier STL pour la pince supérieure pour les seringues Luer lock jetables de 1 mL pour l’extrudeuse de seringues. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 3 : Fichier STL pour l’adaptateur de seringue pour seringues Luer lock jetables de 1 mL pour l’extrudeuse de seringues. Veuillez cliquer ici pour télécharger ce fichier.
Dossier supplémentaire 4 : Fichier STL pour le porte-échantillon de la cuvette. Veuillez cliquer ici pour télécharger ce fichier.
Dossier supplémentaire 5 : Fichier STL pour le porte-échantillon de la fiole de tissu. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 6 : Fichier STL pour la plaque à 6 puits et le lit d’impression de la boîte de Pétri de 35 mm. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 7 : Script personnalisé pour le suivi des particules. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Étapes critiques du protocole
Il est important de s’assurer que lors de la préparation de chaque matrice d’hydrogel, la matrice est fabriquée dans un environnement stérile. Sinon, une contamination peut se produire, qui se manifeste par exemple par des microcolonies (petits sphéroïdes) dans la matrice après plusieurs jours. Pendant le processus de mélange, il est important que toutes les particules d’hydrogel granulaires sèches soient dissoutes. De plus, lors de l’ajustement du pH de chaque matrice d’hydrogel avec le NaOH, les granulés commenceront à gonfler, ce qui augmentera la viscosité de la matrice d’hydrogel, ce qui rendra le mélange plus difficile. L’utilisation du batteur sur socle aidera à s’assurer que le NaOH est bien mélangé à la matrice d’hydrogel. Lors du chargement de chaque suspension bactérienne, des poches d’air peuvent se former dans l’aiguille. Pour éviter ce problème, assurez-vous que la pointe de l’aiguille se trouve toujours dans la suspension bactérienne dans le tube à centrifuger et non au bas du tube ou près de la surface supérieure. Une autre façon de surmonter ce problème est de cultiver de grands volumes de cellules et donc d’avoir de plus grands volumes de suspension bactérienne pour l’impression.
Limitations
Actuellement, lors de l’impression, la faible viscosité de la suspension bactérienne limite les géométries pouvant être imprimées et conduit souvent à la formation d’un biofilm et à sa croissance sur le dessus de la surface de la matrice d’hydrogel en raison des cellules traces. Il existe quelques méthodes potentielles pour surmonter cette limitation, notamment l’augmentation de la viscosité de la suspension bactérienne ou l’optimisation des paramètres de l’imprimante 3D. Pour augmenter la viscosité de la suspension bactérienne, on peut mélanger la suspension bactérienne avec un autre polymère - par exemple, l’alginate, qui a déjà été utilisé pour l’impression 3D de bactéries sur des surfaces planes38. Les paramètres de l’imprimante peuvent être optimisés pour permettre la rétraction du piston de la seringue lors du retrait de l’aiguille de la matrice d’hydrogel granulaire, ce qui aurait le potentiel d’empêcher les cellules de se déposer lors du retrait de l’aiguille de la matrice d’hydrogel.
L’importance de la méthode par rapport aux méthodes existantes ou alternatives
La méthode décrite ici permet d’imprimer des colonies bactériennes dans des matrices d’hydrogel granulaires. Les matrices d’hydrogel granulaires permettent d’étudier l’impact des facteurs environnementaux externes (par exemple, la taille des pores, la déformabilité de la matrice) sur la motilité et la croissance des bactéries. De plus, alors que dans ce travail, LB est utilisé comme milieu de croissance liquide pour gonfler la matrice d’hydrogel, la matrice d’hydrogel peut être gonflée avec d’autres milieux de croissance liquides, y compris des milieux contenant des antibiotiques. Les méthodes précédentes d’étude des bactéries dans des environnements confinés étaient limitées par la durée de l’expérience, la taille du maillage du polymère et la rigidité de la matrice d’hydrogel environnante37,38. Il existe déjà des protocoles pour fabriquer des matrices d’hydrogel granulaires à partir de différents polymères, de sorte que le potentiel d’étude des impacts de différentes conditions environnementales sur la motilité et la croissance des bactéries est vaste. Cette méthode permet d’étudier les bactéries dans des environnements de contrôle qui récapitulent plus facilement les environnements que les bactéries habitent dans le monde réel, tels que le mucus de l’hôte ou le sol. Une autre limitation de nombreuses autres méthodes est l’opacité de la matrice environnante ; cependant, cette approche utilisant des matériaux optiquement transparents offre la possibilité d’explorer, par exemple, le contrôle optogénétique et la structuration des bactéries en 3D.
Au-delà de l’étude de la motilité et de la croissance, la méthode d’impression 3D décrite ici surmonte les limites de nombreuses autres méthodes de bio-impression qui nécessitent le dépôt d’une bio-encre sur un substrat et sont donc limitées dans la hauteur du matériau vivant qu’elles peuvent produire. À l’avenir, ce protocole de bio-impression pourra être étendu pour fabriquer des matériaux biohybrides en mélangeant des polymères avec des cellules formant un biofilm. Les matrices d’hydrogel granulaires permettent d’imprimer en 3D des matériaux vivants plus épais et à plus grande échelle et des géométries plus complexes que de nombreuses autres méthodes actuelles de bio-impression de bactéries. Bien que ce travail n’ait utilisé que V. cholerae et E. coli, d’autres espèces, telles que Pseudomonas aeruginosa, ont également été imprimées en3D avec succès 37. Au-delà de l’impression, l’imprimante peut être adaptée pour effectuer un échantillonnage contrôlé des bactéries après leur croissance afin de voir s’il y a eu des changements génétiques, par exemple.
Déclarations de divulgation
La plateforme expérimentale utilisée pour imprimer en 3D et imager les communautés bactériennes dans cette publication fait l’objet d’une demande de brevet déposée par l’Université de Princeton au nom de Tapomoy Bhattacharjee et S.S.D. (numéro de demande PCT PCT/US/2020/030213).
Remerciements
R.K.B. remercie le soutien du programme présidentiel de bourses de recherche postdoctorale. Ce matériel est également basé sur des travaux soutenus par le programme de bourses de recherche d’études supérieures de la NSF DGE-2039656 (à A.M.H.). A.S.D.-M. et H.N.L. reconnaissent le soutien du Lidow Independent Work/Senior Thesis Fund de l’Université de Princeton. Nous remercions également le laboratoire de Bonnie Bassler pour avoir fourni des souches de V. cholerae. S.S.D. remercie le soutien des subventions de la NSF CBET-1941716, DMR-2011750 et EF-2124863, ainsi que du Eric and Wendy Schmidt Transformative Technology Fund, de la New Jersey Health Foundation, du Pew Biomedical Scholars Program et du Camille Dreyfus Teacher-Scholar Program.
matériels
| Name | Company | Catalog Number | Comments |
| 1 mL cuvettes | VWR | 97000-586 | |
| 1 mL Luer lock syringe | BH Supplies | BH1LL | |
| 10 M NaOH | Sigma-Aldrich | 72068 | |
| 100 nm carboxylated fluorescent polystyrene nanoparticles (FluoSpheres) | Invitrogen, (ThermoFischer Scientific) | F8803 | |
| 15 mL centrifuge tubes | ThermoFischer Scientific | 14-955-237 | |
| 20 G blunt needle | McMaster Carr | 75165A252 | |
| 25 mL tissue culture flasks | VWR | 10861-566 | |
| 3D printer | Lulzbot | LulzBot Mini 2 | |
| 3D printing software | Cura | Cura-Lulzbot | |
| 50 mL centrifuge tubes | ThermoFischer Scientific | 14-955-239 | |
| Agar | Sigma-Aldrich | A1296 | |
| Carbomer Granular Hydrogel Particles | Lubrizol | Carbopol 980NF | dry granules of crosslinked acrylic acid/alkyl acrylate copolymers |
| Centrifuge (2 mL tube capacity) | VWR | 2405-37 | |
| Centrifuge (50 mL tube capacity) | ThermoFischer Scientific | 75007200 | Sorvall (brand) ST 8 (model) |
| Confocal Microscope | Nikon | A1R+ inverted laserscanning confocal microscope | |
| Glass bottom petri dish | Cellvis | D35-10-1-N | |
| Lennox LB (Lubria Broth) | Sigma-Aldrich | L3022 | |
| M8 × 1.25 mm, 150 mm long, Fully Threaded Socket Cap | McMaster Carr | 91290A478 | |
| M8 × 1.25 mm, Brass Thin Hex Nut | McMaster Carr | 93187A300 | |
| Open-source syringe pump | Custom-made | Replistruder 4 | https://www.sciencedirect.com/science/article/pii/S2468067220300791 |
| Petri dish (60 mm round) | ThermoFischer Scientific | FB0875713A | |
| Shear Rheometer | Anton Paar | MCR 501 | |
| Ultrasonic cleaner | VWR | 97043-992 |
Références
- Persat, A., et al. The mechanical world of bacteria. Cell. 161 (5), 988-997 (2015).
- Stoodley, P., Dodds, I., Beer, D. D., Scott, H. L., Boyle, J. D. Flowing biofilms as a transport mechanism for biomass through porous media under laminar and turbulent conditions in a laboratory reactor system. Biofouling. 21 (3-4), 161-168 (2005).
- Ludemann, H., Arth, I., Liesack, W. Spatial changes in the bacterial community structure along a vertical oxygen gradient in flooded paddy soil cores. Applied and Environmental Microbiology. 66 (2), 754-762 (2000).
- Sicard, J. F., Bihan, G. L., Vogeleer, P., Jacques, M., Harel, J. Interactions of intestinal bacteria with components of the intestinal mucus. Frontiers in Cellular and Infection Microbiology. 7, 387 (2017).
- Grice, E. A., Segre, J. A. The skin microbiome. Nature Reviews Microbiology. 9 (4), 244-253 (2011).
- Balzan, S., Quadros, C. D. A., Cleva, R. D., Zilberstein, B., Cecconello, I. Bacterial translocation: Overview of mechanisms and clinical impact. Journal of Gastroenterology and Hepatology. 22 (4), 464-471 (2007).
- Chaban, B., Hughes, H. V., Beeby, M. The flagellum in bacterial pathogens: For motility and a whole lot more. Seminars in Cell & Developmental Biology. 46, 91-103 (2015).
- Datta, S. S., Steinberg, A. P., Ismagilov, R. F. Polymers in the gut compress the colonic mucus hydrogel. Proceedings of the National Academy of Sciences. 113 (26), 7041-7046 (2016).
- Harman, M. W., et al. The heterogeneous motility of the Lyme disease spirochete in gelatin mimics dissemination through tissue. Proceedings of the National Academy of Sciences. 109 (8), 3059-3064 (2012).
- Ribet, D., Cossart, P. How bacterial pathogens colonize their hosts and invade deeper tissues. Microbes and Infection. 17 (3), 173-183 (2015).
- Siitonen, A., Nurminen, M. Bacterial motility is a colonization factor in experimental urinary tract infection. Infection and Immunity. 60 (9), 3918-3920 (1992).
- Lux, R., Miller, J. N., Park, N. H., Shi, W. Motility and chemotaxis in tissue penetration of oral epithelial cell layers by Treponema denticola. Infection and Immunity. 69 (10), 6276-6283 (2001).
- O’Neil, H. S., Marquis, H. Listeria monocytogenes flagella are used for motility, not as adhesins, to increase host cell invasion. Infection and Immunity. 74 (12), 6675-6681 (2006).
- Gill, C. O., Penney, N. Penetration of bacteria into meat. Applied and Environmental Microbiology. 33 (6), 1284-1286 (1977).
- Shirai, H., Datta, A. K., Oshita, S. Penetration of aerobic bacteria into meat: A mechanistic understanding. Journal of Food Engineering. 196, 193-207 (2017).
- Thornlow, D. N., Brackett, E. L., Gigas, J. M., Dessel, N. V., Forbes, N. S. Persistent enhancement of bacterial motility increases tumor penetration: Motility enhances bacterial tumor penetration. Biotechnology and Bioengineering. 112 (11), 2397-2405 (2015).
- Toley, B. J., Forbes, N. S. Motility is critical for effective distribution and accumulation of bacteria in tumor tissue. Integrative Biology. 4 (2), 165-176 (2011).
- Dechesne, A., Wang, G., Gülez, G., Or, D., Smets, B. F. Hydration-controlled bacterial motility and dispersal on surfaces. Proceedings of the National Academy of Sciences. 107 (32), 14369-14372 (2010).
- de Souza, R., Ambrosini, A., Passaglia, L. M. P. Plant growth-promoting bacteria as inoculants in agricultural soils. Genetics and Molecular Biology. 38 (4), 401-419 (2015).
- Turnbull, G. A., Morgan, J. A. W., Whipps, J. M., Saunders, J. R. The role of bacterial motility in the survival and spread of Pseudomonas fluorescens in soil and in the attachment and colonisation of wheat roots. FEMS Microbiology Ecology. 36 (1), 21-31 (2001).
- Watt, M., Kirkegaard, J. A., Passioura, J. B. Rhizosphere biology and crop productivity—a review. Soil Research. 44 (4), 299-317 (2006).
- Adadevoh, J. S. T., Ramsburg, C. A., Ford, R. M. Chemotaxis Increases the Retention of Bacteria in Porous Media with Residual NAPL Entrapment. Environmental Science & Technology. 52 (13), 7289-7295 (2018).
- Adadevoh, J. S. T., Triolo, S., Ramsburg, C. A., Ford, R. M. Chemotaxis Increases the Residence Time of Bacteria in Granular Media Containing Distributed Contaminant Sources. Environmental Science & Technology. 50 (1), 181-187 (2016).
- Ford, R. M., Harvey, R. W. Role of chemotaxis in the transport of bacteria through saturated porous media. Advances in Water Resources. 30 (6-7), 1608-1617 (2007).
- Wang, M., Ford, R. M., Harvey, R. W. Coupled effect of chemotaxis and growth on microbial distributions in organic-amended aquifer sediments: Observations from laboratory and field studies. Environmental Science & Technology. 42 (10), 3556-3562 (2008).
- Amchin, D. B., Ott, J. A., Bhattacharjee, T., Datta, S. S. Influence of confinement on the spreading of bacterial populations. PLoS Computational Biology. 18 (5), e1010063 (2022).
- Moore-Ott, J. A., Chiu, S., Amchin, D. B., Bhattacharjee, T., Datta, S. S. A biophysical threshold for biofilm formation. eLife. 11, e76380 (2022).
- Tittsler, R. P., Sandholzer, L. A. The use of semi-solid agar for the detection of bacterial motility. Journal of Bacteriology. 31 (6), 575-580 (1936).
- Bhattacharjee, T., et al. Polyelectrolyte scaling laws for microgel yielding near jamming. Soft Matter. 14 (9), 1559-1570 (2018).
- Bhattacharjee, T., Datta, S. S. Confinement and activity regulate bacterial motion in porous media. Soft Matter. 15 (48), 9920-9930 (2019).
- Bhattacharjee, T., Datta, S. S. Bacterial hopping and trapping in porous media. Nature Communications. 10 (1), 2075 (2019).
- Bhattacharjee, T., Amchin, D. B., Ott, J. A., Kratz, F., Datta, S. S. Chemotactic migration of bacteria in porous media. Biophysical Journal. 120 (16), 3483-3497 (2021).
- Bhattacharjee, T., Amchin, D. B., Alert, R., Ott, J. A., Datta, S. S. Chemotactic smoothing of collective migration. eLife. 11, e71226 (2022).
- Tashman, J. W., Shiwarski, D. J., Feinberg, A. W. A high performance open-source syringe extruder optimized for extrusion and retraction during FRESH 3D bioprinting. HardwareX. 9, e00170 (2021).
- Crocker, J. C., Grier, D. G. Methods of digital video microscopy for colloidal studies. Journal of Colloid and Interface Science. 179 (1), 298-310 (1996).
- Chatterjee, T., Chatterjee, B. K., Chakrabarti, P. Modelling of growth kinetics of Vibrio cholerae in presence of gold nanoparticles: Effect of size and morphology. Scientific Reports. 7 (1), 9671 (2017).
- Martínez-Calvo, A., et al. Morphological instability and roughening of growing 3D bacterial colonies. Proceedings of the National Academy of Sciences. 119 (43), e2208019119 (2022).
- Lehner, B. A. E., Schmieden, D. T., Meyer, A. S. A Straightforward approach for 3D bacterial printing. ACS Synthetic Biology. 6 (7), 1124-1130 (2017).
- Zhang, Q., et al. Morphogenesis and cell ordering in confined bacterial biofilms. Proceedings of the National Academy of Sciences. 118 (31), e2107107118 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon