Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Alternative Strategie zur Analyse der in vitro Zellinvasion von 3D-Kulturen
In diesem Artikel
Zusammenfassung
Die Invasion ist ein wichtiges biologisches Phänomen bei der Entstehung und dem Fortschreiten von Krebs. Dieser Prozess wird durch nicht-neoplastische Zellen und Bestandteile der Tumormikroumgebung beeinflusst. Das Ziel dieser Arbeit ist es, eine alternative Methode zur Analyse der Tumorzellinvasion in vitro mit Hilfe von dreidimensionalen (3D) Kulturen zu beschreiben.
Zusammenfassung
Die Sphäroidkultur ist ein 3D-Modell, das im Vergleich zu herkömmlichen zweidimensionalen (2D) Kulturen eine verbesserte Replikation der in vivo-Mikroumgebung ermöglicht. Die Invasion ist ein zelluläres Ergebnis, das für die Krebsbiologie von größtem Interesse ist. In diesem Protokoll haben wir eine alternative Strategie zur Bewertung der Invasion von Krebszellen in vitro entwickelt, bei der Heterosphäroide verwendet werden, die aus dem oralen Plattenepithelkarzinom (OSCC), krebsassoziierten Fibroblasten (CAF) und Monozyten bestehen. Diese Heterosphäroide zielen darauf ab, die Tumormikroumgebung (TME) nachzuahmen, einschließlich zweier relevanter nicht-neoplastischer Zelltypen neben den Krebszellen. Jeder Zelltyp wurde mit lebenswichtigen Fluoreszenzmarkern markiert, die vor der Sphäroidbildung in unterschiedlichen Wellenlängen emittierten. Nach ihrer Bildung wurden Heterosphäroide auf eine Schicht aus humaner Leiomyom-abgeleiteter extrazellulärer Matrix im oberen Kompartiment einer mikroporösen Membran ausgesät. Die Invasion wurde in der z-Achse mit Hilfe der konfokalen Mikroskopie beurteilt. Digitale Bilder wurden in den entsprechenden Fluoreszenzkanälen im Abstand von 10 μm aufgenommen, die eine Tiefe von 90 μm in der z-Achse abdecken. Die Analyse wurde mit Hilfe einer Freeware-Bildsoftware durchgeführt, indem die integrierte Fluoreszenzintensität in jedem Bild und Fluoreszenzkanal berechnet wurde. Dieser Ansatz ermöglicht eine dynamischere Analyse von Zellinvasionsmustern in einem mehrschichtigen Kontext sowie die Untersuchung der räumlichen Kolokalisation verschiedener Zelltypen während der Invasion.
Einleitung
Die Invasion ist ein Prozess, bei dem neoplastische Zellen durch die extrazelluläre Matrix in das umgebende Gewebe wandern 1,2. Mit fortschreitendem Tumor werden neoplastische Zellen immer komplexeren Mikroumgebungen ausgesetzt. Die Mikroumgebung des Tumors umfasst verschiedene Bestandteile der extrazellulären Matrix und nicht-neoplastische Zelltypen. Stromazellen, einschließlich Fibroblasten und ansässiger Makrophagen, gestalten die Mikroumgebung um, indem sie extrazelluläre Matrixkomponenten und Wachstumsfaktoren sowie Zytokine sezernieren, die wiederum die neoplastischen Zellfunktionen beeinflussen3. Stroma- und infiltrierende Leukozytenzellen interagieren auch direkt mit neoplastischen Zellen, untereinander und mit der extrazellulären Matrix in einer dreidimensionalen Mikroumgebung. Insgesamt beeinflusst die TME-Zusammensetzung die Wirksamkeit und das Muster der neoplastischen Zellinvasion4.
Die komplizierte Natur der Tumormikroumgebung erfordert die Implementierung wirksamer Methoden zum Verständnis interzellulärer Wechselwirkungen innerhalb des Tumorgewebes. Die meisten In-vitro-Assays, die zur Beurteilung der Invasion verwendet werden, wurden hauptsächlich in traditionellen 2D-Modellen und/oder Einzelzellkulturen durchgeführt 5,6,7,8, was den komplexen Prozess der Invasion stark vereinfacht und mehrere Einschränkungen aufweist, wie z. B. das Fehlen einer Gewebearchitektur, stromader Komponenten und In-vivo-Reproduzierbarkeit. Sphäroidkulturen stellen ein Modell von In-vitro-3D-Kulturen dar, das die Morphologie der Tumormassen nachahmt und Zell-Zell- und Zell-Matrix-Wechselwirkungen unter Einbeziehung physikalischer und biochemischer Merkmale ermöglicht9. Die Verwendung von 3D-Kulturmodellen hat in den letzten Jahrzehnten zugenommen, und es gibt Hinweise darauf, dass verschiedene biologische Ergebnisse, wie z. B. das Ansprechen auf die Behandlung, die Zellmorphologie und die Genexpression, im Vergleich zum 2D-Modell unterschiedlich sind 9,10.
Dieses Protokoll demonstriert eine Methode zur Beurteilung der In-vitro-Invasion in einer humanen extrazellulären Matrix unter Verwendung einer 3D-Sphäroid-Zellkultur. Darüber hinaus setzten wir Heterosphäroide ein, die aus neoplastischen Zellen, Fibroblasten und Monozyten bestehen, um die Mikroumgebung des Tumors mit zwei relevanten nicht-neoplastischen Zelltypen nachzuahmen. Die Abbildung von Zellen durch konfokale Mikroskopie entlang verschiedener Invasionsebenen hin zu einer mikroporösen Membran ermöglicht die Visualisierung dieses Prozesses in einem dynamischeren und vielschichtigeren Kontext.
Protokoll
Die Verwendung von primären menschlichen Zellen wurde von der Institutionellen Ethikkommission für die Humanforschung (CAAE 57895822.0.0000.5416) der Fakultät für Zahnmedizin in Araraquara - UNESP genehmigt. Die Einzelheiten zu den Reagenzien und der in der Studie verwendeten Ausrüstung sind in der Materialtabelle aufgeführt.
1. Vorbereitung der Zellen für die 3D-Zellkultur
HINWEIS: Alle Schritte in diesem Abschnitt müssen innerhalb einer Laminar-Flow-Haube durchgeführt werden.
- Erhalten Sie einzellige Suspensionen der Zellen, die das Heterosphäroid bilden.
HINWEIS: In diesem Experiment erzeugten wir Sphäroide (siehe Schritt 2), bestehend aus OSCC-Zelllinie (SCC9), Fibroblasten (primäre krebsassoziierte Fibroblasten aus Zungenkrebsbiopsien) und Monozyten (primäre Monozyten von gesunden Spendern). Wählen Sie den Zelltyp entsprechend den experimentellen Interessen aus. - Überprüfen Sie die Zellzahl und Lebensfähigkeit mit einem Hämozytometer und einer Trypanblau-Färbung.
HINWEIS: Das Zellverhältnis in diesem Protokoll beträgt 5:3:2 (5.000 neoplastische: 3.000 Fibroblasten: 2.000 Monozyten), insgesamt 10.000 Zellen pro Sphäroide. Optimieren Sie das Verhältnis und die Gesamtzahl der Zellen entsprechend den experimentellen Interessen. - Verdünnen Sie die Zellen separat in 1,5-ml-Röhrchen mit unsupplementiertem Medium, das für jeden Zelltyp geeignet ist (z. B. für neoplastische Zellen - DMEM/F12; für Fibroblasten - DMEM; für Monozyten - RPMI), um eine geeignete Aussaatdichte von 5.000 neoplastischen Zellen pro Well, 3.000 Fibroblasten pro Well und 2.000 Monozyten pro Well oder 500.000 Zellen pro Milliliter zu erreichen.
HINWEIS: Um eine ausreichende Anzahl von Zellen für das Experiment zu gewährleisten, verwenden Sie 20 % mehr als die erforderliche Gesamtmenge, unter Berücksichtigung möglicher Verluste während der Färbe- und Zentrifugationsschritte. - Markieren Sie die verschiedenen Zelltypen getrennt mit unterschiedlichen fluoreszierenden Vitalfärbungen, die über mehrere Generationen in lebenden Zellen verbleiben. Geben Sie optimierte Konzentrationen des Fluoreszenzfarbstoffs in das Medium, das Zellen enthält (z. B. neoplastisch: CFSE - grüner Farbstoff; Fibroblast: CMTPX - roter Farbstoff; Monozyten, Violett BMQC - blauer Farbstoff) und inkubieren Sie 20 Minuten lang bei 37 °C.
HINWEIS: Wählen Sie Fluoreszenzsonden mit unterschiedlichen Anregungs- und Emissionsspektren, um spektrale Überlappungen gemäß den Spezifikationen des zu verwendenden Konfokalmikroskops zu vermeiden. - Zentrifugieren Sie die Zellen bei 400 x g für 5 min bei Raumtemperatur, um ein Zellpellet zu bilden. Aspirieren Sie das Medium mit einer 200-μl-Pipette und resuspendieren Sie es in 1 mL 1x PBS.
- Zentrifugieren Sie 5 Minuten lang bei 400 x g , um ein Zellpellet zu bilden, und resuspendieren Sie die Gesamtzahl der Zellen in 1 ml supplementiertem Kulturmedium entsprechend dem verwendeten Zelltyp/der verwendeten Zelllinie (z. B. neoplastisch - 100 μg/ml Streptomycin, 100 IE/ml Penicillin, 10 % (vol) FBS und 400 μg/ml Hydrocortison; Fibroblasten und Monozyten - 100 μg/ml Streptomycin, 100 I.E./ml Penicillin, 10 % (vol) FBS). Überprüfen Sie die Fluoreszenzintensität am Mikroskop und zählen Sie die Zellen mit Trypanblau-Färbung.
2. 3D Zellkultur
HINWEIS: In dieser Studie wurde die Bioprinting-Magnetfeldmethode11 verwendet, um Heterospheroide zu erzeugen. Dabei werden Zellen mit biokompatiblen magnetischen Nanopartikeln magnetisiert und sanften magnetischen Kräften ausgesetzt. Optimieren Sie das Verhältnis von Nanopartikeln zu Zellen je nach Zelltyp. Alternative Methoden zur Etablierung von Sphäroidkulturen können ebenfalls verwendet werden. Führen Sie die Schritte in diesem Abschnitt in einer Laminar-Flow-Haube durch.
- Fügen Sie magnetische Nanopartikel in einem Verhältnis von 1 μl Nanopartikeln zu 20.000 Zellen hinzu und resuspendieren Sie sie gründlich, indem Sie 10-mal mischen.
- Die Zellen werden bei 400 x g Raumtemperatur 5 Minuten lang zentrifugiert und durch 10-maliges Mischen wieder suspendiert. Wiederholen Sie diesen Zyklus des Zentrifugierens/Resuspension zwei weitere Male.
HINWEIS: Magnetisierte Zellen bilden ein bräunliches Pellet. - Nach Abschluss des letzten Zentrifugationszyklus wird das Medium aspiriert und die Zellen in 34 μl pro Vertiefung (34 μl/5.000 neoplastische Zellen; 34 μl/3.000 Fibroblastenzellen; 34 μl/2.000 Monozytenzellen) von supplementiertem Kulturmedium (DMEM/F12 mit 100 μg/ml Streptomycin, 100 IE/ml Penicillin und 5 % (vol) FBS) für die neoplastischen Zellen resuspendiert.
HINWEIS: Berechnen Sie das Volumen basierend auf der Well-Größe. Für dieses Protokoll wurden 96-Well-Platten mit insgesamt 100 μl Medium pro Well verwendet. - Mischen Sie alle Zellen für die Co-Kultivierung in ein neues Röhrchen und suspendieren Sie sie dann erneut.
- Übertragen Sie 100 μl co-kultivierte Zellen in jede Vertiefung einer Platte mit extrem niedrigem Anheftungspotenzial.
- Das Magnetfeld des Sphäroidantriebs wird unter die Platte gelegt, um die Aggregation und Sphäroidbildung zu induzieren, und für 3 h in den Inkubator (5 % CO2, 37 °C) gestellt.
HINWEIS: Die Zeit der Exposition gegenüber dem Magnetfeld kann je nach Größe oder Zusammensetzung des Sphäroids variieren. - Entfernen Sie das Magnetfeld und stellen Sie die Platte für mindestens 24 Stunden in den Inkubator (5 % CO2, 37 °C), um die Produktion von extrazellulären Matrixproteinen und Zell-zu-Zelle/Zell-zu-Matrix-Wechselwirkungen zu ermöglichen.
3. Herstellung einer mit extrazellulärer Matrix überzogenen mikroporösen Membran
HINWEIS: Führen Sie die Schritte in diesem Abschnitt in einer Laminar-Flow-Haube durch.
- Geben Sie 50 μl der im Handel erhältlichen extrazellulären Matrix mit einer 200-μl-Spitze in die Mitte einer mikroporösen 24-Well-Membran.
HINWEIS: Bewahren Sie die extrazelluläre Matrix auf nassem Eis auf. Berühren Sie nicht die mikroporöse Membran, wenn Sie die Matrize darauf legen. - Entfernen Sie Blasen mit einer sterilen Nadel und stellen Sie sicher, dass die Matrix die gesamte Membranoberfläche bedeckt.
- Legen Sie die Membran für 1 h in den Inkubator (37 °C), um die Gelbildung der extrazellulären Matrix zu ermöglichen.
4. Invasions-Assay
HINWEIS: Führen Sie die Schritte in diesem Abschnitt in einer Laminar-Flow-Haube durch.
- Geben Sie 500 μl DMEM/F12-Kulturmedium, ergänzt mit 10 % fötalem Rinderserum, als Chemoattraktor in die untere Kammer der mikroporösen Membran.
- Entfernen Sie vorsichtig das Medium von den gebildeten Sphäroiden und fügen Sie vorsichtig 50 μl/Vertiefung frisches, nicht supplementiertes DMEM/F12-Medium über das Heterosphäroid hinzu.
HINWEIS: Dieser Schritt kann mit einem Halteantrieb für die Bioprinting-Magnetfeldmethode durchgeführt werden. - Schneiden Sie den Rand einer 200 μl Low-Retention-Spitze mit einer sterilen Schere ab.
HINWEIS: Die Größe der Spitze hängt von der Größe des Sphäroids ab. - Sammeln Sie das Sphäroid und 50 μl Medium mit der Spitze mit niedriger Retention.
HINWEIS: Führen Sie diesen Schritt langsam und vorsichtig aus, um eine Disaggregation des Sphäroids zu vermeiden. - Legen Sie den Sphäroid vorsichtig auf die extrazelluläre Matrix.
HINWEIS: Berühren Sie nicht die gelierte extrazelluläre Matrix. - Vorsichtig werden Tropfen für Tropfen 150 μl nicht supplementiertes DMEM/F12-Medium zugegeben, um ein Gesamtvolumen von 200 μl zu erreichen.
- Stellen Sie die Platte für 48 h in den Inkubator (5 % CO2, 37 °C).
HINWEIS: Die Inkubationszeit kann je nach Zelllinie oder Protokoll variieren.
5. Bilderfassung
- Verwenden Sie ein konfokales Mikroskop für die Bildgebung.
- Richten Sie das Mikroskop basierend auf den Anregungs- und Emissionseigenschaften der verwendeten wichtigen Fluoreszenzfärbungen ein (z. B. CFSE-Farbstoff, 488-nm-Laser und 520-nm-Detektionswellenlänge; Fibroblast: CMTPX-Farbstoff, 561-nm-Laser und 610-nm-Detektionswellenlänge; Monozyten, violetter BMQC-Farbstoff, 405-nm-Laser und 516-nm-Detektionswellenlänge).
- Legen Sie die Platte auf den Mikroskoptisch.
- Lokalisieren Sie Sphäroide/Zellen mit dem Hellfeld bei 10-facher Vergrößerung.
- Stellen Sie den Zoom auf 40-fache Vergrößerung ein.
- Stellen Sie die Laserleistung und die Detektorverstärkung für jeden Fluoreszenzkanal ein.
- Identifizieren Sie den Startpunkt des Z-Stacks als Membranfokalebene, indem Sie die Membranporen mit Hilfe des Hellfelds lokalisieren.
- Nehmen Sie drei Bilder unterhalb und fünf bis acht Bilder oberhalb der Membran entlang der z-Achse für jeden Kanal auf, einschließlich des Hellfelds. Stellen Sie sicher, dass 10 μm Abstände zwischen den Bildern eingehalten werden, um eine Gesamttiefe von 90-120 μm in der z-Achse abzudecken.
- Speichern Sie das Projekt nach dem Generieren der Bilder als .czi-Erweiterungsdatei.
6. Bildextraktion
HINWEIS: Die Verwendung von Bildsoftware wird im Folgenden erläutert.
- Öffnen Sie die .czi-Datei mit der Bildbearbeitungssoftware (siehe Materialtabelle).
- Klicken Sie oben rechts auf Verarbeitung und wählen Sie dann Single aus. Klicken Sie anschließend auf Methode und wählen Sie Bildexport.
- Wählen Sie die Datei unter Eingabe aus.
- Klicken Sie auf die Schaltfläche Parameter , und wählen Sie dann die folgenden Einstellungen aus: Dateityp (Tagged Image File Format - . TIFF), Komprimierung (LZM), Größenänderung (100 %), Anzeigekurve und Kanalfarbe anwenden, einzelne Kanalbilder, Kanalnamen verwenden, Untergruppen definieren (Kanal, alle Kanäle auswählen), Z-Position (alle Kanäle extrahieren), Region (voll), Kacheln (ausgewählte Kacheln exportieren) und Exportieren nach (wählen Sie die Zieldatei aus).
HINWEIS: Um Bilder aus zusammengeführten Kanälen zu extrahieren, wählen Sie die folgenden Einstellungen aus: Dateityp (Tagged Image File Format - . TIFF), Komprimierung (LZM), Größenänderung (100 %), Anzeigekurve und Kanalfarbe anwenden, Bild zusammengeführter Kanäle, Kanalnamen verwenden, Untermengen definieren (Kanal, wählen Sie die spezifischen Kanäle aus, die überlagert werden sollen), Z-Position (alles extrahieren), Region (vollständig), Kacheln (ausgewählte Kacheln exportieren) und Exportieren nach (wählen Sie die Zieldatei aus). - Klicken Sie auf das Symbol Anwenden in der oberen rechten Ecke.
HINWEIS: In diesem Schritt werden gleichzeitig Bilder aus den verschiedenen Kanälen exportiert.
7. Bildanalyse
HINWEIS: Die Verwendung kostenloser Software wird in diesem Schritt erläutert.
- Klicken Sie in der oberen linken Ecke auf Datei , wählen Sie Öffnen, wählen Sie das Bild aus und klicken Sie dann auf OK.
- Klicken Sie auf Analysieren, dann auf Messungen festlegen und wählen Sie Fläche, Integrierte Dichte und Anzeigebeschriftung. Klicken Sie auf OK.
- Klicken Sie auf Analysieren und dann auf Messen.
HINWEIS: Stellen Sie sicher, dass der Bereich aller Bilder identisch ist. "Integrierte Dichte" zeigt zwei Werte an: (1) Raw Integrated Density (RawIntDen): Summe aller Pixelwerte im Bild; (2) Integrierte Dichte (IntDen): Produkt aus mittlerem Grauwert und Fläche. Da das Bild eine RGB-Farbe hat und der Bereich aller Bilder gleich ist, sollten Sie RawIntDen als Fluoreszenzmesswert verwenden. Unterschiede in den Fluoreszenzwerten (Intensität und Menge) zwischen den Bildern können auf Schwankungen der Zellinvasion und der Proliferationsintensität hinweisen. Eine andere Methode, um das Invasionsmuster in der extrazellulären Matrix in Richtung der mikroporösen Membran mit Hilfe von Co-Kulturen verschiedener Zelltypen zu normalisieren, besteht darin, das Verhältnis der RawIntDen-Werte zu berechnen:
Verhältnis RawIntDen =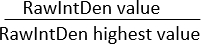
Ein Verhältnis von 1 stellt das Bild/die Position entlang der z-Achse mit der höchsten Invasion/Lokalisierung von Zellen für diesen spezifischen Zelltyp dar.
Ergebnisse
In dieser Studie wird eine alternative Methode zur Beurteilung der Zellinvasion in vitro mit Hilfe von Sphäroiden vorgestellt, die aus verschiedenen Zelltypen bestehen. Es ermöglicht die Analyse von Zellinvasionsmustern in einem vielschichtigen Kontext und die Untersuchung der Zellkolokalisationsdynamik während der Invasion (Abbildung 1).
Die Hellfeldbilder stellen die Lokalisierung von Sphäroiden und Zellen dar, die durch dunkle/schwarze Agglomerate ...
Diskussion
Dreidimensionale Sphäroidkulturen sind ein leistungsfähiger Ansatz, um verschiedene Aspekte der Zellbiologie zu untersuchen. In dieser Studie berichten wir über ein Protokoll zur Bewertung der neoplastischen Zellinvasion mit Heterospheroiden, die die Tumormikroumgebung in einem mehrschichtigen und dynamischeren Ansatz nachahmen. Diese Methode ermöglicht die Beurteilung der Kolokalisation und der Invasionsmuster in Gegenwart von nicht-neoplastischen Stromazellen.
Es gibt einige kritische Sc...
Offenlegungen
Die Autoren erklären, dass sie keine Interessenkonflikte haben.
Danksagungen
Diese Arbeit wurde von der São Paulo Research Foundation unterstützt (FAPESP 20/10544-1 und 20/10664-7). Einige Bilder wurden mit BioRender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 96 Well Bioprinting Kit | Greiner Bio-one | 655840 | Spheroid drive magnetic field. Holding drive. 96 well microplate, cell-repellent surface |
| CellTrace CFSE Cell Proliferation Kit | Invitrogen, ThermoFisher Scientific | C34554 | |
| CellTracker Red CMTPX | Invitrogen, ThermoFisher Scientific | C34552 | |
| CellTracker Violet BMQC Dye | Invitrogen, Thermo Fisher | C10094 | |
| DMEM | Gibco, ThermoFisher Scientific | 31600-034 | |
| DMEM/F12 | Gibco, ThermoFisher Scientific | 12400-024 | |
| Extracellular matrix with Myogel | doi: 10.1186/s12885-015-1944-z | Mix composed of myogel (2.4 mg/mL), collagen (0.8 mg/mL) and non-supplemented medium of neoplastic cell | |
| FBS | Gibco, ThermoFisher Scientific | 12657-029 | |
| Hydrocortisone | Sigma | H088 | |
| ImageJ | National Institutes of Health | Software | |
| Nanoshuttle-PL | Greiner Bio-one | 657841 | Magnetic nanoparticles |
| Needle 23 G | Medix | AHMD004 | 25 x 0.60 mm (23 G x 1) |
| PBS pH 7.2 (1x) | Gibco, ThermoFisher Scientific | 2012-027 | |
| Penicillin-Streptomycin (5,000 U/mL) | Gibco, ThermoFisher Scientific | 15070063 | |
| Pipet Tips | Axygen, Corning Inc. | TF-200-L-R-S | Pipet tip with barrier (filter), 200 µL Low Retention Filter, short, maximum recovery |
| RPMI | Gibco, ThermoFisher Scientific | 31800-022 | |
| TransWell 24 well | Costar, Corning Inc. | 3422 | Transwell Permeable Supports 6.5 mm Insert, 24 well plate; 0.8 µm Polycarbonate Membrane |
| Zen 3.4 (blue edition) | Zeiss Group | Software |
Referenzen
- Steeg, P. S. Tumor metastasis: Mechanistic insights and clinical challenges. Nat Med. 12 (8), 895-904 (2006).
- Friedl, P., Wolf, K. Tumour-cell invasion and migration: diversity and escape mechanisms. Nat Rev Cancer. 3, 362-374 (2003).
- Friedl, P., Alexander, S. Cancer invasion and the microenvironment: Plasticity and reciprocity. Cell. 147 (5), 992-1009 (2011).
- Wu, J. S., et al. Plasticity of cancer cell invasion: Patterns and mechanisms. Transl Oncol. 14 (1), 100899 (2021).
- Pijuan, J., et al. In vitro cell migration, invasion, and adhesion assays: From cell imaging to data analysis. Front Cell Dev Biol. 7, 1-16 (2019).
- Nagy, &. #. 1. 9. 3. ;. G., Székács, I., Bonyár, A., Horvath, R. Simple and automatic monitoring of cancer cell invasion into an epithelial monolayer using label-free holographic microscopy. Sci Rep. 12 (1), 1-13 (2022).
- Kramer, N., et al. In vitro cell migration and invasion assays. Mutat Res Rev Mutat Res. 752 (1), 10-24 (2013).
- Justus, C. R., Leffler, N., Ruiz-Echevarria, M., Yang, L. V. In vitro cell migration and invasion assays. J Vis Exp. (88), e51046 (2014).
- Kapałczyńska, M., et al. 2D and 3D cell cultures - A comparison of different. Arch Med Sci. 14 (4), 910-919 (2016).
- Cacciamali, A., Villa, R., Dotti, S. 3D cell cultures: Evolution of an ancient tool for new applications. Front Physiol. 13, 1-15 (2022).
- Trindade, J., et al. Magnetic 3D cell culture State of the art and current advances. Life Sci. 286, 1-8 (2021).
- Han, S. J., Kwon, S., Kim, K. S. Challenges of applying multicellular tumor spheroids in preclinical phase. Cancer Cell Int. 21 (1), 1-19 (2021).
- Lin, Y. N., et al. Monitoring cancer cell invasion and T-cell cytotoxicity in 3D culture. J Vis Exp. (160), e61392 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten