È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Strategia alternativa per analizzare l'invasione cellulare in vitro di colture 3D
In questo articolo
Riepilogo
L'invasione è un importante fenomeno biologico nello sviluppo e nella progressione del cancro. Questo processo è influenzato dalle cellule non neoplastiche e dai componenti del microambiente tumorale. Lo scopo di questo studio è quello di descrivere un metodo alternativo per analizzare l'invasione delle cellule tumorali in vitro utilizzando colture tridimensionali (3D).
Abstract
La coltura sferoidale è un modello 3D che fornisce una migliore replica del microambiente in vivo rispetto alle tradizionali colture bidimensionali (2D). L'invasione è un esito cellulare di estremo interesse nella biologia del cancro. In questo protocollo, abbiamo ideato una strategia alternativa per valutare l'invasione delle cellule tumorali in vitro, impiegando eterosferoidi costituiti da carcinoma orale a cellule squamose (OSCC), fibroblasti associati al cancro (CAF) e monociti. Questi eterosferoidi mirano a imitare il microambiente tumorale (TME), includendo due tipi di cellule non neoplastiche rilevanti accanto alle cellule tumorali. Ogni tipo di cellula è stato etichettato con marcatori fluorescenti vitali che emettono in lunghezze d'onda distinte prima della formazione degli sferoidi. Una volta formati, gli eterosferoidi sono stati seminati su uno strato di matrice extracellulare derivata dal leiomioma umano nel compartimento superiore di una membrana microporosa. L'invasione è stata valutata sull'asse z utilizzando la microscopia confocale. Le immagini digitali sono state ottenute nei corrispondenti canali fluorescenti a intervalli di 10 μm, coprendo una profondità di 90 μm nell'asse z. L'analisi è stata eseguita utilizzando un software di immagini freeware calcolando l'intensità di fluorescenza integrata in ciascuna immagine e canale di fluorescenza. Questo approccio consente un'analisi più dinamica dei pattern di invasione cellulare in un contesto multistrato, nonché l'esame della co-localizzazione spaziale di diversi tipi di cellule durante l'invasione.
Introduzione
L'invasione è un processo in cui le cellule neoplastiche migrano attraverso la matrice extracellulare nel tessuto circostante 1,2. Con il progredire del tumore, le cellule neoplastiche sono esposte a microambienti sempre più complessi. Il microambiente tumorale comprende vari componenti della matrice extracellulare e tipi di cellule non neoplastiche. Le cellule stromali, compresi i fibroblasti e i macrofagi residenti, rimodellano il microambiente secernendo componenti della matrice extracellulare e fattori di crescita e citochine, che a loro volta influenzano le funzioni delle cellule neoplastiche3. Le cellule stromali e leucocitarie infiltranti interagiscono anche direttamente con le cellule neoplastiche, tra loro e con la matrice extracellulare in un microambiente tridimensionale. Nel complesso, la composizione del TME influenza l'efficacia e il modello di invasione cellulare neoplastica4.
La natura complessa del microambiente tumorale richiede l'implementazione di metodologie efficaci per comprendere le interazioni intercellulari all'interno del tessuto tumorale. La maggior parte dei saggi in vitro utilizzati per valutare l'invasione sono stati condotti principalmente in modelli 2D tradizionali e/o colture di singoli tipi cellulari 5,6,7,8, che semplificano eccessivamente il complesso processo di invasione e presentano molteplici limitazioni, come la mancanza di architettura tissutale, componenti stromali e riproducibilità in vivo. Le colture sferoidi rappresentano un modello di colture 3D in vitro che imita la morfologia della massa tumorale, consentendo interazioni cellula-cellula e cellula-matrice che coinvolgono caratteristiche fisiche e biochimiche9. L'uso di modelli di coltura 3D è cresciuto negli ultimi decenni e ci sono prove che vari risultati biologici, come la risposta al trattamento, la morfologia cellulare e l'espressione genica, sono diversi rispetto al modello 2D 9,10.
Questo protocollo dimostra un metodo per valutare l'invasione in vitro in una matrice extracellulare umana utilizzando colture cellulari sferoidi 3D. Inoltre, abbiamo impiegato eterosferoidi costituiti da cellule neoplastiche, fibroblasti e monociti per imitare il microambiente tumorale con due tipi di cellule non neoplastiche rilevanti. L'imaging di cellule mediante microscopia confocale lungo diversi piani di invasione verso una membrana microporosa consente la visualizzazione di questo processo in un contesto più dinamico e multistrato.
Protocollo
L'uso di cellule umane primarie è stato approvato dal Comitato Etico Istituzionale per la Ricerca Umana (CAAE 57895822.0.0000.5416) della Scuola di Odontoiatria di Araraquara - UNESP. I dettagli dei reagenti e delle attrezzature utilizzate nello studio sono elencati nella tabella dei materiali.
1. Preparazione delle cellule per la coltura cellulare 3D
NOTA: Tutti i passaggi di questa sezione devono essere eseguiti all'interno di una cappa a flusso laminare.
- Ottenere sospensioni unicellulari delle cellule che andranno a formare l'eterosferoide.
NOTA: In questo esperimento, abbiamo generato sferoidi (vedi passaggio 2) costituiti da linea cellulare OSCC (SCC9), fibroblasti (fibroblasti primari associati al cancro da biopsie di cancro della lingua) e monociti (monociti primari da donatori sani). Selezionare il tipo di cellule in base agli interessi sperimentali. - Controllare il numero e la vitalità delle cellule utilizzando un emocitometro e la colorazione con blu di tripano.
NOTA: Il rapporto cellulare in questo protocollo è 5:3:2 (5.000 neoplastici: 3.000 fibroblasti: 2.000 monociti), per un totale di 10.000 cellule per sferoide. Ottimizzare il rapporto e il numero totale di celle in base agli interessi sperimentali. - Diluire separatamente le cellule in provette da 1,5 mL con terreno non integrato appropriato per ciascun tipo di cellula (ad esempio, per le cellule neoplastiche - DMEM/F12; per i fibroblasti - DMEM; per i monociti - RPMI) per ottenere una densità di semina appropriata di 5.000 cellule neoplastiche per pozzetto, 3.000 fibroblasti per pozzetto e 2.000 monociti per pozzetto, o 500.000 cellule per millilitro.
NOTA: Per garantire un numero adeguato di cellule per l'esperimento, utilizzare il 20% in più rispetto al totale richiesto, considerando le potenziali perdite durante le fasi di colorazione e centrifugazione. - Etichettare i diversi tipi di cellule separatamente utilizzando coloranti vitali fluorescenti distinti che rimangono nelle cellule viventi per diverse generazioni. Aggiungere concentrazioni ottimizzate di colorante fluorescente al terreno contenente cellule (ad es. neoplastico: CFSE - colorante verde; fibroblasti: CMTPX - colorante rosso; monocita, Violet BMQC - colorante blu) e incubare a 37 °C per 20 minuti.
NOTA: Selezionare sonde fluorescenti con diversi spettri di eccitazione ed emissione per evitare sovrapposizioni spettrali in base alle specifiche del microscopio confocale che verrà utilizzato. - Centrifugare le celle a 400 x g per 5 minuti a temperatura ambiente per formare un pellet di cellule. Aspirare il terreno utilizzando una pipetta da 200 μL e risospendere in 1 mL di 1x PBS.
- Centrifugare a 400 x g per 5 minuti per formare un pellet cellulare e risospendere il numero totale di cellule in 1 mL di terreno di coltura integrato in base al tipo di cellula/linea cellulare utilizzata (ad esempio, neoplastico - 100 μg/mL di streptomicina, 100 UI/mL di penicillina, 10% (vol) di FBS e 400 μg/mL di idrocortisone; fibroblasti e monociti - 100 μg/mL di streptomicina, 100 UI/mL di penicillina, 10% (vol) FBS). Controllare l'intensità della fluorescenza al microscopio e contare le cellule con colorazione con blu di tripano.
2. 3D coltura cellulare
NOTA: Questo studio ha impiegato il metodo del campo magnetico bioprinting11 per creare eterosferoidi. Qui, le cellule vengono magnetizzate utilizzando nanoparticelle magnetiche biocompatibili e sottoposte a delicate forze magnetiche. Ottimizza il rapporto tra nanoparticelle e cellule in base al tipo di cellula. Possono essere utilizzati anche metodi alternativi per stabilire colture sferoidi. Eseguire i passaggi in questa sezione in una cappa a flusso laminare.
- Aggiungere nanoparticelle magnetiche in un rapporto di 1 μL di nanoparticelle per 20.000 celle e risospendere accuratamente mescolando 10 volte.
- Centrifugare le celle a 400 x g, a temperatura ambiente, per 5 minuti e risospendere mescolando 10 volte. Ripetere questo ciclo di centrifugazione/risospensione altre due volte.
NOTA: Le celle magnetizzate formeranno una pallina di colore brunastro. - Dopo aver completato l'ultimo ciclo di centrifugazione, aspirare il terreno e risospendere le cellule in 34 μL per pozzetto (34 μL/5.000 cellule neoplastiche; 34 μL/3.000 cellule fibroblastiche; 34 μL/2.000 cellule monocitarie) di terreno di coltura integrato (DMEM/F12 contenente 100 μg/mL di streptomicina, 100 UI/mL di penicillina e 5% (vol) di FBS) per le cellule neoplastiche.
NOTA: Calcolare il volume in base alle dimensioni del pozzetto. Questo protocollo utilizzava piastre da 96 pozzetti, per un totale di 100 μL di terreno per pozzetto. - Mescolare tutte le cellule in una nuova provetta per la co-coltura, quindi ri-sospendere.
- Trasferire 100 μl di cellule co-coltivate in ciascun pozzetto di una piastra di attacco ultra-bassa.
- Inserire il campo magnetico dell'azionamento sferoidale sotto la piastra per indurre l'aggregazione e la formazione di sferoidi e posizionarlo nell'incubatore (5% CO2, 37 °C) per 3 h.
NOTA: Il tempo di esposizione al campo magnetico può variare in base alle dimensioni o alla composizione dello sferoide. - Rimuovere il campo magnetico e porre la piastra nell'incubatore (5% CO2, 37 °C) per almeno 24 ore per consentire la produzione di proteine della matrice extracellulare e le interazioni cellula-cellula/cellula-matrice.
3. Preparazione della membrana microporosa rivestita di matrice extracellulare
NOTA: Eseguire i passaggi descritti in questa sezione in una cappa a flusso laminare.
- Aggiungere 50 μl della matrice extracellulare disponibile in commercio al centro di una membrana microporosa a 24 pozzetti utilizzando un puntale da 200 μl.
NOTA: Mantenere la matrice extracellulare su ghiaccio bagnato. Non toccare la membrana microporosa quando si posiziona la matrice sopra. - Rimuovere le bolle utilizzando un ago sterile e assicurarsi che la matrice copra l'intera superficie della membrana.
- Porre la membrana nell'incubatore (37 °C) per 1 ora per consentire la formazione del gel della matrice extracellulare.
4. Saggio di invasione
NOTA: Eseguire i passaggi descritti in questa sezione in una cappa a flusso laminare.
- Aggiungere 500 μl di terreno di coltura DMEM/F12 integrato con il 10% di siero fetale bovino nella camera inferiore della membrana microporosa come chemioattrattivo.
- Rimuovere con cautela il terreno dagli sferoidi formati e aggiungere delicatamente 50 μl/pozzetto di terreno DMEM/F12 fresco non integrato sull'eterosferoide.
NOTA: Questo passaggio può essere eseguito utilizzando un azionamento di tenuta per il metodo del campo magnetico di bioprinting. - Tagliare il bordo di un puntale a bassa ritenzione da 200 μl con una forbice sterile.
NOTA: La dimensione della punta dipenderà dalla dimensione dello sferoide. - Raccogliere lo sferoide e 50 μl di terreno con la punta a bassa ritenzione.
NOTA: Eseguire questo passaggio lentamente e con attenzione per evitare la disgregazione dello sferoide. - Posizionare delicatamente lo sferoide sopra la matrice extracellulare.
NOTA: Non toccare la matrice extracellulare gelificata. - Aggiungere con cautela 150 μl di terreno DMEM/F12 non integrato goccia per goccia per raggiungere un volume totale di 200 μl.
- Posizionare la piastra nell'incubatore (5% CO2, 37 °C) per 48 ore.
NOTA: Il tempo di incubazione può variare a seconda della linea cellulare o del protocollo.
5. Acquisizione delle immagini
- Utilizzare un microscopio confocale per l'imaging.
- Impostare il microscopio in base alle caratteristiche di eccitazione ed emissione dei coloranti fluorescenti vitali utilizzati (ad esempio, colorante CFSE, laser a 488 nm e lunghezza d'onda di rilevamento a 520 nm; fibroblasti: colorante CMTPX, laser a 561 nm e lunghezza d'onda di rilevamento a 610 nm; monocita, colorante BMQC viola, laser a 405 nm e lunghezza d'onda di rilevamento a 516 nm).
- Caricare la piastra sul tavolino del microscopio.
- Localizza sferoidi/celle utilizzando il campo luminoso con ingrandimento 10x.
- Regolare lo zoom su un ingrandimento di 40x.
- Impostare la potenza del laser e il guadagno del rivelatore per ciascun canale fluorescente.
- Identifica il punto di partenza Z-Stack come il piano focale della membrana individuando i pori della membrana utilizzando il campo chiaro.
- Cattura tre immagini sotto e da cinque a otto immagini sopra la membrana lungo l'asse z per ogni canale, incluso il campo luminoso. Garantire intervalli di 10 μm tra le immagini, coprendo una profondità totale di 90-120 μm nell'asse z.
- Salva il progetto come file con estensione .czi dopo aver generato le immagini.
6. Estrazione delle immagini
NOTA: L'utilizzo del software di immagine è spiegato di seguito.
- Aprire il file .czi utilizzando il software di elaborazione delle immagini (vedere Tabella dei materiali).
- Fare clic su Elaborazione nell'angolo in alto a destra, quindi selezionare Singolo. Quindi, fai clic su Metodo e scegli Esportazione immagini.
- Selezionare il file in Input.
- Fare clic sul pulsante Parametri , quindi selezionare le seguenti impostazioni: Tipo di file (Formato file immagine con tag - . TIFF), Compressione (LZM), Ridimensiona (100%), Applica curva di visualizzazione e colore del canale, Immagini dei singoli canali, Usa nomi dei canali, Definisci sottoinsiemi (Canale, seleziona tutti i canali), Posizione Z (estrai tutto), Regione (Completa), Piastrelle (Esporta le tessere selezionate) ed Esporta in (seleziona il file di destinazione).
NOTA: Per estrarre le immagini dei canali uniti, selezionare le seguenti impostazioni: Tipo di file (Formato file immagine con tag - . TIFF), Compressione (LZM), Ridimensiona (100%), Applica curva di visualizzazione e colore del canale, Immagine canali uniti, Usa nomi canali, Definisci sottoinsiemi (Canale, seleziona i canali specifici da sovrapporre), Posizione Z (estrai tutto), Regione (Completa), Piastrelle (Esporta le tessere selezionate) ed Esporta in (seleziona il file di destinazione). - Fare clic sull'icona Applica situata nell'angolo in alto a destra.
NOTA: Questo passaggio esporterà contemporaneamente le immagini dai diversi canali.
7. Analisi delle immagini
NOTA: L'uso del software gratuito è spiegato in questo passaggio.
- Fare clic su File nell'angolo in alto a sinistra, selezionare Apri, scegliere l'immagine, quindi fare clic su OK.
- Fare clic su Analizza, quindi su Imposta misurazioni e selezionare Area, Densità integrata ed Etichetta di visualizzazione. Fare clic su OK.
- Fare clic su Analizza, quindi su Misura.
NOTA: Assicurarsi che l'area di tutte le immagini sia la stessa. "Densità integrata" visualizza due valori: (1) Densità integrata raw (RawIntDen): somma di tutti i valori dei pixel nell'immagine; (2) Densità integrata (IntDen): prodotto del valore medio di grigio e dell'area. Poiché l'immagine è di colore RGB e l'area di tutte le immagini è la stessa, è consigliabile utilizzare RawIntDen come valore di misurazione della fluorescenza. Le differenze nei valori di fluorescenza (intensità e quantità) tra le immagini possono indicare variazioni nell'invasione cellulare e nell'intensità della proliferazione. Un altro metodo per normalizzare il pattern di invasione nella matrice extracellulare verso la membrana microporosa utilizzando co-colture di diversi tipi di cellule consiste nel calcolare il rapporto tra i valori di RawIntDen:
Rapporto RawIntDen =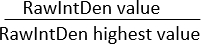
Un rapporto di 1 rappresenta l'immagine/posizione lungo l'asse z con la più alta invasione/localizzazione di cellule per quel tipo di cellula specifico.
Risultati
Questo studio presenta un metodo alternativo per valutare l'invasione cellulare in vitro utilizzando sferoidi composti da diversi tipi di cellule. Consente l'analisi dei pattern di invasione cellulare in un contesto multistrato e l'esame delle dinamiche di co-localizzazione cellulare durante l'invasione (Figura 1).
Le immagini in campo chiaro rappresentano la localizzazione di sferoidi/cellule, identificate da agglomerati scuri/neri (...
Discussione
Le colture di sferoidi tridimensionali sono un approccio potente per studiare vari aspetti della biologia cellulare. In questo studio, riportiamo un protocollo per la valutazione dell'invasione cellulare neoplastica utilizzando eterosferoidi che mimano il microambiente tumorale in un approccio multistrato e più dinamico. Questa metodica consente di valutare la co-localizzazione e i pattern di invasione in presenza di cellule stromali non neoplastiche.
Ci sono alcuni passaggi critici che merit...
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Questo lavoro è stato sostenuto dalla Fondazione per la ricerca di San Paolo (FAPESP 20/10544-1 e 20/10664-7). Alcune immagini sono state generate utilizzando BioRender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| 96 Well Bioprinting Kit | Greiner Bio-one | 655840 | Spheroid drive magnetic field. Holding drive. 96 well microplate, cell-repellent surface |
| CellTrace CFSE Cell Proliferation Kit | Invitrogen, ThermoFisher Scientific | C34554 | |
| CellTracker Red CMTPX | Invitrogen, ThermoFisher Scientific | C34552 | |
| CellTracker Violet BMQC Dye | Invitrogen, Thermo Fisher | C10094 | |
| DMEM | Gibco, ThermoFisher Scientific | 31600-034 | |
| DMEM/F12 | Gibco, ThermoFisher Scientific | 12400-024 | |
| Extracellular matrix with Myogel | doi: 10.1186/s12885-015-1944-z | Mix composed of myogel (2.4 mg/mL), collagen (0.8 mg/mL) and non-supplemented medium of neoplastic cell | |
| FBS | Gibco, ThermoFisher Scientific | 12657-029 | |
| Hydrocortisone | Sigma | H088 | |
| ImageJ | National Institutes of Health | Software | |
| Nanoshuttle-PL | Greiner Bio-one | 657841 | Magnetic nanoparticles |
| Needle 23 G | Medix | AHMD004 | 25 x 0.60 mm (23 G x 1) |
| PBS pH 7.2 (1x) | Gibco, ThermoFisher Scientific | 2012-027 | |
| Penicillin-Streptomycin (5,000 U/mL) | Gibco, ThermoFisher Scientific | 15070063 | |
| Pipet Tips | Axygen, Corning Inc. | TF-200-L-R-S | Pipet tip with barrier (filter), 200 µL Low Retention Filter, short, maximum recovery |
| RPMI | Gibco, ThermoFisher Scientific | 31800-022 | |
| TransWell 24 well | Costar, Corning Inc. | 3422 | Transwell Permeable Supports 6.5 mm Insert, 24 well plate; 0.8 µm Polycarbonate Membrane |
| Zen 3.4 (blue edition) | Zeiss Group | Software |
Riferimenti
- Steeg, P. S. Tumor metastasis: Mechanistic insights and clinical challenges. Nat Med. 12 (8), 895-904 (2006).
- Friedl, P., Wolf, K. Tumour-cell invasion and migration: diversity and escape mechanisms. Nat Rev Cancer. 3, 362-374 (2003).
- Friedl, P., Alexander, S. Cancer invasion and the microenvironment: Plasticity and reciprocity. Cell. 147 (5), 992-1009 (2011).
- Wu, J. S., et al. Plasticity of cancer cell invasion: Patterns and mechanisms. Transl Oncol. 14 (1), 100899 (2021).
- Pijuan, J., et al. In vitro cell migration, invasion, and adhesion assays: From cell imaging to data analysis. Front Cell Dev Biol. 7, 1-16 (2019).
- Nagy, &. #. 1. 9. 3. ;. G., Székács, I., Bonyár, A., Horvath, R. Simple and automatic monitoring of cancer cell invasion into an epithelial monolayer using label-free holographic microscopy. Sci Rep. 12 (1), 1-13 (2022).
- Kramer, N., et al. In vitro cell migration and invasion assays. Mutat Res Rev Mutat Res. 752 (1), 10-24 (2013).
- Justus, C. R., Leffler, N., Ruiz-Echevarria, M., Yang, L. V. In vitro cell migration and invasion assays. J Vis Exp. (88), e51046 (2014).
- Kapałczyńska, M., et al. 2D and 3D cell cultures - A comparison of different. Arch Med Sci. 14 (4), 910-919 (2016).
- Cacciamali, A., Villa, R., Dotti, S. 3D cell cultures: Evolution of an ancient tool for new applications. Front Physiol. 13, 1-15 (2022).
- Trindade, J., et al. Magnetic 3D cell culture State of the art and current advances. Life Sci. 286, 1-8 (2021).
- Han, S. J., Kwon, S., Kim, K. S. Challenges of applying multicellular tumor spheroids in preclinical phase. Cancer Cell Int. 21 (1), 1-19 (2021).
- Lin, Y. N., et al. Monitoring cancer cell invasion and T-cell cytotoxicity in 3D culture. J Vis Exp. (160), e61392 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon