Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Stratégie alternative pour analyser l’invasion cellulaire in vitro de cultures 3D
Dans cet article
Résumé
L’invasion est un phénomène biologique majeur dans le développement et la progression du cancer. Ce processus est influencé par les cellules non néoplasiques et les composants du microenvironnement tumoral. Le but de cette étude est de décrire une méthode alternative pour analyser l’invasion des cellules tumorales in vitro à l’aide de cultures tridimensionnelles (3D).
Résumé
La culture de sphéroïdes est un modèle 3D qui offre une meilleure réplication du microenvironnement in vivo par rapport aux cultures bidimensionnelles (2D) traditionnelles. L’invasion est un résultat cellulaire du plus grand intérêt pour la biologie du cancer. Dans ce protocole, nous avons conçu une stratégie alternative pour évaluer l’invasion des cellules cancéreuses in vitro, en utilisant des hétérosphéroïdes composés d’un carcinome épidermoïde oral (OSCC), de fibroblastes associés au cancer (CAF) et de monocytes. Ces hétérosphéroïdes visent à imiter le microenvironnement tumoral (TME), y compris deux types de cellules non néoplasiques pertinents aux côtés des cellules cancéreuses. Chaque type de cellule a été marqué avec des marqueurs fluorescents vitaux émettant dans des longueurs d’onde distinctes avant la formation des sphéroïdes. Une fois formés, les hétérosphéroïdes ont été ensemencés sur une couche de matrice extracellulaire dérivée d’un léiomyome humain dans le compartiment supérieur d’une membrane microporeuse. L’invasion a été évaluée dans l’axe z à l’aide de la microscopie confocale. Des images numériques ont été obtenues dans les canaux fluorescents correspondants à des intervalles de 10 μm, couvrant une profondeur de 90 μm dans l’axe z. L’analyse a été effectuée à l’aide d’un logiciel d’imagerie gratuit en calculant l’intensité de fluorescence intégrée dans chaque image et canal de fluorescence. Cette approche permet une analyse plus dynamique des modèles d’invasion cellulaire dans un contexte multicouche, ainsi que l’examen de la co-localisation spatiale de différents types de cellules pendant l’invasion.
Introduction
L’invasion est un processus dans lequel les cellules néoplasiques migrent à travers la matrice extracellulaire dans les tissus environnants 1,2. Au fur et à mesure que la tumeur progresse, les cellules néoplasiques sont exposées à des microenvironnements de plus en plus complexes. Le microenvironnement tumoral comprend divers composants de la matrice extracellulaire et des types de cellules non néoplasiques. Les cellules stromales, y compris les fibroblastes et les macrophages résidents, remodèlent le microenvironnement en sécrétant des composants de la matrice extracellulaire et des facteurs de croissance, ainsi que des cytokines, qui à leur tour influencent les fonctions cellulaires néoplasiques3. Les cellules stromales et leucocytaires infiltrantes interagissent également directement avec les cellules néoplasiques, entre elles et avec la matrice extracellulaire dans un microenvironnement tridimensionnel. Dans l’ensemble, la composition des EUT influence l’efficacité et le modèle d’invasion cellulaire néoplasique4.
La nature complexe du microenvironnement tumoral nécessite la mise en œuvre de méthodologies efficaces pour comprendre les interactions intercellulaires au sein du tissu tumoral. La plupart des essais in vitro utilisés pour évaluer l’invasion ont été principalement réalisés dans des modèles 2D traditionnels et/ou des cultures de type unicellulaire 5,6,7,8, ce qui simplifie à l’excès le processus complexe d’invasion et présente de multiples limites, telles que l’absence d’architecture tissulaire, de composants stromaux et de reproductibilité in vivo. Les cultures de sphéroïdes représentent un modèle de cultures 3D in vitro qui imite la morphologie de la masse tumorale, permettant des interactions cellule-cellule et cellule-matrice impliquant des caractéristiques physiques et biochimiques9. L’utilisation de modèles de culture 3D s’est développée au cours des dernières décennies, et il existe des preuves que divers résultats biologiques, tels que la réponse au traitement, la morphologie cellulaire et l’expression des gènes, sont différents par rapport au modèle 2D 9,10.
Ce protocole démontre une méthode d’évaluation de l’invasion in vitro dans une matrice extracellulaire humaine à l’aide de la culture de cellules sphéroïdes 3D. De plus, nous avons utilisé des hétérosphéroïdes composés de cellules néoplasiques, de fibroblastes et de monocytes pour imiter le microenvironnement tumoral avec deux types de cellules non néoplasiques pertinents. L’imagerie des cellules par microscopie confocale le long de différents plans d’invasion vers une membrane microporeuse permet de visualiser ce processus dans un contexte plus dynamique et multicouche.
Protocole
L’utilisation de cellules humaines primaires a été approuvée par le Comité d’éthique de la recherche humaine institutionnelle (CAAE 57895822.0.0000.5416) de l’École de médecine dentaire d’Araraquara - UNESP. Les détails des réactifs et de l’équipement utilisés dans l’étude sont énumérés dans la table des matériaux.
1. Préparation des cellules pour la culture cellulaire 3D
REMARQUE : Toutes les étapes de cette section doivent être effectuées à l’intérieur d’une hotte à flux laminaire.
- Obtenir des suspensions unicellulaires des cellules qui formeront l’hétérosphéroïde.
REMARQUE : Dans cette expérience, nous avons généré des sphéroïdes (voir étape 2) constitués de lignées cellulaires OSCC (SCC9), de fibroblastes (fibroblastes primaires associés au cancer provenant de biopsies de cancer de la langue) et de monocytes (monocytes primaires provenant de donneurs sains). Sélectionnez le type de cellules en fonction des intérêts expérimentaux. - Vérifiez le nombre de cellules et la viabilité à l’aide d’un hémocytomètre et d’une coloration au bleu de trypan.
REMARQUE : Le rapport cellulaire dans ce protocole est de 5:3:2 (5 000 néoplasiques : 3 000 fibroblastes : 2 000 monocytes), soit un total de 10 000 cellules par sphéroïde. Optimiser le ratio et le nombre total de cellules en fonction des intérêts expérimentaux. - Diluer séparément les cellules dans des tubes de 1,5 mL avec un milieu non supplémenté approprié pour chaque type de cellule (p. ex., pour les cellules néoplasiques - DMEM/F12 ; pour les fibroblastes - DMEM ; pour les monocytes - RPMI) afin d’obtenir une densité d’ensemencement appropriée de 5 000 cellules néoplasiques par puits, 3 000 fibroblastes par puits et 2 000 monocytes par puits, ou 500 000 cellules par millilitre.
REMARQUE : Pour garantir un nombre adéquat de cellules pour l’expérience, utilisez 20% de plus que le total requis, en tenant compte des pertes potentielles pendant les étapes de coloration et de centrifugation. - Étiquetez les différents types de cellules séparément à l’aide de colorants vitaux fluorescents distincts qui restent dans les cellules vivantes pendant plusieurs générations. Ajouter des concentrations optimisées de colorant fluorescent au milieu contenant des cellules (p. ex., néoplasique : CFSE - colorant vert ; fibroblaste : CMTPX - colorant rouge ; monocyte, violet BMQC - colorant bleu) et incuber à 37 °C pendant 20 min.
REMARQUE : Choisissez des sondes fluorescentes avec des spectres d’excitation et d’émission différents pour éviter le chevauchement spectral selon les spécifications du microscope confocal qui sera utilisé. - Centrifugez les cellules à 400 x g pendant 5 min à température ambiante pour former une pastille cellulaire. Aspirer le milieu à l’aide d’une pipette de 200 μL et remettre en suspension dans 1 mL de 1x PBS.
- Centrifuger à 400 x g pendant 5 min pour former une pastille cellulaire et remettre en suspension le nombre total de cellules dans 1 mL de milieu de culture supplémenté selon le type de cellule/lignée cellulaire utilisé (p. ex., néoplasique - 100 μg/mL de streptomycine, 100 UI/mL de pénicilline, 10 % (vol) FBS et 400 μg/mL d’hydrocortisone ; fibroblastes et monocytes - 100 μg/mL de streptomycine, 100 UI/mL de pénicilline, 10 % (vol) FBS). Vérifiez l’intensité de la fluorescence au microscope et comptez les cellules avec une coloration au bleu de trypan.
2. 3D culture cellulaire
REMARQUE : Cette étude a utilisé la méthode de champ magnétique de bio-impression11 pour créer des hétérosphéroïdes. Ici, les cellules sont magnétisées à l’aide de nanoparticules magnétiques biocompatibles et soumises à des forces magnétiques douces. Optimiser le rapport nanoparticules/cellules en fonction du type de cellule. D’autres méthodes d’établissement de cultures de sphéroïdes peuvent également être utilisées. Effectuez les étapes de cette section dans une hotte à flux laminaire.
- Ajoutez des nanoparticules magnétiques à raison de 1 μL de nanoparticules pour 20 000 cellules, et remettez complètement en suspension en mélangeant 10 fois.
- Centrifugez les cellules à 400 x g, à température ambiante, pendant 5 min, et remettez-les en suspension en mélangeant 10 fois. Répétez ce cycle de centrifugation/remise en suspension deux fois de plus.
REMARQUE : Les cellules magnétisées formeront une pastille de couleur brunâtre. - Une fois le dernier cycle de centrifugation terminé, aspirer le milieu et remettre les cellules en suspension dans 34 μL par puits (34 μL/5 000 cellules néoplasiques ; 34 μL/3 000 cellules fibroblastiques ; 34 μL/2 000 cellules monocytaires) de milieu de culture supplémenté (DMEM/F12 contenant 100 μg/mL de streptomycine, 100 UI/mL de pénicilline et 5 % (vol) de FBS) pour les cellules néoplasiques.
REMARQUE : Calculez le volume en fonction de la taille du puits. Ce protocole utilisait des plaques de 96 puits, totalisant 100 μL de milieu par puits. - Mélangez toutes les cellules dans un nouveau tube pour la co-culture, puis remettez-les en suspension.
- Transférez 100 μL de cellules co-cultivées dans chaque puits d’une plaque de fixation à très faible épaisseur.
- Insérez le champ magnétique d’entraînement des sphéroïdes sous la plaque pour induire l’agrégation et la formation de sphéroïdes, et placez-le dans l’incubateur (5% CO2, 37 °C) pendant 3 h.
REMARQUE : Le temps d’exposition au champ magnétique peut varier en fonction de la taille ou de la composition du sphéroïde. - Retirez le champ magnétique et placez la plaque dans l’incubateur (5% CO2, 37 °C) pendant au moins 24 h pour permettre la production de protéines de la matrice extracellulaire et les interactions de cellule à cellule/cellule à matrice.
3. Préparation d’une membrane microporeuse recouverte d’une matrice extracellulaire
REMARQUE : Effectuez les étapes de cette section dans une hotte à flux laminaire.
- Ajoutez 50 μL de la matrice extracellulaire disponible dans le commerce au centre d’une membrane microporeuse de 24 puits à l’aide d’une pointe de 200 μL.
REMARQUE : Conservez la matrice extracellulaire sur de la glace humide. Ne touchez pas la membrane microporeuse lorsque vous placez la matrice sur le dessus. - Éliminez les bulles à l’aide d’une aiguille stérile et assurez-vous que la matrice recouvre toute la surface de la membrane.
- Placez la membrane dans l’incubateur (37 °C) pendant 1 h pour permettre la formation de gel de la matrice extracellulaire.
4. Essai d’invasion
REMARQUE : Effectuez les étapes de cette section dans une hotte à flux laminaire.
- Ajouter 500 μL de milieu de culture DMEM/F12 complété par 10 % de sérum fœtal bovin dans la chambre inférieure de la membrane microporeuse comme agent chimioattractant.
- Retirez délicatement le milieu des sphéroïdes formés et ajoutez délicatement 50 μL/puits de milieu DMEM/F12 frais non supplémenté sur l’hétérosphéroïde.
REMARQUE : Cette étape peut être effectuée à l’aide d’un entraînement de maintien pour la méthode de champ magnétique de bio-impression. - Coupez le bord d’une pointe à faible rétention de 200 μL avec des ciseaux stériles.
REMARQUE : La taille de la pointe dépendra de la taille du sphéroïde. - Prélever le sphéroïde et 50 μL de milieu avec l’embout à faible rétention.
REMARQUE : Effectuez cette étape lentement et soigneusement pour éviter la désagrégation du sphéroïde. - Placez doucement le sphéroïde sur la matrice extracellulaire.
REMARQUE : Ne touchez pas la matrice extracellulaire gélifiée. - Ajouter délicatement 150 μL de DMEM/F12 moyen non supplémenté goutte à goutte pour atteindre un volume total de 200 μL.
- Placez la plaque dans l’incubateur (5% CO2, 37 °C) pendant 48 h.
REMARQUE : Le temps d’incubation peut varier en fonction de la lignée cellulaire ou du protocole.
5. Acquisition d’images
- Utilisez un microscope confocal pour l’imagerie.
- Configurez le microscope en fonction des caractéristiques d’excitation et d’émission des colorants fluorescents vitaux utilisés (par exemple, colorant CFSE, laser 488 nm et longueur d’onde de détection 520 nm ; fibroblaste : colorant CMTPX, laser 561 nm et longueur d’onde de détection 610 nm ; monocyte, colorant BMQC violet, laser 405 nm et longueur d’onde de détection 516 nm).
- Chargez la plaque sur la platine du microscope.
- Localisez les sphéroïdes/cellules à l’aide du champ clair à un grossissement de 10x.
- Réglez le zoom sur un grossissement de 40x.
- Réglez la puissance du laser et le gain du détecteur pour chaque canal fluorescent.
- Identifiez le point de départ de la pile Z comme le plan focal de la membrane en localisant les pores de la membrane à l’aide du champ clair.
- Capturez trois images en dessous et cinq à huit images au-dessus de la membrane le long de l’axe z pour chaque canal, y compris le champ clair. Assurez-vous que les images sont intercalées à intervalles de 10 μm, couvrant une profondeur totale de 90 à 120 μm dans l’axe z.
- Enregistrez le projet en tant que fichier d’extension .czi après avoir généré les images.
6. Extraction d’images
REMARQUE : L’utilisation du logiciel d’image est expliquée ci-dessous.
- Ouvrez le fichier .czi à l’aide du logiciel de retouche d’image (voir Table des matériaux).
- Cliquez sur Traitement dans le coin supérieur droit, puis sélectionnez Unique. Ensuite, cliquez sur Méthode et choisissez Exportation d’image.
- Sélectionnez le fichier sous Entrée.
- Cliquez sur le bouton Paramètres , puis sélectionnez les paramètres suivants : Type de fichier (Format de fichier image balisé - . TIFF), Compression (LZM), Redimensionnement (100%), Application de la courbe d’affichage et de la couleur du canal, Images de canal individuelles, Utilisation des noms de canal, Définition de sous-ensembles (Canal, sélection de tous les canaux), Position Z (extraction de tout), Région (Complète), Tuiles (Exporter les tuiles sélectionnées) et Exporter vers (sélection du fichier de destination).
REMARQUE : Pour extraire les images de canal fusionnées, sélectionnez les paramètres suivants : Type de fichier (format de fichier image balisé - . TIFF), Compression (LZM), Redimensionner (100%), Appliquer la courbe d’affichage et la couleur du canal, Image des canaux fusionnés, Utiliser les noms de canaux, Définir des sous-ensembles (Canal, sélectionner les canaux spécifiques à superposer), Position Z (extraire tout), Région (Complète), Tuiles (Exporter les tuiles sélectionnées) et Exporter vers (sélectionner le fichier de destination). - Cliquez sur l’icône Appliquer située dans le coin supérieur droit.
REMARQUE : Cette étape permet d’exporter simultanément les images des différents canaux.
7. Analyse d’images
REMARQUE : L’utilisation du logiciel libre est expliquée dans cette étape.
- Cliquez sur Fichier dans le coin supérieur gauche, sélectionnez Ouvrir, choisissez l’image, puis cliquez sur OK.
- Cliquez sur Analyser, puis sur Définir les mesures, puis sélectionnez Surface, Densité intégrée et Étiquette d’affichage. Cliquez sur OK.
- Cliquez sur Analyser, puis sur Mesurer.
REMARQUE : Assurez-vous que la zone de toutes les images est la même. « Densité intégrée » affiche deux valeurs : (1) Densité intégrée brute (RawIntDen) : Somme de toutes les valeurs de pixels de l’image ; (2) Densité intégrée (IntDen) : produit de la valeur de gris moyenne et de la surface. Étant donné que l’image est de couleur RVB et que la zone de toutes les images est la même, envisagez d’utiliser RawIntDen comme valeur de mesure de fluorescence. Les différences de valeurs de fluorescence (intensité et quantité) entre les images peuvent indiquer des variations dans l’invasion cellulaire et l’intensité de la prolifération. Une autre méthode pour normaliser le modèle d’invasion dans la matrice extracellulaire vers la membrane microporeuse en utilisant des co-cultures de différents types de cellules consiste à calculer le rapport des valeurs RawIntDen :
Rapport RawIntDen =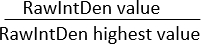
Un rapport de 1 représente l’image/l’emplacement le long de l’axe z avec l’invasion/localisation la plus élevée de cellules pour ce type de cellule spécifique.
Résultats
Cette étude présente une méthode alternative pour évaluer l’invasion cellulaire in vitro à l’aide de sphéroïdes composés de différents types de cellules. Il permet d’analyser les modèles d’invasion cellulaire dans un contexte multicouche et d’examiner la dynamique de co-localisation cellulaire pendant l’invasion (Figure 1).
Les images en fond clair représentent la localisation sphéroïde/cellulaire, identifiée par des agglomérats...
Discussion
Les cultures tridimensionnelles de sphéroïdes sont une approche puissante pour étudier divers aspects de la biologie cellulaire. Dans cette étude, nous rapportons un protocole d’évaluation de l’invasion cellulaire néoplasique à l’aide d’hétérosphéroïdes qui imitent le microenvironnement tumoral dans une approche multicouche et plus dynamique. Cette méthode permet d’évaluer la co-localisation et les schémas d’invasion en présence de cellules stromales non néoplasiques.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu par la Fondation de recherche de São Paulo (FAPESP 20/10544-1 et 20/10664-7). Certaines images ont été générées à l’aide de BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| 96 Well Bioprinting Kit | Greiner Bio-one | 655840 | Spheroid drive magnetic field. Holding drive. 96 well microplate, cell-repellent surface |
| CellTrace CFSE Cell Proliferation Kit | Invitrogen, ThermoFisher Scientific | C34554 | |
| CellTracker Red CMTPX | Invitrogen, ThermoFisher Scientific | C34552 | |
| CellTracker Violet BMQC Dye | Invitrogen, Thermo Fisher | C10094 | |
| DMEM | Gibco, ThermoFisher Scientific | 31600-034 | |
| DMEM/F12 | Gibco, ThermoFisher Scientific | 12400-024 | |
| Extracellular matrix with Myogel | doi: 10.1186/s12885-015-1944-z | Mix composed of myogel (2.4 mg/mL), collagen (0.8 mg/mL) and non-supplemented medium of neoplastic cell | |
| FBS | Gibco, ThermoFisher Scientific | 12657-029 | |
| Hydrocortisone | Sigma | H088 | |
| ImageJ | National Institutes of Health | Software | |
| Nanoshuttle-PL | Greiner Bio-one | 657841 | Magnetic nanoparticles |
| Needle 23 G | Medix | AHMD004 | 25 x 0.60 mm (23 G x 1) |
| PBS pH 7.2 (1x) | Gibco, ThermoFisher Scientific | 2012-027 | |
| Penicillin-Streptomycin (5,000 U/mL) | Gibco, ThermoFisher Scientific | 15070063 | |
| Pipet Tips | Axygen, Corning Inc. | TF-200-L-R-S | Pipet tip with barrier (filter), 200 µL Low Retention Filter, short, maximum recovery |
| RPMI | Gibco, ThermoFisher Scientific | 31800-022 | |
| TransWell 24 well | Costar, Corning Inc. | 3422 | Transwell Permeable Supports 6.5 mm Insert, 24 well plate; 0.8 µm Polycarbonate Membrane |
| Zen 3.4 (blue edition) | Zeiss Group | Software |
Références
- Steeg, P. S. Tumor metastasis: Mechanistic insights and clinical challenges. Nat Med. 12 (8), 895-904 (2006).
- Friedl, P., Wolf, K. Tumour-cell invasion and migration: diversity and escape mechanisms. Nat Rev Cancer. 3, 362-374 (2003).
- Friedl, P., Alexander, S. Cancer invasion and the microenvironment: Plasticity and reciprocity. Cell. 147 (5), 992-1009 (2011).
- Wu, J. S., et al. Plasticity of cancer cell invasion: Patterns and mechanisms. Transl Oncol. 14 (1), 100899 (2021).
- Pijuan, J., et al. In vitro cell migration, invasion, and adhesion assays: From cell imaging to data analysis. Front Cell Dev Biol. 7, 1-16 (2019).
- Nagy, &. #. 1. 9. 3. ;. G., Székács, I., Bonyár, A., Horvath, R. Simple and automatic monitoring of cancer cell invasion into an epithelial monolayer using label-free holographic microscopy. Sci Rep. 12 (1), 1-13 (2022).
- Kramer, N., et al. In vitro cell migration and invasion assays. Mutat Res Rev Mutat Res. 752 (1), 10-24 (2013).
- Justus, C. R., Leffler, N., Ruiz-Echevarria, M., Yang, L. V. In vitro cell migration and invasion assays. J Vis Exp. (88), e51046 (2014).
- Kapałczyńska, M., et al. 2D and 3D cell cultures - A comparison of different. Arch Med Sci. 14 (4), 910-919 (2016).
- Cacciamali, A., Villa, R., Dotti, S. 3D cell cultures: Evolution of an ancient tool for new applications. Front Physiol. 13, 1-15 (2022).
- Trindade, J., et al. Magnetic 3D cell culture State of the art and current advances. Life Sci. 286, 1-8 (2021).
- Han, S. J., Kwon, S., Kim, K. S. Challenges of applying multicellular tumor spheroids in preclinical phase. Cancer Cell Int. 21 (1), 1-19 (2021).
- Lin, Y. N., et al. Monitoring cancer cell invasion and T-cell cytotoxicity in 3D culture. J Vis Exp. (160), e61392 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon