Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Digitale Genschaltkreise auf Basis von CRISPR-Cas-Systemen und Anti-CRISPR-Proteinen
* Diese Autoren haben gleichermaßen beigetragen
Zusammenfassung
CRISPR-Cas-Systeme und Anti-CRISPR-Proteine wurden in das Schema der Booleschen Gates in Saccharomyces cerevisiae integriert. Die neuen kleinen Logikschaltkreise zeigten eine gute Leistung und vertieften das Verständnis sowohl von dCas9/dCas12a-basierten Transkriptionsfaktoren als auch von den Eigenschaften von Anti-CRISPR-Proteinen.
Zusammenfassung
Boolesche Gatter mit synthetischen Genen und digitale Schaltkreise haben ein breites Anwendungsspektrum, von der medizinischen Diagnostik bis zum Umweltschutz. Die Entdeckung der CRISPR-Cas-Systeme und ihrer natürlichen Inhibitoren – der Anti-CRISPR-Proteine (Acrs) – bietet ein neues Werkzeug für den Entwurf und die Implementierung digitaler Schaltkreise für Gene in vivo . Hier beschreiben wir ein Protokoll, das der Idee des "Design-Build-Test-Learn"-Bio-Engineering-Zyklus folgt und dCas9/dCas12a zusammen mit den entsprechenden Acrs verwendet, um kleine Transkriptionsnetzwerke zu etablieren, von denen sich einige wie Boolesche Gatter verhalten, in Saccharomyces cerevisiae. Diese Ergebnisse weisen auf die Eigenschaften von dCas9/dCas12a als Transkriptionsfaktoren hin. Um eine maximale Aktivierung der Genexpression zu erreichen, muss dSpCas9 insbesondere mit einer künstlich hergestellten Gerüst-RNA interagieren, die mehrere Kopien der VP64-Aktivierungsdomäne (AD) sammelt. Im Gegensatz dazu soll dCas12a an der C-Endstation mit dem starken VP64-p65-Rta (VPR) AD fusioniert werden. Darüber hinaus wird die Aktivität beider Cas-Proteine nicht durch eine Erhöhung der Menge an sgRNA/crRNA in der Zelle erhöht. In diesem Artikel wird auch erläutert, wie boolesche Gatter basierend auf der CRISPR-dCas-Acr-Wechselwirkung erstellt werden. Die mit AcrIIA4 fusionierte Hormonbindungsdomäne des humanen Östrogenrezeptors ist der Kern eines NOT-Gates, das auf β-Östradiol reagiert, während AcrVAs, die durch den induzierbaren GAL1-Promotor synthetisiert werden, es ermöglichen, sowohl YES- als auch NOT-Gatter mit Galaktose als Input nachzuahmen. In den letztgenannten Schaltungen zeigte AcrVA5 zusammen mit dLbCas12a das beste logische Verhalten.
Einleitung
Im Jahr 2011 schlugen Forscher eine Berechnungsmethode vor und entwickelten eine entsprechende Software für den automatischen Entwurf digitaler synthetischer Genschaltkreise1. Ein Benutzer musste die Anzahl der Eingänge (drei oder vier) angeben und die Wahrheitstabelle des Schaltkreises ausfüllen. Dies lieferte alle notwendigen Informationen, um den Schaltungsaufbau mit Hilfe von Techniken aus der Elektronik abzuleiten. Die Wahrheitstabelle wurde mit Hilfe der Karnaugh-Map-Methode2 in zwei boolesche Formeln übersetzt. Jede boolesche Formel besteht aus Klauseln, die logische Operationen (Summe oder Multiplikation) zwischen (einem Teil) der Schaltungseingänge und ihren Negationen (den Literalen) beschreiben. Klauseln wiederum werden entweder summiert (OR) oder multipliziert (AND), um den Schaltungsausgang zu berechnen. Jede Schaltung kann nach einer der beiden entsprechenden Formeln realisiert werden: eine in POS-Form (Produkt der Summen) und die andere in SOP-Darstellung (Summe der Produkte). Ersteres besteht aus einer Multiplikation von Klauseln (d. h. booleschen Gattern), die eine logische Summe der Literale enthalten. Letzteres ist im Gegensatz dazu eine Summe von Sätzen, bei denen die Literale multipliziert werden.
Elektrische Schaltkreise können auf einem Steckbrett realisiert werden, indem verschiedene Gatter physisch miteinander verdrahtet werden. Der elektrische Strom ermöglicht den Austausch von Signalen zwischen den Gattern, was zur Berechnung des Ausgangs führt.
In der Biologie ist die Situation komplexer. Ein Boolesches Gatter kann als Transkriptionseinheit (TU; d.h. die Sequenz "Promotor-kodierender Region-Terminator" innerhalb eukaryotischer Zellen) realisiert werden, in der die Transkription oder Translation (oder beides) reguliert wird. So stellen mindestens zwei Arten von Molekülen eine biologische Verdrahtung her: die Proteine des Transkriptionsfaktors und die nicht-kodierenden Antisense-RNAs1.
Ein digitaler Genschaltkreis ist in zwei oder drei Schichten von Gates organisiert, nämlich: 1) die Eingangsschicht, die aus JA- (Puffer) und NICHT-Gattern besteht und die Eingangschemikalien in Verdrahtungsmoleküle umwandelt; 2) die innere Schicht, die aus so vielen TEs besteht, wie es Klauseln in der entsprechenden booleschen Formel gibt. Wenn die Schaltung nach der SOP-Formel entworfen wird, erzeugt jede Klausel in der inneren Schicht den Schaltungsausgang (z. B. Fluoreszenz) in einer sogenannten verteilten Ausgangsarchitektur. Wenn die POS-Formel (Product of Summe) verwendet wird, ist eine 3) letzte Schicht erforderlich, die ein einzelnes multiplikatives Gate enthält, das die Verdrahtungsmoleküle aus der internen Schicht sammelt.
Insgesamt können in der synthetischen Biologie viele verschiedene Schemata für denselben Schaltkreis entworfen werden. Sie unterscheiden sich in der Anzahl und Art der TUs und der Verdrahtungsmoleküle. Um die einfachste Lösung für die Implementierung in Hefezellen zu wählen, ist jedes Schaltungsdesign mit einem Komplexitätswert S verknüpft, der definiert ist als
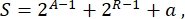
wobei A für die Anzahl der Aktivatoren, R für die Anzahl der Repressoren und a für die Menge der Antisense-RNA-Moleküle steht. Wenn entweder Aktivatoren oder Repressoren im Kreislauf fehlen, ist ihr Beitrag zu S gleich Null. Daher ist es schwieriger, ein Schaltungsschema im Labor (hohes S) zu realisieren, wenn es eine hohe Anzahl orthogonaler Transkriptionsfaktoren erfordert. Das bedeutet, dass neue Aktivatoren und Repressoren de novo entwickelt werden müssen, um die komplette Verdrahtung innerhalb der digitalen Schaltkreise zu realisieren. Prinzipiell können neuartige DNA-bindende Proteine unter Verwendung von Zinkfingerproteinen3 und TAL-Effektoren4 als Matrizen assembliert werden. Diese Option erscheint jedoch zu mühsam und zeitaufwändig; Daher sollte man sich hauptsächlich auf kleine RNAs und Translationsregulation verlassen, um komplexe Genschaltkreise zu finalisieren.
Ursprünglich wurde diese Methode entwickelt, um digitale Schaltkreise in Bakterien herzustellen. In eukaryotischen Zellen ist es in der Tat besser geeignet, anstelle von Antisense-RNAs von microRNAs (miRNAs) oder kleinen interferierenden RNAs (siRNAs) zu sprechen5. Der RNAi-Signalweg ist jedoch in der Hefe S. cerevisiae nicht vorhanden. Daher sollte man sich für vollständig transkriptionelle Netzwerke entscheiden. Angenommen, eine Schaltung benötigt fünf Aktivatoren und fünf Repressoren; Sein Komplexitätswert wäre S = 32. Die Komplexität der Schaltkreise kann reduziert werden, indem die 10 Transkriptionsfaktoren durch ein einzelnes dCas96 (Nuklease-defizientes Cas9) ersetzt werden, das zu einer Aktivierungsdomäne (AD) fusioniert ist. Wie in7 gezeigt, wirkt dCas9-AD als Repressor in Hefe bei der Bindung eines Promotors zwischen der TATA-Box und der TSS (Transkriptionsstartstelle) und als Aktivator, wenn es weit stromaufwärts der TATA-Box bindet. So kann man 10 Transkriptionsfaktoren durch ein einzelnes dCas9-AD-Fusionsprotein und 10 sgRNAs (Single Guide RNAs) ersetzen, was einen Gesamtkomplexitätsscore von S = 11 ergibt. Es ist schnell und einfach, zehn sgRNAs zu synthetisieren, wobei, wie bereits erwähnt, die Assemblierung von 10 Proteinen viel längere und kompliziertere Arbeit erfordern würde.
Alternativ kann man zwei orthogonale dCas-Proteine (z. B. dCas9 und dCas12a) verwenden: eines, um zu einem AD zu fusionieren, und das andere nackt oder in Kombination mit einer Repressionsdomäne. Der Komplexitätswert würde sich nur um eine Einheit erhöhen (S = 12). Daher sind CRISPR-dCas-Systeme der Schlüssel zum Aufbau sehr komplizierter digitaler Genschaltkreise in S. cerevisiae.
In dieser Arbeit wird die Effizienz von dCas9- und dCas12a-basierten Repressoren und Aktivatoren in Hefe eingehend charakterisiert. Die Ergebnisse zeigen, dass sie keine große Menge an sgRNA benötigen, um ihre Aktivität zu optimieren, so dass episomale Plasmide bevorzugt vermieden werden. Darüber hinaus sind dCas9-basierte Aktivatoren weitaus effektiver, wenn eine Scaffold-RNA (scRNA) verwendet wird, die Kopien des VP64 AD rekrutiert. Im Gegensatz dazu funktioniert dCas12a gut, wenn es direkt mit dem starken VPR-AD fusioniert wird. Darüber hinaus erfordert ein synthetisch aktivierter Promotor eine variable Anzahl von Zielstellen, abhängig von der Konfiguration des Aktivators (z.B. drei bei Verwendung von dCas12a-VPR, sechs bei dCas9-VP64 und nur eine mit dCas9 und einer scRNA). Als Repressor erscheint dCas12a prägnanter, wenn es an die kodierende Region und nicht an den Promotor bindet.
Ein Nachteil ist jedoch, dass CRISPR-dCas9/dCas12a nicht direkt mit Chemikalien interagieren. Daher sind sie im Eingabe-Layer möglicherweise nicht von Nutzen. Aus diesem Grund wurden alternative Boolesche Gate-Designs untersucht, die Anti-CRISPR-Proteine (Acrs) enthalten. Acrs wirken auf (d)Cas-Proteine und hemmen deren Funktion8. Daher sind sie ein Mittel, um die Aktivität von CRISPR-(d)Cas-Systemen zu modulieren. In dieser Arbeit werden die Wechselwirkungen zwischen Typ II Acrs und (d)Cas9 sowie Typ V Acrs und (d)Cas12a in S. cerevisiae eingehend analysiert. Da Acrs viel kleiner als Cas-Proteine sind, wurde durch Fusion der hormonbindenden Domäne des humanen Östrogenrezeptors9-HBD(hER)- mit AcrIIA4 ein NOT-Gatter gebildet, das auf das Östrogen β-Östradiol anspricht. Außerdem wurden eine Handvoll JA- und NICHT-Gatter, die dCas12a(-AD) konstitutiv exprimierten, und AcrVAs nach Induktion mit Galaktose realisiert. Derzeit dienen diese Tore nur als Proof of Concept. Sie stellen jedoch auch den ersten Schritt zu einem tiefgreifenden Umdenken des Algorithmus dar, um das computergestützte automatische Design von synthetischen Gen-Digitalschaltkreisen in Hefezellen durchzuführen.
Offenlegungen
Die Autoren erklären kein konkurrierendes finanzielles Interesse.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenCopyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten