Method Article
De la onda T de movilidad de iones-espectrometría de masas: Basic Procedimientos experimentales para el análisis de proteínas complejas
En este artículo
Resumen
De movilidad de iones-espectrometría de masas es una emergente tecnología de fase gaseosa, que separa los iones, sobre la base de su colisión sección transversal y la masa. El método proporciona información en tres dimensiones de la topología general y la forma de complejos de proteínas. Aquí, se describe un procedimiento básico para elaborar un instrumento normativo y la optimización, la calibración de los tiempos de desplazamiento, y la interpretación de los datos.
Resumen
De movilidad de iones (IM) es un método que mide el tiempo que tarda un ion de viajar a través de una célula de presión bajo la influencia de un campo eléctrico débil. La velocidad a la que los iones atraviesan la región de la deriva depende de su tamaño: los iones de gran experimentará un mayor número de colisiones con el gas inerte de fondo (por lo general N 2) y por lo tanto viajan más lentamente a través del dispositivo de mensajería instantánea que los iones que forman una pequeña sección transversal. En general, el tiempo necesario para que los iones a emigrar cuando la fase de gas denso que los separa, de acuerdo a la colisión de la sección transversal (Ω).
Recientemente, la espectrometría de IM fue acoplada a espectrometría de masas y una de onda (la onda T) SYNAPT de movilidad de iones espectrómetro de masas (IM-MS) fue puesto en libertad. La integración de la espectrometría de masas con la movilidad iónica permite una nueva dimensión de la separación de la muestra y la definición, dando un espectro en tres dimensiones (masa de tiempo de carga, la intensidad y la deriva). Esta técnica de separación permite que el solapamiento espectral a disminuir, y permite la resolución de complejos heterogéneos con masas muy similares, o las relaciones masa-carga, pero diferentes momentos a la deriva. Por otra parte, las mediciones de tiempo deriva proporcionar una capa importante de la información estructural, ya que Ω tiene que ver con la forma general y la topología de los iones. La correlación entre los valores de tiempo medido a la deriva y Ω se calcula mediante una curva de calibración generada a partir de las proteínas con calibrador definido secciones 1.
El poder del enfoque de IM-MS radica en su capacidad de definir el embalaje de la subunidad y la forma general de las asambleas de proteínas en concentraciones micromolar, y un corto condiciones fisiológicas. Varios estudios recientes de mensajería instantánea de ambas proteínas individuales 2,3 y no covalentes complejos de proteínas 4-9, logrado demostrar que la estructura de proteínas cuaternario se mantiene en la fase de gas, y destacó el potencial de este enfoque en el estudio de las cadenas proteicas de la geometría desconocida . Aquí, le ofrecemos una descripción detallada de IMS-MS análisis de complejos de proteínas utilizando el SYNAPT (cuadrupolo-Ion de movilidad en tiempo de vuelo) instrumento HDMS (Waters Ltd, el único comercial IM-MS instrumento actualmente disponible) 10. Se describen los pasos básicos de la optimización, la calibración de las secciones transversales de colisión, y los métodos para el procesamiento e interpretación de datos. El último paso del protocolo describe los métodos para calcular valores teóricos Ω. En general, el protocolo no pretende cubrir todos los aspectos de la IM-MS caracterización de conjuntos de proteínas, sino que su objetivo es introducir a los aspectos prácticos del método a los nuevos investigadores en el campo.
Protocolo
El procedimiento se describe se centra únicamente en IM-MS análisis de los complejos de proteínas. Por lo tanto, sugerimos que los investigadores no conocen el campo de la MS estructurales se refieren a los pasos de preparación de muestras, calibración de instrumentos y MS y MS en tándem procedimientos de optimización descritos en Kirshenbaum et al. Http://www.jove.com/index/details 2009. stp? ID = 1954. En general, este protocolo consiste en bajas concentraciones micromolar de complejos (1-20 mM) en un tampón volátiles como el acetato de amonio (0,005 a 1 M, pH 6.8). Teniendo en cuenta que 2.1 l son consumidos por nanoflujo capilar, le sugerimos 10-20 l en un volumen mínimo, para permitir la optimización de las condiciones de la EM.
Parte 1: La adquisición de una movilidad de iones-espectrometría de masa espectro
- Ajuste el espectrómetro de masas en los siguientes modos de operación: Movilidad-TOF, la adquisición de iones positivos, y V-mode.
- Encienda todos los gases (API, la trampa y IMS). Usamos N 2 para la separación de mensajería instantánea, y Ar en la trampa / Transferencia. Recomienda valores iniciales son de 1,5 ml / min para la región trampa, y un flujo de gas de 24 ml / min para el dispositivo de IMS.
- Establecer el rango de adquisición de m / z. De un complejo de proteína desconocida, se sugiere el uso inicial de un amplio rango de masas, que a su vez puede ser reducida a los valores deseados. Al mismo tiempo, ajustar el perfil de MS para la eficiencia de transmisión máxima. Para los grandes complejos, el rango de masas de adquisición se debe establecer a partir de 1.000 - 32.000 m / z, y el perfil de MS en Auto. De lo contrario, el perfil se puede ajustar, de acuerdo con el siguiente cuadro:
m / z moran (%) rampa (%) 960 10 20 3200 30 40 10667 - Compruebe la configuración de RF y, si es necesario, ajuste los valores adecuados para grandes complejos de proteínas, por lo tanto:
Fuente Trampa IMS Transferencia RF Offset 450 380 380 380 De ganancia de RF 0 0 0 0 Límite de RF 450 380 380 380 - Aplicar la tensión capilar (1,050-1,400 V) y la presión nanoflujo baja (0,00 a 0.03 bar). Una vez que se inicia la aspersión, tratar de reducir la presión nanoflujo a un valor mínimo. Además, ajustar la posición de los capilares, con respecto al cono.
- Ajustar los parámetros de adquisición MS, para adquirir un bien resueltos espectro MS: el gradiente de presión a lo largo del instrumento, y el cono de muestreo, así como el cono de extracción, los prejuicios, la trampa y la transferencia de valores posibles, todos deben ser optimizados (detallado en el correspondiente JoVe protocolo Kirshenbaum et al. https://www.jove.com/index/details.stp?ID=1954 2009). Aunque estos parámetros se muestra dependiente de las condiciones que usamos para la adquisición de espectros de masas de iones MS diversos, desde péptido de complejos de proteínas, se resumen en la Tabla 1, (ver Fig. 1.). Para minimizar la activación del complejo de tratar de reducir gradualmente (en intervalos de aproximadamente 10 V) Tensiones del cono de la muestra, la trampa y el sesgo, sin cambiar la posición de la punta.
- Una vez que un espectro de masas óptimo se obtiene el perfil temporal de deriva debe ser ajustado. Al analizar las cadenas proteicas, las condiciones óptimas tanto para mediciones de masa y la movilidad son a menudo incompatibles, por lo tanto, es importante encontrar el equilibrio adecuado entre los dos. En general, la trama de movilidad de iones debe ser optimizado de tal manera que los picos se distribuyen en todo el rango de deriva todo este tiempo, y el perfil de pico es suave, acercándose a una distribución de Gauss (Fig. 2A, 2B). Pico importante asimetría puede estar relacionada con escasa separación de múltiples conformaciones.
- Como regla general, los tres parámetros, T-velocidad de la onda, la altura de la onda T y la tasa de IMS de flujo de gas se puede ajustar para optimizar la separación de la movilidad. El aumento de la velocidad de la onda T se ampliará el tiempo de desplazamiento perfil de distribución, mientras que el aumento de la onda T valores de altura de lo estrecho. Del mismo modo, el aumento del flujo de gas IMS cambiará el perfil de tiempo de desplazamiento hacia valores más altos (el mínimo flujo de IMS de gas debe ser de 10 ml / min). Se recomienda salir dos de las tres variables fijas disponibles, y la optimización de la tercera hasta que el espectro de mensajería instantánea está bien resuelto (Fig. 2B). Para este fin, por la velocidad de la onda T y el flujo de gas a 250 m / s y 24 ml / min, rerespectivamente. Entonces, como punto de partida, establecer la altura de la onda T a 3 V y, en una forma gradual, se aumentan en incrementos de 1 V. En general, los iones más grandes requerirán mayor altura de las olas. Por lo general, no hay necesidad de modificar la presión de IMS, sin embargo, cuando las tensiones de polarización que se requieren altas para el ajuste, la reducción de los gases de IMS permitirá una disminución en el valor de tensión de polarización y, en consecuencia, una reducción en la activación de las proteínas complejas. En general, la resolución máxima de 10 a 12 t / Dt se puede llegar.
- Cuando las condiciones no están optimizados (T baja altura de las olas o de alta velocidad de la onda T y / o presión alta IM), los iones de no atravesar el dispositivo de mensajería instantánea con eficacia, y su viaje puede durar más del tiempo necesario para el ion siguiente paquete para ser lanzado en la celda de movilidad. Como resultado, un paquete de iones de nuevo será liberado de la trampa de la región antes de que el paquete anterior se ha entregado a la región de empuje. Esto conducirá a un "roll-over" efecto, en la que el máximo observado en la primera parte del espectro de tiempo de desplazamiento es idéntica a la de los iones en el borde de la cola (Fig. 2C). Este artefacto puede ser eliminada mediante el aumento de la altura de la onda T, y la disminución de la velocidad de la onda T y la presión de IMS. Además, el tiempo de liberación trampa se puede ajustar. Por otra parte, es importante comprobar que la transferencia de la onda T se ajusta a la altura de al menos 5 V. Para evitar la fuga de iones hacia la célula de IMS, se recomienda que la altura de la trampa de movilidad se mantuvo en niveles máximos (30 V).
- Baja velocidad y alta amplitud de la transferencia de la onda T puede conducir a la "ondulante" deriva del perfil de distribución del tiempo (Fig. 2D). Este artefacto se produce cuando la separación de la movilidad de los iones (iones de llegada / la deriva del tiempo) no se mantiene a través de la transferencia y regiones TOF, debido a la sincronización parcial entre la frecuencia de empuje y la velocidad de transferencia de la onda T. Con el fin de eliminar este efecto, ya sea el momento de empuje o la transferencia de la onda T velocidad debe ser ajustada. Dado que la frecuencia de empuje está relacionado con el rango de masas, este artefacto puede reaparecer cuando este parámetro se cambia. T-altura de ola ejerce un efecto menor, a pesar de su reducción también puede ayudar a eliminar las ondulaciones.
- Una vez que los parámetros antes mencionados están optimizados, los datos del IM-MS se pueden adquirir.
Parte 2: Evaluación condiciones experimentales para garantizar la movilidad de las mediciones de las estructuras nativas
Para lograr una alta resolución picos MS, complejos de proteínas son a menudo activa dentro del espectrómetro de masas, para promover el despojo de aguas residuales y los componentes del tampón 11. Sin embargo, si la energía de activación se incrementa más allá de un valor umbral, desarrollo parcial puede inducir la formación de varios estados intermedios 12, que es poco probable que se corresponden con los nativos, la solución de estado la estructura (Fig. 3A-C). Como resultado de ello, el máximo tiempo de deriva puede ser cambiado y ampliado, lo que refleja la población heterogénea de las estructuras desplegadas.
Con el fin de obtener datos de la deriva tiempo consistente con las estructuras en fase de solución, es esencial para controlar cuidadosamente los voltajes usados para acelerar iones, antes de la separación de mensajería instantánea. Por otra parte, para MS de alta resolución, es preferible aumentar la transferencia en lugar de la tensión de la trampa. Como el dispositivo de mensajería instantánea se coloca, en primer lugar, seguida por la región de transferencia y el analizador TOF, por lo tanto, la activación sigue la medición de mensajería instantánea y los iones no se ve afectado, mientras que la exactitud de MS se puede aumentar.
Para validar que la adquisición de datos se realiza en condiciones que mantienen la estructura original del complejo, se recomienda que los datos se registraron en un rango de condiciones experimentales y la solución, más que de acuerdo con un optimizado solo conjunto de parámetros:
- Incrementar el voltaje capilar y el cono de manera gradual, mientras se monitorea el efecto sobre el espectro de tiempo a la deriva.
- Como en el paso 1, el aumento de la tensión colisión trampa en una forma gradual, y la adquisición de datos en intervalos de 10 V.
- Para identificar las conformaciones desplegadas y evaluar los datos obtenidos, de forma manual induce la disociación del complejo proteína mediante titulación de la muestra con ácido acético en un rango de pH de 2.7, y registrar los datos (Fig. 3B).
Parte 3: La correlación entre los valores de tiempo a la deriva y las áreas transversales
A diferencia de las mediciones convencionales de mensajería instantánea, en la que los valores de la deriva midió el tiempo se relaciona linealmente con Ω, en el sistema IMS de la onda T, la sección transversal se define por un método de calibración. Así, en lugar de una medida absoluta, una correlación exponencial en relación se genera entre los tiempos de desplazamiento medido y 1,13 Ω: 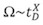
donde t D es el tiempo de desplazamiento medido, y X es la constante de la proporción que se puede extraer de una curva de calibración. La calibración se realizaed mediante la medición de los tiempos de desplazamiento de los iones con conocidos Ω (medido a partir de experimentos de mensajería instantánea convencional).
- Mediciones de tiempo de la deriva se calibran las proteínas desnaturalizadas equina del citocromo C, mioglobina corazón de caballos y bovinos con ubiquitina conocida colisión secciones. Con este fin, las soluciones de 10 M de 49/49/2, la relación de volumen de agua / metanol / ácido acético debe estar preparado (los reactivos utilizados se describen en la Tabla 3).
- Adquirir IM-MS de datos para las proteínas calibrador bajo exactamente las condiciones del instrumento mismo que se utiliza para la proteína diana o complejo de proteínas:
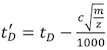 (Parte 1). Todas las tensiones y los valores de presión deben ser idénticos, para preservar los valores de la separación de mensajería instantánea.
(Parte 1). Todas las tensiones y los valores de presión deben ser idénticos, para preservar los valores de la separación de mensajería instantánea. - Para cada estado de carga de las proteínas calibrante extraer el valor de deriva tiempo experimental (t D) (descrito en la Parte 4).
- Corregir cada una de las veces deriva calibrante (t D) (Tabla 2) utilizando la siguiente ecuación:
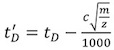 , Donde m / z es la relación masa-carga de los iones observados, y c es el ciclo de trabajo mejorado (EDC) coeficiente de retraso 1. Su valor, por lo general entre 1,4 y 1,6, es el instrumento-dependiente. El valor de la EDC se indica dentro del sistema | Configuración Industriales | Adquisición ficha Configuración.
, Donde m / z es la relación masa-carga de los iones observados, y c es el ciclo de trabajo mejorado (EDC) coeficiente de retraso 1. Su valor, por lo general entre 1,4 y 1,6, es el instrumento-dependiente. El valor de la EDC se indica dentro del sistema | Configuración Industriales | Adquisición ficha Configuración. - Utilizando la sección transversal del Prof. David Clemmer la base de datos:
http://www.indiana.edu/ ~ Clemmer / Investigación / cross% 20section% 20database/Proteins/protein_cs.htm 14 correcta cada uno de los calibrante sección transversal, tanto para el estado de carga de iones y masa reducida.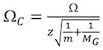
, Donde Ω C es la corrección de sección, Ω es la literatura la sección transversal, z es el estado de carga de iones, m es el peso molecular del ion calibrador, y M G es el peso molecular del gas de fondo de mensajería instantánea (por lo general N 2). - Parcela En (t D) En contra (Ω C) (Fig. 4A).
- La curva resultante se corresponde con la siguiente ecuación:
 Los parámetros X y A puede ser extraído mediante el ajuste de la trama de una relación lineal. El X pendiente corresponde al factor de proporción exponencial, y A representa el constante ajuste determinado.
Los parámetros X y A puede ser extraído mediante el ajuste de la trama de una relación lineal. El X pendiente corresponde al factor de proporción exponencial, y A representa el constante ajuste determinado. - Calcular el coeficiente de correlación de forma r 2, usando la ecuación de Pearson:
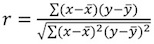 . Los valores aceptables para r 2 es mayor que 0,95 (Fig. 4B). El valor del coeficiente de correlación más baja puede deberse a:
. Los valores aceptables para r 2 es mayor que 0,95 (Fig. 4B). El valor del coeficiente de correlación más baja puede deberse a: - Incompleto desarrollo de los calibradores de proteínas. Esto dará lugar a la ampliación de pico debido a la asamblea heterogénea de estados intermedios.
- En nuestra experiencia, una muestra de edad se puede deteriorar los espectros de mensajería instantánea.
- Diferentes condiciones experimentales utilizadas para las proteínas calibrante diferentes. En este caso, el trazado de los datos para cada proteína por separado debe generar diversos coeficientes de correlación, aunque cada uno de ellos debe ser superior a 0,95.
- Datos imprecisos o incorrectos suavizado y el centrado de la distribución del tiempo a la deriva.
- Error de cálculo.
- Recorrect el tiempo deriva calibrador con el factor determinado exponencial, X, que se deriven en el paso 7:
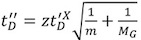
- Como una etapa de validación, replot Ω C vs
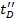 y definir el coeficiente de correlación. Valores superiores a 0,95 son de esperar.
y definir el coeficiente de correlación. Valores superiores a 0,95 son de esperar. - De acuerdo con el procedimiento descrito en el paso 4, corregir el tiempo de desplazamiento medido de la proteína diana o complejo de proteínas:
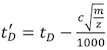
- Calibrar el tiempo de la deriva de la proteína diana / complejo de proteínas utilizando el factor exponencial, X, definida en el paso 7:
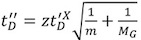
- Calcular Ω de la meta de proteína / proteína compleja con la constante ajuste determinado, A, definida en el paso 7:
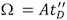 .
. - Para cada condición experimental, los pasos 2 a 13 años deben repetirse. Al definir el área transversal de la proteína desconocido o complejo de proteínas, se recomienda que cada experimento se repitió al menos tres veces, y se determinó la desviación estándar de estas mediciones por triplicado.
Parte 4: Definición de los valores de la deriva el tiempo
El software necesario: MassLynx y Driftscope (Waters).
- Abrir el espectro de IM-MS con el software Driftscope.
- En el menú principal, seleccione View y Cromatograma desmarque, Tiempo de deriva y el espectro (opcional), dejando activas sólo el mapa en 2D que muestra la hora frente a la deriva m / z.
- En la barra de menú, seleccione Mostrar | Opciones | Grupo Editor de pantalla y ajustar los valores umbral de intensidad para minimizar el ruido de fondo (en la mayoría de los casos, este valor se puede definir como Min = 30-40% y Max = 100% de muestras).
- Seleccione Mostrar | Mapa 2D Escala de Intensidad, y tres opciones aparecerán: escala lineal, escala logarítmica y la escala de la raíz cuadrada. Una selección de escala logarítmica (logarítmica transformar los datos) se comprime el código de la intensidad del color, y permitir la aparición simultánea de una amplia gama de intensidades (en comparación con las opciones de raíz lineal y de la plaza, con el que sólo los picos más intensos se hará visible) .
- Desde el panel de la barra de herramientas, utilice el botón Herramienta de selección. Esta opción activará las opciones de selección diferentes, y permitir la selección de la región de referencia en el espectro. La herramienta más precisa es Activar Región de Selecciones de interés, mediante el cual puede ser un borde alrededor de la región de interés, lo que excluye a todos los datos innecesarios y los picos de ruido. Del mismo modo, las opciones de selección ortogonal y la banda son útiles, cuando la región de interés no está rodeado de picos redundantes.
- Una vez que la región de interés seleccionada, utilice el comando ACCEPT Selección actual para eliminar la información innecesaria.
- Exportar los datos a MassLynx, manteniendo la información del tiempo a la deriva.
- Dentro de MassLynx, abra el cromatograma del espectro deriva un ahorro de tiempo, y combinar los contenedores tiempo. El espectro de masas correspondiente se abrirá automáticamente.
- Aplicar la función de suavizado mediante la definición del tamaño de la ventana y el número de parámetros suave (debe ser adaptado específicamente para cada espectro, con valores mínimos).
- Aplicar de referencia resta, si es necesario.
- Centro del espectro y medir la masa, para validar la identidad de proteínas y precisión la masa.
- Para cada estado de la carga, se combinan la gama m / z. El espectro de la deriva de tiempo correspondiente aparecerá automáticamente. Suave y el centro de la deriva perfil de tiempo, y definir el valor de tiempo de deriva, indicando el centro de gravedad de cada pico.
Parte 5: Resultados de Representante

Figura 1. Representación esquemática del instrumento SYNAPT HDMS indicando los principales parámetros ajustables de IMS-MS adquisición. Parámetros experimentales utilizadas para la IM-MS mediciones son etiquetados de acuerdo a su posición dentro del instrumento. El haz de iones es de color rojo, y la presión en cada región se designa mediante un código de color. El panel en la parte inferior muestra la caída de potencial a lo largo del instrumento y las posibles diferencias que definen la trampa y la transferencia de energías de colisión, así como el potencial de sesgo. Todos los potenciales leer-backs se hace referencia a la tensión estática de compensación que se establece normalmente en 120.

Figura 2. De movilidad de iones distribuciones de tiempo de llegada de la proteína Gβυ.
A. Una alta velocidad de la onda T conduce a una distribución estrecha del perfil de tiempo a la deriva. La trama muestra la distribución del tiempo de llegada de los Los estados 16 + (rojo), 15 + (verde), 14 + (azul), y 13 + (magenta) de carga, así como el perfil de la deriva el tiempo total (en negro) de la proteína G βυ.
B. Una desviación óptima del espectro de tiempo con una suave forma de pico de Gauss. Etiquetas de colores similares, como en A.
C. Un "roll-over" efecto que se produce cuando el tiempo necesario para que los iones de atravesar la célula de la movilidad es más lento que el intervalo entre las inyecciones de paquetes de iones de nuevo en el dispositivo. Como resultado, el pico de la deriva tiempo prolongado aparece al principio del espectro. Este efecto puede ser eliminada mediante el aumento de la altura de la onda T, y la disminución de la velocidad de la onda T y la presión de IMS.
'Ondas' D. Artificial se producen cuando la transferencia de T-velocidad de la onda y la frecuencia de empuje son parcialmente sincronizados. Este efecto puede ser superado mediante el ajuste sea la frecuencia de empuje o la velocidad de transferencia de la onda T.
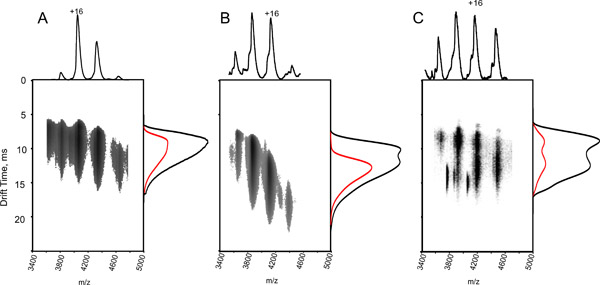
Figura 3. El efecto de la activación de iones y las condiciones de desnaturalización parcial de IM-MS espectros de la hemoglobina. Parcela de tiempo la deriva frente a m / z para el complejo hemoglobina tetramérica, con una solución acuosa de acetato de amonio 10 mM (pH = 7.6) (A, C) y la adición de 0,1% de ácido acético (B). Los datos obtenidos con la trampa de voltaje de energía de colisión de 13 V (A, B) y 35 V (C), aunque en los tres paneles de la espectrometría de masas (proyectado en la parte superior) es similar, con una serie de carga tetramérica centrada a 4.000 m / z el perfil temporal de deriva (proyectado a los lados) es diferente (la distribución total de tiempo de desplazamiento esen negro, y el perfil de + 16 es de color rojo). El tiempo de desplazamiento más largo de la muestra parcialmente desnaturalizado, obtenido en B, y los iones en fase gaseosa activados obtenidos en C, es indicativo de un cierto grado de desarrollo. Esta observación pone de manifiesto que a pesar de que la masa medida corresponde a un complejo intacto, su estructura de la solución se vea interrumpido. Como consecuencia, el control cuidadoso de las condiciones experimentales es necesario.
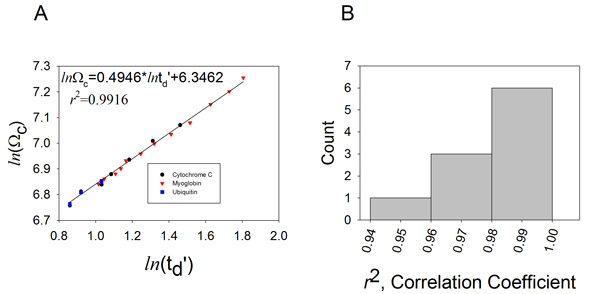
Figura 4. Mediante la generación de una curva de calibración, la deriva mediciones de tiempo y de la colisión secciones pueda establecer una correlación.
A. Los valores medidos de la deriva del tiempo de los estados de carga múltiple de equinos del citocromo C (círculos), mioglobina caballo del corazón (triángulos) y ubiquitina bovina (cuadrados) se representará gráficamente en la literatura los valores de Ω corregido, tanto para el estado de carga de iones y masa reducida. El ajuste de los rendimientos de una función lineal que corresponde a: ln (Ω C) = xln (t D ') + A. El factor exponencial determinada (X), el ajuste determinado por constantes (A), y coeficiente de correlación se muestran en el gráfico de los datos adquiridos a una velocidad de la onda T de 350 m / s, y una altura de ola estática de 11 V. B. Un histograma de la distribución del coeficiente de correlación obtenido a partir de 10 experimentos de calibración consecutivos.
| Proteína de la muestra / parámetros técnicos | GluFibrino- péptido monómero 1,6 kDa | La mioglobina monómero 17 kDa | Hemoglobina tetrámero 67 kDa | Transferrina monómero 80 kDa | GroEL 14-mer 801 kDa |
| La presión de soporte, mBar | 4.4 | 5.0 | 5.1 | 5.1 | 6.5 |
| La presión de la trampa, mBar | 1.6x10 -2 | 2.4x10 -2 | 2.4x10 -2 | 2.6x10 -2 | 2.8x10 -2 |
| Presión de IMS, mBar | 4.4x10 -1 | 4.4x10 -1 | 4.4x10 -1 | 4.4x10 -1 | 4.2x10 -1 |
| Muestreo de tensión de cono, V | 46 | 80 | 80 | 80 | 118 |
| Extracción de tensión de cono, V | 1.7 | 1 | 1 | 1 | 3 |
| Tensión de polarización, V | 20 | 20 | 25 | 25 | 50 |
| Atrapan la energía de colisión, V | 20 | 15 | 15 | 15 | 80 |
| Transferencia de energía de colisión, V | 5 | 12 | 12 | 12 | 15 |
Tabla 1. Condiciones experimentales utilizadas para el análisis de macromoléculas.
| Las proteínas estándar | Masa molecular (m) | Cargos (z) | m / z | Colisión de la sección transversal (en 2) |
| Citocromo C | 12213 | 10 | 1222.3 | 2226 |
| 11 | 1111.3 | 2303 | ||
| 12 | 1018.8 | 2335 | ||
| 13 | 940.5 | 2391 | ||
| 14 | 873.4 | 2473 | ||
| 15 | 815.2 | 2579 | ||
| 16 | 764.3 | 2679 | ||
| 17 | 719.4 | 2723 | ||
| 18 | 679.5 | 2766 | ||
| La mioglobina | 16952 | 11 | 1542.1 | 2942 |
| 12 | 1413.7 | 3044 | ||
| 13 | 1305.0 | 3136 | ||
| 14 | 1211.9 | 3143 | ||
| 15 | 1131.1 | 3230 | ||
| 16 | 1060.5 | 3313 | ||
| 17 | 998.2 | 3384 | ||
| 18 | 942.8 | 3489 | ||
| 19 | 893.2 | 3570 | ||
| 20 | 848.6 | 3682 | ||
| 21 | 808.2 | 3792 | ||
| 22 | 771.6 | 3815 | ||
| Ubiquitina | 8565 | 8 | 1071.6 | 1442 |
| 8 | 1071.6 | 1622 | ||
| 9 | 952.7 | 1649 | ||
| 10 | 857.5 | 1732 | ||
| 11 | 779.6 | 1802 | ||
Tabla 2. Calibrante proteínas y su colisión secciones de los valores determinados por el convencional Medicion IMS 14.
| Dispositivos | Empresa | Número de catálogo |
| SYNAPT HDMS-32K generador de RF | Aguas Ltd. | |
| P-97-Brown Flaming micropipeta extractor | Sutter Instrumentos | P-97 |
| Sputter aplicador | Microscopía electrónica de Ciencias | EMS550 |
| Microscopio binocular | Nikon | |
| Reactivos | Empresa | Número de catálogo |
| Acetato de amonio | Sigma-Aldrich | Sigma, A2706 |
| Csl 99,999% | Sigma-Aldrich | Aldrich, 203033 |
| Metanol | Sigma-Aldrich | Fluka, 34966 |
| El ácido acético | Fisher Scientific | AC12404 |
| Equina mioglobina (de corazón de caballo) | Sigma-Aldrich | M1882 |
| Equina del citocromo c (del corazón de caballo) | Sigma-Aldrich | C-2506 |
| Bovina ubiquitina (de los glóbulos rojos) | Sigma-Aldrich | U6253 |
| Hemoglobina | Sigma-Aldrich | H2625 |
| Gas | Comentarios | |
| Nitrógeno, 99,999% de pureza | 8 cilindros metro cúbico | |
| Argón, 99,999% de pureza | 8.8 meterscylinder cúbicos | |
Tabla 3. Reactivos y equipo.
Discusión
El protocolo aquí descrito permite definir la sección transversal de colisión de las proteínas o complejos de proteínas con un desconocido la estructura tridimensional, con el objetivo de proporcionar información sobre su forma general, el embalaje y la topología de la subunidad. Con este fin, una vez los valores de la sección transversal de colisión se muestran, es necesario convertir estos valores a los elementos estructurales. Este proceso requiere de esfuerzos adicionales experimental, así como el análisis computacional, el cual se describen brevemente a continuación.
Para empezar, se recomienda analizar las proteínas o complejos de proteínas con estructuras conocidas. Estas mediciones pueden proporcionar un útil de control de calidad de la metodología y permitirá evaluar la precisión de los parámetros de adquisición mediante la comparación de los valores de Ω teóricos y medidos. El teórico áreas de corte transversal se puede calcular a partir de la estructura cristalina coordenadas utilizando el software MOBCAL 15,16, que es un software de código abierto basado en FORTRAN que permite la edición de código de acuerdo a las necesidades del operador. Para el funcionamiento de estos cálculos se requiere de modificar el programa de tal manera que el número de cálculos iterativos realizados por la estructura de entrada es mayor y que los archivos de coordenadas que contiene gran número de átomos son aceptados 1.
Una estrategia de IM-MS para la definición de los arreglos topológicos de las subunidades en las asambleas de varios componentes se ha propuesto recientemente 4,6. El método consiste en la vigilancia de las vías de la disociación de las asambleas de proteínas a componentes más pequeños. Esta disociación se logra a través del ajuste de las condiciones de control en fase de solución, lo que da lugar a una distribución de subcomplejos reflejo de los "bloques constructores" de las asambleas. La medición simultánea de los valores de Ω tanto del complejo intacto y productos desmontaje genera restricciones estructurales que se utilizan para el cálculo de los modelos topológicos de los complejos de proteínas. El supuesto básico que subyace a esta metodología es que la subcomplejos generado conservar su lengua materna-como confirmaciones, de hecho, estudios recientes han demostrado que la estructura de la solución de los productos de desmontaje se mantiene y no hay reorganización importante en cualquiera de las soluciones o las fases de gas se han producido 4,6.
El último paso en la asignación de la estructura cuaternaria de iones de proteína de fase de gas del complejo es conveniente los valores de la sección transversal de colisión con los modelos generados por computadora. Métodos de modelización, se emplean con el fin de explorar las diferentes disposiciones posibles topológica de subunidades y su in silico valores Ω se calculan y se comparan con los experimentales. Actualmente, sólo un pocos métodos computacionales se utilizan, como el spheretype de grano grueso, método que se aproxima el diámetro de las subunidades de 1,8. En general, este campo está todavía en sus primeros años de vida y el desarrollo se requiere para hacer este planteamiento genérico, y aplicable a una amplia gama de complejos.
Divulgaciones
Agradecimientos
Los autores agradecen a los miembros del grupo a Sharon por su revisión crítica, y por sus contribuciones al manuscrito. Estamos muy agradecidos por el apoyo de los Programas de Morasha y bikura, la Israel Science Foundation (Grant N º 1823 / 07 y 378/08), el Josef Cohn Minerva Centro de Investigación biomembrana, la Familia Chais Programa de Becas para los nuevos científicos, el Abraham y Sonia Rochlin Fundación, el Fideicomiso de la Familia Wolfson caridad; el Centro Helen y Milton A. Kimmelman de Estructura Biomolecular y de la Asamblea, la finca de Shlomo y Beirzwinsky Sabine; Meil de Botton Aynsley, y Karen Siem, Reino Unido.
Referencias
- Ruotolo, B. T. Ion mobility-mass spectrometry analysis of large proteincomplexes. Nat Protoc. 3 (7), 1139-1152 (2008).
- Scarff, C. A., Thalassinos, K., Hilton, G. R., Scrivens, J. H. Travelling wave ion mobility mass spectrometry studies of protein structure: biological significance and comparison with X-ray crystallography and nuclear magnetic resonance spectroscopy measurements. Rapid Commun Mass Spectrom. 22 (20), 3297-3304 (2008).
- Smith, D. P. Deciphering drift time measurements from travelling wave ion mobility spectrometry-mass spectrometry studies. Eur J Mass Spectrom (Chichester, Eng). 15 (2), 113-130 (2009).
- Leary, J. A. Methodology for measuring conformation of solvent-disrupted protein subunits using T-WAVE ion mobility MS: an investigation into eukaryotic initiation factors. J Am Soc Mass Spectrom. 20 (9), 1699-1706 (2009).
- Lorenzen, K. Determination of stoichiometry and conformational changes in the first step of the P22 tail assembly. J Mol Biol. 379 (2), 385-396 (2008).
- Pukala, T. L. Subunit architecture of multiprotein assemblies determined using restraints from gas-phase measurements. Structure. 17 (9), 1235-1243 (2009).
- van Duijn, E. Chaperonin complexes monitored by ion mobility mass spectrometry. J Am Chem Soc. 131 (4), 1452-1459 (2009).
- Ruotolo, B. T. Evidence for macromolecular protein rings in the absence of bulk water. Science. 310 (5754), 1658-1661 (2005).
- Ruotolo, B. T., Robinson, C. V. Aspects of native proteins are retained in vacuum. Curr Opin Chem Biol. 10 (5), 402-408 (2006).
- Giles, K. Applications of a travelling wave-based radio-frequency-only stacked ring ion guide. Rapid Commun Mass Spectrom. 18 (20), 2401-2414 (2004).
- McKay, A. R., Ruotolo, B. T., Ilag, L. L., Robinson, C. V. Mass measurements of increased accuracy resolve heterogeneous populations of intact ribosomes. J Am Chem Soc. 128 (35), 11433-11442 (2006).
- Ruotolo, B. T. Ion mobility-mass spectrometry reveals long-lived, unfolded intermediates in the dissociation of protein complexes. Angew Chem Int Ed Engl. 46 (42), 8001-8004 (2007).
- Morton, V. L., Stockley, P. G., Stonehouse, N. J., Ashcroft, A. E. Insights into virus capsid assembly from non-covalent mass spectrometry. Mass Spectrom Rev. 27 (6), 575-595 (2008).
- Valentine, S. J., Counterman, A. E., Clemmer, D. E. A database of 660 peptide ion cross sections: use of intrinsic size parameters for bona fide predictions of cross sections. J Am Soc Mass Spectrom. 10 (11), 1188-1211 (1999).
- Mesleh, M. F. Structural information from ion mobility measurements: effects of the long-range potential. J Phys Chem. 100, 16082-16086 (1996).
- Shvartsburg, A. A., Jarrold, M. F. An exact hard-spheres scattering model for the mobilities of polyatomic ions. Chem Phys Lett. 261, 86-91 (1996).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados