Method Article
T-Wellen-Ionen-Mobilitäts-Massenspektrometrie: Basic Experimentelle Verfahren zur Protein Complex Analysis
In diesem Artikel
Zusammenfassung
Ionen-Mobilitäts-Massenspektrometrie ist eine aufstrebende Gas-Phasen-Technologie, die Ionen trennt, auf der Grundlage ihrer Kollision Querschnitt und Masse. Die Methode liefert dreidimensionale Informationen über die gesamte Topologie und Form der Protein-Komplexen. Hier skizzieren wir ein grundlegendes Verfahren für die Geräteeinstellung und Optimierung, Kalibrierung der Drift Zeiten, und die Interpretation der Daten.
Zusammenfassung
Ionen-Mobilitäts-(IM) ist eine Methode, die Zeit für ein Ion durch eine unter Druck stehende Zelle unter dem Einfluss eines schwachen elektrischen Feldes Reise Maßnahmen ergriffen. Die Geschwindigkeit, mit der die Ionen durchqueren die Driftregion hängt von ihrer Größe: große Ionen wird eine größere Zahl von Kollisionen mit dem Hintergrund Inertgas (meist N 2) Erfahrung und damit mehr reisen langsam durch die IM-Gerät als die Ionen, die eine kleinere umfassen Querschnitt. Im Allgemeinen ist die Zeit, die für die Ionen zu wandern wenn die dichten Gasphase trennt sie, nach ihrer Kollision Querschnitt (Ω).
Kürzlich wurde IM-Spektrometrie mit der Massenspektrometrie und ein travelling-wave (T-Welle) SYNAPT Ionen-Mobilitäts-Massenspektrometer (IM-MS) veröffentlicht wurde gekoppelt. Die Integration der Massenspektrometrie mit Ionenbeweglichkeit ermöglicht eine zusätzliche Dimension der Probe Trennung und Definition, was eine dreidimensionale Spektrum (Masse zu Ladung, Intensität und Drift-Zeit). Diese Trennung ermöglicht es den spektralen Überlappung zu verringern, und ermöglicht Auflösung von heterogenen Komplexe mit sehr ähnlichen Masse oder Masse-Ladungs-Verhältnis, aber unterschiedliche Drift Zeiten. Darüber hinaus bieten die Driftzeit Messungen eine wichtige Schicht von strukturellen Informationen, wie Ω um die Form und die Topologie des Ions verbunden ist. Die Korrelation zwischen der gemessenen Driftzeit Werte und Ω berechnet unter Verwendung einer Eichkurve aus Kalibrierlösung Proteine mit definierten Querschnitten 1 erzeugt.
Die Macht der IM-MS Ansatzes liegt in seiner Fähigkeit, die Untereinheit Verpackung und allgemeine Form von Protein-Baugruppen in mikromolaren Konzentrationen zu definieren, und annähernd physiologischen Bedingungen 1. Mehrere neuere Studien IM sowohl einzelner Proteine 2,3 und nicht-kovalente Protein-Komplexe 4-9, erfolgreich demonstriert, dass Protein Quartärstruktur in der Gasphase erhalten bleibt, und wies auf das Potenzial dieses Ansatzes in der Untersuchung von Protein-Baugruppen unbekannter Geometrie . Hier bieten wir Ihnen eine detaillierte Beschreibung der IMS-MS-Analyse von Protein-Komplexen mit dem SYNAPT (Quadrupol-Ion Mobility-Time-of-Flight) HDMS Instrument (Waters Ltd; der einzige kommerzielle IM-MS-Gerät derzeit) 10. Wir beschreiben die grundlegenden Optimierung Schritte, die Kalibrierung von Stoßquerschnitte und Methoden für die Datenverarbeitung und-interpretation. Der letzte Schritt des Protokolls werden Methoden zur Berechnung der theoretischen Ω Werte. Insgesamt hat das Protokoll nicht versuchen, jeden Aspekt des IM-MS Charakterisierung von Protein-Baugruppen decken, sondern es ist ihr Ziel, die praktischen Aspekte der Methode, um neue Forscher auf dem Gebiet einzuführen.
Protokoll
Das Verfahren beschreiben wir konzentriert sich ausschließlich auf IM-MS-Analyse von Protein-Komplexen. Daher empfehlen wir, dass die Forscher mit dem Gebiet der strukturellen MS unbekannt, die Probenvorbereitung, Gerätekalibrierung und MS und Tandem-MS Optimierungsverfahren in Kirshenbaum et al verweisen. 2009 https://www.jove.com/index/details. stp? ID = 1954. In der Regel handelt es sich dabei Protokoll niedrigen mikromolaren Konzentrationen von komplexen (1-20 M) in einem flüchtigen Puffer wie Ammoniumacetat (0,005 bis 1 M, pH 6-8). Angesichts, dass 1-2 ul pro nanoflow Kapillare konsumiert werden, empfehlen wir 10-20 ul als ein minimales Volumen, der Optimierung des MS-Bedingungen zu ermöglichen.
Teil 1: Erwerb eines Ionen-Mobilitäts-Massenspektrometrie Spektrum
- Set dem Massenspektrometer auf den folgenden Betriebsarten: Mobility-TOF, positive Ionen Erwerb und V-Modus.
- Schalten Sie alle Gase (API, Trap und IMS). Wir verwenden N 2 für IM Trennung und Ar für die Trap / Transfer. Empfohlene Startwerte sind 1,5 ml / min für die Trap-Region und einem Gasfluss von 24 ml / min für die IMS-Gerät.
- Stellen Sie die m / z Erfassungsbereich. Für ein unbekanntes Protein-Komplex, empfehlen wir dem ersten Gebrauch eines breiten Massenbereich, die dann auf die gewünschten Werte reduziert werden kann. Parallel dazu stellen Sie die MS-Profil für maximalen Wirkungsgrad der Kraftübertragung. Für große Komplexe, sollte die Übernahme Masse von 1.000 eingestellt werden - 32.000 m / z, und die MS-Profil auf Auto. Andernfalls kann das Profil, nach der folgenden Tabelle werden:
m / z wohnen (%) Rampe (%) 960 10 20 3200 30 40 10667 - Überprüfen Sie die RF-Einstellung und, wenn nötig, anpassen, um geeignete Werte für große Proteinkomplexe, also:
Quelle Falle IMS Transfer RF Offset 450 380 380 380 RF-Gain 0 0 0 0 RF-Limit 450 380 380 380 - Bewerben Kapillar-Spannung (1,050-1,400 V) und geringer nanoflow Druck (0,00 bis 0,03 bar). Einmal sprühen eingeleitet wird, versuchen, die nanoflow Druck auf minimale Werte reduzieren. Darüber hinaus stellen Sie die Position der Kapillare, in Bezug auf den Kegel.
- Passen Sie die MS Aufnahmeparameter, um ein gut aufgelöstes MS-Spektrum zu erwerben: den Druckgradienten entlang des Instruments und die Sampling-Konus sowie die Extraktion Kegel, sollte bias, Trap und Transfer möglichen Einstellungen, die alle optimiert werden (ausführliche in der Verbindung gebracht werden JoVE Protokoll Kirshenbaum et al. 2009 https://www.jove.com/index/details.stp?ID=1954). Obwohl diese Parameter Sample-abhängig sind, die Bedingungen, die wir für den Erwerb von MS-Spektren von verschiedenen Ionen-Massen, von Peptid zu Protein-Komplexe verwendet, sind in Tabelle 1 dargestellt, (siehe auch Abb. 1).. Um die Aktivierung des Komplexes versuchen zu minimieren, schrittweise zu reduzieren (in Schritten von ~ 10 V) wird die Probe Kegel, Trap und Bias-Spannungen, ohne die Position des Peaks.
- Sobald eine optimale Massenspektrum erhalten wird die Drift Zeit-Profil angepasst werden sollten. Bei der Analyse von Protein-Baugruppen, optimale Bedingungen für die Masse und Mobilität Messungen sind oft unvereinbar, daher ist es wichtig, die richtige Balance zwischen den beiden zu schlagen. Insgesamt sollte der Ionen-Mobilitäts-Plot derart optimiert werden, dass die Spitzen über die gesamte Drift Zeitbereich verteilt sind, und die Peak-Profil ist glatt, nähert sich eine Gauß-Verteilung (Abb. 2A, 2B). Signifikante Peak-Asymmetrie kann zu einer schlechten Trennung von mehreren Konformationen bezogen werden.
- In der Regel lassen sich drei Parameter, T-Wellengeschwindigkeit, T-Wellenhöhe und IMS Gasdurchsatz abgestimmt, um die Mobilität Trennung zu optimieren. Die Erhöhung der T-Wellengeschwindigkeit wird erweitert die Driftzeit Distribution Profile, während erhöhte T-Wellenhöhe Werte, die sie verengen wird. Ebenso wird die Erhöhung der IMS Gasstrom die Drift Zeit-Profil in Richtung höherer Werte (die minimale IMS Gasfluss sollte 10 ml / min sein) zu verlagern. Wir schlagen vor, wobei zwei der drei verfügbaren Variablen fixiert und Optimierung der dritten bis zum IM-Spektrum ist gut gelöst (Abb. 2B). Zu diesem Zweck hat die T-Wellen-Geschwindigkeit und den Gasstrom auf 250 m / s und 24 ml / min, respectively. Dann, als Ausgangspunkt, setzen Sie die T-Wellenhöhe bis 3 V und stufenweise erhöhen sie in 1 V-Schritten. In der Regel werden größere Ionen erfordern höhere Wellenhöhen. Normalerweise gibt es keine Notwendigkeit, die IMS Druck ändern, aber wenn hohe Vorspannungen sind für Tuning benötigt wird, reduzieren die IMS-Gas wird eine Abnahme der Vorspannung Wert und damit eine Verringerung der Protein-Komplex Aktivierung zu ermöglichen. Insgesamt können maximal Auflösung von 10-12 t / At erreicht werden.
- Wenn die Bedingungen nicht optimiert sind (niedrige T-Wellen-Höhe oder hohe T-Wellen-Geschwindigkeit und / oder hohem Druck IM), werden die Ionen nicht durch die IM-Gerät effektiv durchziehen, und die Reise kann länger dauern als die Zeit für die nächste Ionen erforderlich Paket in die Mobilität Zelle freigesetzt werden. Als Ergebnis wird ein neuer Ionen-Paket von der Trap-Region freigegeben werden, bevor das vorherige Paket hat den Drücker Region geliefert worden. Dies wird zu einem "roll-over"-Effekt führen, in dem der Spitzenwert im ersten Teil der Driftzeit Spektrum beobachteten identisch mit der die Ionen in der tailing Rand (Abb. 2C) ist. Dieses Artefakt kann durch Erhöhung der T-Wellenhöhe und Verringerung der T-Wellengeschwindigkeit und IMS Druck beseitigt werden. Darüber hinaus kann der Trap-Release-Zeit eingestellt werden. Darüber hinaus ist es wichtig zu überprüfen, ob die Übertragung T-Wellenhöhe von mindestens 5 V. eingestellt ist um das Austreten von Ionen in Richtung der IMS-Zelle zu vermeiden, empfehlen wir, dass die Mobilität trap Höhe, in der Höchstgehalte (30 V) gehalten werden.
- Niedrige Geschwindigkeit und hoher Amplitude des Transfer-T-Wellen können dem "Plätschern" des Driftzeit Distribution Profile (Abb. 2D) führen. Dieses Artefakt tritt auf, wenn die Mobilität Trennung von Ionen (Ionen Ankunft / Driftzeit) ist nicht durch die Transfer-und ToF Regionen aufrechterhalten, durch partielle Synchronisation zwischen den Drücker Frequenz und die Übertragung der T-Welle Geschwindigkeit. Um diesen Effekt zu eliminieren, entweder die Schieber Zeit oder die Übertragung der T-Welle Geschwindigkeit angepasst werden sollte. Da die Schieber-Frequenz, um die Masse Bereich verbunden ist, kann dieses Artefakt wieder, wenn dieser Parameter geändert wird. T-Wellenhöhe übt einen geringen Einfluss, obwohl die Reduktion auch helfen können, um Wellen zu eliminieren.
- Sobald die oben genannten Parameter optimiert sind, können die IM-MS-Daten erworben werden.
Teil 2: Screening experimentellen Bedingungen für die Mobilität Messungen der nativen Strukturen zu gewährleisten
Um hochaufgelöste MS Gipfel zu erreichen, sind Protein-Komplexe oft im Massenspektrometer aktiviert, um das Strippen von Restwasser und Puffer Komponenten 11 zu fördern. Allerdings, wenn die Aktivierungsenergie jenseits eines Schwellenwertes erhöht wird, können partielle Entfaltung induzierte Bildung von mehreren Zwischenstufen 12, die sich kaum für die native, Lösungs-Struktur (Abb. 3A-C) entsprechen werden. Als Ergebnis kann der Driftzeit Gipfel verschoben und verbreitert werden, was die heterogene Population von ungefalteten Strukturen.
Um Driftzeit Daten im Einklang mit flüssiger Phase Strukturen zu erhalten, ist es wichtig, sorgfältig kontrollieren die Spannungen für die Beschleunigung von Ionen verwendet werden, vor IM-Trennung. Darüber hinaus für hohe MS-Auflösung ist es vorzuziehen, um die Übertragung statt der Trap-Spannung zu erhöhen. Da die IM-Gerät positioniert ist, zuerst, gefolgt von der Transfer-Region und der TOF-Analysator, daher folgt die Aktivierung der IM-Messung und die Ionen bleibt unberührt, während die MS Genauigkeit erhöht werden kann.
Um zu überprüfen, Datenerfassung ist unter den Bedingungen, dass die native Struktur des Komplexes zu erhalten durchgeführt, ist es empfehlenswert, die Daten über eine Reihe von experimentellen und Lösung Bedingungen, anstatt nach einem einzigen, optimierten Parameter eingestellt aufgezeichnet werden:
- Erhöhen Kapillare und Konusspannungen stufenweise, während die Überwachung der Auswirkungen auf die Driftzeit Spektrum.
- Wie in Schritt 1, erhöhen Sie die Trap-Kollision Spannung stufenweise, und Messdaten mit 10 V Abständen.
- Zur Identifizierung der ungefalteten Konformationen und Bewertung der erfassten Daten, manuell auslösen Dissoziation des Protein-Komplex durch Titration der Probe mit Essigsäure über einen pH-Bereich von 2-7, und notieren Sie die Daten (Abb. 3B).
Teil 3: Korrelation zwischen Driftzeit Werte und Querschnittsflächen
Im Gegensatz zu herkömmlichen IM-Messungen, in denen die gemessenen Driftzeit Werte linear auf Ω verwandt sind, in der T-Welle IMS-System ist die Querschnittsfläche durch eine Kalibrierung Ansatz definiert. Statt also eine absolute Messung ist eine relative exponentiellen Zusammenhang zwischen der gemessenen Drift Zeiten und Ω 1,13 generiert: 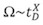
wo t D ist der gemessene Driftzeit und X ist der Anteil Konstante, die aus einer Eichkurve entnommen werden können. Die Kalibrierung wird durchgeführted durch die Messung der Drift der Ionen mit bekannten Ω (gemessen von herkömmlichen IM-Experimente).
- Drift Zeitmessungen kalibriert denaturierte Proteine Pferde Cytochrom C, Pferd Herzen Myoglobin und bovine Ubiquitin mit bekannten Stoßquerschnitte. Zu diesem Zweck Lösungen von 10 uM in 49/49/2, Volumen-Verhältnis, sollte Wasser / Methanol / Essigsäure (verwendeten Reagenzien sind in Tabelle 3 dargestellt) werden.
- Erwerben Sie IM-MS-Daten für die Kalibrierlösung Proteine unter genau den gleichen Instrument Bedingungen für die Ziel-Protein-oder Protein-Komplex verwendet:
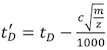 (Teil 1). Alle Spannungen und Druck-Werte sollten identisch sein, um die IM Trennung Einstellungen erhalten bleiben.
(Teil 1). Alle Spannungen und Druck-Werte sollten identisch sein, um die IM Trennung Einstellungen erhalten bleiben. - Für jeden Ladezustand der Kalibrierlösung Proteine extrahieren Sie die experimentelle Drift Zeitwert (t D) (beschrieben in Teil 4).
- Korrigieren Sie jede der Kalibrierlösung Drift Zeiten (t D) (Tabelle 2) mit folgender Gleichung:
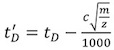 , Wobei m / z ist die Masse-Ladungs-Verhältnis der beobachteten Ionen und c ist die Enhanced Duty Cycle (EDC) Verzögerung Koeffizient 1. Sein Wert, typischerweise zwischen 1,4 und 1,6, ist Instrument abhängig. Acquisition Settings | | Erwerb Registerkarte Setup Die EDC-Wert innerhalb des Systems angezeigt.
, Wobei m / z ist die Masse-Ladungs-Verhältnis der beobachteten Ionen und c ist die Enhanced Duty Cycle (EDC) Verzögerung Koeffizient 1. Sein Wert, typischerweise zwischen 1,4 und 1,6, ist Instrument abhängig. Acquisition Settings | | Erwerb Registerkarte Setup Die EDC-Wert innerhalb des Systems angezeigt. - Mit Prof. David Clemmer Cross-Section-Datenbank:
http://www.indiana.edu/ ~ clemmer / Forschung / cross% 20section% 20database/Proteins/protein_cs.htm 14 richtige jedem der Kalibrierlösung Querschnitt für beide Ionen-Ladezustand und reduzierten Masse.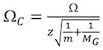
, Wo Ω C ist die korrigierte Querschnitt Ω der Literatur ist ein Querschnitt, z die Ionenladung Zustand ist, ist m das Molekulargewicht des Kalibrier-Ion und M G wird das Molekulargewicht des IM Hintergrund Gas (in der Regel N 2). - Plot In (t D) gegen In (Ω C) (Abb. 4A).
- Die resultierende Kurve entspricht der folgenden Gleichung:
 Die Parameter X und A kann durch den Einbau der Plot um eine lineare Beziehung extrahiert werden. Die Steigung X entspricht der exponentiellen Anteil Faktor und A entspricht dem Fit-bestimmt konstant.
Die Parameter X und A kann durch den Einbau der Plot um eine lineare Beziehung extrahiert werden. Die Steigung X entspricht der exponentiellen Anteil Faktor und A entspricht dem Fit-bestimmt konstant. - Berechnen Sie die fit Korrelationskoeffizient r 2, mit dem Pearson-Gleichung:
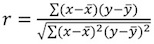 . Akzeptable Werte für r 2 sind größer als 0,95 (Abb. 4B). Ein niedriger Korrelationskoeffizient Wert kann darauf zurückzuführen sein:
. Akzeptable Werte für r 2 sind größer als 0,95 (Abb. 4B). Ein niedriger Korrelationskoeffizient Wert kann darauf zurückzuführen sein: - Unvollständige Entfaltung des Proteins Kalibrierstandards. Dies führt zu Spitzenzeiten Verbreiterung aufgrund der heterogenen Versammlung Zwischenzustände.
- Nach unserer Erfahrung kann eine gealterte Probe verschlechtern sich die IM-Spektren.
- Unterschiedliche experimentellen Bedingungen für die verschiedenen Kalibrierlösung Proteinen verwendet. In diesem Fall Plotten der Daten für jedes Protein getrennt zu erzeugen diverse Korrelationskoeffizienten, obwohl jeder von ihnen sollte höher sein als 0,95.
- Verrauschten Daten und falsche Glätten und Zentrierung der Driftzeit Verteilung.
- Fehler in der Berechnung.
- Recorrect der Kalibrierlösung Driftzeit anhand der ermittelten exponentiellen Faktor X, der in Schritt 7 abgeleitet:
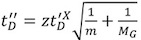
- Als Validierung Schritt Replot Ω C vs
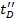 und definieren Sie den Korrelationskoeffizienten. Höhere Werte als 0,95 zu erwarten sind.
und definieren Sie den Korrelationskoeffizienten. Höhere Werte als 0,95 zu erwarten sind. - Nach dem Verfahren in Schritt 4 beschrieben, korrigieren Sie die gemessenen Driftzeit der Ziel-oder Protein-Komplex:
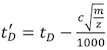
- Kalibrieren Sie die Driftzeit des Zielproteins / Protein-Komplex mit dem exponentiellen Faktor, X, in Step 7 definiert:
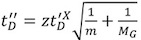
- Berechnen Ω des Zielproteins / Protein-Komplex mit dem fit-ermittelte Konstante, A, in Step 7 definiert:
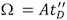 .
. - Für jeden experimentellen Bedingungen, Schritte 2 bis 13 zu wiederholen. Bei der Definition der Querschnittsfläche des unbekannten Protein-oder Protein-Komplex, empfehlen wir, dass jedes Experiment mindestens dreimal wiederholt werden, und die Standardabweichung dieser drei Messungen bestimmt.
Teil 4: Definieren von Drift Zeitwerte
Erforderliche Software: MassLynx und Driftscope (Waters).
- Öffnen Sie die IM-MS-Spektrum mit dem Driftscope Software.
- Aus dem Hauptmenü wählen Sie View und deaktivieren Chromatogramm, Drift Time and Spectrum (optional), so dass nur aktiv, die 2D-Karte angezeigt werden Driftzeit vs m / z
- Von der Menüleiste anzeigen | Optionen | Display Editor-Panel und stellen Intensity Schwellenwerte, die Hintergrundgeräusche zu minimieren (in den meisten Fällen können diese Einstellung als Min = 30-40% und Max = 100% zählt definiert werden).
- Wählen Sie Anzeigen | 2D-Karte Intensity Scale, und drei Optionen angezeigt: Linear Scale, Log-Skala und Quadratwurzel-Skala. Eine Auswahl von Log-Skala (logarithmische Datentransformation) wird die Intensität Farbcode zu komprimieren, und ermöglichen das gleichzeitige Auftreten einer Vielzahl von Intensitäten (im Gegensatz zu den linearen und Quadratwurzel-Optionen gegenüber, bei denen nur die intensivsten Banden sichtbar werden) .
- Von der Symbolleiste Panel Verwenden Selection Tool-Taste. Diese Option aktiviert die verschiedenen Auswahlmöglichkeiten, und ermöglichen die Auswahl der betreffenden Region innerhalb des Spektrums. Die genaueste Werkzeug aktivieren Region of Interest Auswahl, mit denen ein Rahmen um die Region von Interesse gezogen werden, kann dadurch ohne alle unnötigen Daten und Geräuschspitzen. Ebenso sind die Orthogonal und Band Selection-Optionen sinnvoll, wenn die Region von Interesse nicht durch redundante Gipfeln umgeben.
- Nachdem die Region von Interesse ausgewählt ist, verwenden Sie die Accept Aktuelle Auswahl Befehl, um unnötige Informationen zu entfernen.
- Exportieren Sie die Daten zu MassLynx, unter Beibehaltung der Driftzeit Informationen.
- Innerhalb MassLynx, öffnen Sie das Chromatogramm der gespeicherten Driftzeit Spektrum, und kombinieren Sie die Zeitintervalle. Die entsprechende Massenspektrum wird automatisch geöffnet.
- Übernehmen Sie die Glättung Funktion definieren Sie die Fenstergröße und die Anzahl der glatten Parameter (insbesondere sollte für jedes Spektrum abgestimmt werden, mit minimalen Werten).
- Bewerben Baseline Subtraktion, wenn nötig.
- Zentrum des Spektrums und messen die Masse, um Protein Identität und Masse Genauigkeit zu überprüfen.
- Für jeden Ladezustand, kombinieren die m / z-Bereich. Die entsprechenden Driftzeit Spektrum wird automatisch angezeigt. Glatte und in der Mitte der Drift Zeit-Profil, und definieren die Drift Zeitwert durch die Angabe des Schwerpunktes der einzelnen Peaks.
Teil 5: Repräsentative Ergebnisse

Abbildung 1. Schematische Darstellung der SYNAPT HDMS Instrument unter Angabe der wichtigsten einstellbaren Parameter der IMS-MS Akquisition. Experimentelle Parameter für die IM-MS-Messungen eingesetzt werden entsprechend ihrer Position innerhalb des Gerätes gekennzeichnet. Der Ionenstrahl wird in roter Farbe, und der Druck in der jeweiligen Region bezeichnet wird mit einem Farbcode. Das Panel auf der Unterseite zeigt die Potentialgradienten entlang des Instruments und die möglichen Unterschiede der Definition der Trap-und Transfer Kollisionsenergien sowie die Bias-Potential. Alle Potentiale read-Rücken sind die Static Offset-Spannung in der Regel bis 120V eingestellt ist referenziert.

Abbildung 2. Ion Mobilität Ankunftszeit Verteilungen für die Gβυ Protein.
A. Eine hohe T-Wellen-Geschwindigkeit führt zu einer engen Verteilung der Drift Zeit-Profil. Der Plot zeigt die Ankunft zeitliche Verteilung der 16 + (rot), 15 + (grün), 14 + (blau) und 13 + (magenta) Ladezustände, sowie die insgesamt Driftzeit Profil (in schwarz) des G βυ Protein.
B. Eine optimierte Driftzeit Spektrum mit einer glatten Gaussian Peakform. Ähnliche farbige Etiketten wie in A.
C. A 'roll-over "-Effekt, die auftritt, wenn die Zeit für Ionen getroffen werden, um die Mobilität Zelle durchqueren langsamer als das Intervall zwischen den Injektionen von neuen Ionen-Pakete in das Gerät. Als Ergebnis erscheint die erweiterte Driftzeit Höchststand Anfang des Spektrums. Dieser Effekt kann durch Erhöhung der T-Wellenhöhe und Verringerung der T-Wellengeschwindigkeit und IMS Druck beseitigt werden.
D. Artificial 'Wellen' auf, wenn der Transfer-T-Wellengeschwindigkeit und Drücker Frequenz teilweise synchronisiert sind. Dieser Effekt kann durch Verstellen der Schieber entweder Frequenz-oder Transfer-T-Wellengeschwindigkeit überwunden werden.
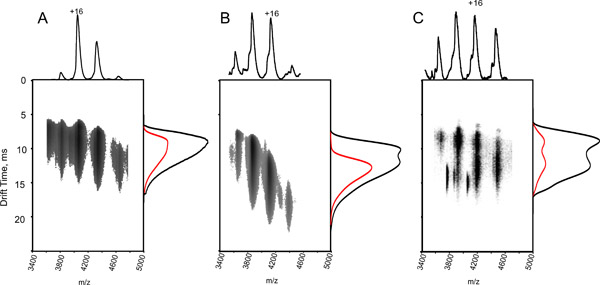
Abbildung 3. Die Wirkung von Ionen-Aktivierung und teilweise denaturierenden Bedingungen auf IM-MS-Spektren von Hämoglobin. Plot von Drift Zeit gegen m / z für das tetramere Hämoglobin-Komplex, mit einer wässrigen Lösung von 10 mM Ammoniumacetat (pH = 7.6) (A, C) und die Zugabe von 0,1% Essigsäure (B). Erfassten Daten mit Trap-Kollisionsenergie Spannung von 13 V (A, B) und 35 V (C) Obwohl in allen drei Platten Massenspektrum (projiziert auf der Oberseite) sieht ähnlich aus, mit einem tetrameren kostenlos Serie bei 4.000 m / z zentriert, die Driftzeit Profil (projiziert auf den Seiten) ist anders (insgesamt Driftzeit Verteilungin schwarz, und die 16 +-Profil ist in rot). Je länger Driftzeit der teildenaturierten Probe, in B erhalten, und die Gas-Phase aktiviert Ionen in C erhalten, ist bezeichnend für eine gewisse Entfaltung. Diese Beobachtung zeigt, dass, obwohl die gemessene Masse entspricht einer intakten Komplex, dessen Struktur in Lösung gestört wird. Als Folge davon ist eine sorgfältige Kontrolle der experimentellen Bedingungen erforderlich.
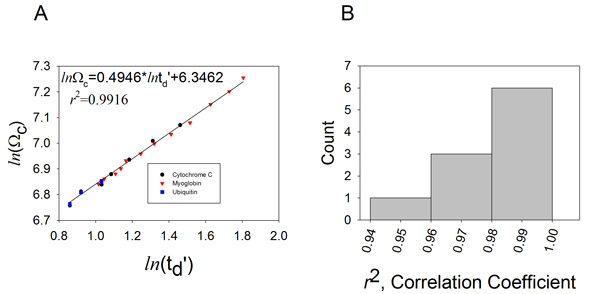
Abbildung 4. Durch die Erzeugung einer Kalibrierungskurve, Drift Zeitmessungen und Stoßquerschnitte korreliert werden kann.
A. Gemessen Driftzeit Werte der mehrfachen Ladungszustände der Pferde Cytochrom C (Kreise), Pferd Herzen Myoglobin (Dreiecke) und Rinder-Ubiquitin (Quadrate) wurden gegen Literatur Ω-Werte für beide Ionen-Ladezustand und reduzierte Masse korrigiert dargestellt. Die Passung ergibt eine lineare Funktion entspricht: ln (Ω C) = XLN (t D ') + A. Der ermittelte exponentielle Faktor (X), fit-ermittelte Konstante (A) und Korrelationskoeffizient werden auf dem Grundstück für Daten mit einer T-Wellen-Geschwindigkeit von 350 m / s und eine statische Wellenhöhe von 11 V. B erworben angezeigt. Ein Histogramm der Korrelationskoeffizient Ausschüttungen aus 10 aufeinander folgenden Kalibrierung Experimenten.
| Protein sample / Technische Parameter | GluFibrino- Peptid Monomer 1,6 kDa | Myoglobin Monomer 17 kDa | Hämoglobin Tetramer 67 kDa | Transferrin Monomer 80 kDa | GroEL 14-mer 801 kDa |
| Sichern Druck, mBar | 4,4 | 5,0 | 5,1 | 5,1 | 6,5 |
| Trap-Druck, mBar | 1.6x10 -2 | 2.4x10 -2 | 2.4x10 -2 | 2.6x10 -2 | 2.8x10 -2 |
| IMS Druck, mBar | 4.4x10 -1 | 4.4x10 -1 | 4.4x10 -1 | 4.4x10 -1 | 4.2x10 -1 |
| Sampling Kegel, V | 46 | 80 | 80 | 80 | 118 |
| Extraction Kegel, V | 1,7 | 1 | 1 | 1 | 3 |
| Bias-Spannung, V | 20 | 20 | 25 | 25 | 50 |
| Trap-Kollision Energie, V | 20 | 15 | 15 | 15 | 80 |
| Transfer Aufprallenergie, V | 5 | 12 | 12 | 12 | 15 |
Tabelle 1. Versuchsbedingungen für die Analyse von Makromolekülen.
| Standard-Protein | Molekulare Masse (m) | Gebühren (z) | m / z | Collision Cross-Section (in 2) |
| Cytochrom C | 12213 | 10 | 1222,3 | 2226 |
| 11 | 1111,3 | 2303 | ||
| 12 | 1018,8 | 2335 | ||
| 13 | 940,5 | 2391 | ||
| 14 | 873,4 | 2473 | ||
| 15 | 815,2 | 2579 | ||
| 16 | 764,3 | 2679 | ||
| 17 | 719,4 | 2723 | ||
| 18 | 679,5 | 2766 | ||
| Myoglobin | 16952 | 11 | 1542,1 | 2942 |
| 12 | 1413,7 | 3044 | ||
| 13 | 1305,0 | 3136 | ||
| 14 | 1211,9 | 3143 | ||
| 15 | 1131,1 | 3230 | ||
| 16 | 1060,5 | 3313 | ||
| 17 | 998,2 | 3384 | ||
| 18 | 942,8 | 3489 | ||
| 19 | 893,2 | 3570 | ||
| 20 | 848,6 | 3682 | ||
| 21 | 808,2 | 3792 | ||
| 22 | 771,6 | 3815 | ||
| Ubiquitin | 8565 | 8 | 1071,6 | 1442 |
| 8 | 1071,6 | 1622 | ||
| 9 | 952,7 | 1649 | ||
| 10 | 857,5 | 1732 | ||
| 11 | 779,6 | 1802 | ||
Tabelle 2. Kalibrierlösung Proteine und ihre Stoßquerschnitte Werte durch konventionelle IMS Messungen 14 bestimmt.
| Devices | Firma | Katalog-Nummer |
| SYNAPT HDMS-32K-RF-Generator | Waters Ltd | |
| P-97 Flaming-Brown Mikropipette puller | Sutter Instruments | P-97 |
| Sputter-Coater | Electron Microscopy Sciences | EMS550 |
| Binokulares Mikroskop | Nikon | |
| Reagenzien | Firma | Katalog-Nummer |
| Ammoniumacetat | Sigma-Aldrich | Sigma, A2706 |
| CsI 99,999% | Sigma-Aldrich | Aldrich, 203033 |
| Methanol | Sigma-Aldrich | Fluka, 34966 |
| Essigsäure | Fisher Scientific | AC12404 |
| Equine Myoglobin (aus Pferdeherz) | Sigma-Aldrich | M1882 |
| Equine Cytochrom c (aus Pferdeherz) | Sigma-Aldrich | C-2506 |
| Bovine Ubiquitin (von roten Blutkörperchen) | Sigma-Aldrich | U6253 |
| Hämoglobin | Sigma-Aldrich | H2625 |
| Gas | Kommentare | |
| Stickstoff, 99,999% rein | 8 kubische Meter Zylinder | |
| Argon, 99,999% rein | 8,8 Kubikmeter meterscylinder | |
Tabelle 3. Reagenzien und Ausrüstung.
Diskussion
Die hier beschriebene Protokoll ermöglicht es, die Kollision Querschnitt von Proteinen oder Protein-Komplexen mit einem unbekannten dreidimensionale Struktur zu definieren, mit dem Ziel der Bereitstellung von Informationen über ihre gesamte Form, Untereinheit Verpackung und Topologie. Zu diesem Zweck einmal Stoßquerschnitt Werte sind dargestellt, ist es notwendig, diese Werte zu strukturellen Details zu konvertieren. Dieser Prozess erfordert zusätzliche experimentelle Bemühungen sowie computergestützte Analyse, die kurz diskutiert werden unten.
Zunächst ist es empfehlenswert, Proteine oder Protein-Komplexen mit bekannten Strukturen zu analysieren. Diese Messungen können eine nützliche Kontrolle der Qualität der Methodik und Genauigkeit Beurteilung des Erwerbs Parameter durch den Vergleich theoretischer und gemessener Ω-Werte zu ermöglichen. Die theoretische Querschnittsflächen kann aus der Kristallstruktur berechnet werden koordiniert mit der MOBCAL 15,16 Software, die eine Open-Source-basierte Software ermöglicht FORTRAN Code-Bearbeitung nach dem Operator benötigt wird. Für den Betrieb solcher Berechnungen ist es erforderlich, das Programm so, dass die Zahl der iterative Berechnungen pro Input-Struktur durchgeführt erhöht wird und dass die Akten koordinieren, die große Zahl der Atome sind 1 akzeptiert zu ändern.
Ein IM-MS-Strategie für die Definition von topologischen Anordnung der Untereinheiten innerhalb Mehrkomponenten-Baugruppen wurde vor kurzem 4,6 vorgeschlagen. Das Verfahren beinhaltet die Überwachung der Dissoziationspfade von Protein-Baugruppen zu kleineren Komponenten. Diese Dissoziation wird durch gezielte Einstellung der Lösungsphase Bedingungen, die Anlass zu einer Verteilung von Subkomplexe reflektiert die "Bausteine" der Baugruppen erreicht. Die gleichzeitige Messung von Ω-Werte sowohl des intakten komplexen Auf-und Abbau-Produkte erzeugt strukturelle Beschränkungen, die dann für die Berechnung der topologischen Modelle der Protein-Komplexen eingesetzt. Die Grundannahme dieser Methode ist, dass die erzeugten Subkomplexe ihrer Muttersprache wie Bestätigungen erhalten, und zwar neuere Studien haben gezeigt, dass die Lösung Struktur der Demontage Produkte erhalten bleibt und keine größeren Umlagerung entweder in Lösung oder Gasphase haben 4,6 aufgetreten.
Der letzte Schritt bei der Zuordnung der Quartärstruktur auf Gas-Phase-Protein-Komplex-Ionen passt die Kollision Querschnitt Werte computergenerierte Modelle. Modeling Ansätze eingesetzt, um die verschiedenen möglichen topologischen Anordnung der Untereinheiten und ihre in-silico-Ω-Werte berechnet und verglichen mit den experimentellen erkunden. Derzeit ist nur ein paar computergestützte Ansätze verwendet werden, wie die spheretype grobkörnigen Methode, die den Durchmesser der Untereinheiten 1,8 annähert. Im Großen und Ganzen ist dieses Feld noch in den frühen Jahren und ein weiterer Ausbau erforderlich, um diesen Ansatz generisch und für eine breite Palette von Komplexen.
Offenlegungen
Danksagungen
Die Autoren danken der Sharon Gruppenmitglieder für die kritische Durchsicht und für ihre Beiträge zu dem Manuskript. Wir sind dankbar für die Unterstützung der Morasha und Bikura Programme, die Israel Science Foundation (Grant Nr. 1823-1807 und 378/08), das Josef Cohn Minerva Center for Biomembrane Research, die Chais Familie Fellows Program für New Scientists, die Abraham und Sonia Rochlin Stiftung, die Familie Wolfson Charitable Trust, die Helen und Milton A. Kimmelman Zentrums für Biomolekulare Struktur und Montage; dem Nachlass von Shlomo und Sabine Beirzwinsky; Meil de Botton Aynsley, und Karen Siem, UK.
Referenzen
- Ruotolo, B. T. Ion mobility-mass spectrometry analysis of large proteincomplexes. Nat Protoc. 3 (7), 1139-1152 (2008).
- Scarff, C. A., Thalassinos, K., Hilton, G. R., Scrivens, J. H. Travelling wave ion mobility mass spectrometry studies of protein structure: biological significance and comparison with X-ray crystallography and nuclear magnetic resonance spectroscopy measurements. Rapid Commun Mass Spectrom. 22 (20), 3297-3304 (2008).
- Smith, D. P. Deciphering drift time measurements from travelling wave ion mobility spectrometry-mass spectrometry studies. Eur J Mass Spectrom (Chichester, Eng). 15 (2), 113-130 (2009).
- Leary, J. A. Methodology for measuring conformation of solvent-disrupted protein subunits using T-WAVE ion mobility MS: an investigation into eukaryotic initiation factors. J Am Soc Mass Spectrom. 20 (9), 1699-1706 (2009).
- Lorenzen, K. Determination of stoichiometry and conformational changes in the first step of the P22 tail assembly. J Mol Biol. 379 (2), 385-396 (2008).
- Pukala, T. L. Subunit architecture of multiprotein assemblies determined using restraints from gas-phase measurements. Structure. 17 (9), 1235-1243 (2009).
- van Duijn, E. Chaperonin complexes monitored by ion mobility mass spectrometry. J Am Chem Soc. 131 (4), 1452-1459 (2009).
- Ruotolo, B. T. Evidence for macromolecular protein rings in the absence of bulk water. Science. 310 (5754), 1658-1661 (2005).
- Ruotolo, B. T., Robinson, C. V. Aspects of native proteins are retained in vacuum. Curr Opin Chem Biol. 10 (5), 402-408 (2006).
- Giles, K. Applications of a travelling wave-based radio-frequency-only stacked ring ion guide. Rapid Commun Mass Spectrom. 18 (20), 2401-2414 (2004).
- McKay, A. R., Ruotolo, B. T., Ilag, L. L., Robinson, C. V. Mass measurements of increased accuracy resolve heterogeneous populations of intact ribosomes. J Am Chem Soc. 128 (35), 11433-11442 (2006).
- Ruotolo, B. T. Ion mobility-mass spectrometry reveals long-lived, unfolded intermediates in the dissociation of protein complexes. Angew Chem Int Ed Engl. 46 (42), 8001-8004 (2007).
- Morton, V. L., Stockley, P. G., Stonehouse, N. J., Ashcroft, A. E. Insights into virus capsid assembly from non-covalent mass spectrometry. Mass Spectrom Rev. 27 (6), 575-595 (2008).
- Valentine, S. J., Counterman, A. E., Clemmer, D. E. A database of 660 peptide ion cross sections: use of intrinsic size parameters for bona fide predictions of cross sections. J Am Soc Mass Spectrom. 10 (11), 1188-1211 (1999).
- Mesleh, M. F. Structural information from ion mobility measurements: effects of the long-range potential. J Phys Chem. 100, 16082-16086 (1996).
- Shvartsburg, A. A., Jarrold, M. F. An exact hard-spheres scattering model for the mobilities of polyatomic ions. Chem Phys Lett. 261, 86-91 (1996).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten