Method Article
T-wave Ion Mobility Spectrometry massa: Basic Procedimentos para Análise Experimental Protein Complex
Neste Artigo
Resumo
Ion espectrometria de mobilidade de massa é uma tecnologia emergente fase gasosa que separa íons, com base em sua colisão transversal e massa. O método fornece informações tridimensionais sobre a topologia geral e forma de complexos de proteínas. Aqui, descrevemos um procedimento básico para configuração e otimização de instrumento, calibração de vezes drift, e interpretação dos dados.
Resumo
De mobilidade iônica (IM) é um método que mede o tempo necessário para um íon de viajar através de uma célula pressurizada sob a influência de um campo elétrico fraco. A velocidade pela qual os íons atravessam a região drift depende de seu tamanho: íons grandes vão experimentar um maior número de colisões com o gás de fundo inerte (geralmente N 2) e, assim, viajar mais devagar através do dispositivo de mensagens instantâneas do que os íons que compõem um menor cross-section. Em geral, o tempo que leva para os íons de migrar embora a fase de gás denso separa-los, de acordo com sua colisão transversal (Ω).
Recentemente, a espectrometria de IM foi acoplado com espectrometria de massa e uma viagem-wave (onda T) Synapt de mobilidade iônica espectrômetro de massa (IM-MS) foi lançado. Integração de espectrometria de massa com mobilidade de íons permite uma dimensão extra de separação e definição da amostra, produzindo um espectro tridimensional (em massa para o tempo de carga, intensidade e drift). Esta técnica permite a separação de sobreposição espectral a diminuir, e permite a resolução de complexos heterogêneos com massa muito semelhante, ou massa-carga índices, mas os tempos drift diferentes. Além disso, as medidas de tempo deriva fornecer uma importante camada de informações estruturais, como Ω está relacionado com a forma geral e topologia do íon. A correlação entre os valores medidos drift tempo e Ω é calculada usando uma curva de calibração gerada a partir de proteínas de calibração definido com seções transversais 1.
O poder da abordagem IM-MS reside na sua capacidade de definir a embalagem subunidade e forma geral das assembleias de proteína em concentrações micromolar, e quase fisiológica condições 1. Vários estudos recentes IM de ambas as proteínas individuais 2,3 e complexos não-covalente de proteínas 4-9, conseguiu demonstrar que a estrutura da proteína quaternária é mantida na fase gasosa, e destacou o potencial dessa abordagem no estudo dos conjuntos de proteínas de geometria desconhecida . Aqui, fornecemos uma descrição detalhada do IMS-MS análise de complexos de proteínas usando o Synapt (Quadrupole-Ion Mobility-Time-of-Flight) HDMS instrumento (Waters Ltd; o único instrumento IM-MS comerciais disponíveis atualmente) 10. Descrevemos os passos básicos de otimização, a calibração de colisão seções transversais, e métodos de processamento de dados e interpretação. A etapa final do protocolo discute métodos de cálculo dos valores teóricos Ω. No geral, o protocolo não tenta cobrir todos os aspectos do IM-MS caracterização de conjuntos de proteínas, mas sim, seu objetivo é apresentar os aspectos práticos do método para novos pesquisadores no campo.
Protocolo
O procedimento que descrevemos se concentra exclusivamente em IM-MS a análise dos complexos de proteína. Portanto, sugerimos que os pesquisadores não familiarizados com o campo da MS estruturais referem-se os passos para preparação de amostras, calibração de instrumentos e procedimentos de MS e MS em tandem de otimização descrito na Kirshenbaum et al. Http://www.jove.com/index/details 2009. stp? ID = 1954. Em geral, este protocolo envolve baixas concentrações micromolar do complexo (1-20 mM) em tampão volátil como o acetato de amónio (0,005-1 M, pH 6-8). Dado que 1-2 mL são consumidos por nanofluxo capilar, sugerimos 10-20 mL como um volume mínimo, para permitir a otimização das condições de MS.
Parte 1: A aquisição de um espectro de espectrometria de mobilidade iônica em massa
- Definir o espectrômetro de massa sobre os seguintes modos de operação: Mobilidade TOF-, a aquisição de íons positivos, e V-mode.
- Ligue todos os gases (API, Trap e IMS). Usamos N 2 para a separação de IM, e Ar para a armadilha / Transfer. Recomenda valores iniciais são de 1,5 ml / min para a região Trap, e um fluxo de gás de 24 ml / min para o dispositivo IMS.
- Definir o intervalo de aquisição de m / z. Para um complexo de proteínas desconhecidas, sugerimos o uso inicial de uma ampla gama de massas, que pode então ser reduzida para os valores desejados. Em paralelo, ajustar o perfil de MS para a eficiência máxima de transmissão. Para complexos de grande porte, a faixa de massa aquisição deve ser ajustado de 1,000 - 32,000 m / z, eo perfil de MS para Auto. Caso contrário, o perfil pode ser definido, de acordo com a seguinte tabela:
m / z habitam (%) rampa (%) 960 10 20 3200 30 40 10667 - Verifique a configuração do RF e, se necessário, ajustar para valores apropriados para grandes complexos de proteínas, assim:
Fonte Armadilha IMS Transferência RF offset 450 380 380 380 Ganho RF 0 0 0 0 Limite de RF 450 380 380 380 - Aplique a tensão capilar (1,050-1,400 V) e pressão nanofluxo baixa (,00-,03 bar). Uma vez que spray é iniciado, tentar reduzir a pressão nanofluxo para um valor mínimo. Além disso, ajustar a posição do capilar, com respeito ao cone.
- Ajustar os parâmetros de aquisição MS, para adquirir um bem-resolvido espectro MS: o gradiente de pressão ao longo do instrumento e do cone de amostragem, bem como a extração de cone, Trap viés, e Transferência de configurações possíveis, todos devem ser otimizados (detalhados no associada JOVE protocolo Kirshenbaum et al. https://www.jove.com/index/details.stp?ID=1954 2009). Embora estes parâmetros são dependentes da amostra, as condições foram utilizados para a aquisição de MS espectros de massas de íons diferentes, de peptídeo a complexos de proteína, são descritos na Tabela 1, (ver também Fig. 1.). Para minimizar a ativação do complexo tentar reduzir gradualmente (em passos de ~ 10 V) tensões na amostra cone Trap, e viés sem alterar a posição do pico.
- Uma vez que um espectro de massa ideal é obtido o perfil de tempo drift deve ser ajustado. Ao analisar conjuntos de proteínas, as condições ideais para ambas as massas e as medidas de mobilidade são muitas vezes incompatíveis, por isso, é importante encontrar o equilíbrio adequado entre os dois. No geral, o enredo mobilidade iônica deve ser otimizado de tal forma que os picos são distribuídos em todo o intervalo de tempo deriva, eo perfil de pico é suave, aproximando-se uma distribuição gaussiana (Fig. 2A, 2B). Pico significativo assimetria pode estar relacionada à separação pobre de conformações múltiplas.
- Como regra geral, três parâmetros, onda T, velocidade da onda T altura e IMS taxa de fluxo de gás pode ser ajustado para otimizar a separação de mobilidade. Aumentando a velocidade da onda T vai alargar o drift perfil de distribuição do tempo, enquanto aumentou os valores da onda T altura vai reduzi-lo. Da mesma forma, aumentando o fluxo de gás IMS vai mudar o perfil do tempo deriva em direção a valores mais elevados (o fluxo de gás mínima IMS deverá ser de 10 ml / min). Sugerimos deixando duas das três variáveis disponíveis fixo, e otimizar o terceiro até o espectro de IM é bem-resolvida (Fig. 2B). Para este fim, defina a velocidade da onda T eo fluxo de gás a 250 m / s e 24 ml / min, rerespectivamente. Então, como ponto de partida, definir a altura da onda T a 3 V e, de uma forma gradual, aumentá-lo em incrementos de 1 V. Em geral, os íons maiores exigirá maiores alturas de onda. Normalmente, não há necessidade de modificar a pressão IMS, no entanto, quando tensões viés de alta são necessárias para o ajuste, reduzindo o gás IMS permitirá uma diminuição no valor da tensão de polarização e, conseqüentemente, uma redução na ativação da proteína complexa. No geral, resolução máxima de 10-12 t / Dt pode ser alcançado.
- Quando as condições não são otimizados (altura da onda T de baixa ou a velocidade da onda T de alta e / ou pressão alta IM), os íons não vai atravessar o dispositivo IM efetivamente, e sua viagem pode demorar mais que o tempo necessário para o íon próxima pacote a ser lançado para dentro da célula de mobilidade. Como resultado, um pacote de íons novo será lançado a partir da região Armadilha antes do pacote anterior tenha sido entregue para a região empurrador. Isto levará a um "roll-over" efeito, em que o pico observado na primeira parte do espectro de tempo deriva é idêntica à dos íons na borda de rejeitos (Fig. 2C). Este artefato pode ser eliminado, aumentando a altura da onda T, e diminuindo a onda T velocidade e pressão IMS. Além disso, o tempo de liberação armadilha pode ser ajustado. Além disso, é importante para validar que a transferência da altura da onda T é definido, pelo menos, 5 V. Para evitar vazamento de íons para a célula de IMS, recomendamos que a altura armadilha mobilidade ser mantida a níveis máximos (30 V).
- Baixa velocidade e alta amplitude de Transferência de ondas T pode levar à "ondulação" do perfil de distribuição drift tempo (Fig. 2D). Este artefato ocorre quando a separação mobilidade dos íons (hora de chegada / drift ion) não é mantida através da transferência e regiões ToF, devido à sincronização parcial entre a frequência empurrador ea transferência de velocidade da onda T. , A fim de eliminar esse efeito, quer a tempo o traficante ou o de transferência de velocidade da onda T deve ser ajustado. Como a freqüência empurrador está relacionado com a faixa de massa, este artefato pode reaparecer quando este parâmetro é alterado. T-wave altura exerce um efeito menor, embora a sua redução pode também ajudar a eliminar ondulações.
- Uma vez que os parâmetros acima referidos são otimizados, os dados IM-MS podem ser adquiridos.
Parte 2: Triagem de condições experimentais para assegurar medidas de mobilidade das estruturas nativas
Para atingir picos altamente resolvido MS, complexos de proteína são frequentemente ativado dentro do espectrômetro de massa, para promover a remoção de água residual e de componentes do tampão 11. No entanto, se a energia de ativação é aumentada além de um valor limite, parcial desdobramento pode ser induzida formando vários estados intermediários 12, que não são susceptíveis de corresponder ao nativo, estrutura de solução do Estado (Fig. 3A-C). Como resultado, o pico do tempo deriva pode ser deslocado e ampliado, refletindo a população heterogênea de unfolded estruturas.
, A fim de obter dados drift tempo consistente com a solução fase-estruturas, é essencial controlar cuidadosamente as tensões usadas para acelerar íons, antes da separação IM. Além disso, de alta resolução de MS é preferível aumentar a transferência ao invés da tensão de Trap. Como o dispositivo de IM está posicionado, em primeiro lugar, seguido pela região de Transferência eo analisador TOF, portanto, a ativação se segue a medição IM e os íons permanece inalterada, enquanto a precisão MS pode ser aumentado.
Para validar a aquisição de dados é realizada sob condições que mantêm a estrutura nativa do complexo, é recomendável que os dados sejam gravados em um intervalo de condições experimentais e solução, em vez de acordo com um único conjunto de parâmetros otimizados:
- Aumentarem a voltagem do capilar e cone de forma gradual, enquanto monitora o efeito sobre o espectro de tempo deriva.
- Como na Etapa 1, aumentar a tensão de colisão armadilha de uma forma gradual, e aquisição de dados em intervalos de 10 V.
- Para identificar as conformações desdobrado e avaliar os dados adquiridos, manualmente induzir a dissociação do complexo protéico pela titulação da amostra com ácido acético em uma faixa de pH de 2-7, e gravar os dados (Fig. 3B).
Parte 3: Correlacionando entre os valores de drift tempo e áreas transversais
Ao contrário das medidas IM convencional, em que os valores de tempo medidos drift são linearmente relacionadas com Ω, no sistema IMS da onda T, a área da seção transversal é definida por uma abordagem de calibração. Assim, ao invés de uma medida absoluta, uma correlação em relação exponencial é gerado entre os tempos medidos e drift 1,13 Ω: 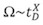
onde t D é o tempo medido drift, e X é a constante de proporção que pode ser extraído de uma curva de calibração. A calibração é realizared medindo os tempos de deriva dos íons com Ω conhecidos (medidos a partir de experimentos IM convencional).
- Medições de tempo deriva são calibrados utilizando proteínas desnaturadas eqüinos citocromo C, mioglobina coração cavalo e bovina com ubiquitina conhecida colisão seções transversais. Para este efeito, as soluções de 10 mM em 49/49/2, relação de volume, água / metanol / ácido acético deve ser preparado (reagentes utilizados estão descritos na Tabela 3).
- IM-MS adquirir dados para as proteínas de calibração em condições exactamente mesmo instrumento usado para a proteína-alvo, ou complexo de proteínas:
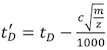 (Parte 1). Todas as tensões e os valores de pressão devem ser idênticos, para preservar as configurações de separação IM.
(Parte 1). Todas as tensões e os valores de pressão devem ser idênticos, para preservar as configurações de separação IM. - Para cada estado de carga das proteínas de calibração extrair o valor de tempo experimental drift (t D) (descrito na Parte 4).
- Correta cada uma das vezes deriva de calibração (t D) (Tabela 2), utilizando a seguinte equação:
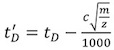 , Onde m / z é a relação massa-carga do íon observado, e c é o Ciclo Avançado (EDC) coeficiente de atraso 1. Seu valor, tipicamente entre 1,4 e 1,6, é o instrumento-dependente. O valor EDC é indicado dentro do Sistema | Configurações Aquisição | guia de instalação de Aquisição.
, Onde m / z é a relação massa-carga do íon observado, e c é o Ciclo Avançado (EDC) coeficiente de atraso 1. Seu valor, tipicamente entre 1,4 e 1,6, é o instrumento-dependente. O valor EDC é indicado dentro do Sistema | Configurações Aquisição | guia de instalação de Aquisição. - Usando Banco de Dados Prof David Clemmer Cross-Section:
http://www.indiana.edu/ ~ Clemmer / Research / cross% 20section% 20database/Proteins/protein_cs.htm 14 correta cada um dos calibração seção transversal para ambos estado de carga de íons e massa reduzida.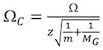
, Onde Ω C é a correção de seção transversal, Ω é a literatura de seção transversal, z é o estado de carga iônica, m é o peso molecular do íon de calibração, e M G é o peso molecular do gás de fundo IM (tipicamente N 2). - Na trama (t D) contra In (Ω C) (Fig. 4A).
- A curva resultante corresponde à seguinte equação:
 O X parâmetros e A pode ser extraído por montar o plano para uma relação linear. O X inclinação corresponde ao fator proporção exponencial e A representa a constante de fit-determinado.
O X parâmetros e A pode ser extraído por montar o plano para uma relação linear. O X inclinação corresponde ao fator proporção exponencial e A representa a constante de fit-determinado. - Calcular o coeficiente de correlação r 2 se encaixam, usando a equação de Pearson:
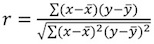 . Valores aceitáveis para r 2 são maiores do que 0,95 (Fig. 4B). Um valor mais baixo coeficiente de correlação pode ser devido a:
. Valores aceitáveis para r 2 são maiores do que 0,95 (Fig. 4B). Um valor mais baixo coeficiente de correlação pode ser devido a: - Incompleta desdobramento da calibradores proteína. Isto levará a uma ampliação de pico devido ao conjunto heterogêneo de estados intermediários.
- Em nossa experiência, uma amostra de idade pode deteriorar-se os espectros de IM.
- Diferentes condições experimentais utilizadas para a calibração proteínas diferentes. Neste caso, traçando os dados separadamente para cada proteína deve gerar diversos coeficientes de correlação, embora cada um deles deve ser superior a 0,95.
- Dados ruidosos e incorretas de alisamento ea centralização da distribuição do tempo de drift.
- Erro de cálculo.
- Recorrect o tempo drift de calibração usando o fator exponencial determinada, X, derivada na etapa 7:
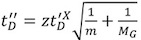
- Como uma etapa de validação, replot Ω C vs
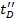 e definir o coeficiente de correlação. Valores superiores a 0,95 são esperadas.
e definir o coeficiente de correlação. Valores superiores a 0,95 são esperadas. - De acordo com o procedimento descrito na Etapa 4, corrigir o tempo de drift medida da proteína-alvo, ou complexo de proteínas:
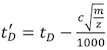
- Calibrar o tempo de deslocamento do alvo complexo de proteína / proteína usando o fator exponencial, X, definido no Passo 7:
- Calcular Ω do alvo complexo proteína / proteína usando a constante fit-determinado, A, definido no Passo 7:
 .
. - Para cada condição experimental, Passos 2-13 deve ser repetido. Ao definir a área da seção transversal da proteína desconhecido ou complexo de proteínas, recomendamos que cada experimento será repetido pelo menos três vezes, eo desvio padrão dessas medições triplicado determinado.
Parte 4: Definindo valores de tempo deriva
Software necessários: MassLynx e Driftscope (Waters).
- Abrir o espectro IM-MS usando o software Driftscope.
- No menu principal, selecione View e desmarque Cromatograma, Time Drift e Spectrum (opcional), deixando ativo somente o mapa em 2D mostrando a hora drift vs m / z.
- Na barra de menu, escolha Display | Opções | Painel Editor de exibição e ajustar os valores limiar de intensidade para minimizar o ruído de fundo (na maioria dos casos, esta configuração pode ser definida como Min% = 30-40 = Max e 100 contagens%).
- Escolha Display | Escala de Intensidade Mapa 2D, e serão apresentadas três opções: Escala Linear, Log Escala e Escala de Raiz Quadrada. A escolha da Escala Log (dados logarítmica transformar) irá comprimir o código de cor, intensidade e permitir o aparecimento simultâneo de uma ampla gama de intensidades (por oposição às opções de raiz linear e quadrada, com a qual apenas os picos mais intensos se tornará visível) .
- A partir do painel da barra de ferramentas, use o botão Ferramenta de Seleção. Esta opção irá ativar as opções de seleção diferentes, e permitem a seleção da região relevante dentro do espectro. A ferramenta mais precisa é ativar Região de Seleções de juros, por meio do qual uma borda pode ser desenhada em torno da região de interesse, excluindo assim todos os dados desnecessários e picos de ruído. Da mesma forma, as opções de seleção Orthogonal e Band são úteis, quando a região de interesse não é cercada por picos redundante.
- Uma vez que a região de interesse é selecionado, use o comando Aceitar seleção atual para remover informações desnecessárias.
- Exportar os dados para MassLynx, mantendo a informação em tempo deriva.
- Dentro MassLynx, abra o cromatograma do espectro salvo drift tempo, e combinar os compartimentos do tempo. O espectro de massa correspondente abrirá automaticamente.
- Aplicar a função de alisamento, definindo o tamanho da janela e número de parâmetros bom (deve ser ajustado especificamente para cada espectro, utilizando-se valores mínimos).
- Aplicar base subtração, se necessário.
- Centro de espectro e medir a massa, para validar a identidade de proteína e precisão em massa.
- Para cada estado de carga, combine a gama m / z. O espectro de tempo correspondente drift aparecerá automaticamente. Suave e centro do perfil temporal de vento, e definir o valor do tempo deriva, indicando o centróide de cada pico.
Parte 5: Resultados Representante

Figura 1. Representação esquemática do instrumento Synapt HDMS indicando os principais parâmetros ajustáveis do IMS-MS aquisição. Parâmetros experimentais para IM-MS medições são rotuladas de acordo com sua posição dentro do instrumento. O feixe de íons é colorido em vermelho, ea pressão em cada região é designada com um código de cores. O painel no fundo ilustra o gradiente de potencial ao longo do instrumento e as potenciais diferenças definir a armadilha e energias de colisão de Transferência, bem como o potencial Bias. Todos os potenciais ler-backs são referenciados à tensão estática offset que é geralmente definido para 120V.

Figura 2. Mobilidade iônica chegada distribuições de tempo para a proteína Gβυ.
A. A velocidade da onda T alta leva a uma distribuição estreita do perfil de tempo deriva. O enredo mostra a distribuição do tempo de chegada do Estados de carga 16 + (vermelho), 15 + (verde), 14 + (azul), e 13 + (magenta), assim como o perfil total de drift tempo (em preto) da proteína G βυ.
B. Uma vez otimizado do espectro de drift com uma forma suave de pico de Gauss. Similar etiquetas de cores como em A.
C. A 'roll-over "efeito, que ocorre quando o tempo necessário para íons de atravessar a célula a mobilidade é mais lento do que o intervalo entre as injeções de pacotes ion nova para o dispositivo. Como resultado, o pico de tempo prolongado drift aparece no início do espectro. Este efeito pode ser eliminado com o aumento da altura da onda T, e diminuindo a onda T velocidade e pressão IMS.
"Ondulações" D. Artificial são causados quando a transferência de velocidade da onda T e freqüência empurrador são parcialmente sincronizado. Este efeito pode ser superada através do ajuste ou a freqüência ou velocidade de transferência empurrador da onda T.
Figura 3. O efeito de ativação de íons e condições parciais de desnaturação em IM-MS espectros de hemoglobina. Plot de tempo deriva em relação m / z para o complexo de hemoglobina tetramérica, usando uma solução aquosa de acetato de amônio 10 mM (pH = 7.6) (A, C) ea adição de 0,1% de ácido acético (B). Os dados adquiridos com Armadilha de tensão de energia de colisão de 13 V (A, B) e 35 V (C) Embora em todos os três painéis do espectro de massa (projetada no topo) é semelhante, com uma série cobrar tetramérica centrada em 4.000 m / z, o perfil temporal drift (projetado nas laterais) é diferente (distribuição do tempo total de drift éem preto, eo perfil de + 16 está em vermelho). O tempo de drift da amostra parcialmente desnaturado, obtido em B, e os íons na fase gasosa activada, obtido em C, é indicativo de algum grau de desdobramento. Esta observação ilustra que, embora a massa medida corresponde a um complexo intacta, a sua estrutura de solução é interrompido. Como conseqüência, o controle cuidadoso das condições experimentais é necessária.
Figura 4. Gerando uma curva de calibração, a deriva medidas de tempo e colisão seções transversais podem ser correlacionados.
A. Medida valores drift tempo do estados de carga múltipla de equinos do citocromo C (círculos), a mioglobina de cavalo coração (triângulos) e bovinos ubiquitina (quadrados) foram plotados contra os valores da literatura Ω corrigido para ambos estado de carga de íons e massa reduzida. O ajuste produz uma função linear correspondente a: ln (Ω C) = XLN (t D ') + A. O fator determinado exponencial (X), encaixam-determinado constante (A), eo coeficiente de correlação são apresentados no gráfico de dados adquiridos a uma velocidade da onda T de 350 m / s, e uma altura de onda estática de 11 V. B. Um histograma das distribuições coeficiente de correlação obtido a partir de 10 experimentos de calibração consecutivos.
| Amostra de proteína / Parâmetros técnicos | GluFibrino- peptídeo monômero 1,6 kDa | Mioglobina monômero 17 kDa | Hemoglobina tetrâmero 67 kDa | Transferrina monômero 80 kDa | GroEL 14-mer 801 kDa |
| Pressão de suporte, mBar | 4,4 | 5,0 | 5,1 | 5,1 | 6,5 |
| Pressão armadilha, mBar | 1.6x10 -2 | 2.4x10 -2 | 2.4x10 -2 | 2.6x10 -2 | 2.8x10 -2 |
| IMS de pressão, mBar | 4.4x10 -1 | 4.4x10 -1 | 4.4x10 -1 | 4.4x10 -1 | 4.2x10 -1 |
| Tensão de amostragem cone, V | 46 | 80 | 80 | 80 | 118 |
| Extração de tensão cone, V | 1,7 | 1 | 1 | 1 | 3 |
| Viés de tensão, V | 20 | 20 | 25 | 25 | 50 |
| Energia armadilha colisão, V | 20 | 15 | 15 | 15 | 80 |
| Transferência de energia de colisão, V | 5 | 12 | 12 | 12 | 15 |
Tabela 1. Condições experimentais utilizadas para a análise de macromoléculas.
| Proteína padrão | Massa Molecular (m) | Encargos (z) | m / z | Colisão cross-section (no 2) |
| Citocromo C | 12213 | 10 | 1222,3 | 2226 |
| 11 | 1111,3 | 2303 | ||
| 12 | 1018,8 | 2335 | ||
| 13 | 940,5 | 2391 | ||
| 14 | 873,4 | 2473 | ||
| 15 | 815,2 | 2579 | ||
| 16 | 764,3 | 2679 | ||
| 17 | 719,4 | 2723 | ||
| 18 | 679,5 | 2766 | ||
| Mioglobina | 16952 | 11 | 1542,1 | 2942 |
| 12 | 1413,7 | 3044 | ||
| 13 | 1305,0 | 3136 | ||
| 14 | 1211,9 | 3143 | ||
| 15 | 1131,1 | 3230 | ||
| 16 | 1060,5 | 3313 | ||
| 17 | 998,2 | 3384 | ||
| 18 | 942,8 | 3489 | ||
| 19 | 893,2 | 3570 | ||
| 20 | 848,6 | 3682 | ||
| 21 | 808,2 | 3792 | ||
| 22 | 771,6 | 3815 | ||
| Ubiquitina | 8565 | 8 | 1071,6 | 1442 |
| 8 | 1071,6 | 1622 | ||
| 9 | 952,7 | 1649 | ||
| 10 | 857,5 | 1732 | ||
| 11 | 779,6 | 1802 | ||
Tabela 2. Proteínas de calibração e suas seções de colisão transversal valores determinados pelo measurments convencionais IMS 14.
| Dispositivos | Companhia | Número de catálogo |
| Synapt HDMS-32K RF gerador | Águas Ltd. | |
| P-97 Flaming-Brown micropipeta extrator | Sutter Instruments | P-97 |
| Sputter coater | Microscopia Eletrônica de Ciências | EMS550 |
| Microscópio binocular | Nikon | |
| Reagentes | Companhia | Número Catálogo |
| Acetato de amônio | Sigma-Aldrich | Sigma, A2706 |
| CsI 99,999% | Sigma-Aldrich | Aldrich, 203033 |
| Metanol | Sigma-Aldrich | Fluka, 34966 |
| Ácido acético | Fisher Scientific | AC12404 |
| Equine mioglobina (de coração de cavalo) | Sigma-Aldrich | M1882 |
| Equine citocromo c (de coração de cavalo) | Sigma-Aldrich | C-2506 |
| Bovina ubiquitina (de glóbulos vermelhos) | Sigma-Aldrich | U6253 |
| Hemoglobina | Sigma-Aldrich | H2625 |
| Gás | Comentários | |
| Nitrogênio, 99,999% puro | 8 cilindros metro cúbico | |
| Argônio, 99,999% puro | 8,8 meterscylinder cúbicos | |
Tabela 3. Reagentes e equipamentos.
Discussão
O protocolo aqui descrito permite definir a seção transversal de colisão de proteínas ou complexos de proteínas com uma estrutura de três desconhecidos dimensional, com o objetivo de fornecer informações sobre sua forma geral, embalagem subunidade e topologia. Para este efeito, uma vez valores colisão seção transversal são retratados é necessário converter esses valores para detalhes estruturais. Este processo requer esforços adicionais experimental bem como a análise computacional, que são brevemente discutidos abaixo.
Para começar, recomenda-se a análise de proteínas ou complexos de proteínas com estruturas conhecidas. Estas medidas podem proporcionar um controle de qualidade útil da metodologia e permitirá avaliação da acurácia dos parâmetros de aquisição, comparando os valores teóricos e medidos Ω. O teórico áreas das secções transversais podem ser calculadas a partir da estrutura de cristal coordenadas usando o software MOBCAL 15,16, que é um software de código aberto baseado em FORTRAN que permite a edição de código de acordo com as necessidades do operador. Para a execução de tais cálculos é necessário modificar o programa de tal forma que o número de cálculos iterativos realizados por estrutura de entrada é maior e que os arquivos de coordenadas contendo grande número de átomos são aceitos 1.
Uma estratégia IM-MS para a definição de arranjos topológicos de subunidades dentro multicomponentes montagens propôs recentemente 4,6. O método envolve o monitoramento de vias dissociação de grupos de proteínas para os componentes menores. Essa dissociação é conseguido através do ajuste das condições controladas fase de solução, que dá origem a uma distribuição de subcomplexes reflexivo da "blocos de construção" das assembléias. A medição simultânea de valores Ω de ambos os complexos intacta a desmontagem e produtos gera restrições estruturais que são então utilizados para calcular os modelos topológicos dos complexos de proteína. O pressuposto básico subjacente a esta metodologia é que o subcomplexes gerada reter seus nativos como confirmações, e na verdade estudos recentes têm demonstrado que a estrutura de solução dos produtos desmontagem é mantida e nenhum rearranjo importante em qualquer solução ou fases de gás têm ocorrido 4,6.
O último passo na atribuição de estrutura quaternária de íons em fase gasosa em proteínas complexas é adequado a colisão valores seção transversal de modelos gerados por computador. Abordagens de modelagem são empregadas, a fim de explorar as diferentes possíveis arranjos topológicos de subunidades e seus valores em silico Ω são calculados e comparados com os experimentais. Atualmente, apenas algumas abordagens computacionais são usados, como o método de granulação grossa spheretype que se aproxima o diâmetro de 1,8 subunidades. No geral, este campo ainda está em seus primeiros anos e desenvolvimento é necessário para fazer essa abordagem genérica e aplicável a uma ampla gama de complexos.
Divulgações
Agradecimentos
Os autores agradecem os membros do grupo Sharon para a sua revisão crítica, e por suas contribuições ao manuscrito. Estamos gratos pelo apoio dos Programas Morasha e Bikura, a Israel Science Foundation (Grant n. º s 378/08 e 1823-1807), o Josef Cohn Minerva Center for Research biomembrana, a Família Chais Fellows Program para novos cientistas, o Abraham e Sonia Rochlin Foundation, o Family Trust Wolfson Charitable, a Helen e Milton A. Kimmelman Centro de Estrutura Biomolecular e Montagem; o espólio de Shlomo e Beirzwinsky Sabine; Meil de Botton Aynsley, e Karen Siem, Reino Unido.
Referências
- Ruotolo, B. T. Ion mobility-mass spectrometry analysis of large proteincomplexes. Nat Protoc. 3 (7), 1139-1152 (2008).
- Scarff, C. A., Thalassinos, K., Hilton, G. R., Scrivens, J. H. Travelling wave ion mobility mass spectrometry studies of protein structure: biological significance and comparison with X-ray crystallography and nuclear magnetic resonance spectroscopy measurements. Rapid Commun Mass Spectrom. 22 (20), 3297-3304 (2008).
- Smith, D. P. Deciphering drift time measurements from travelling wave ion mobility spectrometry-mass spectrometry studies. Eur J Mass Spectrom (Chichester, Eng). 15 (2), 113-130 (2009).
- Leary, J. A. Methodology for measuring conformation of solvent-disrupted protein subunits using T-WAVE ion mobility MS: an investigation into eukaryotic initiation factors. J Am Soc Mass Spectrom. 20 (9), 1699-1706 (2009).
- Lorenzen, K. Determination of stoichiometry and conformational changes in the first step of the P22 tail assembly. J Mol Biol. 379 (2), 385-396 (2008).
- Pukala, T. L. Subunit architecture of multiprotein assemblies determined using restraints from gas-phase measurements. Structure. 17 (9), 1235-1243 (2009).
- van Duijn, E. Chaperonin complexes monitored by ion mobility mass spectrometry. J Am Chem Soc. 131 (4), 1452-1459 (2009).
- Ruotolo, B. T. Evidence for macromolecular protein rings in the absence of bulk water. Science. 310 (5754), 1658-1661 (2005).
- Ruotolo, B. T., Robinson, C. V. Aspects of native proteins are retained in vacuum. Curr Opin Chem Biol. 10 (5), 402-408 (2006).
- Giles, K. Applications of a travelling wave-based radio-frequency-only stacked ring ion guide. Rapid Commun Mass Spectrom. 18 (20), 2401-2414 (2004).
- McKay, A. R., Ruotolo, B. T., Ilag, L. L., Robinson, C. V. Mass measurements of increased accuracy resolve heterogeneous populations of intact ribosomes. J Am Chem Soc. 128 (35), 11433-11442 (2006).
- Ruotolo, B. T. Ion mobility-mass spectrometry reveals long-lived, unfolded intermediates in the dissociation of protein complexes. Angew Chem Int Ed Engl. 46 (42), 8001-8004 (2007).
- Morton, V. L., Stockley, P. G., Stonehouse, N. J., Ashcroft, A. E. Insights into virus capsid assembly from non-covalent mass spectrometry. Mass Spectrom Rev. 27 (6), 575-595 (2008).
- Valentine, S. J., Counterman, A. E., Clemmer, D. E. A database of 660 peptide ion cross sections: use of intrinsic size parameters for bona fide predictions of cross sections. J Am Soc Mass Spectrom. 10 (11), 1188-1211 (1999).
- Mesleh, M. F. Structural information from ion mobility measurements: effects of the long-range potential. J Phys Chem. 100, 16082-16086 (1996).
- Shvartsburg, A. A., Jarrold, M. F. An exact hard-spheres scattering model for the mobilities of polyatomic ions. Chem Phys Lett. 261, 86-91 (1996).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados