Method Article
T-wave mobilità ionica-spettrometria di massa: Basic procedure sperimentali per l'analisi delle proteine complesse
In questo articolo
Riepilogo
Mobilità ionica, spettrometria di massa è un emergente tecnologia in fase gas che separa gli ioni, in base alla loro collisione sezione trasversale e di massa. Il metodo fornisce informazioni tridimensionali sulla topologia generale e la forma di complessi proteici. Qui, delineare una procedura di base per l'impostazione dello strumento e l'ottimizzazione, la taratura dei tempi di deriva, e l'interpretazione dei dati.
Abstract
Mobilità ionica (IM) è un metodo che misura il tempo impiegato per uno ione di viaggiare attraverso una cella sotto pressione sotto l'influenza di un debole campo elettrico. La velocità con cui gli ioni attraversano la regione deriva dipende dalla loro grandezza: gli ioni di grande esperienza sarà un numero maggiore di collisioni con il gas di fondo inerte (in genere N 2) e quindi viaggiare più lentamente attraverso il dispositivo IM rispetto a quelli ioni che costituiscono una piccola sezione trasversale. In generale, il tempo necessario per gli ioni a migrare anche se la fase di gas denso li separa, in base alla loro collisione sezione (Ω).
Recentemente, IM spettrometria stata accoppiata con spettrometria di massa e di un viaggio-onda (onda T) Synapt mobilità ionica spettrometro di massa (IM-MS) è stato rilasciato. L'integrazione di spettrometria di massa con mobilità ionica consente una dimensione extra di separazione del campione e definizione, ottenendo un effetto tridimensionale dello spettro (di massa a tempo di carica, intensità, e deriva). Questa tecnica di separazione consente la sovrapposizione spettrale a diminuire, e consente la risoluzione di complessi eterogenei con massa molto simili, o di massa a carica rapporti, ma i tempi di deriva diverse. Inoltre, le misure deriva tempo forniscono un importante livello di informazioni strutturali, come Ω è legato alla forma complessiva e la topologia dello ione. La correlazione tra i valori misurati deriva tempo e Ω è calcolato utilizzando una curva di calibrazione generata dalle proteine calibrante con definite sezioni 1.
Il potere della IM-MS approccio risiede nella sua capacità di definire l'imballaggio subunità e la forma complessiva delle assemblee proteine a concentrazioni micromolari, e quasi fisiologica condizioni 1. Diversi studi recenti di IM sia singole proteine 2,3 e non covalenti complessi proteici 4-9, ha dimostrato con successo che la struttura della proteina quaternaria è mantenuta in fase gassosa, e ha evidenziato le potenzialità di questo approccio allo studio delle assemblee proteine della geometria sconosciuto . Qui, fornire una descrizione dettagliata di IMS-MS analisi dei complessi proteici con il Synapt (quadrupolo-Ion Mobilità-Time-of-Flight) strumento HDMS (Waters Ltd, l'unica commerciale IM-MS strumento attualmente disponibile) 10. Sono descritte le fasi di ottimizzazione di base, la taratura di collisione sezioni, e metodi per l'elaborazione dei dati e l'interpretazione. La fase finale del protocollo discute metodi di calcolo dei valori teorici Ω. Nel complesso, il protocollo non cerca di coprire ogni aspetto della IM-MS caratterizzazione delle assemblee proteine, anzi, il suo obiettivo è quello di introdurre gli aspetti pratici del metodo di nuovi ricercatori nel campo.
Protocollo
La procedura che descriviamo si concentra esclusivamente su IM-MS analisi dei complessi proteici. Pertanto, suggeriamo che i ricercatori non conoscono con il campo di MS strutturali fanno riferimento alle fasi di preparazione del campione, calibrazione degli strumenti e MS e MS tandem procedure di ottimizzazione descritte in Kirshenbaum et al. Http://www.jove.com/index/details 2009. stp? ID = 1954. In generale, questo protocollo prevede basse concentrazioni micromolari del complesso (1-20 mM) in un buffer di volatili quali l'acetato di ammonio (0,005 - 1 M, pH 6-8). Dato che 1-2 microlitri sono consumati per nanoflussi capillare, suggeriamo 10-20 microlitri come un volume minimo, per consentire l'ottimizzazione delle condizioni di SM.
Parte 1: L'acquisizione di uno ione di mobilità di massa spettrometria spettro
- Impostare lo spettrometro di massa sui seguenti modalità di funzionamento: Mobilità-TOF, l'acquisizione di ioni positivi, e V-mode.
- Accendere tutti i gas (API, Trappola e IMS). Usiamo N 2 per la separazione IM, e Ar per il Trap / trasferimento. Consigliati valori iniziali sono 1,5 ml / min per la regione Trap, e un flusso di gas di 24 ml / min per il dispositivo IMS.
- Impostare la m / z gamma di acquisizione. Per un complesso proteico sconosciuto, si consiglia l'uso iniziale di una vasta gamma di massa, che possono poi essere ridotto a valori desiderati. In parallelo, regolare il profilo di MS per la massima efficienza di trasmissione. Per complessi di grandi dimensioni, la gamma di massa acquisizione dovrebbe essere impostato da 1.000 - 32.000 m / z, e il profilo di MS su Auto. In caso contrario, il profilo può essere impostato secondo la seguente tabella:
m / z abitano (%) rampa (%) 960 10 20 3200 30 40 10667 - Controllare l'impostazione radiofrequenza e, se necessario, regolare di valori appropriati per i complessi di proteine di grandi dimensioni, così:
Fonte Trappola IMS Trasferimento RF Offset 450 380 380 380 RF Gain 0 0 0 0 RF Limite 450 380 380 380 - Applicare la tensione capillare (1,050-1,400 V) e la pressione nanoflussi bassa (0,00-0,03 bar). Una volta spray è iniziata, provare a ridurre la pressione nanoflussi ad un valore minimo. Inoltre, regolare la posizione del capillare, rispetto al cono.
- Regolare i parametri di acquisizione MS, di acquisire uno spettro ben risolto MS: il gradiente di pressione lungo lo strumento, e il cono di campionamento così come il cono di estrazione, pregiudizi, Trappola e Trasferimento impostazioni possibili, dovrebbero essere tutti ottimizzati (dettagliato nel associati JOVE protocollo Kirshenbaum et al. https://www.jove.com/index/details.stp?ID=1954 2009). Anche se questi parametri sono campione-dipendente, le condizioni che abbiamo usato per l'acquisizione di spettri MS delle masse di ioni diversi, dal peptide di complessi proteici, sono riportati nella tabella 1, (vedi anche Fig. 1).. Per ridurre al minimo l'attivazione del complesso cercare di ridurre gradualmente (in passi di 10 V ~) tensioni il cono campione, Trappola e pregiudizi senza modificare la posizione del picco.
- Una volta uno spettro di massa ottimale si ottiene il profilo temporale deriva deve essere regolato. Quando si analizzano gruppi di proteine, le condizioni ottimali sia per la mobilità di massa e le misure sono spesso incompatibili, quindi, è importante trovare il giusto equilibrio tra i due. Nel complesso, la trama mobilità ionica deve essere ottimizzata in modo tale che i picchi sono distribuiti su tutta la gamma tempo di deriva, ed il profilo di picco è liscio, si avvicina ad una distribuzione gaussiana (Fig. 2A, 2B). Picco significativo asimmetria può essere correlato a scarsa separazione di conformazioni multiple.
- Come regola generale, tre parametri, la velocità dell'onda T, T-altezza d'onda e la velocità del flusso di gas IMS possono essere adattati per ottimizzare la separazione mobilità. L'aumento della velocità dell'onda T amplierà il tempo di deriva profilo di distribuzione, mentre un aumento dell'onda T valori di altezza lo stretto. Allo stesso modo, aumentando il flusso del gas IMS si sposterà il profilo temporale deriva verso valori più alti (il minimo flusso di gas IMS dovrebbe essere di 10 ml / min). Vi consigliamo di lasciare due delle tre variabili a disposizione fissa, e ottimizzando il terzo fino a quando lo spettro IM è ben risolto (Fig. 2B). A tal fine, impostare la velocità dell'onda T e il flusso di gas a 250 m / s e 24 ml / min, rispettivamente. Poi, come punto di partenza, impostare l'altezza dell'onda T a 3 V e, in maniera graduale, esso aumenta con incrementi di 1 V. In generale, più grande ioni richiederà altezze dell'onda più alta. Di solito, non vi è alcuna necessità di modificare la pressione IMS, tuttavia, quando tensioni di polarizzazione che richiedono elevate per la regolazione, riducendo il gas IMS permetterà una diminuzione del valore della tensione di polarizzazione e, di conseguenza, una riduzione di attivazione delle proteine complesse. Nel complesso, risoluzione massima di 10-12 t / At può essere raggiunto.
- Quando le condizioni non sono ottimizzate (basso dell'onda T altezza o ad alta velocità dell'onda T e / o pressione alta IM), gli ioni non attraversare attraverso il dispositivo IM in modo efficace, e il loro viaggio può richiedere più il tempo necessario per lo ione prossimo pacchetto per essere rilasciato nella cellula mobilità. Come risultato, un pacchetto di ioni nuovo verrà rilasciato dalla regione Trappola prima che il pacchetto precedente è stato consegnato alla regione pusher. Questo porterà a un 'roll-over' effetto, in cui il picco osservato nella prima parte dello spettro tempo deriva è identica a quella degli ioni nel bordo tailing (Fig. 2C). Questo artefatto può essere eliminato aumentando la T-wave altezza, e diminuendo la velocità dell'onda T e pressione IMS. Inoltre, il tempo di rilascio Trappola può essere regolata. Inoltre, è importante verificare che l'altezza dell'onda T trasferimento è impostata su almeno 5 V. Per evitare la fuoriuscita di ioni verso la cella di IMS, si raccomanda che l'altezza trappola mobilità essere mantenuti a livelli massimi (30 V).
- Bassa velocità ed elevata ampiezza di trasferimento T-onde possono portare alla "increspature" del profilo di distribuzione deriva tempo (Fig. 2D). Questo artefatto si verifica quando la separazione mobilità degli ioni (ioni di arrivo / deriva tempo) non è mantenuta attraverso il trasferimento e regioni ToF, a causa di sincronizzazione parziale tra la frequenza e la spinta dell'onda T velocità di trasferimento. Per eliminare questo effetto, sia il tempo di spinta o il T-velocità dell'onda di trasferimento deve essere regolato. Poiché la frequenza pusher è in relazione alla gamma di massa, questo manufatto può ricomparire quando questo parametro è cambiato. Dell'onda T altezza esercita un effetto minore, anche se la sua riduzione può inoltre contribuire ad eliminare increspature.
- Una volta che i suddetti parametri sono ottimizzati, l'IM-MS dati possono essere acquisiti.
Parte 2: Screening condizioni sperimentali per assicurare misure di mobilità delle strutture native
Per raggiungere picchi molto risolto MS, complessi proteici sono spesso attivati nel spettrometro di massa, per promuovere la rimozione dei residui di acqua e componenti di buffer 11. Tuttavia, se l'energia di attivazione viene aumentata oltre il valore soglia, parziale svolgimento può essere indotta formando 12 diversi stati intermedi, che sono difficilmente corrispondono ai nativi, la soluzione a stato struttura (Fig. 3A-C). Di conseguenza, il picco tempo di deriva può essere spostato e ampliato, riflettendo la popolazione eterogenea di strutture spiegato.
Al fine di ottenere dati in tempo deriva coerente con la soluzione fase strutture, è essenziale per controllare con attenzione le tensioni utilizzate per accelerare gli ioni, prima della separazione IM. Inoltre, per alta risoluzione MS è preferibile aumentare il trasferimento, piuttosto che la tensione di Trap. Come dispositivo di IM è posizionato, per prima, seguita dalla regione di trasferimento e l'analizzatore TOF, quindi, l'attivazione segue la misura IM e gli ioni rimane inalterato, mentre l'accuratezza MS può essere aumentata.
Per convalidare che acquisizione dei dati è effettuata in condizioni di mantenere la struttura originaria del complesso, si raccomanda che i dati da registrare su una gamma di condizioni sperimentali e la soluzione, piuttosto che in base ad un singolo, ottimizzato serie di parametri:
- Aumentare il voltaggio del capillare e cono in modo graduale, monitorando l'effetto sullo spettro tempo di deriva.
- Come nella Fase 1, aumentare la tensione collisione Trappola in maniera graduale, e acquisire i dati a intervalli di 10 V.
- Per identificare le conformazioni spiegato e valutare i dati acquisiti, manualmente indurre dissociazione del complesso proteina titolando il campione con acido acetico in un range di pH 2-7, e registrare i dati (Fig. 3B).
Parte 3: Correlazione tra i valori di tempo di deriva e di sezione
A differenza di misure IM convenzionali, in cui i valori misurati tempo di deriva sono linearmente relative a Ω, nel T-wave sistema IMS, l'area della sezione trasversale è definita da un approccio di calibrazione. Così, piuttosto che una misura assoluta, una correlazione relativa esponenziale è generato tra i tempi di deriva misurato e 1,13 Ω: 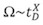
dove t D è il tempo di deriva misurato, e X è la costante proporzione che può essere estratto da una curva di calibrazione. La calibrazione è eseguireed misurando i tempi di deriva degli ioni con nota Ω (misurata dagli esperimenti IM convenzionale).
- Misure di tempo deriva sono calibrati utilizzando proteine denaturate equina citocromo C, cuore mioglobina cavallo e bovino ubiquitina con nota di collisione sezioni. A tal fine, le soluzioni di 10 micron di 49/49/2, rapporto volume, acqua / metanolo / acido acetico deve essere preparato (reagenti utilizzati sono riportati nella tabella 3).
- Acquisire IM-MS dati per le proteine calibrante sotto esattamente le condizioni lo stesso strumento utilizzato per la proteina bersaglio o complesso di proteine:
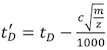 (Parte 1). Tutte le tensioni ed i valori di pressione devono essere identiche, per preservare le impostazioni di separazione IM.
(Parte 1). Tutte le tensioni ed i valori di pressione devono essere identiche, per preservare le impostazioni di separazione IM. - Per ogni stato di carica delle proteine calibrante estrarre il valore sperimentale tempo di deriva (t D) (descritto nella parte 4).
- Corretta per ciascuno dei tempi deriva calibrante (t D) (Tabella 2) utilizzando la seguente equazione:
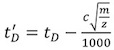 , Dove m / z è la massa-di-carica rapporto tra lo ione osservato, e c è il ciclo di funzionamento avanzato (EDC) ritardo coefficiente 1. Il suo valore, tipicamente tra 1.4 e 1.6, è lo strumento-dipendente. Il valore di EDC è indicato all'interno del Sistema | Impostazioni di acquisizione | Acquisizione scheda Impostazione.
, Dove m / z è la massa-di-carica rapporto tra lo ione osservato, e c è il ciclo di funzionamento avanzato (EDC) ritardo coefficiente 1. Il suo valore, tipicamente tra 1.4 e 1.6, è lo strumento-dipendente. Il valore di EDC è indicato all'interno del Sistema | Impostazioni di acquisizione | Acquisizione scheda Impostazione. - Utilizzando Prof. David Clemmer Cross-Sezione del database:
http://www.indiana.edu/ ~ Clemmer / Ricerca / croce% 20section% 20database/Proteins/protein_cs.htm 14 corretto ciascuno dei calibrante sezione trasversale sia per stato di carica di ioni e massa ridotta.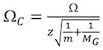
, Dove Ω C è la corretta sezione, Ω è la sezione letteratura, z è lo stato di carica ionica, m è il peso molecolare dello ione calibrante, G e M è il peso molecolare del gas di fondo IM (tipicamente N 2). - Nel plot (t D) contro In (Ω C) (Fig. 4A).
- La curva risultante corrisponde alla seguente equazione:
 I parametri X e A può essere estratta inserendo la trama di una relazione lineare. La X pendenza corrisponde al fattore di proporzione esponenziale e A rappresenta il fit-determinata costante.
I parametri X e A può essere estratta inserendo la trama di una relazione lineare. La X pendenza corrisponde al fattore di proporzione esponenziale e A rappresenta il fit-determinata costante. - Calcolare il coefficiente di correlazione misura r 2, con la formula di Pearson:
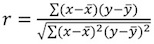 . Valori accettabili per r 2 sono maggiori di 0,95 (Fig. 4B). Un valore più basso coefficiente di correlazione può essere dovuta a:
. Valori accettabili per r 2 sono maggiori di 0,95 (Fig. 4B). Un valore più basso coefficiente di correlazione può essere dovuta a: - Svolgimento incompleta del calibranti proteine. Questo porterà a picco a causa di allargare l'assemblaggio eterogeneo di stati intermedi.
- Nella nostra esperienza, un campione di età compresa possono deteriorare gli spettri IM.
- Dissimili condizioni sperimentali utilizzate per le diverse proteine calibrante. In questo caso, riportando i dati per ogni proteina separatamente dovrebbe generare diversi coefficienti di correlazione, anche se ciascuno di essi deve essere superiore a 0,95.
- Dati rumorosi e scorretta levigatura e centraggio della distribuzione del tempo di deriva.
- Calcolo degli errori.
- Recorrect il tempo calibrante deriva utilizzando il fattore determinato esponenziale, X, derivato al punto 7:
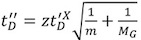
- In una fase di validazione, replot Ω C vs
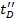 e definire il coefficiente di correlazione. Valori superiori a 0,95 sono previsti.
e definire il coefficiente di correlazione. Valori superiori a 0,95 sono previsti. - Secondo la procedura descritta al punto 4, correggere il tempo misurato deriva della proteina bersaglio o complesso di proteine:
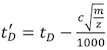
- Calibrare il tempo di deriva del complesso bersaglio proteina / proteina utilizzando il fattore esponenziale, X, definito al punto 7:
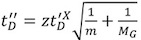
- Calcolare Ω del complesso bersaglio proteina / proteina utilizzando il fit-determinata costante, A, definito al punto 7:
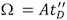 .
. - Per ogni condizione sperimentale, passaggi da 2 a 13 deve essere ripetuto. Nel definire l'area della sezione trasversale della proteina sconosciuta o complesso di proteine, si consiglia di ogni esperimento da ripetere almeno tre volte, e la deviazione standard di queste misurazioni triplice copia determinato.
Parte 4: Definizione dei valori di tempo di deriva
Software richiesto: MassLynx e Driftscope (Waters).
- Aprire l'IM-MS spettro utilizzando il software Driftscope.
- Dal menu principale selezionare View e Cromatogramma deselezionare, tempo di deriva e Spectrum (opzionale), lasciando attiva solo la visualizzazione mappa 2D tempo di deriva vs m / z.
- Dalla barra dei menu, scegliere Visualizza | Opzioni | Pannello Visualizza editor e regolare i valori di soglia di intensità di ridurre al minimo il rumore di fondo (in molti casi, questa impostazione può essere definito come% Min = 30-40 e Max = 100 conteggi%).
- Scegliere Visualizza | Mappa 2D scala di intensità, e appariranno tre opzioni: scala lineare, Log Scala e Scala radice quadrata. Una scelta di scala logaritmica (dati logaritmica trasforma) in grado di comprimere il codice del colore di intensità, e consentire la simultanea comparsa di una vasta gamma di intensità (in contrasto con le opzioni di radice quadrata e lineare, con cui solo i picchi più intensi sarà visibile) .
- Dal pannello di barra degli strumenti, utilizzare lo strumento pulsante di selezione. Questa opzione consente di attivare le varie opzioni di selezione, e consentire la selezione della regione interessata all'interno dello spettro. Lo strumento più preciso è Attiva Regione di selezioni interesse, per mezzo del quale un confine può essere tracciato intorno alla regione di interesse, escludendo così tutti i dati necessari e picchi di rumore. Allo stesso modo, le opzioni di selezione ortogonali e Band sono utili, quando la regione di interesse non è circondato da cime ridondanti.
- Una volta che la regione di interesse è selezionata, utilizzare il comando Accetta corrente selezione di rimuovere le informazioni non necessarie.
- Esportare i dati in MassLynx, pur mantenendo le informazioni tempo di deriva.
- All'interno MassLynx, aprire il cromatogramma dello spettro di deriva di risparmiare tempo, e combinare i bidoni tempo. Lo spettro di massa corrispondente si aprirà automaticamente.
- Applicare la funzione di lisciare definendo le dimensioni della finestra e il numero di parametri liscio (dovrebbe essere regolato in modo specifico per ogni spettro, utilizzando i valori minimi).
- Applicare sottrazione di base, se necessario.
- Centro dello spettro e misurare la massa, per convalidare l'identità delle proteine e la precisione di massa.
- Per ogni stato di carica, combinare la m / z gamma. Il corrispondente spettro di tempo di deriva apparirà automaticamente. Liscio e il centro del tempo deriva profilo, e definire il valore del tempo deriva indicando il baricentro di ogni picco.
Parte 5: Risultati Rappresentante

Figura 1. Rappresentazione schematica dello strumento Synapt HDMS indicando i parametri principali sintonizzabile di IMS-MS acquisizione. Parametri sperimentali utilizzati per IM-MS misurazioni sono etichettati in base alla loro posizione all'interno dello strumento. Il fascio di ioni è colorato in rosso, e la pressione in ogni regione è designato con un codice a colori. Il pannello in basso mostra il gradiente di potenziale lungo lo strumento e le differenze di potenziale che definiscono la trappola e le energie di collisione di trasferimento così come il potenziale di Bias. Tutte le potenzialità di lettura spalle si fa riferimento alla tensione di offset statico che di solito è impostato su 120V.

Figura 2. Mobilità ionica tempo distribuzioni arrivo per la proteina Gβυ.
A. Una elevata velocità dell'onda T-porta ad una stretta distribuzione del profilo tempo di deriva. La trama illustra la distribuzione del tempo di arrivo Stati di carica 16 + (rosso), 15 + (verde), 14 + (blu), e 13 + (magenta), così come il profilo tempo totale di deriva (in nero) della proteina G βυ.
B. Una volta ottimizzato spettro deriva con una superficie liscia forma del picco gaussiano. Etichette colore simile in A.
C. Un 'roll-over' effetto, che si verifica quando il tempo impiegato per gli ioni di attraversare la cellula mobilità è più lento rispetto l'intervallo tra le iniezioni di nuovi pacchetti di ioni nel dispositivo. Di conseguenza, esteso picco tempo di deriva appare all'inizio dello spettro. Questo effetto può essere eliminato aumentando la T-wave altezza, e diminuendo la velocità dell'onda T e pressione IMS.
D. artificiale 'onde' sono causati quando il T-velocità dell'onda di trasferimento e la frequenza pusher sono parzialmente sincronizzati. Questo effetto può essere superato regolando sia la frequenza pusher o Bonifico T velocità dell'onda.
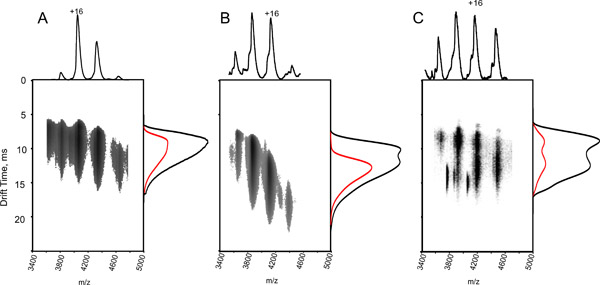
Figura 3. L'effetto di attivazione di ioni e condizioni di parziale denaturazione su IM-MS spettri di emoglobina. Plot del tempo di deriva rispetto a m / z per il complesso emoglobina tetramerica, utilizzando una soluzione acquosa di 10 mM ammonio acetato (pH = 7.6) (A, C) e l'aggiunta di 0,1% di acido acetico (B). I dati acquisiti mediante collisione tensione trappola energetica di 13 V (A, B) e 35 V (C) Anche se in tutti e tre i pannelli lo spettro di massa (proiettata in alto) è simile, con una serie di carica tetramerica centrato a 4.000 m / z, il profilo temporale deriva (proiettato sui lati) è diverso (per un totale di distribuzione è tempo di derivain nero, e il 16 profilo + è in rosso). Il tempo di deriva più lunga del campione parzialmente denaturato, ottenuta in B, e la fase gassosa ioni attivati, ottenuto in C, è indicativo di un certo grado di dispiegarsi. Questa osservazione dimostra che anche se la massa misurata corrisponde a un complesso intatta, la sua struttura soluzione è interrotto. Di conseguenza, attento controllo delle condizioni sperimentali è richiesto.
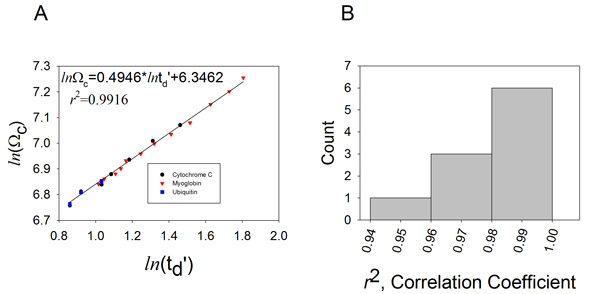
Figura 4. Generando una curva di calibrazione, la deriva misure di tempo e di collisione sezioni possono essere correlati.
A. Valori deriva momento gli stati di carica multipla di equino citocromo C (circoli), il cuore mioglobina cavallo (triangoli) e bovina ubiquitina (quadrati) sono state rilevate in letteratura i valori corretti per Ω sia stato ioni di carica e massa ridotta. L'adattamento produce una funzione lineare, corrispondente: ln (Ω C) = XLN (t D ') + A. Il fattore determinato esponenziale (X), fit-determinata costante (A), e il coefficiente di correlazione vengono visualizzati sul grafico per i dati acquisiti in un T-velocità dell'onda di 350 m / s, e una altezza d'onda statica di 11 V. B. Un istogramma della distribuzione del coefficiente di correlazione ottenuto da 10 esperimenti di calibrazione consecutivi.
| Proteine del campione / Parametri tecnici | GluFibrino- peptide monomero 1,6 kDa | Mioglobina monomero 17 kDa | Emoglobina tetramero 67 kDa | Transferrina monomero 80 kDa | GroEL 14-mer 801 kDa |
| Supporto di pressione, mBar | 4,4 | 5.0 | 5,1 | 5,1 | 6,5 |
| Trappola di pressione, mBar | 1.6x10 -2 | 2.4x10 -2 | 2.4x10 -2 | 2.6x10 -2 | 2.8x10 -2 |
| IMS pressione, mBar | 4.4x10 -1 | 4.4x10 -1 | 4.4x10 -1 | 4.4x10 -1 | 4.2x10 -1 |
| Campionamento tensione di cono, V | 46 | 80 | 80 | 80 | 118 |
| Estrazione tensione di cono, V | 1,7 | 1 | 1 | 1 | 3 |
| Bias di tensione, V | 20 | 20 | 25 | 25 | 50 |
| Trappola energia di collisione, V | 20 | 15 | 15 | 15 | 80 |
| Trasferimento di energia di collisione, V | 5 | 12 | 12 | 12 | 15 |
Tabella 1. Condizioni sperimentali utilizzati per l'analisi di macromolecole.
| Proteine di serie | Massa molecolare (m) | Oneri (z) | m / z | Collisione sezione trasversale (in 2) |
| Citocromo C | 12213 | 10 | 1222,3 | 2226 |
| 11 | 1111,3 | 2303 | ||
| 12 | 1018,8 | 2335 | ||
| 13 | 940,5 | 2391 | ||
| 14 | 873,4 | 2473 | ||
| 15 | 815,2 | 2579 | ||
| 16 | 764,3 | 2679 | ||
| 17 | 719,4 | 2723 | ||
| 18 | 679,5 | 2766 | ||
| Mioglobina | 16952 | 11 | 1542,1 | 2942 |
| 12 | 1413,7 | 3044 | ||
| 13 | 1305,0 | 3136 | ||
| 14 | 1211,9 | 3143 | ||
| 15 | 1131,1 | 3230 | ||
| 16 | 1060,5 | 3313 | ||
| 17 | 998,2 | 3384 | ||
| 18 | 942,8 | 3489 | ||
| 19 | 893,2 | 3570 | ||
| 20 | 848,6 | 3682 | ||
| 21 | 808,2 | 3792 | ||
| 22 | 771,6 | 3815 | ||
| Ubiquitina | 8565 | 8 | 1071,6 | 1442 |
| 8 | 1071,6 | 1622 | ||
| 9 | 952,7 | 1649 | ||
| 10 | 857,5 | 1732 | ||
| 11 | 779,6 | 1802 | ||
Tabella 2. Calibrante proteine e la loro collisione sezioni i valori determinati dal convenzionale tranne del IMS 14.
| Dispositivi | Azienda | Numero di catalogo |
| Synapt HDMS-32K generatore RF | Acque Ltd. | |
| P-97 Flaming-Brown micropipetta estrattore | Sutter Instruments | P-97 |
| Sputter coater | Electron Microscopy Sciences | EMS550 |
| Microscopio binoculare | Nikon | |
| Reagenti | Azienda | Numero di catalogo |
| Acetato di ammonio | Sigma-Aldrich | Sigma, A2706 |
| CsI 99,999% | Sigma-Aldrich | Aldrich, 203033 |
| Metanolo | Sigma-Aldrich | Fluka, 34966 |
| Acido acetico | Fisher Scientific | AC12404 |
| Equine mioglobina (da cuore a cavallo) | Sigma-Aldrich | M1882 |
| Equine citocromo c (da cuore a cavallo) | Sigma-Aldrich | C-2506 |
| Bovina ubiquitina (dai globuli rossi) | Sigma-Aldrich | U6253 |
| Emoglobina | Sigma-Aldrich | H2625 |
| Gas | Commenti | |
| Azoto, purezza del 99.999% | 8 cilindri metro cubo | |
| Argon, 99,999% puro | 8,8 meterscylinder cubi | |
Tabella 3. Reagenti e attrezzature.
Discussione
Il protocollo qui descritto consente di definire la sezione trasversale collisione di proteine o complessi proteici con uno sconosciuto struttura tridimensionale, con l'obiettivo di fornire informazioni sulla loro forma complessiva, l'imballaggio subunità e topologia. A tal fine, una volta valori urto sezione sono descritti, è necessario convertire questi valori per i dettagli strutturali. Questo processo richiede ulteriori sforzi sperimentali e analisi computazionale, che sono brevemente discussi di seguito.
Per cominciare, si consiglia di analizzare le proteine o complessi di proteine con strutture conosciute. Queste misure possono fornire un utile controllo qualità della metodologia e consentirà la valutazione accuratezza dei parametri di acquisizione confrontando teorici e valori misurati Ω. Il teorico aree delle sezioni trasversali possono essere calcolati dalla struttura cristallina coordinate utilizzando il MOBCAL 15,16 software, che è un software open source basato FORTRAN che permette la modifica del codice in base alle esigenze dell'operatore. Per l'esecuzione di tali calcoli è richiesto di modificare il programma in modo che il numero di calcoli iterativi eseguite per ogni struttura di ingresso è aumentata e che i file contenenti coordinare gran numero di atomi sono accettate 1.
IM-MS strategia per la definizione di accordi topologica della subunità all'interno di assemblee multicomponente è stato recentemente proposto 4,6. Il metodo prevede il monitoraggio dei percorsi di dissociazione delle assemblee proteina di componenti più piccoli. Questa dissociazione si ottiene attraverso la regolazione controllata di condizioni fase soluzione, che dà luogo ad una distribuzione di subcomplessi riflettente del "mattoni" delle assemblee. La misurazione simultanea di valori Ω sia del complesso intatto e prodotti smontaggio genera vincoli strutturali che vengono poi utilizzati per il calcolo dei modelli topologica dei complessi proteici. Il presupposto fondamentale di questa metodologia è che il subcomplessi generato conservano la loro madrelingua come conferme, e infatti recenti studi hanno dimostrato che la struttura soluzione dei prodotti smontaggio viene mantenuta e nessun riassetto importanti in entrambe le fasi di soluzione o di gas si sono verificati 4,6.
L'ultimo passo nella cessione della struttura quaternaria di ioni in fase gassosa complesso proteico si addice ai valori sezione collisione ai modelli generati al computer. Approcci di modellazione sono impiegati al fine di esplorare le diverse possibili soluzioni topologiche di subunità e il loro in silico Ω valori sono calcolati e confrontati con quelli sperimentali. Attualmente solo alcuni approcci computazionali sono utilizzati, come il spheretype grana grossa metodo che approssima il diametro della subunità 1,8. Nel complesso, questo campo è ancora agli inizi e l'ulteriore sviluppo è necessario per rendere questo approccio generico ed applicabile ad una vasta gamma di complessi.
Divulgazioni
Riconoscimenti
Gli autori ringraziano i membri del gruppo di Sharon per la loro revisione critica e per il loro contributo al manoscritto. Siamo grati per il sostegno dei Programmi Morasha e Bikura, la Israel Science Foundation (Grant numeri 1823-1807 e 378/08), Josef Cohn Minerva Centro per la Ricerca Biomembrane, il Programma per la Famiglia Chais Fellows nuovi scienziati, Abramo e Sonia Rochlin Fondazione, la famiglia Wolfson Charitable Trust, la Helen e Milton A. Kimmelman Centro per la Struttura Biomolecolari e dell'Assemblea, la tenuta di Shlomo e Sabine Beirzwinsky; Meil de Botton Aynsley, e Karen Siem, Regno Unito.
Riferimenti
- Ruotolo, B. T. Ion mobility-mass spectrometry analysis of large proteincomplexes. Nat Protoc. 3 (7), 1139-1152 (2008).
- Scarff, C. A., Thalassinos, K., Hilton, G. R., Scrivens, J. H. Travelling wave ion mobility mass spectrometry studies of protein structure: biological significance and comparison with X-ray crystallography and nuclear magnetic resonance spectroscopy measurements. Rapid Commun Mass Spectrom. 22 (20), 3297-3304 (2008).
- Smith, D. P. Deciphering drift time measurements from travelling wave ion mobility spectrometry-mass spectrometry studies. Eur J Mass Spectrom (Chichester, Eng). 15 (2), 113-130 (2009).
- Leary, J. A. Methodology for measuring conformation of solvent-disrupted protein subunits using T-WAVE ion mobility MS: an investigation into eukaryotic initiation factors. J Am Soc Mass Spectrom. 20 (9), 1699-1706 (2009).
- Lorenzen, K. Determination of stoichiometry and conformational changes in the first step of the P22 tail assembly. J Mol Biol. 379 (2), 385-396 (2008).
- Pukala, T. L. Subunit architecture of multiprotein assemblies determined using restraints from gas-phase measurements. Structure. 17 (9), 1235-1243 (2009).
- van Duijn, E. Chaperonin complexes monitored by ion mobility mass spectrometry. J Am Chem Soc. 131 (4), 1452-1459 (2009).
- Ruotolo, B. T. Evidence for macromolecular protein rings in the absence of bulk water. Science. 310 (5754), 1658-1661 (2005).
- Ruotolo, B. T., Robinson, C. V. Aspects of native proteins are retained in vacuum. Curr Opin Chem Biol. 10 (5), 402-408 (2006).
- Giles, K. Applications of a travelling wave-based radio-frequency-only stacked ring ion guide. Rapid Commun Mass Spectrom. 18 (20), 2401-2414 (2004).
- McKay, A. R., Ruotolo, B. T., Ilag, L. L., Robinson, C. V. Mass measurements of increased accuracy resolve heterogeneous populations of intact ribosomes. J Am Chem Soc. 128 (35), 11433-11442 (2006).
- Ruotolo, B. T. Ion mobility-mass spectrometry reveals long-lived, unfolded intermediates in the dissociation of protein complexes. Angew Chem Int Ed Engl. 46 (42), 8001-8004 (2007).
- Morton, V. L., Stockley, P. G., Stonehouse, N. J., Ashcroft, A. E. Insights into virus capsid assembly from non-covalent mass spectrometry. Mass Spectrom Rev. 27 (6), 575-595 (2008).
- Valentine, S. J., Counterman, A. E., Clemmer, D. E. A database of 660 peptide ion cross sections: use of intrinsic size parameters for bona fide predictions of cross sections. J Am Soc Mass Spectrom. 10 (11), 1188-1211 (1999).
- Mesleh, M. F. Structural information from ion mobility measurements: effects of the long-range potential. J Phys Chem. 100, 16082-16086 (1996).
- Shvartsburg, A. A., Jarrold, M. F. An exact hard-spheres scattering model for the mobilities of polyatomic ions. Chem Phys Lett. 261, 86-91 (1996).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon