Method Article
L'onde T de mobilité ionique et spectrométrie de masse: Procédures de base expérimentale pour l'analyse de protéines complexes
Dans cet article
Résumé
De mobilité ionique et spectrométrie de masse est une société émergente en phase gazeuse technologie qui sépare les ions, basée sur leur collision section transversale et la masse. La méthode fournit des informations tridimensionnelles sur la topologie et la forme générale de complexes protéiques. Ici, nous présentons une procédure de base pour réglage de l'appareil et l'optimisation, l'étalonnage du temps de dérive, et l'interprétation des données.
Résumé
De mobilité ionique (IM) est une méthode qui mesure le temps pris pour un ion de voyager à travers une cellule de pression sous l'influence d'un champ électrique faible. La vitesse avec laquelle les ions traversent la région de dérive dépend de leur taille: les gros ions connaîtront un plus grand nombre de collisions avec le gaz inerte de fond (généralement N 2) et donc voyager plus lentement à travers le dispositif de messagerie instantanée que ceux des ions qui composent une petite section transversale. En général, le temps qu'il faut pour les ions de migrer si la phase de gaz dense qui les sépare, selon leur collision section transversale (Ω).
Récemment, la spectrométrie de IM a été couplée à la spectrométrie de masse et un voyage-onde (onde T) Synapt mobilité ionique du spectromètre de masse (IM-MS) a été libéré. Intégration spectrométrie de masse avec la mobilité des ions permet une dimension supplémentaire de séparation des échantillons et la définition, ce qui donne un spectre en trois dimensions (masse à temps de charge, l'intensité et la dérive). Cette technique de séparation permet le chevauchement spectral à diminuer, et permet la résolution des complexes hétérogènes avec une masse très semblable, ou masse-charge des ratios, mais les temps de dérive différent. Par ailleurs, les mesures de temps de dérive de fournir une couche importante d'informations structurelles, comme Ω est liée à la forme générale et la topologie de l'ion. La corrélation entre les valeurs mesurées du temps de dérive et Ω est calculée en utilisant une courbe de calibration générée à partir des protéines calibrant avec défini des sections 1.
La puissance de l'approche MI-MS réside dans sa capacité à définir l'emballage sous-unité et la forme globale des assemblages de protéines à des concentrations micromolaires, et proche des conditions physiologiques 1. Plusieurs études récentes de messagerie instantanée à la fois des protéines individuelles 2,3 et complexes protéiques non-covalentes 4-9, démontré avec succès que la structure des protéines quaternaires est maintenue dans la phase gazeuse, et a souligné le potentiel de cette approche dans l'étude des assemblages protéiques de géométrie inconnue . Ici, nous fournir une description détaillée de l'IMS-MS analyse des complexes protéiques en utilisant les Synapt (quadripôle-Ion Mobility-Time-of-Flight) instrument de HDMS (Waters Ltd; le seul commerciales IM-MS instruments actuellement disponibles) 10. Nous décrivons les étapes d'optimisation de base, l'étalonnage de la collision des sections, et les méthodes de traitement des données et leur interprétation. L'étape finale du protocole décrit les méthodes de calcul des valeurs théoriques Ω. Globalement, le protocole ne vise pas à couvrir tous les aspects de la GI-MS caractérisation des assemblages protéiques, mais plutôt son objectif est de présenter les aspects pratiques de la méthode à de nouveaux chercheurs dans le domaine.
Protocole
La procédure que nous décrivons se concentre uniquement sur la GI-SM analyse des complexes protéiques. Par conséquent, nous suggérons que les chercheurs connaissent pas le domaine de la structure MS référer aux étapes de préparation d'échantillon, étalonnage des instruments et des MS et MS tandem procédures d'optimisation décrit dans Kirshenbaum et al. Http://www.jove.com/index/details 2009. stp? ID = 1954. En général, ce protocole implique de faibles concentrations micromolaires des complexes (1-20 uM) dans un tampon volatils tels que l'acétate d'ammonium (0,005 - 1 M, pH 6-8). Etant donné que 1-2 pl sont consommées par nanodébit capillaire, nous vous suggérons de 10-20 ul comme un volume minimal, pour permettre l'optimisation des conditions de la SEP.
Partie 1: Acquisition d'un spectre de spectrométrie de mobilité ionique de masse
- Régler le spectromètre de masse sur les modes de fonctionnement suivants: Mobilité-TOF, l'acquisition d'ions positifs, et V-mode.
- Allumez tous les gaz (API, Piège et IMS). Nous utilisons N 2 pour la séparation de messagerie instantanée, et Ar pour le piège / de transfert. Recommandée valeurs initiales sont de 1,5 ml / min pour la région piège, et un débit de gaz de 24 ml / min pour le périphérique d'IMS.
- Définissez la plage d'acquisition m / z. Pour un complexe protéine inconnue, nous suggérons l'utilisation initiale d'une large gamme de masse, qui peut ensuite être ramenée à la valeur désirée. En parallèle, ajuster le profil de MS pour une efficacité maximale de transmission. Pour de grands complexes, la gamme de masse d'acquisition devrait être réglée de 1000 - 32 000 m / z, et le profil MS sur Auto. Dans le cas contraire le profil peut être configuré selon le tableau suivants:
m / z habitent (%) rampe (%) 960 10 20 3200 30 40 10667 - Vérifiez le réglage RF et, si nécessaire, ajuster les valeurs appropriées pour les complexes protéiques, donc:
Source Piège IMS Transfert RF Offset 450 380 380 380 RF Gain 0 0 0 0 Limite RF 450 380 380 380 - Appliquer la tension capillaire (1,050-1,400 V) et la pression nanodébit faible (0,00 à 0,03 bar). Une fois de pulvérisation est initiée, essayez de réduire la pression nanodébit à une valeur minimale. En outre, d'ajuster la position de la capillarité, par rapport à la conique.
- Ajustez les paramètres d'acquisition MS, d'acquérir un spectre bien résolu MS: le gradient de pression le long de l'instrument, et le cône d'échantillonnage ainsi que le cône d'extraction, de partialité, de piéger et de transfert de paramètres possibles, devraient tous être optimisés (détaillés dans le associées JoVE protocole Kirshenbaum et al. https://www.jove.com/index/details.stp?ID=1954 2009). Bien que ces paramètres sont dépendants de l'échantillon-, les conditions, nous avons utilisé pour l'acquisition de spectres MS de masses d'ions différents, à partir de peptide à des complexes de protéines, sont décrits dans le tableau 1, (voir aussi Fig. 1). Afin de minimiser l'activation du complexe de tenter de réduire progressivement (par pas de 10 V ~) Tensions le cône de l'échantillon, Piège et de partialité, sans changer la position de la pointe.
- Une fois un spectre de masse optimale est obtenue le profil temporel de dérive doit être ajustée. Lors de l'analyse des assemblages protéiques, des conditions optimales pour la masse et la mobilité des mesures sont souvent incompatibles, c'est pourquoi il est important de trouver le bon équilibre entre les deux. Dans l'ensemble, l'intrigue de mobilité ionique doit être optimisé de telle sorte que les sommets sont répartis sur la plage de temps de dérive entière, et le profil de pointe est lisse, approcher une distribution gaussienne (figure 2A, 2B). Pic important asymétrie peut être liée à une mauvaise séparation des conformations multiples.
- En règle générale, trois paramètres, la vitesse de l'onde T, T-hauteur des vagues et le taux d'IMS flux de gaz peut être réglé pour optimiser la séparation de la mobilité. L'augmentation de la vitesse de l'onde T va élargir le profil de distribution de dérive du temps, alors que l'augmentation des valeurs de hauteur de l'onde T va le réduire. De même, l'augmentation du débit de gaz d'IMS se déplacera le profil temporel dérive vers des valeurs plus élevées (le débit de gaz minimum IMS devrait être de 10 ml / min). Nous suggérons de laisser deux des trois variables disponibles fixes, et l'optimisation des tiers jusqu'à ce que le spectre IM est bien résolu (figure 2B). À cette fin, régler la vélocité de l'onde T et le flux de gaz à 250 m / s et 24 ml / min, rérespectivement. Puis, comme un point de départ, définissez la hauteur de l'onde T à 3 V et, de manière progressive, de l'augmenter par incréments de 1 V. En général, les grands ions faudra hauteurs de vagues supérieures. Habituellement, il n'est pas nécessaire de modifier la pression de l'IMS; cependant, quand des tensions de polarisation élevées sont nécessaires pour le tuning, la réduction des gaz IMS permettra une diminution de la valeur tension de polarisation et, par conséquent, une réduction de l'activation de protéines complexes. Globalement, la résolution maximale de 10-12 t / At peut être atteint.
- Lorsque les conditions ne sont pas optimisés (faible hauteur de l'onde T ou haute vitesse de l'onde T et / ou une pression haute IM), les ions ne traversent à travers le dispositif de messagerie instantanée de manière efficace, et leur voyage peut durer plus longtemps que le temps requis pour l'ion suivante paquet à être libérés dans la cellule de mobilité. En conséquence, un paquet d'ions nouvelles seront libérés de la région piège avant que le paquet précédent a été livré à la région poussoir. Cela conduira à un «roll-over" effet, dans lequel le pic observé dans la première partie du spectre de temps de dérive est identique à celle des ions dans le bord de résidus (figure 2C). Cet artefact peut être éliminée en augmentant la hauteur de l'onde T, et en diminuant la vitesse de l'onde T et la pression d'IMS. En outre, le temps de libération piège peut être ajustée. Par ailleurs, il est important de valider que le transfert de l'onde T de hauteur est fixée à au moins 5 V. Pour éviter les fuites d'ions vers la cellule IMS, nous recommandons que la hauteur de mobilité piège être maintenu à des niveaux maximum (30 V).
- Faible vitesse et de grande amplitude du transfert de T-ondes peut conduire à la «ondulant» du profil de distribution de dérive du temps (Fig. 2D). Cet artefact se produit lorsque la séparation de mobilité des ions (ions d'arrivée / de dérive de temps) n'est pas maintenue grâce au transfert et régions ToF, en raison de la synchronisation partielle entre la fréquence poussoir et le transfert de l'onde T de vitesse. Afin d'éliminer cet effet, ni le temps pousseur ou le transfert de l'onde T de vitesse doit être ajustée. Comme la fréquence poussoir est lié à la gamme de masse, cet artefact peut réapparaître lorsque ce paramètre est modifié. T-hauteur des vagues exerce un effet mineur, mais sa réduction peut aussi aider à éliminer les rides.
- Une fois les paramètres précités sont optimisés, les données IM-MS peut être acquise.
Partie 2: Dépistage des conditions expérimentales pour assurer des mesures de mobilité des structures natives
Pour atteindre des pics à haute résolution MS, complexes protéiques sont souvent activés dans le spectromètre de masse, afin de promouvoir le décapage de l'eau résiduelle et des composants du tampon 11. Cependant, si l'énergie d'activation est augmentée au-delà d'un seuil, un dépliement partiel peut être induite formant de multiples états intermédiaires 12, qui sont peu susceptibles de correspondre à la maternelle, la solution de l'état la structure (Fig. 3A-C). En conséquence, le pic de temps de dérive peut être déplacé et élargi, ce qui reflète la population hétérogène des structures déplié.
Afin d'obtenir des données en temps compatible avec la dérive phase solution des structures, il est essentiel de contrôler soigneusement les tensions utilisées pour les ions accélération, avant la séparation de messagerie instantanée. Par ailleurs, pour haute résolution MS, il est préférable d'augmenter le transfert plutôt que la tension Piège. Comme le dispositif de messagerie instantanée est positionné, en premier, suivie par la région de transfert et de l'analyseur TOF, par conséquent, l'activation suit la mesure IM et les ions reste inchangée, tandis que la précision MS peut être augmenté.
Afin de valider que l'acquisition des données est effectuée dans des conditions qui maintiennent la structure native de la complexité, il est recommandé que les données soient enregistrées sur une gamme de conditions expérimentales et la solution, plutôt que selon un seul ensemble de paramètres optimisés:
- Augmenter les tensions capillaires et le cône de manière progressive, tout en surveillant l'effet sur le spectre en temps de dérive.
- Comme dans l'étape 1, augmenter la tension de collision Piège de manière progressive, et d'acquérir des données à intervalles de 10 V.
- Pour identifier les conformations déplié et évaluer les données acquises, manuellement induisent la dissociation du complexe protéique par titrage de l'échantillon avec l'acide acétique sur une gamme de pH de 2-7, et enregistrer les données (figure 3B).
Partie 3: Corrélation entre les valeurs du temps de dérive et transversale des domaines
Contrairement à des mesures de messagerie instantanée classique, dans lequel les valeurs mesurées sont linéairement temps de dérive liés à Ω, dans le système de l'onde T IMS, la section transversale est définie par une approche d'étalonnage. Ainsi, plutôt que comme une mesure absolue, une corrélation relativement exponentielle est généré entre le temps de dérive mesurée et Ω 1,13: 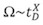
D où t est le temps de dérive mesurée, et X est la constante de la proportion qui peut être extraite à partir d'une courbe de calibration. L'étalonnage est d'effectuered en mesurant le temps de dérive des ions avec connues Ω (mesurée à partir d'expériences de messagerie instantanée classique).
- Mesures de temps de dérive sont calibrés en utilisant des protéines dénaturées équine cytochrome C, la myoglobine de coeur de cheval et de bovin ubiquitine avec les collisions connues sections. À cette fin, les solutions de 10 uM dans 49/49/2, le ratio du volume, de l'eau / méthanol / acide acétique doit être préparé (réactifs utilisés sont décrites dans le tableau 3).
- Acquérir IM-données MS pour les protéines calibrant exactement dans les conditions même instrument utilisé pour la protéine cible ou complexes protéiques:
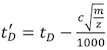 (Partie 1). Toutes les tensions et les valeurs de pression doivent être identiques, afin de préserver les paramètres de séparation de messagerie instantanée.
(Partie 1). Toutes les tensions et les valeurs de pression doivent être identiques, afin de préserver les paramètres de séparation de messagerie instantanée. - Pour chaque état de charge des protéines calibrant extraire la valeur de la dérive expérimentale temps (t D) (voir partie 4).
- Corrigez chacun des temps de dérive calibrant (t D) (tableau 2) en utilisant l'équation suivante:
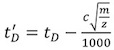 , Où m / z est le rapport masse-charge de l'ion observé, et c est le Cycle d'Amélioration (EDC) 1 coefficient de retard. Sa valeur, généralement entre 1,4 et 1,6, est l'instrument-dépendante. La valeur d'EDC est indiqué dans le Système | Paramètres Acquisition | Acquisition onglet Configuration.
, Où m / z est le rapport masse-charge de l'ion observé, et c est le Cycle d'Amélioration (EDC) 1 coefficient de retard. Sa valeur, généralement entre 1,4 et 1,6, est l'instrument-dépendante. La valeur d'EDC est indiqué dans le Système | Paramètres Acquisition | Acquisition onglet Configuration. - En utilisant le professeur David Clemmer Croix-section Base de données:
http://www.indiana.edu/ ~ Clemmer / Recherche / cross% 20L'article% 20database/Proteins/protein_cs.htm 14 correcte de chacun des calibrant section à la fois pour l'état de charge des ions et une masse réduite.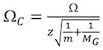
, Où Ω C est la correction de section, Ω est la littérature de section, z est l'état de charge ionique, m est la masse moléculaire de l'ion calibrant, et M G est le poids moléculaire du gaz de fond IM (généralement N 2). - Parcelle En (t D) contre In (Ω C) (Fig. 4A).
- La courbe obtenue correspond à l'équation suivante:
 Les paramètres X et A peuvent être extraites par ajustement de la parcelle à une relation linéaire. Le X pente correspond à la proportion du facteur exponentiel et A représente la constante aménagement déterminée.
Les paramètres X et A peuvent être extraites par ajustement de la parcelle à une relation linéaire. Le X pente correspond à la proportion du facteur exponentiel et A représente la constante aménagement déterminée. - Calculer le coefficient de corrélation R 2 ajustement, en utilisant l'équation de Pearson:
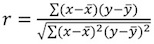 . Les valeurs acceptables pour r 2 sont supérieures à 0,95 (figure 4B). Une valeur inférieure coefficient de corrélation peut être due à:
. Les valeurs acceptables pour r 2 sont supérieures à 0,95 (figure 4B). Une valeur inférieure coefficient de corrélation peut être due à: - Incomplet déroulement de la calibrants protéines. Cela conduira à l'élargissement de pointe en raison de l'assemblage hétérogène des états intermédiaires.
- Dans notre expérience, un échantillon âgés peuvent se détériorer les spectres de messagerie instantanée.
- Dissemblables conditions expérimentales utilisées pour les différentes protéines calibrant. Dans ce cas, tracer les données séparément pour chaque protéine devrait générer des coefficients de corrélation diversifiée, même si chacun d'eux doit être supérieur à 0,95.
- Données bruitées et incorrect de lissage et de centrage de la distribution des temps de dérive.
- Erreur de calcul.
- Recorrect le temps de dérive calibrant utilisant le facteur déterminé exponentielle, X, dérivé de l'étape 7:
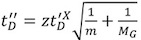
- Comme une étape de validation, replot Ω C vs
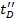 et de définir le coefficient de corrélation. Des valeurs supérieures à 0,95 sont à prévoir.
et de définir le coefficient de corrélation. Des valeurs supérieures à 0,95 sont à prévoir. - Selon la procédure décrite à l'étape 4, corriger le temps de dérive mesurée de la protéine cible ou complexes protéiques:
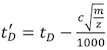
- Calibrer le temps de dérive de la cible protéine / protéine complexe en utilisant le facteur exponentiel, X, défini à l'étape 7:
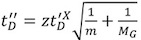
- Calculer Ω de la cible protéine / protéine complexe en utilisant la constante ajustement déterminée, A, défini à l'étape 7:
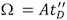 .
. - Pour chaque condition expérimentale, les étapes 2 à 13 devrait être répétée. Lors de la définition de la section transversale de la protéine inconnue ou complexe protéique, nous recommandons que chaque expérience soit répétée au moins trois fois, et la déviation standard de ces trois mesures déterminées.
Partie 4: Définition des valeurs de temps de dérive
Logiciel requis: MassLynx et Driftscope (Waters).
- Ouvrez le spectre IM-MS en utilisant le logiciel Driftscope.
- Dans le menu principal, sélectionnez ViEW et Chromatogramme décocher, Drift Time et Spectrum (en option), ne laissant actives que la carte 2D affichant le temps de dérive par rapport m / z.
- Dans la barre de menu, choisissez Affichage | Options | Panneau d'affichage éditeur et ajuster les valeurs seuil d'intensité pour minimiser le bruit de fond (dans la plupart des cas, ce paramètre peut être défini comme Min = 30-40% et Max = 100 compte%).
- Choisissez Affichage | Échelle d'intensité 2D Map, et trois options apparaissent: échelle linéaire, l'échelle et l'échelle Connexion racine carrée. Un choix de l'échelle logarithmique (données logarithmiques transformer) va compresser le code couleur intensité, et permettre l'apparition simultanée d'une large gamme d'intensités (par opposition aux options de la racine linéaire et carré, avec laquelle seuls les pics les plus intenses deviendra visible) .
- Depuis le panneau barre d'outils, utilisez le bouton Outil de sélection. Cette option va activer les options de sélection différents, et permettre la sélection de la région concernée au sein du spectre. L'outil le plus précis est Activer Région des sélections d'intérêt, au moyen de laquelle une frontière peut être établi autour de la région d'intérêt, ce qui exclut toutes les données inutiles et les pics de bruit. De même, les options de sélection orthogonale et la bande sont utiles, lorsque la région d'intérêt n'est pas entourée de sommets redondants.
- Une fois la région d'intérêt est sélectionné, utilisez la commande Accepter la sélection actuelle de supprimer les informations inutiles.
- Exporter les données à MassLynx, tout en conservant les informations de temps de dérive.
- Dans MassLynx, ouvrez le chromatogramme du spectre en temps de dérive sauvés, et de combiner les bacs le temps. Le spectre de masse correspondant s'ouvre automatiquement.
- Appliquer la fonction de lissage en définissant la taille de fenêtre et le nombre de paramètres en douceur (qui devrait être réglé spécifiquement pour chaque spectre, en utilisant les valeurs minimales).
- Appliquer base de soustraction, si nécessaire.
- Centre du spectre et mesurer la masse, pour valider l'identité des protéines et la précision de masse.
- Pour chaque état de charge, combiner la gamme de m / z. Le spectre de la dérive temporelle correspondante apparaîtra automatiquement. Lisse et le centre du profil de temps de dérive, et de définir la valeur du temps de dérive en indiquant le barycentre de chaque pic.
Partie 5: Résultats Représentant

Figure 1. Représentation schématique de l'instrument Synapt HDMS indiquant les principaux paramètres ajustables de l'IMS-MS acquisition. Les paramètres expérimentaux utilisés pour IM-SM mesures sont étiquetés en fonction de leur position au sein de l'instrument. Le faisceau d'ions est coloré en rouge, et la pression dans chaque région est désigné par un code couleur. Le panneau en bas illustre le gradient de potentiel le long du bord et les différences potentielles de définir le piège et énergies de collision de transfert ainsi que le potentiel de polarisation. Tous les potentiels lisez-backs sont référencées à la tension statique de décalage qui est généralement fixé à 120V.

Figure 2. Ion distributions de mobilité heure d'arrivée pour la protéine Gβυ.
A. Une haute vitesse de l'onde T conduit à une distribution étroite du profil de temps de dérive. L'intrigue illustre la distribution de l'heure d'arrivée États de charge 16 + (rouge), 15 + (vert), 14 + (bleu), et 13 + (magenta), ainsi que le profil temporel dérive totale (en noir) de la protéine G βυ.
B. Un spectre optimisé le temps de dérive avec un pic de forme gaussienne lisse. Étiquettes de couleur similaire à celui de A.
C. Un «roll-over 'effet, qui survient lorsque le temps pris pour des ions de traverser la cellule de mobilité est plus lent que l'intervalle entre les injections de paquets d'ions nouvelles dans l'appareil. En conséquence, le pic de dérive prolongée de temps apparaît au début du spectre. Cet effet peut être éliminé en augmentant la hauteur de l'onde T, et en diminuant la vitesse de l'onde T et la pression d'IMS.
«Ondulations» D. artificiels sont causés lorsque le transfert de l'onde T de vitesse et la fréquence pousseur sont partiellement synchronisée. Cet effet peut être surmontée en ajustant soit la fréquence pousseur ou le transfert de l'onde T de vitesse.
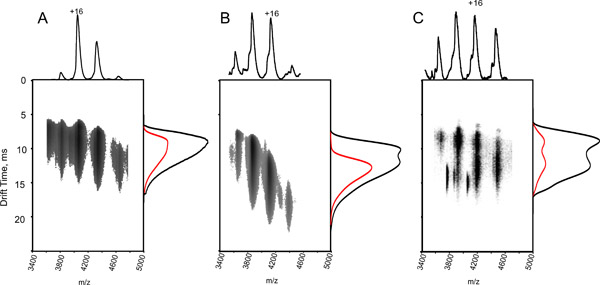
Figure 3. L'effet de l'activation partielle des ions et des conditions de dénaturation de la GI-MS spectres de l'hémoglobine. Terrain de temps de dérive par rapport m / z pour le complexe hémoglobine tétramérique, en utilisant une solution aqueuse d'acétate d'ammonium 10 mM (pH = 7,6) (A, C) et l'ajout d'acide acétique à 0,1% (B). Les données acquises en utilisant la tension de l'énergie de collision de Piège 13 V (A, B) et 35 V (C) Bien que dans les trois panneaux du spectre de masse (projetée sur le dessus) ressemble, avec une série de charge tétramérique centré à 4000 m / z, le profil temporel de dérive (projetées sur les côtés) est différente (au total distribution du temps de dérive esten noir, et le profil 16 + est en rouge). Le temps plus la dérive de l'échantillon partiellement dénaturé, obtenu en B, et les ions en phase gazeuse activé, obtenu en C, est révélatrice d'un certain degré de dévoilement. Cette observation illustre le fait que même si la masse mesurée correspond à un complexe intact, sa structure en solution est perturbé. En conséquence, un contrôle minutieux des conditions expérimentales est requise.
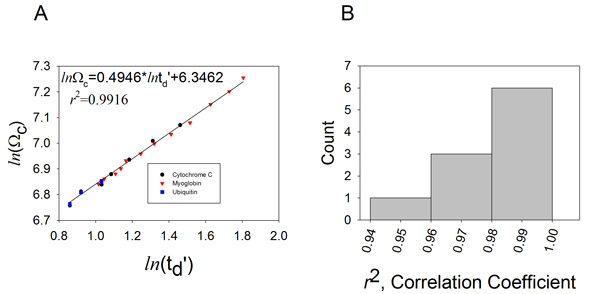
Figure 4. En générant une courbe de calibration, la dérive des mesures de temps et la collision des sections peuvent être corrélées.
A. Les valeurs mesurées du temps de dérive des états de charge multiple des équins cytochrome C (cercles), la myoglobine de coeur de cheval (triangles) et bovin ubiquitine (carrés) ont été tracées par rapport aux valeurs Ω littérature corrigé pour la fois l'état de charge des ions et une masse réduite. L'ajustement des rendements d'une fonction linéaire correspondant à: ln (Ω C) = XLN (t D ') + A. Le facteur déterminé exponentielle (X), l'ajustement déterminé constante (A), et le coefficient de corrélation sont affichés sur la parcelle pour des données acquises à une vitesse de l'onde T de 350 m / s, et une hauteur de vague statique de 11 V. B. Un histogramme des distributions coefficient de corrélation obtenu à partir de 10 expériences d'étalonnage consécutifs.
| Échantillon de protéines / paramètres techniques | GluFibrino- peptide monomère 1,6 kDa | Myoglobine monomère 17 kDa | L'hémoglobine tétramère 67 kDa | La transferrine monomère 80 kDa | GroEL 14-mer 801 kDa |
| Sauvegarde de pression, mBar | 4.4 | 5,0 | 5.1 | 5.1 | 6.5 |
| Piège de pression, mBar | 1.6x10 -2 | 2.4x10 -2 | 2.4x10 -2 | 2.6x10 -2 | 2.8x10 -2 |
| Pression d'IMS, mBar | 4.4x10 -1 | 4.4x10 -1 | 4.4x10 -1 | 4.4x10 -1 | 4.2x10 -1 |
| Tension de cône d'échantillonnage, V | 46 | 80 | 80 | 80 | 118 |
| Tension de cône d'extraction, V | 1.7 | 1 | 1 | 1 | 3 |
| Tension de polarisation, V | 20 | 20 | 25 | 25 | 50 |
| L'énergie de collision Trap, V | 20 | 15 | 15 | 15 | 80 |
| Collisions de transfert d'énergie, V | 5 | 12 | 12 | 12 | 15 |
Tableau 1. Les conditions expérimentales utilisées pour l'analyse des macromolécules.
| Protein Standard | Masse moléculaire (m) | Charges (z) | m / z | Collision section transversale (en 2) |
| Cytochrome C | 12213 | 10 | 1222.3 | 2226 |
| 11 | 1111.3 | 2303 | ||
| 12 | 1018.8 | 2335 | ||
| 13 | 940,5 | 2391 | ||
| 14 | 873,4 | 2473 | ||
| 15 | 815,2 | 2579 | ||
| 16 | 764,3 | 2679 | ||
| 17 | 719,4 | 2723 | ||
| 18 | 679,5 | 2766 | ||
| Myoglobine | 16952 | 11 | 1542.1 | 2942 |
| 12 | 1413.7 | 3044 | ||
| 13 | 1305.0 | 3136 | ||
| 14 | 1211.9 | 3143 | ||
| 15 | 1131.1 | 3230 | ||
| 16 | 1060.5 | 3313 | ||
| 17 | 998,2 | 3384 | ||
| 18 | 942,8 | 3489 | ||
| 19 | 893,2 | 3570 | ||
| 20 | 848,6 | 3682 | ||
| 21 | 808,2 | 3792 | ||
| 22 | 771,6 | 3815 | ||
| Ubiquitine | 8565 | 8 | 1071.6 | 1442 |
| 8 | 1071.6 | 1622 | ||
| 9 | 952,7 | 1649 | ||
| 10 | 857,5 | 1732 | ||
| 11 | 779,6 | 1802 | ||
Tableau 2. Protéines calibrant et de leur collision sections valeurs déterminées par measurments IMS classique 14.
| Périphériques | Société | Numéro de catalogue |
| Synapt HDMS-32K générateur RF | Waters Ltd | |
| P-97 Flaming-Brown micropipette extracteur | Sutter Instruments | P-97 |
| Sputter coucheuse | Sciences Electron Microscopy | EMS550 |
| Microscope binoculaire | Nikon | |
| Réactifs | Société | Numéro de catalogue |
| Acétate d'ammonium | Sigma-Aldrich | Sigma, A2706 |
| Csl 99,999% | Sigma-Aldrich | Aldrich, 203033 |
| Méthanol | Sigma-Aldrich | Fluka, 34966 |
| Acide acétique | Fisher Scientific | AC12404 |
| Equine myoglobine (de coeur de cheval) | Sigma-Aldrich | M1882 |
| Equine cytochrome c (de coeur de cheval) | Sigma-Aldrich | C-2506 |
| Bovine ubiquitine (à partir de globules rouges) | Sigma-Aldrich | U6253 |
| L'hémoglobine | Sigma-Aldrich | H2625 |
| Gaz | Commentaires | |
| L'azote, pur à 99,999% | 8 cylindres mètre cube | |
| Argon, pur à 99,999% | 8,8 meterscylinder cubes | |
Tableau 3. Réactifs et équipements.
Discussion
Le protocole décrit ici permet de définir la section de collision des protéines ou des complexes de protéines avec une structure en trois dimensions inconnues, dans le but de fournir des informations sur leur forme générale, l'emballage sous-unité et de la topologie. À cette fin, une fois les valeurs de collision section transversale sont représentés, il est nécessaire de convertir ces valeurs pour plus de détails structuraux. Ce processus exige des efforts supplémentaires d'expérimentation ainsi que l'analyse informatique, qui sont brièvement discutées ci-dessous.
Pour commencer, il est recommandé d'analyser les protéines ou des complexes de protéines avec des structures connues. Ces mesures peuvent fournir un contrôle de la qualité de la méthodologie utile et permettra évaluation de l'exactitude des paramètres d'acquisition en comparant les valeurs théoriques et mesurées Ω. La théorie Croix zones sectionnelle peut calculer la structure cristalline coordonne utilisant les MOBCAL 15,16 logiciel qui sont une source du logiciel open FORTRAN base permettent édition codes conformément aux besoins conducteur. Pour exécuter ces calculs, il est nécessaire de modifier le programme de telle sorte que le nombre de calculs itératifs effectués par la structure d'entrée est augmentée et que les fichiers contenant de coordonner grand nombre d'atomes sont acceptés 1.
Une stratégie de GI-MS pour la définition des arrangements topologique de sous-unités au sein des assemblées multicomposant a été récemment proposé 4,6. La méthode implique la surveillance des voies de dissociation des assemblages de protéines pour de plus petits composants. Cette dissociation est obtenue par un ajustement des conditions contrôlées en phase solution, qui donne lieu à une répartition des sous-complexes reflet de la «building blocks» des assemblées. La mesure simultanée des valeurs de Ω à la fois des complexes intacts et le démontage des produits génère des contraintes structurelles qui sont ensuite utilisés pour calculer les modèles topologiques des complexes protéiques. L'hypothèse de base de cette méthodologie est que le sous-complexes générées conservent leur native-like confirmations, et en effet des études récentes ont démontré que la structure en solution des produits de démontage est maintenue et aucun réarrangement majeur dans les deux phases de solution ou de gaz ont eu lieu 4,6.
La dernière étape dans la cession de la structure quaternaire des protéines à des ions en phase gazeuse complexe montage des valeurs de collision section transversale de modèles par ordinateur. Approches de modélisation sont employées dans le but d'explorer les différents arrangements possibles topologique de sous-unités et de leurs valeurs dans Ω silico sont calculés et comparés à ceux expérimentaux. Actuellement, seuls quelques approches de calcul sont utilisées, comme le spheretype grossiers méthode qui se rapproche du diamètre de 1,8 unités. Dans l'ensemble, ce domaine est encore à ses débuts et le développement est nécessaire pour faire de cette approche générique et applicable à un large éventail de complexes.
Déclarations de divulgation
Remerciements
Les auteurs remercient les membres du groupe de Sharon pour leur examen critique, et pour leur contribution au manuscrit. Nous sommes reconnaissants de l'appui des programmes et des Morasha Bikura, l'Israel Science Foundation (Grant 378/08 et n ° 1823-1807), Josef Cohn Minerva Centre de recherche biomembrane, la famille Chais Fellows Program pour les nouveaux chercheurs, l'Abraham et Sonia Rochlin Fondation Trust Wolfson famille de bienfaisance, le Helen et Milton A. Kimmelman Center for Structure biomoléculaire et de l'Assemblée; la succession de Shlomo et Sabine Beirzwinsky; Meil de Botton Aynsley, et Karen Siem, Royaume-Uni.
Références
- Ruotolo, B. T. Ion mobility-mass spectrometry analysis of large proteincomplexes. Nat Protoc. 3 (7), 1139-1152 (2008).
- Scarff, C. A., Thalassinos, K., Hilton, G. R., Scrivens, J. H. Travelling wave ion mobility mass spectrometry studies of protein structure: biological significance and comparison with X-ray crystallography and nuclear magnetic resonance spectroscopy measurements. Rapid Commun Mass Spectrom. 22 (20), 3297-3304 (2008).
- Smith, D. P. Deciphering drift time measurements from travelling wave ion mobility spectrometry-mass spectrometry studies. Eur J Mass Spectrom (Chichester, Eng). 15 (2), 113-130 (2009).
- Leary, J. A. Methodology for measuring conformation of solvent-disrupted protein subunits using T-WAVE ion mobility MS: an investigation into eukaryotic initiation factors. J Am Soc Mass Spectrom. 20 (9), 1699-1706 (2009).
- Lorenzen, K. Determination of stoichiometry and conformational changes in the first step of the P22 tail assembly. J Mol Biol. 379 (2), 385-396 (2008).
- Pukala, T. L. Subunit architecture of multiprotein assemblies determined using restraints from gas-phase measurements. Structure. 17 (9), 1235-1243 (2009).
- van Duijn, E. Chaperonin complexes monitored by ion mobility mass spectrometry. J Am Chem Soc. 131 (4), 1452-1459 (2009).
- Ruotolo, B. T. Evidence for macromolecular protein rings in the absence of bulk water. Science. 310 (5754), 1658-1661 (2005).
- Ruotolo, B. T., Robinson, C. V. Aspects of native proteins are retained in vacuum. Curr Opin Chem Biol. 10 (5), 402-408 (2006).
- Giles, K. Applications of a travelling wave-based radio-frequency-only stacked ring ion guide. Rapid Commun Mass Spectrom. 18 (20), 2401-2414 (2004).
- McKay, A. R., Ruotolo, B. T., Ilag, L. L., Robinson, C. V. Mass measurements of increased accuracy resolve heterogeneous populations of intact ribosomes. J Am Chem Soc. 128 (35), 11433-11442 (2006).
- Ruotolo, B. T. Ion mobility-mass spectrometry reveals long-lived, unfolded intermediates in the dissociation of protein complexes. Angew Chem Int Ed Engl. 46 (42), 8001-8004 (2007).
- Morton, V. L., Stockley, P. G., Stonehouse, N. J., Ashcroft, A. E. Insights into virus capsid assembly from non-covalent mass spectrometry. Mass Spectrom Rev. 27 (6), 575-595 (2008).
- Valentine, S. J., Counterman, A. E., Clemmer, D. E. A database of 660 peptide ion cross sections: use of intrinsic size parameters for bona fide predictions of cross sections. J Am Soc Mass Spectrom. 10 (11), 1188-1211 (1999).
- Mesleh, M. F. Structural information from ion mobility measurements: effects of the long-range potential. J Phys Chem. 100, 16082-16086 (1996).
- Shvartsburg, A. A., Jarrold, M. F. An exact hard-spheres scattering model for the mobilities of polyatomic ions. Chem Phys Lett. 261, 86-91 (1996).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon