Method Article
La recuperación de fluorescencia después de photobleaching (FRAP) de fluorescencia de proteínas marcadas en las espinas dendríticas de las neuronas del hipocampo cultivadas
En este artículo
Resumen
FRAP se ha utilizado para cuantificar la movilidad de la proteína verde fluorescente (GFP) de etiquetado proteínas en células cultivadas. Se examinaron las fracciones móviles / inmóviles de la GFP mediante el análisis del porcentaje de recuperación después de fluorescencia photobleaching. En este estudio, FRAP llevó a cabo en las espinas de las neuronas del hipocampo.
Resumen
FRAP se ha utilizado para cuantificar la movilidad de las buenas prácticas agrarias de etiquetado proteínas. El uso de un láser de excitación fuerte, la fluorescencia de la proteína GFP-etiquetados se blanquea en la región de interés. La fluorescencia de la región se recupera cuando el crudo de proteína GFP-etiquetados desde fuera de la región se difunde en la región de interés. La movilidad de la proteína se analiza mediante la medición de la tasa de recuperación de fluorescencia. Esta técnica podría ser usada para caracterizar la movilidad de la proteína y la tasa de rotación.
En este estudio, que expresan el vector (aumento de proteína verde fluorescente) EGFP en cultivos de neuronas del hipocampo. Usando el microscopio confocal Zeiss 710, que photobleach la señal de fluorescencia de la proteína GFP en una sola columna, y luego tomar imágenes de lapso de tiempo para registrar la recuperación de fluorescencia después de photobleaching. Por último, estimar el porcentaje de fracciones móviles e inmóviles de la GFP en las espinas, mediante el análisis de los datos de imagen usando ImageJ y softwares Graphpad.
Este protocolo FRAP muestra cómo realizar un experimento FRAP básica, así como la forma de analizar los datos.
Protocolo
1. Neurona transfección
- Cultura embrionarias neuronas de rata de 18 días (E18) del hipocampo en poli-D-lisina MatTek de 35 mm con fondo de cristal platos 1. En 16-18 días in vitro (DIV), las neuronas transfectar mediante el kit de Clontech CalPhos transfección de mamíferos. En primer lugar, sustituir el medio de cultivo con 1,5 ml de Dulbecco modificado del medio Eagle (DMEM) por 35 mm plato de 0,5 horas antes de la transfección. Salvar el medio de cultivo original en un tubo estéril de 15 ml para más adelante (paso 1.6) el uso.
- Mezcla de 10 mg pEGFP-N1 ADN plásmido con estériles de H 2 O (Clontech) y 12,4 l 2 M solución de calcio (Clontech) para un volumen total de 100 microlitros.
- Añadir la mezcla del paso 1.2) a 100 l 2 × HBS, gota a gota, mientras que 2 × EPF agitación a velocidad media.
- Deja reposar la mezcla a temperatura ambiente durante 20 minutos y luego agregar la mezcla final del paso 1.3) en DMEM-incubaron las neuronas.
- Poner a las neuronas de nuevo en la incubadora a 37 ° C durante 1-1,5 horas.
- Eliminar el fosfato que contienen calcio medio, lavar las células con DMEM tres veces. Antes de regresar la placa de cultivo a la incubadora, el cambio de medio DMEM con el medio de cultivo original.
2. FRAP experimento en una columna
- Las neuronas son utilizados para el experimento FRAP dos a cuatro días después de la transfección.
- Vuelva a colocar el medio de cultivo de los 35 mm con fondo de cristal plato, por añadir inmediatamente pre-calentado solución Tyrode, que contiene (en mM) NaCl 145, KCl 5, HEPES 10, glucosa 10, glicina 0,005, CaCl2 2,6, y MgCl 2 1.3 (pH ajustado a 7,4 con NaOH).
- A Zeiss LSM 710 microscopio confocal se utiliza para el experimento FRAP. El sistema TempModule Zeiss se utiliza para controlar la temperatura (37 ° C), la humedad y el CO 2 (5%) del sistema de trabajo. Asegúrese de que el cilindro de CO 2 está conectado y la botella de agua, que se utiliza para el equilibrio de humedad, se llena de agua.
- Buscar una dendrita transfectadas madura con el objetivo de 100 × (αplan-Apochromat × 100 / 1,46 de aceite). Si las células transfectadas en el plato son escasos, la búsqueda de una célula deseado con el objetivo de 40 × (Plan-Neofluar 40 × / 1,3 de aceite), y luego cambiar a los 100 × objetivo de capturar imágenes.
- Use 5 x zoom óptico y una resolución de 256 × 256 píxeles a la imagen de un pequeño trozo de la dendrita con varias espinas. Para capturar imágenes, utilizar la velocidad nominal 9 (pixel tiempo de permanencia 3,15 microsegundos) que tarda 0,5 segundos para finalizar el análisis. El agujero está en 2μm para obtener fuerte fluorescencia. Al tomar las imágenes, trate de usar la transmisión de láser de baja, por ejemplo, 5.1%, para evitar photobleaching toda la imagen.
- Seleccione la columna vertebral de su interés. En nuestro experimento, elegimos espinas de setas con sus diámetros columna jefe de ~ 1 micra.
- Para hacer un experimento FRAP, tomar 5 imágenes de control antes del blanqueo, después cloro de la columna vertebral de interés 10 veces en la transmisión nominal láser 100% [El poder de láser de argón es de 30 mW, con un 50% (15 mW) en la línea de láser de 488, y mW aproximadamente 4.3 alcanza el microscopio a través de la línea láser 488], y luego capturar una serie de imágenes inmediatamente después de la decoloración. Para este experimento, las imágenes se capturan cada 1 segundo durante 15 segundos después del blanqueamiento. El intervalo de tiempo se ajustará de acuerdo a las diferentes proteínas de la focalización y diferentes diseños experimentales (Fig. 1).
- Guardar las imágenes.
3. Análisis de los datos
- Abrir las imágenes con el software ImageJ.
- Alinee la pila de imágenes utilizando la herramienta de alineación ('pilas - arrastrando los pies', 'plugins' → → 'alinear cortes en la pila de' → 'cuerpo rígido "de" traducción "y / o) en ImageJ, de modo que la columna vertebral de interés no flota , en otras palabras - que permanece en la misma posición en la imagen.
- Medir la intensidad de fluorescencia relativa de la columna vertebral de interés (F s), una región, pero sin blanquear transfectadas (control), y una región de fondo (F b) en las imágenes lapso de tiempo, utilizando la herramienta de la "intensidad v Time Monitor 'de ImageJ (' plugins '→' pilas - arrastrando los pies '→' Intensidad v Time Monitor '). La región de control podría ser una pieza de bien enfocada dendrita distal. La región de fondo es una región no fluorescente.
- Calcular la tasa de photobleaching (r) mediante la comparación de la fluorescencia de la región de control antes de (F c0) y después (F c) photobleaching.
r = F c / F c0. - Normalizar la intensidad de la fluorescencia de la columna de destino (F) de la siguiente manera:
F = (F s - F b) / r. - Ajuste de la curva de la intensidad de la fluorescencia de la columna de destino con una ecuación de una fase exponencial de software GraphPad Prism (Fig. 2) o Other software similar.
- Calcular la fracción móvil (f m) y la fracción inmóvil (f i) por las siguientes ecuaciones:
f m = ∞ F / F 0,
donde F ∞ es la intensidad de la fluorescencia después de la recuperación completa, y F 0 es la intensidad de fluorescencia antes de photobleaching.
f i = 1 - f m.
4. Los resultados representativos:
En este estudio, llevamos a cabo un experimento FRAP en las neuronas del hipocampo madura. A 18-22 DIV, espinas de setas ya están formados. Con nuestro método, los cambios dinámicos de la intensidad de fluorescencia en una pequeña región, como una columna vertebral, se puede grabar.
Para analizar el proceso de recuperación de fluorescencia de EGFP, tomamos 5 imágenes como los controles antes del blanqueamiento y luego una imagen cada 1 segundo inmediatamente después de la decoloración durante 15 segundos. La resolución de la imagen es suficiente para el análisis cuantitativo. Los perfiles de la recuperación de la fluorescencia de etiquetado proteínas de fluorescencia son altamente reproducibles.
También brevemente se muestra cómo definir las fracciones móviles e inmóviles de una proteína de fluorescencia, usando ImageJ y el software GraphPad Prism. El método FRAP y el análisis que mostramos aquí pueden ser ampliamente utilizados en la neurociencia, la biología celular y otros estudios.

Figura 1. FRAP mediciones de EGFP fluorescencia en una columna de una neurona del hipocampo cultivadas. Las puntas de flecha roja indica el tiempo de photobleaching. Las fotografías representan la misma zona antes (Pre) y photobleaching a los 0, 1, 5, 10, 15 segundos después. La región de la columna vertebral, el control y el fondo están marcadas con las letras S, C y B, respectivamente. Las neuronas se mantienen a 37 ° C durante el experimento. Barra de escala, 1 micra.
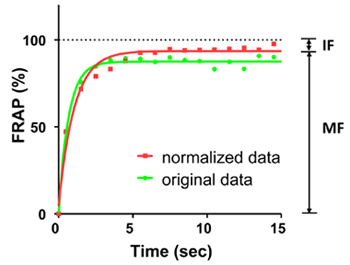
Figura 2. FRAP curvas de EGFP fluorescencia en un periodo de 15 segundos. La línea verde muestra la curva original, la línea roja muestra la curva normalizada. Los puntos en las curvas muestran el FRAP cada 1 segundo. Las curvas se ajustaron a una fase de ecuaciones exponenciales. La media de fluorescencia photobleaching antes se había contado el 100%. En este experimento, la fracción móvil (MF) es del 94% y la fracción inmóvil (SI) es del 6%.
Discusión
Análisis FRAP ha sido ampliamente utilizado in vivo e in vitro estudios 1-2. Esta técnica utiliza comúnmente GFP proteínas de fusión , aunque también se podría utilizar el rojo proteínas de fusión alga 3. Este análisis es sensible y puede ser utilizado para caracterizar la movilidad de las buenas prácticas agrarias de etiquetado proteínas.
Para producir el análisis FRAP sentido, es importante para evitar photobleaching innecesarios antes y durante el experimento FRAP. Hay dos maneras de lograr esto. En primer lugar, el proceso de buscar y observar la neurona experimental debe ser rápida. Sobre todo, la observación de las neuronas con un objetivo 100X durante mucho tiempo significativamente blanquea la fluorescencia. Segunda potencia, y el escaneo láser de alta frecuencia a menudo aumentan la posibilidad de photobleaching. Por lo tanto, será necesario calcular la tasa de photobleaching en una región de control y normalización de la curva del FRAP en la región experimental. El método de normalización se ha descrito en el protocolo (ver paso 3.3-3.5 para más detalles).
El paso photobleaching también es fundamental para garantizar buenos resultados FRAP. En este experimento, blanqueador de la columna vertebral de interés 10 veces al 100% de transmisión de láser. Esta condición es suficiente para blanquear la fluorescencia de una columna a nivel de base en una preparación fija. Por lo tanto, ajustar la intensidad de fluorescencia para el mismo número que de fondo a 0 segundos después de photobleaching. Dependiendo de la velocidad de la primera exploración, una cantidad importante de recuperación de fluorescencia ya puede ser detectado cuando la proteína de interés es muy móvil.
Muchas sondas de la proteína fluorescente se han desarrollado para estudiar la dinámica de las proteínas con el FRAP. Un enfoque complementario es, por ejemplo, para utilizar fotoactivables GFP (PA-GFP), o variantes fotoconvertible. Junto con la técnica FRAP, estas herramientas son cada vez más imprescindible para vivir estudios de células de imágenes 4.6.
Divulgaciones
Agradecimientos
Este trabajo fue apoyado por el Instituto Nacional de la Sordera y Otros Desórdenes de la Comunicación (NIDCD) Programa Intramural.
Materiales
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| 35 mm glass-bottom dishes | MatTek | P35GC-1.0-14-C | ||
| CalPhos Mammalian Transfection Kit | Clontech | 631312 | ||
| pEGFP-N1 plasmid | Clontech | 6085-1 | ||
| Zeiss confocal microscope | Zeiss | LSM 710 | ||
| Zeiss TempModule system | Zeiss | N.A. | ||
| ImageJ software | NIH | N.A. | ||
| Graphpad Prism software | Graphpad software | N.A. |
Referencias
- Zheng, C. Y., Petralia, R. S., Wang, Y. X., Kachar, B., Wenthold, R. J. SAP102 is a highly mobile MAGUK in spines. J Neurosci. 30, 4757-4766 (2010).
- Grati, M. Rapid turnover of stereocilia membrane proteins: evidence from the trafficking and mobility of plasma membrane Ca(2+)-ATPase 2. J Neurosci. 26, 6386-6395 (2006).
- Liu, L. N., Aartsma, T. J., Thomas, J. C., Zhou, B. C., Zhang, Y. Z. FRAP Analysis on Red Alga Reveals the Fluorescence Recovery Is Ascribed to Intrinsic Photoprocesses of Phycobilisomes than Large-Scale Diffusion. PLoS ONE. 4, e5295-e5295 (2009).
- Wiedenmann, J., Nienhaus, G. U. Live-cell imaging with EosFP and other photoactivatable marker proteins of the GFP family. Expert Rev Proteomics. 3, 361-374 (2006).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. J Cell Sci. 120, 4247-4260 (2007).
- Sprague, B. L., McNally, J. G. FRAP analysis of binding: proper and fitting. Trends Cell Biol. 15, 84-91 (2005).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados