Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Desarrollo de programas específicos de células de tipo anti-gp120 del VIH aptámeros para la entrega de siRNA
En este artículo
Resumen
Varios fluoro-2'-aptámeros de ARN del VIH-1BA-L con gp120 nanomol afinidad están aisladas de la biblioteca de ARN por In vitro Procedimiento SELEX. Un nuevo doble función inhibitoria anti-gp120 aptamer-siRNA quimera se crea y se muestra muy prometedora para sistémica anti-VIH.
Resumen
La epidemia mundial de infección por el VIH ha creado una necesidad urgente de nuevas clases de fármacos antirretrovirales. La potente capacidad de interferente pequeño (si) ARN para inhibir la expresión de los transcritos de ARN complementarias está siendo explotado como una nueva clase de terapias para una variedad de enfermedades incluyendo el VIH. Muchos informes anteriores han demostrado que las nuevas estrategias basadas en el ARNi terapéutico anti-HIV/AIDS tienen una considerable promesa, sin embargo, un obstáculo clave para la exitosa aplicación terapéutica y la traducción clínica de siRNAs es la prestación eficiente. Sobre todo, teniendo en cuenta la seguridad y la eficacia de tratamientos basados en el ARNi, es muy conveniente para desarrollar un planteamiento selectivo de la entrega del siRNA intracelulares a las poblaciones de células o tejidos específicos. El VIH-1 proteína gp120, un sobre glicoproteína en la superficie del VIH-1, juega un papel importante en la entrada del virus en las células CD4. La interacción de la gp120 y CD4 que provoca el VIH-1 de entrada e inicia la fusión de células ha sido validado como clínicamente relevante antivirales estrategia para el descubrimiento de fármacos.
Aquí, en primer lugar, discutir la selección e identificación de los 2'-F modificado aptámeros anti-gp120 del VIH ARN. Utilizando un método SELEX filtro de nitrocelulosa convencionales, varios aptámeros nuevo con afinidad nanomolar fueron aislados de un 50 al azar de la biblioteca nt ARN. Con el fin de recabar las especies vinculado con una mayor afinidad, el rigor de la selección está cuidadosamente controlada mediante el ajuste de las condiciones. Los aptámeros seleccionadas pueden unirse específicamente y de forma rápida internaliza en las células que expresan la proteína de envoltura del VIH-1. Además, los aptámeros el único que puede neutralizar el VIH-1 infectividad. Sobre la base de la mejor aptamer A-1, también creamos un nuevo doble función inhibitoria anti-gp120 aptamer-siRNA quimera en la que tanto el aptámero y las partes han siRNA potente contra el VIH actividades. Además, utilizamos la gp120 siRNA aptamer quimeras para la entrega del tipo de células específicas de los siRNA en el VIH-1 las células infectadas. Esta función dual quimera muestra un considerable potencial de la combinación de varios agentes de ácidos nucleicos terapéuticos (aptámero y siRNA) en la supresión de la infección por VIH-1, por lo que las quimeras siRNA aptamer atractivo candidatos terapéuticos para pacientes que no la terapia antirretroviral altamente activa (HAART).
Protocolo
1. Preparación de la biblioteca de ARN
- La biblioteca de ADN a partir contenía 50 nucleótidos de las secuencias aleatorias y se sintetizó por Integrated DNA Technologies (Coralville, Iowa). La única secuencia de ADN de cadena de la biblioteca es oligo 5'-GGG AGG ACG ATG CGG - N 50 - CAG ACG ACT CGC CCG A - 3 '(81 nt). La región de azar está flanqueado por las regiones constantes, que incluyen el promotor T7 para la transcripción in vitro y la etiqueta de un 3 'para la RT-PCR. El 5 'y 3' secuencias constantes son 5 '- TAA GAC TAC TCA CTA TAG GGA GGA CGA TGC GG - 3' (32 mer) y 5 '-GGC GAG TCG TCG TCT G - 3' (16 mer), respectivamente. Preparar una solución madre con agua y almacenar en alícuotas a -20 ° C.
- Amplificar el ADN de cadena simple al azar de la biblioteca oligo (0,4 M) por PCR utilizando 3 M cada uno de los 5 'y 3' primers, junto con 2 mM de MgCl 2 y 200 mM de cada dNTP. A fin de preservar la abundancia de la biblioteca original de ADN, PCR limitar a diez ciclos. Después de la reacción de PCR (10 reacciones, 100 L por reacción), la recuperación de la piscina dsDNA amplificado utilizando un gel QIAquick Kit de purificación (QIAGEN).
- Convertir el ADN de doble cadena que resulta en una biblioteca de ARN utilizando el kit de DuraScription (Epicentro, Madison, WI), de acuerdo a las instrucciones del fabricante. En la mezcla de reacción de transcripción, sustituya CTP y UTP con 2'-F-CTP y 2'-F-UTP para producir ribonucleasa ARN resistente. Por lo general, preparar 20 l de reacción que contiene 1 g de agua purificada plantilla de ADN, 2 l 10 x tampón, 2 l dATP, dGTP 2 l, 2 l 2'-F-dCTP, 2 l 2'-F-dUTP, 2 l TDT y 2 l T7 ARN polimerasa a temperatura ambiente y luego se incuba a 37 ° C durante 6 h. (50 mM de cada dNTP).
- Posteriormente digerir la reacción con DNasa I (1,5 l por 20 l reacción de transcripción T7) para eliminar la plantilla de ADN y se purifica por un 8% de poliacrilamida / 7 M urea gel. Cuantificar el ARN purificado de la biblioteca por espectrofotometría UV.
2. Generación in vitro de aptámeros
- Antes de la selección, preparación del buffer de selección y el tampón de replegamiento. Preparar un tampón HEPES 100 mM Hepes, pH 7,4. El uso de NaOH para ajustar el valor pH y luego almacenar a temperatura ambiente. Preparar un buffer replegado ARN (5xHBS) que contiene 50 mM Hepes pH 7,4, 750 mM NaCl, 5 mM MgCl 2, 5 mM CaCl 2, 13,5 mM KCl. Preparar un buffer de bajo contenido de sal de unión a ARN (10 mM HEPES pH 7,4, 50 mM NaCl, 1 mM CaCl 2, 1 mM MgCl 2, 2,7 mM KCl, 10 mM DTT, 0,01% de BSA y un buffer de alto contenido de sal de unión a ARN (10 mM HEPES pH 7,4, 150 mM NaCl, 1 mM CaCl 2, 1 mM MgCl 2, 2,7 mM KCl, 10 mM DTT, 0,01% de BSA). Guarde estos topes a -20 ° C.
- Realizar la SELEX principalmente como se describe 1-4. Antes de cada ronda de selección, vuelva a plegar las piscinas de ARN en 1xHBS buffer (10 mM HEPES pH 7,4, 150 mM NaCl, 1 mM CaCl2, MgCl2 1 mM, KCl 2,7 mM), se calienta a 95 ° C durante 3 minutos y luego enfriar lentamente a 37 ° C. Continuar la incubación a 37 ° C durante 10 min.
- Por lo general, a fin de minimizar la unión no específica con los filtros de nitrocelulosa, antes de absorber la volvió a doblar las piscinas de ARN para un filtro de nitrocelulosa (HAWP filtro de 0,45 micras) durante 30 minutos, antes de la incubación con el objetivo de VIH-1 Bal proteína gp120.
- Incubar la piscina pre-aprobados ARN con la proteína diana en baja en sal tampón de unión al ARN durante 30 minutos para las rondas de SELEX 1 a 4. Después de la cuarta ronda de SELEX, use un buffer de alto contenido de sal de unión a ARN. Con el progreso de SELEX, reducir la cantidad de proteína gp120 y aumentar el tRNA de levadura competidor con el fin de aumentar el rigor de la aptamer selección.
- Para el primer ciclo de selección, se incuba la piscina pre-aprobados al azar de ARN (40 mg, 1,5 nmol, 9x10 14 moléculas) y el VIH-1 Bal proteína gp120 (0,23 nmol, RNA / proteína 6,5 / 1) en 200 l de baja sal de unión a ARN de amortiguación en una plataforma giratoria a temperatura ambiente durante 30 min.
- Pasar de la reacción a través de un filtro de nitrocelulosa previamente mojado y lavado con 1 ml de tampón de unión.
- Eluir el ARN unido desde el filtro con tampón de elución 200 l (7 M urea y 5 mM EDTA) a 95 ° C durante 5 minutos, seguido de extracción con fenol / cloroformo y la concentración con un Microcon YM-30 columnas.
- Invertir transcribir el grupo recuperó el ARN utilizando el Thermoscript RT-PCR System (Invitrogen) y ampliar de 15 ciclos de PCR.
- Purificar la piscina dsDNA amplificado utilizando un gel QIAquick Kit de purificación y transcribir como se describió anteriormente para la siguiente ronda de la selección.
3. SELEX progreso monitoreado por ensayo de unión de filtro
- Monitorear el progreso de los aptámeros SELEX por ensayo de unión del filtro. El tratamiento de la piscina de ARN con el CIP para quitar la etiqueta de inicio de 5'-trifosfato y después con T4 polinucleótido quinasa y γ-32P-ATP.
- Calor 10 pmol de CIP tratados ARN de la biblioteca a 95 ° C durante 5 minutos y luego enfriar en el hielo. A continuación, añadir 2L de PNK buffer, 1 l de T4 polinucleótido quinasa, 1 l de ácido gamma-P 32-ATP y agua a 20 mL.
- Se incuba a 37 ° C durante 30 minutos, a continuación, añadir 20 mu ^; L de agua y purificar la reacción de los G-50 de la columna. Por último, obtener 40 L de ARN marcado en la concentración final de 250 nM.
- Antes de la prueba, vuelva a plegar el ARN marcado piscinas en 1xHBS buffer (10 mM HEPES pH 7,4, 150 mM NaCl, 1 mM CaCl2, MgCl2 1 mM, KCl 2,7 mM), se calienta a 95 ° C durante 3 minutos y luego enfriar lentamente a 37 ° C. Continuar la incubación a 37 ° C durante 10 min.
- Llevar a cabo una l 100 de la reacción de unión como ejemplo. Incubar el final de la etiqueta del ARN piscina (10 Nm) con proteína gp120 (100 nm) y un exceso molar de 10 veces de la competencia no específico tRNA (100 nm) en el buffer de alto contenido de sal de unión al ARN durante 30 minutos.
- Separar un l 50 de reacción de unión por un filtro de nitrocelulosa previamente mojado.
- Lave el filtro con 2 ml de tampón de unión y el recuento de la radiactividad retenida en el filtro a través de un contador de centelleo de usos múltiples (Beckman Coulter). Como un control de entrada, cuenta el restante 50 l de reacción de unión, al mismo tiempo. Calcular el porcentaje de la ARN retenido en el filtro en el ARN de entrada como la afinidad de unión.
4. La clonación, la secuenciación y los alineamientos
- Después de 11 rondas, si no se observa un mayor enriquecimiento, incluso después de rondas de selección adicional y luego ligar máxima de la piscina ARN ha sido potencialmente alcanzado.
- Invertir transcribir el altamente enriquecido piscina aptamer (12 de piscina ARN), utilizando el Thermoscript RT-PCR System (Invitrogen) y, posteriormente, ampliar el ADNc resultante mediante PCR. Purificar el producto de PCR utilizando un kit de purificación QIAquick Gel (QIAGEN). Clonar el gel purificado el producto de ADN en el vector de clonación TA pCR 2.1 (Invitrogen). En total, inocular 170 clones individuales y aún más su identificación por secuenciación del ADN para obtener las secuencias individuales.
- Clasificar los clones individuales en seis grupos diferentes en función de los alineamientos de las secuencias de aptamer individual. Eligió una secuencia representante de cada grupo (A-1, A-5, A-9, A-12, A-28 y B-68) para la caracterización adicional debido a su abundancia relativa dentro de su grupo.
5. Generación de aptámero y ARN quimera por la transcripción in vitro
- Generan directamente de doble cadena de ADN molde por PCR utilizando 2 M cada uno de los 5 'y 3' primers, junto con 2 mM de MgCl 2 y 200 mM de cada dNTP, y recuperar los productos resultantes de PCR utilizando un gel QIAquick Kit de purificación.
- Transcribe cadena con sentido quimera de sus plantillas de PCR de ADN generados mediante el kit de DuraScription (Epicentro, Madison, WI). En la mezcla de reacción de transcripción, sustituya la canónica CTP y UTP con 2'-F-CTP y 2'-F-UTP para producir ARN que es resistente a la RNasa A la degradación.
- Por lo general, se incuban 20 l de reacción que contenía 1 mg de ADN plantilla, 2 10xbuffer ml, 2 l dATP, dGTP 2 l, 2 l 2'-F-dCTP, 2 l 2'-F-dUTP, 2 l TDT y 2 l T7 RNA polimerasa a 37 ° C durante 6 horas, y posteriormente se purifican con Bio-Spin 30 columnas (Bio-Rad) después de la extracción con fenol y precipitación con etanol.
- Con el fin de evitar la respuesta de interferón, además tratar el ARN transcrito por el CIP para eliminar el inicio de 5'-trifosfato. Incubar total de 60 l de reacción que contiene 3 mg de transcripciones, 6 l de tampón 3 y 0,25 l de CIP a 37 ° C durante 60 min. Después de fenol / cloroformo y precipitación con etanol hecho de exigir, resuspender ARN de pellets en el agua.
- Para preparar las quimeras, se combinan las quimeras albergar sólo el sentido de ARN de cadena con el ARN antisentido apropiado replegado buffer, el calor a 95 ° C durante 3 minutos y dejar enfriar a 37 ° C poco a poco. Continuar la incubación a 37 ° C durante 10 min. Realizar replegado paso a un buffer de remate 1xHBS. Por ejemplo: mezcla de 10 l de 10 mM cadena con sentido quimera, 10 l de 10 mM cadena antisentido y el 5 de amortiguación replegado l (5xHBS) en 25 l sistema.
6. Determinación de las constantes de disociación de los ensayos de gel de cambio
- P 32 final marca el aptámero representante de cada grupo y el capítulo quimeras sentido y luego vuelva a plegar el ARN en tampón 1xHBS como se describió anteriormente.
- Prepare 25 ml de gel al 5% mediante la mezcla de 2,5 ml de solución tampón 10xTBE, con 3,125 ml de acrilamida / bis solución al 40%, 19.375 ml de agua, 150 l de persulfato de amonio al 10% (APS) en solución, y 30 l de TEMED. El gel debe polimeriza en unos 30 min. Retire con cuidado el peine y el uso de una jeringa de 30 ml equipado con una aguja para lavar los pocillos con tampón de (1xTBE).
- Completar el montaje de la unidad de gel y conectarse a una fuente de alimentación. El gel se puede pre-correr durante una hora a 180 V a 4 ° C.
- Serie diluir el VIH-1Bal proteína gp120 con tampón de unión a las concentraciones deseadas. La r finalconcentraciones eaction de gp120 son 0, 1, 5, 10, 20, 40, 80, 160, 320, 640 nm. Incubar una cantidad constante de 5'-P-32 final de la etiqueta del ARN (10 nM) con mayores concentraciones de proteína gp120 en el tampón de unión (un total de 20 l de reacción) sobre una plataforma giratoria a temperatura ambiente durante 30 min.
- Después de la incubación, mezclar 20 l de reacción de unión con 5 l de amortiguación de carga nativo y la carga en un gel de poliacrilamida al 5% no desnaturalización. Preparar un tampón de carga nativo (4x), que contiene 10 mM Tris-HCl, pH 7,5, 1 mM EDTA, 0,1% azul de bromofenol, 0,1% xileno cyanol FF, el 0,1% de Orange G, el 40% de glicerol. Almacenar en alícuotas a -20 ° C.
- Tras la electroforesis (180 V a 4 ° C durante 2 horas, hasta que el colorante secundario funciona en medio de gel), exponer el gel a una imagen de la pantalla de fósforo y cuantificar la radiactividad mediante un escáner tifón.
- Calcular las constantes de disociación usando regresión no lineal curva con un Pad Prism Gráfico.
7. Superficie de la célula-estudios de unión por citometría de flujo
- Generar aptamer fluorescentes y quimeras con el siRNA silenciador kit de marcaje (Ambion). Añadir los siguientes reactivos con el fin de: 22,5 l agua libre de nucleasa, 5 ml de 10 x tampón de etiquetado, 15 l de ARN (5 mg), 7,5 l tinte de etiquetado. Incubar un total de 50 l reacción de marcaje a 37 ° C durante 1 hora.
- Después de la incubación, añadir 5,0 l (0,1 vol) 5 M NaCl y 125 l (2,5 vol) EtOH frío 100%, y mezclar bien. Se incuba a 20 ° C durante 60 min. Centrifugar a velocidad máxima a 4 ° C durante 20 min. Quitar el sobrenadante y pellet lavado con EtOH 175 l 70%. Aire seco en pelets en la oscuridad y luego suspender ARN marcado en 15 l de agua libre de nucleasa.
- Medir la absorbancia de la ARN marcado a 260 nm y en el máximo de absorbancia para el colorante fluorescente. Cálculo de la base: relación de colorante y la concentración de ARN de acuerdo con la calculadora proporcionada por http://www.ambion.com/techlib/append/base_dye.html .
- Mix Cy3 marcado sentido capítulo quimeras y cadena antisentido y replegar en buffer replegado como se describió anteriormente.
- Obtener CHO-WT expresa gp160 y CHO-EE control de las células a través de la investigación del SIDA y el Programa de Referencia Reactivo 5, 6. Se cultivan las células en medio GMEM-S (con deficiencia de glutamina medio mínimo esencial con 400 M metionina sulfoximina (MSX)) (Gibco, Invitrogen). Cultivar las células en un 5% humidificado incubadora de CO 2 a 37 ° C.
- Lave la gp160 CHO-WT o CHO-EE control de las células con tampón de lavado precalentado, trypsinize y desprenderse de las placas. Después de lavar las células dos veces con 500 l de tampón de unión, resupend los gránulos de las células en tampón de unión y se incuba a 37 ° C durante 30 min. Precipitar las células y luego volver a suspender en 50 l de tampón precalentado vinculante que contiene 400 nM Cy3 marcado ARN experimental.
- Después de la incubación a 37 ° C durante 40 minutos, lavar las células tres veces con 500 l de tampón de unión precalentado, y finalmente se resuspenden en 350 l de tampón de unión precalentada a 37 ° C e inmediatamente se analizan por citometría de flujo.
8. La internalización y la localización intracelular de los estudios de células vivas, la microscopía confocal
- Antes de un día de ensayo, hacer crecer la gp160 CHO y CHO-WT-EE control de las células en placa de 35 mm con la siembra de 0.3x10 6 en 2 ml de medio GMEM-S para permitir que aproximadamente el 70% de confluencia en 24 h.
- En el día de los experimentos, lavar las células con 1 ml de PBS precalentado. Y se incuba con 1 ml de pre-calentado medio de cultivo completo durante 30 minutos a 37 ° C.
- Prepare Cy3 marcado siRNA aptamer quimeras como se describió anteriormente. Incubar la replegar aptamer seguir con el siRNA-stick que contiene 5'-Cy3 marcado sentido capítulo para formar el conjugado aptamer stick-siRNA como se describió anteriormente.
- Antes del ensayo, preparar dosis de 0,15 mg / ml de solución de Hoechst 33342 (medio de contraste nuclear de las células vivas, Molecular Probes, Invitrogen, CA) en el agua y almacenar en alícuotas a 4 ° C.
- Tinción de las células mediante el tratamiento con 0,15 mg / ml Hoechst 33342 durante 15 min a 37 ° C. Inmediatamente, lavar la tinta con 1,0 ml de medio fresco dos veces y vuelva a colocar 2 ml de medio fresco precalentado.
- Agregue el marcado con Cy3 aptamer-siRNA quimera a una concentración final de 100 nM en los medios de comunicación e incubar células vivas, la microscopía confocal en un 5% de CO2 microscopio incubadora a 37 ° C.
- Recoger las imágenes cada 15 minutos utilizando un Zeiss LSM 510 Meta invertido dos fotones sistema de microscopio confocal con la inmersión en agua a 40 aumentos.
9. In vitro del VIH-1 y el desafío de ensayo del antígeno p24
- Compra CCRF-CEM células de ATCC. Crecer en RPMI-1640 (Cellgro, Mediatech Inc.), complementado con fetal al 10% de suero bovino (FBS, HyClone), L-glutamina y 1xPen Strep-(Gibco, Invitrogen). Las células de cultivo en un 5% humidificado incubadora de CO 2 a 37 ° C.
- Obtener mononucle sangre periféricaar muestras de donantes sanos del Nacional City of Hope Medical Center (personal de la clínica).
- Aislar PBMCs de sangre total por centrifugación a través de Ficoll-Hypaque solución (Histopaque-1077, Sigma). Agotan las células CD8 (células T-cytotoxic/suppressor) a partir de PBMC usando Dynabeads CD8 (Invitrogen, CA).
- Se cultivan las células en las células T medio activo (BioE, St. Paul, MN). Las células de cultivo en un 5% humidificado incubadora de CO 2 a 37 ° C.
- Obtener el VIH-1 IIIB y NL4-3 virus y virus VIH-1 Bal de la investigación del SIDA y el Programa de reactivos de referencia. Después de la propagación del virus, almacenar en alícuotas a -80 ° C.
- Infectar a las células CCRF-CEM o PBMC humanos con virus de VIH (IIIB, NL4-3 o Bal) (MOI 0,001 o 0,005). Después de 24 horas de post-infección, lave las células tres veces con PBS para eliminar el virus de forma gratuita. Continuar con la cultura de las células infectadas en un 5% de microscopía CO 2 incubadora a 37 ° C durante 4 días.
- Antes de ARN tratamientos, lavar suavemente las células infectadas con PBS tres veces para eliminar el virus de forma gratuita. Incubar 2x10 4 células infectadas y 3x10 4 células no infectadas con replegada ARN experimental a 400 nM concentración final en placas de 96 pozos a 37 ° C (100 l por pocillo, ensayo triplex).
- Recoger el sobrenadante de cultivo (10 l por pocillo) en diferentes momentos (3 d, 5 d, 7 D y D 9) y almacenar a -20 ° C hasta el ensayo p24.
- Realizar los análisis de antígeno p24 utilizando un antígeno p24 del VIH-1 kit de ELISA.
10. La función de detección de siRNA cuantitativos de RT-PCR
- Infectar a las células CCRF-CEM o PBMC humanos con el VIH virus (IIIB, NL4-3 o Bal) y tratar con los ARN experimentales (400 Nm) como se describió anteriormente.
- Después de 7 días de tratamiento, las células de pellets y aislar ARN total con STAT-60. Tratar el ARN total con DNasa I para extraer el ADN genómico y producir cDNA utilizando 2 g de ARN total.
- Mezcle los reactivos siguientes: 8 ml agua libre de nucleasa, 1,5 mL 10 x DNasa de Estabilización, 4 l de ARN (2 mg), inhibidor de 0,5 y 1,0 l RNain l RNasa libre de DNasa I. Incubar un total de 15 l de reacción 37 º C durante 1 horas y el calor a 80 ° C durante 10 min para inactivar DNasa I. Inmediatamente, enfriar la reacción sobre el hielo.
- Añadir 2 imprimación l azar (50 ng / mL) y 1μL dNTP (10 mM) en la mezcla de reacción anterior, y entonces el calor a 65 ° C durante 5 min. Inmediatamente, enfriar la reacción sobre el hielo.
- Añadir los siguientes reactivos: 5 ml 5xFirst cadena de amortiguación; 2,5 ml 0,1 M de DTT, 0,5 mL RNain inhibidor y 1,0 mL MMLV-RT. Incubar un total de 27 mL de reacción a 25 ° C durante 10 minutos y a 37 ° C durante 1 hora. Después de mezcla de calor de la reacción, a 70 ° C durante 15 min para inactivar la transcriptasa inversa y luego enfriar en el hielo. El cDNA está listo para QRT-PCR análisis.
- Analizar la expresión de los genes diana por RT-PCR cuantitativa utilizando 2 x iQ SyberGreen Mastermix y conjuntos de cebadores específicos a una concentración final de 400 nM (ensayo triplex). Use la expresión de GAPDH como control interno para la normalización de los datos qPCR.
11. Los resultados representativos:
1. Nueva aptámeros de ARN del VIH-1 gp120 BaL son aislados y caracterizados.
Como se describe en la sección experimental, una biblioteca de oligonucleótidos de ADN inicial que contiene una región 50 nt al azar, flanqueada por regiones imprimación fija en el 5 'y 3' se amplifica y se transcribe en una piscina de ARN. Esta biblioteca inicial se compone de hasta 10 15 diversas secuencias (1 nmol), que se pliegan en una amplia gama de diferentes estructuras 3-D. La complejidad y diversidad de la biblioteca inicial podría garantizar la presencia de estructuras activas con una buena afinidad de unión a la meta.
Emplear un procedimiento in vitro SELEX (Figura 1) para seleccionar 2'-fluoropirimidinas modificado aptámeros de ARN que se unen selectivamente el R5 cepa de VIH-1 gp120 BaL proteína de la cubierta 7. Como se muestra en la Figura 1, una estrategia de selección de base de nitrocelulosa se realiza para aislar específicos objetivo vinculante para el ARN-no vinculante de las moléculas de ARN. Dado que la proteína se adhiere a la nitrocelulosa, sólo el RNA / proteína complejos o agregados puede ser retenida en la membrana y ARN libre son lavados. Bajo condiciones de desnaturalización, el ARN unido se recuperan y se transcriben de forma inversa a ADNc y luego se amplifica en ADN de doble cadena, y posteriormente transcrita in vitro para crear un nuevo grupo de ARN para el ciclo siguiente selección. El rigor de selección se incrementa al reducir la cantidad de proteína de interés y el aumento de la cantidad de tRNA de la competencia. La cantidad de reserva de ARN, proteínas y tRNA competidor utilizado en cada ronda de selección se muestra en la Tabla 1.
Monitorear el progreso de la selección después de cada ciclo SELEX por el ensayo de unión del filtro. Evaluar la afinidad de unión como el porcentaje de la ARN mantiene en la FILter en el total acumulado de ARN. La partida de billar ARN (1-RNA) sólo muestra un 0,1% de los ARN de entrada retenida en la membrana. Sin embargo, después de nueve rondas de selección de la novena biblioteca del ARN (9-ARN) tiene un 9,72% de la entrada de ARN unido. A pesar de las rondas de selección adicionales se llevaron a cabo, no se observa un mayor enriquecimiento, lo que sugiere que la unión máxima de la piscina del ARN se ha alcanzado (Figura 2A). Similar con el ensayo de unión del filtro, el cambio de ensayo de gel también es una de las estrategias más populares para la determinación de las constantes de disociación. Este procedimiento es fácil y conveniente. Como se muestra en la Figura 2B, los ensayos de gel de cambio confirman aún más la actividad de unión de las piscinas de ARN. Estos resultados demuestran que algunos ligandos con alta especificidad de unión a la proteína diana, sucesivamente enriquecida en estas piscinas ARN ..
Clonar y secuenciar las piscinas aptamer altamente enriquecido (12-ARN). De acuerdo con los alineamientos de las secuencias individuales aptamer clonado, seis grupos diferentes se clasifican como se muestra en la Tabla 2. Alrededor del 40% de los clones (grupos I y II aptámeros) contienen una secuencia conservada: A TTGAGGGACC (A / G) (A / G). Nosotros elegimos una secuencia de representante de cada grupo (por ejemplo: A-1, A-5, A-9, A-12, A-28 y B-68) para la caracterización adicional debido a su abundancia relativa dentro de su grupo. A través de un cambio de ensayo en gel nativo de movilidad, las constantes de disociación (K d) de estos aptámeros representante se calculan (Figura 3). Por ejemplo, A-1, el mejor de los aptámeros, tiene un valor aparente de 52 Kd nM (Figura 3B). Como se muestra en la Figura 3A, estos aptámeros seleccionados de manera selectiva se puede unir con el objetivo de VIH-1 gp120 Bal, pero no el VIH proteína gp120 CM.
2. Anti-gp120 aptamer se une específicamente y es internalizado por las células que expresan gp160 del VIH e inhibir la infección VIH-1 en cultivos celulares.
CHO-gp160 células que expresan establemente la envoltura del VIH gp160 glicoproteína se utilizan para probar para la internalización de unión y de los seleccionados anti-gp120 aptámeros. Estas células no se procesan en gp160 gp120 y gp41, ya que carecen de las proteasas codificadas necesarias para gag sobre su procesamiento. Como control se utiliza la de sus padres CHO-EE línea celular que no expresa gp160. Análisis de citometría de flujo (Figura 4) revelan que el Cy3 marcado aptámeros se unen específicamente a las células CHO-gp160, pero no el control de CHO-EE células. Por otra parte, en tiempo real en células vivas del eje Z microscopía confocal indica que el Cy3 marcado aptamer es selectiva internalizados en el CHO-gp160 células (Figura 4B y 4C) después de 2 horas de incubación, pero no el CHO-EE control de las células ( Figura 4D). Figura 4C muestra también que la aptamer agregados en el citoplasma, lo que sugiere la gp120 aptámeros tal vez entrar en las células mediante endocitosis mediada por receptor.
En el ensayo de VIH-1 reto, el VIH-1 infectadas PBMC células son tratadas con los aptámeros. En diferentes días después del tratamiento con los aptámeros, alícuotas de los medios de comunicación se analizan para los niveles de antígeno viral p24 (Figura 4). Los resultados muestran que los aptámeros anti-gp120 (A-1) inhibe la producción de VIH-1 p24 con una concentración de nano-molar.
3. Anti-gp120 aptamer-siRNA quimera es diseñado y evaluado su eficacia como del tipo de células específicas del sistema de entrega de siRNA
Como se muestra en la Figura 5, y el segmento de la aptamer sentido capítulo de la siRNAs contenida nucleasa resistente 2'-fluoro UTP y 2'-fluoro-CTP y se sintetizan a partir de plantillas dsDNA correspondiente en la transcripción del bacteriófago vitro. Con el fin de aumentar la flexibilidad de la molécula, un engarce de dos nucleótidos (UU) se inserta entre la aptamer y la porción de sustrato Dicer. Para preparar el siRNA que contienen quimeras, in vitro transcrito quimérico polímeros cadena con sentido aptamer se recocido con concentraciones equimolares de un ARN de cadena sin modificar antisentido. Estos datos del ensayo de gel de cambio (Figura 5) y citometría de flujo (Figura 5) indican que las quimeras mantener aproximadamente las mismas afinidades vinculantes como los aptámeros solo. Las imágenes en función del tiempo de tiempo real microscopía confocal (Figura 5D) muestran que Cy3 marcado quimera Ch A-1 puede ser con éxito internalizado en el citoplasma de las células. Como era de esperar, no la absorción de la quimera que se observa con las células CHO-EE de control (Figura 5E).
Del mismo modo, el potencial antiviral de ARN es evaluado por el VIH-1 ensayo de desafío. Los resultados de los análisis del antígeno p24 (Figura 6A y 6B) muestran que ambos aptamer y la quimera inhibir la producción de p24, pero la mayor inhibición se observó con la quimera Ch A-1 del tratamiento.
Para confirmar que el componente de siRNA está funcionando a lo largo de la APTAmer, después de la internalización de la A-1 quimera en las células infectadas Ch, también evaluamos los niveles relativos de la inhibición de la expresión de los genes tat / vuelta por los análisis cuantitativos de RT-PCR la expresión. Nos encontramos con que el tratamiento de las células infectadas con las quimeras es capaz de inducir el silenciamiento del gen tat / rev, mientras que la aptamer sí solo no afecta tat / expresión del gen rev (Figura 6C y 6D). Estos resultados proporcionan más apoyo que el siRNA aptamer entregado desencadenantes RNAi.
| La cantidad de proteína, el ARN de la piscina y tRNA utilizado para la selección | |||||
| SELEX rondas | Proporción de Objetivo / RNA | Proteína gp120 | ARN piscina | Competidor tRNA | Selección de Buffer |
| 1 | 1/6.5 | 229,8 pmol | 1,5 nmol (40,1 mg) | 0 | Bajo contenido de sal de amortiguación SELEX |
| 2 | 1/6.5 | 114,9 pmol | 0,75 nmol (20,1 mg) | 0,25 nmol (6,6 mg) | |
| 3 | 8.1 | 76,6 pmol | 0,625 nmol (16,7 mg) | 0,25 nmol (6,6 mg) | |
| 4 | 8.1 | 76,6 pmol | 0,625 nmol (16,7 mg) | 0,25 nmol (6,6 mg) | |
| 5 | 8.1 | 38,3 pmol | 0,306 nmol (8,18 mg) | 0,5 nmol (13,2 mg) | Elevado de sal de amortiguación SELEX |
| 6 | 8.1 | 38,3 pmol | 0,306 nmol (8,18 mg) | 0,5 nmol (13,2 mg) | |
| 7 | 1 / 10 | 26,8 pmol | 0,268 nmol (7,16 mg) | 0,5 nmol (13,2 mg) | |
| 8 | 1 / 10 | 26,8 pmol | pmol 0,268 nmol (7,16 mg) | 1 nmol (26,4 mg) | |
| 9 | 1 / 10 | 15,3 pmol | 0,153 nmol (4,09 mg) | 1 nmol (26,4 mg) | |
| 10 | 1 / 10 | 15,3 pmol | 0,153 nmol (4,09 mg) | 1,5 nmol (39,6 mg) | |
| 11 | 1/12.5 | 7,66 pmol | 0,096 nmol (2,56 mg) | 2 nmol (52,8 mg) | |
| 12 | 1/12.5 | 7,66 pmol | 0,096 nmol (2,56 mg) | 2 nmol (52,8 mg) | |
Tabla 1. Las condiciones de selección. La cantidad de proteína, el ARN de la piscina y tRNA para cada selección y buffer de selección se indican.
| Grupo | ARN | Secuencias aleatorias | Frecuencia (140 clones) |
| Grupo I | A-1 | AATTGAGGGACCA CGCGCTGCTTGTTGTGATAAGCAG TTTGT CG GATGG | 33 (23,6%) |
| B-7 | AATTGAGGGACCA ACGCGAGGATGTGGATAGTGTGTA TTTGC GT GATGG | 3 | |
| A-32 | AATTGAGGGACCG TTGGTAAAAGCCGGA AATTG AGCT TTTAC GGC GATGG | 5 | |
| B-55 | AATTGAGGTACCGCG TTATTAGGAACA AATTG GAATTCTAAACGC GATGG | 2 | |
| A-24 | AATAGAGGGACC CAGATATAGGCTACACGGATGATGGTGTATCTG GATGG | 1 | |
| B-19 | AATAGAGGAACCG TTTCAGAAGACTACAGGTTAGTCCAATGAAGC GACGG | 1 | |
| B-31 | AATAGAGGGACCG TGGACAATAATTTATGGTCA TTTATTGGCAC GATGG | 1 | |
| Grupo II | A-12 | AGTAGAGGAACCA AGCAATGGATGAATGCAAAAGTGTAAATGCTT GATGG | 10 (7,1%) |
| Grupo III | A-9 | TGAGTTTGGGTAAATTTCCGGTTTCGGTTTACTCACGAAAGATCGGTCGG | 15 (10,7%) |
| Grupo IV | A-28 | TAAAGGAGGGAAGGATGAGACCGCACGAAAAATATCAGCATACG TTTGTG | 10 (7,1%) |
| Grupo V | A-5 | GAAACTAGTTTGAATAATGGTGTAGAGGAGGGTCAATAGTTTCG TTGGTG | 9 (6,4%) |
| Grupo VI | B-68 | ACATAGTAATGACACGGAGGATGGAGAAAAAACAGCCATCTCTTGACGGT | 2 |
| Otros | Secuencia de los huérfanos | 48 |
Tabla 2. La alineación e identificación de los aptámeros de ARN. Tras la ronda 12 ª de la selección, el grupo seleccionado de ARN fue clonado y secuenciado. Después de la alineación de los 140 clones, seis grupos de anti-gp120 aptámeros fueron identificados. Sólo las secuencias aleatorias de las regiones centrales aptamer (5'-3 ') se indican. Los aislamientos se producen con frecuencia se especifican varios.
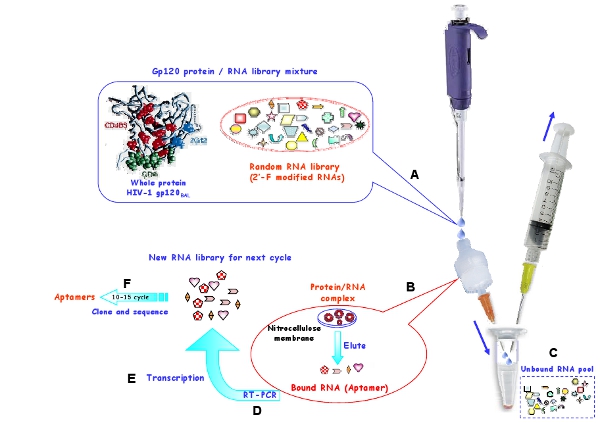
Figura 1: Representación esquemática de la del proceso de selección in vitro utilizando una membrana de nitrocelulosa, para la generación de aptámeros de ARN del VIH-1 Bal proteína gp120. (A) La partida de billar ARN y proteína diana se incubaron a formar complejos. (B) El límite moléculas de ARN fueron retenidos en la membrana y se eluyó de la membrana en condiciones de desnaturalización. (C) El ARN no consolidados fueron arrasadas. (D) El ARN seleccionados fueron transcritas inversa y amplificado por PCR. (E) El ADN correspondiente se transcribe a continuación en nuevo grupo de ARN para los ciclos de selección que viene. (F) Después de 10-15 rondas de selección, los aptámeros seleccionados fueron clonados y secuencia.
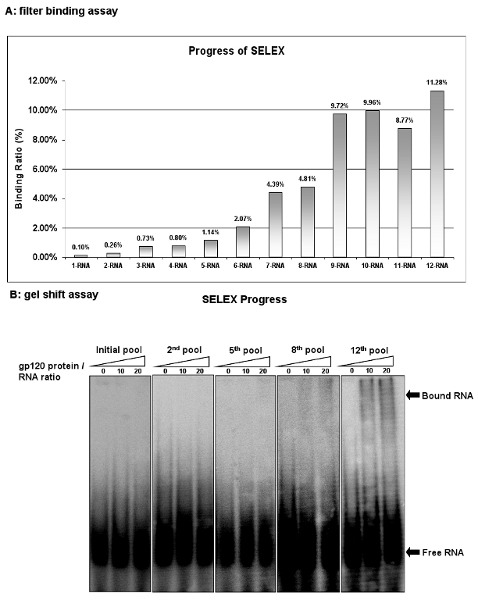
Figura 2: El avance del VIH-1 gp120 selección aptámeros. (A) La actividad de unión de la piscina de ARN en cada ciclo se analizaron mediante un ensayo de unión con el filtro de tRNA de la competencia. Actividades de unión se calcula como el porcentaje de entrada de ARN retenido en el filtro de proteínas / complejo de ARN. (B) La actividad de unión de la piscina de ARN en cada ciclo se analizaron mediante un ensayo de gel de cambio. El 12 de piscina ARN mostraron la mayor actividad de unión.
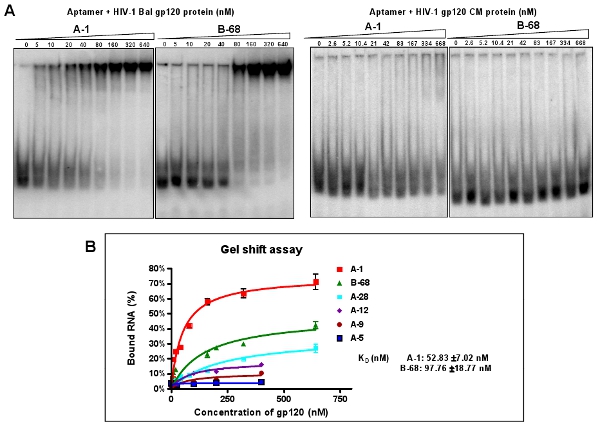
Figura 3: ensayo de actividad de unión de una selección de aptámeros individuales contra el VIH-1 gp120 Bal. (A) El extremo 5 'P 32 etiquetados aptámeros individuales se incubaron con la creciente cantidad de proteína gp120 objetivo o no específicos de la proteína CM. Las mezclas de reacción de unión se analizaron mediante un ensayo de gel de cambio de la movilidad. Aptámero A-1 y B-68 mostró la mejor afinidad de unión con la proteína diana, pero la proteína no CM. Los datos representan el promedio de cuatro repeticiones. (B) la curva de unión de un ensayo de gel de cambio.
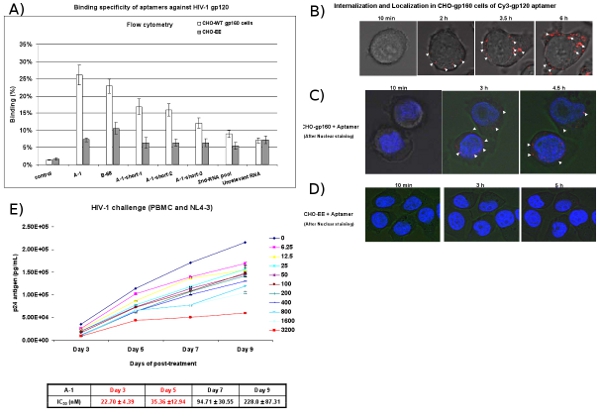
Figura 4: tipo de célula unión específica y estudios de captación de aptámeros. (A) de la unión de la superficie celular de Cy3 marcado ARN se evaluó por citometría de flujo. ARN Cy3 marcado fueron probados por la unión a las células CHO-gp160 y CHO-EE control de las células. Los aptámeros seleccionados mostraron tipo de células afinidad de unión específica. El 2 de ARN ARN piscina e irrelevante se utilizaron como controles negativos. Los datos representan el promedio de tres repeticiones. (B) La internalización de análisis. CHO-gp160 células fueron cultivadas en placas de 35 mm y se incubaron con una concentración de 100 nM Cy3 marcado A-1 en los medios de cultivo para el análisis en tiempo real en vivo microscopía confocal de células. Las imágenes se recogieron en 15 minutos. intervalos con 40 aumentos. (C, D) Localización de análisis. CHO-gp160 células CHO-EE y el control de las células se cultivaron en placas de 35 mm. Antes de la incubación con 100 nM de Cy3 marcado A-1, las células se tiñeron con Hoechst 33342 (medio de contraste nuclear con células vivas) y después se analizan en tiempo real mediante microscopía confocal. (E) El seleccionado anti-gp120 aptámeros inhibir la replicación del VIH-1 en PBMC humanos previamente infectados con el VIH-1 NL4-3 virus. Diferentes concentraciones y el tiempo de pointes se han presentado. IC50 valor fue incluido. Los datos representan el promedio de las mediciones por triplicado de la p24.
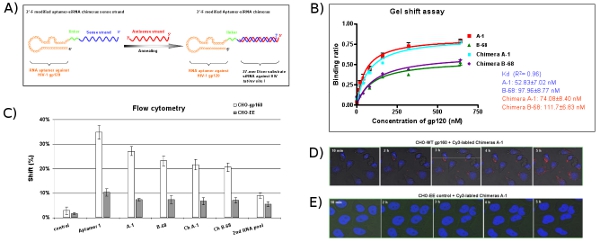
Figura 5: El diseño y la evaluación del sistema de prestación aptamer-siRNA quimera. (A) Esquema siRNA aptamer ARN quimérico: la región del aptámero anti-gp120 es responsable de la unión de la gp120 y el siRNA está orientada a un exón común del VIH-1 tat / rev. La 2'-fluoro modificado aptamer-siRNA cadena de sentido único se co-transcritos, seguido de recocido de la hebra de siRNA antisentido complementario para completar la molécula quimérica. Un enlazador (UU) entre el aptámero y siRNA se indica en verde. (B) El ARN siRNA aptamer quiméricos que tienen valores comparables Kd, así como aptámeros los padres se unen específicamente a la proteína gp120 del VIH Bal. Los datos representan el promedio de tres repeticiones. (C) Tipo de célula-estudios de unión específica de aptámeros. ARN Cy3 marcado fueron probados por la unión a las células CHO-gp160 y CHO-EE control de las células. Celular surenlaces frente a Cy3 marcado ARN fueron evaluados por citometría de flujo. Los aptámeros seleccionados mostraron tipo de células afinidad de unión específica. El 2 de ARN y ARN piscina irrelevantes se utilizaron como controles negativos. Los datos representan el promedio de dos repeticiones. (D, E) La internalización intracelular y análisis de localización. CHO-gp160 células fueron cultivadas en placas de 35 mm y se tiñeron con Hoechst 33342 (medio de contraste nuclear de las células vivas). Posteriormente, las células fueron incubadas en medio de cultivo con una concentración de 100 nM Cy3 marcado quimera en tiempo real en células vivas análisis de microscopía confocal, tal como se ha descrito anteriormente.
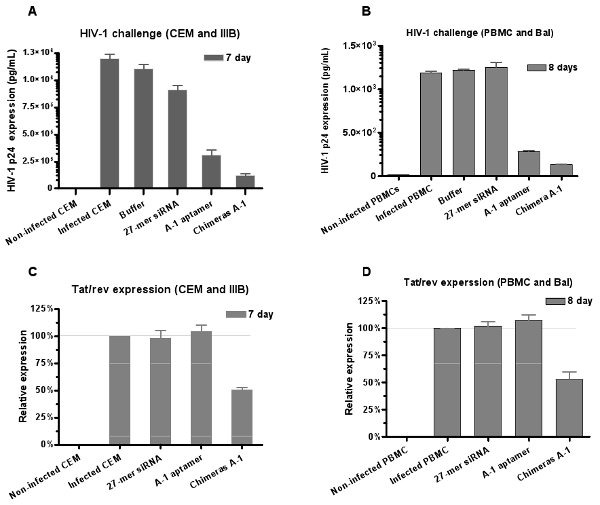
Figura 6: la inhibición dual de la infección VIH-1 mediada por quimeras siRNA aptamer. Tanto anti-gp120 aptamer y quimeras siRNA aptamer neutralizado infección VIH-1 en (A) las células CEM (cepa IIIB) y (B) la cultura PBMCs humano (cepa BAL), respectivamente. Los datos representan el promedio de las mediciones por triplicado de la p24. Las quimeras mostró fuerte inhibición que aptamer solo lo que indica que (C, D) el siRNA emitido por el aptámeros el regulado tat / rev la expresión génica en las PBMC. Los datos representan el promedio de tres repeticiones.
Discusión
Los aptámeros son in vitro desarrollado ácidos nucleicos que asumen específica y estable formas tridimensionales, proporcionando muy específicos, la unión estrecha de moléculas específicas 8. La baja afinidad nanomolar vinculante y especificidad exquisita de aptámeros a sus objetivos que sean versátiles herramientas de diagnóstico, imágenes in vivo, y la terapéutica 9. Con el advenimiento de la tecnología aptamer para la entrega de siRNA objetivo ahora es pos...
Divulgaciones
Agradecimientos
Damos las gracias a Britta Hoehn, Sun Guihua, Soifer Harris y Lisa Scherer útil para los debates. Este trabajo fue apoyado por becas de los Institutos Nacionales de Salud y AI29329 HL07470 otorgado a JJR Los siguientes reactivos fueron obtenidos a través de la Investigación y el Programa de SIDA de los NIH de reactivos de referencia, la División de SIDA, NIAID, NIH: el Cho-EE y de células CHO-gp160 línea, el pNL4-3 luc vector, el VIH-1 gp120 BaL de DAIDS, el NIAID.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios (opcional) |
|---|---|---|---|
| MF-Millipore filtro de membrana | Millipore | HAWP01300 | Tamaño de poro 0,45 m |
| Swinnex portafiltros | Millipore | SX0001300 | 13 mm de diámetro |
| QIAquick Gel Extraction Kit | QIAGEN | 28706 | De purificación de ADN |
| Microcon YM-30 columnas | Millipore | 42410 | ARN concentración |
| Bio-spin columna del 30 de | Bio-Rad | 732-6250 | ARN purificación |
| PCR Taq ADN polimerasa | Sigma-Aldrich | D1806 | |
| Thermoscript RT-PCR sistema | Invitrogen | 11146-024 | |
| DuraScribe Kit T7 transcripción | EpiCentre | DS010925 | |
| dNTP para PCR | Roche | 1 581 295 | |
| El ácido ribonucleico, el traslado de E. coli | Sigma-Aldrich | R1753 | tRNA competidor |
| VIH-1 Ba-L proteína gp120 | la investigación del SIDA y el Programa de reactivos de referencia | 4961 | Proteína diana |
| SiRNA silenciador kit de etiquetado - Cy3 | Ambion | 1632 | |
| Fenol / cloroformo 01/05 solución (pH 4,5) | Ambion | AM9720 | |
| Cloroformo / isopropanol 24 / 1 solución | Sigma | C0549 | |
| Ternero intestinal fosfatasa (CIP) | Nueva Inglaterra BioLab | M0290L | |
| Polinucleótido quinasa T4 | Nueva Inglaterra BioLab | M0201L | |
| Glucógeno | Roche | 10 901 393 001 | ARN precipitación |
| Gamma-P-32 ATP | MP Biomédica | 013502002 | Radiactividad |
| 40% AccuGel 19:01 | Nacional de Diagnóstico | CE-850 | |
| 10xTBE | Nacional de Diagnóstico | CE-860 | |
| N, N, N, N'-tetrametiletilendiamina (TMEMD) | Sigma-Aldrich | T9281 | |
| Persulfato de amonio (APS) | Sigma-Aldrich | A3678 | |
| L-metionina sulfoximina | Sigma-Aldrich | M5379-250 mg | |
| RPMI 1640 los medios de comunicación | Invitrogen | 11835-030 | |
| Solución de bicarbonato sódico, un 7,5% w / v | Invitrogen | 25080-094 | |
| Medio esencial mínimo (MEM) (10 ) | Invitrogen | 11430-030 | |
| MEM no aminoácidos esenciales (100 ) | Invitrogen | 11140-050 | |
| TA kit de clonación con pCR 2.1 | Invitrogen | K2040-01 |
Referencias
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Robertson, D. L., Joyce, G. F. Selection in vitro of an RNA enzyme that specifically cleaves single-stranded DNA. Nature. 344, 467-468 (1990).
- Fitzwater, T., Polisky, B. A SELEX primer. Methods Enzymol. 267, 275-301 (1996).
- Weiss, C. D., White, J. M. Characterization of stable Chinese hamster ovary cells expressing wild-type, secreted, and glycosylphosphatidylinositol-anchored human immunodeficiency virus type 1 envelope glycoprotein. J Virol. 67, 7060-706 (1993).
- Vodicka, M. A. Indicator cell lines for detection of primary strains of human and simian immunodeficiency viruses. Virology. 233, 193-198> (1997).
- Zhou, J. Selection, characterization and application of new RNA HIV gp 120 aptamers for facile delivery of Dicer substrate siRNAs into HIV infected cells. Nucleic Acids Res. , (2009).
- Mayer, G. The chemical biology of aptamers. Angew Chem Int Ed Engl. 48, 2672-2689 (2009).
- Famulok, M., Hartig, J. S., Mayer, G. Functional aptamers and aptazymes in biotechnology, diagnostics, and therapy. Chem Rev. 107, 3715-3743 (2007).
- Chu, T. C., Twu, K. Y., Ellington, A. D., Levy, M. Aptamer mediated siRNA delivery. Nucleic Acids Res. 34, e73-e73 (2006).
- McNamara, J. O., 2nd, . Cell type-specific delivery of siRNAs with aptamer-siRNA chimeras. Nat Biotechnol. 24, 1005-1015 (2006).
- Dassie, J. P. Systemic administration of optimized aptamer-siRNA chimeras promotes regression of PSMA-expressing tumors. Nat Biotechnol. 27, 839-849 (2009).
- Zhou, J., Rossi, J. J. The therapeutic potential of cell-internalizing aptamers. Curr Top Med Chem. 9, 1144-1157 (2009).
- Zhou, J., Li, H., Li, S., Zaia, J., Rossi, J. J. Novel dual inhibitory function aptamer-siRNA delivery system for HIV-1 therapy. Mol Ther. 16, 1481-1489 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados