Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Entwicklung von Zell-spezifischen Anti-HIV-gp120-Aptamere für siRNA
In diesem Artikel
Zusammenfassung
Mehrere 2'-Fluoro RNA-Aptamere gegen HIV-1Ba-L gp120 mit Nanomol Affinität von einer RNA-Bibliothek isoliert durch In-vitro- SELEX Verfahren. Ein neuer Dual-hemmende Funktion anti-gp120 Aptamer-siRNA-Chimäre wird erstellt und zeigt viel versprechend für die systemische Anti-HIV-Therapie.
Zusammenfassung
Die globale Epidemie der HIV-Infektion hat ein dringender Bedarf für neue Klassen von antiretroviralen Wirkstoffen erstellt. Die starke Fähigkeit von small interfering (si) RNAs, die Expression der komplementären RNA-Transkripte verhindern wird als eine neue Klasse von Therapeutika für eine Vielzahl von Krankheiten, einschließlich HIV ausgenutzt. Viele frühere Berichte haben gezeigt, dass neue RNAi-basierte anti-HIV/AIDS therapeutische Strategien sehr vielversprechend sind, jedoch ist ein zentrales Hindernis für die erfolgreiche therapeutische Anwendung und klinische Umsetzung von siRNAs effiziente Lieferung. Besonders, wenn man die Sicherheit und Wirksamkeit von RNAi-basierten Therapeutika, ist es höchst wünschenswert, um eine gezielte intrazelluläre siRNA Ansatz für spezifische Zellpopulationen oder Geweben entwickeln. Die HIV-1 gp120-Protein, einem Glykoproteinumhüllung auf der Oberfläche des HIV-1, spielt eine wichtige Rolle in Eindringen des Virus in CD4-Zellen. Die Interaktion von gp120 und CD4, dass HIV-1-Eintrag löst und initiiert Zellfusion wurde als klinisch relevant anti-virale Strategie für die Wirkstoffforschung validiert.
Im Folgenden stellen wir zunächst besprechen die Auswahl und Identifizierung von 2'-F modifiziert Anti-HIV-gp120 RNA-Aptameren. Mit einem herkömmlichen Nitrocellulosefilter SELEX-Methode wurden mehrere neue Aptamere mit nanomolaren Affinität von einem 50 zufällige nt RNA-Bibliothek isoliert. Um erfolgreich zu erhalten gebundenen Spezies mit höherer Affinität, ist die Auswahl Stringenz sorgfältig durch Einstellen der Bedingungen gesteuert. Die selektierten Aptamere spezifisch binden können und schnell in Zellen, die das HIV-1 Hüllprotein internalisiert werden. Darüber hinaus können die Aptamere allein neutralisieren HIV-1 Infektiosität. Basierend auf den besten Aptamer A-1, erstellen wir auch einen neuartigen dualen hemmende Funktion anti-gp120 Aptamer-siRNA-Chimäre, in denen sowohl die Aptamer und die siRNA Teile haben starke anti-HIV-Aktivitäten. Darüber hinaus nutzen wir die gp120 Aptamer-siRNA Chimären für die Zell-spezifische Auslieferung der siRNA in HIV-1 infizierten Zellen. Diese Doppelfunktion Chimäre zeigt ein erhebliches Potenzial für die Kombination von verschiedenen Nukleinsäure-Therapeutika (Aptamer-und siRNA) in der Unterdrückung von HIV-1-Infektion, so dass die Aptamer-siRNA Chimären attraktive therapeutische Kandidaten für Patienten, bei denen hochaktive antiretrovirale Therapie (HAART).
Protokoll
1. Vorbereitung der RNA-Bibliothek
- Die Ausgangs-DNA-Bibliothek enthielt 50 Nucleotide der zufälligen Sequenzen und wurde von Integrated DNA Technologies (Coralville, Iowa) synthetisiert. Die Einzelstrang-DNA-Oligo-Bibliothek Sequenz 5'-GGG AGG ACG ATG CGG - N 50 - CAG ACG ACT CGC CCG A - 3 "(81 nt). Die zufällige Region wird von konstanten Regionen, die die T7-Promotor für in vitro-Transkription und eine 3 'Tag für RT-PCR gehören flankiert. Die 5 'und 3' konstante Sequenzen 5 '- TAA TAC GAC TCA CTA TAG GGA GGA CGA TGC GG - 3 "(32 mer) und 5'-TCG GGC GAG TCG TCT G - 3" (16 mer) bzw.. Machen Stammlösung mit Wasser und lagern in Aliquots bei -20 ° C.
- Amplify der Einzelstrang-DNA-Oligo random-Bibliothek (0,4 M) durch PCR mit 3 uM jedes 5'-und 3'-Primer, zusammen mit 2 mM MgCl 2 und 200 uM von jedem dNTP. Um die Fülle der ursprünglichen DNA-Bibliothek zu erhalten, zu begrenzen PCR zu zehn Zyklen. Nach der PCR-Reaktionen (10 Reaktionen 100 ul pro Reaktion), wieder das verstärkte dsDNA-Pool mit einem QIAquick Gel Purification Kit (QIAGEN).
- Konvertieren Sie die daraus resultierenden dsDNA zu einer RNA-Bibliothek mit dem DuraScription Kit (Epicentre, Madison, WI) nach den Anweisungen des Herstellers. In der Transkription Reaktionsgemisch, ersetzen CTP und UTP mit 2'-F-CTP und 2'-F-UTP, Ribonuclease resistent RNA produzieren. In der Regel werden 20 ul der Reaktion mit 1 pg gereinigte DNA-Template, 2 ul 10 x Puffer, 2 ul dATP, 2 ul dGTP, 2 ul 2'-F-dCTP, 2 ul 2'-F-dUTP, 2 ul DTT und 2 ul T7-RNA-Polymerase bei Raumtemperatur und dann bei 37 ° C inkubieren für 6 h (50 mM von jedem dNTP).
- Anschließend verdauen die Reaktion mit DNase I (1,5 mL pro 20-ul T7 Transkriptions-Reaktion), um die Template-DNA zu entfernen und durch eine 8% Polyacrylamid / 7 M Harnstoff-Gel zu reinigen. Quantifizierung der gereinigten RNA-Bibliothek durch UV-Spektroskopie.
2. In-vitro-Herstellung von Aptameren
- Vor Auswahl, Vorbereitung der Auswahl-Puffer und Rückfaltung Puffer. Bereiten Sie einen HEPES-Puffer mit 100 mM Hepes, pH 7,4. Verwenden NaOH auf pH-Wert und dann bei Raumtemperatur lagern anzupassen. Bereiten Sie eine RNA Rückfaltungspuffer (5xHBS) mit 50 mM Hepes pH 7,4, 750 mM NaCl, 5 mM MgCl 2, 5 mM CaCl 2; 13,5 mm KCl. Bereiten Sie eine salzarme RNA Bindungspuffer (10 mM HEPES pH 7,4, 50 mM NaCl, 1 mM CaCl 2, 1 mM MgCl 2, 2,7 mM KCl, 10 mM DTT, 0,01% BSA und eine hohe Salz-RNA Bindungspuffer (10 mM HEPES pH 7,4, 150 mM NaCl, 1 mM CaCl 2, 1 mM MgCl 2, 2,7 mM KCl, 10 mM DTT, 0,01% BSA). Bewahren Sie diese Puffer bei -20 ° C.
- Führen Sie die SELEX hauptsächlich als 1-4 beschrieben. Vor jeder Auswahlrunde, falten die RNA-Pools in 1xHBS Puffer (10 mM HEPES pH 7,4, 150 mM NaCl, 1 mM CaCl 2, 1 mM MgCl 2, 2,7 mM KCl), Hitze bis 95 ° C für 3 min und dann langsam abkühlen bis 37 ° C. Weiter Inkubation bei 37 ° C für 10 min.
- Im Allgemeinen, um unspezifische Bindung mit dem Nitrocellulosefilter, pre-Adsorption minimieren refolded RNA-Pools auf ein Nitrocellulosefilter (HAWP Filter, 0,45 um) für 30 min vor der Inkubation mit dem Ziel HIV-1 gp120-Protein Bal.
- Inkubieren Sie die pre-cleared RNA-Pool mit dem Zielprotein in salzarme RNA Bindungspuffer für 30 min für SELEX Runden 1 bis 4. Nach der vierten Runde der SELEX, verwenden Sie ein High-Salz RNA Bindungspuffer. Mit der SELEX Fortschritt, reduzieren die Menge an gp120-Protein und erhöhen die Wettbewerber Hefe tRNA um die Stringenz der Aptamer-Selektion zu erhöhen.
- Für den ersten Zyklus der Auswahl, bebrüten die bereits geklärten random RNA-Pool (40 ug, 1,5 nmol, 9x10 14 Moleküle) und HIV-1 gp120-Protein Bal (0,23 nmol, RNA / Protein-Verhältnis 6,5 / 1) in 200 ul low- Salz RNA Bindungspuffer auf einer rotierenden Plattform bei Raumtemperatur für 30 min.
- Pass die Reaktion durch eine vorbefeuchtete Nitrocellulosefilter und waschen mit 1 mL Bindungspuffer.
- Eluieren der gebundenen RNA aus dem Filter mit 200 ul Elutionspuffer (7 M Harnstoff und 5 mM EDTA) bei 95 ° C für 5 min, durch Phenol / Chloroform-Extraktion und Konzentration mit einer Microcon YM-30-Säule.
- Reverse Transkription der RNA-Pool wieder mit dem ThermoScript RT-PCR System (Invitrogen) und verstärken für 15 PCR-Zyklen.
- Purify das verstärkte dsDNA-Pool mit einem QIAquick Gel Purification Kit und transkribieren, wie oben für die nächste Runde der Auswahl beschrieben.
3. SELEX Fortschritte Filter Bindungsassay überwacht
- Überwachen Sie den Fortschritt SELEX der Aptamere durch Filter Bindungsassay. Behandeln Sie die RNA-Pool mit CIP zur Einleitung 5'-Triphosphat und dann Etikett mit T4-Polynukleotid-Kinase und γ-32P-ATP zu entfernen.
- Hitze 10 pmol des CIP behandelt RNA-Bibliothek bei 95 ° C für 5 min und dann auf dem Eis kühlen. Anschließend fügen Sie 2ul der PNK-Puffer, 1 ul T4-Polynukleotid-Kinase, 1 ul gamma-P 32-ATP und Wasser auf 20 ul.
- Bei 37 ° C für 30 min, dann mit 20 ^ mu; L Wasser und Reinigung der Reaktionsprodukte von G-50-Säule. Schließlich erhalten 40 ul der markierten RNA bei Endkonzentration von 250 nM.
- Vor dem Test, falten die markierte RNA-Pools in 1xHBS Puffer (10 mM HEPES pH 7,4, 150 mM NaCl, 1 mM CaCl 2, 1 mM MgCl 2, 2,7 mM KCl), Hitze bis 95 ° C für 3 min und dann langsam abkühlen 37 ° C. Weiter Inkubation bei 37 ° C für 10 min.
- Führen Sie eine 100 ul der Bindungsreaktion als Beispiel hier. Inkubieren Sie die Ende-markierten RNA-Pool (10 nM) mit gp120-Protein (100 nM) und einer 10-fachen molaren Überschuss von unspezifischen Konkurrenten tRNA (100 nM) in der High-Salz RNA Bindungspuffer für 30 min.
- Separate einem 50 ul der Bindungsreaktion durch eine pre-Nass-Nitrocellulosefilter.
- Waschen Sie den Filter mit 2 ml Bindungspuffer und zählen Sie die Radioaktivität auf dem Filter durch ein Mehrzweck-Szintillationszähler (Beckman Coulter) erhalten. Als Input-Steuerung, zählen die verbleibenden 50 ul der Bindungsreaktion zur gleichen Zeit. Berechnen Sie den Prozent der RNA auf dem Filter in das Eingabefeld RNA als die Bindungsaffinität beibehalten.
4. Klonierung, Sequenzierung und Ausrichtungen
- Nach 11 Runden, wenn keine weiteren Anreicherung ist auch folgende weitere Auswahlrunden beobachtet dann eine maximale Bindung der RNA-Pool hat möglicherweise nicht erreicht.
- Reverse Transkription der hoch angereichertes Aptamer-Pool (12 th RNA-Pool) mit dem ThermoScript RT-PCR System (Invitrogen) und anschließend verstärken die resultierende cDNA durch PCR. Purify das PCR-Produkt mit einem QIAquick Gel Purification Kit (QIAGEN). Clone dem Gel-gereinigten DNA-Produkt in den TA-Klonierungsvektor pCR 2.1 (Invitrogen). Insgesamt impfen 170 einzelne Klone und weiter zu ermitteln, sie durch DNA-Sequenzierung, um einzelne Sequenzen zu bekommen.
- Klassifizieren Sie die einzelnen Klone in sechs verschiedene Gruppen auf die Ausrichtungen der einzelnen Aptamer-Sequenzen. Wählen Sie einen Vertreter Sequenz aus jeder Gruppe (A-1, A-5, A-9, A-12, A-28 und B-68) zur weiteren Charakterisierung durch ihre relative Häufigkeit innerhalb ihrer Gruppe.
5. Generierung von Aptamer-und Chimäre RNAs durch in vitro Transkription
- Direkt generieren doppelsträngigen DNA-Templates mittels PCR unter Verwendung von 2 pM jeweils von 5'-und 3'-Primer, zusammen mit 2 mM MgCl 2 und 200 uM von jedem dNTP, und Wiederherstellung der resultierenden PCR-Produkte mit einem QIAquick Gel Purification Kit.
- Transcribe Chimäre Sense-Strang aus der PCR erzeugten DNA-Templates mit dem DuraScription Kit (Epicentre, Madison, WI). In der Transkription Reaktionsgemisch, ersetzen Sie die kanonische CTP und UTP mit 2'-F-CTP und 2'-F-UTP, um RNA, die resistent gegen RNase A Abbau ist zu produzieren.
- Typischerweise inkubieren 20 ul der Reaktion mit 1 ug DNA-Vorlage, 2 mL 10xbuffer, 2 ul dATP, 2 ul dGTP, 2 ul 2'-F-dCTP, 2 ul 2'-F-dUTP, 2 ul DTT und 2 ul T7-RNA-Polymerase bei 37 ° C für 6 h, und anschließend zu reinigen es mit Bio-Spin 30 Spalten (Bio-Rad) nach Phenolextraktion und Ethanolfällung.
- Um Interferon-Antwort zu vermeiden, weitere Behandlung der transkribierten RNA durch CIP die Einleitung 5'-Triphosphat zu entfernen. Inkubieren insgesamt 60 ul der Reaktion mit 3 ug von Transkripten, 6 ul Puffer 3 und 0,25 ul der CIP bei 37 ° C für 60 min. Nach Phenol / Chloroform exaction und Ethanolfällung resuspendieren RNA-Pellet in Wasser.
- Zur Vorbereitung der Chimären, kombinieren die Chimären beherbergen nur die Sense-Strang-RNA mit der entsprechenden Antisense-RNA in Rückfaltungspuffer, Hitze bis 95 ° C für 3 min und dann abkühlen auf 37 ° C langsam. Weiter Inkubation bei 37 ° C für 10 min. Führen Rückfaltung Schritt zu einer Kreuzblume 1xHBS Puffer. Zum Beispiel: mix 10 ul 10 uM Chimäre Sense-Strang, 10 ul 10 uM Antisense-Strang und 5 ul Rückfaltungspuffer (5xHBS) in 25 ul-System.
6. Bestimmung von Dissoziationskonstanten mittels Gel-Shift-Assays
- Ende P 32 Etikett der Vertreter Aptamer aus jeder Gruppe und Chimären Sense-Strang und dann falten in 1xHBS Puffer RNA wie oben beschrieben.
- Bereiten Sie 25 ml 5%-Gel hergestellt, indem 2,5 ml 10xTBE Puffer mit 3,125 ml 40% Acrylamid / Bis-Lösung, 19,375 ml Wasser, 150 ul 10% Ammoniumpersulfat (APS)-Lösung und 30 ul TEMED. Das Gel sollte in ca. 30 min polymerisieren. Entfernen Sie vorsichtig den Kamm und einen 30-ml-Spritze mit einer Nadel in die Vertiefungen mit Laufpuffer (1xTBE) waschen ausgestattet.
- Vervollständigen Sie die Versammlung des Gels Einheit und eine Verbindung zu einem Netzteil. Das Gel kann für eine Stunde bei 180 V Vorlauf bei 4 ° C.
- Serienmäßig verdünnen HIV-gp120-Protein 1Bal mit Bindungspuffer auf die gewünschten Konzentrationen. Der letzte reaction Konzentrationen von gp120 sind 0, 1, 5, 10, 20, 40, 80, 160, 320, 640 nM. Inkubieren einer konstanten Menge von 5'-P 32-Ende-markierte RNA (10 nM) mit erhöhten Konzentrationen von gp120-Protein in den Bindungspuffer (insgesamt 20 uL-Reaktion) auf einer rotierenden Plattform bei Raumtemperatur für 30 min.
- Nach der Inkubation mix 20 ul der Bindungsreaktion mit 5 ul nativen Ladepuffer und laden in eine 5% nicht-denaturierenden Polyacrylamidgel. Bereiten Sie eine native Ladepuffer (4x) mit 10 mM Tris-HCl, pH 7,5, 1 mM EDTA, 0,1% Bromphenolblau, 0,1% Xylencyanol FF, 0,1% Orange G, 40% Glycerin. Shop in Aliquots bei -20 ° C.
- Nach der Elektrophorese (180 V bei 4 ° C für 2 Stunden, bis die sekundäre Farbstoff läuft Mitte des Gel) Setzen Sie das Gel auf eine Phosphor Bildschirms und Quantifizierung der Radioaktivität mit einem Typhoon-Scanner.
- Berechnen Sie die Dissoziationskonstanten mit nicht-linearen Kurve Regression mit einer Graph Pad Prism.
7. Cell-Oberfläche Bindungsstudien mittels Durchflusszytometrie
- Generieren fluoreszierenden Aptamer und Chimären mit dem Silencer siRNA Labeling Kit (Ambion). Fügen Sie die folgenden Reagenzien in Ordnung: 22,5 ul Nuklease-freiem Wasser, 5 ml 10 x Labeling Puffer, 15 ul RNA (5 ug), 7,5 ul Labeling Dye. Inkubieren insgesamt 50 ul Kennzeichnung Reaktion bei 37 ° C für 1 Stunde.
- Nach der Inkubation add 5,0 ul (0,1 vol) 5 M NaCl und 125 ul (2,5 vol) kaltes 100% EtOH und gründlich mischen. Inkubation bei -20 ° C für 60 min. Zentrifuge mit hoher Geschwindigkeit bei 4 ° C für 20 min. Überstand entfernen und waschen Pellet mit 175 ul 70% EtOH. Air trockene Pellet in der Dunkelheit und dann auszusetzen markierte RNA in 15 ul der Nuklease-freies Wasser.
- Messen Sie die Absorption der markierten RNA bei 260 nm und am Absorptionsmaximum für den Fluoreszenzfarbstoff. Berechnen Sie die Basis: Farbstoff-Verhältnis und RNA-Konzentration nach, um den Rechner bereitgestellt durch http://www.ambion.com/techlib/append/base_dye.html .
- Mix Cy3-markierten Chimären Sense-Strang und Antisense-Strang und wieder zusammenfalten, in Rückfaltungspuffer wie oben beschrieben.
- Erhalten CHO-WT exprimieren gp160 und CHO-EE Kontroll-Zellen durch die Aids-Forschung und Referenzreagens Programm 5, 6. Wachsen Zellen in GMEM-S-Medium (Glutamin-defizienten minimal essential medium mit 400 uM Methioninsulfoximin (MSX)) (Gibco, Invitrogen). Kultur der Zellen in einem befeuchteten 5% CO 2-Inkubator bei 37 ° C.
- Waschen Sie die CHO-WT gp160 oder CHO-EE Kontroll-Zellen mit vorgewärmtem Waschpuffer, trypsinize und lösen von den Platten. Nach dem Waschen die Zellen zweimal mit 500 ul Bindungspuffer, resupend die Zellpellets in Bindungspuffer und bei 37 ° C für 30 min. Pellet-Zellen und dann resuspendieren sie in 50 ul vorgewärmtem Bindungspuffer mit 400 nM Cy3-markierten experimentellen RNAs.
- Nach Inkubation bei 37 ° C für 40 min, waschen Sie die Zellen dreimal mit 500 mL vorgewärmtes Bindungspuffer und schließlich in 350 ul Bindungspuffer resuspendiert vorgewärmt auf 37 ° C und sofort mittels Durchflusszytometrie analysiert.
8. Internalisierung und intrazelluläre Lokalisation Studien von Live-Zell-konfokale Mikroskopie
- Vor 1 Tag von Assay, wachsen die CHO-WT gp160 und CHO-EE Kontroll-Zellen in 35 mm-Platte mit Aussaat in 0.3x10 6 in 2 mL GMEM-S mittel-bis etwa 70% Konfluenz in 24 h erlauben
- Am Tag der Experimente, waschen Sie die Zellen mit 1 ml vorgewärmtem PBS. Und mit 1 ml vorgewärmten vollständigen Wachstumsmedium für 30 min bei 37 ° C.
- Bereiten Cy3-markierten Aptamer-siRNA Chimären wie oben beschrieben. Inkubieren Sie die falten Aptamer-Stick mit dem siRNA-Stick mit 5'-Cy3-markierten Sense-Strang der Aptamer-stick-siRNA konjugierter Form wie oben beschrieben.
- Vor dem Test, bereiten 0,15 mg / ml Lösung von Hoechst 33342 (nuklearen Farbstoff für lebende Zellen, Molecular Probes, Invitrogen, CA) in Wasser und lagern in Aliquots bei 4 ° C.
- Stain die Zellen durch die Behandlung mit 0,15 mg / mL Hoechst 33342 für 15 min bei 37 ° C. Sofort auswaschen Farbstoff mit 1,0 ml frisches Medium zweimal und ersetzen 2 mL vorgewärmtes frisches Medium.
- Fügen Sie die Cy3-markierten Aptamer-siRNA-Chimäre auf 100 nM Endkonzentration in den Medien und inkubieren für Live-cell konfokale Mikroskopie in einer 5% igen CO2-Mikroskopie Brutschrank bei 37 ° C.
- Sammeln Sie die Bilder alle 15 min mit einem Zeiss LSM 510 Meta Inverted 2 Photonen konfokalen Mikroskop-System unter Eintauchen in Wasser bei 40-facher Vergrößerung.
9. In-vitro-HIV-1 Herausforderung und p24-Antigen-Test
- Purchase CCRF-CEM-Zellen von ATCC. Wachsen in RPMI-1640 (CellGro, Mediatech Inc.) ergänzt mit 10% fötalem Rinderserum (FBS, HyClone), L-Glutamin und 1xPen-Strep (Gibco, Invitrogen). Kultur-Zellen in einem befeuchteten 5% CO 2-Inkubator bei 37 ° C.
- Erhalten peripheren Blut mononuclear Proben von gesunden Spendern aus der City of Hope National Medical Center (Klinikpersonal).
- Isolieren PBMCs aus Vollblut durch Zentrifugation durch Ficoll-Hypaque Lösung (Histopaque-1077, Sigma). Führen CD8-Zellen (T-cytotoxic/suppressor-Zellen) aus PBMCs mit Dynabeads CD8 (Invitrogen, CA).
- Wachsen Zellen in T-Zell-aktives Medium (BioE, St. Paul, MN). Kultur-Zellen in einem befeuchteten 5% CO 2-Inkubator bei 37 ° C.
- Erhalten HIV-1 IIIB und NL4-3-Virus und HIV-1 Bal-Virus von der AIDS-Forschung und Referenzreagens Program. Nach Ausbreitung des Virus, speichern in Aliquots bei -80 ° C.
- Infect CCRF-CEM-Zellen oder humanen PBMCs mit HIV-Viren (IIIB, NL4-3-oder Bal) (MOI 0,001 oder 0,005). Nach 24 Stunden post-Infektion, vorsichtig waschen Zellen mit PBS dreimal auf freie Virus zu entfernen. Weiter zur Kultur der infizierten Zellen in einem 5% CO 2-Mikroskopie Brutschrank bei 37 ° C für 4 Tage.
- Vor Behandlungen RNA, vorsichtig waschen die infizierten Zellen mit PBS dreimal auf freie Virus zu entfernen. Inkubieren 2x10 4 infizierten Zellen und 3x10 4-infizierten Zellen mit refolded experimentellen RNAs bei 400 nM Endkonzentration in 96-Well-Platten bei 37 ° C (100 ul pro Well, Triplex-Assay).
- Sammeln Sie die Kulturüberstände (10 ul pro Well) zu verschiedenen Zeitpunkten (3 d, 5 d, 7 d und 9 d) und bei -20 ° C bis p24-Assay.
- Führen Sie die p24-Antigen-Analysen mit einem HIV-1 p24 Antigen ELISA-Kit.
10. Die siRNA-Funktion Erkennung durch quantitative RT-PCR-Assay
- Infect CCRF-CEM-Zellen oder humanen PBMCs mit HIV-Viren (IIIB, NL4-3-oder Bal) und Behandlung mit den experimentellen RNAs (400 nM) wie oben beschrieben.
- Nach 7 Tagen Behandlung, Pellet-Zellen und Isolierung von Gesamt-RNA mit STAT-60. Behandeln Sie die Gesamt-RNA mit DNase I, um genomische DNA zu entfernen und cDNA mit 2 ug Gesamt-RNA.
- Mischen Sie die folgenden Reagenzien: 8 ml Nuklease-freies Wasser, 1,5 mL 10 x DNase-Puffer, 4 ul RNA (2 g), 0,5 ul RNain Inhibitor und 1,0 ul RNase-freie DNase I. inkubieren insgesamt 15 ul Reaktion 37 ° C für 1 Stunde und Wärme bei 80 ° C für 10 min bis DNase I. Unmittelbar inaktivieren, chill die Reaktion auf dem Eis.
- Add 2 ul random primer (50 ng / mL) und 1μL dNTP (10 mM) in das Reaktionsgemisch vor, und dann Wärme bei 65 ° C für 5 min. Unmittelbar, chill die Reaktion auf dem Eis.
- Fügen Sie die folgenden Reagenzien: 5 mL 5xFirst Strang-Puffer, 2,5 mL 0,1 M DTT, 0,5 mL RNain Inhibitor und 1,0 mL MMLV-RT. Inkubieren insgesamt 27 mL Reaktion bei 25 ° C für 10 min und bei 37 ° C für 1 Stunde. Nach der Reaktion Wärme-Gemisch bei 70 ° C für 15 min auf Reverse-Transkriptase zu inaktivieren und dann kalt auf dem Eis. Die cDNA ist bereit für qRT-PCR-Analyse.
- Analyze Expression des Ziel-Gene durch quantitative RT-PCR unter Verwendung von 2 x iQ SyberGreen Mastermix und spezifischen Primer-Sets in einer Endkonzentration von 400 nM (Triplex-Assay). Verwenden Sie die GAPDH-Expression als interne Kontrolle für die Normalisierung der qPCR-Daten.
11. Repräsentative Ergebnisse:
1. New RNA-Aptamere gegen HIV-1 BaL gp120 sind isoliert und charakterisiert.
Wie in den experimentellen Teil, eine erste DNA-Oligonukleotid-Bibliothek mit einem 50 nt random Region durch feste Grundierung Regionen auf der 5 'und 3' Enden wird verstärkt und transkribiert in ein RNA-Pool flankiert beschrieben. Diese erste Bibliothek besteht aus bis zu 10 15 verschiedenen Sequenzen (1 nmol), die in eine Vielzahl von verschiedenen 3-D-Strukturen falten. Die hohe Komplexität und Vielfalt der ursprünglichen Bibliothek könnte garantieren, das Vorhandensein von aktiven Strukturen mit guter Bindungsaffinität zum Ziel.
Verwenden eine in vitro SELEX-Verfahren (Abb. 1) zu 2'-fluor-modifizierten RNA-Aptameren, die selektiv an die R5-Stamm HIV-1 BaL Hüllprotein gp120 7 wählen. Wie in Abbildung 1 gezeigt, ist eine Nitrozellulose-Basis Auswahl Strategie durchgeführt, um spezifische Ziel-bindenden RNAs aus Nicht-bindende RNA-Moleküle zu isolieren. Da das Protein-Sticks auf Nitrozellulose, nur die RNA / Protein-Komplexe oder Aggregate können auf der Membran zurückgehalten werden und frei RNAs werden ausgewaschen. Unter denaturierenden Bedingungen werden die gebundenen RNAs erholt und sind umgekehrt zu cDNA transkribiert und dann verstärkt in dsDNA und anschließend in vitro transkribiert, um eine neue RNA-Pool für die nächste Auswahl-Zyklus zu schaffen. Die Auswahl Stringenz wird durch Verringerung der Menge an Zielprotein und die Erhöhung der Menge an Wettbewerber tRNA erhöht. Die Höhe des RNA-Pools, Protein-und Wettbewerbsanalysen tRNA in jeder Auswahlrunde ist in Tabelle 1 dargestellt.
Überwachen Sie den Fortschritt der Auswahl nach jeder SELEX-Zyklus durch den Filter Bindungsassay. Beurteilen Sie die Bindungsaffinität als Prozent der RNA erhalten auf der FILter in der Gesamt-RNA-Pool. Die Ausgangs-RNA-Pool (1-RNA) zeigt nur 0,1% des Eingangs-RNAs auf die Membran zurückgehalten. Doch nach neun Auswahlrunden neunten RNA-Bibliothek (9-RNA) hat 9,72% der Eingangs-RNA gebunden. Obwohl zusätzliche Auswahlrunden durchgeführt wurden, ist keine weitere Anreicherung beobachtet, was darauf hindeutet, dass eine maximale Bindung des RNA-Pools erreicht wurde (Abbildung 2A). Ähnliche mit dem Filter Bindungsassay, das Gel-Shift-Assay ist auch einer der beliebtesten Strategien zur Bestimmung Dissoziationskonstanten. Dieses Verfahren ist einfach und bequem. Wie in Abbildung 2B gezeigt, Gel-Shift-Assays bestätigen die verbindliche Aktivitäten der RNA-Pools. Diese Ergebnisse zeigen, dass einige Liganden mit hoher Spezifität binden für das Zielprotein nacheinander in dieser RNA-Pools bereichert ..
Clone und die Reihenfolge der hoch angereichertes Aptamer-Pools (12-RNA). Nach der Ausrichtung der einzelnen geklonten Aptamersequenzen sind sechs verschiedene Gruppen eingeteilt, wie in Tabelle 2 dargestellt. Über 40% der Klone (Gruppe I und II Aptamere) enthalten einen konservierten Sequenz: A (A / G) TTGAGGGACC (A / G). Wir wählen einen Vertreter Sequenz aus jeder Gruppe (zum Beispiel: A-1, A-5, A-9, A-12, A-28 und B-68) zur weiteren Charakterisierung durch ihre relative Häufigkeit innerhalb ihrer Gruppe. Durch eine native Gel-Mobility-Shift-Assay werden die Dissoziationskonstanten (K d) dieser repräsentativen Aptamere berechnet (Abbildung 3A). Zum Beispiel hat A-1, das Beste aus der Aptamere, einer scheinbaren Kd-Werte von 52 nM (Abbildung 3B). Wie in Abbildung 3A gezeigt, können diese selektierten Aptamere gezielt mit dem Ziel HIV-1 gp120 bindet Bal, aber nicht die HIV gp120 CM-Protein.
2. Anti-gp120-Aptamer spezifisch bindet und wird von Zellen, die HIV gp160 verinnerlicht und hemmen HIV-1-Infektion in der Zellkultur.
CHO-Zellen gp160 stabil exprimieren die HIV-Envelope-Glykoprotein gp160 werden verwendet, um für die Bindung und Internalisierung der ausgewählten Anti-gp120-Aptamere zu testen. Diese Zellen nicht verarbeitet gp160 in gp120 und gp41, da sie die gag codiert Proteasen für Umschlag Verarbeitung benötigt fehlt. Zur Kontrolle verwenden wir die Eltern CHO-EE-Zelllinie, die nicht exprimiert gp160. Durchflusszytometrische Analysen (Abbildung 4A) zeigen, dass die Cy3-markierte Aptamere binden spezifisch an die CHO-Zellen gp160, nicht aber die Kontrolle CHO-EE-Zellen. Darüber hinaus zeigt Echtzeit-Live-Zelle Z-Achse der konfokalen Mikroskopie, dass die Cy3-markierten Aptamer selektiv in den CHO-Zellen gp160 (Abbildung 4B und 4C) nach 2 Stunden Inkubation verinnerlicht, aber nicht die CHO-EE Kontroll-Zellen ( Abbildung 4D). 4C zeigt auch, dass das Aptamer in das Zytoplasma, die das gp120-Aptamere geben können Zellen über Rezeptor-vermittelte Endozytose schlägt aggregiert.
In der HIV-1-Assay Herausforderung, HIV-1 infizierten PBMC-Zellen mit den Aptameren behandelt. An verschiedenen Tagen nach der Behandlung mit den Aptameren, werden Aliquote der Medien für die virale p24-Antigen-Werten (Abb. 4E) untersucht. Die Ergebnisse zeigen, dass die anti-gp120-Aptamere (A-1) HIV-1 p24-Produktion mit einer Nano-Molare Konzentration hemmt.
3. Anti-gp120-Aptamer-siRNA Chimäre ist so konzipiert und evaluiert seine Wirksamkeit als Zelltyp-spezifische siRNA-System
Wie in Abbildung 5A, die Aptamer-und Sense-Strang Segment der siRNAs enthalten Nuklease-resistenten 2'-Fluoro UTP und 2'-Fluoro CTP gezeigt und sind von den entsprechenden dsDNA Vorlagen von in vitro-Transkription synthetisiert Bakteriophagen. Um die Flexibilität des Moleküls, zwei Nukleotid-Linker zu erhöhen (UU) ist zwischen dem Aptamer und die Dicer Substratabschnittes eingefügt. Zur Vorbereitung der siRNA mit Chimären sind in vitro transkribiert chimären Aptamer-Sense-Strang Polymere mit äquimolaren Konzentrationen eines unmodifizierten Antisense-Strang-RNA-geglüht. Diese Daten stammen aus Gel-Shift-Assay (Abbildung 5B) und Durchflusszytometrie (Abbildung 5C) zeigen, dass die Chimären ungefähr die gleiche Bindungsaffinität wie die Aptamere allein aufrecht zu erhalten. Der zeitliche Verlauf Bilder von real-time konfokale Mikroskopie (Abbildung 5D) zeigen, dass Cy3-markierten Chimäre Ch A-1 erfolgreich in das Zytoplasma der Zellen internalisiert werden. Wie erwartet, ist keine Aufnahme von der Chimäre mit dem CHO-EE Kontroll-Zellen (Abb. 5E) beobachtet.
Ebenso ist die antivirale Potential von RNAs, die von HIV-1-Assay Herausforderung ausgewertet. Die Ergebnisse der HIV p24-Antigen-Analysen (Abbildung 6A und 6B) zeigen, dass sowohl Aptamer und Chimäre p24 hemmen, aber die stärkste Hemmung ist mit der Chimäre Ch A-1-Behandlung beobachtet.
Um zu bestätigen, dass die siRNA-Komponente funktioniert zusammen mit dem aptamer nach Internalisierung der Ch A-1-Chimäre in infizierten Zellen, bewerten wir auch die relative Bedeutung der Hemmung der tat / rev Genexpression durch quantitative RT-PCR-Assays Ausdruck. Wir finden, dass die Behandlung von infizierten Zellen mit den Chimären der Lage zu induzieren Silencing der tat / rev-Gen ist, während die Aptamer allein hatte keinen Einfluss tat / rev Genexpression (Abbildung 6C und 6D). Diese Ergebnisse liefern weitere Unterstützung, dass das Aptamer geliefert siRNA RNAi auslöst.
| Die Menge an Protein, verwendet RNA-Pool und tRNA zur Auswahl | |||||
| SELEX Runden | Verhältnis von Target / RNA | Gp120-Protein | RNA-Pool | Competitor tRNA | Selection Buffer |
| 1 | 1/6.5 | 229,8 pmol | 1,5 nmol (40,1 ug) | 0 | Low Salz SELEX-Puffer |
| 2 | 1/6.5 | 114,9 pmol | 0,75 nmol (20,1 ug) | 0,25 nmol (6,6 ug) | |
| 3 | 1 / 8 | 76,6 pmol | 0,625 nmol (16,7 ug) | 0,25 nmol (6,6 ug) | |
| 4 | 1 / 8 | 76,6 pmol | 0,625 nmol (16,7 ug) | 0,25 nmol (6,6 ug) | |
| 5 | 1 / 8 | 38,3 pmol | 0,306 nmol (8,18 ug) | 0,5 nmol (13,2 ug) | Hohe Salz SELEX-Puffer |
| 6 | 1 / 8 | 38,3 pmol | 0,306 nmol (8,18 ug) | 0,5 nmol (13,2 ug) | |
| 7 | 1 / 10 | 26,8 pmol | 0,268 nmol (7,16 ug) | 0,5 nmol (13,2 ug) | |
| 8 | 1 / 10 | 26,8 pmol | pmol 0,268 nmol (7,16 ug) | 1 nmol (26,4 ug) | |
| 9 | 1 / 10 | 15,3 pmol | 0,153 nmol (4,09 ug) | 1 nmol (26,4 ug) | |
| 10 | 1 / 10 | 15,3 pmol | 0,153 nmol (4,09 ug) | 1,5 nmol (39,6 ug) | |
| 11 | 1/12.5 | 7,66 pmol | 0,096 nmol (2,56 ug) | 2 nmol (52,8 ug) | |
| 12 | 1/12.5 | 7,66 pmol | 0,096 nmol (2,56 ug) | 2 nmol (52,8 ug) | |
Tabelle 1. Die Auswahl Bedingungen. Die Menge an Protein, sind RNA-Pool und tRNA für jede Auswahl und Selektion verwendete Puffer angegeben.
| Gruppe | RNA | Zufällige Folgen | Frequency (140 Klone) |
| Gruppe I | A-1 | AATTGAGGGACCA CGCGCTGCTTGTTGTGATAAGCAG TTTGT CG GATGG | 33 (23.6%) |
| B-7 | AATTGAGGGACCA ACGCGAGGATGTGGATAGTGTGTA TTTGC GT GATGG | 3 | |
| A-32 | AATTGAGGGACCG TTGGTAAAAGCCGGA AATTG AGCT TTTAC GGC GATGG | 5 | |
| B-55 | AATTGAGGTACCGCG TTATTAGGAACA AATTG GAATTCTAAACGC GATGG | 2 | |
| A-24 | AATAGAGGGACC CAGATATAGGCTACACGGATGATGGTGTATCTG GATGG | 1 | |
| B-19 | AATAGAGGAACCG TTTCAGAAGACTACAGGTTAGTCCAATGAAGC GACGG | 1 | |
| B-31 | AATAGAGGGACCG TGGACAATAATTTATGGTCA TTTATTGGCAC GATGG | 1 | |
| Gruppe II | A-12 | AGTAGAGGAACCA AGCAATGGATGAATGCAAAAGTGTAAATGCTT GATGG | 10 (7,1%) |
| Gruppe III | A-9 | TGAGTTTGGGTAAATTTCCGGTTTCGGTTTACTCACGAAAGATCGGTCGG | 15 (10,7%) |
| Gruppe IV | A-28 | TAAAGGAGGGAAGGATGAGACCGCACGAAAAATATCAGCATACG TTTGTG | 10 (7,1%) |
| Gruppe V | A-5 | GAAACTAGTTTGAATAATGGTGTAGAGGAGGGTCAATAGTTTCG TTGGTG | 9 (6,4%) |
| Gruppe VI | B-68 | ACATAGTAATGACACGGAGGATGGAGAAAAAACAGCCATCTCTTGACGGT | 2 |
| Andere | Orphan-Sequenz | 48 |
Tabelle 2. Die Ausrichtung und die Identifizierung von RNA-Aptameren. Nach der 12. Runde der Selektion wurde die ausgewählten RNA-Pool kloniert und sequenziert. Nach der Ausrichtung der alle 140 Klone wurden sechs Gruppen von anti-gp120-Aptamere identifiziert. Nur die zufälligen Sequenzen der Aptamer-Kernregionen (5'-3 ') angegeben sind. Isoliert auftretende mit mehreren Frequenzen angegeben sind.
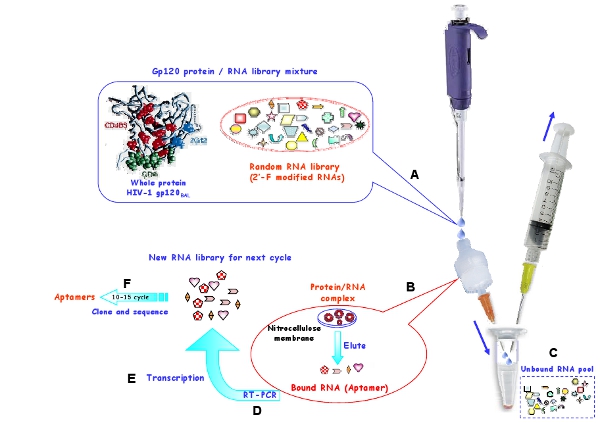
Abbildung 1: Schematische Darstellung der in vitro Auswahlverfahren mit einer Nitrozellulosemembran, zur Erzeugung von RNA-Aptameren für HIV-1 gp120-Protein Bal. (A) Die Ausgangs-RNA-Pool und Zielprotein inkubiert wurden, um komplexe Form. (B) Die gebundene RNA-Moleküle wurden auf der Membran zurückgehalten und eluiert aus der Membran unter denaturierenden Bedingungen. (C) Die ungebundenen RNAs wurden weggespült. (D) Die ausgewählten RNAs wurden revers transkribiert und durch PCR amplifiziert. (E) Die entsprechenden DNA wurde anschließend in neue RNA-Pool für die nächste Auswahl-Zyklen transkribiert. (F) Nach 10-15 Auswahlrunden wurden die selektierten Aptamere geklont und Sequenz.
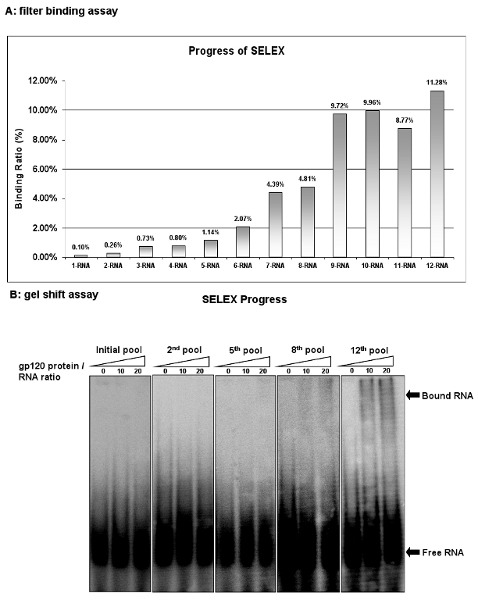
Abbildung 2: Der Fortschritt der HIV-1 gp120 Aptamere Auswahl. (A) Die Bindung Aktivität der RNA-Pool bei jedem Zyklus wurde durch Filter Bindungsassay mit Konkurrenten tRNA analysiert. Binding-Aktivitäten wurden als Prozentsatz der Eingang RNA auf dem Filter zurückgehaltenen in Protein / RNA-Komplex berechnet. (B) Die Bindung Aktivität der RNA-Pool bei jedem Zyklus wurde durch Gel-Shift-Assay analysiert. Die 12 th RNA-Pool zeigte die höchste Bindungsaffinität.
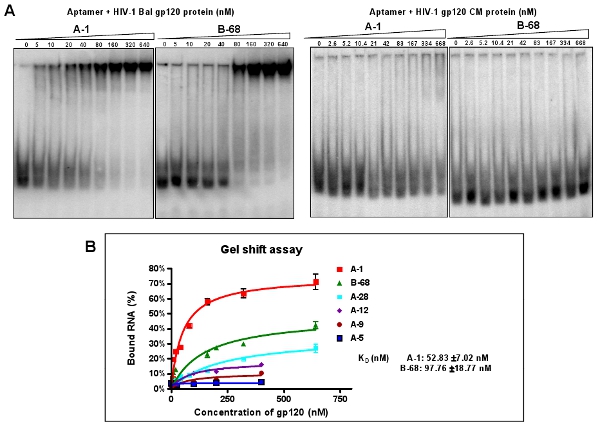
Abbildung 3: Binding Aktivitätstest von ausgewählten einzelnen Aptamere gegen HIV-1 gp120 Bal. (A) Das 5'-Ende P 32 gekennzeichnet individuelle Aptamere wurden mit der zunehmenden Mengen an Ziel-gp120-Protein oder unspezifische CM-Protein inkubiert. Die Bindung Reaktionsmischungen wurden durch eine Gel-Mobility-Shift-Assay analysiert. Aptamer A-1 und B-68 zeigte die beste Bindungsaffinität mit dem Zielprotein, aber nicht CM-Protein. Die Daten repräsentieren den Durchschnitt von vier Wiederholungen. (B) Binding-Kurve aus einem Gel-Shift-Assay.
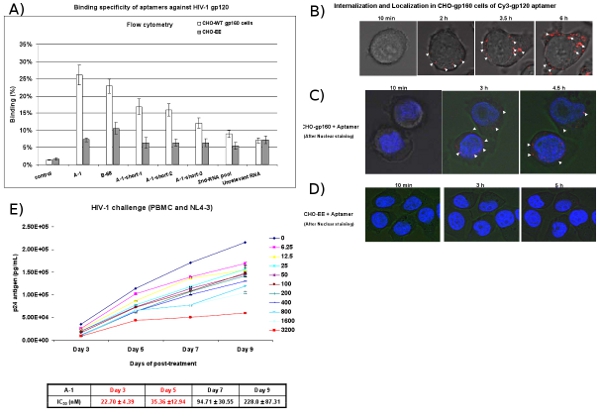
Abbildung 4: Cell-spezifische Bindung und Aufnahme-Studien von Aptameren. (A) Zelloberflächenbindung der Cy3-markierten RNAs wurde mittels Durchflusszytometrie untersucht. Cy3-markierten RNAs wurden für die Bindung an CHO-gp160-Zellen und CHO-EE Kontroll-Zellen getestet. Die selektierten Aptamere zeigten Zelltyp-spezifische Bindungsaffinität. Die 2. RNA-Pool und irrelevant RNAs wurden als negative Kontrollen verwendet. Die Daten repräsentieren den Durchschnitt der drei Wiederholungen. (B) Internalisierung Analyse. CHO-gp160-Zellen wurden in 35 mm-Platten kultiviert und inkubiert mit 100 nM Konzentration von Cy3-markierten A-1 in Kulturmedien für die Echtzeit-Live-cell-konfokale Mikroskopie Analyse. Die Bilder wurden bei 15 min gesammelt. Intervalle mit 40-facher Vergrößerung. (C, D) Localization Analyse. CHO-gp160 Zellen und CHO-EE Kontroll-Zellen wurden in 35 mm-Platten kultiviert. Vor der Inkubation mit 100 nM von Cy3-markierten A-1, wurden die Zellen mit Hoechst 33342 (nuklearen Farbstoff für lebende Zellen) gefärbt und anschließend mit Hilfe der Echtzeit-konfokale Mikroskopie. (E) Der gewählte anti-gp120-Aptamere hemmt HIV-1-Replikation in humanen PBMCs zuvor mit HIV-1 NL4-3-Virus infiziert. Unterschiedliche Konzentrationen und Zeit pointes wurden vorgestellt. IC50-Wert wurde notiert. Daten stellt den Durchschnitt der drei Messungen des p24.
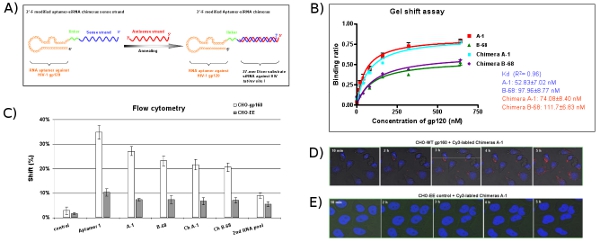
Abbildung 5: Das Design und die Auswertung der Aptamer-siRNA Chimäre Delivery-System. (A) Schematische Aptamer-siRNA chimärer RNAs: im Bereich der anti-gp120 Aptamer ist verantwortlich für die Bindung an gp120 und die siRNA zielt auf eine gemeinsame Exon von HIV-1 tat / rev. Die 2'-Fluoro geändert Aptamer-siRNA Sinne Einzelstrang wurde co-transkribiert, gefolgt von Glühen der komplementären siRNA Antisense-Strang, um das chimäre Molekül abzuschließen. Ein Linker (UU) zwischen dem Aptamer-und siRNA wird in grün angezeigt. (B) Die Aptamer-siRNA chimärer RNAs, die vergleichbar Kd-Werte sowie die elterliche Aptamere binden spezifisch die HIV Bal gp120-Protein. Die Daten repräsentieren den Durchschnitt der drei Wiederholungen. (C) Cell-Typ spezifische Bindung Studien von Aptameren. Cy3-markierten RNAs wurden für die Bindung an CHO-gp160-Zellen und CHO-EE Kontroll-Zellen getestet. Zell-surGesicht Bindungen Cy3-markierten RNAs wurden mittels Durchflusszytometrie untersucht. Die selektierten Aptamere zeigten Zelltyp-spezifische Bindungsaffinität. Die 2. RNA-Pool und irrelevant RNA wurden als negative Kontrollen verwendet. Die Daten repräsentieren den Durchschnitt von zwei Wiederholungen. (D, E) Internalisierung und intrazelluläre Lokalisation analysiert. CHO-gp160-Zellen wurden in 35 mm-Platten kultiviert und wurden mit Hoechst 33342 (nuklearen Farbstoff für lebende Zellen) gefärbt. Anschließend wurden die Zellen in Kulturmedium mit 100 nM Konzentration von Cy3-markierten Chimären für die Echtzeit-Live-cell konfokale Mikroskopie Analyse, wie oben beschrieben inkubiert.
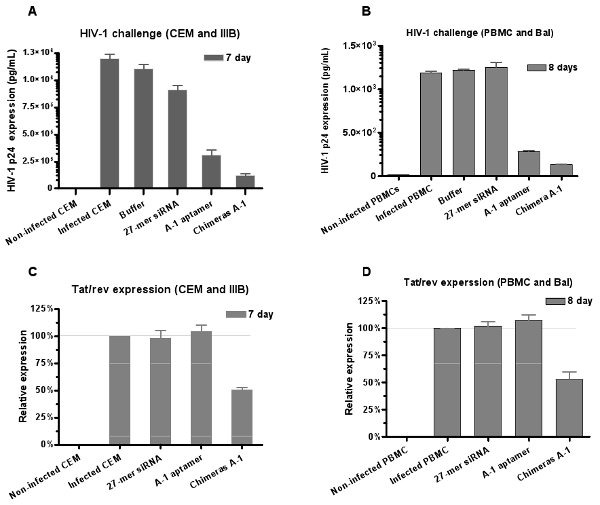
Abbildung 6: Dual Hemmung von HIV-1 Infektion durch Aptamer-siRNA Chimären vermittelt. Sowohl anti-gp120-Aptamer-und Aptamer-siRNA Chimären neutralisiert HIV-1 Infektion in (A) CEM-Zellen (IIIB-Stamm) und (B) humanen PBMCs (BAL Stamm) Kultur, bzw.. Die Daten repräsentieren den Durchschnitt der drei Messungen des p24. Der Chimären zeigten deutlich stärkere Inhibition als Aptamer allein darauf hinweist, dass (C, D) der siRNA durch die Aptamere herunter reguliert tat / rev Genexpression in den PBMCs geliefert. Die Daten repräsentieren den Durchschnitt der drei Wiederholungen.
Diskussion
Aptamere sind in vitro entwickelt Nukleinsäuren, die spezifische und stabile dreidimensionale Formen annehmen, wodurch eine sehr spezifische, enge Bindung an gezielten Moleküle 8. Die niedrigen nanomolaren Bindungsaffinität und exquisite Spezifität der Aptamere, um ihre Ziele machen vielseitige Werkzeuge für Diagnose, Bildgebung in vivo, und Therapeutika 9. Mit dem Aufkommen der Aptamer-Technologie zur gezielten siRNA Anlieferung ist es nun möglich, die Aptamer-Bindung-Funkt...
Offenlegungen
Danksagungen
Wir bedanken uns bei Britta Hoehn, Guihua Sun, Harris Soifer und Lisa Scherer für hilfreiche Diskussionen. Die CHO-EE-und CHO-Zellen gp160: Diese Arbeit wurde durch Zuschüsse der National Institutes of Health AI29329 und HL07470 verliehen Folgende Reagenzien JJR wurden durch die NIH AIDS Research and Reference Reagent Program, Division of AIDS, NIAID, NIH erhalten Unterstützung Linie, die pNL4-3 luc-Vektor; HIV-1 BaL gp120 aus DAIDS, NIAID.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog-Nummer | Kommentare (optional) |
|---|---|---|---|
| MF-Millipore Membranfilter | Millipore | HAWP01300 | Porengröße 0,45 um |
| Swinnex Filterhalter | Millipore | SX0001300 | 13 mm Durchmesser |
| QIAquick Gel Extraction Kit | QIAGEN | 28706 | DNA-Aufreinigung |
| Microcon YM-30 Säule | Millipore | 42410 | RNA-Konzentration |
| Bio-Spin-30 Säulenvolumina | Bio-Rad | 732-6250 | RNA-Reinigung |
| Taq PCR-DNA-Polymerase | Sigma-Aldrich | D1806 | |
| ThermoScript RT-PCR-System | Invitrogen | 11146-024 | |
| DuraScribe T7 Transkription Kit | EpiCentre | DS010925 | |
| dNTP für PCR | Roche | 1 581 295 | |
| Ribonukleinsäure, Transfer vom E.coli | Sigma-Aldrich | R1753 | tRNA Wettbewerber |
| HIV-1 Ba-L gp120-Protein | die AIDS-Forschung und Referenzreagens Programm | 4961 | Zielprotein |
| Silencer siRNA Labeling Kit - Cy3 | Ambion | 1632 | |
| Saure Phenol / Chloroform 5 / 1 Lösung (pH 4,5) | Ambion | AM9720 | |
| Chloroform / Isopropanol 24 / 1 Lösung | Sigma | C0549 | |
| Kalbsdarmphosphatase (CIP) | New England Biolab | M0290L | |
| T4-Polynukleotid-Kinase | New England Biolab | M0201L | |
| Glykogen | Roche | 10 901 393 001 | RNA Niederschläge |
| Gamma-P 32-ATP | MP Biomedical | 013502002 | Radiactivity |
| 40% AccuGel 19.01 | National Diagnostics | EC-850 | |
| 10xTBE | National Diagnostics | EC-860 | |
| N, N, N, N'-Tetramethylethylendiamin (TMEMD) | Sigma-Aldrich | T9281 | |
| Ammoniumpersulfat (APS) | Sigma-Aldrich | A3678 | |
| L-methioine Sulfoximin | Sigma-Aldrich | M5379-250 mg | |
| RPMI Medien 1640 | Invitrogen | 11835-030 | |
| Natriumhydrogencarbonat-Lösung, 7,5% w / v | Invitrogen | 25080-094 | |
| Minimum Essential Medium (MEM) (10 ) | Invitrogen | 11430-030 | |
| MEM nicht essentielle Aminosäure (100 ) | Invitrogen | 11140-050 | |
| TA Cloning-Kit mit pCR 2.1 | Invitrogen | K2040-01 |
Referenzen
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Robertson, D. L., Joyce, G. F. Selection in vitro of an RNA enzyme that specifically cleaves single-stranded DNA. Nature. 344, 467-468 (1990).
- Fitzwater, T., Polisky, B. A SELEX primer. Methods Enzymol. 267, 275-301 (1996).
- Weiss, C. D., White, J. M. Characterization of stable Chinese hamster ovary cells expressing wild-type, secreted, and glycosylphosphatidylinositol-anchored human immunodeficiency virus type 1 envelope glycoprotein. J Virol. 67, 7060-706 (1993).
- Vodicka, M. A. Indicator cell lines for detection of primary strains of human and simian immunodeficiency viruses. Virology. 233, 193-198> (1997).
- Zhou, J. Selection, characterization and application of new RNA HIV gp 120 aptamers for facile delivery of Dicer substrate siRNAs into HIV infected cells. Nucleic Acids Res. , (2009).
- Mayer, G. The chemical biology of aptamers. Angew Chem Int Ed Engl. 48, 2672-2689 (2009).
- Famulok, M., Hartig, J. S., Mayer, G. Functional aptamers and aptazymes in biotechnology, diagnostics, and therapy. Chem Rev. 107, 3715-3743 (2007).
- Chu, T. C., Twu, K. Y., Ellington, A. D., Levy, M. Aptamer mediated siRNA delivery. Nucleic Acids Res. 34, e73-e73 (2006).
- McNamara, J. O., 2nd, . Cell type-specific delivery of siRNAs with aptamer-siRNA chimeras. Nat Biotechnol. 24, 1005-1015 (2006).
- Dassie, J. P. Systemic administration of optimized aptamer-siRNA chimeras promotes regression of PSMA-expressing tumors. Nat Biotechnol. 27, 839-849 (2009).
- Zhou, J., Rossi, J. J. The therapeutic potential of cell-internalizing aptamers. Curr Top Med Chem. 9, 1144-1157 (2009).
- Zhou, J., Li, H., Li, S., Zaia, J., Rossi, J. J. Novel dual inhibitory function aptamer-siRNA delivery system for HIV-1 therapy. Mol Ther. 16, 1481-1489 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten