Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Развитие сотового типа конкретных анти-ВИЧ gp120 аптамеры для доставки миРНК
В этой статье
Резюме
Несколько 2'-фтор РНК аптамеры против ВИЧ-1BA-L gp120 с наномоля близость изолированы от библиотеки РНК В пробирке SELEX процедуры. Новый двойной тормозной функции анти-gp120 аптамер-миРНК химера создается и показывает значительные предпосылки для системных анти-ВИЧ-терапии.
Аннотация
Глобальная эпидемия ВИЧ-инфекции создало насущную необходимость новых классов антиретровирусных препаратов. Мощные способности малых интерферирующих (SI) РНК для подавления экспрессии дополнительных транскриптов РНК в настоящее время используются в качестве нового класса терапевтических средств для целого ряда заболеваний, включая ВИЧ. Многие предыдущие отчеты показали, что роман RNAi основе Анти-ВИЧ/СПИД терапевтических стратегий широкие перспективы, однако основным препятствием для успешного терапевтического применения и клинических перевод siRNAs является эффективной доставки. Особенно, принимая во внимание безопасность и эффективность RNAi основе терапии, крайне желательно разработать целевые внутриклеточной доставки миРНК подход к конкретной популяции клеток или тканей. ВИЧ-1 gp120 протеин, гликопротеин конверт на поверхности ВИЧ-1, играет важную роль в вирусные вступления в клетки CD4. Взаимодействие gp120 и CD4, который вызывает ВИЧ-1, вход и инициирует слияние клеток была подтверждена как клинически значимые антивирусной стратегии для открытия новых лекарств.
При этом, во-первых, мы обсудим выбор и идентификация 2'-F изменение анти-ВИЧ gp120 аптамеров РНК. Использование обычных нитроцеллюлозных фильтров метод SELEX, несколько новых аптамеры с наномолярных близости были выделены из 50 случайных п РНК библиотеку. Для того чтобы успешно получить вид связан с более высоким сродством, выбор жесткости тщательно контролируется путем изменения условий. Выбранных аптамеры могут специфически связываться и быстро внутренним в клетках, экспрессирующих ВИЧ-1 конверт белка. Кроме того, аптамеры только могут нейтрализовать ВИЧ-1 инфекционности. Основываясь на лучших аптамер-1, мы также создаем новый двойной тормозной функции анти-gp120 аптамер-миРНК химеры, в которых оба аптамер и миРНК части обладают мощным анти-ВИЧ. Кроме того, мы используем gp120 аптамер-миРНК химер для камерного типа конкретной доставки миРНК в ВИЧ-1 инфицированных клеток. Эта двойная функция химера показывает значительный потенциал для объединения различных нуклеиновых кислот терапевтических агентов (аптамер и миРНК) для подавления инфекции ВИЧ-1, что делает аптамер-миРНК химеры привлекательной терапевтической кандидатов для пациентов отсутствии высокоактивной антиретровирусной терапии (ВААРТ).
протокол
1. Подготовка РНК библиотеку
- Начиная библиотеки ДНК содержит 50 нуклеотидов случайных последовательностей и был синтезирован Комплексная ДНК технологий (Coralville, штат Айова). Одноцепочечной ДНК олиго библиотеку последовательность 5'-GGG AGG ACG ATG CGG - N 50 - CAG ACG ACT CGC CCG - 3 "(81 п). Случайные региона в окружении постоянной регионов, которые включают в себя промоутер Т7 для экстракорпорального транскрипции и теги 3 'для RT-PCR. 5 'и 3' постоянной последовательности 5 '- ТАА TAC GAC TCA CTA TAG GGA GGA CGA ТГК Г.Г. - 3 "(32 MER) и 5'-TCG GGC GAG TCG TCT G - 3" (16 MER), соответственно. Сделать маточного раствора с водой и хранят в аликвоты при -20 ° C.
- Amplify одноцепочечной ДНК олиго случайных библиотеки (0,4 мкм) методом ПЦР с использованием 3 мкМ каждый из 5'-и 3'-праймеров, наряду с 2 мМ MgCl 2 и 200 мкМ каждого дНТФ. Для того чтобы сохранить обилие оригинальных библиотеки ДНК, ПЦР предел до десяти циклов. После реакции ПЦР (10 реакций, 100 мкл на реакцию), восстанавливать усиливается дцДНК бассейн использованием очистки QIAquick Гель Kit (Qiagen).
- Преобразование в результате двухцепочечной ДНК к РНК с использованием библиотеки DuraScription Kit (Эпицентр, Мэдисон, Висконсин) в соответствии с инструкциями производителя. В реакционной смеси, транскрипции, заменить CTP и UTP с 2'-F-CTP и 2'-F-UTP производить рибонуклеазы устойчивы РНК. Как правило, готовят 20 мкл реакции, содержащей 1 мкг очищенной ДНК-матрицы, 2 мкл 10 х буфера, 2 мкл дАТФ, 2 мкл дГТФ, 2 мкл 2'-F-дЦТФ, 2 мкл 2'-F-dUTP, 2 мкл DTT и 2 мкл Т7 РНК-полимеразы при комнатной температуре, а затем инкубируют при 37 ° C в течение 6 ч. (50 мМ каждого дНТФ.)
- Впоследствии переварить реакции с ДНКазы I (1,5 мкл на 20-мкл реакции транскрипции Т7), чтобы удалить шаблон ДНК и очистить от 8% полиакриламидном / 7 M мочевины геля. Количественная очищенной РНК библиотеку, УФ-спектрофотометрии.
2. В пробирке поколение аптамеров
- До отбора, подготовки буфера выбора и рефолдинг буфера. Подготовка HEPES буфера, содержащего 100 мМ HEPES, рН 7,4. Используйте NaOH для регулировки рН и затем хранить при комнатной температуре. Подготовка буфера РНК рефолдинг (5xHBS), содержащего 50 мМ Hepes рН 7,4, 750 мМ NaCl, 5 мМ MgCl 2, 5 мМ CaCl 2, 13,5 мМ KCl. Подготовка с низким содержанием соли связывание РНК буфера (10 мМ HEPES рН 7,4, 50 мМ NaCl, 1 мМ CaCl 2, 1 мМ MgCl 2, 2,7 мМ KCl, 10 мМ DTT, 0,01% BSA и высокой соли связывание РНК буфера (10 мМ HEPES рН 7,4, 150 мМ NaCl, 1 мМ CaCl 2, 1 мМ MgCl 2, 2,7 мМ KCl, 10 мМ DTT, 0,01% BSA). Храните эти буферы при температуре -20 ° C.
- Выполните SELEX главным образом, как описано 1-4. Перед каждым отборочный тур, refold РНК бассейнов в 1xHBS буфера (10 мМ HEPES рН 7,4, 150 мМ NaCl, 1 мМ CaCl 2, 1 мМ MgCl 2, 2,7 мМ KCl), тепло до 95 ° С в течение 3 мин, а затем медленно охлаждать до 37 ° C. Продолжить инкубации при температуре 37 ° С в течение 10 мин.
- Как правило, для того, чтобы свести к минимуму неспецифического связывания с нитроцеллюлозные фильтры, предварительно адсорбируют сложил РНК пулы нитроцеллюлозы фильтр (HAWP фильтр, 0,45 мкм) в течение 30 мин, до инкубации с целевой ВИЧ-1 Бал gp120 протеина.
- Инкубируйте предварительно очищен РНК бассейн с белком-мишенью в странах с низким соль связывание РНК буфера в течение 30 мин для SELEX раунды 1 до 4. После четвертого раунда SELEX, использование высокого соль связывание РНК буфера. С SELEX прогресса, уменьшить количество белка gp120 и увеличение тРНК дрожжей конкурента с целью увеличения жесткости аптамер выбора.
- За первый цикл отбора, инкубировать предварительно очищен пула случайных РНК (40 мкг, 1,5 нмоль, 9x10 14 молекул) и ВИЧ-1 Бал gp120 белка (0,23 нмоль, соотношение РНК / белок 6.5 / 1) в 200 мкл низким соль связывание РНК буфер на вращающейся платформе, при комнатной температуре в течение 30 мин.
- Pass реакции через предварительно увлажненный нитроцеллюлозный фильтр и промыть 1 мл буфера для связывания.
- Элюции РНК связаны с фильтром 200 мкл буфера элюирования (7 M мочевины и 5 мМ ЭДТА) при 95 ° С в течение 5 мин, после чего фенол / хлороформ добыча и обогащение с микроконтроллер YM-30 колонки.
- Обратный записать восстановленный РНК бассейн использованием ThermoScript RT-PCR системы (Invitrogen) и усилить в течение 15 циклов ПЦР.
- Purify усиливается дцДНК бассейн использованием очистки QIAquick Гель Kit и записать, как описано выше в следующий раунд отбора.
3. SELEX контроля за ходом работы по фильтру анализа связывания
- Монитор SELEX прогресс аптамеры фильтром анализа связывания. Лечить бассейн РНК с CIP, чтобы удалить начало 5'-трифосфата, а затем метку с Т4 полинуклеотидкиназы и γ-32P-АТФ.
- Тепло 10 пмоль CIP лечение РНК библиотеку при 95 ° С в течение 5 мин, затем охладите на льду. Затем добавьте 2мкл PNK буфера, 1 мкл Т4 полинуклеотидкиназы, 1 мкл гамма-P 32-АТФ и воды до 20 мкл.
- Инкубировать при 37 ° С в течение 30 мин, затем добавить 20 ^ му; л воды и очистить реакцию G-50 колонки. Наконец, получить 40 мкл меченых РНК при конечной концентрации 250 нМ.
- До теста, refold меченой РНК бассейнов в 1xHBS буфера (10 мМ HEPES рН 7,4, 150 мМ NaCl, 1 мМ CaCl 2, 1 мМ MgCl 2, 2,7 мМ KCl), тепло до 95 ° С в течение 3 мин, а затем медленно охладить до 37 ° C. Продолжить инкубации при температуре 37 ° С в течение 10 мин.
- Провести 100 мкл обязательной реакции, как например здесь. Инкубируйте конечных меченой РНК бассейн (10 нм) с gp120 белка (100 нм) и 10-кратный молярный избыток неспецифическое конкурента тРНК (100 нм) в области высоких соли связывание РНК буфер на 30 мин.
- Отдельные 50 мкл обязательным реакции предварительно мокрой нитроцеллюлозы фильтр.
- Промыть фильтр с 2 мл буфера для связывания и считать радиоактивности остается на фильтре через многоцелевой сцинтилляционного счетчика (Beckman Coulter). Как входной контроль, подсчет оставшихся 50 мкл Реакцию связывания в то же время. Рассчитать процент РНК остается на фильтре на входе РНК, как сродство.
4. Клонирование, секвенирование и выравнивания
- После 11 раундов, если нет дополнительного обогащения наблюдается даже следующие дополнительные отборочные туры, то максимальное связывание РНК бассейн имеет потенциально была достигнута.
- Обратный транскрибировать обогащенного аптамер бассейн (12 м РНК бассейн) с использованием ThermoScript RT-PCR системы (Invitrogen), а затем усиливает результате кДНК с помощью ПЦР. Purify ПЦР продукта с использованием очистки QIAquick Гель Kit (Qiagen). Клон гель-очищенной ДНК продукта в ТА вектора клонирования ПЦР 2,1 (Invitrogen). В общей сложности, привить 170 индивидуальных клонов и дальнейшей их идентификации путем секвенирования ДНК, чтобы получить отдельных последовательностей.
- Классификация индивидуальных клонов на шесть различных групп, основанных на выравнивание индивидуальных последовательностей аптамеров. Выберите одного представителя последовательность из каждой группы (А-1, А-5, А-9, А-12, А-28 и Б-68) для дальнейшей характеристики из-за их относительной численности в пределах своей группы.
5. Генерация аптамер химера и РНК в транскрипции по пробирке
- Непосредственно генерировать двухцепочечной ДНК-матрицы с помощью ПЦР с использованием 2 мкМ каждый из 5'-и 3'-праймеров, наряду с 2 мМ MgCl 2 и 200 мкМ каждого дНТФ и восстановления в результате ПЦР-продуктов с использованием очистки QIAquick Гель Kit.
- Расшифруйте химера прядь толку от его ПЦР ДНК порожденных шаблонов с помощью DuraScription Kit (Эпицентр, Мэдисон, Висконсин). В реакционной смеси, транскрипции, заменить канонические CTP и UTP с 2'-F-CTP и 2'-F-UTP производить РНК, которая устойчива к РНКазы деградации.
- Как правило, инкубировать 20 мкл реакции, содержащей 1 мкг ДНК шаблон, 2 мл 10xbuffer, 2 мкл дАТФ, 2 мкл дГТФ, 2 мкл 2'-F-дЦТФ, 2 мкл 2'-F-dUTP, 2 мкл DTT и 2 мкл Т7 РНК-полимеразы при температуре 37 ° C в течение 6 ч, а затем очистить его с Био-Spin 30 Столбцы (Bio-Rad) после извлечения фенола и этанола осадков.
- Для того чтобы избежать ответа интерферона, дальнейшее лечение транскрипции РНК CIP, чтобы удалить начало 5'-трифосфата. Инкубируйте всего 60 мкл реакции, содержащий 3 мг стенограммы, 6 мкл буфера 3 и 0,25 мкл CIP при температуре 37 ° С в течение 60 мин. После фенол / хлороформ взыскание и этанола осадков, ресуспендируют РНК гранул в воду.
- Для подготовки химеры, объединить химеры укрывательство только смысловой нити РНК с соответствующей РНК антисмысловых в рефолдинг буфера, тепла до 95 ° С в течение 3 мин и затем охлаждают до 37 ° С медленно. Продолжить инкубации при температуре 37 ° С в течение 10 мин. Выполните рефолдинг шаг буфера увенчением 1xHBS. Например: смешайте 10 мкл 10 мкМ химера нить смысла, 10 мкл 10 мкМ прядь антисмысловых и 5 мкл буфера рефолдинг (5xHBS) в 25 мкл системы.
6. Определение константы диссоциации анализов гель сдвиг
- P конце 32 этикетке представитель аптамеров для каждой группы и химеры смысловой нити, а затем refold РНК в буфере 1xHBS как описано выше.
- Подготовка 25 мл 5% геля путем смешивания 2,5 мл буфера 10xTBE, с 3,125 мл 40% акриламид / бис решение, 19,375 мл воды, 150 мкл 10% персульфат аммония (APS), решение, и 30 мкл TEMED. Гель должен полимеризоваться в 30 мин. Осторожно выньте гребенку и использовать 30-мл шприц оснащен иглой мыть проточной скважин буфер (1xTBE).
- Полное собрание геля устройства и подключения к источнику питания. Гель может быть предварительно запустить в течение часа при 180 В при температуре 4 ° C.
- Серийно разбавить ВИЧ-1Bal gp120 белок с обязательным буфер для желаемой концентрации. Окончательный тeaction концентрации gp120 0, 1, 5, 10, 20, 40, 80, 160, 320, 640 нм. Инкубируйте постоянное количество 5'-P 32-конечных меченой РНК (10 нм) с увеличением концентрации белка gp120 в связывающем буфере (всего 20 мкл реакции) на вращающейся платформе, при комнатной температуре в течение 30 мин.
- После инкубации смесь 20 мкл Реакцию связывания с 5 мкл родного буфера загрузки и загрузки в 5% неденатурирующих полиакриламидном геле. Подготовка родного буфера загрузки (4x), содержащего 10 мМ Трис-HCl, рН 7,5, 1 мМ ЭДТА, 0,1% бромфеноловый синий, 0,1% Ксилол Cyanol FF, 0,1% Оранжевый G, 40% глицерина. Хранить в аликвоты при -20 ° C.
- После электрофореза (180 В при 4 ° С в течение 2 часов, пока вторичный красителя работает на середину гель), подвергают гель для изображения на экране фосфор и количественной оценки радиоактивности использованием Тайфун сканера.
- Рассчитайте константы диссоциации использованием нелинейной кривой регрессии с Призма Pad Graph.
7. Cell-поверхность обязательные исследования методом проточной цитометрии
- Создание флуоресцентные аптамер и химер использовании маркировки Silencer миРНК Kit (Амбион). Добавьте следующие реагенты в порядке: 22,5 мкл нуклеазы без воды, 5 мл 10 х Маркировка буфера, 15 мкл РНК (5 мкг), 7,5 мкл Dye маркировка. Инкубируйте всего 50 мкл реакции маркировки при температуре 37 ° С в течение 1 часа.
- После инкубации, добавить 5,0 мкл (0,1 т.) 5 М NaCl и 125 мкл (2,5 т.) холодного 100% этанола, и тщательно перемешать. Инкубировать при температуре -20 ° С в течение 60 мин. Центрифуга на максимальной скорости при 4 ° С в течение 20 мин. Удалить супернатант и мыть гранул с 175 мкл 70% этанола. Воздух сухой осадок в темной, а затем приостановить меченой РНК в 15 мкл нуклеазы без воды.
- Измерить абсорбцию во меченой РНК при 260 нм и при абсорбции максимум для флуоресцентным красителем. Рассчитать база: соотношение красителя и РНК концентрации по калькулятор по http://www.ambion.com/techlib/append/base_dye.html .
- Смешайте Cy3 меченных химеры нить смысла и антисмысловых нитей и refold в рефолдинг буфера, как описано выше.
- Получить СНО-WT выражения gp160 и СНО-EE клетки контроль через исследований СПИДа и справочные программы реагента 5, 6. Рост клеток в GMEM-S среде (глютамин-дефицитных минимальным необходимой среде с 400 мкМ метионин сульфоксимина (MSX)) (Gibco, Invitrogen). Культуры клеток в увлажненном 5% CO 2 инкубаторе при температуре 37 ° C.
- Вымойте СНО-WT gp160 или СНО-EE клетки управления с нагретого промывочный буфер, trypsinize и оторваться от плиты. После мытья клетки в два раза с 500 мкл буфера для связывания, resupend ячейки гранул в связывающем буфере и инкубировать при температуре 37 ° С в течение 30 мин. Гранул клеток, а затем ресуспендируют их в 50 мкл предварительно нагретого обязательным буфера, содержащего 400 нМ Cy3 меченных экспериментальных РНК.
- После инкубации при температуре 37 ° C в течение 40 мин, промыть клетки три раза с 500 мкл буфера для связывания предварительно нагретого, и, наконец, ресуспендируют в 350 мкл буфера для связывания предварительно нагретого до 37 ° С и сразу же проанализировать с помощью проточной цитометрии.
8. Интернализация и внутриклеточных исследований локализации Live-клетка конфокальной микроскопии
- Прежде, чем каждый день анализа, растут СНО-WT gp160 и СНО-EE клетки контроль в 35-мм пластины с посевом на 0.3x10 6 в 2 средних мл GMEM-S, чтобы около 70% слияния в 24 ч.
- В день эксперимента, мыть клетки с 1 мл предварительно нагретого PBS. И инкубировать с 1 мл подогретого полной среде роста в течение 30 мин при 37 ° C.
- Подготовка Cy3 меченных аптамер-миРНК химеры, как описано выше. Инкубируйте refold аптамер-стик с миРНК-палки содержащих 5'-Cy3 меченных смысловой нити для формирования аптамер-палки миРНК сопряженных, как описано выше.
- До анализа, подготовить 0,15 мг / мл раствора Hoechst 33342 (ядерные краситель для живых клеток, молекулярные зонды, Invitrogen, штат Калифорния) в воде и хранить в аликвот при 4 ° C.
- Пятно клетки путем обработки 0,15 мг / мл Hoechst 33342 в течение 15 мин при 37 ° C. Сразу же, промыть красителя с 1,0 мл свежей среды, в два раза и заменить 2 мл предварительно нагретого свежую среду.
- Добавить Cy3 меченных аптамер-миРНК химера на 100 нМ конечная концентрация в средствах массовой информации и инкубировать живая клетка конфокальной микроскопии в 5% CO2 микроскопии инкубаторе при температуре 37 ° C.
- Сбор изображения каждые 15 минут, используя Zeiss LSM 510 Мета Перевернутый два фотона конфокальной микроскопии системы под водой, погружение на 40-кратном увеличении.
9. В пробирке ВИЧ-1 вызов и p24 анализа антигена
- Покупка УК РФ-CEM клетки АТСС. Растут в RPMI-1640 (Cellgro, Mediatech Inc) с добавлением 10% эмбриональной телячьей сыворотки (FBS, Hyclone), L-глютамин и 1xPen-Strep (Gibco, Invitrogen). Культуры клеток в увлажненном 5% CO 2 инкубаторе при температуре 37 ° C.
- Получить периферической крови mononucleар образцов от здоровых доноров из города Надежда Национальный медицинский центр (клиника персонала).
- Изолировать МНПК из цельной крови путем центрифугирования через Ficoll-Hypaque решение (Histopaque-1077, Sigma). Разрушающим CD8 клеток (T-cytotoxic/suppressor клеток) от МНПК с помощью Dynabeads CD8 (Invitrogen, Калифорния).
- Рост клеток в Т-клеточной активной среды (BioE, Сент-Пол, Миннесота). Культуры клеток в увлажненном 5% CO 2 инкубаторе при температуре 37 ° C.
- Получить ВИЧ-1, IIIB и NL4-3 вируса и ВИЧ-1 Бал вирус из исследований СПИДа и Программа Ссылка реагентов. После распространения вируса, магазин в аликвоты при -80 ° C.
- Infect УК РФ-CEM клетки или человека МНПК вирусами ВИЧ (IIIB, NL4-3 или Бал) (МВД 0,001 или 0,005). Через 24 часа после заражения, аккуратно мыть клетки с PBS три раза для удаления свободного вируса. Продолжайте культуре инфицированных клеток в 5% CO 2 микроскопии инкубаторе при температуре 37 ° С в течение 4 дней.
- До лечения РНК, мягко мыть инфицированных клеток с PBS три раза для удаления свободного вируса. Инкубируйте 2x10 4 инфицированных клеток и 3x10 4 незараженных клеток с сложил экспериментальных РНК при 400 нм конечной концентрации в 96-луночных планшетах при 37 ° C (100 мкл на лунку, анализ триплекса).
- Сбор культуры супернатантов (10 мкл на лунку) в разное время (3 г, 5 г, 7 г и 9 г) и хранят при температуре -20 ° С до p24 анализа.
- Выполнить анализ антигена р24 использования ВИЧ-1 р24 антиген ИФА.
10. Обнаружение миРНК функции количественного ПЦР-анализа
- Infect УК РФ-CEM клетки или человека МНПК с ВИЧ, вирусы (IIIB, NL4-3 или Бал) и лечить с экспериментальными РНК (400 нм), как описано выше.
- После 7 дней лечения, пеллет клетки и изолировать общую РНК с STAT-60. Лечить общую РНК с ДНКазы я для удаления геномной ДНК и кДНК производят с помощью 2 мкг тотальной РНК.
- Смешайте следующие реагенты: 8 мл нуклеазы без воды, 1,5 мл 10 х ДНКазы буфера, 4 мкл РНК (2 мкг), 0,5 мкл ингибитора RNain и 1,0 мкл РНКазы без ДНКазы I. Инкубируйте всего 15 мкл реакции 37 ° С в течение 1 час и тепла при температуре 80 ° С в течение 10 мин для инактивации ДНКазы I. Сразу же, озноб реакции на льду.
- Добавьте 2 мкл случайных праймеров (50 нг / мкл) и 1 мкл дНТФ (10 мМ) в реакционной смеси выше, а затем тепло при температуре 65 ° С в течение 5 мин. Сразу же, озноб реакции на льду.
- Добавить следующие реагенты: 5 мл 5xFirst прядь буфера, 2,5 мл 0,1 М ДТТ, 0,5 мл RNain ингибитора и 1,0 мл MMLV-RT. Инкубируйте всего 27 мл реакции при 25 ° С в течение 10 мин, а при 37 ° С в течение 1 часа. После реакции, тепло смеси при температуре 70 ° С в течение 15 мин для инактивации обратной транскриптазы и затем охладите на льду. КДНК готов к QRT-PCR анализа.
- Анализ экспрессии генов-мишеней количественными RT-PCR с использованием 2 х IQ SyberGreen Mastermix и конкретные наборы грунтовки в конечной концентрации 400 нМ (триплекс анализ). Использование выражения GAPDH как внутреннего контроля для нормализации данных КПЦР.
11. Представитель результаты:
1. Новые РНК аптамеры против ВИЧ-1 БЛ gp120 изолированы и охарактеризованы.
Как описывается в экспериментальной части, начальный библиотеки олигонуклеотидных ДНК, содержащей 50 п случайных регионе окружении фиксированной регионах грунт на 5 'и 3' концов, усиливается и транскрибируется в РНК, бассейн. Этот начальный библиотека состоит из до 10 15 разнообразных последовательности (1 нмоль), которые складываются в широкий спектр различных 3-D структуры. Высокая сложность и разнообразие начальной библиотека может гарантировать наличие активных структур с хорошим сродством к цели.
Применять в пробирке процедуры SELEX (рис. 1), чтобы выбрать 2'-фторпиримидина изменение РНК-аптамеров, которые избирательно связываются R5 штамма ВИЧ-1 БЛ gp120 конверт белка 7. Как показано на рисунке 1, нитроцеллюлозы основе выбора стратегии осуществляется выделить конкретную цель РНК-связывающий от необязательных молекул РНК. Поскольку белок прилипает к нитроцеллюлозы, только РНК / белок комплексы или агрегаты могут быть сохранены на мембране и свободной РНК вымываются. Под денатурирующих условиях, связаны РНК восстанавливаются и обратной транскрипции с кДНК и затем усиливается в двухцепочечной ДНК, а затем в пробирке транскрипции, чтобы создать новый пул РНК для следующего цикла отбора. Выбор строгости увеличивается за счет уменьшения количества белка-мишени и увеличения количества конкурентов тРНК. Количество РНК бассейн, белков и тРНК конкурента, используемых в каждом отборочном туре показано в таблице 1.
Контролировать ход отбора после каждого цикла SELEX фильтром анализа связывания. Оценка сродство как процент от РНК сохраняется на филтер в общем пуле РНК. Начиная РНК бассейн (1-РНК), показывает только 0,1% от входного РНК удерживается на мембране. Тем не менее, после девяти отборочных туров девятого РНК библиотеки (9-РНК) имеет 9,72% входной РНК связаны. Хотя дополнительные отборочные туры были проведены, нет дальнейшего обогащения наблюдается, что свидетельствует о максимальной связывание РНК бассейн был достигнут (рис. 2А). Подобные с фильтром связывания, анализа гель сдвиг также является одним из самых популярных стратегий для определения константы диссоциации. Эта процедура проста и удобна. Как показано на рис 2B, анализы гель сдвиг дальнейшего подтверждения обязательной деятельности бассейнов РНК. Эти результаты показывают, что некоторые лигандов с высокой специфичностью связывания для белка-мишени последовательно обогащенный в этих бассейнах РНК ..
Клонов и последовательность высоко обогащенного аптамер бассейна (12-РНК). По выравнивания отдельных клонированных последовательностей аптамеров, шесть различных групп классифицируются, как показано в таблице 2. Около 40% клонов (группа I и II аптамеры) содержат консервативной последовательности: (/ G) TTGAGGGACC (/ G). Мы выбираем одного представителя последовательность из каждой группы (например, A-1, А-5, А-9, А-12, А-28 и Б-68) для дальнейшей характеристики из-за их относительной численности в пределах своей группы. Через родной анализа подвижности сдвига геля, константы диссоциации (К г) этих представительств аптамеры рассчитываются (рис. 3А). Например, A-1, лучший из аптамеры, имеет очевидный Kd значения 52 нм (рис. 3В). Как показано на рисунке 3A, этих отобранных аптамеров может избирательно связываются с мишенью ВИЧ-1 Бал gp120, но не CM ВИЧ gp120 протеина.
2. Анти-gp120 аптамер специфически связывается и усвоены клеток, экспрессирующих ВИЧ gp160 и ингибировать ВИЧ-1 инфекции в клеточной культуре.
СНО-gp160 клетки стабильно выражения гликопротеина gp160 ВИЧ конверт используются для тестирования для связывания и интернализации выбранного анти-gp120 аптамеров. Эти клетки не обрабатывают gp160 в gp120 и gp41 поскольку они не кляп закодированные протеаз, необходимых для обработки конверта. В качестве контроля мы используем родительских СНО-EE клеточной линии, которая не выразить gp160. Проточной цитометрии анализа (рис. 4А) показывают, что Cy3 меченных аптамеры специфически связываться с gp160 CHO-клеток, но не управления СНО-EE клеток. Кроме того, в режиме реального времени жить-клеток Z-оси конфокальной микроскопии показывают, что Cy3 меченных аптамер избирательно интернализированы в рамках СНО-gp160 клетки (рис. 4B и 4C) после 2 часов инкубации, но не СНО-EE контрольными клетками ( Рис 4D). Рис 4C также показывает, что аптамер агрегированных в цитоплазме, что свидетельствует о gp120 аптамеров, может быть, проникать в клетки через рецептор-опосредованного эндоцитоза.
В ВИЧ-1 Challenge анализа, инфицированных ВИЧ-1-РВМС клетки обрабатывают аптамеров. В разные дни после лечения с аптамеры аликвоты СМИ оценивали на вирусный антиген p24 уровней (рис. 4E). Результаты показывают, что анти-gp120 аптамеры (A-1) ингибирует ВИЧ-1 р24 производства нано-Молярная концентрация.
3. Анти-gp120 аптамер-миРНК химера разработан и оценивать его эффективность в качестве камерного типа конкретной системы доставки миРНК
Как показано на рисунке 5А, аптамер и смысловой нити сегменте siRNAs содержащихся нуклеазы устойчивые 2'-фтор UTP и 2'-фтор CTP и синтезируются из соответствующих шаблонов двухцепочечной ДНК путем в транскрипции бактериофага пробирке. В целях повышения гибкости молекулы, две нуклеотидные компоновщику (UU) вставляется между аптамер и часть Dicer подложки. Для подготовки миРНК содержащие химер, в пробирке транскрипции химерных аптамер смысла полимеров нити отжигаются с эквимолярной концентрации РНК немодифицированных антисмысловых нитей. Эти данные из геля сдвиг анализа (рис. 5B) и проточной цитометрии (рис. 5С) показывают, что химеры поддерживать примерно такой же обязательной, как сродство аптамеров в одиночку. Время курса изображений в реальном масштабе времени конфокальной микроскопии (рис. 5D) показывают, что Cy3 меченных химера Ch-1 может быть успешно интернализованной в цитоплазму клетки. Как и ожидалось, не поглощение химера наблюдается при ЧО-EE контрольными клетками (рис. 5E).
Кроме того, противовирусные потенциал оценивается РНК ВИЧ-1 Challenge анализа. Результаты анализов ВИЧ p24 антигена (рис. 6А и 6Б) показывают, что оба аптамер химера и подавляют p24 производства, но сильные торможение наблюдается при химера Ch-1 лечение.
Чтобы подтвердить, что миРНК компонент функционирует наряду с АПТАМер, следующие интернализации Ch-1 химера в инфицированных клетках, мы также оценить относительный уровень ингибирования зуб / об экспрессии генов путем количественного ПЦР-анализов выражения. Мы считаем, что лечение инфицированных клеток с химерами способен вызвать молчание зуб / об гена, в то время аптамер само по себе не влияет на зуб / об экспрессии генов (рис. 6С и 6D). Эти результаты обеспечивают дополнительную поддержку, что аптамер доставлены миРНК триггеры RNAi.
| Количество белка, РНК и тРНК бассейна используются для выбора | |||||
| SELEX раундов | Отношение Target / РНК | Gp120 белок | РНК бассейн | Конкурент тРНК | Выбор буфера |
| 1 | 1/6.5 | 229,8 пмоль | 1,5 нмоль (40,1 мкг) | 0 | Низкий буфер SELEX соли |
| 2 | 1/6.5 | 114,9 пмоль | 0,75 нмоль (20,1 мкг) | 0,25 нмоль (6,6 мкг) | |
| 3 | 1 / 8 | 76,6 пмоль | 0,625 нмоль (16,7 мкг) | 0,25 нмоль (6,6 мкг) | |
| 4 | 1 / 8 | 76,6 пмоль | 0,625 нмоль (16,7 мкг) | 0,25 нмоль (6,6 мкг) | |
| 5 | 1 / 8 | 38,3 пмоль | 0,306 нмоль (8,18 мкг) | 0,5 нмоль (13,2 мкг) | Высокая буфера SELEX соли |
| 6 | 1 / 8 | 38,3 пмоль | 0,306 нмоль (8,18 мкг) | 0,5 нмоль (13,2 мкг) | |
| 7 | 1 / 10 | 26,8 пмоль | 0,268 нмоль (7,16 мкг) | 0,5 нмоль (13,2 мкг) | |
| 8 | 1 / 10 | 26,8 пмоль | пмоль 0,268 нмоль (7,16 мкг) | 1 нмоль (26,4 мкг) | |
| 9 | 1 / 10 | 15,3 пмоль | 0,153 нмоль (4,09 мкг) | 1 нмоль (26,4 мкг) | |
| 10 | 1 / 10 | 15,3 пмоль | 0,153 нмоль (4,09 мкг) | 1,5 нмоль (39,6 мкг) | |
| 11 | 1/12.5 | 7,66 пмоль | 0,096 нмоль (2,56 мкг) | 2 нмоль (52,8 мкг) | |
| 12 | 1/12.5 | 7,66 пмоль | 0,096 нмоль (2,56 мкг) | 2 нмоль (52,8 мкг) | |
Таблица 1. Условиям отбора. Количество белка, РНК и тРНК бассейн для каждого отбора и выбора буфера указаны.
| Группа | РНК | Случайные последовательности | Частота (140 клоны) |
| Группа I | A-1 | AATTGAGGGACCA CGCGCTGCTTGTTGTGATAAGCAG TTTGT CG GATGG | 33 (23,6%) |
| В-7 | AATTGAGGGACCA ACGCGAGGATGTGGATAGTGTGTA TTTGC GT GATGG | 3 | |
| А-32 | AATTGAGGGACCG TTGGTAAAAGCCGGA AATTG AGCT TTTAC GGC GATGG | 5 | |
| B-55 | AATTGAGGTACCGCG TTATTAGGAACA AATTG GAATTCTAAACGC GATGG | 2 | |
| А-24 | AATAGAGGGACC CAGATATAGGCTACACGGATGATGGTGTATCTG GATGG | 1 | |
| В-19 | AATAGAGGAACCG TTTCAGAAGACTACAGGTTAGTCCAATGAAGC GACGG | 1 | |
| B-31 | AATAGAGGGACCG TGGACAATAATTTATGGTCA TTTATTGGCAC GATGG | 1 | |
| Группа II | А-12 | AGTAGAGGAACCA AGCAATGGATGAATGCAAAAGTGTAAATGCTT GATGG | 10 (7,1%) |
| Группа III | -9 | TGAGTTTGGGTAAATTTCCGGTTTCGGTTTACTCACGAAAGATCGGTCGG | 15 (10,7%) |
| Группа IV | А-28 | TAAAGGAGGGAAGGATGAGACCGCACGAAAAATATCAGCATACG TTTGTG | 10 (7,1%) |
| Группа V | А-5 | GAAACTAGTTTGAATAATGGTGTAGAGGAGGGTCAATAGTTTCG TTGGTG | 9 (6,4%) |
| VI группы | B-68 | ACATAGTAATGACACGGAGGATGGAGAAAAAACAGCCATCTCTTGACGGT | 2 |
| Другие | Сирота последовательность | 48 |
Таблица 2. Выравнивания и идентификации аптамеров РНК. После 12-го тура отбора, выбранного пула РНК был клонирован и секвенирован. После согласования всех 140 клонов, шесть групп по анти-gp120 аптамеры были идентифицированы. Только случайные последовательности аптамер центральных районов (5'-3 ') указаны. Изоляты, происходящие с кратными частотами указаны.
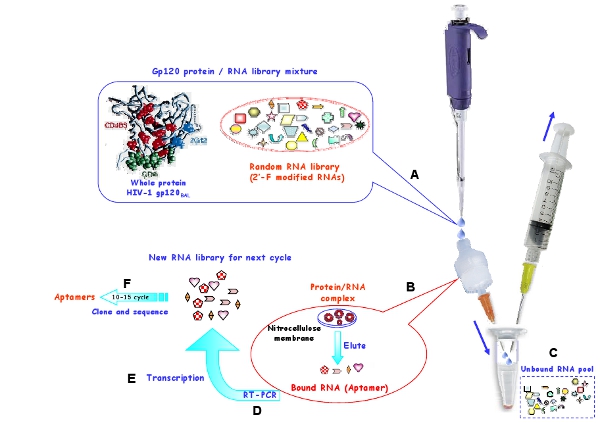
Рисунок 1: Схематическое изображение в процедуре отбора пробирке использованием нитроцеллюлозные мембраны, для получения РНК-аптамеров для ВИЧ-1 Бал gp120 протеина. (А), начиная РНК бассейн и целевого белка, инкубировали в форме комплекса. (B), связанных молекул РНК были сохранены на мембране и вымывают из мембраны в денатурирующих условиях. (C) несвязанного РНК были смыты. (D) выбран РНК была обратной транскрипции и усиливаются с помощью ПЦР. (Е) соответствующих ДНК транскрибируется в дальнейшем новый пул РНК для следующего цикла отбора. (F) Через 10-15 отборочные туры, выбранные аптамеры были клонированы и последовательности.
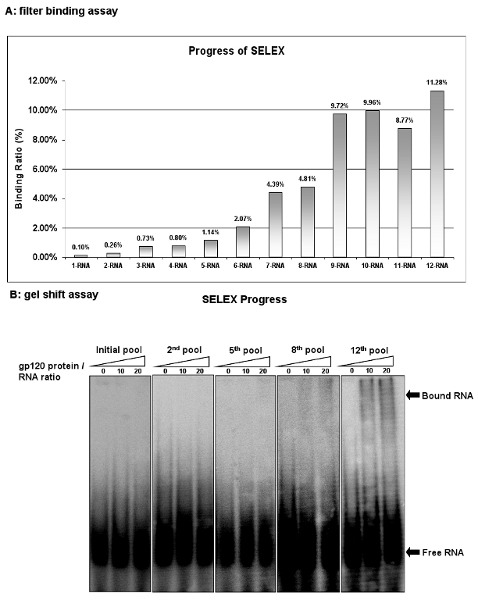
Рисунок 2: прогресс ВИЧ-1 gp120 отбор аптамеров. (А), связывающей активности бассейн РНК в каждом цикле была проанализирована фильтр связывания с конкурентом тРНК. Связывание мероприятия рассчитывается как процент входного РНК остается на фильтре в белок / РНК комплекса. (B), связывающей активности бассейн РНК в каждом цикле была проанализирована гель методом сдвига. 12-й РНК бассейн показали высокий связывающей активности.
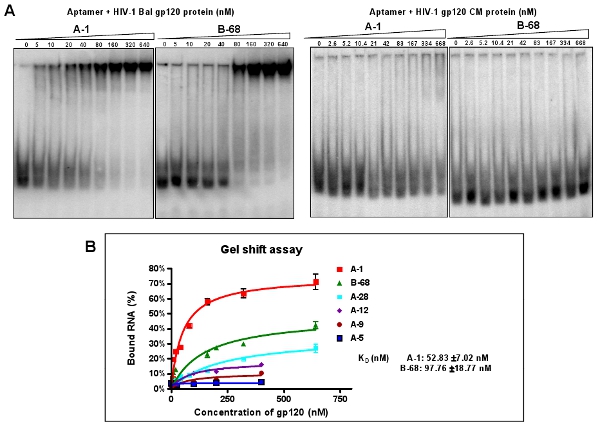
Рисунок 3: Связывание анализа деятельности отдельных выбранных аптамеры против ВИЧ-1 Бал gp120. (А) 5'-конце P 32 помечены отдельных аптамеры инкубировали с увеличением количества целевых gp120 белок или неспецифический CM белка. Обязательные смесей реакции анализировали с помощью сдвига анализа подвижности геля. Аптамеров-1 и В-68 показал лучшее сродство с белка-мишени, но не CM белка. Данные представляют собой среднее из четырех повторяет. (B) Связывание кривой от методом сдвига геля.
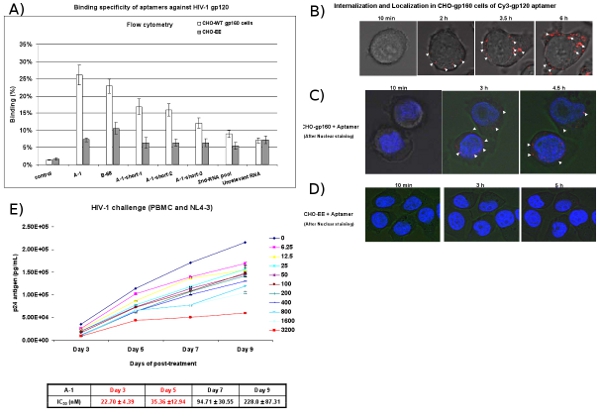
Рисунок 4: Сотовый типа специфического связывания и поглощения исследования аптамеров. () Сотовые поверхности связывание Cy3 меченных РНК оценивали с помощью проточной цитометрии. Cy3 меченных РНК были протестированы на связывание gp160 СНО-СНО клетки и клетки-EE контроля. Выбранных аптамеры показал камерного типа специфического связывания близости. 2-й РНК бассейн и не имеет значения РНК были использованы в качестве отрицательного контроля. Данные представляют собой среднее из трех повторяет. (B) Интернализация анализа. СНО-gp160 Клетки выращивались в 35 мм пластин и инкубировали с 100 нМ концентрации Cy3 меченных-1 в культуре средств массовой информации в режиме реального времени жить-клеток-конфокальной микроскопии анализа. Изображения были собраны в 15 мин. интервалы использованием 40-кратном увеличении. (C, D) Локализация анализа. СНО-gp160 клеток и СНО-EE контрольными клетками выращивали в 35-мм пластин. До инкубации с 100 нМ Cy3 меченных-1, клетки окрашивали Hoechst 33342 (ядерные краситель для живых клеток), а затем анализировали с помощью реального времени конфокальной микроскопии. (Э) выбранного анти-gp120 аптамеры подавляют репликации ВИЧ-1 в человеческой МНПК ранее инфицированных ВИЧ-1 NL4-3 вируса. Различные концентрации и времени пуанты были представлены. IC50 значение было в списке. Данные представляет среднем трех экземплярах измерения p24.
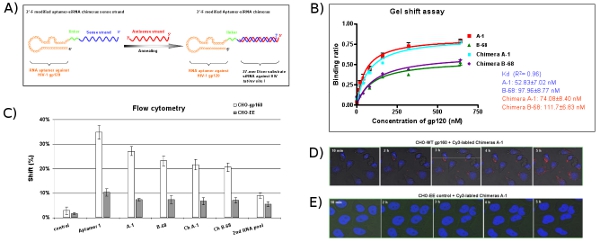
Рисунок 5: проектирование и оценка аптамер-миРНК химера системы доставки. (А) Схема аптамер-миРНК химерные РНК: область анти-gp120 аптамер отвечает за связывание gp120 и миРНК ориентирована на общую экзона ВИЧ-1 зуб / об. 2'-фтор изменение аптамер-миРНК смысле одну прядь была совместно переписана, с последующим отжигом в комплементарной цепи антисмысловых миРНК для завершения химерные молекулы. Компоновщик (UU) между аптамер и миРНК указывается в зеленый цвет. (B) аптамер-миРНК химерные РНК, которые имеют сопоставимые Kd ценностей, а также родительской аптамеры специфически связываются с ВИЧ gp120 Бал белка. Данные представляют собой среднее из трех повторяет. (C) камерного типа специфического связывания исследования аптамеров. Cy3 меченных РНК были протестированы на связывание gp160 СНО-СНО клетки и клетки-EE контроля. Сотовые сюрЛицо привязки Cy3 меченных РНК оценивали с помощью проточной цитометрии. Выбранных аптамеры показал камерного типа специфического связывания близости. 2-й РНК бассейн и не имеет значения РНК были использованы в качестве отрицательного контроля. Данные представляют собой среднее арифметическое двух повторяет. (D, E) Интернализация и внутриклеточной локализации анализы. СНО-gp160 клетки выращивали в 35-мм пластин и окрашивали Hoechst 33342 (ядерные краситель для живых клеток). Затем клетки инкубировали в культуральной среде с 100 нМ концентрации Cy3 меченных химера в режиме реального времени жить-клеточной конфокальной микроскопии анализ, как описано выше.
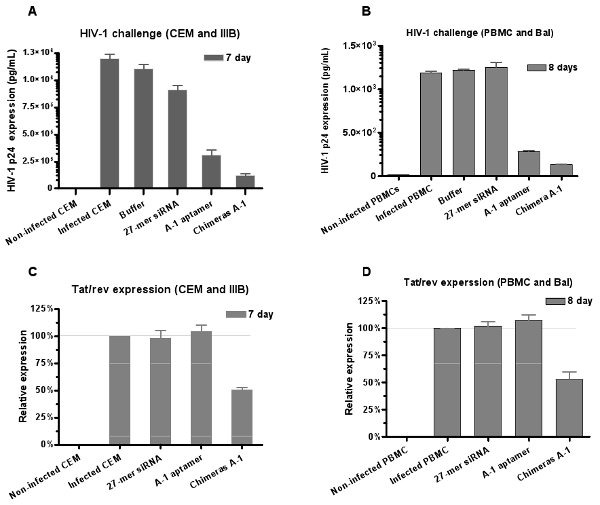
Рисунок 6: Двойное ингибирование ВИЧ-1 инфекции посредничестве аптамер-миРНК химеры. Оба анти-gp120 аптамер и аптамер-миРНК химеры нейтрализованы инфекции ВИЧ-1 в () клеток CEM (IIIB деформации) и (Б) человека МНПК (БАЛ деформации) культуры, соответственно. Данные представляют собой среднее из трех экземплярах измерения p24. Химер показали сильное торможение, чем аптамер только о том, что (С, D) миРНК выступил аптамеры вниз регулируется зуб / об экспрессии генов в МНПК. Данные представляют собой среднее из трех повторяет.
Обсуждение
Аптамеры в пробирке развивались нуклеиновых кислот, которые принимают конкретные и устойчивые трехмерные формы, тем самым обеспечивая высокую конкретные, жесткие привязки к целевой молекулы 8. Низкий наномолярных сродство и изысканные специфику аптамеры к цели сделать их ?...
Раскрытие информации
Благодарности
Мы благодарим Бритта Hoehn, Гуйхуа ВС, Харрис Сойфер и Лиза Шерер за полезные обсуждения. Эта работа была поддержана грантами от Национального института здоровья и AI29329 HL07470 присуждена JJR следующие реагенты были получены через NIH исследований СПИДа и Программа Ссылка реагентов, Отдел СПИДа, NIAID, NIH: СНО-EE и СНО-gp160 ячейки линии; pNL4-3 Люк вектор; ВИЧ-1 БЛ gp120 из DAIDS, NIAID.
Материалы
| Name | Company | Catalog Number | Comments |
| Название реагента | Компания | Номер в каталоге | Комментарии (необязательно) |
|---|---|---|---|
| MF-Millipore мембранный фильтр | Millipore | HAWP01300 | Размер пор 0,45 мкм |
| Swinnex держатель фильтра | Millipore | SX0001300 | Диаметром 13 мм |
| QIAquick Гель Добыча Kit | QIAGEN | 28706 | Очистки ДНК |
| Микроконтроллер YM-30 колонке | Millipore | 42410 | РНК концентрации |
| Био-спин 30 колонки | Bio-Rad | 732-6250 | РНК очистки |
| Taq ПЦР ДНК-полимеразы | Sigma-Aldrich | D1806 | |
| ThermoScript RT-PCR системой | Invitrogen | 11146-024 | |
| DuraScribe T7 транскрипции Kit | Эпицентр | DS010925 | |
| дНТФ для ПЦР | Roche | 1 581 295 | |
| Рибонуклеиновой кислоты, трансфер из E.coli | Sigma-Aldrich | R1753 | тРНК конкурента |
| ВИЧ-1-Ba-L gp120 белок | Исследования СПИДа и Программа Ссылка реагент | 4961 | Белка-мишени |
| Глушитель миРНК маркировке комплекта - Cy3 | Амбион | 1632 | |
| Кислота фенол / хлороформ 5 / 1 раствора (рН 4,5) | Амбион | AM9720 | |
| Хлороформ / Изопропанол 24 / 1 решение | Сигма | C0549 | |
| Теленок кишечной фосфатазы (CIP) | Новая Англия Биолаб | M0290L | |
| Т4 полинуклеотидкиназы | Новая Англия Биолаб | M0201L | |
| Гликоген | Roche | 10 901 393 001 | РНК осадков |
| Гамма-P 32-ATP | Депутат биомедицинских | 013502002 | Radiactivity |
| 40% AccuGel 19:01 | Национальный диагностики | EC-850 | |
| 10xTBE | Национальный диагностики | EC-860 | |
| N, N, N, N-тетраметилэтилендиамина (TMEMD) | Sigma-Aldrich | T9281 | |
| Аммоний персульфат (АПС) | Sigma-Aldrich | A3678 | |
| L-methioine сульфоксимина | Sigma-Aldrich | M5379-250 мг | |
| RPMI СМИ 1640 | Invitrogen | 11835-030 | |
| Раствором бикарбоната натрия, 7,5% м / о | Invitrogen | 25080-094 | |
| Минимально необходимые Средняя (MEM) (10 ) | Invitrogen | 11430-030 | |
| MEM несущественные аминокислоты (100 ) | Invitrogen | 11140-050 | |
| Т. А. Клонирование комплект с ПЦР 2,1 | Invitrogen | K2040-01 |
Ссылки
- Tuerk, C., Gold, L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Robertson, D. L., Joyce, G. F. Selection in vitro of an RNA enzyme that specifically cleaves single-stranded DNA. Nature. 344, 467-468 (1990).
- Fitzwater, T., Polisky, B. A SELEX primer. Methods Enzymol. 267, 275-301 (1996).
- Weiss, C. D., White, J. M. Characterization of stable Chinese hamster ovary cells expressing wild-type, secreted, and glycosylphosphatidylinositol-anchored human immunodeficiency virus type 1 envelope glycoprotein. J Virol. 67, 7060-706 (1993).
- Vodicka, M. A. Indicator cell lines for detection of primary strains of human and simian immunodeficiency viruses. Virology. 233, 193-198> (1997).
- Zhou, J. Selection, characterization and application of new RNA HIV gp 120 aptamers for facile delivery of Dicer substrate siRNAs into HIV infected cells. Nucleic Acids Res. , (2009).
- Mayer, G. The chemical biology of aptamers. Angew Chem Int Ed Engl. 48, 2672-2689 (2009).
- Famulok, M., Hartig, J. S., Mayer, G. Functional aptamers and aptazymes in biotechnology, diagnostics, and therapy. Chem Rev. 107, 3715-3743 (2007).
- Chu, T. C., Twu, K. Y., Ellington, A. D., Levy, M. Aptamer mediated siRNA delivery. Nucleic Acids Res. 34, e73-e73 (2006).
- McNamara, J. O., 2nd, . Cell type-specific delivery of siRNAs with aptamer-siRNA chimeras. Nat Biotechnol. 24, 1005-1015 (2006).
- Dassie, J. P. Systemic administration of optimized aptamer-siRNA chimeras promotes regression of PSMA-expressing tumors. Nat Biotechnol. 27, 839-849 (2009).
- Zhou, J., Rossi, J. J. The therapeutic potential of cell-internalizing aptamers. Curr Top Med Chem. 9, 1144-1157 (2009).
- Zhou, J., Li, H., Li, S., Zaia, J., Rossi, J. J. Novel dual inhibitory function aptamer-siRNA delivery system for HIV-1 therapy. Mol Ther. 16, 1481-1489 (2008).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены